Method Article
פרופיל תאי בתפוקה גבוהה של תרכובות ממוקדות לפירוק חלבונים באמצעות קווי תאי HiBiT CRISPR
In This Article
Summary
פרוטוקול זה מתאר את הגילוי הכמותי של קינטיקה של פירוק חלבונים בתאים חיים שהונדסו באמצעות CRISPR/Cas9 כדי לבטא תג זיהוי חלבון אנדוגני נטול נוגדנים שהתמזג עם חלבון מטרה. הוראות מפורטות לחישוב וקבלת פרמטרי השפלה כמותיים, קצב, Dmax, DC 50 ו- Dmax50 כלולים.
Abstract
תרכובות ממוקדות לפירוק חלבונים, כולל דבקים מולקולריים או פרוטאוליזה המכוונת נגד כימרות, הן שיטה טיפולית חדשה ומלהיבה בגילוי תרופות של מולקולות קטנות. סוג זה של תרכובות גורם לפירוק חלבונים על ידי קירוב לחלבון המטרה ולחלבוני המנגנון E3 ליגאז הדרושים ליוביקוויטינאט ובסופו של דבר לפירוק חלבון המטרה דרך מסלול יוביקוויטין-פרוטאזומלי (UPP). עם זאת, יצירת פרופיל של פירוק חלבון מטרה באופן בעל תפוקה גבוהה נותרה מאתגרת ביותר בהתחשב במורכבות המסלולים התאיים הנדרשים להשגת פירוק. כאן אנו מציגים פרוטוקול ואסטרטגיית סינון המבוססים על שימוש בתיוג אנדוגני CRISPR/Cas9 של חלבוני מטרה עם תג 11 חומצות אמינו HiBiT אשר משלים עם זיקה גבוהה לחלבון LgBiT, כדי לייצר חלבון זוהר. ניתן להשתמש בקווי תאים ממוקדי קריספר אלה עם תגים אנדוגניים כדי למדוד השפלה המושרה על ידי תרכובות במצבים חיים קינטיים בזמן אמת או במצבי ליטית של נקודת קצה על ידי ניטור אות זוהר באמצעות קורא מבוסס צלחת זוהרת. כאן אנו מתארים את פרוטוקולי הסינון המומלצים עבור הפורמטים השונים, וגם מתארים את חישוב פרמטרי ההשפלה העיקריים של קצב, Dmax, DC 50, Dmax50, כמו גם ריבוב עם מבחני כדאיות התא. גישות אלה מאפשרות גילוי וטריאז'ינג מהירים של תרכובות בשלבים מוקדמים תוך שמירה על ביטוי אנדוגני וויסות של חלבוני מטרה ברקע תאי רלוונטי, ומאפשרות אופטימיזציה יעילה של תרכובות טיפוליות עופרת.
Introduction
פירוק חלבונים ממוקד התגלה כאחד האזורים הצומחים ביותר בגילוי תרופות עם מולקולות קטנות, מחוזק מאוד על ידי ההצלחה הטיפולית של תרכובות דבק מולקולריות אימונומודולטוריות (למשל, IMiD) לטיפול בסרטן, ונתוני ניסוי קליניים מוקדמים מבטיחים של פרוטאוליזה המכוונת לתרכובות כימרה 1,2,3,4,5,6,7,8, 9,10,11,12. תרכובות פירוק חלבונים ממוקדות פועלות על ידי קירוב חלבון מטרה עם חלבוני מכונות ליגאז E3 1,2,3,4,5,6,7,8,9,10,11,12 . גיוס זה המושרה על ידי תרכובת של חלבון המטרה לליגאז E3 מוביל ליוביקוויטינציה ופירוק של חלבון המטרה באמצעות מסלול יוביקוויטין פרוטאזומלי (UPP)1,2,3,4,5,6,7,8,9,10,11,12 . מבחינה היסטורית, תוכניות לגילוי תרופות עם מולקולות קטנות הסתמכו על בדיקות ביוכימיות ראשוניות כדי להעריך את הפעילות ולדרג תרכובות סדר. עם זאת, זה היווה אתגר משמעותי עבור משפילים חלבונים ממוקדים שפעילותם הסופית, פירוק באמצעות הפרוטאזום, תלויה במפל של אירועים תאיים 1,2,4,5,6,11,12,13,14,15,16,17, 18. המסלולים המרובים והמורכבות של קומפלקסי חלבונים הנדרשים לפירוק המטרה המוצלחת מחייבים גישות בדיקה תאיות לסינון מוקדם וטרייג'ינג של תרכובות ראשוניות. כיום, הזמינות של טכנולוגיות לניטור פירוק חלבון המטרה באופן בעל תפוקה גבוהה בהקשר של הסביבה התאית חסרה מאוד14. כאן נציג פרוטוקולים להערכת פעילות של התא החי הקינטי בזמן אמת או של השפלה ליטית בנקודת הקצה באמצעות קווי תאי יעד HiBiT המתויגים באופן אנדוגניכ-HiBiT 18,19,20 כדי לנטר את אובדן חלבון המטרה באמצעות מדידה זוהרת לאחר טיפול בתרכובות משפילות10,11,18,19.
כדי להשיג פירוק מוצלח של מטרות טיפוליות ולהרחיב את הפרוטאום הניתן לסימום, התפתחו גישות וסוגים רבים של משפילים שיכולים לכוון למגוון רחב של חלבונים להשמדה, כולל אלה הממוקמים בקרום הפלזמה או בתוכו, ליזוזומים, קרום המיטוכונדריה, ציטופלסמה והגרעין21-57. שני סוגי התרכובות העיקריים שנחקרו בהרחבה הם דבקים מולקולריים וחלבונים המכוונים לסימרה 2,4,5,6,7,12,26. דבקים מולקולריים הם חד-ערכיים, ולכן בדרך כלל קטנים יותר בגודלם, ומאפשרים ממשק אינטראקציה של חלבון-חלבון חדשני עם חלבון מטרה בעת קשירתו לרכיב ליגאז E3 2,12,26. הם בדרך כלל משפילים שנקשרים לרכיב הליגאז E3 של Cereblon (CRBN) 2,12,26,55,56,57. לאחרונה, אם כי דוגמאות חדשות ומלהיבות המשתמשות במכונות ליגאז E3 אחרות כגון DCAF1558,59,60 וגיוס CDK/Cyclin ל- DDB145 מראות את הפוטנציאל להרחבה של סוג זה של תרכובות. לעומת זאת, PROTACs הם מולקולות דו-ערכיות גדולות יותר, המורכבות מליגנד קושר מטרה, לרוב מעכב, המגשר באמצעות מקשר כימי לידית ליגאז E3 1,3,4,5,7,13. ככאלה, תרכובות אלה מסוגלות להיקשר ישירות הן לליגאז E3 והן לחלבון המטרה 1,3,4,5,7,13. חלבונים רבים הוכחו כמתפרקים באמצעות מולקולות דו-ערכיות אלה, וידיות הליגאז E3 הנפוצות ביותר מגייסות CRBN או Von Hippel Lindau (VHL)1,3,4,5,7,13. עם זאת, מספר הידיות הזמינות לגיוס ליגאז E3 בכימרה המכוונת לתכנון פרוטאוליזה גדל במהירות, ומרחיב את היכולות של סוג זה של תרכובות עם פוטנציאל לפגוע במחלקות מטרה מגוונות, כמו גם לשפר את הספציפיות של סוג התא או הרקמה 24,48,61,62 . בשילוב עם הדרישה המינימלית לעסוק בחלבון מטרה, אפילו עם זיקה שולית, תרכובות הפירוק טומנות בחובן הבטחה להרחבת הפרוטאום הניתן לתרופה.
אפיון הדינמיקה התאית של אובדן חלבונים, כמו גם התאוששות פוטנציאלית של חלבונים לאחר הטיפול, הוא קריטי להבנת תפקוד ויעילות תרכובת הפירוק. בעוד שניתן לחקור שינויים ברמת החלבון האנדוגני במערכות תאיות רלוונטיות באמצעות מבחני נוגדנים של כתמים מערביים או ספקטרומטריית מסה, גישות אלה קשות להתאמה לפורמטים של סינון תפוקה גבוהה, יש להן יכולת כימות מוגבלת, או יכולת למדוד שינויים קינטיים בנקודות זמן רבות14. כדי להתמודד עם אתגרים אלה, פיתחנו מערכת זוהרת תאית מבוססת צלחת לניטור שינויים ברמות החלבון האנדוגניות, המשתמשת בהחדרה גנומית באמצעות CRISPR/Cas9 של תג 11 חומצות האמינו, HiBiT, למוקדים של כל יעד השפלה מרכזי18,19,20. פפטיד זה משלים זיקה גבוהה לשותף המחייב שלו, LgBiT, כדי לייצר זוהר בהיר בנוכחות המצע שלו 18,19,20,63, ובכך להפוך את החלבונים האנדוגניים המתויגים האלה לזוהרים בתאים או ליזאטים18,19,20,63 . יחידות האור היחסיות (RLUs) הנמדדות באמצעות מכשיר לומינומטר נמצאות ביחס ישר לרמות חלבון היעד המתויגות 18,19,20,63. עם התפתחותם של מצעי לוציפראז מיוצבים, מדידות ברמת החלבון הקינטי בזמן אמת על פני מסגרות זמן של 24-48 שעות אפשריות 18,53,64. זה מאפשר לקבוע פרופיל השפלה מלא לכל מטרה נתונה בכל ריכוז תרכובת נתון, כולל ניתוח כמותי של קצב הפירוק הראשוני, מקסימום השפלה (Dmax), והתאוששות לאחר טיפול תרכובת18,53. עם זאת, אם מסננים ספריות גדולות של תרכובות השפלה, ניתן לבצע בקלות גם ניתוח נקודות קצה בפורמט 384-well בריכוזי תרופות שונים ובזמנים ייעודיים.
הפרוטוקולים המוצגים בכתב יד זה מייצגים אסטרטגיות סינון תאיות לתרכובות ממוקדות לפירוק חלבונים, החלות על כל סוגי המשפילים. עם זאת, השימוש בקווי תאי HiBiT CRISPR יחד עם פרוטוקולים אלה אינם מוגבלים לפירוק חלבונים, אלא הם כלים כלליים לניטור כל רמת חלבון מטרה אנדוגנית שניתן לווסת לאחר הטיפול כדי לחקור השפעה של תרכובות או אפילו מנגנוני עמידות 20,65,66. תנאי מקדים לשיטות זיהוי מבוססות זוהר אלה הוא קו תאי מטרה HiBiT המתויג באופן אנדוגני כ-CRISPR, שהוא קריטי מכיוון שהוא מאפשר זיהוי זוהר רגיש, תוך שמירה על ביטוי מטרה אנדוגני ותקנות מקדם מקומיות18,19,20. התקדמות משמעותית נעשתה בשימוש ב- CRISRP/Cas9 להחדרת תגים גנומיים, במיוחד במדרגיות 20 וברגישות הגבוהה של זיהוי, בפורמטים שונים כולל מאגרי קריספר או שיבוטים עם החדרות אליליות הטרוזיגוטיות או הומוזיגוטיות18,19,20. שימוש בביטוי אקסוגני של HiBiT או היתוך מדווח אחר בתאים במקום תיוג אנדוגני אפשרי, אך יש לנקוט משנה זהירות באמצעות מערכות עם ביטוי יתר של חלבון14,18. אלה יכולים להוביל לחפצים בהבנת העוצמה האמיתית של תרכובות ודינמיקה של התאוששות חלבונים14,18, כולל לולאות משוב שעתוק פוטנציאליות המופעלות לאחר השפלת המטרה. בנוסף, תרכובות בשלב מוקדם עם עוצמה נמוכה עלולות להתפספס, ולהציג את עצמן כשליליות כוזבות בסינון. מכיוון שאובדן חלבונים עלול לנבוע מרעילות הנגרמת על-ידי תרכובות וממוות של תאים, הפרוטוקולים המתוארים כאן מכילים בדיקות זוהרות או פלואורסצנטיות של תאים מומלצות מאוד, אך אופציונליות, בשילוב עם פרוטוקול הפירוק. ישנם שני חלקים עיקריים לפרוטוקול, נקודת קצה ליטית, והקרנה קינטית של תאים חיים. בתוך כל אחד ממקטעים אלה, נכללות אפשרויות למדידות של כדאיות תאים מרובים בתבניות של נקודות קצה או קינטיות. ניטור השינויים של החלבון האנדוגני המתויג דורש השלמה עם LgBiT בתאים. לכן, סעיף הסינון הקינטי מפנה לפרוטוקולים חשובים להכנסת זה, אשר ניתן להשיג באמצעות ביטוי חולף או יציב והוא חיוני לביצוע מדידות זוהר התא החי. כל הגישות המוצגות כאן מאפשרות סידור דירוג מהיר והערכת פעילות של תרכובות, מה שמאפשר מאמצי סינון תרכובת בשלבים מוקדמים וזיהוי מהיר יותר של משפילי עופרת.
פרוטוקול זה מיועד לחקר תרכובות פירוק בשילוב עם קו תאי HiBiT CRISPR. פרוטוקולים ליצירת תוספות HiBiT CRISPR ליעדים רבים תוארו במספר פרסומים אחרונים18,19,20.
Protocol
1. מחקרי פירוק נקודות קצה עם חלבוני מטרה HiBiT CRISPR בפורמט ליטי עם ניתוח פלואורסצנטי אופציונלי של כדאיות התאים
- הכנה וציפוי של דבק יונקים או קו תא תרחיף
- התאם את צפיפות התאים ל-2.22 x 105/mL על-ידי דילול במדיית תאים מתאימה המשמשת למעבר ולגדילת תאים.
- יש לחלק תאים לצלחות עם מינימום של 3 בארות לכל תנאי ניסוי ובקרה. יש לחלק 90 μL (20,000 תאים) לכל באר של תרחיף תאים ללוחות לבנים של 96 בארות. עבור פורמט של 384 בארות, יש לחלק 36 μL (8,000 תאים) לכל באר של תרחיף תאים ללוחות לבנים של 384 בארות.
- הכנה והוספה של תרכובות
- הכן לוחות תרכובת PROTAC או משפילים בדילול סדרתי בריכוז סופי של פי 1,000 ב-100% DMSO. לאחר מכן דיללו אותו לריכוז סופי של פי 10 במדיום תרבית התאים. הוסף נפח שווה של DMSO למדיום, כדי שישמש כפקד DMSO ללא תרכובת.
- עבור פורמט 96-well להוסיף 10 μL של 10x תרכובת פתרונות בקרה ל 90 μL של תאים. עבור פורמט 384-well להוסיף 4 μL של 10x תרכובת ופתרונות בקרה ל 36 μL של תאים.
- דגירה של הצלחות באינקובטור בטמפרטורה של 37 מעלות צלזיוס ו-5% CO2 למשך פרק הזמן הרצוי או בתנאים אופטימליים לצמיחתם.
הערה: מכיוון שמדובר בבדיקת נקודת קצה, בדיקה של נקודות זמן מרובות תדרוש הכנה של לוחות השפלה נפרדים עבור כל נקודת זמן, כמתואר בשלב 1.1.2 לעיל. זמני הדגירה לאיתור השפלה בתיווך תרכובת הם משתנים מאוד וככל הנראה תלויים גם בריכוז התרכובת. נקודות הזמן הראשוניות המוצעות יהיו 6 שעות ו-24 שעות. - אם מודדים זיהוי זוהר של נקודות קצה ללא מדידת הכדאיות האופציונלית של התא, המשך ישירות לשלב 1.3 להלן. אם אתה מבצע ריבוב עם מדידת כדאיות התא, המשך לסעיף 1.4 הבא להלן.
- מדידה ליטית של תאים
- מיד לפני מדידות ליטי HiBiT, הכינו 2x מגיב זיהוי ליטי על ידי הוספת 20 μL של מצע ליטי ו-10 μL של חלבון LgBiT לכל 1 מ"ל של המאגר הליטי. הכן מספיק מגיב גילוי פי 2 למספר הבארות שיש לבחון, כולל נפח נוסף כדי להסביר שגיאת צנרת (כלומר, מספר בארות + 10%).
- הוסף מגיב זיהוי ליטי מוכן לתאים. עבור פורמט של 96 בארות, הוסף 100 μL של 2x מגיב זיהוי ליטי לכל באר המכילה 100 μL של תאים. עבור פורמט 384-well, הוסף 40 μL של 2x מגיב זיהוי ליטי לכל באר המכילה 40 μL של תאים. מערבבים את הצלחת על מערבל מערבולת microplate במשך 10-20 דקות ב 350 סל"ד.
- מדוד לומינסנציה על לומינומטר המסוגל לקרוא זוהר בלוח של 96 או 384 בארות.
- ריבוב אופציונלי של כדאיות התאים
הערה: שלב זה מבוצע באמצעות ערכת CellTiter-Fluor (CTF) הזמינה מסחרית (ראה טבלת חומרים).- 30-40 דקות לפני מדידת נקודת הקצה הרצויה, הכינו תמיסת ריאגנט לזיהוי כדאיות תאים פי 6 על ידי הוספת 10 μL של המצע ל-2 מ"ל של Buffer הבדיקה. הכינו מספיק מגיב פי 6 לכל באר לבדיקה, כולל נפח נוסף לשגיאת צנרת (כלומר, מספר בארות + 10%).
- מוסיפים את המגיב המוכן לבארות. עבור פורמט של 96 באר הוסף 20 μL של 6x מגיב לכל באר שכבר מכילה נפח של 100 μL. עבור פורמט 384-well להוסיף 8 μL של 6x מגיב לכל באר המכילה 40 μL של תאים. מערבבים לזמן קצר על מערבל מערבולת microplate, ולאחר מכן דגירה של הצלחת במשך 30 דקות באינקובטור של 37 מעלות צלזיוס.
- בנקודת הקצה הרצויה של המדידה (כלומר, 6 או 24 שעות לאחר הטיפול, שלב 1.2.3), מדוד פלואורסצנטיות במכשיר המסוגל לקרוא פלואורסצנציה (380-400nmEx/505nmEm) בפורמט 96 או 384- היטב.
- הכן 2x מגיב לזיהוי ליטי על ידי הוספת 20 μL של מצע ליטי ו-10 μL של חלבון LgBiT לכל 1 מ"ל של חיץ ליטי. הכינו מספיק מגיב גילוי פי 2 למספר הבארות שייבחנו, כולל נפח נוסף כדי להסביר שגיאת צנרת (למשל, מספר בארות + 10%).
- הוסף את מגיב זיהוי ליטי מוכן לבארות. עבור פורמט 96 באר להוסיף 120 μL של 2x מגיב זיהוי ליטי לכל באר כבר מכיל נפח 120 μL. עבור פורמט 384-well להוסיף 48 μL של 2x מגיב זיהוי ליטי לכל באר כבר מכיל נפח 48 μL. מערבבים את הצלחת על מערבל מערבולת microplate במשך 10-20 דקות.
- מדוד לומינסנציה על לומינומטר המסוגל לקרוא לומינסנציה בלוחות באר 96 או 384.
- כימות ההשפלה וכדאיות התא
- ממוצע יחידות האור היחסיות (RLU) מהפקד DMSO בנקודת הזמן שנמדדה. השתמש בערך זה כרמת החלבון הבסיסית של המטרה כדי לחשב השפלה חלקית על-ידי נרמול כל הטיפולים האחרים שנבדקו באותו זמן והצביע על ערך זה. לדוגמה, אם ה-RLU הממוצע עבור בארות הבקרה של DMSO ב-6 שעות היה 10,000, וה-RLU עבור טיפול תרכובת נתון ב-6 שעות היה 5,000, ההתפרקות החלקית תחושב כ-5,000 ÷ 10,000 = 0.5 (משוואה 1).
משוואה 1: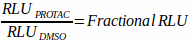
- קבע את אחוזי ההפחתה מ- RLU חלקי:
משוואה 2: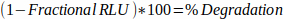
- התווה RLU חלקי או % השפלה בנקודות זמן ספציפיות כדי לדרג את הפעילות של תרכובות.
- לחלופין, נתח נתוני יחידת פלואורסצנטיות יחסית (RFU) למדידת בדיקת כדאיות התא על ידי השוואת הערכים מכל הטיפולים לבקרת DMSO. אם נצפתה ירידה משמעותית ב- RFU עבור כל טיפול ביחס לבקרת ה- DMSO, ניתן לנרמל את נתוני ההשפלה בנוסף לנתוני בדיקת הכדאיות של התא כדי לקבוע שינויים ברמת החלבון ביחס להפסדים בכדאיות התא.
- ממוצע יחידות האור היחסיות (RLU) מהפקד DMSO בנקודת הזמן שנמדדה. השתמש בערך זה כרמת החלבון הבסיסית של המטרה כדי לחשב השפלה חלקית על-ידי נרמול כל הטיפולים האחרים שנבדקו באותו זמן והצביע על ערך זה. לדוגמה, אם ה-RLU הממוצע עבור בארות הבקרה של DMSO ב-6 שעות היה 10,000, וה-RLU עבור טיפול תרכובת נתון ב-6 שעות היה 5,000, ההתפרקות החלקית תחושב כ-5,000 ÷ 10,000 = 0.5 (משוואה 1).
2. פירוק קינטי בזמן אמת של חלבוני מטרה HiBiT CRISPR ובדיקת לומינסנציה אופציונלית של כדאיות התאים
הערה: היכולת לבצע הקרנה קינטית ופירוק דורשת ביטוי משותף של חלבון LgBiT בתא, שתואר קודם לכן18,19,63. ניתן להשיג זאת באמצעות העברה חולפת של וקטור LgBiT, שימוש ב- BacMam LgBiT, או על ידי ביצוע החדרת HiBiT CRISPR לקו תאים יציב LgBiT.
- ציפוי של קווי תאים דבקים.
- הסר מדיום מבקבוק התא על ידי שאיפה, שטף תאים עם DPBS, נתק תאים עם 0.05% טריפסין-EDTA, ואפשר לתאים להתנתק מתחתית הצלוחית. עבור קווי תאי השעיה, המשך לסעיף 2.2.
- נטרול טריפסין באמצעות מדיום תרבית תאים המכיל סרום, לערבב כדי לאסוף ולתרן תאים, ולהעביר את תרחיף התא לצינור חרוטי.
- סובבו את התאים ב-125 x g למשך 5 דקות. יש להשליך את מדיום תרבית התאים ולחזור בנפח שווה של מדיום תרבית תאים טריים.
- תאי צלחת ללוחות בדיקה עם מינימום של בארות משולשות לכל תנאי ניסוי ובקרה. עבור ספירת תבנית של 96 בארות כדי להעריך את צפיפות התא, התאם את הצפיפות ל-2 x 105 תאים למ"ל במדיום הבדיקה וחלק 100 μL (20,000 תאים) לכל באר בלוח של 96 בארות. עבור ספירת תבניות של 384 בארות כדי להעריך את צפיפות התאים, התאם את הצפיפות ל- 4.44 x 105 תאים למ"ל במדיום הבדיקה וחלק 18 μL (8,000 תאים) לכל באר.
- לוחות דגירה ב 37 מעלות צלזיוס, 5% CO2 לילה או בתנאים אופטימליים לצמיחתם.
- ציפוי תאי השעיה
- התאם את צפיפות התאים ל-2.22 x 105 תאים /מ"ל במדיום בלתי תלוי CO2 בתוספת 10% FBS ו-1x Endurazine (דילול 1:100 של מגיב המניות).
- תאי צלחת ללוחות בדיקה עם מינימום של 3 בארות לכל תנאי ניסוי ובקרה. עבור פורמט של 96 באר יש לחלק 90 μL (20,000 תאים) לכל באר. עבור פורמט 384-well יש לחלק 36 μL (8,000 תאים) לכל באר.
הערה: עבור קווי תאים תלויים בעלי אות נמוך לרקע (S:B), לדוגמה, בעת עבודה עם מאגרי קריספר ולא עם שיבוטים, ניתן להגדיל את הלומינסנציה על ידי הגדלת מספר התאים המצופים, עד 100,000 תאים/באר בפורמט 96 בארות, או 40,000 תאים/באר בפורמט 384-well.
- מבחני פירוק קינטיים באמצעות תאי HiBiT CRISPR המבטאים LgBiT
- עבור תאי תרחיף שכבר מכילים אנדוראזין, שנכלל בשלב הציפוי ב-2.2., המשך ישירות לשלב 2.3.3. עבור קווי תאים דבקים להכין פתרון ננו-גלו אנדוראזין. עבור פורמט 96-well, הכינו פתרון 1x של Endurazine על ידי דילול מגיב מניות 1:100 למדיום עצמאי CO2 בתוספת 10% FBS. עבור פורמט 384-well, הכינו תמיסה 2x של Endurazine על ידי דילול מגיב מניות 1:50 למדיום בלתי תלוי CO2 בתוספת 10% FBS.
- הוסף תמיסת Endurazine לכל באר של תאים דבקים. עבור 96-טוב פורמט לשאוף מדיום ולהוסיף 90 μL של 1x Endurazine פתרון. עבור פורמט 384-well, הוסף 18 μL של 2x תמיסת Endurazine ל 18 μL של תאים. אל תשאף מדיום שכן בדיקת ההשפלה מבוצעת בתערובת של 50:50 של מדיום תרבות ומדיום עצמאי CO2 בפורמט 384-well.
- תרחיף דגירה או לוחות תאים דבקים המכילים Endurazine במשך 2.5 שעות באינקובטור ב 37 °C ו 5% CO2 כדי לאפשר luminescence שיווי משקל.
- הכן ריכוז של פי 10 של טיטרציית PROTAC לבדיקה במדיום CO2-עצמאי והוסף 10 μL לכל באר של צלחת 96-well או 4 μL עבור צלחת 384-well. עבור תרכובות עם יעילות לא ידועה, ריכוז סופי של 1-10 μM בנקודה הגבוהה ביותר מומלץ כנקודת התחלה.
- אסוף מדידות קינטיות של זוהר בלומינומטר שיווי משקל מראש ל-37 מעלות צלזיוס לתקופה שבין 0-48 שעות. ניתן להתאים אישית את מרווחי הזמן של המדידה עבור כל ניסוי, אך ניסוי ראשוני מומלץ יהיה מדידות לומינסנציה כל 5-15 דקות למשך 24 שעות או פרק הזמן הרצוי.
- כדאיות תאים אופציונלית באותו ניתוח מולטיפלקס לאחר מדידה קינטית סופית
הערה: בדיקה זו מבוצעת עם ערכת CellTiter-Glo (CTG) הזמינה מסחרית (ראה טבלת חומרים).- ריאגנט CTG שיווי משקל לטמפרטורת החדר.
- לאחר מדידת ההשפלה בנקודת הזמן האחרונה של הניתוח הקיני, הוסיפו 100 μL (צלחת 96-well) או 40 μL (צלחת 384-well) של הריאגנט לכל באר של הצלחת, וערבבו על שייקר צלחת ב-500-700 סל"ד (צלחת 96-well) או מערבל מערבולת microplate (צלחת 384-well) למשך 5 דקות.
- דגירה של הצלחת בטמפרטורת החדר למשך 30 דקות כדי לאפשר תסיסה של תאים ומרווה של אות HiBiT.
- מדוד את עוצמת האור הכוללת על לומינומטר על-ידי ביצוע המלצת היצרן.
- כימות פרופילי השפלה קינטית
- באמצעות מדידות הזוהר הקינטיות שנאספו, נרמלו את ה-RLUs הגולמיים עבור כל ריכוז PROTAC למצב DMSO ממוצע משוכפל בכל נקודת זמן כדי לקחת בחשבון את השינויים בריכוז הפורימאזין החופשי לאורך זמן. חישוב RLU חלקי באמצעות משוואה 1.
משוואה 1: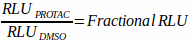
- מתוך עקומות ההשפלה, התאם מודל דעיכה מעריכית של רכיב יחיד באמצעות משוואה 2 לחלק ההשפלה הראשוני של כל עקומה עד לנקודה שבה הנתונים מגיעים לרמה.
הערה: ייתכן שיהיה מועיל לא לכלול מההתאמה את נקודות הנתונים הראשונות מכיוון שייתכן שיהיה פיגור קצר לפני שנצפתה השפלה.
משוואה 2: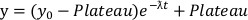
- מתוך משוואה 2, קבעו את הפרמטר ƛ, המייצג את קבוע קצב ההתפרקות, ואת הרמה, המייצגת את כמות החלבון הנמוכה ביותר שנותרה.
- חישוב Dmax, שהוא הסכום החלקי המרבי של חלבון מושפל ומחושב כ-1-Plateau.
- התווה Dmax עבור כל ריכוז של PROTAC כדי לקבוע עקומת עוצמת השפלה שאינה תלויה בזמן.
- קבע את ערך Dmax50 עבור העלילה ב 2.3.5 כדי לנתח את היעילות של תרכובות.
הערה: כדי לקבועDC 50 בנקודת זמן מסוימת, התווה את אחוזי הירידה המחושבים עבור כל ריכוז בזמן שנבחר. ניתן לציין זאת כ- DC 50 t = 4 שעות או DC50 t = 12 שעות.
- באמצעות מדידות הזוהר הקינטיות שנאספו, נרמלו את ה-RLUs הגולמיים עבור כל ריכוז PROTAC למצב DMSO ממוצע משוכפל בכל נקודת זמן כדי לקחת בחשבון את השינויים בריכוז הפורימאזין החופשי לאורך זמן. חישוב RLU חלקי באמצעות משוואה 1.
תוצאות
כדי להדגים ניתוח השפלה ליטית של נקודת קצה בריכוז יחיד, מספר חלבוני מטרה של CDK; CDK2, CDK4, CDK7 ו-CDK10 תויגו באופן אנדוגני עם HiBiT ב-C-terminus שלהם בתאי HEK293 וטופלו בריכוז של 1 מיקרומטר של PROTAC מבוסס פאן-קינאז קרבלון, TL12-18654 (איור 1A). רמת חלבון ה-CDK נמדדה בנקודות זמן שונות, ונקבע ה-RLU השברירי ביחס לבקרת ה-DMSO (איור 1A). כל חלבון CDK הראה דרגות שונות של התדרדרות בתגובה לטיפול התרכובת ולנקודות הזמן השונות (איור 1A). כדי להבין כיצד חלבוני CDK הושוו זה לזה באופן ישיר במונחים של אובדן חלבונים, ה-RLUs השבריריים באיור 1A חושבו כאחוזי פירוק כוללים והתוו עבור כל נקודת זמן באיור 1B. זה מראה שאפילו בנקודות זמן מוקדמות, 2 או 4 שעות, חלק מבני משפחת CDK מראים רמות גבוהות של השפלה שממשיכות במגמת עלייה לאורך זמן (איור 1B).
כדי להדגים ניתוח פירוק קינטי, כל אחד מהחלבונים בני משפחת BET; BRD2, BRD3 ו-BRD4 תויגו באופן אנדוגני עם HiBiT ב-N-terminus שלהם בתאי HEK293 המבטאים ביציבות את החלבון LgBiT18. אלה טופלו לאחר מכן בשלושה ריכוזים שונים של ה-PROTACs של פאן-BET; ה-dBET650 המבוסס על המוח (איור 2A) וה-ARV-77141 המבוסס על VHL (איור 2B). מדידות קינטיות נאספו במשך 24 שעות, ומהפרופילים בכל ריכוז, ההבדלים בתגובת בני המשפחה של BET ניכרים בקלות. היכולת של BRD2 ליזום תגובת התאוששות מהירה יותר לאחר טיפול תרכובת השפלה (איור 2A,B) נצפתה בעבר עם PROTACs פאן-BET אחרים, והיא ככל הנראה נובעת מתגובת משוב שעתוק תחרותית לתהליך הפירוק18.
ניתן לבצע הן ניתוח נקודת קצה והן ניתוח קינטי עם טיפולי תגובה במינון מורכב מלא. מוצגים באיור 3 פרופילי פירוק של תגובת מינון קינטית של טיפול בתאי Ikaros/IKZF1-HiBiT CRISPR Jurkat המבטאים ביציבות חלבון LgBiT עם ארבע תרכובות דבק מולקולריות שונות 2,26,55,57; לנלידומיד (איור 3A), איברדומיד (CC-220) (איור 3B), תלידומיד (איור 3C) ופומלידומיד (איור 3D). משפילים אלה מראים הבדלים משמעותיים בתגובת ההשפלה בין התרכובות, כמו גם בכל סדרות הריכוזים (איור 3).
כדי להעריך באופן כמותי את ההשפלה ואת סדר הדירוג של התרכובות באיור 3, פרופילי התגובה של המינון שימשו לחישוב פרמטרים מרכזיים של השפלה, כולל קצב ההשפלה (איור 4A), Dmax (איור 4B) וערכי Dmax50 (איור 4B). ניתוחים אלה מראים כי לאיברדומיד (CC-220) ולפומלידומיד יש שיעורי פירוק ראשוניים מהירים דומים מאוד (איור 4A), אך לאיברדומיד (CC-220) יש את העוצמה הגבוהה ביותר כפי שנראה בעבר במחקרים אורתוגונליים55,57 (איור 4B). מכיוון שאיברדומיד מפגין עוצמה כה גבוהה, וכל הריכוזים שנבדקו מראים ירידה של יותר מ-50%, הערך Dmax50 המתקבל עבור איברדומיד מייצג הערכה המבוססת על המגבלה בהתאמה מדויקת של הנתונים. מהגרפים באיורים 3C,D ו-4B, לא לנלידומיד ולא תלידומיד פוגעים ביעד Ikaros/IKZF1 עד להשלמתו בריכוזים הגבוהים ביותר שנבדקו. בשל מעט מאוד השפלה שנצפתה עם תלידומיד, עקבות ההשפלה לא יכלו להתאים במדויק למודל דעיכה מעריכי, ולכן קצב ההשפלה לא היה מכמת לטיפול זה. עבור המשפיל החזק ביותר, איברדומיד (CC-220)55,57 (איור 4B). מבחני ריבוי הכדאיות של התאים לא הראו ירידה בכדאיות התא עבור הריכוזים שנבדקו (איור 4C).
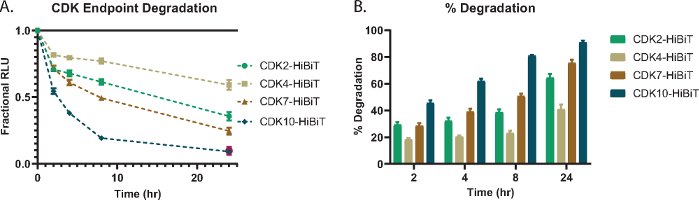
איור 1: השפלה ורעילות של נקודות קצה של CDK עם פאן-קינאז PROTAC, TL12-18654. (A) פאנל נבחר של חלבוני מטרה אנדוגניים מסוג CDK שהתמזגו עם HiBiT ב-C-terminus באמצעות CRISPR/Cas9 והוערכו לפירוק עם טיפול של 1 μM TL12-186 PROTAC54 ב-2 שעות, 4 שעות, 8 שעות ו-24 שעות. ערכים מיוצגים כ- RLU חלקי ביחס לפקד DMSO הנמדד בכל נקודת זמן. קווי שגיאה מייצגים SD של הממוצע של 3 שכפולים טכניים. (B) אחוזי הפירוק של פאנל חלבוני המטרה של CDK המחושבים מתוך (A) המייצגים את כמות ההשפלה של כל אחד מבני המשפחה שנצפו בנקודות זמן של 2, 4, 8 ו-24 שעות. קווי שגיאה מייצגים SD של הממוצע של 3 שכפולים טכניים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
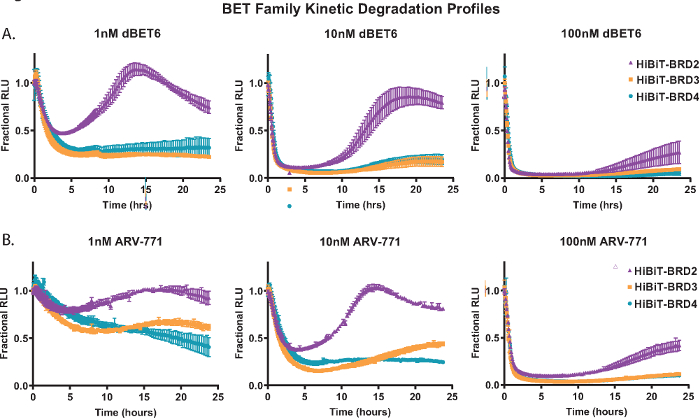
איור 2: יצירת פרופיל סלקטיביות של השפלה קינטית של בני משפחת BET עם משפילים של BET, dBET650 ו-ARV-77141. פרופילי השפלה קינטיים של בני משפחת BET אנדוגניים, BRD2, BRD3 ו- BRD4, המתויגים עם HiBiT במסוף N באמצעות CRISPR/Cas9 עם טיפול בריכוזים בודדים של 1 ננומטר (משמאל), 10 ננומטר (באמצע), או 100 ננומטר (מימין) dBET650 (A) או ARV-77141 (B) PROTACs. ערכים מיוצגים כ- RLU שברים המחושבים מתוך פקד DMSO בכל נקודת זמן קינטית. קווי שגיאה מייצגים SD של הממוצע של 4 שכפולים טכניים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
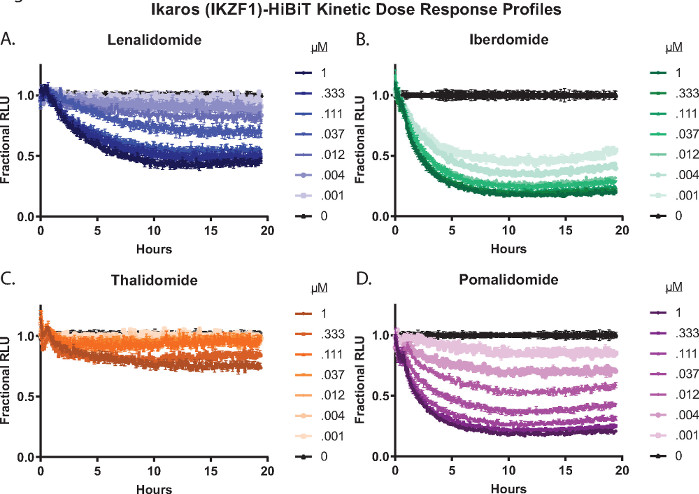
איור 3: פרופילי תגובה של מינון קינטי לפירוק תאים חיים של Ikaros/IKZF1-HiBiT עם לוח דבק מולקולרי 2,26,55,57. תאי Jurkat המבטאים ביציבות את החלבון LgBiT הונדסו באמצעות CRISPR/Cas9 כדי לתייג את ה-C-terminus של איקרוס/IKZF1 עם פפטיד HiBiT. התאים טופלו בסדרת ריכוזי תגובה של 8 נקודות כולל DMSO של ארבע תרכובות דבק מולקולריות שונות 2,26,55,57: (A) לנלידומיד, (B) איברדומיד (CC-220), (C) תלידומיד, או (D) פומלידומיד. לומינסנציה נמדדה כל 5 דקות במשך 19.5 שעות בסך הכל. נתוני יחידת אור יחסית (RLU) מ- (A-D) הומרו ל- RLU חלקי כמתואר בשלב 2.4.1 וגרפים כפונקציה של זמן. קווי שגיאה מייצגים SD של 3 שכפולים טכניים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
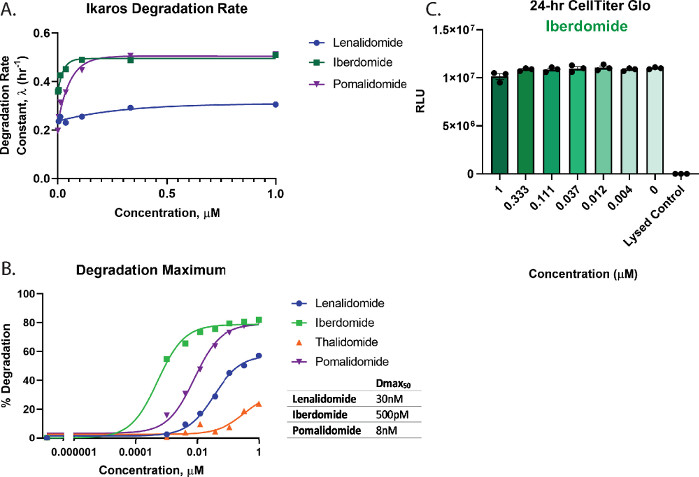
איור 4: חישוב קצב הפירוק ו-Dmax50 עבור Ikaros/IKZF1-HiBiT, ומבחני בריאות תאים מרובים. נתוני השפלה קינטית מאיור 3 שימשו לחישוב פרמטרים של השפלה כמותית. (A) שיעורי הפירוק ו-(B) ערכי ההשפלה המרביים (Dmax) מוצגים בכל ריכוז תרופה עבור תרכובות הדבק המולקולרי שצוינו 2,26,55,57. (B) ערכי Dmax50 עבור כל תרכובת חושבו באמצעות מודל מינון-תגובה עם שיפוע היל מוגבל של 1, אשר ניתן להשתמש בו כדי לדרג תרכובות השפלה סדר עבור מטרה. (C) מבחני הכדאיות של התאים עם האיברדומיד (CC-220)55,57 תגובת מינון הפירוק מאיור 3B בוצעו כמדידת נקודת קצה עם השלמת מדידות ההשפלה הקינטיות. קווי שגיאה מייצגים SD של 3 שכפולים טכניים. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
Discussion
אנו מציגים כאן שתי שיטות לסינון פעילות תרכובת השפלה בפורמט ליטי של נקודת קצה או במצב קינטי של תאים חיים. גישות אלה מבוססות על אותם עקרונות מדידה זוהרים אך מספקות רמות שונות של פירוט והבנה. הבחירה בשתי הגישות תהיה תלויה ככל הנראה ביעדי הסינון ובגודל הספרייה המורכבת. עבור חפיסות הקרנה מורכבות גדולות או מסכים ראשיים כדי לצפות בכל פגיעה הניתנת לזיהוי, סינון ליטי של נקודות קצה מציע תאימות רגישה ויעילה לתפוקה גבוהה שבה גישות אחרות של נקודות קצה, כגון כתם מערבי או ספקטרומטריית מסה, יכולות להיות לא מעשיות או קשות להתאמה14. נקודת מוצא למסכים אלה יכולה להתבצע עם מספר מוגבל של ריכוזים ונקודות זמן. הריכוזים ההתחלתיים המומלצים לבדיקה הם בטווח של 100 nM-10 μM, כדי להסביר משפילים ראשוניים בעלי עוצמה נמוכה, חדירות ירודה, או במקרים מסוימים עם תרכובות חזקות מאוד, נוכחות של אפקט וו. עוד מומלץ לבדוק לפחות שתי נקודות זמן שונות כדי לקבוע השפלה מוקדמת ב-4-6 שעות והשפלה סמויה או מתמשכת ב-18-24 שעות. תרכובות המפגינות עוצמת פירוק גבוהה ומנגנון מטרה נצפות בקלות בטווח זמן של 4-6 שעות, בעוד שפירוק או אובדן חלבון לכאורה שנצפה רק בנקודות זמן מאוחרות יותר יכול לנבוע ממגוון מנגנונים. מומלץ מאוד לעקוב אחר כדאיות התאים הן בנקודות זמן מוקדמות והן בנקודות זמן מאוחרות, כך שניתן יהיה לנתק את אובדן החלבון מאובדן עקב מוות של תאים. בדומה לכל סוג של בדיקה זוהרת או פלואורסצנטית, קיים פוטנציאל לתרכובות בתוך ספריות להפריע או לעכב אותות, ולכן, ניסויי מעקב אורתוגונליים עם תרכובות עופרת באמצעות היתוך לא קשור או גישות חלופיות לניטור רמת החלבון יהיו חשובים להערכת אובדן RLU במבחנים אלה קשור ישירות לפירוק חלבון המטרה.
היכולת לסנן בתבנית קינטית של תאים חיים לאורך פרקי זמן ממושכים מסתמכת במידה רבה על אות הבדיקה לרקע (S:B). גורמים התורמים ל-S:B כוללים את רמת הביטוי של חלבון המטרה עצמו, שיכולה להתפרש על פני מספר סדרי גודל, היעילות של ביטוי LgBiT בקו התא שנבחר להחדרת פפטידים, והזמינות של היעד המתויג להשלמה במתחמי הילידים השונים שלו. קבענו דרישת ניתוק כללית המורכבת מ- S:B של 15 כדי למדוד בהצלחה השפלה במצב קינטי עם Endurazine או Vivazine. ה-S:B נקבע על ידי מדידת האות הבסיסי של התאים הערוכים ב-HiBiT המבטאים במשותף LgBiT ביחס לתאי הורים לא ערוכים המבטאים LgBiT בלבד בנוכחות מצעי תאים חיים מסוג Endurazine או Vivazine. Vivazine יפיק אות זוהר גבוה יותר אך יתפורר מהר יותר מאשר Endurazine ועשוי להגביל את קליטת האות ל-24 שעות או פחות. יתר על כן, S:B יכול גם להיות תלוי מאוד אם בריכות קריספר או שיבוטים משמשים. עבור מטרות בקווי תאים שהם נוחים יותר ובעלי יעילות גבוהה להנדסת CRISPR/Cas9, אוכלוסיית מאגר קריספר הטרוגנית של תאים ערוכים עשויה להיות בעלת S:B מספקת לניתוח קינטי. עבור מטרות בקווי תאים קשים יותר שבהן אינטגרציה גנומית פחות יעילה באמצעות קריספר מביאה למאגרים עם S:B נמוך, ייתכן שיהיה צורך לבודד שיבוטים של CRISPR כדי להעשיר אוכלוסיות ערוכות ולהשיג S:B גבוה מספיק לניתוח קינטי. עבור כל אחד מהתרחישים הללו, אם S:B קטן מ-15 עם מצעי אנדוראזין או ויווזין, מומלץ לבצע סינון ליטי של נקודת קצה.
להבנה ואפיון טובים יותר של תרכובות, כולל קביעת פרופיל פירוק עם פרמטרים כמותיים, אנליזה קינטית בזמן אמת בתאים חיים היא גישת הסינון המומלצת14,18. בדומה לניתוח נקודות קצה שנדון לעיל, סינון קינטי ראשוני יכול להיעשות עם מספר מוגבל של ריכוזים בטווח של 100nM-10μM באופן בעל תפוקה גבוהה. בפורמט של 384 בארות, ניתן לסנן בקלות יותר מ-100 תרכובות במשולש בריכוז אחד על צלחת אחת. פרופילי ההתפרקות המתקבלים יספקו הדרכה לא רק לגבי מידת ההשפלה שנצפתה, אלא גם לגבי קצב ההתפרקות, משך ההתפרקות וההתאוששות הפוטנציאלית של החלבון14,18 (איור 2 ואיור 3). הצורות של פרופיל ההשפלה מניבות גם הן מידע בעל ערך. משפילים ספציפיים וחזקים מראים לעתים קרובות אובדן מהיר ראשוני של חלבון המטרה לרמה תוך שעות ספורות18,53, בעוד שמנגנונים אחרים כגון משוב שעתוק או רעילות תרכובת גורמים בדרך כלל לאובדן ליניארי יותר של החלבון לאורך זמן. פרטים וניואנסים אלה מתפספסים עם ניתוח ליטי של נקודות קצה, ועם ניתוח בזמן אמת במשך 24-48 שעות, אין צורך לחזות את הזמן כדי ללכוד את ה- Dmax האמיתי בתוך קבוצות של תרכובות חדשות או לא ידועות.
קינטיקה בזמן אמת מאפשרת גם סינון יעיל של תגובת מינון כדי להבין טוב יותר את יעילות התרכובת, כיצד ריכוז התרכובות משפיע על קצב הפירוק הראשוני, ומציעה אפשרויות לדירוג תרכובות על סמך יותר מפרמטר אחד. מדידות קלאסיות של עוצמת השפלה כוללות חישובי DC50 בנקודת זמן מסוימת בהתבסס על מקסימום השפלה לכאורה. לעומת זאת, הגישה הקינטית שלנו להערכת עוצמה משלבת את מקסימום ההשפלה האמיתית בכל ריכוז, ללא קשר לזמן שבו היא מתרחשת בזמן18. אנו קוראים למדידה זו של עוצמת ההשפלה הקינטית, Dmax5018. ניתוח באופן זה לוקח בחשבון תרכובות שעלולות ליזום השפלה לאט יותר בריכוזים נמוכים יותר ולכן לוקח זמן רב יותר לאחר הטיפול להגיע ל- Dmax שלהן. זה יכול להיות אינפורמטיבי במיוחד לדרג תרכובות הן על קצב השפלה והן על Dmax. עבור המשפילים החזקים ביותר זה יבדיל עוד יותר בין משפילים איטיים, אך חזקים, לבין אלה שהם גם מהירים וגם חזקים. יחד, הן הסינון הקינטי והן הסינון הקינטי של תאים חיים תוך שימוש בקווי תאי HiBiT CRISPR הן גישות רבות עוצמה המניבות תמונה מקיפה יותר של פירוק חלבונים ממוקד, תפקוד תרכובת, ומאפשרות את תהליך הסינון מהערכת הפעילות הראשונית ועד לאופטימיזציה כימית במורד הזרם באמצעות שיפור פרמטרים מרכזיים של פירוק.
Disclosures
תאגיד פרומגה הוא הבעלים המסחרי על ידי הקצאת פטנטים של טכנולוגיות ויישומים HiBiT ו- NanoLuc.
Acknowledgements
K.M.R, S.D.M, M.U. ו- D.L.D הם כולם עובדי תאגיד פרומגה
Materials
| Name | Company | Catalog Number | Comments |
| CellTiter-Glo 2.0 reagent | Promega | G9241 | Cell Viability luminescent assay |
| CellTiter-Fluor Cell Viability Assay | Promega | G6080 | Cell Viability fluorescent assay |
| CO2-independent medium | ThermoFisher | 18045-088 | Cell culture |
| DMSO | Sigma Aldrich | D2650 | For compound dilution and control |
| DPBS | Gibco | 14190 | Cell culture |
| Fetal Bovine Serum | Seradigm | 89510-194 | Cell culture |
| HEK293 LgBiT stable cell line | Promega | N2672 | For complementation with HiBiT to generate luminescence |
| HiBiT CRISPR mammalian cell line | Promega | https://www.promega.com/crispr-tpd | |
| Hygromycin B solution | Gibco | 10-687-010 | Cell culture |
| LgBiT BacMam | Promega | CS1956C01 | For complementation with HiBiT to generate luminescence |
| LgBiT Expression Vector | Promega | N2681 | For complementation with HiBiT to generate luminescence |
| Luminometer Plate Reader | Luminomenter capable of measuring luminescence and fluorescence (e.g. GloMax Discover System, Promega GM3000) | ||
| NanoGlo Endurazine live cell substrate | Promega | N2570 | Kinetic HiBiT reagent |
| NanoGlo Vivazine live cell substrate | Promega | N2580 | Kinetic HiBiT reagent |
| NanoGlo HiBiT Lytic Detection system | Promega | N3030 | Enpoint lytic HiBiT reagent |
| Opti-MEM Reduced Serum Medium, no phenol red (ThermoFisher) | ThermoFisher | 11058-021 | Cell culture |
| Tissue culture plates, white, 96 well plate | Costar | 3917 | Cell culture |
| Tissue culture plates, white, 384 well plate | Corning | 3570 | Cell culture |
| Trypsin/EDTA | Gibco | 25300 | Cell culture |
References
- Burslem, G. M., Crews, C. M. Proteolysis-targeting chimeras as therapeutics and tools for biological discovery. Cell. 181 (1), 102-114 (2020).
- Chamberlain, P. P., Hamann, L. G. Development of targeted protein degradation therapeutics. Nature Chemical Biology. 15 (10), 937-944 (2019).
- Churcher, I. Protac-induced protein degradation in drug discovery: Breaking the rules or just making new ones. Journal of Medicinal Chemistry. 61 (2), 444-452 (2018).
- Ciulli, A., Farnaby, W. Protein degradation for drug discovery. Drug Discovery Today: Technologies. 31, 1-3 (2019).
- Crews, C. M. Inducing protein degradation as a therapeutic strategy. Journal of Medicinal Chemistry. 61 (2), 403-404 (2018).
- Cromm, P. M., Crews, C. M. Targeted protein degradation: from chemical biology to Drug Discovery. Cell Chemical Biology. 24 (9), 1181-1190 (2017).
- Deshaies, R. J. Protein degradation: Prime time for PROTACs. Nature Chemical Biology. 11 (9), 634-635 (2015).
- Lai, A. C., Crews, C. M. Induced protein degradation: an emerging drug discovery paradigm. Nature Reviews Drug Discovery. 16 (2), 101-114 (2017).
- Ottis, P., Crews, C. M. Proteolysis-targeting chimeras: Induced protein degradation as a therapeutic strategy. ACS Chemical Biology. 12 (4), 892-898 (2017).
- Wu, T., et al. Targeted protein degradation as a powerful research tool in basic biology and drug target discovery. Nature Structural and Molecular Biology. 27, 605-614 (2020).
- Hanan, E. J., et al. Monomeric targeted protein degraders. Journal of Medicinal Chemistry. , (2020).
- Collins, I., Wang, H., Caldwell, J. J., Chopra, R. Chemical approaches to targeted protein degradation through modulation of the ubiquitin-proteasome pathway. Biochemical Journal. 474 (7), 1127-1147 (2017).
- Carmony, K. C., Kim, K. B. PROTAC-induced proteolytic targeting. Methods in Molecular Biology. 832, 627-638 (2012).
- Daniels, D. L., Riching, K. M., Urh, M. Monitoring and deciphering protein degradation pathways inside cells. Drug Discovery Today: Technologies. 31, 61-68 (2019).
- Gu, S., Cui, D., Chen, X., Xiong, X., Zhao, Y. PROTACs: An emerging targeting technique for protein degradation in drug discovery. Bioessays. 40 (4), 1700247 (2018).
- Neklesa, T. K., Winkler, J. D., Crews, C. M. Targeted protein degradation by PROTACs. Pharmacology and Therapy. 174, 138-144 (2017).
- Raina, K., Crews, C. M. Targeted protein knockdown using small molecule degraders. Current Opinion in Chemical Biology. 39, 46-53 (2017).
- Riching, K. M., et al. Quantitative live-cell kinetic degradation and mechanistic profiling of PROTAC mode of action. ACS Chemical Biology. 13 (9), 2758-2770 (2018).
- Schwinn, M. K., et al. CRISPR-mediated tagging of endogenous proteins with a luminescent peptide. ACS Chemical Biology. 13 (2), 467-474 (2018).
- Schwinn, M. K., Steffen, L. S., Zimmerman, K., Wood, K. V., Machleidt, T. A simple and scalable strategy for analysis of endogenous protein dynamics. Science Reports. 10 (1), 8953 (2020).
- Bensimon, A., et al. Targeted degradation of SLC transporters reveals amenability of multi-pass transmembrane proteins to ligand-induced proteolysis. Cell Chemical Biology. 27 (6), 728-739 (2020).
- Bondeson, D. P., et al. Lessons in PROTAC design from selective degradation with a promiscuous warhead. Cell Chemical Biology. 25 (1), 78-87 (2018).
- Buckley, D. L., et al. HaloPROTACS: Use of small molecule PROTACs to induce degradation of HaloTag fusion proteins. ACS Chemical Biology. 10 (8), 1831-1837 (2015).
- Bulatov, E., Ciulli, A. Targeting Cullin-RING E3 ubiquitin ligases for drug discovery: structure, assembly and small-molecule modulation. Biochemical Journal. 467 (3), 365-386 (2015).
- Burslem, G. M., et al. The advantages of targeted protein degradation over inhibition: An RTK case study. Cell Chemical Biology. 25 (1), 67-77 (2018).
- Chamberlain, P. P., et al. Evolution of cereblon-mediated protein degradation as a therapeutic modality. ACS Medicinal Chemistry Letters. 10 (12), 1592-1602 (2019).
- Crew, A. P., et al. Identification and characterization of Von Hippel-Lindau-recruiting Proteolysis Targeting Chimeras (PROTACs) of TANK-Binding Kinase 1. Journal of Medicinal Chemistry. 61 (2), 583-598 (2018).
- DeMars, K. M., Yang, C., Castro-Rivera, C. I., Candelario-Jalil, E. Selective degradation of BET proteins with dBET1, a proteolysis-targeting chimera, potently reduces pro-inflammatory responses in lipopolysaccharide-activated microglia. Biochemical and Biophysical Research Communication. 497 (1), 410-415 (2018).
- Erb, M. A., et al. Transcription control by the ENL YEATS domain in acute leukaemia. Nature. 543 (7644), 270-274 (2017).
- Farnaby, W., et al. BAF complex vulnerabilities in cancer demonstrated via structure-based PROTAC design. Nature Chemical Biology. 15 (7), 672-680 (2019).
- Gadd, M. S., et al. Structural basis of PROTAC cooperative recognition for selective protein degradation. Nature Chemical Biology. 13 (5), 514-521 (2017).
- Gechijian, L. N., et al. Functional TRIM24 degrader via conjugation of ineffectual bromodomain and VHL ligands. Nature Chemical Biology. 14 (4), 405-412 (2018).
- Gustafson, J. L., et al. Small-Molecule-Mediated Degradation of the Androgen Receptor through Hydrophobic Tagging. Angewandte Chemie International Edition England. 54 (33), 9659-9662 (2015).
- Kerres, N., et al. Chemically induced degradation of the oncogenic transcription factor BCL6. Cell Reports. 20 (12), 2860-2875 (2017).
- Lohbeck, J., Miller, A. K. Practical synthesis of a phthalimide-based Cereblon ligand to enable PROTAC development. Bioorganic and Medicinal Chemistry Letters. 26 (21), 5260-5262 (2016).
- Lu, J., et al. Hijacking the E3 ubiquitin ligase cereblon to efficiently target BRD4. Chemical Biology. 22 (6), 755-763 (2015).
- Lu, M., et al. Discovery of a Keap1-dependent peptide PROTAC to knockdown Tau by ubiquitination-proteasome degradation pathway. Eurupean Journal of Medicinal Chemistry. 146, 251-259 (2018).
- Nabet, B., et al. The dTAG system for immediate and target-specific protein degradation. Nature Chemical Biology. 14 (5), 431-441 (2018).
- Nowak, R. P., et al. Plasticity in binding confers selectivity in ligand-induced protein degradation. Nature Chemical Biology. 14, 706-714 (2018).
- Powell, C. E., et al. Chemically induced degradation of Anaplastic Lymphoma Kinase (ALK). Journal of Medicinal Chemistry. 61 (9), 4249-4255 (2018).
- Raina, K., et al. PROTAC-induced BET protein degradation as a therapy for castration-resistant prostate cancer. Proceedings of National Academy of Science U. S. A. 113 (26), 7124-7129 (2016).
- Sakamoto, K. M., et al. Protacs: chimeric molecules that target proteins to the Skp1-Cullin-F box complex for ubiquitination and degradation. Proceeding National Academy Science. 98 (15), 8554-8559 (2001).
- Sakamoto, K. M., et al. Development of Protacs to target cancer-promoting proteins for ubiquitination and degradation. Molecular Cell Proteomics. 2 (12), 1350-1358 (2003).
- Schiedel, M., et al. Chemically induced degradation of Sirtuin 2 (Sirt2) by a proteolysis targeting chimera (PROTAC) based on sirtuin rearranging ligands (SirReals). Journal of Medicinal Chemistry. 61 (2), 482-491 (2018).
- Slabicki, M., et al. The CDK inhibitor CR8 acts as a molecular glue degrader that depletes cyclin K. Nature. , (2020).
- Smith, B. E., et al. Differential PROTAC substrate specificity dictated by orientation of recruited E3 ligase. Nature Communications. 10 (1), 131 (2019).
- Sun, B., et al. BET protein proteolysis targeting chimera (PROTAC) exerts potent lethal activity against mantle cell lymphoma cells. Leukemia. 32 (2), 343-352 (2018).
- Tong, B., et al. A Nimbolide-based kinase degrader preferentially degrades oncogenic BCR-ABL. ACS Chemical Biology. 15 (7), 1788-1794 (2020).
- Winter, G. E., et al. Drug Development. Phthalimide conjugation as a strategy for in vivo target protein degradation. Science. 348 (6241), 1376-1381 (2015).
- Winter, G. E., et al. BET Bromodomain proteins function as master transcription elongation factors independent of CDK9 recruitment. Molecular Cell. 67 (1), 5-18 (2017).
- Zengerle, M., Chan, K. H., Ciulli, A. Selective Small Molecule Induced Degradation of the BET Bromodomain Protein BRD4. ACS Chemical Biology. 10 (8), 1770-1777 (2015).
- Zhang, C., et al. Proteolysis targeting chimeras (PROTACs) of anaplastic lymphoma kinase (ALK). European Journal of Medicinal Chemistry. 151, 304-314 (2018).
- Zoppi, V., et al. Iterative design and optimization of initially inactive proteolysis targeting chimeras (PROTACs) identify VZ185 as a potent, fast, and selective von Hippel-Lindau (VHL) based dual degrader probe of BRD9 and BRD7. Journal of Medicinal Chemistry. 62 (2), 699-726 (2019).
- Huang, H. T., et al. A chemoproteomic approach to query the degradable kinome using a multi-kinase degrader. Cell Chemical Biology. 25 (1), 88-99 (2018).
- Bjorklund, C. C., et al. Iberdomide (CC-220) is a potent cereblon E3 ligase modulator with antitumor and immunostimulatory activities in lenalidomide- and pomalidomide-resistant multiple myeloma cells with dysregulated CRBN. Leukemia. 34 (4), 1197-1201 (2020).
- Matyskiela, M. E., et al. SALL4 mediates teratogenicity as a thalidomide-dependent cereblon substrate. Nature Chemical Biology. 14 (10), 981-987 (2018).
- Matyskiela, M. E., et al. A cereblon modulator (CC-220) with improved degradation of Ikaros and Aiolos. Journal of Medicinal Chemistry. 61 (2), 535-542 (2018).
- Bussiere, D. E., et al. Structural basis of indisulam-mediated RBM39 recruitment to DCAF15 E3 ligase complex. Nature Chemical Biology. 16 (1), 15-23 (2020).
- Du, X., et al. Structural basis and kinetic pathway of RBM39 recruitment to DCAF15 by a sulfonamide molecular glue E7820. Structure. 27 (11), 1625-1633 (2019).
- Ting, T. C., et al. Aryl sulfonamides degrade RBM39 and RBM23 by recruitment to CRL4-DCAF15. Cell Reports. 29 (6), 1499-1510 (2019).
- Hughes, S. J., Ciulli, A. Molecular recognition of ternary complexes: a new dimension in the structure-guided design of chemical degraders. Essays in Biochemistry. 61 (5), 505-516 (2017).
- Schapira, M., Calabrese, M. F., Bullock, A. N., Crews, C. M. Targeted protein degradation: expanding the toolbox. Nature Review Drug Discovery. 18 (12), 949-963 (2019).
- Dixon, A. S., et al. NanoLuc complementation reporter optimized for accurate measurement of protein interactions in cells. ACS Chemical Biology. 11 (2), 400-408 (2016).
- Gilan, O., et al. Selective targeting of BD1 and BD2 of the BET proteins in cancer and immuno-inflammation. Science. 368 (6489), 387-394 (2020).
- Oh-Hashi, K., Furuta, E., Fujimura, K., Hirata, Y. Application of a novel HiBiT peptide tag for monitoring ATF4 protein expression in Neuro2a cells. Biochemical Biophysical Report. 12, 40-45 (2017).
- Ottis, P., et al. Cellular resistance mechanisms to targeted protein degradation converge toward impairment of the engaged ubiquitin transfer pathway. ACS Chemical Biology. 14 (10), 2215-2223 (2019).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved