Method Article
הרכבה הגנום ההיברידית דה נובו לייצור גנומים שלמים של חיידקי השתן באמצעות טכנולוגיות רצף קצרות וארוכות
In This Article
Summary
פרוטוקול זה מפרט גישה מקיפה עבור כתות, רצף, ואת הרכב הגנום ההיברידי דה נובו של חיידקי השתן. הוא מספק הליך לשחזור ליצירת רצפי גנום מעגליים שלמים שימושיים בחקר יסודות גנטיים כרומוזומליים ואקסטרכרומוזומליים התורמים לקולוניזציה של שתן, פתוגנזה והפצת התנגדות מיקרוביאלית.
Abstract
רצפי גנום שלמים מספקים נתונים בעלי ערך להבנת המגוון הגנטי וגורמי ההתיישבות הייחודיים של חיידקי השתן. נתונים אלה עשויים לכלול אלמנטים גנטיים ניידים, כגון plasmids ו phage חוץכרומוזומלי, התורמים להפצת התנגדות מיקרוביאלית ומסבך עוד יותר את הטיפול בדלקת בדרכי השתן (UTI). בנוסף למתן רזולוציה עדינה של מבנה הגנום, הגנום הסגור והשלם מאפשר את הגנומיה ההשוואתית המפורטת ואת הניתוחים האבולוציוניים. הדור של גנומים שלמים דה נובו כבר זמן רב משימה מאתגרת בשל מגבלות של טכנולוגיית ריצוף זמין. רצף הדור הבא (NGS) המשויך מפיק קריאות קצרות באיכות גבוהה, וכתוצאה מכך מורכבים גנום מדויקים אך מקוטעים. להיפך, רצף Nanopore מספק קריאות ארוכות של איכות נמוכה יותר בדרך כלל המוביל להרכבות שלמות מועדות לשגיאות. שגיאות כאלה עלולות לעכב מחקרי שיוך כלל-גנומיים או לספק תוצאות ניתוח משתנות מטעות. לכן, גישות היברידיות המשלבות קריאות קצרות וארוכות התגלו כשיטות אמינות להשגת גנומים חיידקיים סגורים מדויקים ביותר. כאן דווח שיטה מקיפה לתרבות של חיידקי שתן מגוונים, זיהוי מינים על ידי ריצוף גנים rRNA 16S, הפקת DNA גנומי (gDNA), ויצירת קריאות קצרות וארוכות על ידי פלטפורמות NGS וננופור, בהתאמה. בנוסף, שיטה זו מתארת צינור ביואינפורמטי של אלגוריתמים לבקרת איכות, הרכבה וחיזוי גנים ליצירת רצפי גנום שלמים מובאים. שילוב של כלים ביואינפורמטיים מאפשר בחירה של נתוני קריאה באיכות גבוהה להרכבה גנומית היברידית וניתוח במורד הזרם. הגישה היעילה של הרכב הגנום ההיברידי דה נובו המתואר בפרוטוקול זה עשויה להיות מותאמת לשימוש בכל חיידק פולחן.
Introduction
המיקרוביום בדרכי השתן הוא תחום מחקר מתפתח שניפץ תפיסה מוטעית ארוכה של עשרות שנים כי דרכי השתן סטריליות אצל אנשים בריאים. חברי המיקרוביוטה בדרכי השתן עשויים לשמש לאיזון סביבת השתן ולמנוע זיהום בדרכי השתן (UTI)1,2. חיידקים אורופתיגניים פולשים לדרכי השתן ומפעילים מנגנוני ארס מגוונים כדי לעקור את המיקרוביוטה המקומית, ליישב את האורותליום, להתחמק מתגובות חיסוניות ולנטרל לחצים סביבתיים3,4. שתן הוא מדיום מוגבל בחומרים מזינים יחסית המאופיין באוסמולריות גבוהה, זמינות מוגבלת של חנקן ופחמימות, חמצון נמוך ו- pH נמוך5,6,7. שתן נחשב גם אנטי מיקרוביאלי, המורכב ריכוזים גבוהים של אוריאה מעכבת ופפטידים מיקרוביאליים כגון קטלייצידין האדם LL-378. מנגנוני חקירה המועסקים הן על ידי חיידקים מקומיים והן אורופטוגנים כדי ליישב את דרכי השתן היא קריטית להבנת בריאות דרכי השתן ולפיתוח אסטרטגיות חדשות לטיפול ב- UTI. יתר על כן, כמו הכישלון של טיפולים מיקרוביאליים קו החזית הופך נפוץ יותר, חשוב יותר ויותר לפקח על הפצת אלמנטים גנטיים ניידים הנושאים דטרמיננטיות התנגדות מיקרוביאלית בתוך אוכלוסיות של חיידקים בדרכי השתן9,10.
כדי לחקור גנוטיפים ופנוטיפים של חיידקי השתן, התרבות המוצלחת שלהם ורצף הגנום המלא לאחר מכן (WGS) הוא הכרחי. שיטות תלויות תרבות נחוצות כדי לזהות ולזהות חיידקים קיימא בדגימות שתן11. תרבית שתן קלינית סטנדרטית כוללת ציפוי שתן על אגר דם כבשים 5% (BAP) ו MacConkey אגר ודגרה אירובית ב 35 °C (5 °F) עבור 24 שעות12. עם זאת, עם סף זיהוי של ≥105 CFU / mL13, חברים רבים של מיקרוביוטה השתן אינם מדווחים על ידי שיטה זו. טכניקות פולחן משופרות כגון תרבית שתן כמותית משופרת (EQUC)11 משתמשות בשילובים שונים של נפחי שתן שונים, זמני דגירה, מדיה תרבותית ותנאים אטמוספריים כדי לזהות חיידקים שבדרך כלל חסרים על ידי תרבית שתן סטנדרטית. מתואר בפרוטוקול זה היא גרסה שונה של EQUC, המכונה כאן פרוטוקול תרבית שתן משופרת שונה, המאפשר culturing של חיידקים בדרכי השתן מגוונים אורופטוגנים באמצעות מדיה סלקטיבית ותנאים אטמוספריים אופטימליים אבל הוא לא כמותי מטבעו. הבידוד המוצלח של חיידקי השתן מאפשר הפקת דנ"א גנומי (gDNA) עבור WGS במורד הזרם והרכבה גנומית.
מכלולי הגנום, המורכבים המלאים בפרט, מאפשרים גילוי של גורמים גנטיים שעשויים לתרום להתיישבות, תחזוקת נישה ונדיפות בקרב חיידקי מיקרוביוטה ואורופתוגניים מקומיים כאחד. מכלולי גנום טיוטה מכילים מספר מגוון של רצפים רציפים (רציפים) שעשויים להכיל שגיאות רצף וחסר מידע כיוון. בהרכבת גנום מלאה, הן הכיוון והן הדיוק של כל זוג בסיס אומתו14. יתר על כן, השגת רצפי גנום שלמים מספקת תובנה על מבנה הגנום, המגוון הגנטי, ואלמנטים גנטיים ניידים15. קריאות קצרות לבדן עשויות לזהות נוכחות או היעדר גנים חשובים, אך אינן עשויות להצביע על ההקשר הגנומי שלהם16. עם הפעלת טכנולוגיות רצף קריאה ארוכה כגון אוקספורד Nanopore ו PacBio, יצירת מכלולי דה נובו סגורים של גנומים חיידקיים כבר לא דורש שיטות מאומצות כגון סגירה ידנית של מכלולי דה נובו על ידי מולטיפלקס PCR17,18. השילוב של הדור הבא של רצף קריאה קצרה וטכנולוגיות רצף של Nanopore לקריאה ארוכה מאפשר לדור קל של מכלולי גנום חיידקיים מדויקים, שלמים וסגורים בעלות נמוכה יחסית19. רצף קריאה קצרה מייצר מכלולי גנום מדויקים אך מקוטעים המורכבים בדרך כלל מממוצע של 40-100 קונטיבים, בעוד רצף Nanopore מייצר קריאות ארוכות של כ 5-100 kb באורך כי הם פחות מדויקים אבל יכול לשמש פיגומים להצטרף contigs ולפתור סינטטוני גנומי. גישות היברידיות המשתמשות בטכנולוגיות לקריאה קצרה והן בטכנולוגיות קריאה ארוכה יכולות לייצר גנומים חיידקיים מדויקים ומלאים19.
המתואר כאן הוא פרוטוקול מקיף לבידוד וזיהוי של חיידקים משתן אנושי, מיצוי DNA גנומי, רצף, והרכבה גנומית מלאה באמצעות גישת הרכבה היברידית. פרוטוקול זה מספק דגש מיוחד על השלבים הדרושים לשינוי נכון של קריאות שנוצרו על ידי רצף קריאה קצרה וקריאה ארוכה להרכבה מדויקת של כרומוזום חיידקי סגור ואלמנטים חוץ-כרומוזומליים כגון פלסמידים.
Protocol
חיידקים היו בתרבית משתן שנאסף מנשים בהסכמה כחלק ממחקרים שאושרו על ידי ועדת הבדיקה המוסדית 19MR0011 (UTD) ו- STU 032016-006 (UTSW).
1. תרבית שתן משופרת שונה
הערה: כל צעדי התרבות חייבים להתבצע בתנאים סטריליים. עקר את כל המכשירים, הפתרונות והמדיה. נקו את אזור העבודה עם 70% אתנול, ואז הקימו מבער בונזן ועבדו בזהירות קרוב ללהבה כדי להפחית את סיכויי הזיהום. לחלופין, ארון בטיחות ביולוגית מסוג II עשוי לשמש לשמירה על סביבה סטרילית. ללבוש ציוד מגן אישי מתאים (PPE) כדי למנוע חשיפה חיידקים פתוגניים פוטנציאליים.
- ציפוי שתן מלא גלצל ובידוד מושבה
- הפשר שתן המצויד בגליצריל בטמפרטורת החדר (RT). לאחר הפשרה, מערבולת המדגם עבור 5 s לערבב. בצינורות מיקרוצנטריפוגה סטריליים, להכין 1:3 ו 1:30 דילול של השתן סטרילי 1x פוספט-חוצץ מלוח (PBS) לנפח סופי של 100 μL.
הערה: שתן מלאי גליצל מוכן על ידי ערבוב 500 μL של שתן לא דולל ו 500 μL של 50% גלילסול סטרילי cryovials ולאחסן ב -80 °C (80 °F). - צלחות אגר חמות מראש ב 37 °C (5 °F) במשך 15 דקות לפני השימוש. אנא עיינו באיור 1 לסוגי מדיה ולתנאי תרבות המתאימים לסוגי חיידקי השתן הנפוצים. מערבבים היטב את השתן המדולל על ידי צנרת לפני הציפוי, צלחת 100 μL של השתן המדולל על צלחת אגר הרצויה ולהפיץ את המדגם באמצעות חרוזי זכוכית סטריליים. צלחת 100 μL של 1x PBS דילול על צלחת נפרדת כמו בקרת צמיחה.
הערה: אם מנסים לתרבות מינים אורופתיוגנייםנפוצים(למשל, Escherichia coli, Klebsiella spp., Enterococcus faecalis וכו '), מומלץ להשתמש אגר כרומוגניים ( טבלת חומרים ) כפי שהוא מאפשר זיהוי קל של מינים חיידקיים אורופתיגניים (איור 1). חומצה קוליסטין Nalidixic (CNA) או MRS אגר שימושיים לבידוד מינים גראם חיובי בררני (למשל, לקטובצילוס spp.) משתן ידוע להכיל אורופטוגנים גרם שלילי, אשר עשויים להערים על המינים בררניים אגרים שאינם סלקטיביים. - לדגור את הצלחת הפוכה במצב האטמוספרי הרצוי ב 35 °C (35 °C) לתקופה של 24 שעות עבור אורופטוגנים ו 3-5 ימים עבור חיידקים בררניים(איור 1).
- לאחר תקופת הדגירה, להסיר את הצלחות מן האינקובטור. מכל צלחת, בחר את המושבות המציגות צבע ייחודי, מורפולוגיה או דפוסים המוליטיים.
- פס מחדש את מושבת החיידקים באמצעות לולאה סטרילית על אגר המתאים ודגורת הצלחת הפוכה במשך 2-5 ימים באטמוספירה הרצויה כדי להשיג מושבות מבודדות היטב.
הערה: אם משתמשים BAP לתרבות ראשונית, תיקון מושבות על אגר כרומוגניים עשוי לספק מידע שימושי על ההטרוגניות של אוכלוסיית החיידקים במדגם.
- הפשר שתן המצויד בגליצריל בטמפרטורת החדר (RT). לאחר הפשרה, מערבולת המדגם עבור 5 s לערבב. בצינורות מיקרוצנטריפוגה סטריליים, להכין 1:3 ו 1:30 דילול של השתן סטרילי 1x פוספט-חוצץ מלוח (PBS) לנפח סופי של 100 μL.
- פולחן במרק נוזלי וגליצל-גרביים חיידקים מבודדים
- לאחר שהמושבות המבודדות התואמות את המורפולוגיה של מושבת ההורים מתקבלות, בחרו מושבה אחת וחסן ל-3 מ"ל של מרק נוזלי באמצעות לולאת חיסון סטרילית. עיין באיור 1 למרק המסוגל לתמוך בצמיחת המיקרוביוטה נפוצה בדרכי השתן. לאטום את צלחות אגר עם parafilm ולאחסן אותם ב 4 °C (4 ימים). לדגור על תרבויות נוזליות בתנאים האטמוספריים הרצויים במשך 1-5 ימים עד שהתרבות היא מעוכבת בעליל.
- לאחר הצמיחה הוא ציין, מערבולת התרבות, ולאחר מכן להוסיף 1 מ"ל של תרבות הלילה כדי 500 μL של גליצרל סטרילי 50% בקריוביאלי 2 מ"ל; לאטום ולערבב בעדינות על ידי היפוך. הכן שתי מניות גליצריל עבור כל מושבה (אחד משמש כגיבוי) ולאחסן ב -80 °C (80 °F).
2. זיהוי מינים חיידקיים על ידי רצף גן סנגר 16S rRNA
הערה: זהות מיקרוביאלית יכולה להיות מאושרת לחילופין באמצעות זמן יינון לייזר בסיוע מטריצה של ספקטרומטר מסה טיסה (MALDI-TOF)20.
- תגובת שרשרת מושבה-פולימראז (PCR)
- הכן 25 μL של תגובת PCR בצינורות PCR על ידי הוספת 12.5 μL של 2x טאק פולימראז מאסטר מיקס, 0.5 μL של 10 μM 8F פריימר, 0.5 μL של 10 μM 1492R פריימר(שולחן החומרים),ו 11.5 μL של מים ללא נוקלאז21.
הערה: אם אתם מבצעים PCR עבור דגימות מרובות, בצעו תערובת ראשית של תערובת טאק פולימראז, פריימרים ומים סטריליים ללא גרעין. ואז aliquot 25 μL לתוך כל צינור PCR. - כדי לבצע מושבה-PCR, החלק מושבה מבודדת היטב מן הפס מחדש באמצעות קיסם סטרילי או קצה פיפטה. resuspend המושבה בתערובת תגובת PCR מוכן בשלב 2.1.1. מערבבים בעדינות. לאסוף את הנוזל בתחתית הצינור על ידי סיבוב מהיר ב 2000 x g.
הערה: ודאו שהדגימה נקייה מבועות אוויר. כלול דוגמה ללא פקד תבנית (NTC) המכילה את תערובת התגובה של PCR בלבד. - מניחים את צינורות המדגם בתרמוסקלר ולהפעיל את התוכנית הבאה: 95 °C (55 °F) במשך 3 דקות; 40 מחזורים של: 95 °C (55 °F) ל-30 מעלות צלזיוס, 51 °C (50 °F) ל-30 מעלות צלזיוס ו-72 °C (72°F) למשך דקה 30; 72 °C (72 °F) למשך 10 דקות; החזק ב 10 °C (50 °F).
- הכן 25 μL של תגובת PCR בצינורות PCR על ידי הוספת 12.5 μL של 2x טאק פולימראז מאסטר מיקס, 0.5 μL של 10 μM 8F פריימר, 0.5 μL של 10 μM 1492R פריימר(שולחן החומרים),ו 11.5 μL של מים ללא נוקלאז21.
- מיצוי ג'ל וזיהוי מינים
- עם השלמת ריצת ה- PCR, בדוק את מוצר ה- PCR על ג'ל אגרוז 1% שהוכן במאגר 0.5x Tris-Borate-EDTA (TBE). לפני הליהוק של הג'ל, מוסיפים אתידיום ברומיד (EtBr). לאחר מכן, להטיל את הג'ל באמצעות מסרקים עבור בארות להחזיק לפחות 20 נפח מדגם μL.
זהירות: EtBr הוא סוכן intercalating חשוד להיות מסרטן. תמיד ללבוש כפפות PPE בעת טיפול בו ולהיפטר מחומרים המכילים EtBr על פי הנחיות המוסד. - כאשר הג'ל מוגדר, מניחים את הג'ל במיכל האלקטרופורזה מלא במאגר TBE 0.5x ומסירים את המסרק. לטעון את סולם 1 kb בבאר הראשונה ו 10-20 μL של תגובת PCR לתוך הבא. הפעל ב 100-140 V עד לפתרון. דמיינו את הג'ל תחת אור UV ואשרו את נוכחותה של רצועה מוגדרת בבירור ב- ~ 1.5 kb הנעדרת בבאר NTC.
אזהרה: קרני UV מזיקות לעור ולעיניים, השתמשו בשומר מתאים בעת הדמיית הג'ל ולובש PPE מתאים.
הערה: PCR המושבה עשוי להיות לא מוצלח עבור חיידקים מסוימים; המשך עם PCR מ gDNA מבודד היא אפשרות חלופית22. - Excise את רצועות ~ 1.5 kb באמצעות סכין גילוח ולהעביר את ג'ל חתכים לתוך צינורות microcentrifuge נקי. המשך עם פרוטוקול מיצוי ג'ל בהתאם להוראותהיצרן (טבלת חומרים). למדוד את הריכוז של DNA מטוהר על ידי ספקטרופוטומטר microvolume.
הערה: ריכוז >10 ננוגרם /μL רצוי, ו A260/280 בין 1.7-2.0 מקובל. - הכן שתי תגובות רצף סנגר עבור כל מדגם, אחד באמצעות 8F והשני באמצעות פריימר 1492R במים ללא נוקלאז על פי ההנחיות של כל שירות רצף סנגר שנבחר.
- לאחר קבלת נתוני הרצף, העלה את רצפי הדנ"א לאתר האינטרנט של כלי חיפוש היישור המקומי הבסיסי של NCBI (BLAST) (blast.ncbi.nlm.nih.gov/Blast.cgi), בחר Nucleotide BLAST (blastn), בחר את רצפי ה- RNA rRNA / ITS 16S ריבוזומלי (חיידקים וארכיאה) והפעל את תוכנית Megablast. ניתן לזהות את הבידוד על-ידי הפגיעה באיכות הגבוהה ביותר בהפניה ממסד הנתונים.
הערה: מינים חיידקיים מסוימים מפגינים זהות גבוהה ברצפי rRNA 16S שלהם ועשויים להיות מובדלים בשיטה זו בלבד. דגימה תדרוש הומולוגיה DNA ניתוחים ביוכימיים כדי להבחין בביטחון חברים מאותו סוג23.
- עם השלמת ריצת ה- PCR, בדוק את מוצר ה- PCR על ג'ל אגרוז 1% שהוכן במאגר 0.5x Tris-Borate-EDTA (TBE). לפני הליהוק של הג'ל, מוסיפים אתידיום ברומיד (EtBr). לאחר מכן, להטיל את הג'ל באמצעות מסרקים עבור בארות להחזיק לפחות 20 נפח מדגם μL.
3. הפקת דנ"א גנומי (gDNA)
הערה: סעיף זה משתמש ריאגנטים ועמודות ספין המסופקים בערכת החילוץ gDNA המוזכרת בטבלת החומרים להפקת תשואה גבוהה של DNA גנומי איכותי ממיני חיידקים מגוונים. להלן שינויים מומלצים והוראות.
- הכן ריאגנטים של ערכה לפי הוראות היצרן.
- הכן תרבויות של 3-10 מ"ל במרק סטרילי מתאים (איור 1) על ידי חנק חיידקים ממושבות מבודדות היטב לתקשורת ודגרה בטמפרטורה ובלחץ האטמוספרי שצוין באיור 1 עד לצמיחה מספקת.
- לאחר הדגירה, למדוד את הצפיפות האופטית ב 600 ננומטר (OD600)של התרבות באמצעות ספקטרופוטומטר24.
- הכן את המדגם לכימות על ידי דילול תרביות לילה ביחס של 1:10. כלול ריק של מדיה תרבותית סטרילית למדידה גם כן. חשב את הצפיפות האופטית על-ידי חיסור הקריאה הריקה מקריאת המדגם והכפלה בגורם הדילול של עשר.
- באמצעות מדידת OD600 ויחס OD600 ל- CFU / mL שנקבע מראש עבור המינים, לחשב כמה מיליליטרים של תרבות נחוצים כדי להשיג 2 x 109 תאים.
- צנטריפוגה נפח התרבות הנדרש במשך 5 דקות ב 5000 x g לכדור. שאפו את supernatant ו resuspened הכדור ב 200 μL חיץ TE קר (מראש צמרמורת על קרח בתחילת ההליך).
- צנטריפוגה הדגימה במשך 2 דקות ב 5000 x g. הסר את supernatant, ולאחר מכן resuspend הכדור ב 180 μL של חוצץ תמיסת אנזימטית (ELB) ולהוסיף 20 μL של RNase מבושל מראש (10 מ"ג / מ"ל). לקבלת תמוגה יעילה של חיידקים גראם-חיוביים, יש להוסיף 18 מיקרו-אל של מוטנוליזין (25 קילוואט/מ"ל). מערבולת היטב, ולאחר מכן לדגור על הדגימות ב 37 °C (50 °F) על סיבוב במשך 2 שעות.
הערה: מומלץ להשתמש ELB המתואר בפרוטוקול היצרן עבור חיידקים גרם חיובי ו גרם שלילי. - המשך בהתאם להוראות היצרן.
הערה: חזור על שלבי ההעמקה עבור פעם אחת או פעמיים נוספות כדי להשיג תפוקת gDNA נוספת, אם תרצה. - להעריך את האיכות של gDNA שחולץ כפי שהורה בסעיף 4 ולאחסן gDNA ב 4 °C (70 °F) אם זה ישמש בתוך שבוע אחד. לחלופין, שמור gDNA ב-20 °C לאחסון לטווח ארוך.
4. הערכת איכות ה-GDNA שחולץ
- כדי להעריך את האיכות על ידי אלקטרופורזה ג'ל, להכין 1% ג'ל אגרוז כמתואר בסערה 2.2. הכן את המדגם בצינור נקי: לערבב 1-2 μL של gDNA שחולץ ו 3 μL של צבע טעינה 2x על parafilm. הפעל את הג'ל פעם טעון, ולאחר מכן לדמיין אותו תחת אור UV.
הערה: חילוץ gDNA מוצלח יתברר על ידי להקה נפרדת בחלק העליון של הג'ל והכפשה מינימלית (איור 2A). מריחה מעידה על גימה. אם לא ברורה רצועת gDNA ו/או מריחה היא משמעותית, חזור על חילוץ gDNA. שקול להפחית את זמני הדגירה ב RNase A ו Proteinase K. אם נצפות שתי רצועות סביב 1.5-3 kb, הדבר מצביע על זיהום RNA(איור 2B). הכן RNase A טריים וחזור על החילוץ. - כדי להעריך את האיכות על ידי ספקטרופוטומטר מיקרו-וולטום, מדוד את ריכוז ה-gDNA ואת יחס הספיגה A260/280 על ידי ספקטרופוטומטר מיקרו-וולטום. ריכוזים >50 ננוגרם /μL ו- A260/280 בין 1.7-2.0 מקובלים.
הערה: תפוקת gDNA נמוכה עשויה לנבוע מקלט נמוך, קלט גבוה, זיהום של גרקלאזים, תמוגה לא מספקת. יחסי ספיגה מעל הטווח מצביעים על זיהום RNA. מיצוי חוזר אם איכות gDNA ירודה. - כדי להעריך את האיכות על ידי פלואורומטר, בצע את הוראות היצרן לכמת את ריכוז gDNA באמצעות ערכת בדיקת רגישות גבוהה ומכשיר פלואורומטר(שולחן החומרים). ריכוז >50 ננוגרם / μL רצוי.
5. הדור הבא של הדור הבא בהכנה קצרת קריאה והכנת ספריה
הערה: ניתן לבצע רצף לקריאה קצרה בכלים שונים באורכים ובהכוונה ברורים לקריאה. רצף 150 bp (300 מחזור) מומלץ עבור WGS חיידקי. הן הכנת הספרייה והן הרצף עשויים להיות במיקור חוץ למתקני ליבה או למעבדות מסחריות.
- הכן ספריית רצף בהתאם להוראות היצרן (טבלת חומרים). עקוב אחר ריכוז ספריית הטעינה הסופית המומלץ של היצרן; עם זאת, שינוי מומלץ הוא לטעון את הספרייה המאגדת ב- 1.8 pM ליצירת קריאה אופטימלית במכשירי NextSeq.
- למרות האופציה, השתמש ביואנלייזר(טבלת חומרים)כדי להעריך את התפלגות שברי הספרייה במאגר ולוודא שגודל הקטע הוא 600 bp בממוצע.
6. הכנת ספריית רצף מיניון ננופור
- הכן את ספריית הרצף בהתאם לפרוטוקול היצרן (טבלת חומרים). שימוש בשתי ערכות הרחבת ברקוד מאפשר ריבוי של עד 24 דגימות בתא זרימה יחיד. מומלץ לבצע הכנת ספריה בשני חלקים, 12 דגימות בכל פעם בעת מולטיפלקסינג 24 דגימות. ניתן לאגד את כל 24 הדגימות כמתואר להלן.
הערה: דגימות עשויות להיות מאוחסנות ב 4 °C (5 °F) לילה עם סיום קשירת ברקוד מקורי - זה מספק נקודת עצירה בפרוטוקול, במידת הצורך. בסוף סעיף קשירת הברקוד המקורי של פרוטוקול הכנת הספרייה, מומלץ לאסוף כמויות שווימולריות של כל דגימה עד למסת ה- DNA המרבית (ng) האפשרית.- כדי לעשות זאת, לכמת את כל הדגימות הבאות קשירת ברקוד באמצעות פלואורומטר (טבלת חומרים) לפי הוראות היצרן. להעריך את נפח המדגם עם ריכוז dsDNA הנמוך ביותר ולאחר מכן לחשב את הסכום הכולל dsDNA נמצא במדגם זה. השתמש במספר זה כדי לקבוע את הכמויות ההזוותיות של כל הדגימות האחרות שיאחדו יחד.
הערה: מכיוון שהחישוב השוומור ימקסם את כמות ה- dsDNA המאוחס ובכך יניב מאגר בנפח גבוה (>65 μL), יש צורך בניקוי כדי לרכז את הבריכה.
- כדי לעשות זאת, לכמת את כל הדגימות הבאות קשירת ברקוד באמצעות פלואורומטר (טבלת חומרים) לפי הוראות היצרן. להעריך את נפח המדגם עם ריכוז dsDNA הנמוך ביותר ולאחר מכן לחשב את הסכום הכולל dsDNA נמצא במדגם זה. השתמש במספר זה כדי לקבוע את הכמויות ההזוותיות של כל הדגימות האחרות שיאחדו יחד.
- ניקוי וריכוז בבריכת dsDNA
- הוסף נפח של 2.5x של חרוזים פרמגנטיים (שולחן החומרים) למאגר הדנ"א, ולאחר מכן החלק בעדינות את הצינור כדי לערבב את התוכן. מניחים את הצינור ברוטציה במשך 5 דקות ב RT. ספין את המדגם ב 2000 x g וכדור על מגנט.
- מוסיפים 250 μL טרי מוכן 70% אתנול (במים ללא גרעין), דואג לא להפריע לכדור. שאפו את האתנול וחזרו על שטיפת האתנול פעם אחת.
- לאחר השאיפה השנייה, לסובב את המדגם ב 2000 x g ולהניח אותו בחזרה על המגנט. פיפטה את כל אתנול שיורית ולאפשר לדגימה להתייבש במשך כ 30 s.
- הסר את הצינור מן המגנט ו resuspend הכדור ב 60-70 μL של מים ללא גרקלאז. דגירה ב RT במשך 2 דקות. גלם את המדגם על המגנט עד elute הוא ברור, ולאחר מכן להסיר את elute ולהעביר לתוך צינור microcentrifuge 1.5 מ"ל נקי.
- לכמת את הבריכה המרוכזת באמצעות פלואורומטר, ולאחר מכן להכין aliquot להמשיך לשלב קשירת המתאם: להכין 700 ננוגרם של המדגם בנפח הסופי 65 μL. שמור על שאר הבריכה ב 4 °C (7 °F) עבור ריצה שנייה כדי להסתיים לאחר הריצה הראשונה הסתיימה.
- המשך עם קשירת מתאם בהתאם להנחיית היצרן וטען את הדגימה בתא הזרימה. התחל את ריצת הרצף.
הערה: שאיפה אוויר ~ 200 μL של מאגר אחסון מיציאת התחלה של תא הזרימה לפני טעינת המדגם. הדבר קריטי עבור תימוי תאי הזרימה המוצלחים וטעינת הדגימה. השתמש בפיפטה p1000 וטיפים בעת ציור והפקדת פתרונות דרך היציאה הראשונית של תא הזרימה.
- רצף את הספריה בהתאם להוראות היצרן.
- פתח את תוכנת ההפעלה לרצף ולחץ על התחל. הזן שם עבור הניסוי, המינוח המומלץ כולל את תאריך ההפעלה ואת שם המשתמש. לחץ על המשך לבחירת ערכות, בחר בערכת ההכנה המתאימה לספריה ובחבילות הרחבה של ברקוד המשמשות ולאחר מכן לחץ על המשך כדי להפעיל אפשרויות.
- התאם את אורך הריצה ל- 48 שעות אם הוא מתכנן להכין ספריה מספקת להפעלה שנייה (אחרת השאר כברירת מחדל 72 שעות). לחץ על המשך להתקשרות בסיסית.
- בדוק את אפשרות basecalling Config: Basecalling מהיר וודא ש- Barcoding מוגדר כזמין כך שקבצי FASTQ פלט יקצצו את רצפי הברקוד ויהפכו לספריות נפרדות המבוססות על ברקוד. לחץ על המשך לפלט.
- בחר היכן לשמור נתוני ריצוף פלט. צפה לכ-30-50 ג'יגה-בתים של נתונים אם רק שמירת פלט FASTQ ו->500 Gb של נתונים אם גם שומרת את פלט FAST5. בטל את הסימון באפשרות הסינון Qscore: 7 | אורך קריאה: לא מסונן אם הוא מתכנן להמשיך בסינון המתואר בסעיף 7.2, השאר אחרת מסומן ולהתאים את אורך הקריאה ל- 200.
- לחץ על המשך להפעיל את תוכנית ההתקנה וסקור את כל ההגדרות. אם ההגדרות נכונות, לחץ על התחל, אחרת לחץ על אחורה ובצע את כל ההתאמות הדרושות.
- במידת הרצון, ניתן לשטוף את תא הזרימה בהתאם להוראות היצרן ולהיטען מחדש עם המאגר הנותר. חזור על השלבים ב- 6.2 עבור המאגר הנותר לאחר השלמת ההפעלה הראשונה ותא הזרימה נשטף.
הערה: בעת הגדרת הריצה השנייה, התאם את מתח ההטיה ל- -250 mV לפי המלצות היצרן לתאי זרימה ששימשו בעבר בריצות מעל 48 שעות.
7. הערכה והכנת קריאות
הערה: מבנה ספריה מומלץ מתואר באיור 4. צור את הספריות הנמצאות בשולחן העבודה, כלומר, Long_Reads, Short_Reads ו- Trimmed_Reads, לפני שתמשיך עם שלבי החישוב שלהלן.
- קריאות קצרות (איור 3)
הערה: קריאות קצרות נוצרות בתבנית FASTQ. הקבצים מכילים 4000 קריאות מרביות לכל FASTQ. לעתים קרובות הם מכווצים (ארכיון .gz) ומאורגנים בקבצים מרובים. בהתאם לפלטפורמה, ברקודים נחתכים בדרך כלל. תוכניות מסוימות מקבלות קבצים בתבנית מכווצת, אחרות עשויות לדרוש את החילוץ שלהם לפני הייבוא. הקריאות חייבות לעבור שלבי בקרת איכות (QC) כדי להבטיח דיוק נתונים במהלך הרכבת הגנום. אם שולחן העבודה של CLC Genomics אינו זמין, תוכניות חלופיות עשויות לשמש לקיצוץ וקריאה קצרה של QC כגון Trimmomatic25 או חתוך גוון בשפע (https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/) לגיזום ו- FastQC (http://www.bioinformatics.babraham.ac.uk/projects/fastqc/) להערכת איכות הקריאה. כיסוי קריאה קצר ממוצע, המוערך על ידי הכפלת מספר הקריאות לפי אורך הקריאה הממוצע וחלוקה לפי גודל הגנום, מומלץ להיות >100x.- פתח את תוכנת שולחן העבודהשלGenomics ( טבלת חומרים ) וייבוא את כל קבצי FASTQ קצרי הקצה. קבצים משויבים ייווצרו באופן אוטומטי.
- צור תיקיה חדשה תחת CLC_Data על-ידי לחיצה על חדש בסרגל הכלים העליון ובחירה בתיקיה... כדי לאחסן את הקבצים. תן שם לתיקיה כרצונך, מוסכמה מומלצת משתמשת במזהה לדוגמה. שמור את כל הפלט מהשלבים הבאים לתיקיה זו.
- בסרגל הכלים העליון, לחץ על לחצן ייבוא ובחר אילומינה... נווט אל כל הקבצים שנקראו קצרים ובחרו התואמים לדוגמה. ודא שהאפשרות קריאות משויפות נבחרה ובטל את הסימון באפשרות הסר קריאות שנכשלו. לחץ על הבא, בחר שמורולחץ שוב על הבא. בחר לשמור את הקבצים המיובאים בתיקיה החדשה שנוצרה בשלב הקודם ולחץ על סיום.
- צור רשימת רצף של כל הקבצים המשויים עבור הבידוד; פעולה זו תצרר נתוני קריאה לקובץ יחיד לפשטות הניתוח.
- בסרגל הכלים העליון, לחץ על לחצן חדש ובחר רשימת רצף... ברשימת הספריות מימין, בחר את הקבצים שיש לערוץ והשתמש בחצים כדי להעביר אותם לרשימת הקבצים שנבחרו מימין. לחץ על הבא, בחר שמורולחץ שוב על הבא. בחר לשמור את רשימת הרצף ולחץ על סיום.
- לאחר יצירת רשימת הרצף, שנה את שמה באופן מיידי למזהה לדוגמה.
- הפעל את הכלי QC לקריאות רצף ברשימת הרצף: הליך זה יעריך את פרמטרי האיכות הכוללים של הקריאות שנוצרו על-ידי NGS לקריאה קצרה.
- חפש את הכלי QC לרצף קריאות בתפריט ארגז הכלים (חלון מימין למטה). לחץ פעמיים על הכלי ולאחר מכן בחר את רשימת הרצף שיש לנתח ולחץ על הבא.
- ודא שכל אפשרויות הפלט מסומנות ובחר שמור תחת טיפול בתוצאות. לחץ על הבא וציין כדי לשמור את קבצי הפלט ולאחר מכן לחץ על סיום.
- הפעל את הכלי Trim Reads ברשימת הרצף: החיתוך ייעשה בהתבסס על איכות, אורך ואי בהירות. תהליך זה מניח שהברקודים המשמשים לרצף קוצצו לפני שלב זה.
- חפשו את הכלי חיתוך קריאות בארגז הכלים (חלון מימין למטה). לחץ פעמייםעל חיתוך קריאות ולאחר מכן בחר את רשימת הרצף שיש לנתח ולחץ על הבא.
- חיתוך איכות: הגדר את מגבלת ציון האיכות ל- 0.01 והותיר נוקלאוטידים מעורפלים ב- 2. לחץ על הבא.
הערה: ניתן להתאים פרמטרים לפי שיקול דעת המשתמש; אלה ההגדרות המומלצות. - בטל את הסימון של חיתוך מתאם קריאה אוטומטי (בצע זאת רק אם מתאמים נחתכו מהקריאות לפני הייבוא ל- CLC). לחץ על הבא וסמן את מחק קריאות מתחת לאורך, השתמש בברירת המחדל 15.
- לחץ על הבא, סמן יצירת דוחולאחר מכן בחר שמור. לחץ על הבא וציין היכן לשמור את קבצי הפלט. לחץ על סיום.
- ייצוא רשימת הרצף החתוך: ההרכבה והניתוח ההיברידיים הבאים יושלמו מחוץ ל- CLC ודורשים ייצוא של קבצים קצוצים לקריאה קצרה.
- מתוך ניווט הספריה מימין למעלה, בחר את הקובץ החתוך שנוצר בשלב 7.1.4 ולאחר מכן לחץ על ייצוא בסרגל הכלים העליון. בחר Fastq עבור סוג קובץ הייצוא ולחץ על הבא. בדוק יצא רשימת רצף מזווג לשני קבצים. לאחר מכן, לחץ על הבא ובחר את הספריה Trimmed_Reads לייצוא הקבצים. לחץ על סיום. ודא שהקבצים החתוך לקריאה קצרה יוצאו בהצלחה כשני קבצים (R1 ו- R2) עם הסיומת .fastq.
הערה: יש לייצא את רשימת הרצף החתוך לשני קבצים, המוגדרים בדרך כלל על-ידי CLC כ- R1 ו- R2. הדבר קריטי מכיוון שהרכבה היברידית במורד הזרם דורשת להגדיר קלט נתונים לקריאה קצרה ככזה. - שנה את שמות הקבצים המיוצאים, נא להימנע משימוש ברווחים ובתווים מיוחדים בשמות קבצים. לפשטות trimmed_short_file תבנית מומלצת. אר-1.פאסטק.
- מתוך ניווט הספריה מימין למעלה, בחר את הקובץ החתוך שנוצר בשלב 7.1.4 ולאחר מכן לחץ על ייצוא בסרגל הכלים העליון. בחר Fastq עבור סוג קובץ הייצוא ולחץ על הבא. בדוק יצא רשימת רצף מזווג לשני קבצים. לאחר מכן, לחץ על הבא ובחר את הספריה Trimmed_Reads לייצוא הקבצים. לחץ על סיום. ודא שהקבצים החתוך לקריאה קצרה יוצאו בהצלחה כשני קבצים (R1 ו- R2) עם הסיומת .fastq.
- פתח את תוכנת שולחן העבודהשלGenomics ( טבלת חומרים ) וייבוא את כל קבצי FASTQ קצרי הקצה. קבצים משויבים ייווצרו באופן אוטומטי.
- קריאות ארוכות (MinION) (איור 3)
הערה: הצינור הבא להכנת קריאות רצף ארוכות (MinION) להרכבה היברידית משתמש בתוכניות NanoFilt ו- Nanostat26 המבוצעות על-ידי שורת הפקודה. התקן את הכלים לפני שתמשיך והכר את היסודות של UNIX כדי לבצע פקודות אלה. מומלץ למסופי ברירת מחדל ולמעטפת Bash. מדריך שיעורים עבור פקודות מסוף נפוצות ושימוש נמצא ב- Software Carpentry27. ההוראות שלהלן מניחות כי הקבצים שנוצרו ייקראו עם המינוח של הברקוד (NB01, NB02 וכו ') ונשמרו בספריה Long_Reads. לחלופין, ניתן לבצע סינון קריאה באמצעות MinKNOW בעת הגדרת הפעלת הרצף. כיסוי קריאה ארוך ממוצע מומלץ להיות >100x. אורך הקריאה הממוצע המומלץ הוא >2000 bp; לכן, מספר הקריאות הארוכות הדרושות נמוך ממספר הקריאות הקצרות.- צור ספריות חדשות עבור כל ברקוד המשמש בהפעלה (ברקוד01, ברקוד02 וכו') בתוך הספריה Long_Reads(איור 4). העתק את כל קבצי ה- .fastq התואמים לכל ברקוד לתיקיה המתאימה. שלב את כל קבצי .fastq עבור כל ברקוד מכל ריצה.
- פתח את Terminal ונווט אל ספריות הברקוד בספריה Long_Reads באמצעות הפקודה cd: cd Desktop/Long_Reads/ברקוד01
- לכנס את כל קבצי .fastq לכל ברקוד לקובץ .fastq יחיד על ידי ביצוע הפקודה הבאה: cat *.fastq > NB01.fastq
הערה: פקודה זו משלבת את כל הקריאות מכל אחד מקבצי FASTQ ל- FASTQ אחד גדול ובודד בשם NB01.fastq. - השתמש NanoStat כדי להעריך את איכות הקריאה של המדגם על ידי ביצוע הפקודה הבאה: NanoStat --fastq NB01.fastq
- הקלט את התוצאות על-ידי העתקת הפלט לקובץ טקסט או Word לעיון עתידי.
- השתמש NanoFilt כדי לסנן MinION קורא ביטול קריאות עם Q < 7 ואורך < 200 על ידי ביצוע הפקודה: NanoFilt -q 7 -l 200 bp NB01.fastq | gzip > NB01 _trimmed.fastq.gz
- הפעל NanoStat על הקובץ החתוך שנוצר בשלב 7.2.6 על ידי ביצוע הפקודה: NanoStat -fastq NB01 _trimmed.fastq.gz
- הקלט את התוצאות על-ידי העתקת הפלט לקובץ טקסט או Word והשווה לתוצאות שלב 7.2.4 כדי להבטיח שהסינון הצליח (טבלה 1).
- חזור על שלבים 7.2.2 עד 7.2.8 עבור כל ברקוד המשמש בריצת הרצף.
הערה: הקובץ NB01_trimmed.fastq.gz שנוצר בשלב 7.2.6 ישמש להרכבה היברידית.
8. יצירת הרכבה גנומית היברידית
הערה: צינור ההרכבה הבא משתמש ב- Unicycler19,28,29,30 כדי לשלב קריאות קצרות וארוכות שהוכנו בסעיפים 7.1 ו- 7.2 ( איור3). התקן את Unicycler ואת יחסי התלות שלו ובצע את הפקודות שלהלן. מניחים שקבצים הנקראים זמן קצר שיוצאו בשלב 7.1.5 נקראים trimmed_short_file. אר-1.פאסטק trimmed_short_file. R2.fastq לפשטות.
- ארגן את הקבצים שנקראו זמן קצר ואת הקבצים שנקראו זמן רב בספריה אחת בשם Trimmed_Reads. הספריה חייבת להכיל את הדברים הבאים:
- קובץ .fastq.gz עבור קריאות ארוכות חתוכות (נוצר בשלב 7.2.6).
- שני קבצי .fastq (R1 ו- R2) לקריאות קצרות חתוכות (שנוצרו בשלב 7.1.5).
- נווט אל הספריה Trimmed_Reads המאחסנת את קבצי הקריאה באמצעות הפקודה cd בטרמינל: cd Desktop/Trimmed_Reads
- פעם בספריה הנכונה, תסגור את שני קבצי הקריאה הקצרים כך שהם גם בתבנית .fastq.gz על-ידי ביצוע הפקודה הבאה: gzip trimmed_short_file. R1.fastq
- חזור על שלב 8.2 הן עבור R1 והן עבור R2. ודא שכל קבצי הקריאה נמצאים כעת בתבנית .fastq.gz וודא שכל הקבצים תואמים לאותו בידוד.
- התחל את ההרכבה ההיברידית באמצעות חד-ציקלילר על-ידי הפעלת הפקודה הבאה:
חד-קליט -1 trimmed_short_file. R1.fastq.gz -2 trimmed_short_file. R2.fastq.gz -l NB01 _trimmed.fastq.gz -o unicycler_output_directory
הערה: -o מציין את הספריה שבה תישמר פלט יוניציקלר, Unicycler ייצור ספריה זו לאחר ביצוע הפקודה; אל תיצור את הספריה מראש. זמן הריצה משתנה בהתאם לעוצמה החישובית של המחשב המשמש, כמו גם גודל הגנום ומספר הקריאות. פעולה זו עשויה להימשך בין השעות 4 שעות ל-1 או 2 ימים. פרוטוקול זה בוצע על מכונת CentOS Linux 7 עם זיכרון RAM של 250 ג'יגה בייט, מעבד Intel Xeon (R) עם ליבות מעשיות של 2.5 ג'יגה-הרץ ו-48 ליבות וירטואליות. לחלופין, מחשבים אישיים עם זיכרון RAM של 16 ג'יגה-בתים ומעבדי 6 ליבות ב- 2.6 GHz יכולים לחשב הרכבות אלה בזמן עיבוד ארוך יותר. - לאחר השלמת ההפעלה, סקור את קובץ .log unicycler כדי להבטיח שאין שגיאות - הקלט את המספר, הגודל והמצב (שלם, לא שלם) של ההסתבפויות שנוצרו.
- אם מזוהים קונטיגים לא שלמים (מסומנים כלא שלמים ביומן יוניציקלר), הפעל מחדש את Unicycler במצב מודגש על-ידי הוספת הדגל הבא לפקודה בשלב 8.4: --mode מודגש.
הערה: מצב מודגש יוריד את סף האיכות המקובל עבור גשרי קריאה ארוכה במהלך ההרכבה; זה עשוי להניב הרכבה מלאה, אבל איכות ההרכבה עשויה להיות מופחתת. מומלץ להשתמש במצב מודגש רק בעת הצורך וכראיה ראשונית להצטרפות קונטיג שיאושרו מאוחר יותר על ידי PCR.
- אם מזוהים קונטיגים לא שלמים (מסומנים כלא שלמים ביומן יוניציקלר), הפעל מחדש את Unicycler במצב מודגש על-ידי הוספת הדגל הבא לפקודה בשלב 8.4: --mode מודגש.
9. הערכת איכות ההרכבה
הערה: הפרוטוקול הבא משתמש בתחבושת31 וב- QUAST32, שתי תוכניות שיש להגדיר לפני השימוש (איור 2 ואיור 4). התחבושת אינה דורשת התקנה לאחר ההורדה ו- QUAST דורש היכרות עם שימוש בסיסי בשורת הפקודה. מומלץ גם להעריך את שלמות הגנום באמצעות אורתולוגיות אוניברסליות בעלות העתק יחיד (BUSCO)33.
- תחבושת: לחץ על קובץ. לאחר מכן, בחר טען גרף ובחר את הקובץ assembly.gfa שנשמר unicycler_output_directory שנוצר על-ידי Unicycler בשלב 8.4. לאחר טעינה, לחץ על לחצן צייר גרף בסרגל הכלים השמאלי ובדוק כיצד הקונטיגים (הנקראים צמתים) מחוברים ומאורגנים כדי להעריך אם ההרכבה הושלמה (איור 5).
הערה: הרכבות שלמות מיוצגות על-ידי קונטיגסים מעגליים בודדים המקושרים בשני קצותיה (איור 5A,B). להרכבות שלא הושלמו יש קונטיגים מרובים המקושרים זה לזה או שהם ליניאריים(איור 5C). קונטיגס ליניארי קטן לא יכול להיות שלם כפי שהם עשויים להצביע על אלמנטים חוץ-כרומוזומאליים ליניאריים. כיסוי, המכונה גם עומק, יצוין בתחבושת ומייצג את השפע היחסי של הרצף לכרומוזום, מנורמל ב Unicycler ל 1x. - QUAST
- בתוך המסוף, נווט לתיקיה המאחסנת את פלט Unicycler באמצעות הפקודה cd: cd Desktop/Trimmed_Reads/unicycler_output_directory
הערה: רווחים אינם מותרים בנתיב שבו ממוקמת ההרכבה, כלומר, אין ספריות המובילות לפלט יוניציקלר שיכולות לכלול רווחים על שמם. לחלופין, העתק את הקובץ assembly.fasta לשולחן העבודה לקבלת גישה נוחה. - הפעל את QUAST על-ידי ביצוע הפקודה הבאה: assembly quast.fasta -o quast_output_directory
- סקור את הדוחות שנוצרו על-ידי QUAST בספריה quast_output_directory.
- בתוך המסוף, נווט לתיקיה המאחסנת את פלט Unicycler באמצעות הפקודה cd: cd Desktop/Trimmed_Reads/unicycler_output_directory
10. ביאור גנום
הערה: צינור הביאורים שלהלן משתמש ב- Prokka34, כלי שורת פקודה שיש להתקין לפני השימוש. לחלופין, השתמש Prokka דרך אוטומטי GUI K-Base(טבלת חומרים)או גנומים ביאורים באמצעות שרת האינטרנט RAST35. אם הפקדת גנומים לתוך NCBI, הם יירו באופן אוטומטי באמצעות צינור ביאור הגנום הפרוקריוטי (PGAP)36.
- נווט בתוך המסוף לתיקיה המאחסנת את פלט יוניציקלר באמצעות הפקודה cd (ראה שלב 9.2.1). לאחר מכן, הפעל את Prokka על ידי ביצוע הפקודה הבאה: prokka -קידומת sample_ID -להקדים את prokka_output_directory ההרכבה.fasta
הערה: --קידומת תציין את כל קבצי הפלט בהתבסס על sample_ID שצוין. --outdir ייצור ספריית פלט בשם שצוין שבו יישמרו כל קבצי הפלט של Prokka; אל תיצור ספריית פלט עבור Prokka מראש. - סקור את הביאורים על-ידי פתיחת טבלת ה- .tsv ו/או על-ידי העלאת קובץ ה- .gff שנוצר לתוכנת ניתוח רצף כדי להציג באופן חזותי ולנתח את הביאורים (איור 6).
- סוגים מסוימים של ביאורים יכולים להיווצר בהתאם לגורמים גנטיים של עניין. מומלץ להתחיל עם הכלים הידידותיים למשתמש על המרכז לאפידמיולוגיה גנומית (www.genomicepidemiology.org/) שרת אינטרנט לניתוח ראשוני37,38,39,40,41. כלים נוספים לזיהוי מערכות CRISPR-cas ופרואפאז' זמינים (איור 3)42,43.
11. שיטות מומלצות לדמוקרטיזציה של נתונים
- במידת האפשר, הפקד את כל נתוני הקריאה הגולמיים, כמו גם גנומים שהורכבו במאגר ציבורי כגון ארכיון הקריאה של רצף NCBI (SRA) וג'נבנק. הגנומים מובאים באופן אוטומטי באמצעות צינור PGAP במהלך תהליך התצהיר של NCBI.
תוצאות
פרוטוקול זה עבר אופטימיזציה לתרבות ולרצף של חיידקי שתן השייכים לגנים המפורטים באיור 1. לא כל חיידקי השתן ניתנים לתחכים בשיטה זו. אמצעי התקשורת והתנאים התרבותיים מצוינים על-ידי הסוג באיור 1. הערכות אלקטרופורזה של ג'ל למופת של שלמות gDNA מתוארות באיור 2. סקירה כללית של צינור הביו-ינפורמטיקה לעיבוד קריאה, הרכבת גנום וביאור מתוארת באיור 3. מדריך למבנה מדריך למדריך חישובי מסופק באיור 4 כדי לפשט את הבנת הפרוטוקול ולספק מסגרת לארגון מוצלח. יתר על כן, כלולים גנומים שלמים מייצגים של שני Klebsiella spp., K. דלקת ריאות ו K. oxytoca, שנוצרו על ידי פרוטוקול זה. ייצוג של מכלולים אלה ניתן באיור 5 וכולל גם דוגמה חלקית נוספת לגנום ק. דלקת ריאות. סקירה מפורטת של כל גנום מלא עם ביאור מלא מוצגת באיור 6. לבסוף, סיכום של סטטיסטיקת קריאה רצף מסופק בטבלה 1 כדי להציע הבנה רחבה של נתונים גולמיים וגזוזים מספיקים ליצירת מכלולי גנום סגור באיכות גבוהה. בנוסף, הפרמטרים העיקריים של שני הנציגים משלימים את Klebsiella spp. הגנום רשום. גנומים ונתונים גולמיים הופקדו בג'בנק תחת ה- BioProject PRJNA683049.
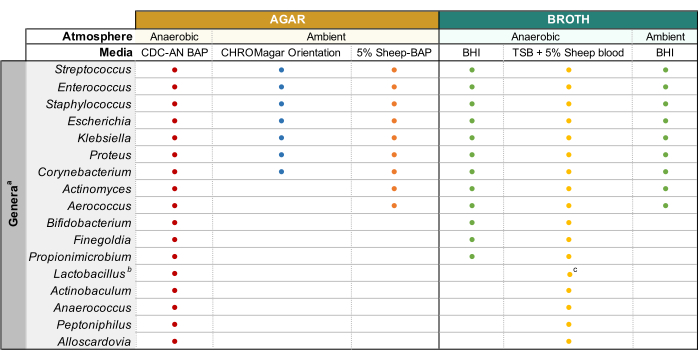
איור 1: תרבית שתן משופרת שונה של סוגים שונים בדרכי השתן. תרשים עבור אגר מרק נוזלי שעשוי לשמש תרבות ג'נדרה בדרכי השתן מגוונת. כל culturing מוצע להתבצע ב 35 °C (5 °F) כמתואר בסעיבה 1.1. עיגולים מייצגים מדיה המתאימה לכת סוג מסוים, צבעים נבחרו באופן שרירותי כדי להבחין בין סוג מדיה אחד למשנהו. CDC-AN BAP (אדום), אגר דם כבשים Anaerobe CDC; 5% כבשים-BAP (כתום), אגר דם כבשים; BHI (ירוק), עירוי לב המוח; TSB (צהוב), ציר סויה טריפטי; אוריינטציה CHROMagar (כחול). גרדנרלה וגינליס צריך להיות תרבותי על HBT Bilayer G. אגר סלקטיבי נרתיקי באווירה מיקרואורופילית ותחת דרישות תרבות מרק מיוחדת44. bלקטובצילוס iners צריך להיות תרבותי על 5% צלחות ארנב-BAP ומרק NYCIII באטמוספירה מיקרואורופילית. cלקטובצילוס spp. עשוי להיות תרבותי על MRS בתנאים מיקרואורופיליים. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
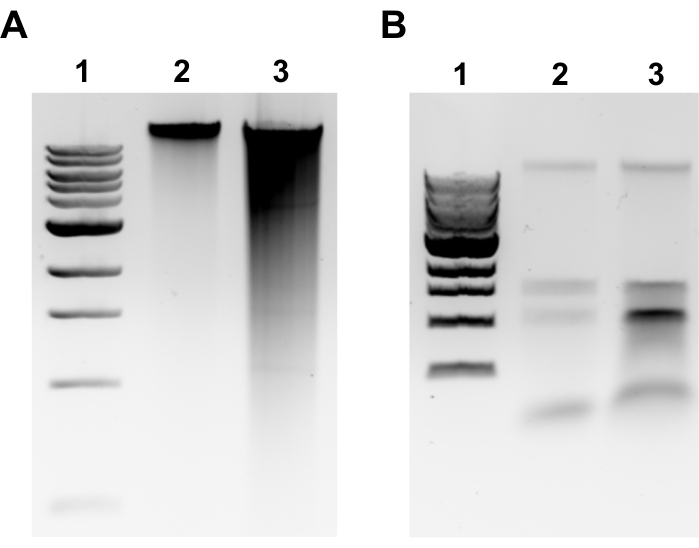
איור 2: תמונות ג'ל אגרוז של מיצוי דנ"א גנומי. תמונות ג'ל מייצגות המתארות את תוצאות החילוץ של gDNA. (A)מסלול 1: 1 kb סולם, נתיב 2: gDNA שלם המייצג חילוץ מוצלח, נתיב 3: מריחה המציינת gDNA מקוטע. (B)מסלול 1: סולם 1 kb, נתיבים 2 ו - 3: זיהום rRNA מסומן על ידי שתי רצועות בין 1.5 kb ו 3 kb. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
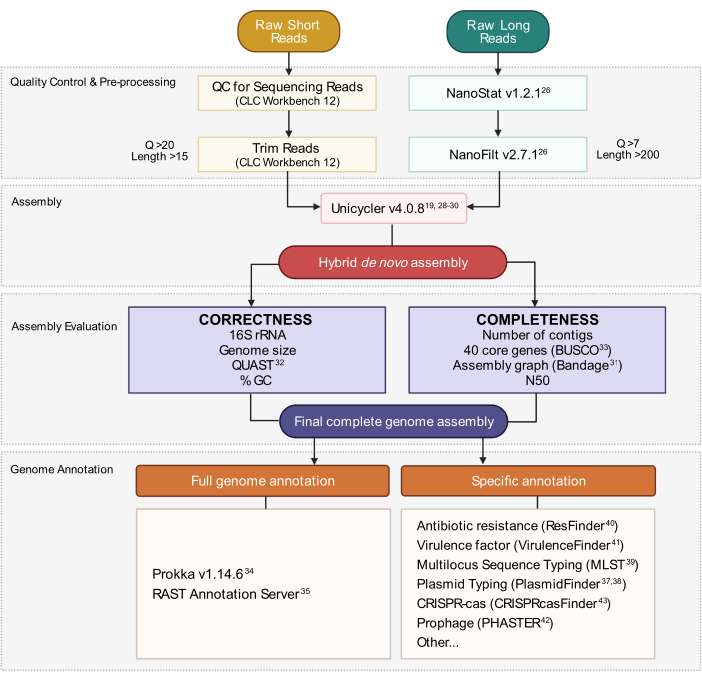
איור 3: זרימת עבודה של הרכבת גנום היברידי. סכמטי של שלבים מקריאת בקרת איכות ועיבוד מקדים לבאור הרכבה. חיתוך קריאה מסיר קריאות מעורפלות ואיכותיות. פרמטרי ציון Q ואורך מסומנים ומייצגים את הקריאות שנשמרות. הרכבה משתמשת הן בקריאה קצרה והן ארוכה כדי ליצור הרכבה גנום היברידי דה נובו. איכות ההרכבה מוערכת בהתבסס על שלמות ונכונות באמצעות כלים ופרמטרים שצוינו. הרכב הגנום הסופי הוא ביאור לכל הגנים וללוק העניין הספציפי. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
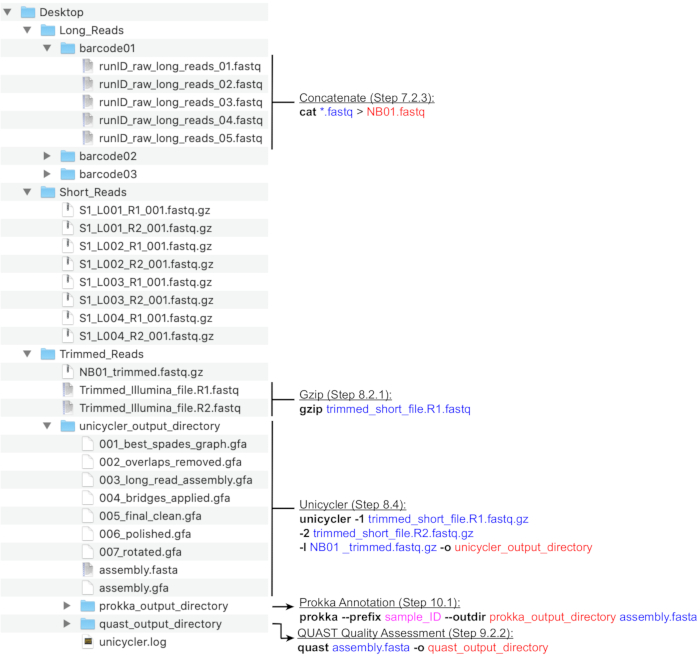
איור 4: מדריך מבנה מדריך לספריות ביו-ינפורמטיקה. סכמטי של ארגון הספריות והקבצים המומלצים לעיבוד קריאות קצרות וארוכות, הרכבה היברידית וביאור גנום ו- QC. שלבי עיבוד נתונים של שורת הפקודה מסומנים לצד קבצים וספריות תואמים. יצירת פקודות ודגלים (מודגשים), קבצי קלט (כחול), קבצי פלט או ספריות (אדום), קלט משתמש כגון מוסכמה למתן שמות לקבצים (magenta). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
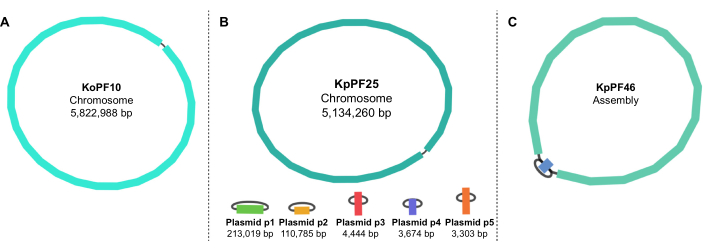
איור 5: גרפי הרכבה של הגנום לפי תחבושת. גרפי הרכבה של גנום של (A) Klebsiella oxytoca KoPF10 ו - (B) Klebsiella דלקת ריאות KpPF25 והרכבה גנומית לא שלמה של (C) קלבסילה דלקת ריאות KpPF46. הגנום המלא של KoPF10 מדגים כרומוזום סגור יחיד ואת הגנום המלא של KpPF25 מורכב כרומוזום סגור וחמישה plasmids סגור. הכרומוזום הלא שלם של KpPF46 מורכב משני קונטיגים מחוברים. הרכבה היברידית חד-icycler de novo יוצרת גרף הרכבה הממחישה על ידי תחבושת. גרף ההרכבה מספק סכמטי פשטני של הגנום, המציין כרומוזום סגור או פלסמידים על ידי מקשר המחבר שני קצוות של קונטיליג יחיד. נוכחותו של יותר מ- contig אחד מחובר מצביעה על הרכבה לא שלמה. גודל ועומק Contig ניתן לציין גם תחבושת. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
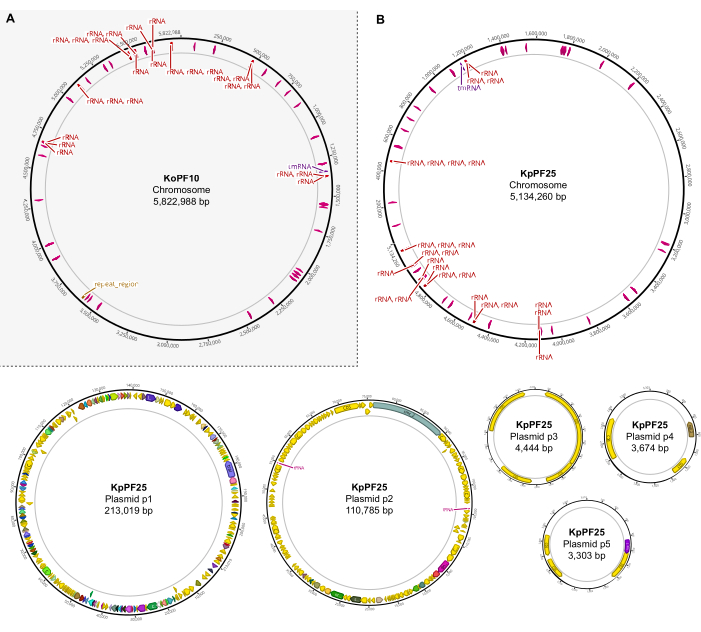
איור 6: מפות גנום שלמות של מכלולים היברידיים מובאים. מפות הרכבה שנוצרו על ידי Geneious Prime עבור הגנום המלא של (A) K. oxytoca KoPF10 ו - (B) K. דלקת ריאות KpPF25 המציגות גנים מובאים המסוינים על ידי חצים צבעוניים לאורך עמוד השדרה של הפלסמיד. כרומוזומים מראים רק גנים rRNA ו- tRNA לפשטות. ביאורים גנומיים בוצעו באמצעות Prokka כפי שצוין בסעיף 10 של פרוטוקול זה. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
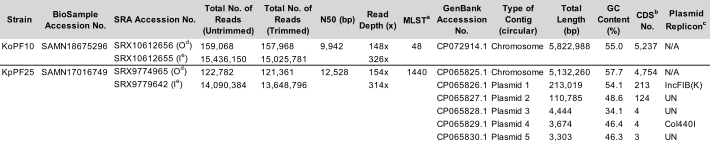
טבלה 1: נציג Klebsiella spp. מאפייני הרכבה מלאה. פרמטרי ההרכבה של זן K. oxytoca KoPF10 ו- K. דלקת ריאות זן KpPF25. מספרי גישה עבור הנתונים שהופקדו על NCBI מסופקים. מספר הקריאות לפני ואחרי החיתוך מצוינות עבור שתי טכנולוגיות הריצוף. N50 מסופק לקריאות ארוכות רק מאז קריאות קצרות הן באורך מבוקר. Replicon Plasmid חזה באמצעות PlasmidFinder v2.1 Enteroebacteriaceae מסד נתונים עם פרמטרים להגדיר 80% זהות ו 60% אורך. סוג רצף MLST, מולטילוקוס. b תקליטורים, רצפי קידוד. c Replicon Plasmid חזה באמצעות PlasmidFinder v2.1 Enterobacteriaceae מסד נתונים עם פרמטרים להגדיר 80% זהות ו 60% אורך. ד אוקספורד ננופור טכנולוגיות (ONT) הפקידו נתוני קריאה. e Illumina הפקידה נתוני קריאה. נא לחץ כאן כדי להוריד טבלה זו.
Discussion
פרוטוקול הרכבת הגנום ההיברידי המקיף המתואר כאן מציע גישה יעילה לפולחן מוצלח של מיקרוביוטה ואורופתוגנים מגוונים בדרכי השתן, והרכבה מלאה של הגנום שלהם. WGS מוצלח של גנומים חיידקיים מתחיל בבידוד של חיידקים מגוונים ולעיתים בררניים על מנת לחלץ את הדנ"א הגנומי שלהם. עד כה, פרוטוקולי תרבית השתן הקיימים או חסר את הרגישות הדרושה כדי לזהות מינים רבים בדרכי השתן או לערב גישות ארוכות ונרחבות הדורשות זמן ומשאבים ארוכים11. הגישה של תרבית השתן המשופרת שהשתנתה המתוארת מציעה פרוטוקול פשוט אך מקיף לבידוד מוצלח של חיידקים השייכים ל -17 סוגים נפוצים בדרכי השתן, כולל מינים פתוגניים או מועילים, הן חיידקים אירוביים או אנאירוביים מחייבים. זה בתורו מספק את החומר ההתחלתי הדרוש לרצף והרכבה מדויקים של גנומים חיידקיים ולניסויים פנוטיפיים קריטיים, התורמים להבנת בריאות השתן ומחלות. יתר על כן, גישה זו של תרבות שונה מספקת אבחנה קלינית מוגדרת יותר של מיקרואורגניזמים קיימא שנמצאו בדגימות שתן ומאפשרת את הבנקאות הביולוגית שלהם למחקרים גנומיים עתידיים. עם זאת, פרוטוקול זה אינו ללא מגבלות. זה עשוי לדרוש זמני דגירה ארוכים בהתאם לאורגניזם, כמו גם שימוש במשאבים כגון תא היפוקסיה או אינקובטורים מבוקרים כי לא יכול להיות זמין. השימוש GasPaks אנאירובי מציע פתרון חלופי אבל אלה הם יקרים ולא תמיד לייצר סביבה מתמשכת ומבוקרת. לבסוף, הטיית תרבות כמו גם מגוון מדגם עשוי לאפשר אורגניזמים מסוימים אורופטוגנים כדי להאיץ חיידקים בררניים. למרות מגבלות אלה, תרבות של חיידקי שתן מגוונים מתאפשרת על ידי גישה זו.
רצף גנומי צבר פופולריות עם קידום טכנולוגיות רצף הדור הבא אשר הגדילו מאוד הן את התשואה והן את הדיוק של ריצוף נתונים14,15. יחד עם פיתוח אלגוריתמים לעיבוד נתונים והרכבה דה נובו, רצפי גנום שלמים נמצאים בקצות אצבעותיהם של טירונים ומדענים מומחים כאחד15,45. הידע של ארגון הגנום הכולל המסופק על ידי גנומים שלמים מציע תובנות אבולוציוניות וביולוגיות חשובות, כולל שכפול גנים, אובדן גנים והעברת גנים אופקית14. בנוסף, גנים החשובים להתנגדות מיקרוביאלית ולוויקולנטיות ממוקמים לעתים קרובות באלמנטים ניידים, אשר בדרך כלל אינם נפתרים בהרכבי גנום טיוטה15,16.
הפרוטוקול כאן עוקב אחר גישה היברידית לשילוב של נתוני רצף מפלטפורמות לקריאה קצרה וקריאה ארוכה כדי ליצור מכלולי גנום שלמים. בעוד התמקדות בגנומים חיידקיים בדרכי השתן, הליך זה עשוי להיות מותאם חיידקים מגוונים ממקורות בידוד שונים. צעדים קריטיים בגישה זו כוללים ביצוע טכניקה סטרילית נאותה וניצול תנאי מדיה ותרבות מתאימים לבידוד של חיידקי שתן טהורים. יתר על כן, החילוץ של gDNA שלם, תשואה גבוהה הוא חיוני ליצירת נתונים רצף ללא קריאות מזהמות שעלולות לעכב את הצלחת ההרכבה. פרוטוקולי הכנת הספרייה הבאים הם קריטיים ליצירת קריאות איכות באורך ועומק מספיקים. לכן, יש חשיבות מוחלטת לטפל ב- gDNA בזהירות במהלך הכנת הספרייה לרצף ארוך במיוחד, שכן היתרון הגדול ביותר של טכנולוגיה זו הוא יצירת קריאות ארוכות ללא הגבלת אורך עליון תיאורטית. כמו כן מפורטים מקטעים לבקרת האיכות המתאימה (QC) של קריאות רצף שמבטלות נתונים רועשים ומשפרות את תוצאת ההרכבה.
למרות בידוד DNA מוצלח, הכנת הספרייה, ורצף, הטבע של ארכיטקטורה גנומית של מינים מסוימים עדיין עשוי להוות מכשול ליצירת הרכבה גנומית סגורה45,46. רצפים חוזרים מסבכים לעתים קרובות את חישוב ההרכבה ולמרות נתוני קריאה ארוכים, אזורים אלה עשויים להיפתר בביטחון נמוך, או כלל לא. לפיכך, קריאות ארוכות צריכות להיות ארוכות בממוצע יותר מהאזור החוזר הגדול ביותר בגנום או הכיסוי חייב להיות גבוה (>100x)19. גנומים מסוימים עשויים להישאר שלמים ודורשים גישות ידניות להשלמה. עם זאת, גנומים היברידיים מורכבים בדרך כלל מפחות קונטיגים מאשר גנומים טיוטה קצרי קריאה. התאמת פרמטרי ברירת המחדל של אלגוריתם ההרכבה או ביצוע ניתוקים מחמירים יותר עבור קריאת QC עשוי לעזור. לחלופין, גישה מוצעת אחת היא למפות קריאות ארוכות לאזורים הלא שלמים בחיפוש אחר ראיות לנתיב ההרכבה הסביר ביותר, ולאחר מכן לאשר את הנתיב המשתמש ברצף PCR וסנגר של האזור המוגבר. מיפוי קריאות באמצעות Minimap2 מוצע ותחבושת מציעה כלי שימושי להדמיה של קריאות ממופות לאורך קונטיגים מורכבים המספקים ראיות לחיבור contig47.
אתגר נוסף ליצירת גנומים שלמים טמון בהיכרות ובנוחות עם כלי שורת פקודה. כלים ביואינפורמטיים רבים מפותחים כדי להציע הזדמנויות חישוביות לכל משתמש; עם זאת, הניצול שלהם מסתמך על הבנה עם היסודות של יוניקס ותכנות. פרוטוקול זה נועד לספק הוראות מפורטות מספיק כדי לאפשר לאנשים ללא ניסיון קודם בשורת הפקודה ליצור מכלולי גנום סגורים ולהביא להם ביאורים.
Disclosures
למחברים אין מה לחשוף.
Acknowledgements
אנו מודים לד"ר מוטוזה ג'ובידה איסלאם וד"ר לוק ג'ויס על תרומתם לפרוטוקול זה. ברצוננו גם להכיר באוניברסיטת טקסס במרכז הגנום של דאלאס עבור המשוב והתמיכה שלהם. עבודה זו מומנה על ידי קרן וולש, מספר פרס AT-2030-20200401 ל- N.J.D., על ידי המכונים הלאומיים לבריאות, מספר הפרס R01AI116610 ל- K.P., ועל ידי יו"ר פלסיה וג'ון קיין בבריאות האישה, המוחזק על ידי P.E.Z.
Materials
| Name | Company | Catalog Number | Comments |
| Equipment: | |||
| Bioanalyzer 2100 | Agilent | G29398A | Optional but recommended |
| Centrifuge | Eppendorf | -- | Any centrifuge for spinning conicals and microcentrifuge tubes (e.g. Models 5810R/5424R) |
| Electrophoresis | BioRad Laboratories | 1645070 | |
| Gel Imaging System | BioRad Laboratories | ChemiDoc models | |
| Incubator | ThermoFisher Scientific | -- | Any CO2 Incubator (e.g. Thermo Forma model 3110) |
| Magnetic Rack | New England BioLabs | S15095 | 12-tube rack |
| MinION | Oxford Nanopore Technologies | -- | |
| Nanodrop | ThermoFisher Scientific | ND-ONE-W | |
| NextSeq 500 | Illumina | SY-415-1002 | Other Illumina models are acceptable |
| Plate Reader | BioTek | -- | Synergy H1 |
| Qubit fluorometer | ThermoFisher Scientific | Q33238 | |
| Rotator | Benchmark Scientific | H2024 | |
| Thermocycler | ThermoFisher Scientific | -- | Any thermocycler for PCR reactions (e.g. ProFlex PCR system) |
| Materials: | |||
| 10X Phosphate Buffered Saline (PBS) | Fisher Scientific | BP3991 | |
| 10X TBE buffer | -- | -- | 1M Tris,1M Boric Acid,0.2M EDTA (pH 8.0) |
| 1429R primer | Sigma Aldrich (Custom oligos) | -- | GGTTACCTTGTTACGACTT |
| 1kb Ladder | VWR | 101228-494 | |
| 1M Tris-Cl (pH 7.5) | ThermoFisher Scientific | 15567027 | |
| 6x Loading dye | Fisher Scientific | NC0783588 | |
| 8F primer | Sigma Aldrich (Custom oligos) | -- | AGAGTTTGATCCTGGCTCAG |
| Agar | Fisher Scientific | BP1423-2 | |
| Agarose | BioRad Laboratories | 63001 | |
| AMPure XP Beads | Beckman Coulter | A63880 | |
| Anaerobe Pouch System - GasPak EZ | BD Diagnostic Systems | B260683 | |
| Boric Acid | Fisher Scientific | A73-500 | |
| Brain Heart Infusion Broth | BD Diagnostic Systems | 212304 | |
| CDC Anaerobe 5% Sheep Blood Agar | BD Diagnostic Systems | L007357 | |
| CHROMagar Orientation | BD Diagnostic Systems | PA-257481.04 | |
| DNeasy Blood & Tissue | QIAGEN | 69504 | |
| DreamTaq Master Mix | ThermoFisher Scientific | K1081 | |
| Dry Anaerobic Indicator Strips | BD Diagnostic Systems | 271051 | |
| EDTA | Fisher Scientific | S311-500 | |
| Ethanol 200 Proof | Sigma Aldrich | E7023 | For molecular biology |
| Ethidium Bromide | ThermoFisher Scientific | BP130210 | |
| Flow cell priming kit | Oxford Nanopore Technologies | EXP-FLP002 | |
| Flow cell wash kit | Oxford Nanopore Technologies | EXP-WSH003 | |
| Gel Extraction Miniprep Kit | BioBasic | BS654 | |
| Ligation sequencing kit | Oxford Nanopore Technologies | SQK-LSK109 | |
| Lysozyme | Research Products International Corp | L381005.05 | |
| Mutanolysin | Sigma Aldrich | M9901-5KU | |
| Native barcoding expansion 1-12 | Oxford Nanopore Technologies | EXP-NBD104 | |
| NEB Blunt/TA Ligase Master Mix | New England BioLabs | M0367L | |
| NEBNext FFPE DNA Repair Mix | New England BioLabs | M6630L | |
| NEBNext quick ligation buffer | New England BioLabs | B6058S | |
| NEBNext Ultra II End repair / dA-tailing module | New England BioLabs | E7546L | |
| Nextera DNA CD Indexes | Illumina | 20018708 | |
| Nextera DNA Flex Library Prep - (M) Tagmentation | Illumina | 20018705 | |
| Nuclease-free water | Sigma Aldrich | W4502 | |
| Qubit 1X dsDNA HS Assay Kit | ThermoFisher Scientific | Q33230 | |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| Quick T4 DNA Ligase | New England BioLabs | E6056L | |
| R9 Flow cell | Oxford Nanopore Technologies | FLO-MIN106D | |
| RNase A | ThermoFisher Scientific | EN0531 | |
| Sheep Blood | Hemostat Laboratories | DS13250 | |
| TE buffer | -- | -- | 10mM Tris, 1mM EDTA (pH 8.0) |
| Triton X-100 | Sigma Aldrich | T8787 | |
| Tryptic Soy Broth | BD Diagnostic Systems | 211825 | |
| Software & Bioinformatic Tools: | |||
| Bandage | -- | -- | https://rrwick.github.io/Bandage/ |
| Center for Genomic Epidemiology | -- | -- | http://www.genomicepidemiology.org/ |
| CLC Genomics Workbench 12 | QIAGEN | -- | |
| CRISPRcasFinder | -- | -- | https://crisprcas.i2bc.paris-saclay.fr/ |
| FastQC | -- | -- | https://www.bioinformatics.babraham.ac.uk/projects/fastqc/ |
| Geneious Prime | Geneious | -- | |
| gVolante (BUSCO) | -- | -- | https://gvolante.riken.jp/ |
| Kbase Prokka Wrapper | -- | -- | https://kbase.us/applist/apps/ProkkaAnnotation/annotate_contigs/release |
| Minimap2 | -- | -- | https://github.com/lh3/minimap2 |
| MinKNOW | Oxford Nanopore Technologies | -- | |
| NanoFilt | -- | -- | https://github.com/wdecoster/nanofilt |
| NanoStat | -- | -- | https://github.com/wdecoster/nanostat |
| PHASTER | -- | -- | https://phaster.ca/ |
| Prokka | -- | -- | https://github.com/tseemann/prokka |
| QUAST | -- | -- | http://quast.sourceforge.net/quast |
| Trim Galore | -- | -- | https://www.bioinformatics.babraham.ac.uk/projects/trim_galore/ |
| Trimmomatic | -- | -- | http://www.usadellab.org/cms/?page=trimmomatic |
| Unicycler | -- | -- | https://github.com/rrwick/Unicycler#necessary-read-length |
References
- Brubaker, L., Wolfe, A. The urinary microbiota: a paradigm shift for bladder disorders. Current Opinion in Obstetrics & Gynecology. 28 (5), 407-412 (2016).
- Neugent, M. L., Hulyalkar, N. V., Nguyen, V. H., Zimmern, P. E., De Nisco, N. J. Advances in understanding the human urinary microbiome and its potential role in urinary tract infection. mBio. 11 (2), (2020).
- Klein, R. D., Hultgren, S. J. Urinary tract infections: microbial pathogenesis, host-pathogen interactions and new treatment strategies. Nature Reviews. Microbiology. 18 (4), 211-226 (2020).
- Horsley, H., et al. Enterococcus faecalis subverts and invades the host urothelium in patients with chronic urinary tract infection. PLoS One. 8 (12), 83637 (2013).
- Reitzer, L., Zimmern, P. Rapid growth and metabolism of uropathogenic Escherichia coli in relation to urine composition. Clinical Microbiology Reviews. 33 (1), 00101-00119 (2019).
- Snyder, J. A., et al. Transcriptome of uropathogenic Escherichia coli during urinary tract infection. Infection and Immunity. 72 (11), 6373-6381 (2004).
- Ipe, D. S., Horton, E., Ulett, G. C. The basics of bacteriuria: Strategies of microbes for persistence in urine. Frontiers in Cellular and Infection Microbiology. 6, 14 (2016).
- Babikir, I. H., et al. The impact of cathelicidin, the human antimicrobial peptide LL-37 in urinary tract infections. BMC Infectious Diseases. 18 (1), 17 (2018).
- Jancel, T., Dudas, V. Management of uncomplicated urinary tract infections. The Western Journal of Medicine. 176 (1), 51-55 (2002).
- Ventola, C. L. The antibiotic resistance crisis: part 1: causes and threats. P & T. 40 (4), 277-283 (2015).
- Price, T. K., et al. The clinical urine culture: Enhanced techniques improve detection of clinically relevant microorganisms. Journal of Clinical Microbiology. 54 (5), 1216-1222 (2016).
- Kass, E. H. Asymptomatic infections of the urinary tract. Transactions of the Association of American Physicians. 69, 56-64 (1956).
- Garcia, L. S. . Clinical microbiology procedures handbook. 3rd edn. , (2010).
- Fraser, C. M., Eisen, J. A., Nelson, K. E., Paulsen, I. T., Salzberg, S. L. The value of complete microbial genome sequencing (you get what you pay for). Journal of Bacteriology. 184 (23), 6403-6405 (2002).
- Chen, Z., Erickson, D. L., Meng, J. Benchmarking hybrid assembly approaches for genomic analyses of bacterial pathogens using Illumina and Oxford Nanopore sequencing. BMC Genomics. 21 (1), 631 (2020).
- Greig, D. R., Dallman, T. J., Hopkins, K. L., Jenkins, C. MinION nanopore sequencing identifies the position and structure of bacterial antibiotic resistance determinants in a multidrug-resistant strain of enteroaggregative Escherichia coli. Microbial Genomics. 4 (10), 000213 (2018).
- Carraro, D. M., et al. PCR-assisted contig extension: stepwise strategy for bacterial genome closure. Biotechniques. 34 (3), 626-628 (2003).
- Tettelin, H., Radune, D., Kasif, S., Khouri, H., Salzberg, S. L. Optimized multiplex PCR: efficiently closing a whole-genome shotgun sequencing project. Genomics. 62 (3), 500-507 (1999).
- Wick, R. R., Judd, L. M., Gorrie, C. L., Holt, K. E. Unicycler: Resolving bacterial genome assemblies from short and long sequencing reads. PLoS Computational Biology. 13 (6), 1005595 (2017).
- Singhal, N., Kumar, M., Kanaujia, P. K., Virdi, J. S. MALDI-TOF mass spectrometry: an emerging technology for microbial identification and diagnosis. Frontiers in Microbiology. 6, 791 (2015).
- Turner, S., Pryer, K. M., Miao, V. P., Palmer, J. D. Investigating deep phylogenetic relationships among cyanobacteria and plastids by small subunit rRNA sequence analysis. The Journal of Eukaryotic Microbiology. 46 (4), 327-338 (1999).
- Weisburg, W. G., Barns, S. M., Pelletier, D. A., Lane, D. J. 16S ribosomal DNA amplification for phylogenetic study. Journal of Bacteriology. 173 (2), 697-703 (1991).
- Janda, J. M., Abbott, S. L. 16S rRNA gene sequencing for bacterial identification in the diagnostic laboratory: pluses, perils, and pitfalls. Journal of Clinical Microbiology. 45 (9), 2761-2764 (2007).
- Stevenson, K., McVey, A. F., Clark, I. B. N., Swain, P. S., Pilizota, T. General calibration of microbial growth in microplate readers. Science Reports. 6, 38828 (2016).
- Bolger, A. M., Lohse, M., Usadel, B. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics. 30 (15), 2114-2120 (2014).
- De Coster, W., D'Hert, S., Schultz, D. T., Cruts, M., Van Broeckhoven, C. NanoPack: visualizing and processing long-read sequencing data. Bioinformatics. 34 (15), 2666-2669 (2018).
- Wilson, G., et al. The UNIX Shell. Zenodo. , (2019).
- Bankevich, A., et al. SPAdes: a new genome assembly algorithm and its applications to single-cell sequencing. Journal of Computational Biology. 19 (5), 455-477 (2012).
- Vaser, R., Sovic, I., Nagarajan, N., Sikic, M. Fast and accurate de novo genome assembly from long uncorrected reads. Genome Research. 27 (5), 737-746 (2017).
- Walker, B. J., et al. Pilon: an integrated tool for comprehensive microbial variant detection and genome assembly improvement. PLoS One. 9 (11), 112963 (2014).
- Wick, R. R., Schultz, M. B., Zobel, J., Holt, K. E. Bandage: interactive visualization of de novo genome assemblies. Bioinformatics. 31 (20), 3350-3352 (2015).
- Gurevich, A., Saveliev, V., Vyahhi, N., Tesler, G. QUAST: quality assessment tool for genome assemblies. Bioinformatics. 29 (8), 1072-1075 (2013).
- Simao, F. A., Waterhouse, R. M., Ioannidis, P., Kriventseva, E. V., Zdobnov, E. M. BUSCO: assessing genome assembly and annotation completeness with single-copy orthologs. Bioinformatics. 31 (19), 3210-3212 (2015).
- Seemann, T. Prokka: rapid prokaryotic genome annotation. Bioinformatics. 30 (14), 2068-2069 (2014).
- Aziz, R. K., et al. The RAST server: rapid annotations using subsystems technology. BMC Genomics. 9, 75 (2008).
- Tatusova, T., et al. NCBI prokaryotic genome annotation pipeline. Nucleic Acids Research. 44 (14), 6614-6624 (2016).
- Carattoli, A., Hasman, H. PlasmidFinder and In Silico pMLST: Identification and Typing of Plasmid Replicons in Whole-Genome Sequencing (WGS). Methods in Molecular Biology. 2075, 285-294 (2020).
- Carattoli, A., et al. In silico detection and typing of plasmids using PlasmidFinder and plasmid multilocus sequence typing. Antimicrobial Agents and Chemotherapy. 58 (7), 3895-3903 (2014).
- Larsen, M. V., et al. Multilocus sequence typing of total-genome-sequenced bacteria. Journal of Clinical Microbiology. 50 (4), 1355-1361 (2012).
- Bortolaia, V., et al. ResFinder 4.0 for predictions of phenotypes from genotypes. The Journal of Antimicrobial Chemotherapy. 75 (12), 3491-3500 (2020).
- Joensen, K. G., et al. Real-time whole-genome sequencing for routine typing, surveillance, and outbreak detection of verotoxigenic Escherichia coli. Journal of Clinical Microbiology. 52 (5), 1501-1510 (2014).
- Arndt, D., et al. PHASTER: a better, faster version of the PHAST phage search tool. Nucleic Acids Research. 44 (1), 16-21 (2016).
- Couvin, D., et al. CRISPRCasFinder, an update of CRISRFinder, includes a portable version, enhanced performance and integrates search for Cas proteins. Nucleic Acids Research. 46 (1), 246-251 (2018).
- Totten, P. A., Amsel, R., Hale, J., Piot, P., Holmes, K. K. Selective differential human blood bilayer media for isolation of Gardnerella (Haemophilus) vaginalis. Journal of Clinical Microbiology. 15 (1), 141-147 (1982).
- Nagarajan, N., Pop, M. Sequence assembly demystified. Nat Reviews. Genetics. 14 (3), 157-167 (2013).
- Phillippy, A. M., Schatz, M. C., Pop, M. Genome assembly forensics: finding the elusive mis-assembly. Genome Biology. 9 (3), 55 (2008).
- . Unicycler Wiki Available from: https://github.com/rrwick/Unicycler/wiki (2017)
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved