A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בידוד תאי אנדותל רשתית מורין לריצוף הדור הבא
In This Article
Summary
פרוטוקול זה מתאר שיטה לבידוד תאי אנדותל רשתית לאחר הלידה הממוטבת לתפוקת התאים, טוהר וכדאיות. תאים אלה מתאימים לגישות ריצוף מהדור הבא.
Abstract
השיפורים האחרונים בריצוף הדור הבא קידמו את הידע של חוקרים בביולוגיה מולקולרית ותאית, כאשר מספר מחקרים חשפו פרדיגמות חדשניות בביולוגיה של כלי הדם. יישום שיטות אלה על מודלים של התפתחות כלי הדם דורש אופטימיזציה של טכניקות בידוד תאים מרקמות עובריות ולאחר הלידה. תפוקת התאים, הכדאיות והטוהר צריכים כולם להיות מקסימליים כדי להשיג תוצאות מדויקות וניתנות לשחזור מגישות ריצוף מהדור הבא. מודל כלי הדם ברשתית של עכברים ילודים משמש חוקרים לחקר מנגנונים של התפתחות כלי דם. חוקרים השתמשו במודל זה כדי לחקור מנגנונים של אנגיוגנזה ומפרט גורל עורקי-ורידי במהלך היווצרות כלי דם והבשלתם. יישום טכניקות ריצוף מהדור הבא כדי לחקור את מודל התפתחות כלי הדם ברשתית דורש אופטימיזציה של שיטה לבידוד תאי אנדותל ברשתית שממקסמת את תפוקת התאים, את הכדאיות ואת הטוהר. פרוטוקול זה מתאר שיטה לבידוד, עיכול וטיהור של רקמת רשתית מורין באמצעות מיון תאים המופעל על ידי פלואורסצנציה (FACS). התוצאות מצביעות על כך שאוכלוסיית תאי האנדותל CD31+/CD45 המטוהרת על ידי FACS מועשרת מאוד לביטוי גנים של תאי אנדותל ואינה מציגה שינוי בכדאיות במשך 60 דקות לאחר FACS. כלולות תוצאות מייצגות של גישות ריצוף מהדור הבא על תאי אנדותל שבודדו בשיטה זו, כולל ריצוף RNA בתפזורת וריצוף RNA חד-תאי, המוכיחות כי שיטה זו לבידוד תאי אנדותל ברשתית תואמת ליישומי ריצוף מהדור הבא. שיטה זו של בידוד תאי אנדותל ברשתית תאפשר טכניקות ריצוף מתקדמות כדי לחשוף מנגנונים חדשים של התפתחות כלי הדם.
Introduction
יכולת התפוקה הגבוהה של ריצוף חומצות גרעין באמצעות גישות ריצוף מהדור הבא קידמה מאוד את הידע של החוקרים בביולוגיה מולקולרית ותאית. טכניקות מתקדמות אלה כוללות ריצוף RNA של תעתיק שלם, ריצוף DNA של אזורים ממוקדים לזיהוי פולימורפיזמים של נוקלאוטידים בודדים (SNPs), ריצוף DNA של גורמי שעתוק מאוגדים בריצוף מיצוי חיסוני של כרומטין (ChIP), או אזורי כרומטין פתוחים ב- Assay לריצוף כרומטין נגיש לטרנספוזאז (ATAC), וריצוף RNA חד-תאי1 . בביולוגיה של כלי הדם, התקדמות זו אפשרה לחוקרים להבהיר מנגנונים מורכבים של התפתחות ומחלות, יחד עם הבחנה בין דפוסי ביטוי גנים לאורך רצף של פנוטיפים משתנים 2,3. ניסויים עתידיים יכולים להגדיר מנגנונים מורכבים על ידי שילוב ריצוף הדור הבא עם מודלים מוערכים של התפתחות כלי דם, אך השיטות להכנת דגימות צריכות להיות תואמות לטכניקות הריצוף המתקדמות.
האיכות, הדיוק והשכפול של גישות ריצוף מהדור הבא תלויים בשיטת הכנת הדגימה. בעת בידוד תת-קבוצה של תאים או יצירת תרחיפים חד-תאיים מרקמות, שיטות עיכול וטיהור אופטימליות חיוניות למקסום מספר התא, הכדאיות והטוהר של אוכלוסיית התאים 4,5. זה דורש איזון בשיטת העיכול: עיכול חזק נחוץ כדי לשחרר תאים מהרקמה ולקבל מספיק תאים עבור גישות במורד הזרם, אבל הכדאיות התא תושפע לרעה אם העיכול חזק מדי 6,7. בנוסף, טוהר אוכלוסיית התאים נחוץ לתוצאות חזקות ולניתוח מדויק של נתונים, שניתן להשיג באמצעות FACS. זה מדגיש את החשיבות של אופטימיזציה של שיטות בידוד תאים כדי ליישם ריצוף הדור הבא על מודלים מבוססים של התפתחות כלי הדם.
מודל מאופיין היטב לחקר התפתחות כלי הדם הוא מודל התפתחות כלי הדם ברשתית. כלי הדם ברשתית מורין מתפתחים לאחר הלידה במקלעת שטחית דו-ממדית, עם הנבטה אנגיוגנית ראשונית מעצב הראייה הנראית ביום שלאחר הלידה (P)3, חזית אנגיוגנית עם תאי גבעול וקצה והבשלה ראשונית של כלי הדם הנראית לעין ב-P6, והבשלה של מקלעת כלי הדם הנראית לאחר P9 8,9. במהלך השיפוץ של מקלעת כלי הדם הראשונית, תאי האנדותל עוברים אפיון לכיוון פנוטיפים עורקיים, נימיים וורידיים בכלי דם שונים כדי ליצור רשת מחזור דם10,11. לכן, שיטה זו מאפשרת לחוקרים לדמיין היווצרות מקלעת כלי דם אנגיוגנית ומפרט עורקי-ורידי אנדותל והתבגרות בנקודות זמן שונות במהלך ההתפתחות9. בנוסף, מודל זה מספק שיטה לחקירת ההשפעות של מניפולציה מהונדסת על אנגיוגנזה והתפתחות מקלעת כלי הדם, אשר יושמה לחקר התפתחות כלי הדם, מומים עורקיים-ורידיים וניאו-וסקולריזציה המושרה על ידי חמצן 12,13,14,15,16 . על מנת לשלב גישות ריצוף מהדור הבא עם מודל פיתוח כלי הדם ברשתית, יש צורך בפרוטוקול אופטימלי לבידוד תאי אנדותל מרקמת הרשתית.
פרוטוקול זה מתאר שיטה אופטימלית לעיכול רקמת רשתית מעכברים ב-P6 כדי למקסם את תפוקת התאים, טוהר וכדאיות. רקמת הרשתית מבודדת מעכברי P6, מתעכלת במשך 20 דקות, מוכתמת חיסונית עבור CD31 ו-CD45, ומטוהרת באמצעות FACS כדי לבודד תרחיף של תא בודד של תאי אנדותל תוך כשעתיים וחצי (איור 1A). נמצא כי תאי אנדותל אלה שומרים על כדאיות גבוהה במשך 60 דקות לאחר הבידוד17, מה שמאפשר הכנה לספרייה לשיטות ריצוף מהדור הבא. בנוסף, תוצאות מייצגות מסופקות עבור תוצאות בקרת איכות ובקרת איכות של FACS משתי שיטות ריצוף נפרדות מהדור הבא המשתמשות בפרוטוקול בידוד זה: ריצוף RNA שלם של תעתיק וריצוף RNA של תא יחיד. שיטה זו מאפשרת להשתמש בגישות ריצוף מהדור הבא בשילוב עם מודל כלי הדם ברשתית כדי להבהיר מנגנונים חדשים של התפתחות כלי הדם.
Protocol
הוועדות המוסדיות לטיפול ושימוש בבעלי חיים של אוניברסיטת ייל ואוניברסיטת וירג'יניה אישרו את כל הניסויים בבעלי חיים המפורטים בפרוטוקול זה.
1. השג עיני עכבר לבידוד הרשתית
- הכינו PBS קר כקרח אחד והוסיפו 500 מיקרון לכל באר של צלחת בת 48 בארות.
- הרדמת עכברים ילודים ביום השישי שלאחר הלידה (P6) בהתאם להנחיות המוסדיות המאושרות. לצורך ניסוי זה, המלטות של כ-4-8 עכברים ילודים מורדמות ב-P6 באמצעות שאיפת איזופלורן למשך שלוש דקות לפחות לאחר המעצר הנשימתי, ולאחר מכן עריפה.
- הסר את העיניים מכל אחד מהעכברים. יש לחתוך את העור ואת הממברנה מעל העין על ידי חיתוך בניצב לעפעף באמצעות מספריים לנתיחה. לאחר מכן, השתמש במלקחיים כדי ללחוץ בעדינות כלפי מטה מעל ומתחת לעין, כך שהעין תזוז אל מחוץ לשקע.
- צובטים בזהירות מתחת לעין עם המלקחיים וחותכים את עצב הראייה שמחזיק את העין מחוברת. לאחר מכן, הניחו כל עין בצלחת 48 בארות ב-PBS המוכן 1x קר כקרח עד לסיום הקציר.
הערה: השתמש באר אחת לכל עכבר, כאשר שתי העיניים מעכבר יחיד נמצאות באותה באר.
- צובטים בזהירות מתחת לעין עם המלקחיים וחותכים את עצב הראייה שמחזיק את העין מחוברת. לאחר מכן, הניחו כל עין בצלחת 48 בארות ב-PBS המוכן 1x קר כקרח עד לסיום הקציר.
2. לבודד רקמת רשתית עכבר
- מלאו צלחת פטרי, מרופדת במשטח דיסקציה בתחתית, ב-500 מיקרולטר של PBS קר כקרח כדי להטביע את העיניים ולהניח אותה תחת מיקרוסקופ דיסקציה המוגדר להגדלה של פי 4.0.
- להשעות את העיניים על כרית דיסקציה באמצעות פיפטה העברה עם קצה רחב כדי לא לפגוע בעיניים.
הערה: ודא שהעיניים מכוסות לחלוטין ב-PBS.
- להשעות את העיניים על כרית דיסקציה באמצעות פיפטה העברה עם קצה רחב כדי לא לפגוע בעיניים.
- באמצעות שני מלקחיים עדינים, יש להחזיק את עצב הראייה עם אחד המלקחיים לייצוב, ובעזרת המלקחיים השניים לנקב חור דרך החדר הקדמי שבו הקרנית והסקלרה מתחברות (איור 1B). קורעים את החור במעגל כ-75% מהדרך סביב הקרנית.
- בזמן שאתם עדיין מחזיקים את עצב הראייה עם אחד המלקחיים, השתמשו במלקחיים השניים כדי לקרוע בעדינות את הסקלרה מרקמת הרשתית.
- כנ"ל, השתמש במלקחיים השניים כדי לקרוע בעדינות את הגוף הזגוגי של רקמת הרשתית.
- שוב, באמצעות המלקחיים השניים, הסר בעדינות את העדשה ואת כלי מקלעת היאלואיד שלא ירדו כאשר הגוף הזגוגי הוסר. כדי לעשות זאת, להגיע לתוך הרשתית עם מלקחיים פתוחים עד כמעט לגעת ברשתית, לסגור את המלקחיים כדי לתפוס את העדשה ואת כלי מקלעת hyaloid, ולמשוך את המלקחיים החוצה מן הרשתית. ניתן לחזור על כך עד להסרת כל כלי הדם.
הערה: מקלעת ההיאלואיד דומה למבנה ברור דמוי רשת.
- מלאו 2 מ"ל צינורות מיקרוצנטריפוגה ב-500 מיקרו-ליטר של PBS קר כקרח והניחו את הרשתיות בצינורות. בודדו את כל הרשתיות מהעיניים והכניסו אותן ל-PBS קר כקרח לפני שתמשיכו הלאה.
הערה: השתמש בצינור אחד לכל עכבר, כאשר שתי רשתיות מאותו עכבר חולקות צינור. אם רקמת הרשתית קרועה, להעביר אותו במספר חתיכות. הזמן הכולל של כריתת רקמת הרשתית עבור המלטה של עכברים צריך לקחת 15-60 דקות, החל בהמתת חסד וכלה בבידוד סופי של רקמת הרשתית.
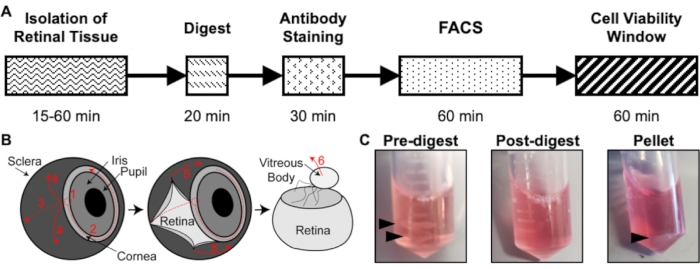
איור 1: סקירה כללית של פרוטוקול הבידוד. (A) סכמת ציר הזמן של הבידוד עם זמן משוער לכל שלב: בידוד רקמת הרשתית, תקציר, צביעת נוגדנים, FACS וחלון כדאיות התא. (B) מדריך שלב אחר שלב לבידוד רקמת הרשתית מהעין, עם שלבי דיסקציה ממוספרים: 1) קרנית נוקבת, 2) קרנית דמעות, 3) דמעה סקלרה, 4) הסרת סקלרה מהרשתית, 5) הסרה נוספת של סקלרה ורקמת חיבור מהרשתית, 6) הסרת הגוף הזגוגי וכלי הזגוגית מהרשתית. (C) תמונות מייצגות של רקמת הרשתית במהלך שלבי עיכול שונים: קדם-עיכול, פוסט-עיכול, גלולה (חיצים שחורים מדגישים רקמת רשתית או כדור תא). פורסם מחדש באישור חבקין ואחרים פורסם ב- S. Karger AG, באזל17. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
3. לעכל רקמת רשתית לתרחיף של תא בודד
- הכינו תמיסת עיכול של 500 μL לכל שתי רשתיות מבודדות. הוסף FBS וקולגןאז סוג II ל-DMEM לריכוז סופי של 10% FBS ו-1 מ"ג/מ"ל קולגן מסוג II. מערבבים ומחממים את התמיסה ל-37 מעלות צלזיוס באמצעות אמבט מים.
- הסר עודפי PBS מצינורות המיקרוצנטריפוגה של 2 מ"ל על ידי פיפטציה זהירה. השאירו מספיק PBS כדי לכסות לחלוטין את רקמת הרשתית, כ -100 μL בכל צינור.
- הוסיפו 500 μL מתמיסת העיכול לכל צינור עם רקמת רשתית (איור 1C).
- השתמש בפיפטור P1000 ובקצה פיפטה כדי לבצע פיפטה במעלה ובמורד רקמת הרשתית בתמיסת העיכול חמש פעמים.
- לדגור את תערובת העיכול באמבט מים 37 מעלות צלזיוס במשך 20 דקות.
- השתמש בפיפטור P1000 ובקצה פיפטה כדי לקטר את תערובת העיכול למעלה ולמטה כל 5 דקות. לאחר הדגירה, רקמת הרשתית התמוססה לתרחיף של תא בודד, כך שתערובת העיכול צריכה להיות עכורה (איור 1C).
4. ספירת תאים
- הניחו צנטריפוגה שולחנית בטמפרטורה של 4 מעלות צלזיוס והניחו בתוכה את צינורות תערובת העיכול. גלולה את תערובת העיכול על ידי צנטריפוגה ב 375 x גרם במשך 5 דקות.
- הסר בזהירות את תמיסת העיכול העל-טבעית על-ידי פיפטציה. אל תפריעו לכדור התא (איור 1C). השהה מחדש את הכדור ב-500 μL של PBS קר כקרח 1x על ידי ערבוב עדין למעלה ולמטה עם הפיפטה.
- לספור את התאים עם hemocytometer תחת מיקרוסקופ.
הערה: ספירת התאים היא בערך 1 x 106 תאים לכל שתי רשתיות. - ודא שהתאים עוברים החייאה כראוי על ידי ערבוב עדין, ולאחר מכן aliquot 20 μL של תרחיף התא לשלושה צינורות לצביעת בקרה (צינור 1: בקרת IgG, צינור 2: בקרת CD31, וצינור 3: בקרת CD45), כפי שתואר ונעשה בו שימוש בעבר17,18.
- מניחים צנטריפוגה שולחנית בטמפרטורה של 4 מעלות צלזיוס ומניחים בתוכה צינורות המכילים תאים. גלולה את התאים על ידי צנטריפוגה ב 375 x גרם במשך 5 דקות.
5. אימונוסטין התאים עם נוגדנים
- הכינו 100 μL של חיץ צביעה לכל שתי רשתיות מוכתמות בתוספת ארבעה צינורות בקרה. הוסיפו FBS, HEPES ו-D-גלוקוז למאגר HBSS לריכוז סופי של 10% FBS, 10 mM HEPES ו-1 מ"ג/מ"ל D-גלוקוז.
- השג נוגדנים מצומדים פלואורסצנטיים כנגד CD31 ו- CD45. הוסיפו את הנוגדנים בדילול של 1:100 לתוך חיץ הכתמים (צינור 1: נוגדן IgG בלבד, צינור 2: נוגדן CD31 בלבד, וצינור 3: נוגדן CD45 בלבד). הוסיפו 1 μL של נוגדן לכל 100 μL של חיץ מכתים לקבלת ריכוז נוגדנים סופי של 2 מיקרוגרם למ"ל.
- הסר בזהירות את PBS מכדור התא השטוף על ידי פיפטציה.
- יש להשעות את הכדור ב-100 μL של תמיסת צביעת נוגדנים לכל 0.5 x 106 תאים .
- דגרו את התרחיף החד-תאי עם תמיסת צביעת הנוגדנים למשך 30 דקות על קרח בחושך. הקש על הצינורות כל 10 דקות כדי לערבב בעדינות את התאים.
6. התכוננו למיון תאים המופעל על ידי פלואורסצנציה
- מניחים צנטריפוגה שולחנית בטמפרטורה של 4 מעלות צלזיוס ומניחים בתוכה את הצינורות המכילים תאים. תאי גלולה על ידי צנטריפוגה ב 375 x גרם במשך 5 דקות.
- הסר את הסופר-נטנט ושלח מחדש את התאים האלה ב-500 μL של 1x PBS לשטיפה.
- מניחים צנטריפוגה שולחנית בטמפרטורה של 4 מעלות צלזיוס ומניחים בתוכה את הצינורות המכילים תאים. תאי גלולה על ידי צנטריפוגה ב 375 x גרם במשך 5 דקות
- הכן 1.5 מ"ל של מאגר FACS (1% FBS ב- PBS אחד).
- הסר את הסופר-נאטנט והשעה מחדש את גלולת התא ב-300 μL של FACS Buffer על-ידי ערבוב עדין עם פיפטטור.
- הוסף פרופידיום יודיד (PI) לצינורות הדגימה המכילים את מאגר FACS לריכוז סופי של 0.5 מיקרוגרם למ"ל. PI משמש כסמן כדאיות.
- העבר ושלב את מתלי התאים למבחנות של 5 מ"ל באמצעות מכסה הצמדה של מסננת תאים. מכסה ההצמדה של מסננת התא צריך להכיל מסנן של 35 מיקרומטר, וניתן לצינור את מתלי התא ישירות על המסנן.
- שמור את צינורות FACS על קרח בחושך והעבר אותם לממיין התאים.
7. הגדר את מכשיר ה-FACS
- התקן זרבובית של 100 מיקרומטר במכשיר FACS.
הערה: זרבובית בגודל זה מצמצמת את נפח האיסוף וממקסמת את צפיפות התאים המבודדים. - הכן 250 μL של PBS 1x לתוך צינור microcentrifuge 1.5 מ"ל כמו צינור איסוף להיות מותקן לתוך מכשיר FACS.
8. לבודד תאי אנדותל בני קיימא באמצעות FACS
- הפעל את מכשיר ה-FACS ואת המחשב. לחץ על סמל תוכנת FACS כדי לפתוח את תוכנת FACS במחשב כדי להפעיל ולהפעיל את מכשיר FACS. בצע בדיקות בקרת איכות שגרתיות.
- טען את הדגימות המוכתמות בבקרה למכשיר FACS כדי להתאים את הצירים. התחל עם תאי נוגדן IgG בלבד כדי ליצור ולהתאים FSC ו- SSC, לאחר מכן נוגדן CD31 רק כדי ליצור ולהתאים CD31, ולאחר מכן נוגדן CD45 רק כדי ליצור ולהתאים CD45. הקלט נתונים מדוגמאות פקדים.
- טען תאי דגימה מוכתמים במכשיר FACS והפעל את הדגימה.
- שער התאים בהתבסס על פרמטרים של פיזור קדימה ופיזור צדדי (FSC-A ו-SSC-A, בהתאמה) (איור 2A).
הערה: הפרמטרים FSC-A ו- SSC-A משמשים לבחירת תאים בהתבסס על גודל, צפיפות, גרעיניות, מאפייני משטח ומקדם שבירה. - תאי שער על-ידי FSC-A ו-FSC-H כדי לזהות כפילי תאים ולאסוף רק תאים בודדים (איור 2A).
הערה: הפרמטרים FSC-A ו- FSC-H משמשים לבחירת תאים בודדים בהתבסס על העיקרון שבמהלך פיזור קדימה, כפילי תאים יהיו טיפות המכילות יחס שטח לגובה גדול יותר. - תאי שער על-ידי PI ו-SSC-A כדי לזהות תאים בני קיימא (איור 2A).
הערה: תאים בני קיימא יהיו שליליים PI. - צור שער CD31+/CD45 עם פקדים ותאי שער לפי CD31 ו-CD45 (איור 2A,B).
- הכנס צינור איסוף עם 250 μL של מאגר FACS.
- התחל למיין תאים חיוביים ל- CD31 / CD45-שליליים לתוך צינור האיסוף המותקן.
- שמור את צינורות האיסוף על קרח לניתוח נוסף. ניתן לעבד דוגמאות עבור יישומי רצף מהדור הבא בשלב1 זה.
9. בצע בדיקת כדאיות
- יש לערבב 10 μL של תאים עם 10 μL של 0.4% תמיסת טריפאן כחולה כדי להעריך את כדאיות התא.
- ספרו תאים בני קיימא על ידי העברת התערובת המוכתמת על מגלשת המוציטומטר. מקם את המגלשה מתחת למיקרוסקופ לקבלת ספירות כדאיות מדויקות. ספירת תאים כחולים כלא ברי קיימא וספירת תאים צלולים כבני קיימא.
- חלק את ספירת התאים הקיימת בספירת התאים הכוללת כדי לחשב את אחוז הכדאיות.
10. בצע בדיקת ביטוי גנים
- להשיג 10,000-20,000 תאים לכל דגימה ולבצע בידוד RNA כדי לנתח ביטוי גנים של תאי אנדותל ברשתית מורין. בידוד RNA מתבצע באמצעות ערכה זמינה מסחרית המשתמשת במשקעי RNA מבוססי אתנול, ולאחר מכן צנטריפוגה ועמודי ממברנה כדי לשטוף ולחמוק RNA מטוהר מליזאט התא. ניתן לעבד דוגמאות עבור יישומי רצף מהדור הבא בשלב1 זה.
- המירו את הרנ"א ל-cDNA באמצעות אנזים טרנסקריפטאז הפוך וריאגנטיםתומכים 19.
- כימות באמצעות צבע מחייב DNA ומכונת PCR כמותית (qPCR)20. טען דגימות cDNA בלוח qPCR של 384 בארות עם פריימרים כדי להגביר גנים מעניינים. נפח התגובה צריך להסתכם ב-10 μL, כולל צבע קושר DNA, שישמש לכימות הגברה של גנים.
- השתמש בפריימרים מסוג CD31, VE-Cadherin, CD45 ו-β-actin קדימה ואחורה כדי למדוד את ביטוי הגנים.
- השתמש בדוגמה עבור יישומי רצף מהדור הבא1.
תוצאות
עיכול רקמת הרשתית וחיסון עבור CD31 ו-CD45 גורם לאוכלוסייה הניתנת לזיהוי של תאי אנדותל מסוג CD31+/CD45 לאחר מיון לתאים, תאים בודדים וחיוניות (איור 2A). CD45 immunostaining נדרש כדי לחסל CD31+/CD45+ תאים, הכוללים טסיות דם וכמה לויקוציטים21. בכל ניסוי יש לבצע בקרות כדי להראות את ספציפיות ה?...
Discussion
פרוטוקול זה מתאר שיטה לבידוד תאי אנדותל מרקמת רשתית מורין לאחר הלידה, אשר עברה אופטימיזציה למספר תאים גבוה, טוהר וכדאיות. טוהר התאים מתקבל על ידי בידוד FACS של אוכלוסיות תאי אנדותל מתרחיף חד-תאי מעוכל על ידי CD31+/CD45- אימונוסטינינג. איכות הבידוד מכמתת במבחנים לכדאיות על ידי צביעה כחולה של טריפא...
Disclosures
למחברים אין גילויים רלוונטיים.
Acknowledgements
תודה למתקן הציטומטריה של זרימת ייל, למתקן הליבה של ציטומטריה של זרימה באוניברסיטת וירג'יניה, למרכז ייל לאנליזה גנומית, ולליבת ניתוח הגנום והטכנולוגיה של אוניברסיטת וירג'יניה על המאמץ, המומחיות והייעוץ שלהם בתרומה לניסויים המוצגים. מחקר זה מומן על ידי מענקי NIH ל- N.W.C. (T32 HL007224, T32 HL007284), S.C. (T32 HL007284), K.W. (R01 HL142650) ו- K.K.H. (R01 HL146056, R01 DK118728, UH3 EB025765).
Materials
| Name | Company | Catalog Number | Comments |
| 2 mL Eppendorf safe-lock tubes | USA Scientific | 4036-3352 | |
| 5 ml Falcon Test Tubes with Cell Strainer Snap Cap | Corning | 352235 | |
| 60 mm Non TC-treated Culture Dish | Corning | 430589 | |
| APC Rat Anti-Mouse CD31 | BD Biosciences | 551262 | |
| APC Rat IgG2a κ Isotype Control | BD Biosciences | 553932 | |
| BD FACSChorus Software | BD Biosciences | FACSCHORUS | |
| BD FACSMelody Cell Sorter | BD Biosciences | FACSMELODY | |
| Collagenase Type II | Sigma-Aldrich | 234115 | |
| Costar 48-well Clear TC-treated Multiple Well Plates, Individually Wrapped, Sterile | Corning | 3548 | |
| D-Glucose | Gibco | A2494001 | |
| Disposable Graduated Transfer Pipettes | Fisher Scientific | 12-711-9AM | |
| Dissecting Pan Wax | Carolina | 629100 | |
| Dissection scissors | Fine Science Tools | 14085-08 | |
| Dissection Stereo Microscope M165 FC | Leica | M165FC | |
| Dulbecco's Modified Eagle Medium (DMEM) | Gibco | 11965-052 | |
| Dulbecco’s Phosphate Buffered Saline (PBS) | Gibco | 14190144 | |
| Eppendorf Flex-Tubes Microcentrifuge Tubes 1.5 mL | Sigma-Aldrich | 22364120 | |
| Fetal Bovine Serum (FBS) | Gemini Bio | 100-106 | |
| Fine dissection forceps | Fine Science Tools | 11250-00 | |
| Hank's Buffered Salt Solution (HBSS) | Gibco | 14175095 | |
| HEPES (1M) | Gibco | 15630130 | |
| iScript cDNA Synthesis Kit | Bio-Rad | 1708890 | |
| Isoflurane, USP | Covetrus | 11695067772 | |
| Isotemp General Purpose Deluxe Water Bath | Fisher Scientific | FSGPD20 | |
| Primer: ActB_Forward: 5’- agagggaaatcgtgcgtgac -3’ | Integrated DNA Technologies | N/A | |
| Primer: ActB_Reverse: 5’- caatagtgatgacctggccgt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Forward: 5’- gagcccaatcacgtttcagttt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD31_Reverse: 5’- tccttcctgcttcttgctagct -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Forward: 5’- gggttgttctgtgccttgtt -3’ | Integrated DNA Technologies | N/A | |
| Primer: CD45_Reverse: 5’- ctggacggacacagttagca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Forward: 5’- tcctctgcatcctcactatcaca -3’ | Integrated DNA Technologies | N/A | |
| Primer: VE-Cadherin_Reverse: 5’- gtaagtgaccaactgctcgtgaat -3’ | Integrated DNA Technologies | N/A | |
| Propidium iodide | Sigma-Aldrich | P4864 | |
| RNeasy Plus Mini Kit | Qiagen | 74134 | |
| Sorvall Legend Micro 21R Centrifuge, Refrigerated | ThermoFisher | 75002477 | |
| SYBR-Green iTaq Universal SYBR Green Supermix | Bio-Rad | 172-5120 | |
| Trypan Blue Solution | ThermoFisher | 15250061 | |
| V450 Rat Anti-Mouse CD45 | BD Biosciences | 560501 | |
| V450 Rat IgG2b, κ Isotype Control | BD Biosciences | 560457 |
References
- Slatko, B. E., Gardner, A. F., Ausubel, F. M. Overview of next-generation sequencing technologies. Current Protocols in Molecular Biology. 122 (1), 59 (2018).
- Chavkin, N. W., Hirschi, K. K. Single cell analysis in vascular biology. Frontiers in Cardiovascular Medicine. 7, 42 (2020).
- Ma, F., Hernandez, G. E., Romay, M., Iruela-Arispe, M. L. Single-cell RNA sequencing to study vascular diversity and function. Current Opinion in Hematology. 28 (3), 221-229 (2021).
- Potter, A. S., Potter, S. S., S, Dissociation of tissues for single-cell analysis. Methods in Molecular Biology. 1926, 55-62 (2019).
- Braga, F. A. V., Miragaia, R. J. Tissue handling and dissociation for single-cell RNA-seq. Single Cell Methods: Methods in Molecular Biology. 1979, 9-21 (2019).
- Brink, S. C., et al. Single-cell sequencing reveals dissociatin-induced gene expression in tissue subpopulations. Nature Methods. 14 (10), 935-936 (2017).
- Skulska, K., Wegrzyn, A. S., Chelmonska-Soyta, A., Chodaczek, G. Impact of tissue enzymatic digestion on analysis of immune cells in mouse reproductive mucosa with a focus on gammadelta T cells. Journal of Immunological Methods. 474, 112665 (2019).
- Connolly, S., Hores, T., Smith, L., D'Amore, P. Characterization of vascular development in the mouse retina. Microvascular Research. 36 (3), 275-290 (1988).
- Crist, A., Young, C., Meadows, S. Characterization of arteriovenous identity in the developing neonate mouse retina. Gene Expression Patterns: GEP. 23-24, 22-31 (2017).
- dela Paz, N. G., D'Amore, P. A. Arterial versus venous endothelial cells. Cell and Tissue Research. 335 (1), 5-16 (2009).
- Fang, J. S., Hirschi, K. K. Molecular regulation of arteriovenous endothelial cell specification. F1000Res. 8, (2019).
- Smith, L. E., et al. Oxygen-induced retinopathy in the mouse. Investigative Ophthalmology and Visual Science. 35 (1), 101-111 (1994).
- Pitulescu, M. E., Schmidt, I., Benedito, R., Adams, R. H. Inducible gene targeting in the neonatal vasculature and analysis of retinal angiogenesis in mice. Nature Protocols. 5 (9), 1518-1534 (2010).
- Ruiz, S., et al. A mouse model of hereditary hemorrhagic telangiectasia generated by transmammary-delivered immunoblocking of BMP9 and BMP10. Scientific Reports. 6, 37366 (2016).
- Fang, J. S., et al. Shear-induced Notch-Cx37-p27 axis arrests endothelial cell cycle to enable arterial specification. Nature Communication. 8 (1), 2149 (2017).
- Ola, R., et al. SMAD4 prevents flow induced arterial-venous malformations by inhibiting Casein Kinase 2. Circulation. 138 (21), 2379-2394 (2018).
- Chavkin, N. W., Walsh, K., Hirschi, K. K. Isolation of highly purified and viable retinal endothelial cells. Journal of Vascular Research. 58 (1), 49-57 (2020).
- Hulspas, R., O'Gorman, M. R. G., Wood, B. L., Gratama, J. W., Sutherland, D. R. Considerations for the control of background fluorescence in clinical flow cytometry. Cytometry PartB: Clinical Cytometry. 76 (6), 355-364 (2009).
- Bachman, J. Reverse-transcription PCR (RT-PCR). Methods in Enzymology. 530, 67-74 (2013).
- Green, M. R., Sambrook, J. Quantification of RNA by real-time Reverse Transcription-Polymerase Chain Reaction (RT-PCR). Cold Spring Harbour Protocols. 2018 (10), (2018).
- Liu, L., Shi, G. P. CD31: beyond a marker for endothelial cells. Cardiovascular Research. 94 (1), 3-5 (2012).
- Zarkada, G., et al. Specialized endothelial tip cells guide neuroretina vascularization and blood-retina-barrier formation. Developmental Cell. 56 (15), 2237-2251 (2021).
- Su, X., Sorenson, C. M., Sheibani, N. Isolation and characterization of murine retinal endothelial cells. Molecular Vision. 9, 171-178 (2003).
- Benedito, R., et al. The notch ligands Dll4 and Jagged1 have opposing effects on angiogenesis. Cell. 137 (6), 1124-1135 (2009).
- Daneman, R., et al. Wnt/beta-catenin signaling is required for CNS, but not non-CNS, angiogenesis. Proceedings of the National Academy of Sciences of the United States of America. 106 (2), 641-646 (2009).
- Okabe, K., et al. Neurons limit angiogenesis by titrating VEGF in retina. Cell. 159 (3), 584-596 (2014).
- Crist, A. M., Lee, A. R., Patel, N. R., Westhoff, D. E., Meadows, S. M. Vascular deficiency of Smad4 causes arteriovenous malformations: a mouse model of Hereditary Hemorrhagic Telangiectasia. Angiogenesis. 21 (2), 363-380 (2018).
- Kim, Y. H., Choe, S. W., Chae, M. Y., Hong, S., Oh, S. P. SMAD4 deficiency leads to development of arteriovenous malformations in neonatal and adult mice. Journal of the American Heart Association. 7 (21), 009514 (2018).
- Ma, W., et al. Absence of TGFbeta signaling in retinal microglia induces retinal degeneration and exacerbates choroidal neovascularization. eLife. 8, 42049 (2019).
- Luo, W., et al. Arterialization requires the timely suppression of cell growth. Nature. 589 (7842), 437-441 (2021).
- Lawson, N. D., Vogel, A. M., Weinstein, B. M. sonic hedgehog and vascular endothelial growth factor act upstream of the Notch pathway during arterial endothelial differentiation. Developmental Cell. 3 (1), 127-136 (2002).
- Larrivee, B., et al. ALK1 signaling inhibits angiogenesis by cooperating with the Notch pathway. Developmental Cell. 22 (3), 489-500 (2012).
- Wythe, J. D., et al. ETS factors regulate Vegf-dependent arterial specification. Developmental Cell. 26 (1), 45-58 (2013).
- Davis, D. M., Purvis, J. E. Computational analysis of signaling patterns in single cells. Seminars in Cell and Developmental Biology. , 35-43 (2015).
- Gaudet, S., Miller-Jensen, K. Redefining signaling pathways with an expanding single-cell toolbox. Trends in Biotechnology. 34 (6), 458-469 (2016).
- Aibar, S., et al. SCENIC: single-cell regulatory network inference and clustering. Nature Methods. 14 (11), 1083-1086 (2017).
- Trapnell, C., et al. The dynamics and regulators of cell fate decisions are revealed by pseudotemporal ordering of single cells. Nature Biotechnology. 32 (4), 381-386 (2014).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved