Method Article
מידול אתר פעיל אנזים באמצעות תוכנה חופשית להדמיה מולקולרית
In This Article
Summary
מיומנות מרכזית בדוגמנות ביומולקולרית היא הצגת וביאור אתרים פעילים בחלבונים. טכניקה זו מודגמת באמצעות ארבע תוכניות פופולריות בחינם להדמיה מקרומולקולרית: iCn3D, Jmol, PyMOL, ו UCSF כימרהX.
Abstract
כישורי הדמיה ביומולקולריים הם בעלי חשיבות עליונה להבנת מושגי מפתח במדעי הביולוגיה, כגון יחסי מבנה-פונקציה ואינטראקציות מולקולריות. תוכניות שונות מאפשרות ללומד לתפעל מבנים תלת-ממדיים, ומודל ביומולקולרי מקדם למידה פעילה, בונה מיומנויות חישוביות ומגשר על הפער בין תמונות דו-ממדיות של ספרי לימוד לשלושת ממדי החיים. מיומנות קריטית בתחום זה היא לדגמן אתר פעיל בחלבון, המציג חלקים של המקרומולקולה שיכולים לקיים אינטראקציה עם מולקולה קטנה, או ליגנד, באופן שמראה אינטראקציות מחייבות. בפרוטוקול זה, אנו מתארים תהליך זה באמצעות ארבע תוכניות מידול מקרומולקולרי זמין באופן חופשי: iCn3D, Jmol / JSmol, PyMOL, ו UCSF ChimeraX. מדריך זה מיועד לסטודנטים המבקשים ללמוד את היסודות של תוכנית ספציפית, כמו גם מדריכים המשלבים מודלים ביומולקולריים בתוכנית הלימודים שלהם. הפרוטוקול מאפשר למשתמש לדגמן אתר פעיל באמצעות תוכנית ויזואליזציה ספציפית, או לדגום כמה מהתוכניות החינמיות הזמינות. המודל שנבחר לפרוטוקול זה הוא גלוקוזקינאז אנושי, איזופורם של האנזים הקסוקינאז, אשר מזרז את הצעד הראשון של גליקוליזה. האנזים קשור לאחד המצעים שלו, כמו גם אנלוגי מצע לא תגובתי, המאפשר למשתמש לנתח אינטראקציות בקומפלקס הקטליטי.
Introduction
הבנת הייצוגים של העולם המולקולרי היא קריטית כדילהיות מומחהבמדעים הביומולקולריים 1 , כי הפרשנות של תמונות כאלה היא המפתח להבנת הפונקציה הביולוגית2. ההיכרות של הלומד עם macromolecules בדרך כלל מגיעה בצורה של תמונות דו ממדיות של קרום התא, אברונים, macromolecules, וכו 'אבל המציאות הביולוגית היא כי אלה הם מבנים תלת ממדיים, והבנה של המאפיינים שלהם דורש דרכים לדמיין ולחלץ משמעות מודלים 3D.
בהתאם, התפתחות האוריינות החזותית הביומולקולרית בקורסי מדעי החיים המולקולריים בחטיבה העליונה זכתה לתשומת לב, כאשר מספר מאמרים דיווחו על החשיבות והקשיים של הוראה והערכה של מיומנויות הדמיה1,3,4,5,6,7,8,9 . המענה למאמרים אלה היה עלייה במספר ההתערבויות בכיתה, בדרך כלל בתוך סמסטר במוסד אחד, שבו תוכניות ויזואליזציה מולקולרית ומודלים משמשים למיקוד מושגים קשים2,10,11,12,13,14,15 . בנוסף, החוקרים ביקשו לאפיין כיצד סטודנטים משתמשים בתוכניות הדמיה ביומולקולרית ו /או במודלים כדי לגשת לנושא מסוים16, 17,18,19. הקבוצה שלנו, BioMolViz, תיארה מסגרת המחלקת נושאים על גבי אוריינות חזותית למטרות למידה ויעדים כדי להנחות התערבויות כאלה20,21, ואנחנו מובילים סדנאות להכשיר את הפקולטה להשתמש במסגרת בעיצוב לאחור של הערכות כדי למדוד מיומנויות אוריינות חזותית22.
במרכז כל העבודה הזאת היא מיומנות קריטית: היכולת לתפעל מבנים של macromolecules באמצעות תוכניות להדמיה ביומולקולרית. כלים אלה פותחו באופן עצמאי באמצעות מגוון פלטפורמות; לכן, הם יכולים להיות ייחודיים למדי בפעולה ובשימוש שלהם. זה מחייב הוראות ספציפיות לתוכנית, וזיהוי של תוכנית שהמשתמש מרגיש בנוח עם חשוב כדי להקל על המשך היישום.
מעבר ליסודות הבסיסיים ביותר של מניפולציה של מבנים בתלת-ממד (סיבוב, בחירה ושינוי המודל), מטרה מרכזית היא לדגמן את האתר הפעיל של חלבון. תהליך זה מאפשר ללומד לפתח את הבנתו בשלושה נושאים מקיפים המתוארים על ידי מסגרת BioMolViz: אינטראקציות מולקולריות, ליגנדים / שינויים, ויחסי מבנה-פונקציה20,21.
ארבע אפשרויות פופולריות של תוכניות להדמיה ביומולקולרית כוללות: Jmol / JSmol23, iCn3D24, PyMOL25, ו UCSF כימרה26,27. אנו מעודדים את החדשים של כימרה להשתמש ב- UCSF ChimeraX, הדור הבא של תוכנית ההדמיה המולקולרית של כימרה, שהיא הגרסה הנתמכת כיום של התוכנית.
בפרוטוקול זה, אנו מדגימים כיצד להשתמש בכל אחת מארבע התוכניות הללו כדי לדגמן את האתר הפעיל של גלוקוקינאז אנושי עם קומפלקס אנלוגי מצע מאוגד (מזהה PDB: 3FGU), ולהציג מדידות כדי להמחיש אינטראקציות מחייבות ספציפיות28. המודל מייצג קומפלקס קטליטי של האנזים. כדי ללכוד את האתר הפעיל במצב טרום קטליזה, אנלוגי לא הידרוליזה של ATP היה קשור לאתר הפעיל glucokinase. אסתר חומצה-אדנילט זרחנית זו (ANP) מכילה קשר זרחן-חנקן במקום הצמדת החמצן הזרחן הרגילה במיקום זה. האתר הפעיל מכיל גם גלוקוז (מסומן BCG בדגם) ומגנזיום (מסומן MG). בנוסף, יש יון אשלגן (K) במבנה, הנובע אשלגן כלורי המשמש ממס התגבשות. יון זה אינו קריטי לתפקוד ביולוגי והוא ממוקם מחוץ לאתר הפעיל.
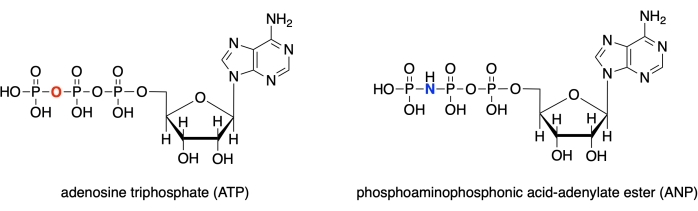
איור 1: מבני ATP/ANP. מבנה אדנוסין טריפוספט (ATP) בהשוואה לאסתר חומצה אדנילט פוספונמית (ANP). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
הפרוטוקול מדגים את הבחירה של ליגנדים קשורים של קומפלקס אנלוגי מצע וזיהוי של שאריות אתר פעיל בתוך 5 Å של המתחם הכרוכות, אשר לוכד חומצות אמינו ומולקולות מים מסוגל לעשות אינטראקציות מולקולריות רלוונטיות, כולל אינטראקציות הידרופובי ואן דר Waals.
התצוגה מופעלת בתחילה כדי להראות את רוב החלבון בייצוג מצויר, עם שאריות חומצות אמינו האתר הפעילות בייצוג מקל כדי להראות את האטומים הרלוונטיים של החלבון ולהדגיש את האינטראקציות המולקולריות. לאחר שלב 3 של הפרוטוקול עבור כל תוכנית, ייצוגים אלה יושמו ותצוגת החלבון דומה בתוכניות על פני תוכניות (איור 2). בסוף הפרוטוקול, קריקטורת החלבון מוסתרת כדי לפשט את התצוגה, ולהתמקד באתר הפעיל.
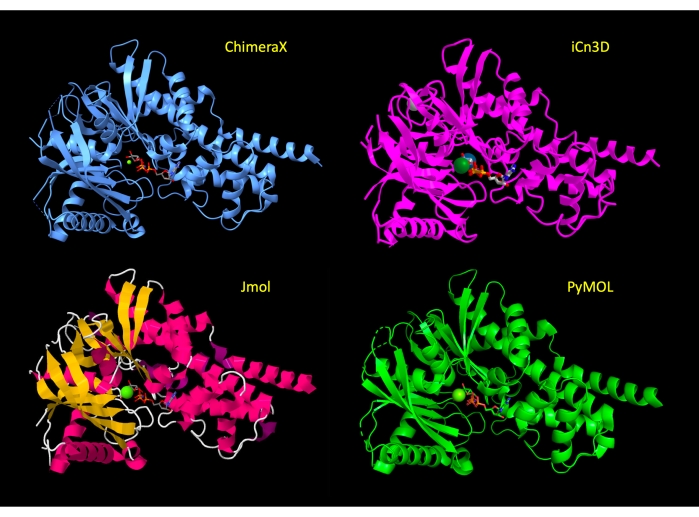
איור 2:השוואת מבנה בין תוכניות. השוואה של המבנה של 3FGU בכל תוכנית לאחר השלב התאמת הייצוג (שלב 2 או 3 של כל פרוטוקול). אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
צביעת CPK מוחלת על חומצות אמינו האתר הפעיל ולידות קשורות29,30. ערכת צביעה זו מבחינה אטומים של יסודות כימיים שונים במודלים מולקולריים המוצגים בתור, מקל, כדור ומקל, וייצוגים ממלאי מקום. מימן הוא לבן, חנקן הוא כחול, חמצן הוא אדום, גופרית היא צהובה, וזרחן הוא כתום בתכנית הצביעה CPK. באופן מסורתי, שחור משמש לפחמן, אם כי בשימוש מודרני, צביעת פחמן עשוי להשתנות.
אטומי מימן אינם נראים במבני קריסטל, אם כי כל אחת מהתוכניות הללו מסוגלת לחזות את מיקומם. הוספת אטומי המימן למבנה מקרומולקולרי גדול יכולה לטשטש את התצוגה, ולכן הם אינם מוצגים בפרוטוקול זה. בהתאם לכך, קשרי מימן יוצגו על ידי מדידה ממרכז שני הטרואטומים (למשל, חמצן לחמצן, חמצן לחנקן) במבנים אלה.
סקירות כלליות של תוכניות
ממשקי משתמש גרפיים להורדה (GUIs): PyMOL (גירסה 2.4.1), כימראקס (גירסה 1.2.5) ו- Jmol (גירסה 1.8.0_301) הם כלי מידול מולקולריים מבוססי GUI. שלושת הממשקים האלה כוללים שורות פקודה לקלט קוד מוקלד; רבות מאותן יכולות זמינות באמצעות תפריטים ולחצנים ב- GUI. תכונה נפוצה בשורת הפקודה של תוכניות אלה היא שהמשתמש רשאי לטעון ולבצע מחדש פקודות קודמות באמצעות מקשי החצים למעלה ולמטה בלוח המקשים.
GUIs מבוסס אינטרנט: iCn3D (I-see-in-3D) הוא מציג מבוסס WebGL לצפייה אינטראקטיבית של מבנים וכימיקלים תלת-ממדיים של מקרומולקולריים באינטרנט, ללא צורך בהתקנת יישום נפרד. הוא אינו משתמש בשורת פקודה, למרות שגירסת האינטרנט המלאה כוללת יומן פקודות הניתן לעריכה. JSmol היא גירסת JavaScript או HTML5 של Jmol לשימוש באתר אינטרנט או בחלון דפדפן אינטרנט, והוא דומה מאוד בפעולה ל- Jmol. ניתן להשתמש ב- JSmol כדי ליצור ערכות לימוד מקוונות, כולל הנפשות.
פרוטאופדיה31,32, FirstGlance ב Jmol33, וממשק האינטרנט JSmol (JUDE) בבית הספר להנדסה של מילווקי למידול ביומולקולרי הם דוגמאות לסביבות עיצוב מקוונות מבוססות Jmolכאלה 34. ויקי Proteopedia הוא כלי הוראה המאפשר למשתמש לדגמן מבנה macromolecule וליצור דפים המציגים מודלים אלה בתוך אתר35. כלי העריכה של סצנות Proteopedia, שנבנה באמצעות JSmol, משלב GUI עם תכונות נוספות שאינן זמינות ב- Jmol GUI.
Jmol ו- iCn3D מבוססים על שפת התכנות Java; JSmol משתמשת ב- Java או ב- HTML5, ו- PyMOL ו- ChimeraX מבוססות על שפת התכנות של פייתון. כל אחת מהתוכניות האלה טוענת קבצי מאגר נתוני חלבון, שניתן להוריד מבנק נתוני החלבון RCSB תחת מזהה PDB אלפאנומרי בן 4 ספרות36,37. סוגי הקבצים הנפוצים ביותר הם קבצי מאגר נתוני חלבון (PDB) המכילים את סיומת .pdb וקובץ מידע קריסטלוגרפי (CIF או mmCIF) המכיל את סיומת .cif. CIF המחליף את PDB כסוג הקובץ המוגדר כברירת מחדל עבור מאגר נתוני החלבון, אך שתי תבניות הקובץ פועלות בתוכניות אלה. תיתכנו הבדלים קלים באופן שבו הרצף/מבנה מוצגים בעת שימוש ב- CIF בניגוד לקבצי PDB; עם זאת, הקבצים פועלים באופן דומה וההבדלים לא יטופלו בפירוט כאן. מסד הנתונים של מידול מולקולרי (MMDB), תוצר של המרכז הלאומי למידע ביוטכנולוגיה (NCBI), הוא תת-קבוצה של מבני PDB שאליהם נקשר מידע קטגורי (למשל, תכונות ביולוגיות, תחומי חלבון שמורים)38. iCn3D, מוצר של NCBI, מסוגל לטעון קבצי PDB המכילים את נתוני MMDB.
כדי להציג מודל, המשתמש יכול להוריד את הקובץ הרצוי מהדף הייעודי Protein Data Bank עבור המבנה (לדוגמה, https://www.rcsb.org/structure/3FGU), ולאחר מכן להשתמש בתפריט קובץ נפתח של התוכנית כדי לפתוח את המבנה. כל התוכניות מסוגלות גם לטעון קובץ מבנה ישירות דרך הממשק, ושיטה זו מפורטת בפרוטוקולים.
כימרהX, Jmol ו- PyMOL GUIs מכילים כל אחד מהם אחד או יותר של הקונסולה שניתן לשנות את גודלו על-ידי גרירת הפינה. iCn3D ו- JSmol כלולים לחלוטין בדפדפן אינטרנט. בעת שימוש ב-iCn3D, ייתכן שהמשתמש יצטרך לגלול בתוך החלונות המוקפצים כדי לחשוף את כל פריטי התפריט, בהתאם לגודל המסך והרזולוציה.
הפרוטוקולים המפורטים כאן מספקים שיטה פשוטה להצגת האתר הפעיל של האנזים באמצעות כל תוכנית. יש לציין כי קיימות דרכים מרובות לביצוע השלבים בכל תוכנית. לדוגמה, ב- ChimeraX, ניתן לבצע את אותה משימה באמצעות תפריטים נפתחים, סרגל הכלים בחלק העליון או שורת הפקודה. משתמשים המעוניינים ללמוד תוכנית ספציפית בפירוט מוזמנים לחקור את ההדרכות המקוונות, המדריכים והוויקי הזמינים עבור תוכניות אלה39,40,41,42,43,44,45,46.
מדריכים וערכות לימוד קיימות עבור תוכניות אלה מציגים את הפריטים בפרוטוקול זה כמשימות נפרדות. כדי להציג אתר פעיל, על המשתמש לסנתז את הפעולות הנדרשות מהמדריכים וערכות הלימוד השונות. כתב יד זה מעצים את ערכות הלימוד הקיימות הזמינות על-ידי הצגת פרוטוקול ליניארי למידול אתר פעיל עם תוויות עם אינטראקציות מולקולריות, ומספק למשתמש לוגיקה למידול אתרים פעיל שניתן להחיל על מודלים ותוכניות אחרים.
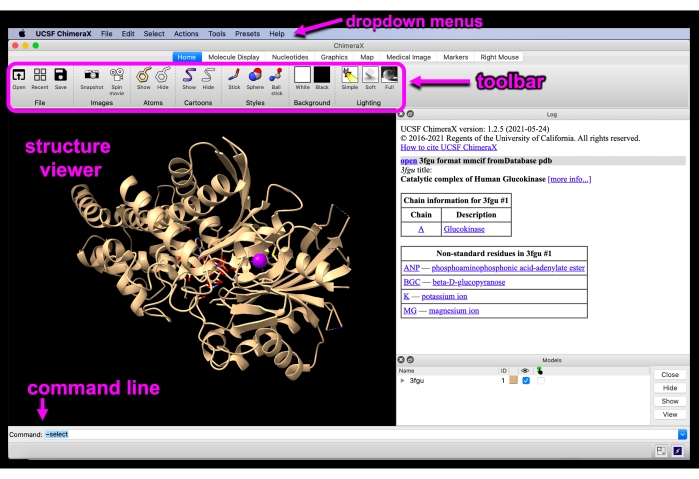
איור 3: כימרקס GUI. ממשק ממשק ממשק ממשק משתמש גרפי של ChimeraX עם התפריטים הנפתחים, סרגל הכלים, מציג המבנה ו שורת הפקודה המסומנים בתווית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
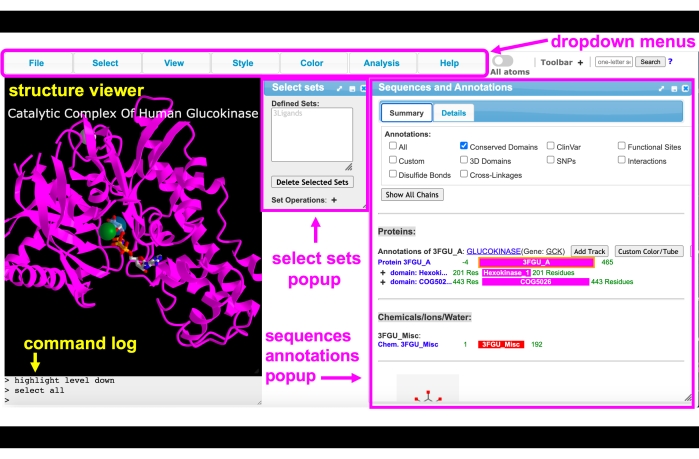
איור 4: iCn3D GUI. iCn3D ממשק ממשק GUI עם התפריטים הנפתחים, סרגל הכלים, מציג המבנים, יומן הפקודות, בחירת ערכות מוקפצות ותפריטים מוקפצים של רצף וביאורים המסומנים בתווית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
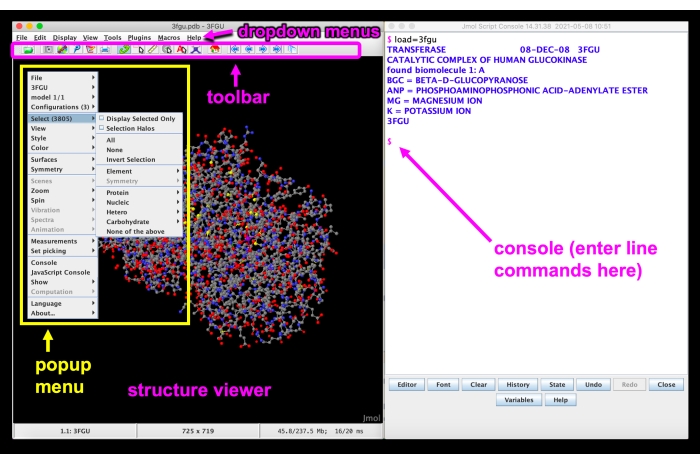
איור 5: ג'מול מג"ב. ממשק Jmol GUI עם התפריטים הנפתחים, סרגל הכלים, מציג המבנה, התפריט המוקפץ וקו המסוף/פקודה המסומנים בתווית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
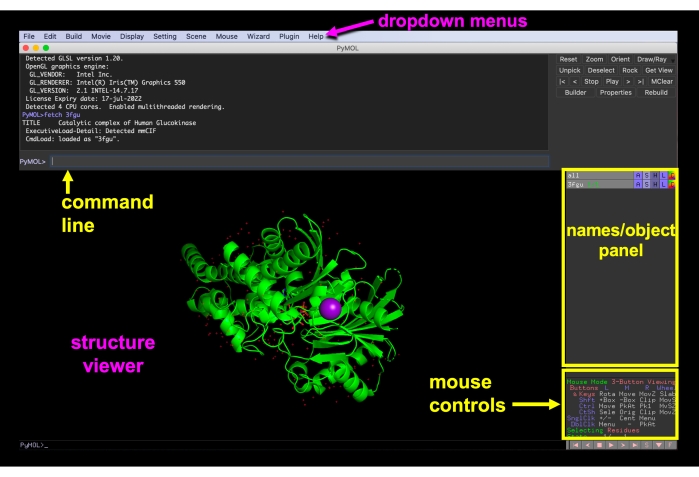
איור 6: PyMOL GUI. ממשק PyMOL GUI עם התפריטים הנפתחים, מציג המבנים, שמות/לוח אובייקטים, תפריט פקדי עכבר ושורה פקודה עם תווית. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Protocol
הערה: הפרוטוקול עבור כל תוכנית מתואר בעשרה שלבים מקיפים, (1) טעינת המבנה לתוכנית, (2) זיהוי הליגנדים באתר הפעיל, (3) התאמת הייצוג, (4) בחירת שאריות בתוך 5 Å כדי להגדיר אתר פעיל, (5) הצגת האינטראקציות של האנזים עם ליגנדים האתר הפעיל, (6) הצגת שרשראות הצד כמקלות והצגת / התאמת מולקולות המים הפעילות באתר, (7) פישוט המבנה, (8) תיוג ליגנדים ושרשראות צד מכובסות במימן, (9) שמירת העיבוד בכל שלב כדי לחזור לעבוד עליו או לשתף עם אחרים, (10) שמירת תמונה להטבעה או להדפסה. שלבים 1, 4 ו- 7-10 זהים עבור כל פרוטוקול; עם זאת, בשל הפעולה הייחודית של כל תוכנית, פרוטוקולים מסוימים מבוצעים ביעילות רבה יותר כאשר שלבים 2/3 ו- 5/6 מתחלפים.
1. פרוטוקול כימרה UCSF
הערה: פקדי משטח עקיבה ועכבר. כדי לסובב, לחץ וגרור או השתמש בגרירה בשתי אצבעות (עכבר: לחץ וגרור שמאלה). כדי להגדיל, לצבוט ולהפיץ (Mac) או לשלוט + תנועה בשתי אצבעות (מחשב) (עכבר: גלגל גלילה). כדי לתרגם (כלומר, להזיז את המבנה כולו) לחץ על האפשרות + לחץ וגרור (Mac) או shift + לחץ וגרור (PC) (עכבר: לחץ וגרור באמצעות לחצן העכבר הימני). כדי למרכז מחדש, השתמש בתפריטים הנפתחים בחלק העליון של הממשק כדי ללחוץ על פעולות > תצוגה.
- טעינת המבנה לתוך כימרהX: בשורת הפקודה הממוקמת בתחתית ה- GUI שלפניו "Command:", הקלד:
פתוח 3fgu
הערה: לאחר הזנת פקודת שורה מוקלדת כלשהי, הקש על חזור בלוח המקשים כדי לבצע אותה. - זיהוי הליגנדים באתר הפעיל: ודא שיש שני ייצוגים, סרט מצויר ומקלות. באמצעות העכבר, לסובב / להגדיל את החלבון כדי לדמיין בצורה הטובה ביותר את ligands המוצגים ליד מרכז החלבון, אשר מוצגים כמו מקלות. רחף מעל ליגנד כדי להציג את שמו.
- התאמת הייצוג: השתמש/י בפקודות בteps שלהלן כדי לצבוע מחדש את החלבון והליגנדים, למרוח את צבעי ה-CPK על אטומים שאינם פחמן ולאחר מכן לבטל את הבחירה בבחירה. חלקים נבחרים של המולקולה להיות מודגש בירוק.
- השתמש בתפריטים הנפתחים בחלק העליון של הממשק כדי לשנות את הצביעה: לחץ על פעולות > צבע > קורנפלור כחול. לאחר מכן, לחץ על בחר מבנה > > ליגנד. כדי לבחור את הצבע, לחץ על פעולות > צבע > אפור. כדי להחיל צביעת CPK, לחץ על בחר > הכלולאחר מכן לחץ על פעולות > צבע > על ידי Heteroatom. לבסוף, נקה את הבחירה על-ידי לחיצה על בחר > נקה.
הערה: ניתן לנקות את הבחירה גם על-ידי הקשה על פקד ולחיצה ברקע השחור של מציג המבנה או בשורת הפקודה על-ידי הקלדה: ~select. כברירת מחדל, עבור רוב המבנים המכילים 1-4 שרשראות, ChimeraX יציג באופן אוטומטי מולקולות מים ושאריות חומצות אמינו בתוך 3.6 Å של ליגנדים ויונים. - השתמש בתפריט הנפתח כדי להסתיר את האטומים המוצגים כעת על-ידי לחיצה על פעולות > אטומים/אג"ח > הסתר.
- השתמש בתפריט הנפתח כדי להציג את הליגנדים ואת Mg יון באתר הפעיל על ידי לחיצה על בחר מבנה > > Ligand. לאחר מכן, לחץ על פעולות > אטומים / אג"ח > הצג. לאחר מכן, לחץ על בחר שאריות > > MG, ולאחר מכן על פעולות > אטומים / אג"ח > הצג. כדי לנקות את הבחירה, לחץ על בחר > נקה.
הערה: לאחר ביצוע הבחירה עם התפריט הנפתח, שלב 1.3.3 עשוי להתבצע על-ידי לחיצה על הלחצנים הסתר והצג בסרגל הכלים אטומים.
- השתמש בתפריטים הנפתחים בחלק העליון של הממשק כדי לשנות את הצביעה: לחץ על פעולות > צבע > קורנפלור כחול. לאחר מכן, לחץ על בחר מבנה > > ליגנד. כדי לבחור את הצבע, לחץ על פעולות > צבע > אפור. כדי להחיל צביעת CPK, לחץ על בחר > הכלולאחר מכן לחץ על פעולות > צבע > על ידי Heteroatom. לבסוף, נקה את הבחירה על-ידי לחיצה על בחר > נקה.
- בחירת שאריות בתוך 5 Å כדי להגדיר אתר פעיל: במציג המבנה, כדי לבחור את פקד הקשת ligands + הזזה וביצוע העכבר לחץ על כל אטום או קשר יחיד בכל אחד משלושת הליגנדים, כלומר, BCG, ANP, ו- Mg.
- לאחר מכן, לחץ על מקש החץ למעלה במקלדת עד שכל האטומים של שלושת הליגנדים מסומנים בזוהר ירוק. הגדר בחירה זו לשימוש עתידי על-ידי לחיצה בתפריט הנפתח בחר > הגדר בורר. בתפריט הנפתח, הקלד:
ליגנדים כדי לתת שם לבחירה הנוכחית ולאחר מכן ללחוץ על אישור.
הערה: לחיצה על החץ למעלה יותר מדי פעמים בשלב 1.4.1 תבחר את החלבון כולו. במקרה זה, לחץ על לחצן החץ למטה עד לבחירת האטומים של שלושת הליגנדים בלבד. - השתמש בתפריט הנפתח כדי לבחור את השאריות בתוך 5 Å של ligands: לחץ על בחר > אזור. בחלון המוקפץ שמופיע, החלף את התפריט הנפתח בחר לשאריותוודא שהממיר העליון מסומן (בדוק את המרחק הנמוך מ- (<) והגדר ל- 5.0 Å). לאחר מכן, לחץ על אישור. רק שאריות שנמצאות במרחק של פחות מ-5 Å יודגשו.
הערה: ניתן לפשט את שלבים 1.4-1.4.2 בהרחבה באמצעות שורת הפקודה, על-ידי הקלדה:
שם ליגנדים קפואים :BGC:MG:ANP
בחר אזור ligands 5 להרחיב שאריות אמיתיות נכון
- לאחר מכן, לחץ על מקש החץ למעלה במקלדת עד שכל האטומים של שלושת הליגנדים מסומנים בזוהר ירוק. הגדר בחירה זו לשימוש עתידי על-ידי לחיצה בתפריט הנפתח בחר > הגדר בורר. בתפריט הנפתח, הקלד:
- הצגת שרשראות הצד כמקלות והצגה/התאמה של מולקולות המים הפעילות באתר: השתמש בתפריט הנפתח כדי להציג את הבחירה והמרכז ולהגדיל את התצוגה של הבחירה על-ידי לחיצה על פעולות > אטומים/אג"ח > הצג כדי להציג אותם. כדי למרכז את הבחירה, לחץ על פעולות > תצוגה. לאחר מכן, כדי לנקות את הבחירה, לחץ על בחר > נקה או לחץ במקום כלשהו בשטח הריק .
- הצגת האינטראקציות של האנזים עם ליגנדים האתר הפעיל: השתמש בתפריטים הנפתחים ולחץ על בחר > בוררים המוגדרים על ידי המשתמש > Ligands. לאחר מכן, לחץ על כלים > ניתוח מבנה > H-אג"ח. בחלון המוקפץ, ודא ש'הגבל לפי בחירה' מסומנת, התפריט הנפתח מוגדר כ'עם קצה אחד לפחות נבחר' ו'בחר אטומים' נבדק ולאחר מכן לחץ על אישור. כדי לנקות את הבחירה, לחץ על בחר > נקה.
הערה: סמן את התיבה תווית מרחק כדי לראות אורכי אג"ח ב- Å; עם זאת, זה עושה את הנוף עסוק מאוד. לבסוף, באפשרותך לשנות את צבע קשרי ה- H על-ידי לחיצה על התיבה צבע ובחירת צבע חדש בחלון המוקפץ. - פישוט המבנה: באמצעות סרגל הכלים המצויר העליון כדי להסתיר את הקריקטורה או לחץ על התפריט הנפתח: פעולות > קריקטורה > הסתר.
- תיוג ליגנדים ושרשראות צד מנוקבים במימן: השתמש בעכבר כדי לבחור שאריות מימן המחוברות לליגנדים (המחוברות על ידי הקווים המקווקו), כמו בשלב 1.4. לאחר מכן, בתפריטים הנפתחים, לחץ על פעולות > תווית > שאריות > שם משולב. לאחר מכן, לחץ על בחר > בוררים המוגדרים על-ידי המשתמש > ליגנדים. לאחר מכן, לחץ על פעולות > תווית > שאריות > כבוי. לבסוף, נקה את הבחירה, על-ידי לחיצה על בחר > נקה.
- שמירת העיבוד בכל שלב כדי לחזור לעבוד עליו או לשתף עם אחרים: בתפריט הנפתח לחץ על קובץ > שמור. בחר מיקום, הזן שם קובץ ולחץ על שמור.
הערה: ודא שהתבנית מוגדרת ל: הפעלת ChimeraX *.cxs. - שמירת תמונה להטבעה או להדפסה: תחילה השתמש בעכבר כדי לכוון את המולקולה כרצונך. שנה את צבע הרקע ללבן על-ידי הקלדה בשורת הפקודה:
הגדרת bgצביעה לבנה
לבסוף, לחץ על סמל התמונה בסרגל הכלים. התמונה תשמור בשולחן העבודה.
הערה: צבע רקע זמין גם בתפריט הנפתח; ב- Mac, לחץ על העדפות > UCSF ChimeraX; במחשב, לחץ על הגדרות > מועדפים > רקע.
2. פרוטוקול iCn3D
הערה: פקדי משטח עקיבה ועכבר: כדי לסובב, לחץ וגרור (עכבר: לחץ וגרור שמאלה). כדי להגדיל, לצבוט ולהפיץ (עכבר: לסובב את גלגל הגלילה). כדי לתרגם (כלומר, להזיז את המבנה כולו) לחץ וגרור בשתי אצבעות (עכבר: לחץ וגרור באמצעות לחצן העכבר הימני). כדי למרכז מחדש, רחף מעל תצוגה בתפריטים הנפתחים העליונים ולאחר מכן לחץ על מרכז בחירה.
- טעינת המבנה ל- iCn3D: נווטו למציג המבנה 3D מבוסס האינטרנט של iCn3D והקלדו 3FGU בתיבה קלט MMDB או מזהה PDB כדי לטעון את הקובץ.
- זיהוי הליגנדים באתר הפעיל: רחף מעל ניתוח בתפריט הנפתח ולאחר מכן לחץ על Seq. וביאורים. הרצפים, במקרה זה חלבונים וכימיקלים / יונים / מים, מוצגים בטבלה מוערמת. גלול מטה כדי לראות את האתר הפעיל ליגנדים ANP, BGC ו- Mg המפורטים. במציג המבנה, רחף מעל הליבנדים באתר הפעיל (המוצגים כמקלות במרכז קריקטורת החלבון) כדי להציג את שמותיהם.
- התאמת הייצוג: אין צורך בהתאמות ראשוניות עבור פרוטוקול זה.
- בחירת שאריות בתוך 5 Å כדי להגדיר אתר פעיל: כדי לבחור את הליגנדים, השתמש בתפריט הנפתח בחר ולחץ על בחר בתלת-ממד. ודא כי שאריות נבדק .
- כדי לבחור את הליגנדים, החזק את לחצן ALT במחשב או בלחצן האפשרות ב- Mac ולחץ על הליגנד הראשון (למשל, BCG) באמצעות העכבר או משטח המגע. לאחר מכן, לחץ על בקרה ולחץ על ליגנדים ANP ו- MG כדי להוסיף אותם לבחירה.
הערה: הליגנדים יודגשו בצהוב כשהם נבחרים. - שמור בחירה זו באמצעות התפריט הנפתח: לחץ על בחר > שמור בחירה והשתמש בלוח המקשים כדי להזין שם בחלון המוקפץ (למשל, 3Ligands), ולאחר מכן לחץ על שמור. החלון הנפתח בחר ערכות יופיע כעת.
הערה: אם הבחירה שגויה, לחץ על בחר > נקה בחירה. - בחר את השאריות בתוך 5 Å של ligands: בתפריט הנפתח, לחץ על בחר > לפי מרחק. בתפריט המוקפץ שמופיע, שנה את הפריט השני (Sphere עם רדיוס), ל- 5 Å על-ידי הקלדה בבלוק. לחץ על המילה הצגבקופסה ולאחר מכן סגור את החלון על-ידי לחיצה על סימן הצלב בפינה השמאלית העליונה.
הערה: בתפריט המוקפץ המופיע בשלב 2.4.3, השאר את הסט הראשון עם הקלט "נבחר" ואת הקבוצה השנייה כ"לא נבחרה". שים לב שהאטומים/המבנים בתוך 5 Å הופכים מסומנים בזוהר צהוב בעת לחיצה על הצג. - שמור את האתר הפעיל 5 Å באמצעות התפריט הנפתח: רחף מעל בחר ולחץ על שמור בחירה, הזן שם בחלון המוקפץ באמצעות המקלדת (למשל, 5Ang) ולחץ על שמור.
- לאחר מכן צור בחירה חדשה המשלבת את שתי הקבוצות (5Ang ו- 3Ligands): בתפריט המוקפץ בחירת ערכות, הקישו ctrl (PC) או הקישו על הפקודה (Mac) 5Ang ו- 3Ligands. לחץ על בחר > שמור בחירה, השתמש בלוח המקשים כדי להקליד שם (למשל, 5AFull) ולאחר מכן לחץ על שמור.
- כדי לבחור את הליגנדים, החזק את לחצן ALT במחשב או בלחצן האפשרות ב- Mac ולחץ על הליגנד הראשון (למשל, BCG) באמצעות העכבר או משטח המגע. לאחר מכן, לחץ על בקרה ולחץ על ליגנדים ANP ו- MG כדי להוסיף אותם לבחירה.
- מראה את האינטראקציות של האנזים עם ליגנדים האתר הפעיל כגון קשרי מימן: לרחף מעל ניתוח בתפריט הנפתח ולחץ על אינטראקציות. יופיע תפריט מוקפץ מקיף של כל האינטראקציות הלא-שיתוף.
- בטל הכל מלבד תיבות הסימון "קשרי מימן" ו"גשר מלח / יוני". לחץ על 3Ligands כדי לבחור את הסט הראשון ו 5Ang עבור הסט השני. לחץ על הטקסט בקופסה שקורא אינטראקציות תצוגה תלת-ממדיות. סגור את החלון על-ידי לחיצה על השלט הצולב בפינה השמאלית העליונה.
הערה: אנשי הקשר/אינטראקציות מייצגים ככל הנראה אינטראקציית דיפול המושרה בדיפול, מה שלעתים קרובות הופך את התצוגה לאומה. אם תרצה, לשנות את המרחק עבור כל סוג של אינטראקציה. - כדי להציג קשרי מימן בלבד, לחץ על 5המול בחלון המוקפץ של ערכות נבחרות. לאחר מכן, רחף מעל ניתוח בתפריט הנפתח ולאחר מכן לחץ על Chem. איגוד > הצג.
- בטל הכל מלבד תיבות הסימון "קשרי מימן" ו"גשר מלח / יוני". לחץ על 3Ligands כדי לבחור את הסט הראשון ו 5Ang עבור הסט השני. לחץ על הטקסט בקופסה שקורא אינטראקציות תצוגה תלת-ממדיות. סגור את החלון על-ידי לחיצה על השלט הצולב בפינה השמאלית העליונה.
- הצגת שרשראות הצד כמקלות והצגה/התאמה של מולקולות המים הפעילות באתר: השתמש בתפריט המוקפץ Select קובע ולחץ על 5AFull. לאחר מכן, בתפריטים הנפתחים, לחץ על סגנון > שרשראות צדדיות > מקל. להחלת צביעת CPK לחץ על צבע > אטום. לבסוף, לחץ על סגנון > מים > כדורים (אם אתה מעדיף מולקולות מים גדולות יותר).
- פישוט המבנה: בתפריט המוקפץ Select sets, לחץ על 5AFull. לאחר מכן, בתפריטים הנפתחים, לחץ על הצג > הצג בחירה (רק כדי לראות את אתר האיגוד 5AFull). לאחר מכן, לחץ על סגנון > חלבונים > מקל (כדי להציג את שרשרת החלבון כמו מקל במקום סרט).
- כדי לצבוע את אטומי הפחמן של הליגנדים בצבע מנוגד, לחצו על כימיקלים בחלון הנפתח 'בחר ערכות'. לאחר מכן, בתפריט הנפתח, לחץ על הצג > הצג בחירה. לחיצה הבאה על בחר > בחר על 3D (להבטיח "אטום" מסומן). באמצעות הפקדים המתוארים בשלב 2.4.1, השתמש בעכבר ובמקלדת כדי לבחור את כל אטומי הפחמן ב- BGC וב- ANP. לאחר מכן, בתפריט הנפתח לחץ על צבע > Unicolor > ציאן > ציאן.
- כדי להפעיל מחדש את האתר הפעיל כולו, השתמש בחלון המוקפץ בחר ערכות כדי ללחוץ על 5AFull. לאחר מכן, בתפריט הנפתח, לחץ על הצג > הצג בחירה.
- תיוג ליגנדים ושרשראות צד מימן מכובסות: השתמש בחלון המוקפץ Select sets כדי לבחור Interface_allולאחר מכן, בתפריט הנפתח, לחץ על ניתוח > תווית > לפי שאריות ומספר.
הערה: יהיה עליך לבחירה מחדש לפי שאריות ומספר בכל פעם שתרצה להוסיף תווית, למרות שפריט התפריט כבר ייבדק מתווית קודמת. - שמירת העיבוד בכל שלב כדי לחזור לעבוד עליו או לשתף עם אחרים: בתפריט הנפתח, לחץ על קובץ > שתף קישור. העתק את כתובת ה- URL הקצרה (לדוגמה: https://structure.ncbi.nlm.nih.gov/icn3d/share.html?r83NqCz41bu7cmcs8) והדבק אותה בדפדפן.
- שמירת תמונה להטבעה או להדפסה: בתפריט הנפתח, לחץ על בחר > החלף מצב סימון. לאחר מכן, לחץ על סגנון > רקע > לבן. לבסוף, לחץ על קובץ > שמור קבצים > iCn3D PNG Image ולבחור את הגודל הרצוי.
3. פרוטוקול חמול
הערה: פקדי משטח עקיבה ועכבר: כדי לסובב, לחץ וגרור (עכבר: לחץ וגרור שמאלה). כדי להגדיל את התצוגה: גלול אנכית באמצעות שתי אצבעות (עכבר: הזזה + לחיצה שמאלית + גרור אנכית). כדי לתרגם (כלומר, להזיז את המבנה כולו) פקד + alt + לחץ וגרור (מחשב), שליטה + אפשרות + לחץ וגרור (Mac). כדי למרכז מחדש: הסט + לחיצה כפולה בשטח הריק של חלון מציג המבנה.
- טעינת המבנה לתוך Jmol: השתמש בתפריט הנפתח בחלק העליון של GUI כדי להגדיר את סביבת העבודה עם המבנה על ידי לחיצה על קובץ > מסוף. לאחר מכן, לחץ על קובץ > לקבל PDB. בחלון המוקפץ, הקלד: 3fgu
לאחר מכן, לחץ על אישור.
הערה: לחלופין, השתמש במסוף Jmol כדי לטעון את המבנה, על ידי הקלדה: עומס = 3fgu
הערה: לאחר הזנת פקודת שורה מוקלדת כלשהי, הקש על חזור בלוח המקשים כדי לבצע אותה. - התאמת הייצוג: פתח את התפריט המוקפץ על-ידי לחיצה באמצעות לחצן העכבר הימני (או על הפקד + לחץ) במקום כלשהו בחלון מציג המבנה.
- כדי לשנות את החלבון לייצוג מצויר, בתפריט המוקפץ, לחצו על 'בחר >'בחירת Halos' . לאחר מכן, לחץ על בחר חלבון > > הכל. לבסוף, לחץ על סגנון > ערכת > קריקטורה.
הערה: הילה של בחירה מציבה קו מיתאר צהוב (זוהר) סביב כל האטומים שנבחרו. - השתמש בתפריט הנפתח העליון כדי להסתיר את המים על-ידי לחיצה על הצג > בחר > מים. לאחר מכן, לחץ על הצג > אטום > ללא, ולבסוף לחץ על הצג > בחר > ללא.
- כדי לשנות את החלבון לייצוג מצויר, בתפריט המוקפץ, לחצו על 'בחר >'בחירת Halos' . לאחר מכן, לחץ על בחר חלבון > > הכל. לבסוף, לחץ על סגנון > ערכת > קריקטורה.
- זיהוי הליגנדים באתר הפעיל: השתמש בעכבר כדי להגדיל את התצוגה באתר הפעיל ולאחר מכן השתמש בפקודות בteps המשניות כדי להציג את הליגנדים כמקלות.
הערה: שמות ליגנד מופיעים במסוף Jmol בעת טעינת הקובץ. ניתן גם להציג שמות ליגנד מאוגדים באמצעות התפריט המוקפץ, על-ידי לחיצה על בחר > הטרו > על ידי HETATM.- רחף מעל הניגנדים עם העכבר כדי להציג את שמותיהם. האתר הפעיל נמצא בסמוך למרכז המבנה; הליגנדים MG, BGC ו- ANP ממוקמים באתר הפעיל.
- בחר את ה- BCG וה- ANP של ליגנדים: באמצעות מסוף Jmol, הקלד:
בחר BGC, ANP - כדי להציג את ה- BCG וה- ANP של ligand כמקלות, השתמש בתפריט המוקפץ ולחץ על סגנון > ערכת > מקלות.
- בחירת שאריות בתוך 5 Å כדי להגדיר אתר פעיל: במסוף Jmol, הקלד את הפקודה הבאה כדי לבחור אטומים בתוך 5 Å משלושת הליגנדים:
בחר בתוך (5, (bgc,anp,mg))- כדי לבחור שאריות מלאות של חומצות אמינו, הקלד את הטקסט הבא בקונסולה והקש Enter
בחר בתוך (קבוצה, נבחר)
הערה: קונסולת Jmol היא הדרך הטובה ביותר לבחור את השאריות בתוך 5 Å.
- כדי לבחור שאריות מלאות של חומצות אמינו, הקלד את הטקסט הבא בקונסולה והקש Enter
- הצגת שרשראות הצד כמקלות והצגה/התאמה של מולקולות המים הפעילות באתר: לחץ באמצעות לחצן העכבר הימני כדי להעלות את התפריט המוקפץ, ורחף מעל סגנון > ערכת > מקלות.
הערה: שלב 3.5 מציג את הרשתות הצדדיות הפעילות של האתר בייצוג מקל. עדיין יהיו כמה הילה ריקה במבנה המייצגות את מולקולות המים באתר הפעיל.- במסוף Jmol, בצע מחדש את הפקודה הבאה:
בחר בתוך (5, (bgc,anp,mg))
הערה: כדי לבצע מחדש פקודה, לחץ בתוך המסוף ולאחר מכן השתמש במקשי החצים בלוח המקשים עד שמופיעה פקודה זו ולחץ על Enter כדי לבצע אותה מחדש. - כדי להציג את אטומי מולקולת המים, הסר את הליבנדים והחלבון מהבחירה על-ידי הקלדת שתי הפקודות הבאות:
בחר הסר חלבון קבוצתי
בחר להסיר הטרו קבוצתי ולא מים - כדי להציג את מולקולות המים, לחץ בתפריט הנפתח הצג. רחף מעל אטום ולחץ על 20% ואן דר ואלס. יוני המגנזיום הירוקים עדיין יוצגו כמקלות. הצג את יון המגנזיום בייצוג התחום הנפוץ יותר על-ידי הקלדת הפקודות הבאות בקונסול Jmol:
בחר Mg
מילוי שטח 50% - צביעה מחדש של הליגנדים כדי להבחין ביניהן לבין החלבון: בקונסולת Jmol, הקלד את הטקסט הבא כדי לבצע פקודה שצבעת מחדש את הליגנדים בערכת צבעים בהירה יותר:
בחר (bgc,anp) ופחמן; צבע [211,211,211]
בחר (bgc, anp) וחמצן; צבע [255,185,185]
בחר (bgc, anp) וחנקן; צבע [150,210,255]
בחר (bgc,anp) וזרחן; צבע [255,165,75]
בחר מ"ג; צבע פליגרין
- במסוף Jmol, בצע מחדש את הפקודה הבאה:
- הצגת האינטראקציות של האנזים עם ליגנדים האתר הפעיל: באמצעות קונסולת Jmol, בצע כל שורה של הפקודה הבאה:
להגדיר קשירה (ANP, BGC, MG)
בחר בתוך (5, (bgc,anp,mg))
בחר להסיר הטרו קבוצתי ולא מים- כדי להציג קווים להמחשת קשרי מימן, הקלד פקודה זו בקונסולת Jmol:
לחבר 3.3 (קשירה ו (חמצן או חנקן)) (נבחר (חמצן או חנקן)) צהוב יתד
לאחר מכן, שנה את עובי השורות על-ידי הקלדת הפקודה הבאה במסוף:
בחר הכל; יתד 0.1; בחר ללא
- כדי להציג קווים להמחשת קשרי מימן, הקלד פקודה זו בקונסולת Jmol:
- פישוט המבנה: כדי להסתיר את הקריקטורה של החלבון ולנקות את הבחירה, בקונסולת Jmol, הקלד:
בחר הכל; קריקטורה כבויה; בחר ללא - תיוג ליגנדים ושרשראות צד מימן ממולאות: בחלון המוקפץ, לחץ על הגדר איסוף > בחר אטום. לחץ על אטום באחד משאריות מימן מכובס. חומצות האמינו ומספרי השאריות מופיעים בקונסולה. לאחר מכן, השתמש במסוף כדי להקליד תווית, לדוגמה:
תווית דבק-256 - שמירת העיבוד בכל שלב כדי לחזור לעבוד עליו או לשתף עם אחרים: בתפריט העליון, לחץ על סמל המצלמה. הקלד שם קובץ ובחר מיקום לשמירה.
הערה: קובץ JPEG מיוצא (.jpg) מכיל את המידע עבור תמונה כפי שהיא מופיעה בחלון התצוגה בעת הייצוא, כמו גם את המצב הנוכחי של המודל. כדי לטעון מחדש את הדגם, פתח את Jmol וגרור את קובץ JPEG שנשמר לחלון התצוגה Jmol. - שמירת תמונה להטבעה או להדפסה: במסוף Jmol, צבע מחדש את הרקע ללבן, על-ידי הקלדה:
רקע לבן
כמו בשלב 3.9, לחץ על סמל המצלמה ושמור את הקובץ.
4. פרוטוקול PyMOL
הערה: פקדי משטח עקיבה ועכבר: כדי לסובב, לחץ וגרור (עכבר: לחץ וגרור שמאלה). כדי להגדיל את התצוגה, לצבוט ולהתפשטות (עכבר: לחץ באמצעות לחצן העכבר הימני וגרור). כדי לתרגם (כלומר, להזיז את המבנה כולו), שלוט + לחץ וגרור (עכבר: פקודה + לחיצה שמאלית וגרור). למרכז מחדש, גנו לחלונית העצמים הימנית ולחצו על > או על מרכז.
- טעינת המבנה לתוך PyMOL: בשורת הפקודה ליד החלק העליון של GUI (לפני "PyMOL>"), הקלד:
להביא 3FGU
הערה: לאחר הזנת פקודת שורה מוקלדת כלשהי, הקש על חזור בלוח המקשים כדי לבצע אותה. - התאמת הייצוג: בחלונית השמות/האובייקטים בצד ימין של חלון PyMOL, מימין ל- "3FGU" לחץ על H > Waters.
- זיהוי הליגנדים באתר הפעיל: הפעל תחילה את מציג הרצף על-ידי לחיצה על התפריט הנפתח העליון: הצג > רצף.
- גלול את הסרגל האפור ימינה עד שתמצא את שמות הליגנד (BCG, ANP, MG, K).
הערה: ישנם שני ייצוגים, סרט מצויר ומקלות; הליגנדים מוצגים כמקלות. ודא שמצב הבחירה בפקדי העכבר בחלונית השמאלית התחתונה מוגדר למצב תצוגה של שאריות ו-3 לחצנים על-ידי לחיצה על שמות אלה כדי לעבור בין האפשרויות. - באמצעות העכבר, סובב והגדל את התצוגה כדי להפוך את הליבנדות לגלויות.
- גלול את הסרגל האפור ימינה עד שתמצא את שמות הליגנד (BCG, ANP, MG, K).
- בחירת שאריות בתוך 5Å כדי להגדיר אתר פעיל: כדי לבחור את ligands באתר הפעיל, לחץ על כל אחד מהם (BCG, ANP, MG) במציג המבנה. בחירה חדשה מופיעה בחלונית 'שמות/אובייקטים'; מימין לאובייקט חדש זה בשם "sele", לחץ על לחצן A ולאחר מכן לחץ על שנה שם בתפריט המוקפץ.
הערה: כדי לנקות בחירה לא רצויה, לחץ על החלל הריק במציג המבנה כדי לבטל את הבחירה.- באמצעות המקלדת, מחק את האותיות "sele" המופיעות בצד השמאלי העליון של חלון מציג המבנה, ובמקומו, הקלד:
ליגנדים
הערה: ניתן לבצע שלבים 4.4-4.4.1 באמצעות שורת הפקודה; סוג:
ליגנדים סליל, resn BGC + ANP + MG - השתמש בבחירה זו כדי להגדיר את האזור סביב הליגנדים על-ידי שכפולו הראשון, לחץ על ליגנדים > A > כפול. לאחר מכן, לחץ על sel01 > > שנה
באמצעות לוח המקשים, מחק את האותיות "se101" והקלד:
פעיל - שנה בחירה זו כדי להציג שאריות בתוך 5 Å: בחלונית השמות/האובייקטים, לחץ על > פעיל > שנה > הרחב > ב- 5 A, Residues. לאחר מכן, כדי להראות שאריות אלה כמו מקלות, לחץ על פעיל > S > שוש > מקלות. לבסוף, לחץ על הרווח הריק במציג המבנה כדי לנקות את הבחירה.
הערה: שלב 4.4.3 יכול להיעשות באמצעות שורת הפקודה, הקלד:
סל פעיל, byres כל בתוך 5 של ליגנדים
הצג מקלות, פעיל
- באמצעות המקלדת, מחק את האותיות "sele" המופיעות בצד השמאלי העליון של חלון מציג המבנה, ובמקומו, הקלד:
- הצגת שרשראות הצד כמקלות והצגה/התאמה של מולקולות המים הפעילות באתר: בחלונית השמות/האובייקטים לחץ על ליגנדים > A > כפול. כדי לשנות את שם הבחירה, לחץ על Sel02 > > שנה את שם הקטע הנבחר. מחק את האותיות בתפריט שינוי השם המופיע בצד השמאלי העליון של מציג המבנה והקלד:
active_water- כדי להתאים את הבחירה החדשה כך שתכיל מולקולות מים פעילות באתר, לחץ על active_water > > שנה > סביב אטומים > בתוך 4 אנגסטרום. כדי לשנות זאת עוד יותר ולהגביל למולקולות מים, לחץ על active_water > > שנה > להגביל > לממס. לבסוף, לחץ על active_water > > קבוע מראש > כדור ומקל.
הערה: ה- GUI מאפשר בחירה בתוך 4 Å; פקודות קו מאפשרות בחירה של מרחק מתאים יותר של 3.3 Å עבור מימן מליטה מולקולות מים. לא ניתן להגדיר את ה-van der Waals של הכדורים ב-GUI, אך בחירת "הכדור והמקל" קרובה ל-0.5 Å.
הערה: שלבים 4.5-4.5.1 עשויים להתבצע באמצעות שורת הפקודה, על-ידי הקלדת כל שורה בקוד הבא:
בחר active_water, ((ליגנדים) בסביבות 3.3) ו- (resn HOH)
הצג ספירות, active_water
אלטר active_water, vdw=0.5
לבנות מחדש
- כדי להתאים את הבחירה החדשה כך שתכיל מולקולות מים פעילות באתר, לחץ על active_water > > שנה > סביב אטומים > בתוך 4 אנגסטרום. כדי לשנות זאת עוד יותר ולהגביל למולקולות מים, לחץ על active_water > > שנה > להגביל > לממס. לבסוף, לחץ על active_water > > קבוע מראש > כדור ומקל.
- מראה את האינטראקציות של האנזים עם ליגנדים האתר הפעיל. התקרבות באתר הפעיל על ידי לחיצה על > פעילה > זום. כדי למצוא את קשרי הקוטב בין הליגנדים לאתר הפעיל, לחצו על ליגנדים > > מצא > קשרים קוטביים > לכל האטומים. הצג מרחקים כתוויות על-ידי לחיצה על תוויות > S ligands_polar_contacts >.
- פישוט המבנה: להסתיר את הקריקטורה של החלבון, אשר מסתיר את החלק של החלבון שאינו באתר הפעיל, על ידי לחיצה על 3FGU > H > קריקטורה בלוח השמות / אובייקט. לאחר מכן, הסתר את התוויות של אורך קשר המימן על-ידי לחיצה על ligands_polar_contacts > H > תוויות בחלונית השמות/האובייקט.
- כדי לצבוע את הליגנדים כדי להבדיל אותם מהחלבון, לחץ על ליגנדים > C > By Element > CHNOS ובחר באפשרות שבה "C" הוא ציאן (כחול בהיר).
הערה: ניתן לבצע את שלב 4.7.1 באמצעות שורת הפקודה. סוג:
צבע ציאן, ליגנדים
צבע אטומי, ליגנדים ואלפים C
- כדי לצבוע את הליגנדים כדי להבדיל אותם מהחלבון, לחץ על ליגנדים > C > By Element > CHNOS ובחר באפשרות שבה "C" הוא ציאן (כחול בהיר).
- תיוג ליגנדים ושרשראות צד מימן מכובסות: בחלונית השמות/האובייקטים, על הכפתורים שמימין לכל שם אובייקט, לחץ על > L > שאריות .
- שמירת העיבוד בכל שלב כדי לחזור לעבוד עליו או לשתף עם אחרים: בתפריט הנפתח לחץ על קובץ > שמור הפעלה בשם. לאחר מכן, בחר מיקום בחלון המוקפץ, הקלד שם קובץ ולחץ על שמור.
- שמירת תמונה להטבעה או להדפסה: תחילה, שנה את הרקע ללבן בתפריט הנפתח על-ידי לחיצה על הצג > רקע > לבן. יצא את התמונה כקובץ חדש, על-ידי לחיצה על קובץ > ייצוא תמונה > PNG.
תוצאות
פרוטוקול שבוצע בהצלחה עבור כל אחת מהתוכניות יביא למודל מולקולרי המוגדל באתר הפעיל, עם שאריות אתר פעילות ולידות המוצגות כמקלות, קריקטורת החלבון מוסתרת ולידות המוצגות עם ערכת צבעים מנוגדת. שאריות חומצת אמינו אינטראקציה צריך להיות מתויג עם המזהים שלהם, ו מימן מליטה ואינטראקציות יוניות מוצג עם קווים. נוכחותן של תכונות אלה יכולה להיקבע על ידי בדיקה חזותית של המודל.
כדי להקל על בדיקה זו ולאפשר למשתמש לקבוע אם הוא ביצע את שלבי הפרוטוקול כראוי, סיפקנו דמויות מונפשות המציגות תמונה של המבנה לאחר כל שלב. עבור כימרהX, iCn3D, Jmol ו- PyMOL, זה מאויר באיורים 7-10, בהתאמה.
איור 7: פלט פרוטוקול כימרהX. דמות אנימציה הממחישה את השלבים 1.1-1.8 של פרוטוקול כימרהX. אנא לחץ כאן כדי להוריד נתון זה.
איור 8: פלט פרוטוקול iCn3D. איור מונפש הממחיש את השלבים 2.1-2.8 של פרוטוקול iCn3D. אנא לחץ כאן כדי להוריד נתון זה.
איור 9: פלט פרוטוקול Jmol. דמות אנימציה הממחישה את השלבים 3.1-3.8 של פרוטוקול Jmol. אנא לחץ כאן כדי להוריד נתון זה.
איור 10: פלט פרוטוקול PyMOL. דמות אנימציה הממחישה את השלבים 4.1-4.8 של פרוטוקול PyMOL. אנא לחץ כאן כדי להוריד נתון זה.
השגיאה הנפוצה ביותר שיכולה להשפיע על התוצאה של פרוטוקולים אלה היא בחירה שגויה, וכתוצאה מכך חלק מהמבנה מוצג בעיבוד לא רצוי. זוהי בדרך כלל תוצאה של לחיצה שגויה, על המבנה עצמו או באחד מלחצני תפריט התצוגה. דוגמה לתוצאה תת-אופטימלית תהיה מודל המכיל שאריות מחוץ לאתר הפעיל המוצג כמקלות. המשתמש יכול להתחיל לנתח אם שגיאה זו התרחשה על-ידי בדיקה חזותית של השאריות המוצגות כמקלות והבטחה שהם נמצאים בקרבת ליגנדים הפעילים של האתר. שיטה מתקדמת להערכת האם השאריות המוצגות נמצאות בטווח של 5Å מהליגנדים הפעילים של האתר היא להשתמש בכלי המדידה המובנים בכל תוכנית כדי למדוד את המרחק בין ליגנד סמוך לשאריות האתר הפעילות. כלי המדידה הם מעבר להיקף כתב היד הזה; עם זאת, אנו מעודדים משתמשים מעוניינים לחקור את ערכות הלימוד המקוונות הרבות המפרטות סוג זה של ניתוח.
אנו מציגים דוגמה ספציפית לביצוע תת-אופטימלי של פרוטוקול זה, הנובעת מלחיצה שגויה בחלונית שמות/אובייקטים ב- PyMOL. שגיאה זו מציגה את החלבון כולו כמקלות, במקום להציג רק את האתר הפעיל באמצעות ייצוג זה, כפי שמודגם באיור 11.
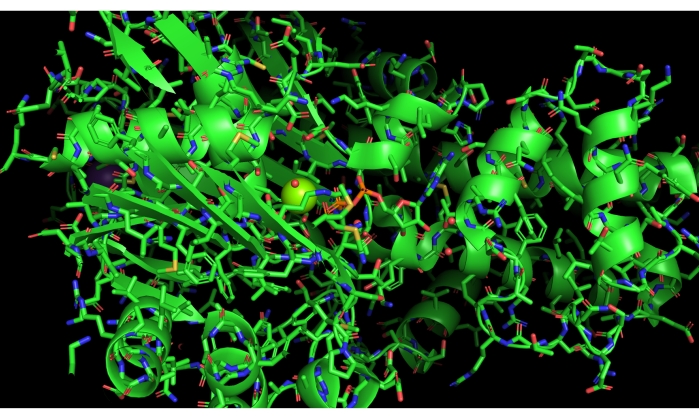
איור 11: תוצאה שלילית. דוגמה לתוצאה שלילית. בחירת קריקטורה מלאה ב- PyMOL והצגת מקלות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
כדי לפתור בעיות, המשתמש יצטרך להסתיר את המקלות עבור הדגם כולו (שכותרתו 3FGU בחלונית השמות/האובייקטים), ולאחר מכן להציג את ייצוג המקל עבור הבחירה בשם "פעילה", באמצעות לחצנים/פקודות הסתרה והצגה ב- PyMOL. שחזור המודל מסוג זה של שגיאה הוא פשוט יחסית ברגע שהמשתמש מסוגל ליצור בחירות מתאימות עבור חלקים שונים של המודל ולהציג ולהסתיר אותם ביעילות. מפתה להפעיל מחדש את הפרוטוקול ולעבוד על השלבים בפעם אחרת; עם זאת, אנו מעודדים את המשתמש לא לפחד ללכת "מחוץ לתסריט" ולהתנסות עם המודל. מניסיוננו, עבודה באמצעות שגיאות תצוגה מקלה על התקדמות בהבנת תוכנית הדוגמנות.
תצוגה זה לצד זה של הפלט הסופי מפרוטוקול שבוצע בהצלחה עבור כל תוכנית מוצגת באיור 12. התצוגות מכוונות באופן דומה כדי לאפשר למשתמש להשוות את המראה של המודלים שנוצרו בתוכניות שונות.
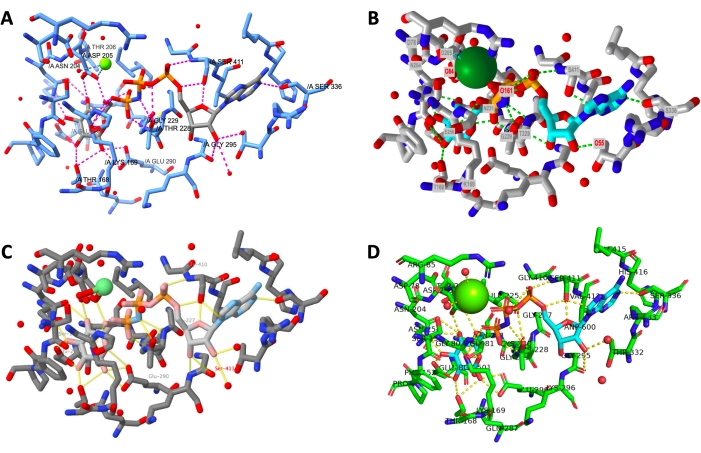
איור 12: השוואת מבנה סופית בין תוכניות. השוואה של המבנה של כל עיבוד אתר פעיל בסוף הפרוטוקול. A: כימרהקס, B: iCn3D, ג: Jmol, D: PyMOL. תווית האתר הפעילה PyMOL כוללת את כל שאריות האתר הפעילות ואת הליבנדים. לתפוקות האחרות יש רק שרשראות צד מימן מתויגות. אנא לחץ כאן כדי להציג גירסה גדולה יותר של איור זה.
Discussion
פרוטוקול זה מתאר תהליך בן עשרה שלבים למידול של אתר פעיל אנזים, החל על ארבע תוכניות פופולריות למידול ביומולקולרי. השלבים הקריטיים של הפרוטוקול הם: זיהוי הליגנדים באתר הפעיל, בחירת שאריות בתוך 5 Å להגדרת אתר פעיל, והצגת האינטראקציות של האנזים עם ליגנדים האתר הפעיל. הבחנה בין הליבנדים הרלוונטיים לתפקוד הביולוגי היא בעלת חשיבות עליונה, שכן זה מאפשר למשתמש להגדיר את שאריות חומצת האמינו בתוך 5 Å שיכול לשחק תפקיד בכריכת ליגנדים. לבסוף, שימוש בתוכנית להצגת אינטראקציות מולקולריות מאפשר למשתמש לפתח את הכישורים הדרושים כדי להבין את האינטראקציות המולקולריות המקדמות כריכה.
מגבלה של פרוטוקולי מידול מולקולרי מבוססי מחשב היא התלות בפקודות ובתחביר ספציפיים. בעוד פרוטוקולים ביוכימיים עשויים להיות סובלניים לשינויים קטנים בהליך, חקירות מבוססות מחשב עשויות להניב מוצרים סופיים שונים לחלוטין אם ההליך אינו דבק מקרוב. הדבר חשוב במיוחד בעת שימוש בממשקי שורת פקודה שבהם נדרש תחביר ספציפי לתוכנית כדי להשיג פלט מסוים, ושינוי חסר משמעות לכאורה בפיסוק או באותיות רישיות עלול לגרום לפקודה להיכשל. ישנם אתרי Wiki ומדריכים שונים עבור כל תוכנית, שבהם משתמש יכול למצוא ולפתור בעיות של קלט שורת פקודה; המשתמש צריך לשים לב היטב לפרטים של תחביר הפקודה. למרות שרוב תוכניות ההדמיה המולקולרית כוללות פקודות ביטול, בשל המורכבות של הממשקים, פקודת הביטול לא תמיד הופכת בנאמנות את השלב האחרון שהוצא להורג. לכן, שמירת מצב העבודה הנוכחי לעתים קרובות מעודדת, במיוחד עבור משתמשים חדשים.
מגבלות נוספות יכולות לנבוע מהנתונים המשמשים ליצירת המודל עצמו. בעוד שהסטנדרטים הטבועים בבנק נתוני החלבון מבטיחים רמה מסוימת של עקביות, משתמשים בתוכניות הדמיה מולקולרית נתקלים לעתים קרובות בהשפעות בלתי צפויות בעיבוד חלבונים. ראשית, רוב המבנים נקבעים באמצעות קריסטלוגרפיה של קרני רנטגן, המספקת דגם יחיד של החלבון; עם זאת, מבני NMR מורכבים לעתים קרובות מדגמים מרובים שניתן לדמיין אחד בכל פעם. שנית, מבנים שנקבעו מניסויי קריסטלוגרפיה או מיקרוסקופ אלקטרונים קריוגניים עשויים להכיל אטומים שאת מיקומם לא ניתן להבהיר ולהופיע כניסולים בייצוגים מסוימים של החלבון. מבני חלבון עשויים להיות קונפורמציות חלופיות של שרשראות צד, אשר, כאשר מוצג עיבוד מקל, מופיעים כמו שתי קבוצות בולטות את אותו עמוד השדרה של חומצת אמינו. אפילו חלקים קצרים של עמוד השדרה עשויים להיות בעלי קונפורמציות חלופיות כאלה, ולעיתים ליגנדים ממוקמים באתר הפעיל ביותר מהתאמה מחייבת אחת.
עבור מבנה גביש, הקואורדינטות 3D שהופקדו כוללות את כל הרכיבים של היחידה האסימטרית, המספקת מספיק מידע כדי לשחזר את היחידה החוזרת של גבישי חלבון. לפעמים, מבנה זה יכיל שרשראות חלבון נוספות בהשוואה לצורה הפעילה ביולוגית של החלבון (למשל, מוטציית המוגלובין עוברית, PDB ID: 4MQK). לעומת זאת, ייתכן שתוכניות מסוימות לא יטענו באופן אוטומטי את כל השרשראות של היחידה הפעילה ביולוגית. לדוגמה, הפרוטאז הראשי SARS-CoV2 (מזהה PDB: 6Y2E) טוען מחצית מהעומעם הפעיל ביולוגית (המורכב משתי שרשראות חלבון) כאשר הוא מובא באמצעות הפקודות המתוארות בפרוטוקול זה ב- ChimeraX, PyMOL ו- Jmol. למרות ששינוי קל של הפקודה יטען את העמעם הפעיל ביולוגית, שיקול זה עשוי שלא להיות פשוט עבור משתמש תוכנית הדוגמנות טירון. בעיה אחרת שיכולה להתעורר היא בזיהוי האתר הפעיל או במצע עצמו. ניסויים קריסטלוגרפיים מתבצעים באמצעות מגוון מולקולות, אשר עשוי להיות מודל לתוך המבנה הסופי. לדוגמה, מולקולות סולפט עשויות לקשור אתרי כריכה פוספט באתר הפעיל, או שהם עשויים לאגד אזורים אחרים שאינם רלוונטיים למנגנון. מולקולות אלה עשויות לטשטש את הזיהוי הנכון של האתר הפעיל עצמו ואף עשויות להציע לתלמיד שהן חלק מהמנגנון.
ככל הנראה, המשתמש ירצה להחיל הליך זה על אתרים פעילים/מחייבים אחרים. כדי ליישם פרוטוקול זה בעבודה העתידית הכרוכה בניתוח של אתרים פעילים חדשים של חלבונים, המשתמש יצטרך לזהות אילו מהליגנדים הכבולים רלוונטיים לתפקוד. חלק מהליגנדים אינם קשורים לתפקוד החלבון, ובמקום זאת הם תוצאה של תנאי הממס או התגבשות המשמשים לביצוע הניסוי (למשל, יון האשלגן הנמצא במודל 3FGU). יש לזהות את ליגנדים המפתח על ידי התייעצות עם כתב היד המקורי. עם תרגול, ובמידת הצורך, הבנה של תחביר הפקודה של הקו, המשתמש יוכל ליישם את הפרוטוקול עבור תוכנית הדוגמנות הרצויה על כל אתר פעיל אנזים, ולדגמן מקרומולקולקולים אחרים לפי בחירתם.
זיהוי וניתוח של מצעים ולידות קשורים הוא מרכזי בהבהרת מנגנונים מולקולריים ומאמצי תכנון תרופות מבוססי מבנה, אשר הובילו ישירות לשיפורים בטיפולים למחלות, כולל תסמונת חיסונית נרכשת (איידס) ו- COVID-1947,48,49,50,51,52 . בעוד שתוכניות ויזואליזציה מולקולרית בודדות מציעות ממשקים וחוויות משתמש שונות, רובן מציעות תכונות דומות. חשוב לפיתוח אוריינות הדמיה ביומולקולרית כי סטודנטים ביוכימיה ברמה העליונה להכיר הדמיה מבנה ואת הכלים כדי ליצור תמונות כאלה4,20,53. זה מאפשר לתלמידים להתקדם מעבר לפרשנות של תמונות דו ממדיות בספרי לימוד ומאמרים בכתבי עת ולפתח בקלות רבה יותר השערות משלהם מנתונים מבניים54, אשר יכין מדענים מתפתחים לטפל בבעיות בריאות הציבור בעתיד ולשפר את ההבנה של תהליכים ביוכימיים.
לסיכום, פרוטוקול זה מפרט מידול אתרים פעיל באמצעות ארבע תוכניות מידול מובילות של מקרומולקולרית בחינם. הקהילה שלנו, BioMolViz, מאמצת גישה לא ספציפית לתוכנה לדוגמנות ביומולקולרית. נמנענו באופן ספציפי מביקורת או השוואה של תכונות התוכנית, אם כי משתמש שדגם כל תוכנית יגלה סביר להניח שהוא מעדיף היבטים מסוימים של מידול מקרומולקולרי בתוכנית אחת לעומת אחרת. אנו מזמינים את הקוראים להשתמש במסגרת BioMolViz, המפרטת את יעדי הלמידה והיעדים המבוססים על הדמיה ביומולקולרית הממוקדים בפרוטוקול זה, ולחקור משאבים להוראת ולמידה של הדמיה ביומולקולרית באמצעות אתר הקהילה BioMolViz http://biomolviz.org.
Disclosures
המחברים מצהירים כי אין להם אינטרסים פיננסיים רלוונטיים או חומריים הקשורים למחקר המתואר במאמר זה.
Acknowledgements
מימון עבודה זו ניתן על ידי הקרן הלאומית למדע:
שיפור מענק החינוך STEM לתואר ראשון (פרס #1712268)
רשתות תיאום מחקר לתואר ראשון בחינוך לביולוגיה לתואר ראשון (פרס # 1920270)
אנו מודים לקרסטן ת'יס, PhD, אוניברסיטת ווסטפילד, על דיונים מועילים על Jmol.
Materials
| Name | Company | Catalog Number | Comments |
| ChimeraX (Version 1.2.5) https://www.rbvi.ucsf.edu/chimerax/ | |||
| Computer | Any | ||
| iCn3D (web-based only: https://www.ncbi.nlm.nih.gov/Structure/icn3d/full.html) | |||
| Java (for Jmol) https://java.com/en/download/ | |||
| Jmol (Version 1.8.0_301) http://jmol.sourceforge.net/ | |||
| Mouse (optional) | Any | ||
| PyMOL (Version 2.4.1 - educational): https://pymol.org/2 educational use only version: https://pymol.org/edu/?q=educational |
References
- Loertscher, J., Green, D., Lewis, J. E., Lin, S., Minderhout, V. Identification of threshold concepts for biochemistry. CBE Life Sciences Education. 13 (3), 516-528 (2014).
- Jaswal, S. S., O’Hara, P. B., Williamson, P. L., Springer, A. L. Teaching structure: Student use of software tools for understanding macromolecular structure in an undergraduate biochemistry course: Teaching structure in undergraduate biochemistry. Biochemistry and Molecular Biology Education. 41 (5), 351-359 (2013).
- Tibell, L. A. E., Rundgren, C. -. J. Educational challenges of molecular life science: Characteristics and implications for education and research. CBE Life Sciences Education. 9 (1), 25-33 (2010).
- Schönborn, K. J., Anderson, T. R. The importance of visual literacy in the education of biochemists. Biochemistry and Molecular Biology Education. 34 (2), 94-102 (2006).
- Anderson, T. R. Bridging the educational research-teaching practice gap: The importance of bridging the gap between science education research and its application in biochemistry teaching and learning: Barriers and strategies. Biochemistry and Molecular Biology Education. 35 (6), 465-470 (2007).
- Schönborn, K. J., Anderson, T. R. Bridging the educational research-teaching practice gap: Foundations for assessing and developing biochemistry students’ visual literacy. Biochemistry and Molecular Biology Education. 38 (5), 347-354 (2010).
- Bateman, R. C., Craig, P. A. Education corner: A proficiency rubric for biomacromolecular 3D literacy. PDB Newsletter. 45, 5-7 (2010).
- Mnguni, L., Schönborn, K., Anderson, T. Assessment of visualization skills in biochemistry students. South African Journal of Science. 112, 1-8 (2016).
- Craig, P. A., Michel, L. V., Bateman, R. C. A survey of educational uses of molecular visualization freeware. Biochemistry and Molecular Biology Education. 41 (3), 193-205 (2013).
- Loertscher, J., Villafañe, S. M., Lewis, J. E., Minderhout, V. Probing and improving student’s understanding of protein α-Helix structure using targeted assessment and classroom interventions in collaboration with a faculty community of practice. Biochemistry and Molecular Biology Education. 42 (3), 213-223 (2014).
- Abualia, M., et al. Connecting protein structure to intermolecular interactions: A computer modeling laboratory. Journal of Chemical Education. 93 (8), 1353-1363 (2016).
- Carvalho, I., Borges, A. D. L., Bernardes, L. S. C. Medicinal chemistry and molecular modeling: An integration to teach drug structure–activity relationship and the molecular basis of drug action. Journal of Chemical Education. 82 (4), 588 (2005).
- Forbes-Lorman, R. M., et al. Physical models have gender-specific effects on student understanding of protein structure-function relationships. Biochemistry and Molecular Biology Education. 44 (4), 326-335 (2016).
- Terrell, C. R., Listenberger, L. L. Using molecular visualization to explore protein structure and function and enhance student facility with computational tools. Biochemistry and Molecular Biology Education. 45 (4), 318-328 (2017).
- Zhang, S., et al. Structure-based drug design of an inhibitor of the SARS-CoV-2 (COVID-19) main protease using free software: A tutorial for students and scientists. European Journal of Medicinal Chemistry. 113390, (2021).
- Roberts, J. R., Hagedorn, E., Dillenburg, P., Patrick, M., Herman, T. Physical models enhance molecular three-dimensional literacy in an introductory biochemistry course. Biochemistry and Molecular Biology Education. 33 (2), 105-110 (2005).
- Jenkinson, J., McGill, G. Visualizing protein interactions and dynamics: Evolving a visual language for molecular animation. CBE Life Sciences Education. 11 (1), 103-110 (2012).
- Bussey, T. J., Orgill, M. What do biochemistry students pay attention to in external representations of protein translation? The case of the Shine–Dalgarno sequence. Chemistry Education Research and Practice. 16 (4), 714-730 (2015).
- Harle, M., Towns, M. H. Students’ understanding of primary and secondary protein structure: Drawing secondary protein structure reveals student understanding better than simple recognition of structures. Biochemistry and Molecular Biology Education. 41 (6), 369-376 (2013).
- Dries, D. R., et al. An expanded framework for biomolecular visualization in the classroom: Learning goals and competencies. Biochemistry and Molecular Biology Education. 45 (1), 69-75 (2017).
- Procko, K., et al. Meeting report: BioMolViz workshops for developing assessments of biomolecular visual literacy. Biochemistry and Molecular Biology Education. 49 (2), 278-286 (2021).
- Wang, J., et al. iCn3D, a web-based 3D viewer for sharing 1D/2D/3D representations of biomolecular structures. Bioinformatics. 36 (1), 131-135 (2020).
- PyMOL . . The PyMOL Molecular Graphics System. Version 2.0. , (2021).
- Goddard, T. D., et al. UCSF ChimeraX: Meeting modern challenges in visualization and analysis. Protein Science. 27 (1), 14-25 (2018).
- Pettersen, E. F., et al. UCSF ChimeraX: Structure visualization for researchers, educators, and developers. Protein Science. 30 (1), 70-82 (2021).
- Petit, P., et al. The active conformation of human glucokinase is not altered by allosteric activators. Acta Crystallographica. Section D. 67 (11), 929-935 (2011).
- Corey, R. B., Pauling, L. Molecular models of amino acids, peptides and proteins. Review of Scientific Instruments. 24, 621-627 (1953).
- Koltun, W. L. Precision space-filling atomic models. Biopolymers. 3 (6), 665-679 (1965).
- Hodis, E., et al. Proteopedia - a scientific 'wiki' bridging the rift between three-dimensional structure and function of biomacromolecules. Genome Biology. 9 (8), 1-10 (2008).
- Prilusky, J., et al. Proteopedia: A status report on the collaborative, 3D web-encyclopedia of proteins and other biomolecules. Journal of Structural Biology. 175 (2), 244-252 (2011).
- . FirstGlance in Jmol Available from: https://www.bioinformatics.org/firstglance/fgij/ (2021)
- Jmol User Design Environment (JUDE). MSOE Centerfor BioMolecular Modeling Available from: https://cbm.msoe.edu/modelingResources/jmolUserDesignEnvironment/#forward (2021)
- Castro, C. R., et al. A practical guide to teaching with Proteopedia. Biochemistry and Molecular Biology Education. 49 (5), 707-719 (2021).
- Berman, H. M., et al. The protein data bank. Nucleic Acids Research. 28, 235-242 (2000).
- . The Protein Data Bank Available from: https://www.rcsb.org/ (2021)
- Wang, Y., et al. MMDB: 3D structure data in Entrez. Nucleic Acids Research. 28 (1), 243-245 (2000).
- . iCn3D Help Page Available from: https://www.ncbi.nlm.nih.gov/Structure/icn3d/docs/icn3d_help.html (2021)
- . MSOE Center for BioMolecular Modeling Jmol Training Guide Available from: https://cbm.msoe.edu/modelingResources/jmolTrainingGuide/started.html (2021)
- . Jmol/JSmol Interactive Scripting Documentation Available from: https://chemapps.stolaf.edu/jmol/docs/ (2021)
- . PyMOL Wiki Available from: https://pymolwiki.org/index.php/Main_Page (2021)
- . PyMOL Advanced Scripting Workshop by Schrödinger Available from: https://pymol.org/tutorials/scripting/index.html (2021)
- . UCSF ChimeraX User Guide Available from: https://www.cgl.ucsf.edu/chimerax/docs/user/index.html (2021)
- . UCSF ChimeraX Tutorials Available from: https://www.rbvi.ucsf.edu/chimerax/tutorials.html (2021)
- Kuntz, I. D. Structure-based strategies for drug design and discovery. Science. 257 (5073), 1078-1082 (1992).
- Hubbard, R. E. . Structure-based drug discovery: an overview. , (2006).
- Patrick, G. L. . An introduction to medicinal chemistry, 6th ed. , (2017).
- Van Montfort, R. L., Workman, P. Structure-based drug design: aiming for a perfect fit. Essays in Biochemistry. 61 (5), 431-437 (2017).
- Holdgate, G. A., Meek, T. D., Grimley, R. L. Mechanistic enzymology in drug discovery: a fresh perspective. Nature Reviews. Drug Discovery. 17 (2), 115-132 (2018).
- Wang, M. Y., et al. SARS-CoV-2: structure, biology, and structure-based therapeutics development. Frontiers in Cellular and Infection Microbiology. 10, (2020).
- White, B., Kim, S., Sherman, K., Weber, N. Evaluation of molecular visualization software for teaching protein structure differing outcomes from lecture and lab: Differing outcomes from lecture and lab. Biochemistry and Molecular Biology Education. 30 (2), 130-136 (2002).
- Canning, D. R., Cox, J. R. Teaching the structural nature of biological molecules: Molecular visualization in the classroom and in the hands of students. Chemistry Education Research and Practice. 2 (2), 109-122 (2001).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved