A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
הערכה של יוביקוויטילציה של מצע על ידי E3 יוביקוויטין-ליגאז בליסאטים של תאי יונקים
In This Article
Summary
אנו מספקים פרוטוקול מפורט לבדיקת יוביקוויטילציה של מצע מסוים ויוביקוויטין-ליגאז E3 בתאי יונקים. קווי תאים HEK293T שימשו לביטוי יתר של חלבונים, המצע הפוליוביקוויטילטיבי טוהר מליאסטים של תאים על ידי מיצוי חיסוני, ונפתר ב- SDS-PAGE. אימונובלוטינג שימש כדי לדמיין את השינוי הפוסט-תרגומי הזה.
Abstract
יוביקוויטילציה היא שינוי פוסט-תרגומי המתרחש בתאים אאוקריוטים שהוא קריטי לוויסות של מספר מסלולים ביולוגיים, כולל הישרדות תאים, התפשטות והתמיינות. זהו תהליך הפיך המורכב מהצמדה קוולנטית של יוביקוויטין לסובסטרט באמצעות תגובת מפל של לפחות שלושה אנזימים שונים, המורכבים מ-E1 (אנזים הפעלת יוביקוויטין), E2 (אנזים מצומד יוביקוויטין) ו-E3 (אנזים יוביקוויטין-ליגאז). קומפלקס E3 ממלא תפקיד חשוב בזיהוי מצעים וביוביקוויטילציה. כאן מתואר פרוטוקול להערכת יוביקוויטילציה של מצע בתאי יונקים באמצעות קו-טרנספקציה חולפת של פלסמיד המקודד את המצע שנבחר, יוביקוויטין ליגאז E3 ויוביקוויטין מתויג. לפני התזה, התאים שעברו טרנספקטציה מטופלים במעכב הפרוטאזום MG132 (קרבובנזוקסי-לאו-לאו-לוצינל) כדי למנוע השפלה של פרוטאסומלי במצע. יתר על כן, תמצית התאים מוגשת למיצוי חיסוני בקנה מידה קטן (IP) כדי לטהר את המצע הפוליוביקוויטילטיבי לצורך זיהוי מאוחר יותר על ידי כתם מערבי (WB) באמצעות נוגדנים ספציפיים לתג יוביקוויטין. לפיכך, מתואר פרוטוקול עקבי ולא מסובך לבדיקת יוביקוויטילציה בתאי יונקים כדי לסייע למדענים לטפל ביוביקוויטילציה של מצעים ספציפיים ובליגאזות יוביקוויטין E3.
Introduction
שינויים לאחר התרגום (PTMs) הם מנגנון חשוב בנוגע לוויסות חלבונים, החיוני להומאוסטזיס של התא. יוביקוויטילציה של חלבונים היא שינוי דינמי ומורכב שיוצר מגוון של אותות שונים וכתוצאה מכך מספר תוצאות תאיות באורגניזמים אאוקריוטים. יוביקוויטילציה היא תהליך הפיך המורכב מהצמדת חלבון יוביקוויטין המכיל 76 חומצות אמינו למצע, המתרחש במפל אנזימטי המורכב משלוש תגובות נפרדות1. הצעד הראשון מאופיין בהפעלת יוביקוויטין, התלויה בהידרוליזה של ATP כדי ליצור יוביקוויטין המקושר לתיואסטר באנרגיה גבוהה בין יוביקוויטין C-terminus לבין שאריות הציסטאין הנמצאות באתר הפעיל של האנזים E1. לאחר מכן, האוביקוויטין מועבר לאנזים E2 ויוצר קומפלקס דמוי תיואסטר עם יוביקוויטין. לאחר מכן, האוביקוויטין מחובר באופן קוולנטי לסובסטרט על ידי E2, או לעתים קרובות יותר, על ידי האנזים E3, שמזהה את הסובסטרט 2,3 ומתקשר איתו. לעיתים, אנזימי E4 (גורמי התארכות שרשרת יוביקוויטין) נחוצים כדי לקדם מכלול שרשרת מולטי-יוביקוויטין3.
ליוביקוויטין יש שבעה שאריות ליזין (K6, K11, K27, K29, K33, K48 ו-K63), מה שמאפשר היווצרות של שרשראות פוליוביקוויטין שיוצרות חוליות נפרדות כדי לייצר מבנים תלת-ממדיים שונים שעומדים להיות מזוהים על ידי מספר חלבונים משפיעים 4,5. לפיכך, סוג שרשרת הפוליוביקוויטין שהוכנסה למצע חיוני כדי להחליט על גורל התא שלו 6,7,8. יתר על כן, המצע יכול להיות גם בכל מקום דרך שאריות N-terminal שלו הנקראות N-degrons. יוביקוויטין-ליגאזות E3 ספציפיות אחראיות לזיהוי N-degron, ומאפשרות פוליוביקוויטילציה של שאריות ליזין סמוכות9.
כיום, ישנם יותר מ -40 מצעים שונים ספציפיים ל- SCF המאופיינים. בין אלה, ניתן למצוא מווסתים מרכזיים של מספר מסלולים ביולוגיים, כולל התמיינות והתפתחות תאים, כמו גם הישרדות תאים ומוות, 10,11,12,13. לפיכך, זיהוי של מצעים ספציפיים של כל יוביקוויטין-ליגאז E3 חיוני לתכנון מפה מקיפה של אירועים ביולוגיים שונים. אף על פי שהזיהוי של מצעים אמיתיים הוא מאתגר מבחינה ביוכימית, השימוש בשיטות מבוססות ביוכימיה מתאים מאוד להערכת ספציפיות השרשרת וההבחנה בין מונו לפוליוביקוויטילציה14. מחקר זה מתאר פרוטוקול מלא לבדיקת יוביקוויטילציה באמצעות קו תאי היונק HEK293T המבטא יתר על המידה את המצע UXT-V2 (מבוטא בכל מקום דמוי פרפולדין דמוי מלווה איזופורם 2) עם קומפלקס E3 יוביקוויטין-ליגאז SCF(Fbxo7). UXT-V2 הוא גורם משותף חיוני לאיתות NF-κB, וברגע שחלבון זה מופל בתאים, הוא מעכב את הפעלת NF-κB המושרה על ידי TNF-α11. לפיכך, כדי לזהות UXT-V2 פוליוביקוויטילציה, מעכב הפרוטאזום MG132 משמש מכיוון שיש לו את היכולת לחסום את הפעילות הפרוטאוליטית של תת-היחידה 26S של קומפלקס הפרוטאזום15. יתר על כן, תמצית התא מוגשת ל- IP בקנה מידה קטן כדי לטהר את המצע, תוך שימוש בנוגדן ספציפי המשותק לשרף אגרוז לצורך זיהוי מאוחר יותר על ידי WB באמצעות נוגדנים נבחרים. פרוטוקול זה שימושי מאוד כדי לאמת יוביקוויטילציה של מצע בסביבה התאית, וניתן גם להתאים אותו לסוגים שונים של תאי יונקים ולקומפלקסים אחרים של יוביקוויטין-ליגאז E3. עם זאת, יש צורך לאמת את המצע שנבדק באמצעות בדיקת יוביקוויטילציה במבחנה גם כן, שכן שני הפרוטוקולים משלימים זה את זה לגבי זיהוי מצעים אמיתיים.
Protocol
הערה: סקירה כללית של פרוטוקול בדיקת יוביקוויטילציה בתאי יונקים מיוצגת באיור 1.
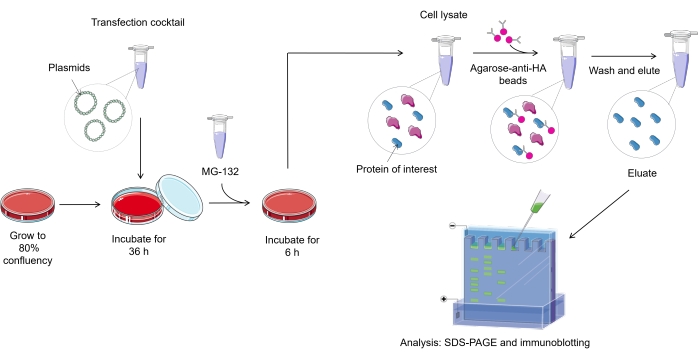
איור 1. סקירה כללית של הליך בדיקת יוביקוויטילציה. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
1. תרבית תאים
- הגדל את קו תאי HEK293T בצלחת תרבית 100 מ"מ שטופלה ב-TC למפגש של 80%-90% במדיית גדילה (Dulbecco's Modified Eagle's Medium (DMEM) עתיר גלוקוז בתוספת 10% סרום בקר עוברי (FBS) ופניצילין (100 יחידות), סטרפטומיצין (100 מיקרוגרם) ו-L-גלוטמין (0.292 מ"ג/מ"ל)). דגירה של התרבית בטמפרטורה של 37 מעלות צלזיוס בחממת תרביות תאים לחות ב-5% CO2.
- כדי להעביר את התאים, שאפו את המדיה מצלחת התרבית באמצעות פיפטה סרולוגית. שטפו את התאים פעם אחת עם 1 מ"ל של מי מלח מעוקרים 1x עם מאגר פוספט (PBS 1x).
- נתק את התאים על ידי הוספת 1 מ"ל של תמיסת טריפסין/EDTA (חומצה אתילנדיאמינט-אצטית). דגירה של המנה בטמפרטורה של 37 מעלות צלזיוס למשך 5 דקות. החייאת התאים באמצעות פיפטה סרולוגית ב-2 מ"ל של מדיית גדילה.
- מעבירים את מתלי התא לצינור וצנטריפוגה טריים ונקיים של 15 מ"ל ב-500 x גרם למשך 5 דקות בטמפרטורת החדר (RT). מוציאים את חומר העל על ידי שפיכתו החוצה בזהירות. יש לבצע החייאה עדינה של כדור התא ב-3 מ"ל של מדיית גדילה על ידי צנרת למעלה ולמטה כדי לקבל תרחיף תאים הומוגני.
- העבר 1 מ"ל של תרחיף התא לתבשיל תרבית 100 מ"מ שטופל ב-TC המכיל 9 מ"ל של מדיית גדילה.
הערה: אם התאים נמצאים בתנאים טובים, כל צלחת תרבית של HEK293T, שבה המפגש הוא 80%-90%, יכולה ליצור שלוש מנות תרבית עם מפגש של 80% יומיים לאחר המעבר.
2. טרנספקציה של תאים
הערה: לא מומלץ לבצע טרנספקטציה של תרבית התאים אם המפגש שאליו הגיעו הוא פחות מ-80%.
- לפני הטרנספקציה, יש לאשר אם התאים נקיים מזיהום ובמפגש הולם עבור טרנספקציות חולפות.
- עבור כל דגימת טרנספקציה, הכן את קומפלקסי הדנ"א-פוליאתילנימין (PEI 1 מיקרוגרם/מיקרול ב-pH 7.2) באופן הבא:
- דילול 3 מיקרוגרם של כל פלסמיד ב-100 מיקרוגרם ליטר של opti-MEM הפחתתי את מדיום הסרום ללא תוספת וערבבתי אותו בעדינות על ידי צנרת התמיסה למעלה ולמטה.
הערה: כאן, התאים עברו טרנספקציה עם 4 מיקרוגרם של כל פלסמיד: וקטור ריק (pcDNA3) או מבני FLAG-Fbxo7 ו- UXT-V2-HA, עם או בלי 6xHis-myc-ubiquitin. תכולת הדנ"א הכוללת הייתה 12 מיקרוגרם. - להפשיר את ה-PEI ב-RT ולהוסיף אותו לתמיסה לאחר שיעור של 3 μL של PEI לכל 1 מיקרוגרם של דנ"א. הומוגניזציה של התמיסה על ידי צנרת למעלה ולמטה. לאחר מכן לדגום אותו במשך 15 דקות ב- RT כדי לאפשר היווצרות של מתחמי DNA-PEI.
הערה: היחס האופטימלי של נפח PEI לכמות DNA משתנה בהתאם לקו התאים שנבחר.
- דילול 3 מיקרוגרם של כל פלסמיד ב-100 מיקרוגרם ליטר של opti-MEM הפחתתי את מדיום הסרום ללא תוספת וערבבתי אותו בעדינות על ידי צנרת התמיסה למעלה ולמטה.
- מוסיפים את הנפח הכולל של מתחמי DNA-PEI לכל מנה המכילה את תרבית התאים ומערבבים אותה בעדינות על ידי נדנדת הצלחת קדימה ואחורה. דגירה של התאים בטמפרטורה של 37 מעלות צלזיוס בחממת תרביות תאים לחות ב-5% CO2.
- החלף את מדיום הגדילה לאחר 5 שעות, מכיוון שחשיפה ממושכת ל-PEI עלולה להיות רעילה לתאי HEK293T. דגירה של התאים בטמפרטורה של 37 מעלות צלזיוס בחממת תרביות תאים לחות ב-5% CO2 למשך 36 שעות.
3. ליזה תאית ומיצוי חיסוני
- לאחר תקופת הדגירה ו-6 שעות לפני התזה של התאים, טפלו בתאים שעברו טרנספקציה עם 10 מיקרומטר של מעכב הפרוטאזום MG-132. שוב, הדגירה של התאים בטמפרטורה של 37 מעלות צלזיוס בחממת תרביות תאים לחות ב-5% CO2.
- שאפו את המדיה מכל צלחת תרבות באמצעות פיפטה סרולוגית ושטפו אותה פעם אחת עם 1 מ"ל של 1x PBS. נתקו את התאים על ידי הוספת 1 מ"ל של טריפסין ודגירה של המנה בטמפרטורה של 37 מעלות צלזיוס למשך 5 דקות. החייאת התאים ב-1 מ"ל של מדיית גדילה.
- העבירו את מתלי התא לצינור 15 מ"ל רענן ונקי והצנטריפוגה שלו ב-500 x גרם למשך 5 דקות ב-RT.
- מוציאים את חומר העל על ידי שפיכתו החוצה בזהירות. יש לבצע החייאה עדינה של גלולת התאים ב-200 μL של מאגר ליזה NP-40 קר כקרח (50 mM Tris-HCl pH 7.2, 225 mM KCl ו-1% NP-40), בתוספת קוקטייל מעכבי פרוטאז ופוספטאז (10 mM NaF ו-1 mM Na3VO4), ולהעביר את התמיסה למיקרו-צינורית נקייה של 1.5 מ"ל.
- דגירה של התא ליזאט במשך 30 דקות על קרח. לאחר הדגירה, צנטריפוגה התא lysates ב 16,900 x g במשך 20 דקות ב 4 °C (64 °F).
- בינתיים, לאזן את חרוזי האגרוז-אנטי-HA עם חיץ ליזה NP-40 קר כקרח. השתמש ב-15 μL של חרוזי אגרוז-אנטי-HA עבור כל דגימה. לשטוף את החרוזים עם 200 μL של חיץ lysis NP-40 על ידי פעימות אותו בצינור microcentrifuge ב 3,000 x g במשך דקה אחת ב 4 °C (66 °F). עם פיפטה, לשאוף ולהשליך את supernatant בזהירות רבה; חזור על תהליך זה שלוש פעמים. לאחר מכן, השאירו את החרוזים מאוזנים על קרח עד לשימוש.
- לאחר צנטריפוגה התא lysates, לשחזר את supernatant. לכמת את תכולת החלבון בליזאט הכולל בשיטת ברדפורד16.
- ודא שכל דגימה הנתונה לתופעה חיסונית מציגה כמות שווה של חלבון. דגירה את הנפח הדרוש של ליאזט התא עם חרוזי האגרוז-אנטי-HA המשוזנים למשך 4 שעות, תוך סיבוב עדין באינקובטור מסתובב בטמפרטורה של 4 מעלות צלזיוס, מה שמאפשר ל- UXT-V2-HA להיקשר לחרוזי האגרוז-אנטי-HA.
- אסוף את החרוזים האגרוזים-אנטי-HA על ידי פעימות שלהם בצינור microcentrifuge ב 3,000 x g במשך דקה אחת ב 4 °C (64 °F). בזהירות לשאוף ולהשליך את supernatant. שטפו את החרוזים שלוש פעמים עם חיץ ליזיס של תאי NP-40 קרים כקרח ופעמיים עם חיץ FLAG/HA קר כקרח (10 mM Hepes pH 7.9, 15 mM MgCl2, 225 mM KCl ו-0.1% NP-40).
- לאחר הכביסה הסופית, הסירו את כל הסופרנאטנט בזהירות באמצעות פיפטה והורידו את החלבון הפוליוביקוויטילטיבי עם פפטיד HA (300 מיקרוגרם/מ"ל) מדולל על חיץ FLAG/HA. דגירה של חרוזי אגרוז-אנטי-HA עם פפטיד HA למשך שעה אחת ב-4 מעלות צלזיוס בפלטפורמת שייקר מתנדנדת.
- סובבו את החרוזים בטמפרטורה של 3,000 x גרם למשך 2 דקות בטמפרטורה של 4 מעלות צלזיוס ופשטו בזהירות את הסופרנטנט המכיל את החלבונים הרב-יוביקוויטינאטים. במידת הצורך, אחסנו את ה-eluate במיקרו-טיוב טרי ונקי בטמפרטורה של -20 מעלות צלזיוס.
- פתרו את האלואטים והתא ליסאט ב-10% SDS-PAGE (נתרן דודציל סולפט-פוליאקרילאמיד ג'ל אלקטרופורזה)17 ואימונובלוטציה.
- במחקר זה בוצעה העברה רטובה WB. עבור סוג זה של העברה, מניחים את הג'ל בכריך העברה המורכב מנייר-ג'ל-נייר-מסנן-ממברנה של מסנן נייר, מרפדים אותו ברפידות ולוחצים אותו יחד על ידי רשת תמיכה. מקם מערכת זו אנכית במיכל מלא במאגר העברה ובין אלקטרודות חוטי נירוסטה /פלטינה. ההעברה מתרחשת במשך 90 דקות ב-150 וולט במאגר העברה רטוב (גליצין 192 mM, tris-base 25 mM, 0.025% SDS, 20% מתנול).
הערה: מאחר שתמציות התאים כומתו (שלב 3.7), הפעל SDS-PAGE עם כמות שווה של חלבון עבור כל דגימה. כמו כן, כדי לפתור את eluate, הפעל נפחים שווים של כל דגימה. - לחקור את קרום האימונובלוט באמצעות נוגדנים נבחרים 11. ודא כי אות מריחה מזוהה באלואט מהמצע הפוליוביקוויטילטיבי שנמשך בתהליך ה- IP על ידי שימוש בנוגדן נגד myc בדגימות המכילות את הליגאז מסוג E3 מסוג בר, את המצע ואת myc-ub. בליזאט של התא (קלט), ודא שהאות מהמצע שנבחר, חלבון Fbxo7, חלבונים שעברו יוביקוויטילציה וחלבון משק בית (למשל, GAPDH ו-β-actin) יזוהו כדי להבטיח את אותה כמות של חלבונים בכל נתיב.
הערה: הדילול עבור כל נוגדן שבו נעשה שימוש הוכן על פי הוראות היצרן.
תוצאות
UXT (תעתיק המבוטא בכל מקום) הוא חלבון דמוי פרפולדין היוצר קומפלקסים של קיפול חלבונים המתבטאים בכל מקום ברקמות עכברים ובני אדם כגון לב, מוח, שרירי שלד, שליה, לבלב, כליות וכבד18. תוארו שני איזופורמים של שחבור של UXT, הנקראים UXT-V1 ו-UXT-V2, המבצעים פונקציות ומיקומים תת-תאיים נפרדים. UXT-V1 מקו?...
Discussion
יוביקוויטילציה היא שינוי פוסט-תרגומי חיוני המווסת את רמותיהם של מספר חלבונים וממלא תפקיד מכריע במסלולי איתות ותהליכים ביולוגיים רבים, ומבטיח סביבה תוך-תאית בריאה. מערכת יוביקוויטין-פרוטאזום (UPS) היא אחד המוקדים העיקריים של המחקר הפרמצבטי האחרון, המספק את האפשרות לייצב מדכאי גידולים או לג...
Disclosures
המחברים מצהירים כי אין ניגוד עניינים.
Acknowledgements
F.R.T נתמך על ידי מענק FAPESP מספר 2020/15771-6 ו- CNPq Universal 405836/2018-0. P.M.S.P ו-V.S נתמכים על ידי CAPES. C.R.S.T.B.C נתמך על ידי מלגת FAPESP מספר 2019/23466-1. אנו מודים לסנדרה R. C. Maruyama (FAPESP 2016/20258-0) על התמיכה בחומר.
Materials
| Name | Company | Catalog Number | Comments |
| 1.5 mL microtube | Axygen | PMI110-06A | |
| 100 mm TC-treated culture dish | Corning | 430167 | |
| 15 mL tube | Corning | 430766 | |
| 96-well plate | Cralplast | 655111 | |
| Agarose-anti-HA beads | Sigma-Aldrich | E6779 | |
| Anti Mouse antibody | Seracare | 5220-0341 | Goat anti-Mouse IgG |
| Anti Rabbit antibody | Seracare | 5220-0337 | Goat anti-Rabbit IgG |
| Anti-Actin antibody | Sigma-Aldrich | A3853 | Dilution used: 1:2000 |
| Anti-Fbxo7 antibody | Sigma-Aldrich | SAB1407251 | Dilution used: 1:1000 |
| Anti-HA antibody | Sigma-Aldrich | H3663 | Dilution used: 1:1000 |
| Anti-Myc antibody | Cell Signalling | 2272 | Dilution used: 1:1000 |
| Bradford reagent | Sigma-Aldrich | B6916-500ML | |
| BSA | Sigma-Aldrich | A9647-100G | Bovine Serum Albumin |
| Cell incubator | Nuaire | NU-4850 | |
| Centrifuge | Eppendorf | 5804R | 500 x g for 5 min |
| ChemiDoc | BioRad | ||
| Digital pH meter | Kasvi | K39-2014B | |
| Dulbecco’s Modified Eagle’s Medium | Corning | 10-017-CRV | High glucose |
| Fetal bovine serum | Gibco | F4135 | Filtrate prior use |
| HA peptide | Sigma-Aldrich | I2149 | |
| HEK293T cells | ATCC | CRL-3216 | |
| Hepes | Gibco | 15630080 | |
| KCl | VWR Life Science | 0365-500G | |
| Kline rotator | Global Trade Technology | GT-2OIBD | |
| MG-132 | Boston Biochem | I-130 | |
| Microcentrifuge | Eppendorf | 5418R | |
| Na3VO4 (Ortovanadato) | |||
| NaF | |||
| Nitrocellulose blotting membrane | GE Healthcare | 10600016 | |
| NP40 (IGEPAL CA-630) | Sigma-Aldrich | I8896-100ML | |
| Optical microscope | OPTIKA microscopes | SN510768 | |
| Opti-MEM | Gibco | 31985-070 | |
| pcDNA3 | Invitrogen | V79020 | For mammalian expression |
| pcDNA3-2xFlag-Fbxo7 | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI | |
| pcDNA3-2xFlag-Fbxo7-ΔF-box | Kindly donated by Dr. Marcelo Damário | Tag 2xFlag (N-terminal). Restriction enzymes: EcoRI and XhoI. Δ335-367 | |
| pcDNA3-UXTV2-HA | Kindly donated by Dr. Marcelo Damário | Tag HA (C-terminal). Restriction enzymes: EcoRI and XhoI | |
| pCMV-6xHis-Myc-Ubiquitin | Kindly donated by Dr. Marcelo Damário | Tag 6x-His-Myc (N-terminal). Restriction enzymes: EcoRI and KpnI | |
| Pen Strep Glutamine 100x | Gibco | 10378-016 | |
| Phosphate buffered saline 10x | AccuGENE | 51226 | To obtain a 1x PBS, dilute the 10x PBS into ultrapure water |
| Polyethylenimine (PEI) | Sigma-Aldrich | 9002-98-6 | |
| Ponceau S | VWR Life Science | 0860-50G | |
| Protease inhibitor cocktail SIGMAFAST | Sigma-Aldrich | S8820 | |
| Rocking Shaker | Kasvi | 19010005 | |
| SDS-PAGE system | BioRad | 165-8004 | |
| Solution Homogenizer | Phoenix Luferco | AP-22 | |
| Trizma base | Sigma-Aldrich | T6066-500G | |
| Trypsine (TrypLe Express) | Gibco | 12605-028 | |
| Western Blotting Luminol Reagent | Santa Cruz Biotechnology | SC-2048 |
References
- Popovic, D., Vucic, D., Dikic, I. Ubiquitination in disease pathogenesis and treatment. Nature Medicine. 20 (11), 1242-1253 (2014).
- Callis, J. The ubiquitination machinery of the ubiquitin system. The Arabidopsis Book. 12, 0174 (2014).
- Koegl, M., et al. A novel ubiquitination factor, E4, is involved in multiubiquitin chain assembly. Cell. 96 (5), 635-644 (1999).
- French, M. E., Koehler, C. F., Hunter, T. Emerging functions of branched ubiquitin chains. Cell Discovery. 7 (1), 6 (2021).
- Komander, D., et al. Molecular discrimination of structurally equivalent Lys 63-linked and linear polyubiquitin chains. EMBO Reports. 10 (5), 466-473 (2009).
- Clague, M. J., Urbé, S. Ubiquitin: Same molecule, different degradation pathways. Cell. 143 (5), 682-685 (2010).
- Davies, B. A., et al. Vps9p CUE domain ubiquitin binding is required for efficient endocytic protein traffic. Journal of Biological Chemistry. 278 (22), 19826-19833 (2003).
- Raasi, S., Wolf, D. H. Ubiquitin receptors and ERAD: A network of pathways to the proteasome. Seminars in Cell and Developmental Biology. 18 (6), 780-791 (2007).
- Pan, M., et al. Structural insights into Ubr1-mediated N-degron polyubiquitination. Nature. 600 (7888), 334-338 (2021).
- Raducu, M., et al. SCF (Fbxl17) ubiquitylation of Sufu regulates Hedgehog signaling and medulloblastoma development. The EMBO Journal. 35 (13), 1400-1416 (2016).
- Spagnol, V., et al. The E3 ubiquitin ligase SCF(Fbxo7) mediates proteasomal degradation of UXT isoform 2 (UXT-V2) to inhibit the NF-κB signaling pathway. Biochimica et Biophysica Acta - General Subjects. 1865 (1), 129754 (2021).
- Teixeira, F. R., et al. Gsk3β and Tomm20 are substrates of the SCFFbxo7/PARK15 ubiquitin ligase associated with Parkinson's disease. Biochemical Journal. 473 (20), 3563-3580 (2016).
- Tan, M. K. M., Lim, H. J., Bennett, E. J., Shi, Y., Harper, J. W. Parallel SCF adaptor capture proteomics reveals a role for SCFFBXL17 in NRF2 activation via BACH1 repressor turnover. Molecular Cell. 52 (1), 9-24 (2013).
- van Wijk, S. J., Fulda, S., Dikic, I., Heilemann, M. Visualizing ubiquitination in mammalian cells. EMBO Reports. 20 (2), 1-18 (2019).
- Kisselev, A. F., Goldberg, A. L. Proteasome inhibitors: From research tools to drug candidates. Chemistry and Biology. 8 (8), 739-758 (2001).
- Bradford, M. A. Rapid and sensitive method for the quantitation of microgram quantities of protein utilizing the principle of protein-dye binding. Analytical Biochemistry. 72 (1-2), 248-254 (1976).
- Laemmli, U. K. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. 228, 726-734 (1970).
- Schröer, A., Schneider, S., Ropers, H. -. H., Nothwang, H. G. Cloning and characterization of UXT, a novel gene in human Xp11, which is widely and abundantly expressed in tumor tissue. Genomics. 56 (3), 340-343 (1999).
- Huang, Y., et al. UXT-V1 facilitates the formation of MAVS antiviral signalosome on mitochondria. The Journal of Immunology. 188 (1), 358-366 (2012).
- Huang, Y., et al. UXT-V1 protects cells against TNF-induced apoptosis through modulating complex II formation. Molecular Biology of the Cell. 22 (8), 1389-1397 (2011).
- Sun, S., et al. UXT is a novel and essential co-factor in the NF-κB transcriptional enhanceosome. The Journal of Cell Biology. 178 (2), 231-244 (2007).
- Huang, X., Dixit, V. M. Drugging the undruggables: Exploring the ubiquitin system for drug development. Cell Research. 26 (4), 484-498 (2016).
- Rajkumar, S. V. Multiple myeloma: 2020 update on diagnosis, risk-stratification and management. American Journal of Hematology. 95 (5), 548-567 (2020).
- Hideshima, T., et al. The proteasome inhibitor PS-341 inhibits growth, induces apoptosis, and overcomes drug resistance in human multiple myeloma cells. Cancer Research. 61 (7), 3071-3076 (2001).
- Tietsche, V., et al. New proteasome inhibitors in the treatment of multiple myeloma. Hematology, Transfusion and Cell Therapy. 41 (1), 76-83 (2018).
- Vassilev, L. T., et al. In vivo activation of the p53 pathway by small-molecule antagonists of MDM2. Science. 303 (5659), 844-848 (2004).
- Kuiken, H. J., et al. Identification of F-box only protein 7 as a negative regulator of NF-kappaB signalling. Journal of Cellular and Molecular Medicine. 16 (9), 2140-2149 (2012).
- Yuan, N., et al. Bafilomycin A1 targets both autophagy and apoptosis pathways in pediatric B-cell acute lymphoblastic leukemia. Haematologica. 100 (3), 345-356 (2015).
- Iconomou, M., Saunders, D. N. Systematic approaches to identify E3 ligase Substrates. Biochemical Journal. 473 (22), 4083-4101 (2016).
- Zhang, Z. R., Bonifacino, J. S., Hegde, R. S. Deubiquitinases sharpen substrate discrimination during membrane protein degradation from the ER. Cell. 154 (3), 609-622 (2013).
- Hunter, T. The age of crosstalk: Phosphorylation, ubiquitination, and beyond. Molecular Cell. 28 (5), 730-738 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved