A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
צינור לחקר המבנים ומסלולי האיתות של קולטני ספינגוזין 1-פוספט
In This Article
Summary
S1P מפעיל את ההשפעות הפיזיולוגיות המגוונות שלו באמצעות תת-משפחת קולטני S1P (S1PRs). כאן, צינור מתואר כדי להרחיב על המבנים והתפקוד של S1PRs.
Abstract
ליזופוספוליפידים (LPLs) הם ליפידים ביו-אקטיביים הכוללים ספינגוזין 1-פוספט (S1P), חומצה ליזופוספטידית וכו '. S1P, תוצר מטבולי של ספינגוליפידים בקרום התא, הוא אחד ה-LPLs המאופיינים בצורה הטובה ביותר המווסת מגוון תגובות פיזיולוגיות תאיות באמצעות מסלולי איתות המתווכים על ידי קולטני ספינגוזין 1-פוספט (S1PRs). זה רמז לכך שמערכת האיתות S1P-S1PRs היא מטרה טיפולית פוטנציאלית יוצאת דופן להפרעות, כולל טרשת נפוצה (MS), הפרעות אוטואימוניות, סרטן, דלקת ואפילו COVID-19. S1PRs, תת-קבוצה קטנה ממשפחת הקולטן המצומד לחלבון G מסוג A (GPCR), מורכבת מחמישה תתי-סוגים: S1PR1, S1PR2, S1PR3, S1PR4 ו-S1PR5. עם זאת, היעדר מידע מבני מפורט מעכב את גילוי התרופה המכוון ל-S1PRs. כאן, יישמנו את שיטת המיקרוסקופיה הקריו-אלקטרונית כדי לפתור את המבנה של קומפלקס S1P-S1PRs, והבהרנו את מנגנון ההפעלה, זיהוי תרופות סלקטיבי וצימוד חלבון G באמצעות מבחנים פונקציונליים מבוססי תאים. קולטני ליזופוספוליפידים אחרים (LPLR) ו- GPCRs יכולים גם הם להיחקר באמצעות אסטרטגיה זו.
Introduction
ספינגוזין-1-פוספט (S1P), תוצר מטבולי של ספינגוליפידים בקרום התא, היא מולקולת איתות ליזופוספטידית הנמצאת בכל מקום ומערבת פעילויות ביולוגיות שונות, כולל סחר בלימפוציטים, התפתחות כלי דם, שלמות האנדותל וקצב לב 1,2,3. S1P מפעיל את ההשפעות הפיזיולוגיות המגוונות שלו באמצעות חמישה תת-סוגים של קולטני S1P (S1PRs 1-5); S1PRs נמצאים במגוון רקמות ומפגינים העדפות ייחודיות לחלבוני Gבמורד הזרם 4,5. S1PR1 מוצמד בעיקר לחלבון Gi, אשר לאחר מכן מעכב את ייצור cAMP; S1PR2 ו-S1PR3 משולבים עם Gi, Gq ו-G12/13, ואות מתמר S1PR4 ו-S1PR5 דרך Gi ו-G12/136.
איתות S1P-S1PR הוא יעד טיפולי קריטי למחלות מרובות, כולל הפרעות אוטואימוניות7, דלקת8, סרטן9 ואפילו COVID-1910. בשנת 2010, פינגולימוד (FTY720) קיבל רישיון כתרופה ראשונה מסוגה המכוונת נגד S1PRs לטיפול בטרשת נפוצה חוזרת (MS)11. עם זאת, הוא מסוגל להיקשר לכל S1PRs למעט S1PR2, בעוד שקשירה לא ספציפית ל- S1PR3 גורמת לבצקת של קליפת המוח, כיווץ כלי הדם והסימפונות ודליפת אפיתל הריאה12. כאסטרטגיה חלופית להגברת הסלקטיביות הטיפולית, נוצרו ליגנדות ספציפיות לתת-סוג עבור הקולטן. סיפונימוד (BAF312) אושרה בשנת 2019 לטיפול בטרשת נפוצהחוזרת 13; הוא מכוון ביעילות ל-S1PR1 ול-S1PR5, בעוד שאין לו זיקה ל-S1PR3, מה שמפגין פחות תופעות לוואי בקליניקה14. בשנת 2020, מנהל המזון והתרופות האמריקאי אישר אוזנימוד לטיפול בטרשת נפוצה15. דווח כי אוזנימוד מחזיק בסלקטיביות גדולה פי 25 עבור S1PR1 מאשר עבור S1PR516. יש לציין כי בהקשר של מגפת COVID-19 הנוכחית, התגלה כי תרופות אגוניסטיות המכוונות ל- S1PRs עשויות לשמש לטיפול ב- COVID-19 על ידי שימוש בטכניקות טיפול אימונומודולטוריות17. בהשוואה לפינגולימוד, האוזנימוד הראה עליונות בהורדת הסימפטומים בחולי COVID-19 וכעת הוא עובר ניסויים קליניים10. הבנת הבסיס המבני והתפקוד של S1PRs מניחה בסיס משמעותי לפיתוח תרופה המכוונת באופן סלקטיבי ל- S1PRs18.
טכניקות רבות משמשות לחקר המידע המבני של ביו-מקרומולקולות, כולל קריסטלוגרפיה של קרני רנטגן, תהודה מגנטית גרעינית (NMR) ומיקרוסקופיית אלקטרונים (EM). נכון למרץ 2022, ישנם יותר מ -180,000 מבנים שהופקדו בבנק נתוני החלבון (PDB), ורובם נפתרו על ידי קריסטלוגרפיה של קרני רנטגן. עם זאת, עם מבנה הרזולוציה הכמעט אטומית הראשונה של TPRV1 (רזולוציה 3.4 Å) שדווחה על ידי ייפאן צ'נג ודיוויד יוליוס בשנת 201319, מיקרוסקופיית קריו-אלקטרונים (cryo-EM) הפכה לטכניקה מרכזית עבור מבני חלבונים, והמספר הכולל של מבני EM PDB היה יותר מ -10,000. התחומים פורצי הדרך הקריטיים הם פיתוח מצלמות חדשות להדמיה המכונות מצלמות לגילוי אלקטרונים ישירים ואלגוריתמים חדשים לעיבוד תמונה. Cryo-EM חוללה מהפכה בביולוגיה של מבנים ובגילוי תרופות מבוססות מבנה בעשורה-20 האחרון. מכיוון שהבנת האופן שבו קומפלקסים מקרומולקולריים ממלאים את תפקידיהם המורכבים בתא החי היא נושא מרכזי במדעי הביולוגיה, לקריו-EM יש פוטנציאל לחשוף קונפורמציות של קומפלקסים מולקולריים דינמיים, במיוחד עבור חלבונים טרנס-ממברניים21. קולטנים מצומדים לחלבון G (GPCRs) הם משפחת-העל הגדולה ביותר של חלבוני טרנס-ממברנה והמטרות של יותר מ-30% מהתרופות המשווקות כיום22. הפיתוח של cryo-EM תרם לפרץ של מבנים ברזולוציה גבוהה של קומפלקסים של חלבוני GPCR-G, המאפשרים קביעה מבנית של מטרות 'בלתי פתירות' שעדיין אינן נגישות לניתוח קריסטלוגרפי של קרני רנטגן בתכנון תרופות23. לפיכך, יישום cryo-EM מספק הזדמנות לקבוע את המבנה התלת-ממדי של GPCRs בתנאים כמעט מקומיים ברזולוציה אטומית קרובה לרזולוציה אטומית24. ההתקדמות ב- cryo-EM מאפשרת לדמיין יסודות מכניסטיים של גירוי או עיכוב GPCR, ותועלת נוספת בחשיפת אתרי הקשירה החדשים ליצירת תרופות ממוקדות GPCR25.
בהסתמך על הצעדים האדירים של טכנולוגיית cryo-EM, זיהינו מבנים של מתחמי איתות S1PR1-, S1PR3 ו-S1PR5-Gi מיוסרים לאחרונה26,27. בבני אדם, S1PRs נמצאים ברקמות שונות ומפגינים העדפות ייחודיות לחלבוני Gבמורד הזרם 4,5. S1PR1 מוצמד בעיקר לחלבון Gi, אשר לאחר מכן מעכב ייצור של 3′,5′-מחזורי של אדנוזין מונופוספט (cAMP). S1PR3 ו-S1PR5 מסוגלים גם לצימוד עם Gi 6,28. מכיוון שהפעלת קולטן מצומד Gi מפחיתה את הייצור של cAMP29, הוכנסה בדיקת cAMP לעיכוב Gi כדי למדוד את השפעות העיכוב של cAMP ללכידת שינויים פונקציונליים26,27. באמצעות גרסה מוטנטית של Photinus pyralis luciferase שבה הוכנס חלבון קושר cAMP, בדיקת cAMP זו מציעה שיטה פשוטה ואמינה לניטור פעילות GPCR באמצעות שינויים בריכוז cAMPתוך תאי 30. זוהי בדיקה תפקודית רגישה ולא רדיואקטיבית וניתן ליישם אותה כדי לנטר את האיתות בזמן אמת במורד הזרם של מגוון רחב של GPCRs למטרות גילוי תרופות31.
כאן, מסופק סיכום של השיטות הקריטיות לפתרון מצבי ההפעלה וזיהוי התרופות של S1PRs, כולל בעיקר מניפולציות cryo-EM ובדיקת cAMP של עיכוב Gi. מאמר זה נועד לספק הדרכה ניסיונית מקיפה לחקירות נוספות של המבנים והתפקודים של GPCRs.
Protocol
1. טיהור קומפלקס חלבונים S1PRs-G
- כדי לטהר את קומפלקס החלבון S1PRs-G האנושי, שכפלו את ה-cDNAs של S1PR1 חסר שאריות C-terminal 338-382, S1PR3 מסוג פראי, S1PR5 שנחתך עם 345-398 ב-C-terminus, ואת ה-Gi1 מהסוג הפראי לווקטור pFastBac1 ול-cDNAs של Gβ1 ו-Gγ2 מהסוג הפראי לווקטור pFastBacdual (טבלת חומרים).
הערה: כל המבנים עבור S1PRs מכילים גם את רצף האותות haemagglutinin (HA) ואחריו תג אפיטופ דגל ב-N-terminus ותג 10x שלו ב-C-terminus. כמו כן, רצף דנ"א סינתטי (טבלת חומרים) לתרגום ליזוזים T4 (T4L) הוכנס לתוך N-terminus של S1PRs על מנת להקל על ביטוי קולטן וטיהור. - הכנת קידוד baculovirus S1PRs, Gi1 ו- Gβ1γ2
- הוסיפו את הווקטורים הרקומביננטיים ל-50 μL של Escherichia coli (E. coli) המוסמך DH5α המאוחסן בצינור של 1.5 מ"ל בטמפרטורה של -80 מעלות צלזיוס, ודגרו על קרח למשך 30 דקות.
- הלם חום התאים בטמפרטורה של 42 מעלות צלזיוס למשך 90 שניות, העבר אותם מיד לקרח וצנן אותם למשך 2 דקות.
- יש לנער את השפופרת בטמפרטורה של 37°C למשך 3-5 שעות לאחר נטילת תוסף עם 300 μL של ציר ליזוגני (LB) בינוני. צלחת 100 μL של תאים לצלחת אגר LB ודגירה ב 37 מעלות צלזיוס על ידי שמירה חשוכה במשך 48 שעות.
- לחסן את המושבה הלבנה ל-5 מ"ל של מדיום LB המכיל 50 מיקרוגרם/מ"ל קנאמיצין, 10 מיקרוגרם/מ"ל טטרציקלין, ו-7 מיקרוגרם/מ"ל גנטמיצין, ותרבית בטמפרטורה של 37 מעלות צלזיוס למשך 16 שעות.
- יש לבודד את הבקמיד הרקומביננטי באמצעות ערכת המיניפרפ פלסמיד (טבלת חומרים) בהתאם להוראות היצרן, לייצור נגיף ה-P0 baculovirus.
הערה: לפני השימוש, הבקמיד המטוהר נותח על ידי PCR עם פריימרים pUC/M13 קדימה ואחורה. עבור ה- PCR, מספר מחזורים = 30 מחזורים, טמפרטורת התכה = 58 ° C, וזמן הארכה = 1 דקה לכל 1 Kb. - הכן P0 baculovirus כפי שתואר בפרוטוקול מוקדםיותר 32.
- תרבית את תאי sf9 (מדיום ESF921) בלוחות של שש בארות וודא שהתאים נמצאים בשלב היומן (1.0-1.5 x 106 תאים למ"ל).
- יש לדלל 8 μL של מגיב טרנספקציה של baculovirus ב-100 μL של המדיום של גרייס, ולדגור במשך 30 דקות בטמפרטורת החדר. יש לדלל 10 מיקרוגרם של הבקמיד הרקומביננטי ב-100 μL של המדיום של גרייס, ולערבב בעדינות. ערבבו את הבקמיד המדולל עם מגיב ההעברה של baculovirus מדולל, ערבבו בעדינות ודגרו במשך 30 דקות בטמפרטורת החדר.
- מוסיפים את התערובת (בקמיד וריאגנט) לתאים (שלב 1.2.6.1), ומצפים אותם בטמפרטורה של 27 מעלות צלזיוס למשך 3 שעות.
- הסר את המדיום של גרייס והחלף אותו ב-2 מ"ל של מדיום תרבית תאים ESF921. צלחת שש באר ב 27 °C ולאסוף ESF921 תרבית תאים בינוני לאחר 5 ימים לאחר טרנספקציה.
- צנטריפוגה ב 500 x גרם ב 4 מעלות צלזיוס במשך 10 דקות כדי להסיר את התאים ואת הפסולת. מעבירים את הסופרנאטנט לצינורות 2 מ"ל. זהו מלאי P0 baculovirus.
- לבודד מלאי וירוסים P1
- העבירו 30 מ"ל של תאי sf9 לבקבוק חרוטי, תרבית בטמפרטורה של 27 מעלות צלזיוס עם רעידות ב-270 סל"ד, וודאו שהתאים מגיעים לשלב יומן (1.0-1.5 x 106 תאים /מ"ל).
- הוסיפו 2 מ"ל של מלאי וירוסים P0 לבקבוק ונערו ב-270 סל"ד למשך 4 ימים בטמפרטורה של 27 מעלות צלזיוס.
- מעבירים את התאים לצינור של 50 מ"ל, צנטריפוגה בגודל 1,800 x גרם למשך 10 דקות ב-4 מעלות צלזיוס כדי להסיר את התאים והפסולת, ומעבירים את הסופר-נאטנט לצינורות של 50 מ"ל. זהו מלאי P1 baculovirus.
- הגבר את מלאי baculoviral
- חזור על שלב 1.2.7 באמצעות 50 מ"ל של תאי sf9 בשלב היומן (1.0-1.5 x 106 תאים למ"ל) ו-1 מ"ל של מלאי P1.
- אחסן את מלאי ה-P2 baculovirus שנוצר ב-4 מעלות צלזיוס, והגן עליו מפני אור.
הערה: אין להגביר את נגיף ה-baculovirus ללא הגבלת זמן, שכן המוטנטים המזיקים נוצרו עם כל מעבר.
- ביטוי של קומפלקס חלבונים S1PRs-G
- תרבית sf9 תאי חרקים להגיע 2.5 x 10 6 תאים / מ"ל צפיפות, להדביק יחד עם P2 baculovirus קידוד S1PRs, Gi1, ו Gβ1γ2 ביחס נפח של 1:2:1, ותרבית שוב ב 27 °C במשך 48 שעות.
- אסוף את התאים על ידי צנטריפוגה ב 700 x g ב 4 ° C למשך 15 דקות, להקפיא אותם בחנקן נוזלי, ולאחסן אותם ב -80 °C לשימוש.
- טיהור חלבונים
- הפשירו את גלולת התא שהושגה בשלב 1.3 בטמפרטורת החדר, ולאחר מכן החזירו אותה למאגר הליזיס (20 mM HEPES pH 7.5, 50 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2) בתוספת 100 מיקרוגרם / מ"ל בנזמידין, 100 מיקרוגרם / מ"ל לאופטין, 100 מיקרוגרם / מ"ל אפרוטינין, 25 mU / mL אפירז, ו 10 μM אגוניסט. ערבבו את תרחיף התאים בטמפרטורת החדר למשך שעתיים כדי לגרום להיווצרות קומפלקס החלבון S1PRs-G.
הערה: אפירז הוא דיפוספוהידרולאז ATP. זה מזרז את הסרת גמא פוספט מ ATP ואת ביתא פוספט מ ADP. - מעבירים את התמיסה לצינורות, צנטריפוגה ב 70,000 x גרם במשך 10 דקות, ולהסיר את supernatant בזהירות. יש להשהות את הכדור בחיץ מסיס (20 mM HEPES pH 7.5, 100 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2, 0.5% (w/v) LMNG, 0.1% (w/v) CHS, 1% (w/v) נתרן כולאט, 10% (v/v) גליצרול).
- מעבירים את המתלים לכוס Dounce והומוגניים את הכדור במלואו. יש להוסיף לתרחיף אגוניסט של 10 מיקרומטר, 4 מ"ג של scFv16, 100 מיקרוגרם/מ"ל בנזמידין, 100 מיקרוגרם/מ"ל לאופטין, 100 מיקרוגרם/מ"ל אפרוטינין ואפיראז של 25 mU/mL, ולערבב בטמפרטורה של 4 מעלות צלזיוס למשך שעתיים.
הערה: שלבי ההומוגניזציה של הכדורים חיוניים לייצור קומפלקס החלבון GPCR-G. - מעבירים את התמיסה לצינורות, וצנטריפוגה ב-100,000 x גרם למשך 30 דקות ב-4 מעלות צלזיוס.
- שיווי משקל מראש של שרף הדגל עם מאגר הכביסה (20 mM HEPES pH 7.5, 100 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2, 10 μM אגוניסט, 0.0375% (w/v) LMNG, 0.0125% (w/v) GDN, 0.01% (w/v) CHS).
- מעבירים את הסופרנטנט לצינורות, ודוגרים עם שרף הדגל ב-4 מעלות צלזיוס למשך שעתיים.
- העמיסו את התערובת הנ"ל על עמוד הזכוכית ושטפו את העמוד עם 50 מ"ל של חיץ כביסה בתוספת 100 מיקרוגרם/מ"ל בנזמידין, 100 מיקרוגרם/מ"ל לאופטין ו-100 מיקרוגרם/מ"ל אפרוטינין.
- אלוקים את העמודה עם 10 מ"ל של חיץ אלוציה המכיל 20 mM HEPES pH 7.5, 100 mM NaCl, 5 mM EDTA, 200 מיקרוגרם / mL פפטיד דגל, 10 μM אגוניסט, 0.0375% (w / v) LMNG, 0.0125% (w / v) GDN, 0.01% (w / v) CHS, 100 מיקרוגרם / מ"ל בנזמידין, 100 מיקרוגרם / מ"ל leupeptin, ו 100 מיקרוגרם / מ"ל אפרוטינין.
- אסוף את קומפלקס החלבון S1PRs-G והתרכז עד 1 מ"ל באמצעות רכז חיתוך של 100 kDa ב-1,300 x גרם ב-4 °C. סנן דרך מסנן 0.22 μM, וצנטריפוגה ב 13,000 x g במשך 10 דקות ב 4 °C כדי להסיר את האגרגטים.
- טען את קומפלקס החלבון S1PRs-G על עמודת סינון ג'ל כרומטוגרפיה (SEC) של אי-הכללת גודל בשיווי משקל מראש עם חיץ SEC המכיל 20 mM HEPES pH 7.5, 100 mM NaCl, אגוניסט 10 μM, 100 Μm TCEP, 0.00375% (w/v) LMNG, 0.00125% (w/v) GDN ו-0.001% (w/v) CHS בקצב זרימה של 0.5 מ"ל/דקה ב-4 °C.
- אסוף את שברי השיא והתרכז באמצעות רכז חיתוך של 100 kDa ב- 1,300 x g ב- 4 °C עבור cryo-EM.
- הפשירו את גלולת התא שהושגה בשלב 1.3 בטמפרטורת החדר, ולאחר מכן החזירו אותה למאגר הליזיס (20 mM HEPES pH 7.5, 50 mM NaCl, 5 mM MgCl 2, 5 mM CaCl2) בתוספת 100 מיקרוגרם / מ"ל בנזמידין, 100 מיקרוגרם / מ"ל לאופטין, 100 מיקרוגרם / מ"ל אפרוטינין, 25 mU / mL אפירז, ו 10 μM אגוניסט. ערבבו את תרחיף התאים בטמפרטורת החדר למשך שעתיים כדי לגרום להיווצרות קומפלקס החלבון S1PRs-G.
2. מיקרוסקופיית אלקטרונים לפתרון מבנה S1PRs
- איסוף נתונים
- כדי להכין את רשתות הקריו-EM, החזק רשתות Au R1.2/1.3 של 300 רשתות למשך 10 שניות ופריקה זוהרת למשך 60 שניות ב-15 mA באמצעות מערכת ניקוי פריקה זוהרת.
- בצע ויטריפיקציה לדוגמה כפי שתואר קודם לכן33,34. במסוף הקפאת הצלילה, הגדר את הטמפרטורה ל- 4 °C ואת הלחות היחסית ל- 100% עבור סביבת העבודה התאית. השתמש בכוח כתם עבור 0, זמן המתנה של 0 שניות, זמן כתם של 2 או 3 שניות, וזמן ניקוז של 0 שניות. זה בדרך כלל דורש רק 3 μL של הדגימה בריכוזים של 5-10 מ"ג / מ"ל עבור vitrification יחיד.
- קליפ וטען רשתות להרכבת רשת אוטומטית, טען הרכבה אוטומטית של הרשת לתוך Nanocab, והעמיס Nanocab לתוך המיקרוסקופ על ידי autoloader כפי שתואר קודםלכן 35. איכות דגימת מסך עם תוכנת EPU234. בדרך כלל, הנתונים שנאספו באזורים של עובי קרח מתאים היו טובים יותר.
- אסוף נתוני cryo-EM כפי שתואר בפירוט קודםלכן 35. עבור קולטן S1P, הגדר את ההיסט defocus בין -1.0 μm ו- -1.8 μm עם מינון אלקטרונים חשיפה של 50-65 e-/Å2. עבור מתחמי S1PR1-Gi, אסוף ערימת סרטים באופן אוטומטי באמצעות תוכנת EPU2 במצב ספירה עם גלאי K2 בזמן חשיפה כולל של 2 שניות, קצב הקלטה של חמש מסגרות גולמיות לשנייה, ומינון כולל של 56 e-/Å2 כדי להפיק 35 פריימים לערימה.
הערה: בדרך כלל, יש צורך ביותר מ-5,000 סרטים כדי לבנות מחדש את מבנה הקולטן.
- עיבוד הנתונים באמצעות שילוב של RELION36 ו-cryoSPARC37 כדי לקבל מפת צפיפות קריו-EM אידיאלית. השתמש ב- RELION-3.1_gpu_ompi4 כדי לעבד את הנתונים בתחילה הכוללים פעולות דומות כפי שתואר קודם לכן34.
- במסוף מערכת Linux, הזן את ספריית האב של ספריית אחסון הנתונים.
- הזן את הפקודה relion במסוף כדי לפתוח את ממשק המשתמש הגרפי (GUI) של RELION.
הערה: אם זו הפעם הראשונה שממשק משתמש גרפי של RELION נפתח בספריה זו, יופיע חלון הנחיה; לחץ על כן. - לחץ על ייבוא בסרגל הפונקציות בצד ימין של ממשק המשתמש הגרפי של RELION כדי לייבא את הנתונים הגולמיים לתוך RELION.
- באפשרות סרטים/מיקרופונים, בחר כן לייבוא סרטים/מיקרוגרפים גולמיים?, הזן את נתיב הנתונים בשדה קובצי קלט Raw (מומלץ להשתמש בתווים כלליים) ובחר כן עבור האם אלה סרטים מרובי מסגרות?. הזן את גודל הפיקסלים של סרטים בשדה גודל פיקסל (Angstrom), את מתח ההפעלה של המיקרוסקופ (ב- kV) בשדה מתח (kV) ואת הסטייה הכדורית של המיקרוסקופ בשדה סטייה כדורית (מ"מ). אלה הם הפרמטרים שנרשמו בזמן איסוף הנתונים.
- באפשרות פועל , שנה את שם התור בהתאם לשרת שבו פועלת התוכנית (פונקציות אחרות צריכות גם לשנות פרמטר זה). השאר את הפרמטרים האחרים בערכי ברירת המחדל שנקבעו על-ידי RELION. לבסוף, כאשר כל הפרמטרים מזוהים כראוי, לחץ על RUN! כדי להפעיל את התוכנית.
- השתמש בפונקציית תיקון התנועה ליישור כל המסגרות38.
- באפשרות קלט / פלט , לחץ על עיון ובחר את הפלט של פונקציית ייבוא בשם movies.star כקלט של קלט סרטים STAR קובץ. הזן מנה למסגרת בשדה מינון למסגרת (e/A2) השווה למנה הכוללת חלקי מספר המסגרות. בחר לא עבור שמור סכום של ספקטרום הספק?.
- באפשרות Motion, הזן 250 עבור B-factor, 5.5 עבור מספר התיקונים X,Y ו- 2 עבור מסגרות קבוצה (ודא את המינון של קבוצה >3). אם הנתונים לא הופנו לרווחים במהלך תקופת האיסוף, יש צורך בתמונת הפניה לרווח שניתן להשיג על ידי רכישת אזור רשת ריק. בחר לא לשימוש ביישום של RELION עצמה? והזן את הספרייה המכילה את קובץ ההפעלה של MOTIONCOR2 39בשדה ההפעלה MOTIONCOR2.
- באפשרות פועל , בחר את מספר ה- MPI וההליך המשנה המתאימים בהתאם לעוצמת המחשוב של השרת; כאן, MPI = 8 וחוטים = 3 שימשו.
- השתמש בפונקציית הערכת CTF לוויסות תמונת cryo-EM של דגימה מזוגגת40. באפשרות קלט/פלט, לחץ על עיון ובחר את הפלט של תנועה cor בשם micrographs.star מתוקן כקלט של קלט סרטים STAR קובץ. באפשרות Gctf, בחר כן עבור UseGctf במקום זאת?.
- השתמש בפונקציית בחירת תת-ערכה כדי להסיר מיקרוגרפים עם ערך של rlnCtfMaxResolutin >4.
- באפשרות קלט/פלט , לחץ על עיון הממוקם בצד ימין של OR בחר מתוך micrographs.star ובחר את הפלט של CtfFind בשם micrographs_ctf.star כקלט. באפשרות ' ערכת משנה ', בחר כן עבור ' בחר' בהתבסס על ערכי מטא-נתונים? והזן 4 לערך המטא-נתונים המרבי להסרת נתונים פגומים.
- ליקוט ידני: בחר כמה תמונות באופן ידני עבור הבחירה והסיווג הראשוניים.
- באפשרות קלט/פלט , לחץ על עיון ובחר micrographs_selected.star מספריית הבחירה הקודמת (שלב 2.2.6) כקלט.
- לחץ על RUN! (חלון קופץ). לחץ על קובץ בפינה השמאלית העליונה של החלון החדש ולחץ על היפוך בחירה כדי לבטל את הבחירה של כל התמונות. סמן את תיבת הבחירה השמאלית ביותר בשורה המתאימה של כל ערך ולחץ על בחר כדי לבדוק את התמונות ובחר ~ 500 תמונות טובות. לחץ על קובץ > שמור בחירה כדי לשמור את התמונות שנבחרו ולסגור את החלון.
- קטיף אוטומטי: חבילות תוכנה אוטומטיות לאיסוף חלקיקים הן שימושיות וחזקות41.
- באפשרות קלט /פלט , לחץ על דפדף מימין למיקרוגרפים של קלט לאיסוף אוטומטי ובחר micrographs_selected.star מספריית ManualPick הקודמת (שלב 2.2.7) כקלט. האלגוריתם Laplacian-of-Gaussian משמש בהתחלה, לכן בחר כן עבור OR: השתמש ב- Laplacian-of-Gaussian.
- באפשרות Laplacian, הגדר את הקוטר המינימלי עבור מסנן LoG (A) ל- 80 ואת הקוטר המרבי עבור מסנן LoG (A) ל- 130. באפשרות בחירה אוטומטית, הגדר את המרחק המינימלי בין חלקיקים (A) ל- 65, ובחר כן לשימוש בהאצת GPU אם ניתן לגשת ל- GPU.
- חלצו חלקיקים לשלבים הבאים.
- באפשרות קלט/פלט , לחץ על דפדף מימין לקובץ STAR של מיקרוגרף ובחר את micrographs_selected.star משלב 2.2.6. לחץ על דפדף מימין לקואורדינטות קלט ובחר את cords_suffix_autopick.star משלב 2.2.8.
- באפשרות ' חילוץ ', בחרו 'כן ' באפשרות 'שינוי קנה מידה של חלקיקים ' והגדירו את הגודל (פיקסלים) שגודלו השתנה מחדש ל-128 כדי להאיץ שלבים מאוחרים יותר.
- סיווג דו-ממדי לסיווג ראשוני של חלקיקים
- באפשרות קלט /פלט , לחץ על דפדף מימין לקובץ קלט תמונות STAR ובחר את החלקיקים.star משלב 2.2.9. באפשרות 'מיטוב ', קבעו את מספר המחלקות על 100 ואת קוטר המסיכה (A) על 140.
- באפשרות 'מחשוב ', הגדר את מספר החלקיקים במאגר ל-10, הזן את הספריה שנמצאת בכונן מקומי מהיר (לדוגמה, כונן SSD) בשדה העתק חלקיק לספריית הגירוד , ובחר כן לשימוש בהאצת GPU? לקבלת מהירות עיבוד מהירה יותר.
- בחירת תת-ערכה לבחירת תוצאות דו-ממדיות טובות כתבניות לבחירת חלקיקים
- באפשרות קלט/פלט , לחץ על דפדף מימין למחלקות נבחרות מתוך model.star ובחר את הפלט של שלב 2.2.10 בשם run_it025_model.star כקלט. לחץ על RUN!. בחלון המוקפץ, סמן את מיין תמונות ב - ומיין הפוך? ולחץ על תצוגה!.
- בחר תוצאות דו-ממדיות מייצגות טובות כהפניה להפניה של פונקציית הבחירה האוטומטית .
הערה: התוצאות שנבחרו מאופסות באדום. תוצאות הסיווג הדו-ממדיות הטובות והרעות מוצגות מאוחר יותר. - לחץ באמצעות לחצן העכבר הימני ובחר שמור מחלקות נבחרות.
- השתמש בתבנית לסיבוב השני של בחירה אוטומטית. באפשרות קלט /פלט , לחץ על דפדף מימין למיקרוגרפים קלט לבחירה אוטומטית ובחר micrographs_selected.star משלב 2.2.6. לחץ על דפדף מימין להפניות דו-ממדיות, בחר class_averages.star משלב 2.2.11, ובחר לא עבור OR: השתמש ב- Laplacian-of-Gaussian.
- בצע חילוץ חלקיקים שוב באמצעות coord_suffix_autopick.star משלב 2.2.12 ו- micrographs_selected.star משלב 2.2.6.
- בצע שוב סיווג דו-ממדי באמצעות particles.star משלב 2.2.13.
- בצע שוב בחירת תת-ערכה באמצעות run_it025_optimiser.star משלב 2.2.14.
הערה: יש לבחור את כל התמונות הדו-ממדיות עם קווי מתאר ברורים וצורות נכונות. - בצע חילוץ חלקיקים באופן הבא. באפשרות קלט/פלט , לחץ על דפדף מימין לקובץ STAR של מיקרוגרף, בחר את micrographs_selected.star משלב 2.2.6, ובחר כן עבור או לחלץ מחדש חלקיקים מזוקקים?. לחץ על דפדף מימין לקובץ STAR של חלקיקים מזוקקים ובחר את החלקיקים.star משלב 2.2.15.
- מודל ראשוני תלת-ממדי ויצירת מפת ייחוס: באפשרות קלט /פלט , לחץ על דפדף מימין לקובץ STAR של תמונות קלט ובחר את החלקיקים.star משלב 2.2.16. קבעו את מספר המחלקות ל - 1, ואת קוטר המסיכה (A) ל- 140 באפשרות ' מיטוב' .
- סיווג תלת-ממדי ויצירת מפה תלת-ממדית ראשונית: באפשרות קלט /פלט , לחץ על דפדף מימין לקובץ STAR של תמונות קלט ובחר את החלקיקים.star משלב 2.2.16. לחץ על דפדף מימין למפת הפניה ובחר את initial_model.mrc משלב 2.2.17. קבעו את מספר המחלקות על 4-6, ואת קוטר המסיכה (A) על 140 באפשרות ' מיטוב' .
- יצירת מסיכות: בחר מפות תלת-ממד טובות משלב 2.2.17 כקלט באפשרות קלט/פלט. קבעו את סף הבינאריזציה הראשונית על 0.05 (התאימו בהתאם לפלט), הרחיבו את הסף הבינארי הזה של פיקסלים רבים ל- 3, והוסיפו קצוות רכים של פיקסלים רבים אלה ל- 8 באפשרות 'מסיכה'.
- השתמש ב- cryoSPARC לעיבוד הבא.
- צור סביבת עבודה חדשה ולחץ על בונה משרות עבור העבודה הראשונה.
- כדי לייבא את ערימת החלקיקים, הזן את נתיב החלקיקים (שלב 2.2.16) בשדה מטא-נתיב של חלקיקים ואת נתיב הסרט (שלב 2.2.16) בשדה נתיב נתוני חלקיקים .
הערה: הפרמטרים מתח האצה (kV), סטייה כדורית (מ"מ) וגודל פיקסל (אנגסטרום) זהים לקודמיהם. ערך ניגודיות משרעת (שבר) הוא 0.1. - יבא אמצעי אחסון תלת-ממדיים על-ידי הזנת הנתיב של אמצעי האחסון התלת-ממדי הטוב ביותר בשלב 2.2.18 בנתיב נתוני אמצעי האחסון ובחירה באפשרות מפה עבור סוג אמצעי האחסון המיובא.
- יבא מסיכה על-ידי הזנת נתיב המסיכה (שלב 2.2.19) בנתיב נתוני אמצעי האחסון ובחירה באפשרות מסיכה עבור סוג אמצעי האחסון המיובא.
- עידון לא אחיד: בצע את הפלטים של שלבים 2.2.22, 2.2.23 ו- 2.2.24 כקלט.
הערה: פונקציה זו שימושית מאוד עבור חלבוני ממברנה.- גרור את הפלט של שלב 2.2.22 (imported_particles) כקלט של חלקיקי עידון לא אחיד (חלקיק), את הפלט של שלב 2.2.23 (imported_volume_1) כקלט של נפח (נפח) של עידון לא אחיד, ואת הפלט של שלב 2.2.24 (imported_mask_1) כקלט של מסיכת עידון לא אחיד (מסיכה).
הערה: לפעמים ניתן להשיג תוצאות טובות יותר ללא מסכה. - לחץ על תור כדי להתחיל בעיבוד.
- גרור את הפלט של שלב 2.2.22 (imported_particles) כקלט של חלקיקי עידון לא אחיד (חלקיק), את הפלט של שלב 2.2.23 (imported_volume_1) כקלט של נפח (נפח) של עידון לא אחיד, ואת הפלט של שלב 2.2.24 (imported_mask_1) כקלט של מסיכת עידון לא אחיד (מסיכה).
- הפעל את שלבים 2.2.18-2.2.25 לקבלת תוצאות טובות יותר. באמצעות הסדרה לעיל של עיבוד, רזולוציה טובה S1PR-Gi 3D מפה ניתן להשיג.
3. בדיקת עיכוב cAMP בתיווך S1PRs-Gi
הערה: ניסוי עיכוב ה-cAMP בתיווך S1PRs-Gi חולק למספר חלקים, ולהלן הליכים ניסיוניים מפורטים. עקרון הניסוי והתהליך הניסויי הכללי מוצגים בצורה של תרשים זרימה באיור 1.
- בניית פלסמידים
- שיבוט משנה של ה-cDNAs של S1PR1, S1PR3 ו-S1PR5 הווקטוריים לתוך הווקטור pcDNA3.1+ עם רצף אותות HA ואחריו תג דגל ב-N-terminus (טבלת חומרים).
הערה: מוטציות עבור כל הקולטנים נוצרו באמצעות ערכת מוטגנזה (טבלת חומרים).
- שיבוט משנה של ה-cDNAs של S1PR1, S1PR3 ו-S1PR5 הווקטוריים לתוך הווקטור pcDNA3.1+ עם רצף אותות HA ואחריו תג דגל ב-N-terminus (טבלת חומרים).
- הכנת הפלסמיד
- הוסף את הווקטורים הרקומביננטיים pcDNA3.1+ או את פלסמיד החיישן (טבלת החומרים) ל-50 μL של Escherichia coli (E. coli) המוסמך DH5α המאוחסן בצינור של 1.5 מ"ל בטמפרטורה של -80°C בנפרד, ודגירה על קרח למשך 30 דקות. הלם חום התאים בטמפרטורה של 42 מעלות צלזיוס למשך 90 שניות, העבר אותם מיד לקרח וצנן אותם למשך 2 דקות.
הערה: פלסמיד החיישן המסופק על ידי ערכת הבדיקה של cAMP לעיכוב Gi (טבלת חומרים) מבטא גן לוציפראז שונה המתמזג עם תחום קשירת cAMP ומגביר את פעילות הזוהר כאשר cAMP קשור. - יש לנער את השפופרת בטמפרטורה של 37°C למשך שעה אחת לאחר נטילת תוסף עם 300 μL של ציר ליזוגני (LB) בינוני. צלחת 100 μL של תאים לצלחת אגר LB ודגירה ב 37 מעלות צלזיוס על ידי שמירה בחושך במשך 48 שעות. לחסן את המושבה הלבנה לתוך 5 מ"ל של מדיום LB המכיל 100 מיקרוגרם / מ"ל אמפיצילין, ותרבית ב 37 מעלות צלזיוס במשך 16 שעות.
- לבודד דנ"א באמצעות ערכת מיניפרפ פלסמיד (טבלת חומרים) בהתאם להוראות היצרן; הפלסמיד היה בריכוז של מעל 350 ng/μL עם הערך של A260/A280 בין 1.7 ל-1.9.
- הוסף את הווקטורים הרקומביננטיים pcDNA3.1+ או את פלסמיד החיישן (טבלת החומרים) ל-50 μL של Escherichia coli (E. coli) המוסמך DH5α המאוחסן בצינור של 1.5 מ"ל בטמפרטורה של -80°C בנפרד, ודגירה על קרח למשך 30 דקות. הלם חום התאים בטמפרטורה של 42 מעלות צלזיוס למשך 90 שניות, העבר אותם מיד לקרח וצנן אותם למשך 2 דקות.
- תרבית תאים
- צלחת את תאי CHO-K1 בצלחת פטרי 10 ס"מ, תרבית אותם באינקובטור 37 מעלות צלזיוס עם 5% CO2, וקצור אותם כאשר המונולייר הוא במפגש של 80%-90%.
- שאפו את מדיום הצמיחה של תאי CHO-K1, הוסיפו 4 מ"ל של 0.05% טריפסין-EDTA שחומם מראש בטמפרטורה של 37 מעלות צלזיוס על צלחת הפטרי בעדינות, ודגרו במשך 15 שניות. לאחר מכן, הוסף 4 מ"ל של מדיום צמיחה המורכב מ- F12 בינוני + 10% FBS.
- נתקו את התאים ממשטח צלחת הפטרי על ידי התנדנדות עדינה והקשה על דופן צלחת פטרי. למלא צינור חרוטי עם השעיה התא. מערבבים ומקפטים באיטיות כדי להסיר בעדינות את גושי התאים.
- תאים צנטריפוגה ב 250 x גרם במשך 5 דקות בטמפרטורת החדר, לשאוף supernatant, ו החייאה עם 3 מ"ל של PBS. חזור על שלב זה.
- קבע את מספר התא עם המציטומטר, ותאי צנטריפוגה ב 250 x גרם במשך 5 דקות בטמפרטורת החדר.
- שאפו למאגר PBS והחזירו תאי CHO-K1 עם 3 מ"ל של מדיום גדילה המורכב מ-F12 בינוני ו-10% FBS.
- הוסף 2 מ"ל של מדיום הצמיחה המורכב מ- F12 בינוני ו- 10% FBS לכל באר של צלחת שש הבארות, וזרע 150 μL של תרחיף תאים לתוך כל באר כדי לשמור על התאים בצפיפות של 1.5 x 105 תאים / מ"ל. דגירה של צלחת שש בארות באינקובטור תרבית רקמה 37 מעלות צלזיוס עם 5% CO2 במשך כ -24 שעות.
- טרנספקציה חולפת
- דילול 2 מיקרוגרם של DNA (שלב 3.2.3) לתוך 200 μL של מאגר מגיב transfection (טבלת חומרים). מערבבים על ידי מערבולת במשך 10 שניות ומסתובבים לזמן קצר לפני השימוש.
הערה: כאן, 2 מיקרוגרם של DNA מכיל 0.5 מיקרוגרם של וקטור קולטן (S1PR1, S1PR3 או S1PR5) ו 1.5 מיקרוגרם של פלסמיד חיישן. - יש להוסיף 4 μL של מגיב הטרנספקציה (טבלת חומרים), מערבולת במשך 10 שניות, ולהסתובב לזמן קצר לפני השימוש. דגירה במשך 15 דקות בטמפרטורת החדר.
- יש לטפטף באיטיות 200 μL של תערובת טרנספקציה לתוך כל באר (המכילה תאי CHO-K1) כדי להתפזר באופן שווה. יש לנער בעדינות את צלחת שש הבארות כדי להבטיח ערבוב יסודי.
- החלף את מדיום הטרנספקציה לאחר 4-6 שעות במדיום צמיחת התאים המורכב ממדיום F12 + 10% FBS, והחזר את צלחת שש הבארות לאינקובטור.
- קציר תאים 24-48 שעות לאחר ההעברה.
- לעכל את תאי CHO-K1 על הבאר עם 500 μL של 0.05% טריפסין-EDTA (מחומם מראש ב 37 מעלות צלזיוס) במשך 15 שניות ולהוסיף 1 מ"ל של מדיום צמיחה המורכב F12 בינוני + 10% FBS. נתקו את התאים מפני השטח של הבאר על ידי נדנדה והקשה עדינה על דופן הבאר.
- מעבירים את מתלה התא לצינור חרוטי, וצנטריפוגה בגודל 250 x גרם למשך 5 דקות בטמפרטורת החדר. שופכים את הסופר-נטנט וקוצרים את התאים הטרנספקטיביים.
הערה: לפני קביעת האות הפלואורסצנטי, בדוק את רמות הביטוי של פני השטח של קולטנים על ידי ELISA כפי שתואר קודםלכן 26.
- דילול 2 מיקרוגרם של DNA (שלב 3.2.3) לתוך 200 μL של מאגר מגיב transfection (טבלת חומרים). מערבבים על ידי מערבולת במשך 10 שניות ומסתובבים לזמן קצר לפני השימוש.
- שיווי משקל עם מלח D-לוציפרין-אשלגן (טבלת חומרים)
- השהה את התאים שנקטפו (24-48 שעות לאחר הטרנספקציה) עם 3 מ"ל של חיץ בדיקה באופן מיידי (כלומר, תמיסת המלח המאוזנת של האנק (HBSS) המכילה 10 mM HEPES pH 7.4), עם דילול v/v נוסף של מלח D-לוציפרין-אשלגן.
- הוסף 90 μL של תרחיף התא לכל באר של צלחת 96 באר באמצעות פיפטה רב ערוצית ולערבב בעדינות.
- לדגור במשך 40 דקות בטמפרטורת החדר.
- קביעת אות פלואורסצנטי
- הכן מראש 10 mM פתרונות מלאי של Siponimod מומס ב- DMSO ובצע דילול סדרתי באמצעות מאגר HBSS המכיל 25 μM פורסקולין לפני גירוי ליגנד.
הערה: למעט קבוצת הביקורת ללא ליגנד, לשאר יש טווח שיפוע ריכוז של 10-11-10-5 mol/L. - מריץ עם 10 μL (לכל באר) של תמיסה אגוניסטית בריכוזים שונים במשך 30 דקות.
- ספור את אותות הזוהר בקורא מיקרו-לוחות באמצעות פרמטרי התוכנה המשויכים (טבלת חומרים) באופן הבא. בחר Luminescence עבור שיטת זיהוי, נקודת קצה עבור סוג קריאה וסיב Luminescence עבור סוג אופטיקה. הגדר את רווח האופטיקה ל- 255.
הערה: כל מדידה חזרה על עצמה בלפחות שלושה ניסויים בלתי תלויים, כל אחד במשולש. - קבל את הערכים של האות הפלואורסצנטי, ייבא את הנתונים לתוכנית גיליון אלקטרוני ועבד את הנתונים באמצעות פונקציית תגובת המינון של רגרסיה לא ליניארית (התאמת עקומה).
- הכן מראש 10 mM פתרונות מלאי של Siponimod מומס ב- DMSO ובצע דילול סדרתי באמצעות מאגר HBSS המכיל 25 μM פורסקולין לפני גירוי ליגנד.
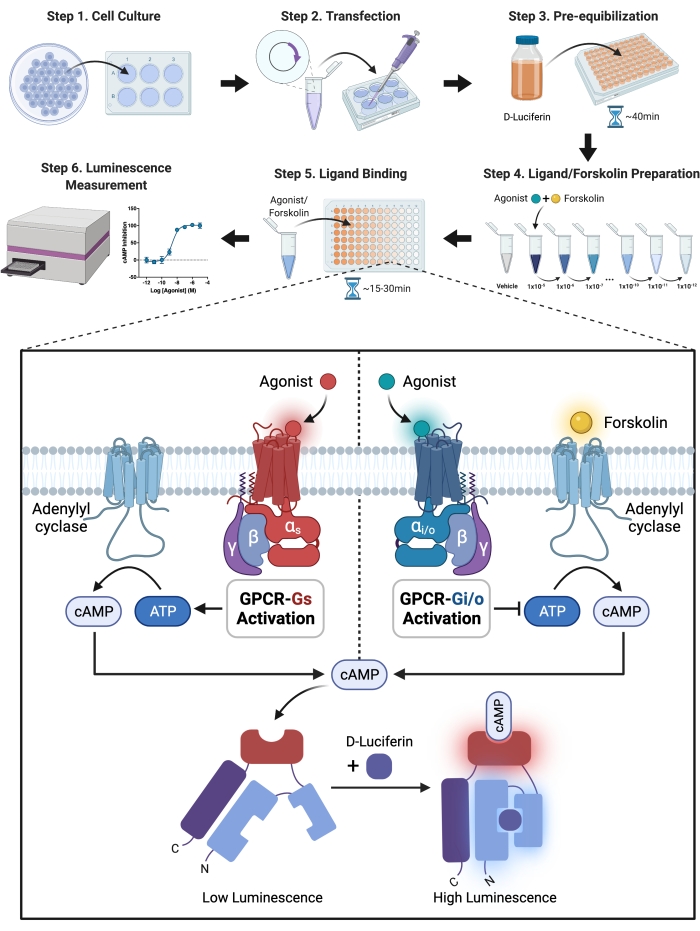
איור 1: המחשה סכמטית של הניסוי. מדריך מפורט מבחינת שלבים להגדרה ולביצוע ניסיוניים. בקצרה, הקולטן והלוציפראז המהונדס התבטאו באופן זמני בתאי CHO-K1 על ידי טרנספקציה של הקולטן ופלסמיד חיישן לתוך התאים עם מגיב טרנספקציה. התאים הושעו בתמיסת HBSS עם מלח D-לוציפרין-אשלגן, מצע לוציפראז, ונזרעו לצלחת של 96 בארות לאחר 24 שעות. כדי לאפשר חדירה לתוך התאים, D-לוציפרין חייב להיות שיווי משקל מראש עם התאים. האנזים החמצוני לוציפראז הופך לוציפרין לאוקסילוציפרין ופולט אור. לוציפראז שונה, לעומת זאת, מייצר אור באמצעות תגובה כימית רק כאשר הוא קשור ל- cAMP, ולעוצמת האור יש קשר חיובי לרמות cAMP בתאים. רמות ה- cAMP הוסדרו עם GPCR שהופעל על ידי אגוניסט. קולטנים מצומדים ל-Gi הפחיתו את רמות ה-cAMP, מה שהצריך הוספת פורסקולין כדי להפעיל את האדניל ציקלאז בניסוי cAMP של עיכוב Gi. אנא לחץ כאן כדי להציג גרסה גדולה יותר של נתון זה.
תוצאות
לפני הקפאת הדגימה של קומפלקס S1PRs-Gi, יש להפריד את הדגימה המטוהרת על ידי כרומטוגרפיה של אי הכללת גודל (SEC) ולנתח אותה באמצעות כרומטוגרפיית סינון ג'ל. איור 2 מציג את קומפלקס S1PR3-Gi כדוגמה. חלק השיא של קומפלקס החלבון ההומוגני GPCR-G היה ממוקם בדרך כלל ב~10.5 מ"ל של הכרומטוגרפיה של אי-הכלל?...
Discussion
פרוטוקול זה מתאר צינור ראשי לקביעת המבנים של S1PRs על ידי cryo-EM ומדידת עוצמת ההפעלה של S1PRs על ידי בדיקת עיכוב cAMP בתיווך Gi. כמה צעדים חיוניים להצלחת הניסוי.
כדי לטהר את קומפלקס S1PRs-Gi, יש לשים לב יותר לאיכות הנגיף ולבריאותם של תאי sf9 . הביטוי של הקולטן מופחת באופן דרמטי בתאי sf9
Disclosures
למחברים אין ניגודי עניינים.
Acknowledgements
הנתונים של קומפלקס S1PRs-Gi נקטפו במרכז Cryo-EM במערב סין באוניברסיטת סצ'ואן ובמרכז Cryo-EM באוניברסיטה הדרומית למדע וטכנולוגיה (SUSTech) ועובדו במרכז המחשוב בעל הביצועים הגבוהים Duyu באוניברסיטת סצ'ואן. עבודה זו נתמכה על ידי הקרן למדעי הטבע של סין (32100965 ללוס אנג'לס, 32100988 ל- W.Y., 31972916 ל- Z.S.) וקרן המחקר לפוסט-דוקטורט במשרה מלאה של אוניברסיטת סצ'ואן (2021SCU12003 עד L.C.)
Materials
| Name | Company | Catalog Number | Comments |
| 0.05% trypsin-EDTA | GIBCO | Cat# 25300054 | |
| 0.22 µM filter | Thermo Fisher Scientific | Cat# 42213-PS | |
| 100 kDa cut-off concentrator | Thermo Fisher Scientific | Cat# 88533 | |
| 6-well plate | Corning | Cat# 43016 | |
| 96-well plate | Corning | Cat# 3917 | |
| Aprotinin | Sigma-Aldrich | Cat# 9087-70-1 | |
| Apyrase | NEB | Cat# M0398S | |
| Baculovirus transfection reagent | Thermo Fisher Scientific | Cat# 10362100 | For the preparation of P0 baculovirus |
| Benzamidine | Sigma-Aldrich | Cat# B6506 | |
| CHO-K1 | ATCC | N/A | |
| CHS | Sigma-Aldrich | Cat# C6512 | |
| CryoSPARC | Punjani, A., et al.,2017 | https://cryosparc.com/ | |
| DH5α competent E.coli | Thermo Fisher Scientific | Cat# EC0112 | |
| D-Luciferin-Potassium Salt | Sigma- Aldrich | Cat# 50227 | |
| DMSO | Sigma- Aldrich | Cat# D2438 | |
| EDTA | Thermo Fisher Scientific | Cat# S311-500 | |
| ESF921 cell culture medium | Expression Systems | Cat# 96-001 | |
| Excel | microsoft | N/A | |
| F12 medium | Invitrogen | Cat# 11765 | |
| FBS | Cell Box | Cat# SAG-01U-02 | |
| Flag resin | Sigma- Aldrich | Cat# A4596 | |
| Forskolin | APExBIO | Cat# B1421 | |
| Gctf | Zhang, 2016 | https://www.mrc-lmb.cam.ac.uk/kzhang/Gctf/ | |
| GDN | Anatrace | Cat# GDN101 | |
| Gel filtration column | GE healthcare | Cat# 28990944 | |
| Gen5 3.11 | BIO-TEK | N/A | |
| Gentamicin | Solarbio | Cat# L1312 | |
| GloSensor cAMP assay kit | Promega | Cat# E1291 | Gi-inhibition cAMP assay kit |
| GloSensor plasmid | Promega | Cat# E2301 | Sensor plasmid |
| Grace’s medium | GIBCO | Cat# 11595030 | |
| GraphPad Prism 8 | Graphpad | N/A | |
| HBSS | Thermo Fisher Scientific | Cat# 88284 | |
| HEPES | Sigma- Aldrich | Cat# H4034 | |
| jetPRIME Reagent | Polyplus Transfection | Cat# 114-15 | transfection reagent |
| Janamycin | Solarbio | Cat# K1030 | |
| LB medium | Invitrogen | Cat# 12780052 | |
| Leupeptin | Sigma-Aldrich | Cat# L2884 | |
| LMNG | Anatrace | Cat# NG310 | |
| MotionCor2 | (Zheng et al., 2017) | https://emcore.ucsf.edu/ucsf-software | |
| NanoCab | Thermo Fisher Scientific | Cat# 1121822 | |
| PBS | Invitrogen | Cat# 14190-144 | |
| pcDNA3.1-HA-FLAG-S1PRs | GenScript | N/A | |
| pFastBac1-Gαi | GenScript | N/A | |
| pFastBac1-HA-FLAG-T4L-S1PRs-His10 | GenScript | N/A | |
| pFastBacdual-Gβ1γ2 | GenScript | N/A | |
| PureLink HiPure Plasmid Miniprep Kit | Invitrogen | Cat# K210003 | For the preparation of plasmids and P0 baculovirus |
| Q5 site-Directed Mutagenesis kit | NEB | Cat# E0554S | For the preparation of plasmids |
| Quantifoil | Quantifoil | Cat# 251448 | |
| RELION-3.1 | (Zivanov et al., 2018) | https://www2.mrc-lmb.cam.ac.uk/relion | |
| S1PRs cDNA | addgene | N/A | |
| scFv16 | Invitrogen | Cat# 703976 | |
| Sf9 | Expression Systems | N/A | |
| Siponimod | Selleck | Cat# S7179 | |
| sodium cholate | Sigma-Aldrich | Cat# C1254 | |
| Synergy H1 microplate reader | BIO-TEK | N/A | |
| Synthetic T4L DNA (sequence) | N/A | N/A | Aacatcttcgagatgctgcgcatcgacgaagg cctgcgtctcaagatttacaagaataccgaagg ttattacacgattggcatcggccacctcctgaca aagagcccatcactcaacgctgccaagtctga actggacaaagccattggtcgcaacaccaac ggtgtcattacaaaggacgaggcggagaaac tcttcaaccaagatgtagatgcggctgtccgtgg catcctgcgtaatgccaagttgaagcccgtgt atgactcccttgatgctgttcgccgtgcagcctt gatcaacatggttttccaaatgggtgagaccgg agtggctggttttacgaactccctgcgcatgctcc agcagaagcgctgggacgaggccgcagtga atttggctaaatctcgctggtacaatcagacacc taaccgtgccaagcgtgtcatcactaccttccg tactggaacttgggacgcttac |
| TCEP | Thermo Fisher Scientific | Cat# 77720 | |
| Tetracycline | Solarbio | Cat# T8180 | |
| Vitrobot Mark IV | Thermo Fisher Scientific | N/A |
References
- Verstockt, B., et al. Sphingosine 1-phosphate modulation and immune cell trafficking in inflammatory bowel disease. Nature Reviews: Gastroenterology & Hepatology. , 1-16 (2022).
- Rosen, H., Stevens, R. C., Hanson, M., Roberts, E., Oldstone, M. B. Sphingosine-1-phosphate and its receptors: structure, signaling, and influence. Annual Review of Biochemistry. 82, 637-662 (2013).
- Cartier, A., Hla, T. Sphingosine 1-phosphate: Lipid signaling in pathology and therapy. Science. 366 (6463), 5551 (2019).
- Jozefczuk, E., Guzik, T. J., Siedlinski, M. Significance of sphingosine-1-phosphate in cardiovascular physiology and pathology. Pharmacological Research. 156, 104793 (2020).
- Kihara, Y., Maceyka, M., Spiegel, S., Chun, J. Lysophospholipid receptor nomenclature review: IUPHAR Review 8. British Journal of Pharmacology. 171 (15), 3575-3594 (2014).
- Bryan, A. M., Del Poeta, M. Sphingosine-1-phosphate receptors and innate immunity. Cellular Microbiology. 20 (5), 12836 (2018).
- Pelletier, D., Hafler, D. A. Fingolimod for multiple sclerosis. New England Journal of Medicine. 366 (4), 339-347 (2012).
- Obinata, H., Hla, T. Sphingosine 1-phosphate and inflammation. International Immunology. 31 (9), 617-625 (2019).
- Pyne, N. J., Pyne, S. Sphingosine 1-phosphate and cancer. Nature Reviews: Cancer. 10 (7), 489-503 (2010).
- Abu-Farha, M., et al. The role of lipid metabolism in COVID-19 virus infection and as a drug target. International Journal of Molecular Sciences. 21 (10), 3544 (2020).
- Chun, J., Kihara, Y., Jonnalagadda, D., Blaho, V. A. Fingolimod: lessons learned and new opportunities for treating Multiple Sclerosis and other disorders. Annual Review of Pharmacology and Toxicology. 59, 149-170 (2019).
- Murakami, A., et al. Sphingosine 1-phosphate (S1P) regulates vascular contraction via S1P3 receptor: investigation based on a new S1P3 receptor antagonist. Molecular Pharmacology. 77 (4), 704-713 (2010).
- Cao, L., et al. Siponimod for multiple sclerosis. Cochrane Database of Systematic Reviews. 11, (2021).
- Scott, L. J. Siponimod: a review in secondary progressive Multiple Sclerosis. CNS Drugs. 34 (11), 1191-1200 (2020).
- Lamb, Y. N. Ozanimod: first approval. Drugs. 80 (8), 841-848 (2020).
- Scott, F. L., et al. Ozanimod (RPC1063) is a potent sphingosine-1-phosphate receptor-1 (S1P1) and receptor-5 (S1P5) agonist with autoimmune disease-modifying activity. British Journal of Pharmacology. 173 (11), 1778-1792 (2016).
- McGowan, E. M., Haddadi, N., Nassif, N. T., Lin, Y. Targeting the SphK-S1P-SIPR pathway as a potential therapeutic approach for COVID-19. International Journal of Molecular Sciences. 21 (19), 7189 (2020).
- O'Sullivan, C., Dev, K. K. The structure and function of the S1P1 receptor. Trends in Pharmacological Sciences. 34 (7), 401-412 (2013).
- Liao, M., Cao, E., Julius, D., Cheng, Y. Structure of the TRPV1 ion channel determined by electron cryo-microscopy. Nature. 504 (7478), 107-112 (2013).
- Bai, X. C., McMullan, G., Scheres, S. H. How cryo-EM is revolutionizing structural biology. Trends in Biochemical Sciences. 40 (1), 49-57 (2015).
- Murata, K., Wolf, M. Cryo-electron microscopy for structural analysis of dynamic biological macromolecules. Biochimica et Biophysica Acta General Subjects. 1862 (2), 324-334 (2018).
- Zhang, M., et al. Cryo-EM structure of an activated GPCR-G protein complex in lipid nanodiscs. Nature Structural & Molecular Biology. 28 (3), 258-267 (2021).
- Renaud, J. P., et al. Cryo-EM in drug discovery: achievements, limitations and prospects. Nature Reviews: Drug Discovery. 17 (7), 471-492 (2018).
- Ishchenko, A., Gati, C., Cherezov, V. Structural biology of G protein-coupled receptors: new opportunities from XFELs and cryoEM. Current Opinion in Structural Biology. 51, 44-52 (2018).
- Yang, D., et al. G protein-coupled receptors: structure- and function-based drug discovery. Signal Transduction and Targeted Therapy. 6 (1), 7 (2021).
- Yuan, Y., et al. Structures of signaling complexes of lipid receptors S1PR1 and S1PR5 reveal mechanisms of activation and drug recognition. Cell Research. 31 (12), 1263-1274 (2021).
- Zhao, C., et al. Structural insights into sphingosine-1-phosphate recognition and ligand selectivity of S1PR3-Gi signaling complexes. Cell Research. 32 (2), 218-221 (2022).
- Xu, Z., et al. Structural basis of sphingosine-1-phosphate receptor 1 activation and biased agonism. Nature Chemical Biology. 18, 281-288 (2022).
- Liu, Y. F., Ghahremani, M. H., Rasenick, M. M., Jakobs, K. H., Albert, P. R. Stimulation of cAMP synthesis by Gi-coupled receptors upon ablation of distinct Galphai protein expression. Gi subtype specificity of the 5-HT1A receptor. Journal of Biological Chemistry. 274 (23), 16444-16450 (1999).
- Buccioni, M., et al. Innovative functional cAMP assay for studying G protein-coupled receptors: application to the pharmacological characterization of GPR17. Purinergic Signalling. 7 (4), 463-468 (2011).
- Wang, F. I., Ding, G., Ng, G. S., Dixon, S. J., Chidiac, P. Luciferase-based GloSensor cAMP assay: Temperature optimization and application to cell-based kinetic studies. Methods. , (2021).
- Audet, M., et al. Small-scale approach for precrystallization screening in GPCR X-ray crystallography. Nature Protocols. 15 (1), 144-160 (2020).
- Sgro, G. G., Costa, T. R. D. Cryo-EM grid preparation of membrane protein samples for single particle analysis. Frontiers in Molecular Biosciences. 5, 74 (2018).
- White, J. B. R., et al. Single particle cryo-electron microscopy: from sample to structure. Journal of Visualized Experiments. (171), e62415 (2021).
- Thompson, R. F., Iadanza, M. G., Hesketh, E. L., Rawson, S., Ranson, N. A. Collection, pre-processing and on-the-fly analysis of data for high-resolution, single-particle cryo-electron microscopy. Nature Protocols. 14 (1), 100-118 (2019).
- Fernandez-Leiro, R., Scheres, S. H. W. A pipeline approach to single-particle processing in RELION. Acta Crystallographica Section D. 73 (6), 496-502 (2017).
- Punjani, A., Rubinstein, J. L., Fleet, D. J., Brubaker, M. A. cryoSPARC: algorithms for rapid unsupervised cryo-EM structure determination. Nature Methods. 14 (3), 290-296 (2017).
- Brilot, A. F., et al. Beam-induced motion of vitrified specimen on holey carbon film. Journal of Structural Biology. 177 (3), 630-637 (2012).
- Zheng, S. Q., et al. MotionCor2: anisotropic correction of beam-induced motion for improved cryo-electron microscopy. Nature Methods. 14 (4), 331-332 (2017).
- Zhang, K. Gctf: Real-time CTF determination and correction. Journal of Structural Biology. 193 (1), 1-12 (2016).
- Scheres, S. H. Semi-automated selection of cryo-EM particles in RELION-1.3. Journal of Structural Biology. 189 (2), 114-122 (2015).
- Liu, S., et al. Differential activation mechanisms of lipid GPCRs by lysophosphatidic acid and sphingosine 1-phosphate. Nature Communications. 13 (1), 731 (2022).
- Duan, J., et al. Cryo-EM structure of an activated VIP1 receptor-G protein complex revealed by a NanoBiT tethering strategy. Nature Communications. 11 (1), 4121 (2020).
- DiIorio, M. C., Kulczyk, A. W. A robust single-particle cryo-electron microscopy (cryo-EM) processing workflow with cryoSPARC, RELION, and Scipion. Journal of Visualized Experiments. (179), e63387 (2022).
- Pradelles, P., Grassi, J., Chabardes, D., Guiso, N. Enzyme immunoassays of adenosine cyclic 3',5'-monophosphate and guanosine cyclic 3',5'-monophosphate using acetylcholinesterase. Analytical Chemistry. 61 (5), 447-453 (1989).
- Jiang, L. I., et al. Use of a cAMP BRET sensor to characterize a novel regulation of cAMP by the sphingosine 1-phosphate/G13 pathway. Journal of Biological Chemistry. 282 (14), 10576-10584 (2007).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved