A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בידוד וקטורי ריפוי גנטי מהדור הבא באמצעות הנדסה, ברקוד וסינון של גרסאות קפסיד הקשורות לנגיף אדנו (AAV)
In This Article
Summary
יצירת ספריית תצוגה פפטידית AAV ותיקוף לאחר מכן באמצעות ברקוד של מועמדים עם תכונות חדשות ליצירת AAV מהדור הבא.
Abstract
וקטורי העברת גנים הנגזרים מווירוס הקשור לאדנו (AAV) הם אחד הכלים המבטיחים ביותר לטיפול במחלות גנטיות, עדות לכך היא עידוד נתונים קליניים ואישור של מספר טיפולים גנטיים AAV. שתי סיבות עיקריות להצלחה של וקטורי AAV הן: (i) בידוד מוקדם של סרוטיפים נגיפיים טבעיים שונים בעלי תכונות ייחודיות, ו-(ii) הקמתן של טכנולוגיות רבות עוצמה להנדסה המולקולרית שלהם וייעוד מחדש בתפוקה גבוהה. חיזוק נוסף לפוטנציאל של טכניקות אלה מיושמות לאחרונה אסטרטגיות לברקוד קפסידים AAV נבחרים ברמת ה- DNA וה- RNA, המאפשרים ריבוד מקיף ומקביל שלהם in vivo בכל האיברים העיקריים וסוגי התאים בחיה אחת. כאן, אנו מציגים צינור בסיסי המקיף קבוצה זו של דרכים משלימות, תוך שימוש בתצוגת פפטיד AAV כדי לייצג את הארסנל המגוון של טכנולוגיות הנדסת קפסיד זמינות. בהתאם לכך, אנו מתארים תחילה את השלבים המרכזיים ליצירת ספריית תצוגה פפטידית AAV לבחירת in vivo של מועמדים בעלי תכונות רצויות, ולאחר מכן הדגמה כיצד לברקוד את גרסאות הקפסיד המעניינות ביותר לסינון משני in vivo. לאחר מכן, אנו מדגימים את המתודולוגיה ליצירת ספריות לריצוף הדור הבא (NGS), כולל הגברת ברקוד וקשירת מתאמים, לפני שנסיים בסקירה כללית של השלבים הקריטיים ביותר במהלך ניתוח נתוני NGS. מכיוון שהפרוטוקולים המדווחים כאן הם רב-תכליתיים וניתנים להתאמה, חוקרים יכולים לרתום אותם בקלות להעשרת גרסאות הקפסיד AAV האופטימליות במודל המחלה המועדף עליהם וליישומי ריפוי גנטי.
Introduction
טיפול בהעברת גנים הוא החדרת חומר גנטי לתאים כדי לתקן, להחליף או לשנות את החומר הגנטי התאי כדי למנוע, לטפל, לרפא או לשפר מחלות. העברת גנים, הן in vivo והן ex vivo, מסתמכת על מערכות העברה שונות, לא ויראליות וויראליות. וירוסים התפתחו באופן טבעי כדי להתמיר ביעילות את תאי המטרה שלהם ויכולים לשמש כווקטורי מסירה. בין הסוגים השונים של וקטורים נגיפיים המשמשים בריפוי גנטי, וירוסים הקשורים באדנו נמצאים בשימוש הולך וגובר, בשל חוסר הפתוגניות שלהם, בטיחותם, אימונוגניות נמוכה, והכי חשוב יכולתם לקיים ביטוי ארוך טווח, לא משלב 1,2,3. הטיפול הגנטי AAV הניב הישגים ניכרים בעשור האחרון; שלושה טיפולים אושרו על ידי סוכנות התרופות האירופית ומנהל המזון והתרופות האמריקאי לשימוש בבני אדם 3,4. כמו כן, נערכים מספר ניסויים קליניים לטיפול במגוון מחלות, כגון המופיליה, מחלות שרירים, לב ונוירולוגיה, כפי שנסקר במקום אחר3. למרות עשרות שנים של התקדמות, תחום הריפוי הגנטי חווה בשנים האחרונות שורה של כישלונות4, ובראשם מקרי מוות בניסויים קליניים5 שהוקפאו עקב רעילות המגבילה מינון, במיוחד עבור רקמות מסיביות, כגון שריר, או קשות להשגה, כגון מוח6.
וקטורי AAV הנמצאים כיום בשימוש בניסויים קליניים שייכים לסרוטיפים הטבעיים עם כמה יוצאים מן הכלל1. הנדסת AAV מציעה את ההזדמנות לפתח וקטורים עם ספציפיות ויעילות מעולה של איברים או תאים. בשני העשורים האחרונים, מספר גישות יושמו בהצלחה, כגון תצוגת פפטידים, החלפת לולאה, ערבוב DNA קפסיד, PCR נוטה לשגיאות ועיצוב ממוקד, כדי ליצור גרסאות AAV בודדות או ספריות שלהם עם תכונות מגוונות7. לאחר מכן הם נתונים לסבבים מרובים של אבולוציה מכוונת כדי לבחור את הווריאנטים בתוכם עם התכונות הרצויות, כפי שנסקר במקומות אחרים 1,3. מכל אסטרטגיות האבולוציה של הקפסיד, ספריות AAV לתצוגת פפטידים, היו בשימוש הנרחב ביותר, בשל כמה תכונות ייחודיות: קל יחסית ליצור אותן, והן יכולות להשיג מגוון גבוה ורצף בתפוקה גבוהה, המאפשר לעקוב אחר האבולוציה שלהן.
ספריות ה-AAV הראשונות להחדרת פפטיד בהצלחה תוארו לפני כמעט 20 שנה. באחד הראשונים, Perabo et al.8 בנו ספרייה של קפסידים AAV2 מותאמים, שבה מאגר של אוליגונוקלאוטידים שנוצרו באופן אקראי הוכנס לפלסמיד במיקום המתאים לחומצת אמינו 587 של חלבון הקפסיד VP1, בציר המשולש הבולט מהקפסיד. באמצעות זיהום משותף של אדנו-וירוס, ספריית AAV התפתחה באמצעות סבבי בחירה מרובים, והווריאנטים הסופיים הממוקדים מחדש הוכחו כמסוגלים להמיר קווי תאים עמידים ל-AAV28 ההורי. זמן קצר לאחר מכן, Müller et al.9 הציגו את המערכת הדו-שלבית לייצור ספריות, שיפור משמעותי לפרוטוקול. בתחילה, ספריית הפלסמיד, יחד עם פלסמיד עוזר אדנו-ויראלי, משמשים לייצור ספריית AAV המכילה קפסידים כימריים. ספריית מעבורת AAV זו משמשת להדבקת תאים בריבוי נמוך של זיהום (MOI), במטרה להציג גנום נגיפי אחד לכל תא. זיהום משותף עם אדנווירוס מבטיח את הייצור של AAVs עם גנום תואם קפסיד9. כעשור לאחר מכן, דלקארה10 השתמש באבולוציה מכוונת in vivo כדי ליצור את גרסת 7m8. גרסה זו כוללת החדרת 10 חומצות אמינו (LALGETTRPA), שלוש מהן פועלות כמקשרות, ומכוונות ביעילות לרשתית החיצונית לאחר הזרקה תוך ויטריאלית10. קפסיד מהונדס זה הוא סיפור הצלחה יוצא דופן, שכן הוא אחד הקפסידים המהונדסים הבודדים שהגיעו למרפאה עד כה11.
התחום חווה דחיפה שנייה עם כניסתן של טכניקות ריצוף מהדור הבא (NGS). שני פרסומים של Adachi et al.12 בשנת 2014 ומ- Marsic et al.13 בשנת 2015, הראו את כוחה של NGS לעקוב אחר התפלגות ספריות קפסיד AAV ברקודיות בדיוק גבוה. כמה שנים לאחר מכן, ה-NGS של אזורים מקודדים הותאם לאזור החדרת הפפטיד כדי לעקוב אחר התפתחות הקפסיד. Körbelin et al.14 ביצעו סריקה מונחית NGS כדי לזהות קפסיד מבוסס AAV2 ממוקד ריאות. ניתוח NGS סייע לחשב שלושה ציוני דירוג: ציון ההעשרה בין סבבי הבחירה, ציון הספציפיות הכללי לקביעת ספציפיות הרקמות, ולבסוף הציון המשולב14. מעבדת Gradinaru15 פרסמה באותה שנה את מערכת האבולוציה הממוקדת AAV (CREATE) מבוססת Cre-recombination, המאפשרת בחירה ספציפית לסוג התא. במערכת זו, ספריית הקפסיד נושאת מתג Cre-inversible, מכיוון שהאות polyA מוקף בשני אתרי loxP. ספריית AAV מוזרקת לאחר מכן בעכברי Cre, שם אות ה- polyA הפוך רק בתאי Cre+, ומספק את התבנית לקשירה של פריימר PCR הפוך עם הפריימר הקדמי בתוך גן הקפסיד. הצלת PCR ספציפית מאוד זו אפשרה את זיהוי ה- AAV-PHP. גרסת B שיכולה לחצות את מחסום הדם-מוח15. מערכת זו התפתחה בהמשך ל-M-CREATE (Multiplexed-CREATE), שבה שולבו NGS וייצור ספריות סינתטיות בצנרת16.
גרסה משופרת מבוססת RNA של מערכת זו ממעבדת מגווייר17, iTransduce, מאפשרת סלקציה ברמת הדנ"א של קפסידים המתמרים תאים באופן פונקציונלי ומבטאים את הגנום שלהם. הגנום הנגיפי של ספריית התצוגה הפפטידית מורכב מגן Cre תחת שליטתו של מקדם הנמצא בכל מקום וגן קפסיד תחת שליטתו של מקדם p41. הספרייה מוזרקת לעכברים שיש להם קלטת loxP-STOP-loxP במעלה הזרם של tdTomato. תאים שהותמרו עם גרסאות AAV המבטאות את הגנום הנגיפי ולכן Cre מבטאים tdTomato, ובשילוב עם סמני תאים, ניתן למיין ולבחור17. באופן דומה, Nonnenmacher et al.18 ו- Tabebordbar et al.19 הציבו את ספריית הגנים של הקפסיד תחת שליטתם של מקדמים ספציפיים לרקמות. לאחר הזרקה במודלים שונים של בעלי חיים, נעשה שימוש ב-RNA נגיפי כדי לבודד את גרסאות הקפסיד.
גישה חלופית היא להשתמש בברקוד כדי לתייג ספריות capsid. מעבדת Björklund20 השתמשה בגישה זו לספריות קפסיד החדרת פפטידי ברקוד ופיתחה את האבולוציה הווקטורית AAV רציונלית ברקודית (BRAVE). בפלסמיד אחד, קלטת Rep2Cap משובטת לצד טרנסגן מבטא טרמינלי הפוך (ITR), חלבון פלואורסצנטי צהוב (YFP) המבטא ברקוד. באמצעות שימוש באתרי loxP בין סוף המכסה לתחילת הברקוד, רקומבינציה של In vitro Cre יוצרת מקטע קטן מספיק עבור NGS, ובכך מאפשרת שיוך של החדרת פפטיד עם הברקוד הייחודי (טבלת חיפוש, LUT). ייצור AAV מתבצע באמצעות ספריית פלסמיד והברקודים המבוטאים ב-mRNA מוקרנים לאחר יישום in vivo , שוב עם NGS20. כאשר ספריות הקפסיד מורכבות מווריאנטים של גן הקפסיד כולו (כלומר, ספריות ערבוב), יש להשתמש בריצוף קריאה ארוכה. מספר קבוצות השתמשו בברקודים כדי לתייג ספריות מגוונות אלה, מה שמאפשר NGS עם עומק קריאה גבוה יותר. מעבדת קיי21 תייגה ספריות ערבוב קפסיד מגוונות ביותר עם ברקודים במורד הזרם של אות cap polyA. בשלב ראשון נוצרה ספריית פלסמיד מקודדת, וספריית הגנים של הקפסיד המעורבב שוכפלה לתוכה. לאחר מכן נעשה שימוש בשילוב של MiSeq (קריאה קצרה, עומק קריאה גבוה יותר) ו- PacBio (קריאה ארוכה, עומק קריאה נמוך יותר) NGS כמו גם ריצוף Sanger כדי ליצור את LUT21 שלהם. ב-2019, אוגדן ועמיתיו ממעבדת צ'רץ'22 תיארו את כשירות הקפסיד AAV2 עבור פונקציות מרובות באמצעות ספריות שהיו להן מוטציות, הוספות ומחיקות נקודתיות בודדות בכל מיקום, מה שאיפשר בסופו של דבר תכנון מונחה מכונה. עבור יצירת הספרייה, מקטעים קטנים יותר של גן הקפסיד סונתזו, תויגו בברקוד, רוצפו מהדור הבא, ולאחר מכן שובטו לגן הקפסיד המלא. נתוני NGS שימשו ליצירת LUT. לאחר מכן הוקרנה הספרייה באמצעות ברקודים בלבד ורצף קריאה קצר, אשר בתורו מאפשר עומק קריאה גבוה יותר22.
ספריות ברקודיות שימשו בעיקר לסינון מאגר של וריאנטים ידועים, טבעיים ומהונדסים לאחר מספר סבבי בחירה של ספריות קפסיד או ללא תלות במחקר אבולוציה של קפסיד. היתרון של ספריות כאלה הוא ההזדמנות לסנן קפסידים מרובים, תוך צמצום מספר בעלי החיים ומזעור השונות בין בעלי חיים. המחקרים הראשונים שהכניסו טכנולוגיה זו לתחום ה-AAV פורסמו לפני כמעט עשור. מעבדת נקאי 12 תייגה 191 מוטנטים כפולים באלנין המכסים חומצות אמינו 356 עד 736 על VP1 מ-AAV9 עם זוג ברקודים של12 נוקלאוטידים. באמצעות NGS, הספרייה הוקרנה in vivo עבור כריכת גלקטוז ותכונות אחרות12. מרסיק ועמיתיו תיארו את ההתפלגות הביולוגית של גרסאות AAV באמצעות ניתוח דו-קני שנה לאחר מכן13. מחקר עדכני יותר שנערך בקרב פרימטים לא אנושיים השווה את ההתפלגות הביולוגית במערכת העצבים המרכזית של 29 קפסידים באמצעות נתיבי מסירה שונים23. המעבדה שלנו פרסמה לאחרונה מסכי ספריית AAV מקודדים של 183 גרסאות שכללו AAV טבעיים ומהונדסים . מסכים אלה ברמת הדנ"א והרנ"א הובילו לזיהוי של וריאנט AAV מיוטרופימאוד 24 בעכברים, כמו גם אחרים המציגים ספציפיות גבוהה מסוג תא במוח העכבר25.
כאן, אנו מתארים את המתודולוגיה המשמשת בעבודה זו ומרחיבים אותה כדי לכלול סינון של ספריות תצוגה פפטידים AAV. זה כולל את הדור של ספריות תצוגה פפטידי AAV2, שיטת PCR טיפתית דיגיטלית (dd-PCR) לכימות ולבסוף צינור NGS לניתוח גרסאות AAV, המבוסס בחלקו על עבודתם של Weinmann ועמיתיו24. לבסוף, תיאור של הדור של ספריות AAV ברקוד ואת צינור NGS המשמש באותו פרסום מסופק.
Protocol
1. הכנת ספריית תצוגה פפטידית אקראית AAV2 7-mer
הערה: להכנת ספריית תצוגה של פפטידים אקראיים AAV2, סנתז את האוליגונוקלאוטידים המנוונים כדנ"א חד-גדילי, המר אותו לדנ"א דו-גדילי, עכל, קשר לפלסמיד המקבל ואלקטרופורט.
- תכנון אוליגונוקלאוטידים מנוונים
- סדרו את האוליגונוקלאוטידים המנוונים והימנעו מהטיה קודונית. באוליגונוקלאוטיד 5' CAGTCGGCCAG AG W GGC (X01)7 GCCCAGGCGGCTGACGAG 3', X01 מתאים ל-20 קודונים, שכל אחד מהם מקודד לאחת מ-20 חומצות האמינו. ה-W יכול להיות A או T, ולייצר את הקודונים AGA או AGT, המקודדים את חומצות האמינו ארגינין (R) או סרין (S).
- הזמינו את פריימר ההגברה: 5' CTCGTCAGCCGCCTGG 3' (ראו איור 1 לפרטים). זה מייצר את תוספת החלבון הבאה: R/S G X7. המגוון התיאורטי מחושב כדלקמן: 1 x 2 x 207 = 2.56 x 109 גרסאות ייחודיות.
הערה: יש לציין כי מגוון זה עשוי להיות מוגבל על ידי יעילות הטרנספורמציה.
- סינתזת גדיל שני
- השהה מחדש את שני האוליגונוקלאוטידים (אוליגונוקלאוטידים מנוונים ופריימר הגברה) לריכוז סופי של 100 מיקרומטר עם חיץ TE.
- עבור תגובת PCR, הגדר תגובה של 50 μL עם 1 μL של כל פריימר, 10 μL של החיץ, 1.5 μL של DMSO, 0.5 μL של dNTPs (10 mM), 0.5 μL של Hi-fidelity Hot Start Polymerase II, ו- 35.5 μL של מים ללא נוקלאז.
- העבירו את התגובה לתרמוסייקלר והפעילו שלב טרום דגירה במשך 10 שניות ב-98°C, ואחריו שלושה מחזורים של 10 שניות ב-98°C, 30 שניות ב-59°C ו-10 שניות ב-72°C, ואז 5 דקות ב-72°C ושלב קירור סופי.
- לטהר את התגובה באמצעות ערכה להסרת נוקלאוטידים ולטהר פנימה 100 μL של מים נטולי נוקלאז.
- אשרו את היעילות של סינתזת הגדיל השני על-ידי ניתוח על ביואנלייזר (ראו איור 2). נתח את הגודל והטוהר של העלון הדו-גדילי על ידי טעינת 1 μL של התגובה לשבב מיקרופלואידי מערכת ריאגנטים DNA 1000 בהתאם להוראות היצרן. ערכה זו מותאמת למדידת הגודל והריכוז של מקטעי DNA דו-גדיליים בין 25-1,000 bps.
- השהה מחדש את שני האוליגונוקלאוטידים (אוליגונוקלאוטידים מנוונים ופריימר הגברה) לריכוז סופי של 100 מיקרומטר עם חיץ TE.
- עיכול של הכנס וקטור פלסמיד
- עיכול 85 μL של העלון המטוהר עם 10 μL של חיץ 10x ו-5 μL של אנזים BglI בנפח תגובה סופי של 100 μL (ראה איור 1 לפרטים). לדגור ב 37 °C (77 °F) במשך הלילה. לטהר באמצעות ערכה להסרת נוקלאוטידים, לטהר 50 μL של מים נטולי נוקלאז, ולכמת באמצעות סוג "אוליגו DNA" בספקטרופוטומטר.
- עיכול 10 מיקרוגרם של פלסמיד AAV (pRep2Cap2_PIS)26 (גנום נגיפי מאגף ITR) עם 20 μL של חיץ 10x ו-10 μL של אנזים SfiI בנפח תגובה סופי של 200 μL (ראו איור 1 לפרטים). יש לדגור ב-50 מעלות צלזיוס למשך הלילה. לטהר את הווקטור על ג'ל אגרוז 1% באמצעות ערכת מיצוי ג'ל ואחריו שלב טיהור נוסף באמצעות ערכת טיהור DNA. לכמת את הריכוז בספקטרופוטומטר.
- קשירת אינסרט לווקטור
- Ligate 955 ng של וקטור פלסמיד עם 45 ng של תוספת עם 2 μL של חיץ ו 2 μL של ליגאז בתגובת קשירה 20 μL. יש לדגור ב-16°C למשך הלילה, ולאחר מכן 10 דקות ב-70°C כדי להשבית את הליגאז בחום.
- טרנספורמציה, חישוב סיבוכיות והכנת ספריית פלסמיד
- לטהר את התגובה עם ערכת טיהור DNA בהתאם להוראות היצרן. יש להצניע את התגובה בכ-80% מהנפח ההתחלתי של מים נטולי נוקלאז ולאחסן על קרח לצורך טרנספורמציה לאחר מכן.
- התמירו תאים בעלי יכולת חשמלית: הפשירו בקבוקון אחד של תאים אלקטרו-מוכשרים על קרח למשך 10 דקות. לאחר מכן יש להוסיף 1-2 μL של תגובת הקשירה המטוהרת ל-30 μL (בקבוקון אחד) של תאים אלקטרו-כשירים ולערבב על ידי הקשה עדינה. לאחר מכן, העבירו בזהירות את תערובת התא/דנ"א לקובט אלקטרופורציה מקורר מראש ברווח של 1 מ"מ מבלי להכניס בועות אוויר.
- אלקטרופורט באמצעות ההגדרות הבאות: 1800 וולט, 600 Ω ו- 10 מיקרופרנהייט. בתוך 10 שניות של פולס אלקטרופורציה, להוסיף 970 μL של מדיה התאוששות שחוממה מראש (מסופק עם תאים electrocompetent) לקובט ולערבב על ידי pipetting. לבסוף, להעביר את התאים לצינור מיקרו צנטריפוגה לדגור במשך 1 שעה ב 37 ° C ב 250 סל"ד. כדי להשיג את הגיוון הרצוי, בצעו 10-100 תגובות, ולאחר הדגירה, אגרו את כל התגובות בצינור אחד.
- חשב את המגוון על ידי דילול 10 μL של הטרנספורמציות המאוחדות פי 10, 100 או 1,000 ב- PBS ופיזור 100 μL על צלחות אגר מזין המכילות את האנטיביוטיקה המתאימה (75 מ"ג / מ"ל של אמפיצילין). דוגרים על צלחות האגר למשך הלילה בטמפרטורה של 37 מעלות צלזיוס ולאחר מכן סופרים את המושבות על צלחות האגר.
- חשב את המגוון התיאורטי כדלקמן:
מגוון מקסימלי תיאורטי = 10 x גורם דילול x מספר מושבות x מספר תגובות אלקטרופורציה.
הערה: כדי לאשר את איכות הספרייה, רצף לפחות 20 מושבות על ידי ריצוף Sanger. רוב השיבוטים צריכים להכיל תוספת, וכולם צריכים להיות ייחודיים. - יש לחסן 400-1,000 מ"ל של LB בינוני המכיל את האנטיביוטיקה המתאימה עם שאר הטרנספורמציות המצטברות ולדגור למשך הלילה ב-37°C, 180 סל"ד.
- הכנת ספריית פלסמיד
- מתרבית הלילה, הכינו ציר גליצרול (ערבבו כמויות שוות של תרבית חיידקים ותמיסת גליצרול 50% במים נטולי נוקלאז והקפיאו בטמפרטורה של -80°C) וטיהרו את ספריית הפלסמיד באמצעות ערכת מקסי פלסמיד.
- הפקת ספרייה ויראלית AAV
- הכינו את הספרייה הוויראלית כמתואר לעיל27. העבר את ספריית הפלסמיד (pRep2Cap2_PI, תוספת פפטידים) יחד עם פלסמיד אדנו-עוזר לתאי HEK293T באמצעות מגיב טרנספקציה כגון פוליאתילנימין (PEI).
- לאסוף את התאים לאחר 3 ימים ולהכפיף אותם לשלושה מחזורים של הקפאה-הפשרה. לטהר את הליזט הנגיפי באמצעות אולטרה-צנטריפוגה הדרגתית של צזיום כלוריד, ולאחר מכן חילופי חיץ ל- PBS, ולבסוף לרכז את החלקיקים הנגיפיים.
- טיטרציה וקטורית AAV באמצעות dd-PCR
- דילול סדרתי של 2 μL של מלאי וקטור AAV ב-198 μL של מים נטולי נוקלאז כדי להניב דילול סופי של 1:106 . מערבבים היטב בכל פעם באמצעות פיפטה של 200 μL. הוסף פקד אחד ללא תבנית (NTC) כפקד שלילי.
הערה: ניתן לבדוק דילולים נוספים נמוכים או גבוהים יותר (1:105-1:107). - הכינו תערובת פריימר-בדיקה 20x. הוסף 3.6 μL של כל אחד מהפריימרים של 100 μM (קדימה ואחורה, Rep2 ו- ITR), 1 μL כל אחד מבדיקות 100 μM dd-PCR (Rep2 ו- ITR), ו- 3.6 μL של מים נטולי נוקלאז לצינור צנטריפוגה של 1.5 מ"ל.
הערה: ספריית AAV נמדדת באמצעות ערכת פריימר-בדיקה ממוקדת טרנסגן (Rep2) שזוהתה באמצעות בדיקה המסומנת בתווית FAM, וערכת בדיקת פריימר ממוקדת ITR שזוהתה באמצעות בדיקה המסומנת בתווית HEX. - הכינו תגובת PCR של 22 μL על ידי הוספת 5.5 μL של דגימה, 1.1 μL של תערובת פריימר-בדיקה 20x, 11 μL של תערובת על dd-PCR עבור בדיקות (ללא dUTP), ו-4.4 μL של מים נטולי נוקלאז. זה מניב ריכוזים של 900 ננומטר ו 250 ננומטר עבור פריימרים ובדיקה, בהתאמה.
- צור את הטיפות באמצעות מחולל טיפות, העבר את התגובה לצלחת 96 בארות, מקם את הצלחת לתוך thermocycler, והפעל שלב דנטורציה במשך 10 דקות ב 94 ° C, ואחריו 40 מחזורים של 30 s ב 94 ° C ו 1 דקה ב 58 ° C. לאחר מכן, השבת בחום את הפולימראז למשך 10 דקות ב-98°C והוסף שלב קירור אחרון. קרא את התגובות בקורא טיפות והמשך לניתוח28.
- פתח את קובץ צלחת dd-PCR שנשמר באמצעות תוכנת הניתוח. השתמש בכלי סף בכרטיסייה 1D Amplitude (משרעת פלואורסצנטית לעומת מספר אירוע) כדי להפריד את הטיפות השליליות והחיוביות עבור כל ערוץ, באמצעות NTC כקו מנחה, ולייצא את הנתונים לקובץ csv.
- כדי לחשב את הריכוז הווקטורי, חשב תחילה את גורם התיקון CF באמצעות הנוסחה:
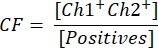
CF קובע את שיעור הטיפות החיוביות עבור הטרנסגן [חיובי] שהן חיוביות עבור שניהם, טרנסגן ו- ITR [Ch1+ Ch2+], כדי להבטיח זיהוי של חלקיקים וקטוריים פונקציונליים. כעת ניתן לחשב את הריכוז הווקטורי הסופי c באמצעות המשוואה הבאה: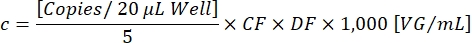
DF הוא גורם הדילול (1:10,5-1:10,7 , כפי שנקבע קודם לכן). העותקים לכל תגובה של 20 μL/well מתאימים ל-5 μL של הדגימה המדוללת. פקטור 1,000 מתקן את הסולם ל-VG/mL (גנום נגיפי/מ"ל). תוצאת טיטרציה לדוגמה מודגמת בטבלה 1 ובאיור 3.
- דילול סדרתי של 2 μL של מלאי וקטור AAV ב-198 μL של מים נטולי נוקלאז כדי להניב דילול סופי של 1:106 . מערבבים היטב בכל פעם באמצעות פיפטה של 200 μL. הוסף פקד אחד ללא תבנית (NTC) כפקד שלילי.
- ניתוח הספרייה הוויראלית AAV על ידי NGS
- הגבר את מקטע החדרת הפפטיד 96-נוקלאוטיד על-ידי הגדרת תגובת PCR של 20 μL באמצעות ערכת הגהה של פולימראז (2x; ראה איור 4). הוסף 1 μL של מלאי AAV המכיל 1 x 108 vg, 0.5 μL של כל אחד 100 μM פריימר (NGS_forward ו NGS_reverse), ו 10 μL של תערובת האנזימים לתגובה. כוונן את הנפח הסופי ל-20 מיקרוליטר עם מים נטולי נוקלאז.
- העבירו את התגובה לתרמוסייקלר והפעילו שלב דנטורציה במשך 3 דקות ב-98°C, ואחריו 30-35 מחזורים של 10 שניות ב-98°C, 10 שניות ב-59°C ו-20 שניות ב-72°C, ולאחר מכן 5 דקות ב-72°C ושלב קירור סופי.
- לטהר את הדגימות באמצעות ערכת טיהור PCR. כמת את הריכוז בספקטרופוטומטר והפעל ג'ל אגרוז 3% כדי לוודא טוהר וגודל מקטע.
- עיבוד קטעי PCR באמצעות מערכת הספרייה לערכת דגימות במורכבות נמוכה בהתאם להוראות היצרן להכנת ספריית NGS. בצע את תגובת התיקון הסופית עם מקטע PCR של 30 ננוגרם, ולאחר מכן קשירת מתאם והגברת PCR למשך 10 מחזורים. השתמש בערכת טיהור PCR לטיהור התגובות.
- עיבוד המוצרים הסופיים על גבי ביואנלייזר לאימות הגודל והטוהר, באמצעות ערכת ריאגנטים DNA בהתאם להוראות היצרן.
- לכמת את האמפליקונים באמצעות פלואורומטר ולאגד אותם. כמת שוב את ספריית ה- NGS הסופית המאוגמת על פלואורומטר (בהתאם להוראות היצרן) ואמת את האיכות בביואנלייזר.
- רצף את ספריות ה- NGS במצב קצה יחיד (SE), באמצעות ערכת פלט גבוהה של 75 מחזורים, עם אורך קריאה של 84 ואינדקס 1 מתוך 8.
הערה: ריצוף הדוגמאות במאמר זה בוצע במתקן GeneCore של EMBL היידלברג (http://www.genecore.embl.de/). - נתח את נתוני ריצוף ה- NGS באמצעות Python 3 ו- biopython. ניתן למצוא את הקבצים בכתובת https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022 (לחילופין בכתובת https://doi.org/10.5281/zenodo.7032215). ניתוח NGS מורכב משני שלבים.
- בשלב הראשון, חפש בקבצי הרצפים רצפים העונים על קריטריונים מסוימים (נוכחות רצפי זיהוי הצמודים לאתר ההכנסה) (ראה איור 4, שלב 1.9.8.5). הדבר נעשה באמצעות סקריפט (Script#1) וקובץ תצורה המספק את המידע הדרוש. לאחר זיהוי הרצף הנכון, התוכנית מחלצת ומאחסנת את הרצף בקובץ הפלט, שהוא קובץ txt בעל שם זהה לקובץ הרצף.
- השלב השני הוא ניתוח קבצי הפלט. הרצפים בספרייה מתחילים בכל אחד מששת הנוקלאוטידים (AGWggc, W =A/T) בתשע חומצות האמינו. בהתבסס על רצף התחלה זה, הפפטיד מתורגם. פעולה זו יוצרת את קובצי הפלט המכילים את גרסאות הפפטיד (PV).
- הכן שתי תיקיות: Script ו- Data. לתיקייה Data, העתק את הקבצים הדחוסים ב- gzip הנובעים מהרצף. לתיקייה Script, העתק את הקבצים הבאים, קובץ Python: Script#1_DetectionExtraction_JoVE_Py3.py; קובץ Python: Script#2_PV_extraction_and_ranking_Py3.py; קובץ תצורה: Barcode_Script_JoVE.conf; וקובץ טבלת חיפוש (LUT): Zuordnung.txt.
- לפני הפעלת קבצי ה- Script, ערוך את הקבצים הבאים בתיקיה Script. פתח את הקובץ "Zuordnung.txt" והוסף בשתי עמודות המופרדות באמצעות טאבים, את שמות קובצי gzip (עמודה 1) ואת השם הסופי הרצוי (עמודה 2; ערכים המופרדים באמצעות טאבים).
הערה: קבצי txt לדוגמה נמצאים בתיקייה "PV_analysis_script" של GitHub. הקבצים שסופקו בתיקייה GitHub מוכנים לניתוח של שלושה נתונים לדוגמה מהספריה לעיל: xaa.txt.gz, xab.txt.gz ו- xac.txt.gz. קבצי הפלט מסופקים גם כן. - שנה את המשתנים הבאים בקובץ התצורה "Barcode_Script_JoVE.conf":
my_dir = "~/נתונים/"
filename_sample_file = "~/Script/Zuordnung.txt"
המשתנים הספציפיים לרצף: BCV_size = 27, BCVשמאל = TCCAGGGCCAG, BCVימין = GCCCAGG, BCVloc = 30,שולי BCV = 8, BCVleft_revcomp = GCCGCCTGGGC, BCVright_revcomp = CTGGCCC ו- BCVloc_revcomp = 41 (ראה איור 4 לפרטים). - השתמש בפקודה הבאה כדי לקרוא לזיהוי ולחילוץ של רצף המשתנים:
>python3 ~/Script#1_DetectionExtraction_JoVE_Py3.py ~/Barcode_Script_JoVE.conf
הערה: הפלט הוא קובצי txt עם רצפי הדנ"א שחולצו ומספר הקריאות שלהם. הכותרת של קובץ זה מכילה נתונים סטטיסטיים (כלומר, המספר הכולל של קריאות וקריאות שחולצו). נתונים אלה מועברים לקבצים הבאים. נתוני txt אלה הם קבצי הקלט של Script#2, שבהם רצפי הדנ"א מתורגמים, מדורגים ומנותחים. - בצע חילוץ וניתוח PV באמצעות הפקודה הבאה:
>python3 ~/Script#2_PV_extraction_and_ranking_Py3.py ~/Barcode_Script_JoVE.conf - נתח את קבצי פלט הטקסט של Script#2. קבצי הפלט של Script#2 נקראים באמצעות העמודה השנייה של LUT ב- "Zuordnung.txt" עם סיומות המבוססות על סוג הניתוח.
הערה: ודא ששלושת קובצי הפלט מכילים נתונים סטטיסטיים בשורות הראשונות ("# של קריאות PV חוקיות", "# של קריאות PV לא חוקיות" ו- "# של קריאות PV ייחודיות"), עמודה ראשונה עם האינדקס של כל רצף DNA מקבצי הקלט txt (פלט של Script#1), והעמודות הבאות: (1) "... analyzed_all.csv": "דגימה:" (רצף DNA), "#" (מספר קריאות), "Frw או Rev" (קריאה קדימה או הפוכה), ו- "PVs" (רצף פפטידי מתורגם). הרצפים הלא חוקיים כוללים "NA" ו- "not valid" בשתי העמודות האחרונות. (2) "... analyzed_validSeq.csv": זהה לקובץ הקודם, מסונן לאיתור רצפים חוקיים. (3) "... analyzed_PV.csv": "PVs" (רצף פפטידי מתורגם), "#" (מספר קריאות) ו- "count" (ספירות FRW ו- rev בקבצים הקודמים מוזגו והספירה ניתנת 1 או 2). - הצג באופן חזותי את קבצי הפלט באמצעות תוכנה זמינה המבוססת על צרכי המשתמש.
2. AAV2 בחירת ספריית תצוגה פפטידית אקראית של 7 מר
- השתמש בספריית AAV לאחר כימות ובקרת איכות (סעיף 1) לאבולוציה מכוונת במודל בחירה כדי לבחור באופן איטרטיבי מועמדים בעלי תכונות רצויות (ראה איור 5)16,18,21.
הערה: מועמדים אלה משמשים לאחר מכן ליצירת ספריית ברקוד כמתואר להלן בסעיף 3.
3. הכנה וניתוח של ספריית קפסיד AAV מקודדת
הערה: לאחר זיהוי קבוצה של קפסידים AAV ספציפיים ויעילים פוטנציאליים במסך התצוגה של הפפטידים, אמת את הפונקציונליות של רצפי הפפטידים שזוהו והשווה אותם לקבוצה של גרסאות APV capsid AAV נפוצות או מתוארות היטב. לשם כך, רצף הקפסיד מוכנס למבנה עוזר Rep/Cap ללא ITR.
- הפקת ספריית AAV ברקודית
- בצע ייצור AAV רקומביננטי עבור כל וריאנט קפסיד באמצעות מערכת שלושת הפלסמיד, כפי שתואר קודם לכן24.
הערה: כדי להבדיל בין גרסאות הקפסיד השונות, פלסמיד הטרנסגן של כתב ITR מכיל ברקוד ייחודי באורך של 15 נוקלאוטידים. הברקוד ממוקם ב-3' UTR (אזור לא מתורגם) בין החלבון הפלואורסצנטי הצהוב המשופר (EYFP) לבין אות ה-polyA (ראו איור 6A). ביטוי EYFP מונע על ידי מקדם ציטומגלווירוס (CMV) חזק בכל מקום המספק רמות מספיקות של תעתיקי RNA. - תכנון ברקודים באורך 15 נוקלאוטידים עם הומופולימרים של פחות משלושה נוקלאוטידים, תכולת GC של <65%29, ומרחק המינג גדול מארבעה נוקלאוטידים24.
- מייצרים כל קפסיד בנפרד בשילוב עם פלסמיד טרנסגני הנושא ברקוד ייחודי. בדרך זו, כל וריאנט קפסיד מתויג בברקוד ייחודי המאפשר מעקב ספציפי (ראה איור 6B).
- בצע ייצור AAV רקומביננטי עבור כל וריאנט קפסיד באמצעות מערכת שלושת הפלסמיד, כפי שתואר קודם לכן24.
- טיטרציה וקטורית AAV באמצעות dd-PCR
- בצע את הטיטרציה AAV כפי שתואר קודם לכן בסעיף 1.8, על-ידי החלפת זוג הפריימר Rep2 בזוג הפריימר YFP.
- כמת את הפקות AAV הבודדות ואסוף כמויות שוות של כל הפקה כדי ליצור את ספריית הברקוד הסופית.
- כמת שוב את הספרייה הסופית כדי לבדוק את הריכוז והאיכות הסופיים (ראה איור 7).
- ספריית AAV מקודדת ביישום vivo
- החל את ספריית AAV המקודדת באופן שיטתי על מערכת המודל הנבחרת (למשל באופן מערכתי בעכברים24).
- אספו רקמות על ומחוץ למטרה (כלומר, כבד, ריאות, לב, דיאפרגמה, שריר חלק, תריסריון, לבלב, מעי גס, שרירי הזרוע, השחלות, קיבה, אוזן פנימית, כליות, אבי העורקים הבטני, אבי העורקים החזי, מוח, שומן חום ולבן וטחול) או סוגי תאים על סמך הניסוי. להקפיא אותם ב -80 ° C, לחלץ את ה- DNA/RNA, וליישם ניתוח כימות NGS, כמתואר בסעיף הבא.
- מיצוי DNA/RNA
- חלצו את הדנ"א והרנ"א מהרקמות המעניינות באמצעות ערכת המיני DNA/RNA.
- מניחים חתיכה קטנה של הרקמה של עניין (1 מ"מ3, כ 5 מ"ג) בצינור תגובה 2 מ"ל.
- הוסף 350 μL של חיץ ליזיס מעורבב עם β-mercaptoethanol (1%) וחרוזי פלדה 5 מ"מ לרקמה (לטפל בדגימות עם β-mercaptoethanol מתחת מכסה אדים).
- הומוגניזציה של הרקמה ברקמה במשך 45 שניות ב 40 הרץ.
- יש להוסיף 10 μL של פרוטאינאז K (10 מ"ג/מ"ל) ולדגור במשך 15 דקות ב-55°C תוך כדי רעידות ב-400 סל"ד.
- צנטריפוגה ב 20,000 x גרם במשך 3 דקות בטמפרטורת החדר, לאסוף את supernatant, ולהמשיך עם פרוטוקול היצרן של ערכת DNA/RNA.
- פצלו את שלב הכביסה לשני שלבים עם 350 מיקרוליטר של חיץ כביסה בכל שלב. בין שלבי השטיפה הללו, עיכלו את שאריות הדנ"א שעל העמוד עם DNase I נטול RNase. הוסיפו 80 μL של תמיסת DNase I, שהוכנה על פי הוראות היצרן, על העמוד ודגרו בטמפרטורת החדר למשך 15 דקות.
- Elute RNA/DNA מהעמודה עם מים נטולי נוקלאז. אחסן את הרנ"א המבודד ב -80 ° C ואת gDNA ב -20 ° C.
- סינתזת cDNA
- העבירו את דגימות ה-RNA לסבב נוסף של טיפול DNase I למשך 15-30 דקות (לסילוק מלא של דנ"א מזהם מדגימות ה-RNA) לפני תגובת השעתוק ההפוך. הוסף 1 μL של תמיסת DNase I, 4 μL של חיץ (מסופק עם הערכה), ומים ללא nuclease לנפח סופי של 40 μL עד 212 ng של RNA. יש לדגור במשך 30 דקות בטמפרטורת החדר ולהשבית את החום ב-70°C למשך 10 דקות.
- לסנתז cDNA, באמצעות 150 ng של RNA באמצעות ערכה על פי הוראות היצרן. כלול פקדים ללא התמליל ההפוך, כדי להבטיח היעדר DNA נגיפי מזהם מהדגימה. cDNA מאוחסן ב -20 ° C.
הערה: כמות RNA הקלט לשעתוק לאחור אופטימלי עשויה להשתנות בהתאם לסוג הרקמה וליעילות ההולכה הצפויה ברקמה המתאימה.
- ניתוח הספרייה הנגיפית AAV (in-vivo) על ידי NGS
- כדי להשיג עומק רצף גבוה בעלות נמוכה, בצע NGS באמצעות ריצוף Illumina כמתואר קודם לכן (סעיף 1.9). הגבירו את רצף הברקוד, ולאחר מכן קשרו את מתאמי הרצף לאמפליקון.
- בשל אורך הקריאה הקצר וקשירת מתאמי הרצף משני צידי האמפליקון, בעת התכנון, בדוק כי האמפליקון קטן מספיק כדי להבטיח נוכחות של רצף הברקוד בתוך קריאת NGS. לצורך ריצוף הברקודים בתוך הגנום הנגיפי והתעתיקים הנגיפיים, אמפליקון ה-PCR מתוכנן להיות באורך של 113 bp (ראה איור 8).
- הגבר את האזור הברקודי עם הפריימרים BC-seq קדימה ו- BC-seq הפוך. הכן את תגובת ה-PCR הבאה: 0.5 μL של פולימראז DNA באיכות גבוהה, 10 μL של 5x buffer, 0.25 μL של כל פריימר 100 μM (BC-seq fw/BC-seq rv), ו-1 μl של 10 mM dNTPs. השתמש ב- 25 ננוגרם של cDNA או DNA/תגובה כתבנית והתאם את הנפח הסופי ל- 50 μL עם מים נטולי נוקלאז.
- הכינו את תערובת האב PCR מתחת למכסה מנוע PCR נקי כדי למנוע זיהום. השתמש בתנאי הרכיבה הבאים: 30 שניות ב- 98 ° C, ולאחר מכן 40 מחזורים ב- 98 ° C במשך 10 שניות ו- 72 ° C במשך 20 שניות, וצעד אחרון של 5 דקות ב- 72 ° C.
- כלול בקרות PCR כדי לאשר את היעדר ה- DNA המזהם בתמהיל האב של PCR. עבור דגימות cDNA, כלול את הפקדים ללא תעתיק הפוך. לבסוף, כלול דוגמה עם ספריית הקלט AAV. מידע זה ישמש ליצירת קובץ Normalization_Variant.txt המשמש בניתוח.
- ודא את גודל מקטע ה- PCR של כל דגימה על ידי אלקטרופורזה בג'ל לפני טיהור PCR. האחרון מושג על ידי שימוש בחרוזים מגנטיים הזמינים מסחרית או במערכות טיהור DNA מבוססות עמודות (ראה טבלת חומרים).
- הכן את ספריית NGS באמצעות מערכת הספרייה לדגימות בעלות מורכבות נמוכה בהתאם להוראות היצרן, כמתואר בסעיף 1.9.
- קבע את ריכוז ה- DNA באמצעות ערכת dsDNA HS ונתח את איכות הספרייה כמתואר קודם לכן (סעיף 1.9.6), ולאחר מכן איגום. כמת את הספרייה המאוגמת על פלואורומטר והערך את האיכות בביואנלייזר.
- ביצוע ריצוף NGS כמתואר בסעיף 1.9.7.
- כמת על ידי qPCR את מספר העותק של הטרנסגן (גנום ויראלי) ואת גן משק הבית כדי להעריך את התפלגות הספרייה המאוגמת בין רקמות או איברים על ה- DNA.
- הגדר תגובת qPCR של 30 μL באופן הבא, כדי לקבוע את מספר העותק של EYFP (טרנסגן) ו- GAPDH (גליצראלדהיד 3-פוספט דהידרוגנאז, גן משק בית):
- הכינו תערובת פריימר/בדיקה 60x עבור EYFP (1.5 μM YFP_fw, 1.5 μM YFP_rv ו-0.6 μM YFP_probe; ראו טבלת חומרים). השתמש בתערובת פריימר/בדיקה GAPDH (ראה טבלת חומרים) כדי לקבוע את מספר העותק של הגן של עוזרת הבית. הגדר את התגובה על קרח.
- הכינו תערובת מאסטר PCR (15 μL, ראו טבלת חומרים) והוסיפו תערובת פריימר/בדיקה 60x (0.5 μL) לכל הדגימות והתקנים (כדי לחשב מספרי העתקה עבור התקנים, השתמשו בקישור הבא: http://cels.uri.edu/gsc/cndna.html). הגדר את התגובה על קרח.
- מעבירים 15.5 מיקרוליטר של תערובת האב לצלחת של 96 בארות ומוסיפים 14.5 מיקרוליטר של דגימה (75 ננוגרם של ריכוז DNA כולל) או סטנדרטי לבאר המתאימה. אטמו את הצלחת בעלת 96 הבארות בעזרת נייר כסף, מערבולות וסחררו לזמן קצר.
- מעבירים 10 μL מכל דגימה לצלחת של 384 בארות בכפילויות. אטמו את הצלחת בנייר כסף וסובבו ב 800 x גרם במשך 5 דקות ב 4 °C.
- לדגור את תערובת התגובה thermocycler באמצעות טמפרטורה התחלתית של 50 ° C במשך 2 דקות, ואחריו שלב ההפעלה הראשוני של 10 דקות ב 95 ° C. בצע 40 מחזורים של דנטורציה ב 95 ° C במשך 15 שניות ו חישול / הרחבה ב 60 ° C במשך 1 דקה24.
- כדי לקבל את מספר הגנומים הדיפלואידיים (dg), השתמש במספר העתק GAPDH וחלק בשניים. לאחר מכן, קח את הערך של מספר העתק EYFP וחלק במספר dg, וכתוצאה מכך גנום וקטורי לכל גנום דיפלואידי (vg/dg). השתמש בערך זה כדי ליצור את קובץ Normalization_Organ.txt עבור הניתוח הביואינפורמטיקה.
- בצע את ניתוח נתוני ריצוף NGS כמו Weinmann et al.24שימוש בקוד מותאם אישית ב- Python3 (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022)., תהליך העבודה כולל זיהוי רצפי ברקוד המונחים על ידי רצפי איגוף, אורכם ומיקומם (Script#1_BarcodeDetection.py), כמו גם ניתוח של העשרת ברקוד ופיזורו על פני קבוצת הרקמות (Script#2_BarcodeAnalysis.py).
- זהה ברקוד והקצה אותם לגרסאות AAV. מקם את נתוני הרצף כקבצי fastq מאוחסנים בארכיון בספרייה אחת (לדוגמה, "Data_to_analyze"). קובץ נתוני הרצף של ספריית הקלט נכלל בספריה זו ומשמש רק לחישוב יחסי הקפסיד בספריית הקלט.
- לפני הפעלת הסקריפט, צור שני קובצי טקסט המופרדים באמצעות טאבים: קובץ גרסאות Capsid (ראה קובץ לדוגמה "Variants.txt") עם רצפי הברקוד שהוקצו לשמות וריאנטים של AAV capsid, וקובץ הזיהום (ראה "Contaminations.txt") עם רצפי ברקוד שמקורם בזיהום אפשרי (ברקודים אחרים הזמינים במעבדה תורמים לזיהום).
- לבסוף, ערוך את קובץ התצורה "Barcode_Script.conf" כך שיכלול את המידע הבא: נתיב לתיקייה עם נתוני רצף (לדוגמה, "Data_to_analyze"), רצף אזורי איגוף של הברקודים, מיקומם וגודל החלון לזיהוי ברקוד (בדומה ל- 1.9.8.5, ראה איור 8).
- השתמש בפקודה הבאה כדי לקרוא לזיהוי ברקוד עם נתיבים שסופקו לקבצי Script#1_BarcodeDetection.py ותצורה:
>python3 ~/Script#1_BarcodeDetection.py ~/Barcode_Script.conf
הערה: הפלט של ביצוע Script#1_BarcodeDetection.py הוא קבצי טקסט עם ספירות קריאה לכל משתנה capsid וכן המספר הכולל של קריאות ששוחזרו מהנתונים הגולמיים. - הערך את ההתפלגות של קפסידים AAV ברקודים בין רקמות או איברים, על ידי הפעלת Script#2_BarcodeAnalysis.py יחד עם קבצי txt הבאים:
- בקובץ "Zuordnung.txt", הקצו את השם לכל קובץ txt שהתקבל מהפעלת זיהוי הברקוד לשם רקמה/איבר: שמות קובצי txt בעמודה הראשונה ושמות רקמות/איברים מתאימים בהקצאה המופרדת בטאבים.
הערה: לדוגמה, היכנס לתיקייה "דוגמה" (https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022). יש לציין כי שם הרקמה/איבר יכול לכלול תווים המגדירים מדידת cDNA או gDNA ומספר שכפול ביולוגי (M1, M2 וכו'). - צור קובץ טקסט "organs.txt" עם רשימת השמות עבור איברי יעד ON ו- OFF, המתאימים לשמות שניתנו בקובץ המשימה "Zuordnung.txt" (ראה תיקיית "דוגמה": https://github.com/grimmlabs/AAV_GrimmLab_JoVE2022).
- צור קובצי טקסט המופרדים באמצעות טאבים "Normalization_Organ.txt" ו- "Normalization_Variant.txt" עם ערכים מנורמלים עבור כל גרסאות הקפסיד וכל האיברים/רקמות. בעמודה הראשונה של הקובץ "Normalization_Organ.txt", כתוב את השמות שניתנו עבור כל איבר (כמו בקובץ המשימה "Zuordnung.txt") ובעמודה השנייה את ערכי הנורמליזציה עבור הרקמות המתאימות, שנוצרו בסעיף 3.6.11.
- מלא את העמודה הראשונה של הקובץ "Normalization_Variant.txt" ברשימת שמות הקפסיד ואת העמודה השנייה בערכים המנורמלים של ספירות הקריאה עבור כל Capsid בספריה המאוגמת (ניתן לחשב נורמליזציה בהתבסס על קובץ הפלט txt עבור ספריית הקלט הנובעת מהסקריפט הראשון).
- ערוך את קובץ התצורה על-ידי ציון הנתיבים המלאים לכל הקבצים הנוספים שהוזכרו לעיל. הפעל את Script#2_BarcodeAnalysis.py בתור:
>python3 /script#2_BarcodeAnalysis.py ~/Barcode_Script.conf
הערה: סקריפט ניתוח הברקוד מפיק מספר קבצים: קובצי טקסט עם ערכי ריכוז יחסי (RC) של התפלגות Capsid ברקמות שונות בהתבסס על שלבי נורמליזציה מרובים שתוארו קודם לכן, וקובץ הגיליון האלקטרוני המשלב נתוני קובץ טקסט לנתוני מטריצה ממוזגים. האחרון יכול לשמש ניתוח אשכולות ויזואליזציה. - הצג את הנתונים באופן חזותי ובצע ניתוח אשכולות של נתוני המטריצה על מנת להבחין בתכונות הקפסיד ולהעריך את הדמיון ביניהם בהתבסס על פרופילי RC על פני רקמות. השתמש בסקריפט הנוסף PCA_heatmap_plot. R ממוקם במאגר:
>Rscript --וניל ~/PCA. R ~ / ריכוז יחסי.xls
הערה: הסקריפט לוקח קבצים יחסיים.xls כקלט ומפיק שתי חלקות של מפת חום היררכית של אשכולות וניתוח רכיבים ראשיים (PCA). - כדי לשנות חלקות (צירים של מפת חום, רכיבים עיקריים של PCA) או פרמטרים png (צבע, גודל, תיוג), פתח את סקריפט R ופעל לפי ההוראות המופיעות בסעיפים עם הערות.
- בקובץ "Zuordnung.txt", הקצו את השם לכל קובץ txt שהתקבל מהפעלת זיהוי הברקוד לשם רקמה/איבר: שמות קובצי txt בעמודה הראשונה ושמות רקמות/איברים מתאימים בהקצאה המופרדת בטאבים.
תוצאות
יצירת ספריית תצוגה של פפטיד AAV2. כצעד ראשון לקראת הבחירה של AAV מהונדס, הדור של ספריית פלסמיד מתואר. תוספת הפפטיד מיוצרת באמצעות פריימרים מנוונים. להפחתת השילוב של קודונים באלה בין 64 ל -20 יש את היתרונות של חיסול קודוני עצירה והקלה על ניתוח NGS, על ידי הפחתת מגוון הספרייה על הדנ"א אך לא ע...
Discussion
בפרוטוקול זה, מתוארים השלבים הדרושים להנדסת קפסיד AAV של תצוגת פפטיד ולהקרנת ספריית AAV עם ברקוד, כמו גם לניתוח ביואינפורמטי של הרכב הספרייה וביצועי הקפסיד. פרוטוקול זה מתמקד בצעדים המאפשרים ניתוח ביואינפורמטי של ספריות מסוג זה, מכיוון שרוב מעבדות הווירולוגיה מפגרות בכישורי תכנות כדי להתאי...
Disclosures
D.G. הוא מייסד שותף של AaviGen GmbH. D.G. ו- K.R. הם ממציאים של בקשת פטנט תלויה ועומדת הקשורה ליצירת גרסאות קפסיד AAV חומקות חיסון. לשאר המחברים אין מה לחשוף.
Acknowledgements
D.G. מעריכה מאוד את תמיכתה של קרן המחקר הגרמנית (DFG) באמצעות מרכזי המחקר השיתופיים של DFG SFB1129 (Projektnummer 240245660) ו-TRR179 (Projektnummer 272983813), כמו גם על ידי המרכז הגרמני לחקר זיהומים (DZIF, BMBF; TTU-HIV 04.819).
Materials
| Name | Company | Catalog Number | Comments |
| Amplification primer | ELLA Biotech (Munich, Germany) | - | Second-strand synthesis of oligonucleotide insert |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | DNA fragment validation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | DNA fragment validation |
| AllPrep DNA/RNA Mini Kit | Qiagen (Venlo, Netherlands) | 80204 | DNA/RNA extraction |
| Agilent DNA 1000 Reagents | Agilent Technologies (Santa Clara, CA, USA) | 5067-1504 | NGS Library preparation |
| Agilent 2100 Bioanalyzer System | Agilent Technologies (Santa Clara, CA, USA) | G2938C | NGS Library preparation |
| BC-seq fw: | IDT (San Joce, CA, CA, USA) | ATCACTCTCGGCATGGACGAGC | NGS Library preparation |
| BC-seq rv: | IDT (San Joce, CA, CA, USA) | GGCTGGCAACTAGAAGGCACA | NGS Library preparation |
| β-Mercaptoethanol | Millipore Sigma (Burlington, MA, USA) | 44-420-3250ML | DNA/RNA extraction |
| BglI | New England Biolabs (Ipswich, MA, USA) | R0143 | Digestion of double-stranded insert |
| C1000 Touch Thermal Cycler | Bio-Rad (Hercules, CA, USA) | 1851196 | dd-PCR cycler |
| dNTPS | New England Biolabs (Ipswich, MA, USA) | N0447S | NGS Library preparation |
| ddPCR Supermix for probes (no dUTP) | Bio-Rad (Hercules, CA, USA) | 1863024 | dd-PCR supermix |
| Droplet Generation Oil for Probes | Bio-Rad (Hercules, CA, USA) | 1863005 | dd-PCR droplet generation oil |
| DG8 Cartridges for QX100 / QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864008 | dd-PCR droplet generation cartridge |
| DG8 Cartridge Holder | Bio-Rad (Hercules, CA, USA) | 1863051 | dd-PCR cartridge holder |
| Droplet Generator DG8 Gasket | Bio-Rad (Hercules, CA, USA) | 1863009 | dd-PCR cover for cartridge |
| ddPCR Plates 96-Well, Semi-Skirted | Bio-Rad (Hercules, CA, USA) | 12001925 | dd-PCR 96-well plate |
| E.cloni 10G SUPREME Electrocompetent Cells | Lucigen (Middleton, WI, USA) | 60081-1 | Electrocompetent cells |
| Electroporation cuvettes, 1mm | Biozym Scientific (Oldendorf, Germany) | 748050 | Electroporation |
| GAPDH primer/probe mix | Thermo Fischer Scientific (Waltham, MA, USA) | Mm00186825_cn | Taqman qPCR primer |
| Genepulser Xcell | Bio-Rad (Hercules, CA, USA) | 1652660 | Electroporation |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystems (Waltham, MA, USA) | 4368814 | cDNA reverse transcription |
| ITR_fw | IDT (San Joce, CA, USA) | GGAACCCCTAGTGATGGAGTT (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_rv | IDT (San Joce, CA, USA) | CGGCCTCAGTGAGCGA (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR primer |
| ITR_probe | IDT (San Joce, CA, USA) | HEX-CACTCCCTCTCTGCGCGCTCG-BHQ1 (https://signagen.com/blog/2019/10/25/qpcr-primer-and-probe-sequences-for-raav-titration/) | dd-PCR probe |
| Illumina NextSeq 500 system | Illumina Inc (San Diego, CA, USA) | SY-415-1001 | NGS Library sequencing |
| KAPA HiFi HotStart ReadyMix (2X)* | Roche AG (Basel, Switzerland) | KK2600 07958919001 | NGS sample prepration |
| MagnaBot 96 Magnetic Separation Device | Promega GmbH (Madison, WI, USA) | V8151 | Sample prepration for NGS library |
| NanoDrop 2000 spectrophotometer | Thermo Fischer Scientific (Waltham, MA, USA) | ND-2000 | Digestion of double-stranded insert |
| NGS_frw | Sigma-Aldrich (Burlinght, MA, USA) | GTT CTG TAT CTA CCA ACC TC | NGS primer |
| NGS_rev | Sigma-Aldrich (Burlinght, MA, USA) | CGC CTT GTG TGT TGA CAT C | NGS primer |
| NextSeq 500/550 High Output Kit (75 cycles) | Illumina Inc (San Diego, CA, USA) | FC-404-2005 | NGS Library sequencing |
| Ovation Library System for Low Complexity Samples Kit | NuGEN Technologies, Inc. (San Carlos, CA, USA) | 9092-256 | NGS Library preparation |
| PX1 Plate Sealer | Bio-Rad (Hercules, CA, USA) | 1814000 | dd-PCR plate sealer |
| Pierceable Foil Heat Seal | Bio-Rad (Hercules, CA, USA) | 1814040 | dd-PCR sealing foil |
| Phusion High-Fidelity DNA-Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F530S | Second-strand synthesis of oligonucleotide insert |
| PEI MAX - Transfection Grade Linear Polyethylenimine Hydrochloride (MW 40,000) | Polysciences, Inc. (Warrington, PA, USA) | 24765-1G | AAV library preparation |
| ProNex Size-Selective Purification System | Promega GmbH (Madison, WI, USA) | NG2002 | Sample prepration for NGS library |
| Phusion Hot Start II Polymerase | Thermo Fischer Scientific (Waltham, MA, USA) | F549L | NGS Library preparation |
| Proteinase K | Roche AG (Basel, Switzerland) | 5963117103 | DNA/RNA extraction |
| pRep2Cap2_PIS | ITR-Rep2Cap2-ITR vector. Peptide insertion site within the Cap2 ORF, manufactured/prepared in the lab | ||
| QX200 Droplet Generator | Bio-Rad (Hercules, CA, USA) | 1864002 | dd-PCR droplet generator |
| QX200 Droplet Reader | Bio-Rad (Hercules, CA, USA) | 1864003 | dd-PCR droplet analysis |
| QIAquick Nucleotide Removal Kit | Qiagen (Venlo, Netherlands) | 28306 | Second-strand synthesis of oligonucleotide insert purification |
| QIAquick Gel Extraction Kit | Qiagen (Venlo, Netherlands) | 28704 | Plasmid vector purification |
| QIAGEN Plasmid Maxi Kit | Qiagen (Venlo, Netherlands) | 12162 | Plasmid library DNA preparation |
| Qiaquick PCR Purification kit | Qiagen (Venlo, Netherlands) | 28104 | Sample prepration for NGS library |
| Qubit fluorometer | Invitrogen (Waltham, MA, USA) | Q32857 | NGS Library preparation |
| Qubit dsDNA HS | Thermo Fischer Scientific (Waltham, MA, USA) | Q32851 | NGS Library preparation |
| QuantiFast PCR Master Mix | Qiagen (Venlo, Netherlands) | 1044234 | Taqman qPCR |
| rep_fw | IDT (San Joce, CA, USA) | AAGTCCTCGGCCCAGATAGAC | dd-PCR primer |
| rep_rv | IDT (San Joce, CA, USA) | CAATCACGGCGCACATGT | dd-PCR primer |
| rep_probe | IDT (San Joce, CA, USA) | FAM-TGATCGTCACCTCCAACA-BHQ1 | dd-PCR probe |
| RNase-free DNase | Qiagen (Venlo, Netherlands) | 79254 | DNA/RNA extraction |
| SfiI | New England Biolabs (Ipswich, MA, USA) | R0123 | Digestion of vector |
| 5 mm, steel Beads | Qiagen (Venlo, Netherlands) | 69989 | DNA/RNA extraction |
| TRIMER-oligonucleotides | ELLA Biotech (Munich, Germany) | - | Degenerate oligonucleotide |
| T4 Ligase | New England Biolabs (Ipswich, MA, USA) | M0202L | Plasmid library ligation |
| TissueLyserLT | Qiagen (Venlo, Netherlands) | 85600 | DNA/RNA extraction |
| YFP_fw | IDT (San Joce, CA, USA) | GAGCGCACCATCTTCTTCAAG | dd-PCR primer |
| YFP_rv | IDT (San Joce, CA, USA) | TGTCGCCCTCGAACTTCAC | dd-PCR primer |
| YFP_probe | IDT (San Joce, CA, USA) | FAM-ACGACGGCAACTACA-BHQ1 | dd-PCR probe |
| Zymo DNA Clean & Concentrator-5 (Capped) | Zymo research (Irvine, CA, USA) | D4013 | Vector and Ligation purification |
References
- Wang, D., Tai, P. W. L., Gao, G. Adeno-associated virus vector as a platform for gene therapy delivery. Nature Reviews Drug Discovery. 18 (5), 358-378 (2019).
- Muhuri, M., Levy, D. I., Schulz, M., McCarty, D., Gao, G. Durability of transgene expression after rAAV gene therapy. Molecular Therapy. 30 (4), 1364-1380 (2022).
- Li, C., Samulski, R. J. Engineering adeno-associated virus vectors for gene therapy. Nature Reviews Genetics. 21 (4), 255-272 (2020).
- Kuzmin, D. A., et al. The clinical landscape for AAV gene therapies. Nature Reviews Drug Discovery. 20 (3), 173-174 (2021).
- Mullard, A. Gene therapy community grapples with toxicity issues, as pipeline matures. Nature Reviews Drug Discovery. 20 (11), 804-805 (2021).
- Nature Biotechnology. Gene therapy at the crossroads. Nature Biotechnology. 40 (5), 621 (2022).
- Becker, P., et al. Fantastic AAV Gene Therapy Vectors and How to Find Them-Random Diversification, Rational Design and Machine Learning. Pathogens. 11 (7), 756 (2022).
- Perabo, L., et al. In vitro selection of viral vectors with modified tropism: the adeno-associated virus display. Molecular Therapy. 8 (1), 151-157 (2003).
- Muller, O. J., et al. Random peptide libraries displayed on adeno-associated virus to select for targeted gene therapy vectors. Nature Biotechnology. 21 (9), 1040-1046 (2003).
- Dalkara, D., et al. In vivo-directed evolution of a new adeno-associated virus for therapeutic outer retinal gene delivery from the vitreous. Science Translational Medicine. 5 (189), (2013).
- Sahel, J. A., et al. Partial recovery of visual function in a blind patient after optogenetic therapy. Nature Medicine. 27 (7), 1223-1229 (2021).
- Adachi, K., Enoki, T., Kawano, Y., Veraz, M., Nakai, H. Drawing a high-resolution functional map of adeno-associated virus capsid by massively parallel sequencing. Nature Communications. 5, 3075 (2014).
- Marsic, D., Mendez-Gomez, H. R., Zolotukhin, S. High-accuracy biodistribution analysis of adeno-associated virus variants by double barcode sequencing. Molecular Therapy-Methods & Clinical Development. 2, 15041 (2015).
- Korbelin, J., et al. Pulmonary targeting of adeno-associated viral vectors by next-generation sequencing-guided screening of random capsid displayed peptide libraries. Molecular Therapy. 24 (6), 1050-1061 (2016).
- Deverman, B. E., et al. Cre-dependent selection yields AAV variants for widespread gene transfer to the adult brain. Nature Biotechnology. 34 (2), 204-209 (2016).
- Ravindra Kumar, S., et al. Multiplexed Cre-dependent selection yields systemic AAVs for targeting distinct brain cell types. Nature Methods. 17 (5), 541-550 (2020).
- Hanlon, K. S., et al. Selection of an efficient AAV vector for robust CNS transgene expression. Molecular Therapy-Methods & Clinical Development. 15, 320-332 (2019).
- Nonnenmacher, M., et al. Rapid evolution of blood-brain-barrier-penetrating AAV capsids by RNA-driven biopanning. Molecular Therapy-Methods & Clinical Development. 20, 366-378 (2021).
- Tabebordbar, M., et al. Directed evolution of a family of AAV capsid variants enabling potent muscle-directed gene delivery across species. Cell. 184 (19), 4919-4938 (2021).
- Davidsson, M., et al. A systematic capsid evolution approach performed in vivo for the design of AAV vectors with tailored properties and tropism. Proceedings of the National Academy of Sciences. 116 (52), 27053-27062 (2019).
- Pekrun, K., et al. Using a barcoded AAV capsid library to select for clinically relevant gene therapy vectors. Journal of Clinical Investigation Insight. 4 (22), (2019).
- Ogden, P. J., Kelsic, E. D., Sinai, S., Church, G. M. Comprehensive AAV capsid fitness landscape reveals a viral gene and enables machine-guided design. Science. 366 (6469), 1139-1143 (2019).
- Kondratov, O., et al. A comprehensive study of a 29-capsid AAV library in a non-human primate central nervous system. Molecular Therapy. 29 (9), 2806-2820 (2021).
- Weinmann, J., et al. Identification of a myotropic AAV by massively parallel in vivo evaluation of barcoded capsid variants. Nature Communications. 11 (1), 5432 (2020).
- Kremer, L. P. M., et al. High throughput screening of novel AAV capsids identifies variants for transduction of adult NSCs within the subventricular zone. Molecular Therapy-Methods & Clinical Development. 23, 33-50 (2021).
- Borner, K., et al. Pre-arrayed pan-AAV peptide display libraries for rapid single-round screening. Molecular Therapy. 28 (4), 1016-1032 (2020).
- Kienle, E., et al. Engineering and evolution of synthetic adeno-associated virus (AAV) gene therapy vectors via DNA family shuffling. Journal of Visualized Experiments. (62), e3819 (2012).
- Furuta-Hanawa, B., Yamaguchi, T., Uchida, E. Two-dimensional droplet digital PCR as a tool for titration and integrity evaluation of recombinant adeno-associated viral vectors. Human Gene Therapy Methods. 30 (4), 127-136 (2019).
- Lyons, E., Sheridan, P., Tremmel, G., Miyano, S., Sugano, S. Large-scale DNA barcode library generation for biomolecule identification in high-throughput screens. Scientific Reports. 7 (1), 13899 (2017).
- Korbelin, J., et al. Optimization of design and production strategies for novel adeno-associated viral display peptide libraries. Gene Therapy. 24 (8), 470-481 (2017).
- Korbelin, J., Trepel, M. How to successfully screen random adeno-associated virus display peptide libraries in vivo. Human Gene Therapy Methods. 28 (3), 109-123 (2017).
- Herrmann, A. K., et al. A robust and all-inclusive pipeline for shuffling of adeno-associated viruses. American Chemical Society Synthetic Biology. 8 (1), 194-206 (2019).
- Choudhury, S. R., et al. In vivo selection yields AAV-B1 Capsid for central nervous system and muscle gene therapy. Molecular Therapy. 24 (7), 1247-1257 (2016).
- Buschmann, T., Bystrykh, L. V. Levenshtein error-correcting barcodes for multiplexed DNA sequencing. BMC Bioinformatics. 14, 272 (2013).
- Buschmann, T. DNABarcodes: an R package for the systematic construction of DNA sample tags. Bioinformatics. 33 (6), 920-922 (2017).
- Li, B., et al. A comprehensive mouse transcriptomic BodyMap across 17 tissues by RNA-seq. Scientific Reports. 7 (1), 4200 (2017).
- Clarner, P., et al. Development of a one-step RT-ddPCR method to determine the expression and potency of AAV vectors. Molecular Therapy-Methods & Clinical Development. 23, 68-77 (2021).
- Zolotukhin, S., Vandenberghe, L. H. AAV capsid design: A Goldilocks challenge. Trends in Molecular Medicine. 28 (3), 183-193 (2022).
- Brown, D., et al. deep parallel characterization of AAV tropism and AAV-mediated transcriptional changes via single-cell RNA sequencing. Frontiers in Immunology. 12, 730825 (2021).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved