A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
ניתוח מרחבי ברזולוציה גבוהה של רמות ביטוי גנים בגידים
In This Article
Summary
מאמר זה מתאר כיצד לבצע פרוטוקול אופטימלי באתרו עבור גידים. שיטה זו דנה בהכנת רקמות, חדירות חתך, תכנון בדיקה ושיטות הגברת אותות.
Abstract
בשנים האחרונות פותחו פרוטוקולים רבים לתעתיק ברזולוציה גבוהה בתחומים רבים ושונים של רפואה וביולוגיה. עם זאת, רקמות עשירות במטריצה, ובמיוחד גידים, נותרו מאחור בשל מספר התאים הנמוך שלהם, כמות ה-RNA הנמוכה לתא ותכולת המטריצה הגבוהה, מה שהפך אותם למסובכים לניתוח. אחד הכלים האחרונים והחשובים ביותר של תא בודד הוא ניתוח מרחבי של רמות ביטוי גנים בגידים. לכלים מרחביים אלה של RNA יש חשיבות גבוהה במיוחד בגידים לאיתור תאים ספציפיים של אוכלוסיות חדשות ולא ידועות, לאמת תוצאות RNA-seq של תא בודד ולהוסיף הקשר היסטולוגי לנתוני ה-RNA-seq של תא בודד. שיטות חדשות אלו יאפשרו ניתוח RNA בתאים בעלי רגישות יוצאת דופן וזיהוי מטרות RNA של מולקולה בודדת ברמת התא הבודד, מה שיסייע לאפיין מולקולרית גידים ולקדם את חקר הגידים.
במאמר שיטה זה, נתמקד בשיטות הזמינות לניתוח רמות ביטוי גנים מרחביות בחתכים היסטולוגיים על ידי שימוש במבחני הכלאה חדשניים באתרם כדי לזהות RNA מטרה בתוך תאים שלמים ברמות תא בודד. ראשית, נתמקד כיצד להכין את רקמת הגיד לבדיקות השונות הזמינות וכיצד להגביר אותות ספציפיים למטרה ללא רעשי רקע אך עם רגישות גבוהה וסגוליות גבוהה. לאחר מכן, המאמר יתאר שיטות חדירה ספציפיות, עיצובי הבדיקה השונים ואסטרטגיות הגברת האותות הקיימות כיום. שיטות ייחודיות אלו לניתוח רמות שעתוק של גנים שונים ברזולוציה של תא בודד יאפשרו זיהוי ואפיון של תאי רקמת הגיד באוכלוסיות צעירות ומבוגרות של מודלים שונים של בעלי חיים ורקמות גידים אנושיות. שיטה זו תסייע גם בניתוח רמות ביטוי גנים ברקמות עשירות במטריצות אחרות כגון עצמות, סחוס ורצועות.
Introduction
גידים הם רקמות חיבור המאפשרות העברת כוח בין שריר לעצם1. מבחינה התפתחותית, טנוציטים ציריים נגזרים מתאים מזנכימליים בתוך הסקלרוטום של הסומיטים2; גידי הגפיים נובעים מהמזודרם של הצלחת הרוחבית; וגידים גולגולתיים נובעים משושלת הפסגה העצבית הגולגולתית 3,4. ניתן לאפיין את הגיד על ידי ביטוי של גורם השעתוק של סקלרקסיס5, אם כי מספר סמנים ממלאים גם תפקיד מפתח בהתפתחות הגידים, כולל טנומודולין, מוהוק ותגובת גדילה מוקדמת 1/2 6,7,8,9.
למרות הסמנים המעטים הידועים של הגיד, באופן כללי, אפיון מעמיק יותר נותר מאתגר מכיוון שהגיד מכיל תאים המשתרעים על פני שיפוע של תכונות ביומכניות. מהצומת המיוטנדינית, אמצע הגיד באמצע הגוף והאנטזה המסוידת יותר, תאי הגיד שוכנים במטריצות חוץ-תאיות הנעות בתכונות מתיחה. מכיוון שהגיד חייב לעמוד במתח מתיחה המוטל על ידי ההבדל בחוזק המכני בין רקמה רכה וקשה, הארגון המרחבי של התאים בגיד חשוב במיוחד לתפקודו. עם זאת, מעט ידוע על תת-אוכלוסיות גידים אלה.
ניתן להשתמש בכלים טרנסקריפטומיים מרחביים רבים ברזולוציה גבוהה כדי להתחיל להבהיר תת-אוכלוסיות תאים, כולל אך לא רק, RNA Seq של תא בודד או הכלאה באתר . עם זאת, בעוד שמבחני פרופיל מרחבי אלה עוזרים לחשוף ביטוי RNA על פני רקמה לאחר מיקרו-דיסקציה או חתך, שיטות אלו יכולות להיות מאתגרות כאשר הן מבוצעות על רקמת הגיד. גידים הם רקמות עשירות במטריקס המורכבות מכמעט 86% קולגן במסה יבשה10, מה שמקשה על חילוץ התאים לריצוף. בשל הסיבוכים בבידוד תאים מהמטריצה, האופי ההיפו-תאי של גיד11 וספירת ה-RNA הנמוכה יחסית, הגיד הוא רקמה קשה לניתוח.
במאמר זה, אנו מציגים שיטה לאופטימיזציה של מבחני הכלאה חדשניים באתרם כדי למנף אותם לגידים על ידי מתן שיטות הכנת רקמות, חדירות ותכנון בדיקה. בשילוב עם טכנולוגיות ריצוף קיימות, זה עשוי לעזור לחוקרים לאפיין מרחבית תת-אוכלוסיות של גידים על פני גידים מתפתחים, בוגרים או פצועים עם רגישות וספציפיות מוגברת לבדיקה.
Access restricted. Please log in or start a trial to view this content.
Protocol
כל הניסויים בבעלי חיים בוצעו בהתאם להנחיות הוועדה המוסדית לטיפול ושימוש בבעלי חיים (IACUC) ו-AAALAC. הניסויים בוצעו תחת פרוטוקול #2013N000062 מאושר בבית החולים הכללי של מסצ'וסטס. במחקר זה נעשה שימוש בעכברי C57BL/J6 (בני 5 שבועות ו-P0). עיין בטבלת החומרים לפרטים הקשורים לכל החומרים, הריאגנטים והמכשירים המשמשים בפרוטוקול זה.
1. הכנה וקיבוע לדוגמא
- המתת חסד של עכברים בתא CO2 ואחריו פריקת צוואר הרחם כשיטה משנית להמתת חסד. לאחר מכן, השתמש במספריים כדי לחתוך את הגפיים האחוריות של העכבר לאורך מפרק הירך12. בבקבוקון נצנוץ, טבלו את הגפה האחורית של העכבר בתמיסת פרפורמלדהיד (PFA) מספקת כדי לכסות לחלוטין את הדגימה, וקבעו את הרקמה למשך 24 שעות.
הערה: במקום 4% PFA, ניתן להשתמש גם ב-10% פורמלין לקיבוע. לאחר הקיבוע, ניתן לאחסן את הדגימה במי מלח עם חוצץ פוספט (PBS) עד לעיבוד נוסף. על ידי קיבוע כל הגפה האחורית, אנו מסוגלים לשמר את המתח על הגיד ויש לנו הזדמנות לבחון את האנתזה, את גוף האמצע של הגיד או את הצומת המיוטנדיני. - לאחר הוצאת ה-PFA מבקבוקון הנצנוץ, שטפו את הדגימה למשך 3 x 10 דקות עם 1x PBS על ידי הוספת מספיק PBS כדי לכסות את הדגימה. לאחר מכן, טבלו את הגפיים האחוריות ב-0.5 מ' EDTA ב-4 מעלות צלזיוס עם תסיסה למשך 1-2 שבועות, והחליפו ב-EDTA טרי כל 2-3 ימים.
- לאחר הסרת ה-EDTA, שטפו את הדגימה למשך 3 x 10 דקות עם 1x PBS, ייבשו את הדגימות ב-70% EtOH והטמיעו אותן בפרפין בכיוון הרצוי. השתמש בטכניקות חיתוך FFPE קונבנציונליות כדי לחתוך את הרקמה בעובי של 3 מיקרומטר ולהעביר את החלקים לשקופית מיקרוסקופ מטופלת וטעונה (ראה טבלת חומרים)13.
הערה: ניתן לאחסן חלקים ב-4 מעלות צלזיוס לפני שתמשיך לשלבים הבאים. - דה-פאראפיניזציה
- אופים את השקופיות על פלטה חמה בחום של 60 מעלות צלזיוס למשך שעה ואז מניחים לשקופיות להגיע לטמפרטורת החדר (RT). כדי להסיר את שעוות הפרפין מהשקופיות, הניחו את השקופיות על מתלה שקופיות והורידו אותן לכלי מכתים מלא בקסילן. חזור על הפעולה בקסילן טרי למשך 3 x 5 דקות.
זהירות: קסילן מסוכן; טפל בזהירות. - כדי להחזיר לחות לחלקים, טבלו את השקופיות בדברים הבאים למשך 5 דקות לכל שטיפה, הכל ב-RT: שטפו 2x ב-100% EtOH, 1x ב-75% EtOH מדולל ב-dH2O, 1x ב-50% EtOH מדולל ב-PBS, 1x ב-25% EtOH מדולל ב-PBS ו-2x ב-PBS.
הערה: כל שלבי ההתייבשות נעשים עם השקופיות על מתלה שקופיות ועם פתרונות EtOH השונים בכתמת כלים.
- אופים את השקופיות על פלטה חמה בחום של 60 מעלות צלזיוס למשך שעה ואז מניחים לשקופיות להגיע לטמפרטורת החדר (RT). כדי להסיר את שעוות הפרפין מהשקופיות, הניחו את השקופיות על מתלה שקופיות והורידו אותן לכלי מכתים מלא בקסילן. חזור על הפעולה בקסילן טרי למשך 3 x 5 דקות.
2. התאמת פרוטוקול RNAscope (ISH ממוסחר)14
- הכנת פתרונות ובדיקות
- הכן תמיסת TEG לטיפול מקדים בדגימות חתוכות על ידי שילוב של 25 מ"מ של Tris-HCl ב-pH 8, 10 מ"מ של EDTA ו-50 מ"מ של גלוקוז. הכינו נפח מספיק כדי לטפל בכל הדגימות או להכין עודפים; לעקר מסנן ולאחסן ב-RT; מערבבים את התמיסה כראוי לפני השימוש בה.
- הכן מספיק מאגר כביסה 1x (ראה טבלת החומרים) עבור כל השטיפות ואחסן אותו ב-RT.
- באמבט מים או חממה של 40 מעלות צלזיוס, יש לחמם את הבדיקות המעניינות למשך 10 דקות. לאחר מכן, הכינו את הבדיקות לפי פרוטוקול ISH14.
- טיפול מקדים בדגימות
- במקום להשתמש בתמיסת הטיפול המומלצת, טבלו את השקופיות בכלי מכתים המכיל מאגר TEG למשך 4 שעות בטמפרטורה של 60 מעלות צלזיוס.
הערה: ניתן להאריך את הדגירה של מאגר TEG עד 6 שעות אם הרקמה צפופה במיוחד או אם אחזור אנטיגן של מוטיב קשירת ה-RNA המעניין מאתגר. במקרה כזה ניתן להוסיף כאן נקודת עצירה, את המגלשות לייבש ולשמור במקרר 4 מעלות למשך הלילה. - מוציאים את הדגימות מהמאגר ונותנים להן להתייבש. לאחר מכן, צייר סביב הדגימות בעזרת עט מחסום הידרופובי, טבל אותן בכלי מכתים אחר המכיל פרוטאז IV, ודגר אותן בתנור למשך 45 דקות בחום של 40 מעלות צלזיוס.
הערה: אנו ממליצים על פרוטאז IV לעכברים בוגרים. אם משתמשים בעכברים צעירים יותר, כגון P0, אנו ממליצים להשתמש באנזים עדין יותר, כגון פרוטאז III, למשך 30 דקות. בעת פתרון בעיות לגילאים שונים, יש להקפיד על תקינות הגיד ולבדוק אם יש השפלה. אם הרקמה מתנתקת או מתפרקת, צמצם את זמן הדגירה או שנה את הפרוטאז בו משתמשים. - שטפו דגימות שוב במאגר TEG למשך 30 דקות ולאחר מכן הכלאו את הבדיקות בהתאם לפרוטוקול ISH14.
- הוסף 50-100 מיקרוליטר של מגיב הרכבה על גבי הדגימה. הניחו כיסוי על גבי הדגימות למיקרוסקופיה. בעת הדמיה, השתמש בהגדלה גבוהה (אובייקט פי 40 ומעלה) כדי view האות.
- במקום להשתמש בתמיסת הטיפול המומלצת, טבלו את השקופיות בכלי מכתים המכיל מאגר TEG למשך 4 שעות בטמפרטורה של 60 מעלות צלזיוס.
3. התאמת פרוטוקול HCR ISH15
- פוסט קיבוע
- הכן 4% PFA (ב-PBS), תמיסת PBT (0.1% Tween-20 ב-PBS) ו-Proteinase K ב-PBS (ריכוז מלאי של 10 מ"ג/מ"ל, ריכוז סופי של 5 מיקרוגרם/מ"ל).
- כדי להתחיל את שלב התיקון הראשון, טבלו את השקופיות בכלי מכתים המכיל 4% PFA למשך 5 דקות; לאחר מכן, הסר את תמיסת ה-4% PFA ואחסן אותה לשימוש בשלבים מאוחרים יותר. שטפו את השקופיות למשך 2 x 5 דקות בתמיסת PBT והסירו את תמיסת ה-PBT. מניחים את השקופיות בכלי מכתים המכיל תמיסת Proteinase K (5 מיקרוגרם/מ"ל) למשך 5 דקות ושוטפים אותן בתמיסת PBT 2 x 5 דקות.
- התחל את שלב ה-postfix השני וטבול את השקופיות ב-4% PFA למשך 5 דקות.
הערה: זהו אותו 4% PFA שנשמר משלב 3.1.2. - שטפו את השקופיות למשך 3 x 5 דקות בתמיסת PBT.
הערה: כל שלבי הפוסט-קיבוע נעשים עם השקופיות על מתלה שקופיות ובכלים מכתימים המכילים את הפתרונות המתאימים.
- אצטילציה
- הכן את תמיסת האצטילציה המכילה 625 מיקרוליטר של אנהידריד אצטי, 3.3 מ"ל של מאגר טריאתנולמין (TEA) של 1 M ו-246 מ"ל של ddH2O. הקפידו לערבב היטב ולהשתמש מיד לאחר ההכנה.
- הנח את השקופיות בתמיסת האצטילציה למשך 10 דקות ולאחר מכן שטוף 3 x 5 דקות בתמיסת PBT. שטפו את השקופיות ב-ddH2O ותנו להן להתייבש באוויר למשך 30 דקות. לאחר מכן, צייר סביב הדגימות בעזרת עט מחסום הידרופובי.
הערה: כל שלבי האצטילציה נעשים עם השקופיות על מתלה שקופיות ובכלים מכתימים המכילים את התמיסות המתאימות
- הכלאה
- מחממים מראש את מאגר ההכלאה ל-37 מעלות צלזיוס ומחממים תא לח ל-37 מעלות צלזיוס.
זהירות: המאגר מכיל פורממיד, חומר מסוכן. אם השקופיות אינן יבשות מספיק, כתם את הקצוות במגבון מעבדה כדי להסיר עודפי dH2O. - הכן פתרונות בדיקה על ידי הוספת 0.4 pmol מכל תערובת בדיקה ל-100 מיקרוליטר של מאגר הכלאה.
הערה: אם אתה משתמש בהדמיית dHCR, השתמש בריכוז גבוה יותר של בדיקה כדי לשפר את יעילות הכלאת הבדיקה. - הכן 4 ליטר של מאגר נתרן ציטראט נתרן כלורי (20x SSC) על ידי שילוב של 3 M NaCl (701.1 גרם NaCl בנפח סופי של 4 ליטר) ו-0.3 מ' של Na3CH6H5O7•2H2O (352.8 גרם) ולאחר מכן התאמת ה-pH הסופי ל-7 על ידי הוספת HCl או 10 N NaOH. הוסף מספיק H2O כדי להגיע ל-4 L. הכן 5x SSCT על ידי דילול 20x SSC ל-5x והוספת מספיק 10% Tween 20 כך שהוא יהיה 0.1% מהנפח הכולל.
- הוסף 200 מיקרוליטר של מאגר הכלאה לדגימה, הנח את השקופיות בתא לח ותן לשקופיות לדגור למשך 10 דקות. הסר את תמיסת ההכלאה ונקז את המאגר העודף על המגלשה על ידי ספיגת הקצוות במגבון מעבדה.
- הוסף 100 מיקרוליטר מתמיסת הבדיקה המוכנה על גבי הדגימה והנח כיסוי על הדגימה. יש לדגור בתנור למשך הלילה או למשך כ-12-16 שעות בתא עם לחות בתמיסה המכילה 5x SSC ו-50% פורממיד. הגדר את הטמפרטורה ל-37 מעלות צלזיוס.
הערה: מכיוון שהדגירה היא לילה, הוספת הכיסוי היא כדי למזער את האידוי. - השאירו מינון של תמיסת SSCT פי 5 באמבט מים של 37 מעלות צלזיוס ותוספת שנייה ב-RT. צפו את נפחי הכמות הנדרשות על סמך מספר המדגם ומספר השטיפות בשלב הבא.
- מחממים מראש את מאגר ההכלאה ל-37 מעלות צלזיוס ומחממים תא לח ל-37 מעלות צלזיוס.
- כביסה
- באמצעות תמיסת 5x SSCT שחוממה מראש בעבר, הכינו 75% מאגר כביסה/25% 5x SSCT, 50% מאגר כביסה/50% 5x SSCT, ו-25% מאגר כביסה/75% 5x SSCT.
- כדי להסיר בדיקות עודפות, טבלו את השקופיות בסדרה למשך 15 דקות לכביסה ב-37 מעלות צלזיוס ב-75% מאגר כביסה/25% 5x SSCT, 50% מאגר כביסה/50% 5x SSCT, ו-25% מאגר כביסה/75% 5x SSCT. אפשר לכיסויים לצוף מהדגימות.
- דגרו את השקופיות 2 x 15 דקות עם 100% 5x SSCT ולאחר מכן טבלו אותן ב-5x SSCT למשך 5 דקות ב-RT.
הערה: ניתן לבצע את שלבי הכביסה באמצעות השקופיות על מתלה שקופיות ובכלים מכתימים המכילים את התמיסות המתאימות או עם התמיסות שנוספו כטיפות.
- הגברה
- הכן 6 pmol של סיכת ראש h1 ו-6 pmol של סיכת ראש h2 על ידי קירור מהיר של 2 מיקרוליטר של ציר של 3 מיקרומטר (מחממים ב-95 מעלות צלזיוס למשך 90 שניות ומצננים ל-RT). הגן על סיכות השיער מפני אור במהלך תהליך זה.
הערה: סיכות שיער HCR h1 ו-h2 מסופקות במאגר אחסון סיכת ראש ומוכנות לקירור בהצמדה. הצמד סיכות שיער מגניבות h1 ו-h2 בצינורות נפרדים. - יבש את השקופיות על ידי ספיגת הקצוות שלהן במגבון מעבדה. לאחר מכן, הוסף 200 מיקרוליטר של מאגר הגברה על גבי הדגימה והנח אותו בתא לח למשך 30 דקות ב-RT. הכן את תערובת סיכת הראש על ידי הוספת סיכות שיער h1 מקוררות וסיכות שיער h2 מקוררות ל-100 מיקרוליטר של מאגר הגברה ב-RT.
- הסר את מאגר ההגברה ונקז את עודפי הנוזל על השקופית על ידי ספיגת הקצוות במגבון מעבדה. מוסיפים 100 מיקרוליטר מתערובת סיכות הראש שהוכנה קודם לכן ומניחים פרפילם על גבי הדגימה. דגרו את המגלשות למשך 4 שעות לפחות או לילה בתא חשוך ולח ב-RT.
- כדי להסיר סיכות שיער עודפות, טבלו את השקופיות ב-5x SSCT בכלי מכתים ודגרו ב-RT למשך 30 דקות. חזור על הכביסה ב-SSCT טרי פי 5 למשך 30 דקות, ובפעם השלישית למשך 5 דקות.
- יבש את השקופיות על ידי ספיגת הקצוות במגבון מעבדה והוסף 50-100 מיקרוליטר של מגיב הרכבה על גבי הדגימה. הניחו כיסוי על גבי הדגימות למיקרוסקופיה. בעת הדמיה, השתמש בהגדלה גבוהה (אובייקט פי 40 ומעלה) כדי view האות.
- הכן 6 pmol של סיכת ראש h1 ו-6 pmol של סיכת ראש h2 על ידי קירור מהיר של 2 מיקרוליטר של ציר של 3 מיקרומטר (מחממים ב-95 מעלות צלזיוס למשך 90 שניות ומצננים ל-RT). הגן על סיכות השיער מפני אור במהלך תהליך זה.
Access restricted. Please log in or start a trial to view this content.
תוצאות
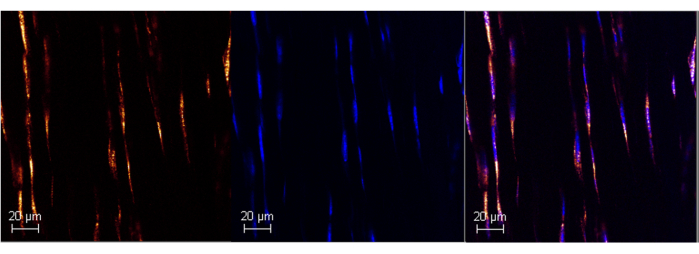
איור 1: ביטוי רנ"א פולי A בגיד אכילס של עכבר בוגר באמצעות RNAScope. תמונה מייצגת של תיוג Poly A מוצלח בגיד אכילס של עכבר (פאנל שמאלי) באמצעות בדיקת ISH הממוסחרת. לוקליזציה עם DAPI מאשרת את ?...
Access restricted. Please log in or start a trial to view this content.
Discussion
במאמר זה, אנו מתארים שינויים שנעשו כדי למנף את כלי ה-ISH הקיימים כך שניתן להשתמש בהם ברקמת גיד עם רמה גבוהה של ספציפיות ורגישות. מכיוון שהגיד הוא רקמה צפופה מאוד, לעתים קרובות יש לבצע התאמות פרוטוקול כדי להשיג דרגות דומות של חדירת בדיקה וספציפיות. שיטות חדירה ספציפיות אלה ו...
Access restricted. Please log in or start a trial to view this content.
Disclosures
למחברים אין ניגודי אינטרסים לחשוף.
Acknowledgements
המחברים מודים לג'נה גאלוויי ולחברי מעבדת גאלוויי על תמיכתם ועידודם בפיתוח ובפתרון בעיות של פרוטוקולים אלה.
Access restricted. Please log in or start a trial to view this content.
Materials
| Name | Company | Catalog Number | Comments |
| 1 M triethanolamine buffer | |||
| 10% Formalin solution | |||
| 10% Tween-20 | |||
| 20x Saline Sodium Citrate buffer | |||
| 4% PFA | |||
| ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 | ACD | 323100 | |
| Acetic Anhydride | |||
| Axio Imager Microscope | ZEISS | ||
| C57BL/J6 mice | JAX ID: 000664 | ||
| Coverslips | Fisher | 12-541-042 | |
| ddH2O | |||
| ETDA | Thermofisher | AM9262 | |
| EtOH | |||
| Glucose | VWR Chemicals BDH | BDH9230-500G | |
| HCR RNA-FISH Bundle | Molecular Instruments Inc. | ||
| HybEZ II Hybridization System | ACD | ||
| Immedge Barrier Pen | Vector Laboratories | H4000 | |
| Leica SPE Confocal Microscope | Leica | ||
| Parafilm | Fisher | ||
| Phosphate-buffered saline (PBS, 1x) | Invitrogen | AM9625 | Dilute 10x PBS in milli-Q water to get 1x solution |
| Protease IV | |||
| Proteinase K | Roche | 3115836001 | |
| RNAscope H2O2 and Protease Reagents | ACD | PN 322381 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V3 |
| RNAscope Multiplex Fluorescent Detection Kit | ACD | PN 323110 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V2 |
| RNAscope Target Retrieval reagents | ACD | 322000 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V4 |
| RNAscope Wash Buffer | ACD | PN 310091 | Included in ACD RNAscope Fluorescent Multiplex Fluorescent Reagent Kit V5 |
| RNAscope Probe Diluent | ACD | 300041 | |
| Slide holder | StatLab | 4465A | |
| Staining Dish with Lid | StatLab | LWS20WH | |
| Superfrost Plus Microscope slides | Fisher | 1255015 | treated, charged slides |
| Tris-HCl | |||
| Xylene | Sigma-Aldrich | 534056-4L |
References
- Sharma, P., Maffulli, N. Tendon injury and tendinopathy: healing and repair. Journal of Bone and Joint Surgery. 87 (1), 187-202 (2005).
- Brent, A. E., Schweitzer, R., Tabin, C. J. A somitic compartment of tendon progenitors. Cell. 113 (2), 235-248 (2003).
- Noden, D. M. The role of the neural crest in patterning of avian cranial skeletal, connective, and muscle tissues. Developmental Biology. 96 (1), 144-165 (1983).
- Chen, J. W., Galloway, J. L. The development of zebrafish tendon and ligament progenitors. Development. 141 (10), 2035-2045 (2014).
- Schweitzer, R., et al. Analysis of the tendon cell fate using Scleraxis, a specific marker for tendons and ligaments. Development. 128 (19), 3855-3866 (2001).
- Tsai, S., Nödl, M., Galloway, J. Bringing tendon biology to heel: Leveraging mechanisms of tendon development, healing, and regeneration to advance therapeutic strategies. Developmental Dynamics. 250 (3), 393-413 (2021).
- Kannus, P., et al. Location and distribution of non-collagenous matrix proteins in musculoskeletal tissues of rat. The Histochemical Journal. 30 (11), 799-810 (1998).
- Kannus, P. Structure of the tendon connective tissue. Scandinavian Journal of Medicine & Science in Sports. 10 (6), 312-320 (2000).
- Thorpe, C. T., Birch, H. L., Clegg, P. D., Screen, H. R. C. The role of the non-collagenous matrix in tendon function. International Journal of Experimental Pathology. 94 (4), 248-259 (2013).
- Lin, T. W., Cardenas, L., Soslowsky, L. J. Biomechanics of tendon injury and repair. Journal of Biomechanics. 37 (6), 865-877 (2004).
- Grinstein, M., et al. A distinct transition from cell growth to physiological homeostasis in the tendon. eLife. 8, e48689(2019).
- Villaseñor, S., Grinstein, M. Two-photon microscopy for the study of tendons. Journal of Visualized Experiments. , e65853(2023).
- Qin, C., et al. The cutting and floating method for paraffin-embedded tissue for sectioning. Journal of Visualized Experiments. (139), e58288(2018).
- Wang, F., et al. RNAscope®: A Novel In Situ RNA Analysis Platform for Formalin-Fixed Paraffin-Embedded Tissues. Journal of Molecular Diagnostics. 14 (1), 22-29 (2012).
- Choi, H. M. T., et al. Third-generation situ hybridization chain reaction: multiplexed, quantitative, sensitive, versatile, robust. Development. 145 (12), 165753(2018).
- Jackson, D. J., Herlitze, I., Hohagen, J. A whole mount in situ hybridization method for the gastropod mollusc Lymnaea stagnalis. Journal of Visualized Experiments. (109), e53968(2016).
- Young, A. P., Jackson, D. J., Wyeth, R. C. A technical review and guide to RNA fluorescence in situ hybridization. PeerJ. 8, e8806(2020).
Access restricted. Please log in or start a trial to view this content.
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved