A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
בדיקת כימיה של צטיל קליק למדידת אצטילטרנספראז היסטון 1 אצטילציה
In This Article
Summary
בדיקות כימיות מהירות ומדויקות לסינון מעכבים ספציפיים הן כלי חשוב בארסנל פיתוח התרופות. כאן, אנו מציגים בדיקה כימית מדרגית של קליק אצטיל-קליק כדי למדוד את העיכוב של פעילות אצטילציה HAT1.
Abstract
HAT1, הידוע גם בשם היסטון אצטילטרנספראז 1, ממלא תפקיד מכריע בסינתזה של כרומטין על ידי ייצוב ואצטילציה של H4 מתהווה לפני הרכבת נוקלאוזומים. הוא נדרש לצמיחת הגידול במערכות שונות, מה שהופך אותו למטרה פוטנציאלית לטיפול בסרטן. כדי להקל על זיהוי תרכובות שיכולות לעכב פעילות אנזימטית של HAT1, פיתחנו בדיקת אצטיל-קליק לסינון מהיר. בבדיקה פשוטה זו, אנו משתמשים ב- HAT1/Rbap46 רקומביננטי, אשר מטוהר מתאים אנושיים פעילים. השיטה משתמשת באנלוגי אצטיל-CoA 4-pentynoyl-CoA (4P) בגישה כימיית קליק. זה כרוך בהעברה אנזימטית של ידית אלקין דרך תגובת אצילציה תלוית HAT1 לפפטיד H4 N-terminal ביוטינילציה. לאחר מכן הפפטיד שנלכד משותק על לוחות נויטרווידין, ולאחר מכן מתפקד כימיית קליק עם ביוטין-אזיד. לאחר מכן, גיוס סטרפטווידין-פרוקסידאז משמש לחמצון אדום אמפלקס, וכתוצאה מכך פלט פלואורסצנטי כמותי. על ידי החדרת מעכבים כימיים במהלך תגובת האצילציה, אנו יכולים לכמת עיכוב אנזימטי בהתבסס על הפחתה של האות הפלואורסצנטי. חשוב לציין, תגובה זו ניתנת להרחבה, ומאפשרת סינון בתפוקה גבוהה של מעכבים פוטנציאליים לפעילות אנזימטית HAT1.
Introduction
בין האצטילטרנספראזות האיקריוטיות הרבות, HAT1 היה ההיסטון אצטילטרנספראז הראשוני שבודד 1,2,3. מחקרים מאוחרים יותר ביססו היטב את תפקידו המרכזי בשכפול הכרומטין, במיוחד בסינתזה של נוקלאוזומים חדשים במהלך שלבS 4. מאמצי המחקר שלנו הובילו להכרה כי HAT1 מגורה מאוד על ידי טיפול בגורם גדילה אפידרמלי (EGF) בתאי חלב5. יתר על כן, התברר כי HAT1 נדרש להתרבות תאים מהירה והיווצרות גידולים in vivo 6,7,8,9. הנתונים מצביעים על כך ש- HAT1 הוא קריטי בתיאום תהליכים אנאבוליים ואפיגנטיים לחלוקת תאים, ומניע את צמיחת הגידול.
HAT1 di-acetylates זנב אמינו-טרמינלי של היסטון H4 על ליזין 5 ו 12 בקומפלקס עם חלבון מלווה Rbap46, אשר קושר את ההיסטון ומציג את האמינו-טרמינוס ל- HAT1. טטרמרים היסטון או דיזומים10, יחד עם HAT1/Rbap46 ומלווי היסטוןאחרים 11, מיובאים לאחר מכן לגרעין. לאחר מכן היסטונים משוחררים כדי להיות מופקדים במזלג השכפול, או באתרים אחרים כדי לתמוך בהפעלה או דיכוי של גנים. הפונקציה של סימן הדי-אצטילציה HAT1 על היסטון H4 אינה מובנת במלואה. סביר להניח שהוא מוסר במהירות בטווח של 15-30 דקות על ידי פעולה של deacetylases היסטון 12,13,14,15 לאחר H4 מוכנס לכרומטין. לפיכך, סימן הדי-אצטילציה HAT1 אינו מופץ בכרומטין ועשוי שלא לשמש תפקיד אפיגנטי אמיתי, אם כי תפקיד בגיוס אנזימים משנים כרומטין-לכרומטין המתהווה הועלה12. כמו כן, HAT1 אינו אצטילט כרומטין; פעילותו מוגבלת להיסטונים מסיסים.
הפיתוח של מעכבי אצטילטרנספראז היסטון במולקולות קטנות התעכב על ידי בדיקות לא ספציפיות ובעלות תפוקה נמוכה, ולעתים קרובות הביא ליצירת תרכובות תגובתיות ביולוגיות16,17. בדיקת תקן הזהב למדידת פעילויות אצטילטרנספראז דורשת שימוש ב- 3H-אצטיל-coA, המגביל את התפוקה ודורש קרינה. עם זאת, לאחרונה, מעכבי אצטילטרנספראז ספציפיים וחזקים מאוד של מולקולות קטנות המכוונות ל- CBP/p30018 ו- KAT6A/B19,20 תוארו ואושרו באמצעות שימוש ב- 3H-אצטיל-CoA. בהמשך, בדיקות משופרות כדי להשיג תפוקה טובה יותר ולהימנע מסכנות מעבדה מתוכננות.
ההתקדמות האחרונה בניטור אצטילציה21 השתמשה בכימיית קליקים כדי לאפשר ניטור תגובה אנזימטית. ישנם מגוון קודמנים תומכי קליק הנגישים במסלולים סינתטיים פשוטים או זמינים לרכישה שניתן לשלב בתגובות אנזימים. תגובות אלה מתבצעות בדרך כלל במערכות רקומביננטיות, אם כי בדיקות מבוססות תאים אפשריות גם22. היתרון של גורמים משלימים ומצעים התומכים בלחיצה הוא שהסינון יכול למדוד ישירות את פעילות האנזים ללא צורך במערכות קריאה מצומדות שלעתים קרובות מוטרדות על ידי תרכובות סינון ודורשות שלבי טיפול נוספים. זה מאפשר טיפולים מעכבים רק בשלב האנזימטי, בעוד שכל שלבי התפקוד והזיהוי במורד הזרם מתבצעים לאחר שטיפה נרחבת כדי להסיר תרכובות, ובכך מגבילים את הפוטנציאל להתרחשות הפרעות בבדיקה. יתרונות אלה הופכים את העיצוב של בדיקות מותאמות קליק לעדיף על פני בדיקות מצומדות המסתמכות בדרך כלל על זיהוי של קואנזים חופשי A.
שיקול חשוב אחד הוא הקבלה של גורמים משלימים התומכים בקליקים לאתר הפעיל של האנזים. ייתכן שגורמים משלימים קיימים המותאמים לשימוש בקליקים לא יהיו תואמים באופן מלא לאתר הפעיל הממוטב עבור הגורם המשותף המקורי. מידע מבני ומידול יכולים לשמש לתכנון תחליפי חומצות אמינו כדי להגדיל את האתר הפעיל כדי לשלב מצעים שהשתנו23. זה עשוי לאפשר סינון עם קינטיקה אנזימית משופרת ורמות נמוכות יותר של סובסטרט ואנזימים. החיסרון של גישה זו הוא שכיסים קטליטיים שהשתנו עשויים שלא לזהות מעכבים המקיימים אינטראקציה חזקה עם האנזים המקומי. בסופו של דבר, נדרש שילוב של גישות כדי לזהות ולאמת מעכבי אנזימים פוטנציאליים.
במאמר זה אנו מתארים שיטה שפותחה לטיהור והערכה של פעילות האנזים HAT1 באמצעות קו-פקטור קליק 4-פנטינואיל-CoA24. בדיקה זו (איור 1) משתמשת ברצף האנזים המקורי בקומפלקס עם החלבון השותף הנדרש Rbap46, אשר הוכח כמגביר את פעילות האנזים. טיהור האנזים מתאי האדם מאפשר הפעלת אנזים בצלולו, מה שעשוי לשמר שינויים מעוררים לאחר התרגום החשובים לפעילות אנזימית מלאה. תכנון ואופטימיזציה של בדיקות אנזימים רקומביננטיים עבור מסכים כימיים בעלי תפוקה גבוהה שימשו בהצלחה לזיהוי ואפיון מעכבי מולקולות קטנות HAT1.
Protocol
1. שיטה 1: ייצור וטיהור קומפלקס רקומביננטי HAT1/Rbap46
- הפשרה, שחזור והרחבה של תאי HEK293f
- הפשירו 1-10 מיליון תאי יונקים HEK293f26 לתוך 30 מ"ל בסגנון חופשי 293 מדיה בבקבוק 100 מ"ל. יש לדגור ב-8% CO2 ב-37°C תוך כדי סיבוב ב-60 סל"ד.
- למחרת, ספרו ובדקו את כדאיות התא, ולאחר מכן התאימו את מהירות הסיבוב ל-120 סל"ד. יש להרחיב לתרבית של 300 מ"ל בצלוחית של 1 ליטר, תוך שמירה על צפיפות זריעה של 500,000 תאים/מ"ל ופיצול תאים לפני שהצפיפות עולה על 3 x 106 תאים/מ"ל.
- טרנספקציה HEK293f
- זרע 5 x 105 תאים / מ"ל בתרבית 300 מ"ל בבקבוק 1 ליטר ותרבית במשך 24 שעות. למחרת, ספרו תאים כדי לוודא שהם בין 7.5 x 105 עד 1.2 x 106 תאים / מ"ל.
- הכינו תערובת של 300 מיקרוגרם של pHEK-FLAG-HAT1 ו-300 מיקרוגרם של DNA פלסמיד pHEK-Rbap46 ב-30 מ"ל של PBS. הוסף 1.2 מ"ל של פוליאתילנימין (PEI) ל- DNA/PBS וערבב. יש לדגור במשך 20 דקות ב-RT. הוסף את תערובת DNA/PBS/PEI לתרבית של 300 מ"ל ודגור ב-37°C למשך 48 שעות ב-120 סל"ד.
- קצירת תאים
- ספור תאים כדי לוודא שהם נופלים בין 1.7 x 106 עד 2.5 x 106 תאים/מ"ל. בצפיפות תאים זו, הוסף פורסקולין לריכוז סופי של 12.5 מיקרומטר כדי להפעיל HAT1 ולדגור על תאים במשך 30 דקות, 37°C, 120 סל"ד.
- גלולה את התאים ב 300 x גרם במשך 5 דקות, לשטוף פעם אחת עם 30 מ"ל של PBS, ולהקפיא בזק נוזל N2. יש לאחסן בטמפרטורה של -80°C עד לטיהור החלבון, או להמשיך ישירות לטיהור.
- HAT1/Rbap46 קומפלקס טיהור חרוזי דגל
- ליז תאים ולהכין תמצית חלבון.
- הכינו את מאגר הליזה על ידי הוספת טבליה אחת של קוקטייל מעכב פרוטאז (PIC) ל-50 מ"ל של RSB-500 (20 mM Tris-HCl pH 7.5, 500 mM NaCl, 25 mM MgCl2).
- הפשירו את גלולת התא מתרבית של 300 מ"ל על קרח וליז עם 40 מ"ל של חיץ ליזה קר כקרח עם 0.1% טריטון X-100. יש לסחור על קרח ואז לסחור כלפי מטה במהירות של 10,000 x גרם למשך 10 דקות ב-4°C. אספו את כל הסופרנאטנט לתוך צינור אחד של 50 מ"ל על קרח, שהוא כעת תמצית החלבון.
- הכינו חרוזי דגל.
- פצל את חרוזי FLAG לארבעה צינורות חרוטיים של 15 מ"ל על ידי הוספת 400 μL של תרחיף חרוזי אגרוז M2-FLAG לכל צינור המכיל 5 מ"ל של 0.1 M גליצין pH 3.5, 0.01% Triton X-100. נערו כל צינור במרץ במשך 5 שניות ביד.
- פולס צנטריפוגה במשך 30 שניות ב 1000 x גרם ב 4 ° C, לשאוף supernatant, לשטוף פעמיים עם 5 מ"ל של RSB-500 + 0.1% Triton X-100 (Tx-100; ללא מעכב פרוטאז), ולדגור על קרח.
- בצע FLAG immunoprecipitation.
- הוסף 10 מ"ל של תמצית חלבון לכל 15 מ"ל צינור חרוטי המכיל חרוזים שטופים. לדגור על הצינורות ב 4 ° C עם סיבוב הפוך לפחות 90 דקות או לילה.
- לשטוף את החרוזים כרוכים 5x ב 10 מ"ל של RSB-500 + 0.1% Tx-100. עבור כל שטיפה, הפוך את הצינור 2x-5x כדי להשהות חרוזים, ולאחר מכן גלולה ב 1000 x גרם במשך 1 דקה ב 4 ° C. לבסוף, יש לשטוף 1x ב-RSB-100 (20 mM Tris-HCl pH 7.5, 100 mM NaCl, 25 mM MgCl2) + 0.1% Tx-100.
- FLAG peptide elution: יש להקפיד על קומפלקס HAT1 ב-1.5 מ"ל של Elution Buffer (EB) המכיל 0.5 מ"ג/מ"ל פפטיד FLAG. יש לדגור בטמפרטורה של 4°C למשך שעה אחת לפחות או למשך הלילה. צנטריפוגו את החרוזים המדוללים ב 1000 x גרם במשך 30 שניות ב 4 ° C ולאסוף את supernatant, אשר מכיל את קומפלקס HAT1 / Rbap46 מטוהר.
- יש לרכז חלבון ולהסיר פפטיד FLAG.
- הוסף את ההדבקה לצינור מסנן ניתוק דלתון של 20 מ"ל, 10,000. הבא את עוצמת הקול עד 15 מ"ל עם EB. יש לסחור במהירות של 2,500 x גרם במשך 15 דקות ב-4°C, ולחזור על הפעולה פעמיים עם 15 מ"ל נוספים של EB בכל פעם.
- שחזר את הפולט הסופי מצינור המסנן ובדוק את ריכוז החלבון עד A260. צפו לריכוז של 1 מ"ג חלבון סה"כ ב-6 מ"ל EB (לכל תרבית התחלתית של 300 מ"ל). בדוק את טוהר החלבון על ידי SDS-PAGE, Coomassie, ו immunoblotting, ולאחסן aliquots ב -80 ° C.
- ליז תאים ולהכין תמצית חלבון.
2. שיטה 2: עקומה סטנדרטית של לחץ אצטיל-קליק HAT1
- הכנת עקומה סטנדרטית
- סנתז פפטיד H4 N-terminal בקרה חיובית עם הרצף: SCRG[Pra]GGKGLG[Pra]GGAKRHRKVLRGG[Lys(Biotin)], כאשר [Pra] מציין Propargylglycine.
- השהה מחדש את פפטיד Pra ל 0.1 מ"ג / מ"ל ב DMSO. ערבבו את פפטיד Pra עם פפטיד היסטון H4 ביוטינילציה (1-23-GGK-ביוטין) כדי ליצור עקומה סטנדרטית (איור 2; נפחים שנמצאים בטבלה 1). ניתן להכין עקומות סטנדרטיות טריות לפני קשירת הצלחת או, אם הן נעשות מראש, לערבב אותן עם 20 μL של 8 M אוריאה ולאחסן אותן בטמפרטורה של -20°C עד לשלב 3.3.
3. שיטה 3: בדיקת אצטיל-לחץ HAT1
- הרכבת תגובות אצטילציה עם מעכבי בדיקה
- להרכיב תגובות אצטילציה בשכפול בצינורות PCR של 0.2 מ"ל או לוחות PCR של 96 באר מהרכיבים הבאים: פפטיד היסטון H4 ביוטינילציה (1-23-GGK-ביוטין) מושעה מחדש ב-DMSO ל-0.1 מ"ג/מ"ל (34.8 מיקרומטר), אנזים HAT1 מדולל מראש ב-EB, חיץ 20x (1M Tris pH 8.5, 0.1% NP40), 2 mM DTT, 4-pentynoyl-CoA מומס במים ל-1 מ"ג/מ"ל (1 מ"ל). תגובה של 20 μL כוללת 10 μL של אנזים, 1 μL של פפטיד H4, 1 μL של חיץ 20x, 1 μL של DTT מעורבב מראש ו aliquoted בארות של צלחת PCR 96 בארות על קרח.
- הוסף 1 μL של DMSO (בקרה שלילית) או תרכובת בדיקה מומסת ב 1-10 μM, או H4K12CoA25 (או מעכב בקרה חיובית מתאים), לכל באר. מערבבים על ידי פיפטינג עדין ודגרים במשך 10 דקות על קרח כדי לאפשר לאנזים: קומפלקסים מעכבים להיווצר.
- המשך תגובת אצטילציה
- ערבבו 2 μL של 4-pentynoyl-CoA עם 4 μL מים, ולאחר מכן הוסיפו לבארות. ערבבו בעדינות על ידי פיפט, צנטריפוגה ב 300 x גרם במשך 30 שניות ב RT כדי לאסוף את התכולה, ולדגור ב 37 ° C במשך 1 שעה בצינורות מכוסים או צלחות עם נייר כסף לסגירה חוזרת.
- יש לעבד את התכולה ישירות עבור מוצרי תגובה או להרוות עם 20 μL של 8 M urea ולאחסן ב -20 °C עד לעיבוד.
- קשירת פפטיד לצלחת Neutravidin
- הוסף תוכן תגובה עם או בלי אוריאה לאלבומין בסרום בקר (BSA) צלחות שחורות חסומות מראש מצופות נויטרווידין 96 בארות המכילות 80 מיקרוליטר PBST (PBS + 0.1% Tween-20) לכל באר.
- הוסף פפטידים עקומות סטנדרטיים בשכפול לבארות שלהם המכילות 80 μL של PBST. קשרו פפטידים עם טלטול אורביטלי עדין למשך שעה אחת בטמפרטורת החדר (RT).
- שטיפה: לאחר השלמת הקשירה, שאפו את הנוזל מהבארות ושטפו את הבארות עם 200 μL של PBST 3x 15 פעימות (נפח שבץ 180 μL) באמצעות מכונת כביסה.
- תגובת כימיית קליק
- הכינו את הריאגנטים לתגובת הקליק באופן הבא: 100 mM Tris(3-hydroxypropyltriazolymethyl) ליגנד אמין (THPTA) במים ו-20 mM CuSO4 במים. THPTA מונע הידרוליזה מזורזת Cu(II) ומרווה רדיקלים ופרוקסידים הנוצרים מ-O2/Cu/ascorbate.
- הוסף את תערובת THPTA/Cu ל- 300 mM נתרן אסקורבט במים ו- 2.5 mM ביוטין אזיד ב- DMSO. תגובה בלחיצה אחת מכילה 140 μL של PBS, 10 μL של THPTA, 10 μL של CuSO4, 10 μL של נתרן ascorbate, 20 μL של ביוטין-אזיד. יש לערבב תמיד את ריאגנטים הקליק טריים, ולאחר מכן לפזר 190 μL לכל באר, לאטום את הצלחת ולדגור בטמפרטורה של 37°C למשך שעה אחת.
- שטיפה: שאפו את הנוזל מהבארות ושטפו את הצלחת 3x עם PBST (נפח שבץ 180 μL, 15 פעימות לכל שטיפה).
- קשירת סטרפטאבידין-חזרת פרוקסידאז: לדלל סטרפטווידין-HRP (0.224 מ"ג/מ"ל) 1:10 בסטרפטאווידין (0.224 מ"ג/מ"ל), ולאחר מכן לדלל עוד יותר 1:1000 ב-PBST. מוסיפים Steptavidin-HRP: תערובת סטרפטאווידין (100 μL לבאר) ודגרים ב-RT למשך שעה אחת עם טלטול אורביטלי עדין.
- יש לשטוף 3x עם PBST כמו בשלבים הקודמים.
- חמצון אדום של Amplex
- שלב את ריאגנטי הזיהוי האדומים של Amplex באופן הבא: 4.45 מ"ל של חיץ NaHPO4 (1x = 50 mM ריכוז סופי), 50 μL אמפלקס אדום (20 mM מדולל ב- DMSO), 500 μL של H2O 2 מדולל2.
- דללו את H2O2 מ-30% מהמניות ל-3% במאגר 1x NaHPO4 , ואז הוסיפו 22.7 μL של 3% H2O2 לתוך 977 μL של 1x NaHPO4 buffer (זהו H2O2 המשמש לתגובת האמפלקס). הוסף 100 μL של תערובת התגובה האדומה של אמפלקס לכל באר, דגור ב- RT למשך 30 דקות מוגן מפני אור, ולאחר מכן זהה עירור/פליטה פלואורסצנטית 571/585 ננומטר באמצעות קורא לוחות פלואורסצנטי סטנדרטי.
- חישוב עיכוב אנזימים: חישוב אחוזי עיכוב (איור 3) לפי הנוסחה הבאה:
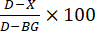 , כאשר D הוא הערך הפלואורסצנטי של תגובות בקרה המטופלות ב-DMSO בלבד, X הוא ערך התגובות המטופלות בתרכובות בדיקה, ו-BG הוא הערך של בארות רקע (בקרת פפטיד H4, ללא תוספת אנזים).
, כאשר D הוא הערך הפלואורסצנטי של תגובות בקרה המטופלות ב-DMSO בלבד, X הוא ערך התגובות המטופלות בתרכובות בדיקה, ו-BG הוא הערך של בארות רקע (בקרת פפטיד H4, ללא תוספת אנזים). - עקומת מינון: בחר תרכובות בדיקה שנמצאו כמעכבות את פעילות האנזים HAT1 לבדיקות חוזרות, כאשר התרכובת מדוללת סדרתית. לקבוע את דילולי המתחם באופן אמפירי. לדוגמה, התחל עם ריכוז מלאי של 2 mM, ולאחר מכן דלל סדרתית 1:3 עבור שמונה דילולים. לאחר מכן יש לדלל ביחס של 1:20 למבחני אנזימים המניבים מינון עליון של 100 מיקרומטר.
- התוויית העקומה: השתמש ברגרסיה של הריבועים הפחותים כדי להתאים לעקומות עיכוב מנה-תגובה (תוך שימוש בערכי עיכוב אחוזים משלב 3.10) בתוכנת ניתוח נתונים (GraphPad Prism) ולגזור ערכי IC50 עבור כל תרכובת (איור 4).
- תגובת אצטילציה עם אצטיל-CoA:
- השתמש באפשרות זו כדי לאשר את פעילות האנזים עם הקו-פקטור המקומי אצטיל-CoA במקום 4-פנטינואיל-CoA. בצע בדיקות אצטילציה HAT1 כמתואר בשלבים 3.1-3.2 עם אצטיל-CoA במקום 4-pentynoyl-CoA.
- זהו את תוצרי התגובה על קרומי ניטרוצלולוז (1-2 μL), אפשרו להם להתייבש, ואז נקודו עם נוגדנים נגד H4-ליזין-12-אצטיל או אנטי-H4-ליזין-5-אצטיל. כמת את אות האימונובלוט על ידי צפיפות.
תוצאות
עקומות סטנדרטיות בכפילות (16 בארות) צריכות להיכלל בכל צלחת כדי להבטיח ביצועי בדיקה נאותים. נתוני עקומה סטנדרטיים צריכים להיות מוגדרים בצורת טבלה, עם טווח של 100% עד 0% בהתאם ליחס בין פפטיד המכיל Pra לפפטיד H4 מקורי בתמיסה (טבלה 1). האות האדום של Amplex יהיה הגבוה ביותר בבארות פפטיד H4 100% / 0% טבע...
Discussion
בעשור האחרון, כימיית הקליק הפכה לבולטת20, ואפשרה תכנון מדויק של מבנים כימיים אינטראקציה. בהקשר זה, קשרים קוולנטיים ביו-אורתוגונליים שונים21 התגלו כאפשרויות מבטיחות ליצירת קומפלקסים בסביבתם הטבעית. כימיית קליקים משתמשת בזוגות של קבוצות פונקציונליות המציגות תגובו...
Disclosures
הגשנו פטנטים המתארים את בדיקת האצטיל-קליק HAT1 (PCT/US20/29395). J.J.G. מדווח על הסכמי ייעוץ עם Sharma Therapeutics, LLC ו-Guidepoint ותמיכה במחקר (למוסד שלו) מ-Hummingbird Biosciences.
Acknowledgements
אנו מודים לג'ורג 'ז'נג על מתן H4K12CoA. אנו מודים לחברי מעבדת גרובר על דיונים מועילים ומשוב. אנו מודים לתמיכה של NIH/NCI (1K08CA245024), CPRIT (RR200090) וקרן V (V2022-022).
Materials
| Name | Company | Catalog Number | Comments |
| 4P CoA | Cayman Chemical | 10547 | Click chemistry co-factor |
| Amplex Red | Fisher Sci | A12222 | Fluorescence substrate |
| Biotin-PEG-Azide | Alfa Aesar | J64996MC | Click chemistry |
| Copper Sulfate | Sigma-aldrich | 7758-98-7 | Click chemistry |
| DMSO | Fisher Scientific | 67-68-5 | diluent |
| DTT | Acros Organics | 03-12-3483 | reducting agent |
| Forskolin | VWR | 102987-310 | Protein expression |
| Freestyle 293 Expression Medium | Thermo Fisher | 12338018 | Media |
| Freestyle 293-F cells | Thermo Fisher | R790-07 | Protein expression |
| H4-peptide/1-23-GGK-biotin | Anaspec | AS65097 | peptide substrate |
| HEPES | Sigma-aldrich | 7365-45-9 | EB buffer |
| Hydrogen peroxide 30% solution | Sigma-aldrich | Z00183-99-0 | initiator |
| M2 FLAG antibody slurry | Millipore-Sigma | A2220 | Protein purification |
| Macrosep 10K Filter (Pall Lab) | VWR | 89131-980 | Protein purification |
| Neutravidin Plate | Thermo Sci | 15127 | BSA-pre-blocked |
| NP40 (IGEPAL) | MP Biomedical | 198596 | 20x buffer |
| pHEK-293 plasmid | Takara Bio | 3390 | Protein expression |
| Phosphate Buffered Saline 10x | Alfa Aesar | Z00082-33-6 | wash buffer |
| Pra peptide | Genscript | Custom synthesis | biotinylated |
| Sodium Ascorbate | Sigma-aldrich | 134-03-2 | Click chemistry |
| Sodium chloride | Sigma-aldrich | 7647-14-5 | EB buffer |
| Sodium phosphate | VWR International | 7558-80-7 | buffer |
| Streptavidin | EMD Millipore | 189730 | competitor |
| Streptavidin-HRP | Cell Signaling | 3999S | enzyme |
| THPTA ligand | Fisher Sci | 1010-500 | Click chemistry |
| Tris base | Sigma-aldrich | 77-86-1 | 20x buffer |
| Triton-X 100 | VWR International | 9002-93-1 | EB buffer |
| Tween-20 | Sigma-aldrich | 9005-64-5 | Wash buffer |
| Urea | Sigma-Aldrich | 57-13-6 | quencher |
References
- Kleff, S., et al. Identification of a gene encoding a yeast histone H4 acetyltransferase. J Biol Chem. 270 (42), 24674-24677 (1995).
- Parthun, M. R., Widom, J., Gottschling, D. E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism. Cell. 87 (1), 85-94 (1996).
- Verreault, A., et al. Nucleosomal DNA regulates the core-histone-binding subunit of the human Hat1 acetyltransferase. Curr Biol. 8 (2), 96-108 (1998).
- Parthun, M. R. Histone acetyltransferase 1: more than just an enzyme. Biochim Biophys Acta. 1819 (3-4), 256-263 (2013).
- Gruber, J. J., et al. HAT1 coordinates histone production and acetylation via H4 promoter binding. Mol Cell. 75 (4), 711-724 e5 (2019).
- Fan, P., et al. Overexpressed histone acetyltransferase 1 regulates cancer immunity by increasing programmed death-ligand 1 expression in pancreatic cancer. J Exp Clin Cancer Res. 38 (1), 47 (2019).
- Xia, P., et al. MicroRNA-377 exerts a potent suppressive role in osteosarcoma through the involvement of the histone acetyltransferase 1-mediated Wnt axis. J Cell Physiol. 234 (12), 22787-22798 (2019).
- Yang, G., et al. Histone acetyltransferase 1 is a succinyltransferase for histones and non-histones and promotes tumorigenesis. EMBO Rep. 22 (2), e50967 (2021).
- Xue, L., et al. RNAi screening identifies HAT1 as a potential drug target in esophageal squamous cell carcinoma. Int J Clin Exp Pathol. 7 (7), 3898-3907 (2014).
- Zhang, W., et al. Structural plasticity of histones H3-H4 facilitates their allosteric exchange between RbAp48 and ASF1. Nat Struct Mol Biol. 20 (1), 29-35 (2013).
- Campos, E. I., et al. The program for processing newly synthesized histones H3.1 and H4. Nat Struct Mol Biol. 17 (11), 1343-1351 (2010).
- Agudelo Garcia, P. A., et al. Identification of multiple roles for histone acetyltransferase 1 in replication-coupled chromatin assembly. Nucleic Acids Res. 45 (16), 9319-9335 (2017).
- Nagarajan, P., et al. Histone acetyl transferase 1 is essential for mammalian development, genome stability, and the processing of newly synthesized histones H3 and H4. PLoS Genet. 9 (6), e1003518 (2013).
- Annunziato, A. T. Assembling chromatin: the long and winding road. Biochim Biophys Acta. 1819 (3-4), 196-210 (2013).
- Annunziato, A. T., Seale, R. L. Histone deacetylation is required for the maturation of newly replicated chromatin. J Biol Chem. 258 (20), 12675-12684 (1983).
- Dahlin, J. L., et al. Assay interference and off-target liabilities of reported histone acetyltransferase inhibitors. Nat Commun. 8 (1), 1527 (2017).
- Baell, J. B., Miao, W. Histone acetyltransferase inhibitors: where art thou. Future Med Chem. 8 (13), 1525-1528 (2016).
- Lasko, L. M., et al. Discovery of a selective catalytic p300/CBP inhibitor that targets lineage-specific tumours. Nature. 550 (7674), 128-132 (2017).
- Baell, J. B., et al. Inhibitors of histone acetyltransferases KAT6A/B induce senescence and arrest tumour growth. Nature. 560 (7717), 253-257 (2018).
- Falk, H., et al. An efficient high-throughput screening method for MYST family acetyltransferases, a new class of epigenetic drug targets. J Biomol Screen. 16 (10), 1196-1205 (2011).
- He, M., et al. Chemical biology approaches for investigating the functions of lysine acetyltransferases. Angew Chem Int Ed Engl. 57 (5), 1162-1184 (2018).
- Lipchik, A. M., et al. A peptide-based biosensor assay to detect intracellular Syk kinase activation and inhibition. Biochemistry. 51 (38), 7515-7524 (2012).
- Song, J., et al. Chemoproteomic profiling of protein substrates of a major lysine acetyltransferase in the native cellular context. ACS Chem Biol. 17 (5), 1092-1102 (2022).
- Gaddameedi, J. D., et al. Acetyl-click screening platform identifies small-molecule inhibitors of histone acetyltransferase 1 (HAT1). J Med Chem. 66 (8), 5774-5801 (2023).
- Ngo, L., Brown, T., Zheng, Y. G. Bisubstrate inhibitors to target histone acetyltransferase 1 (HAT1). Chem Biol Drug Des. 93 (5), 865-873 (2019).
- Parker, C. G., Pratt, M. R. Click chemistry in proteomic investigations. Cell. 180 (4), 605-632 (2020).
- Islam, K. The bump-and-hole tactic: Expanding the scope of chemical genetics. Cell Chem Biol. 25 (10), 1171-1184 (2018).
- Radziwon, K., Weeks, A. M. Protein engineering for selective proteomics. Curr Opin Chem Biol. 60, 10-19 (2021).
- Rich, R. L., Myszka, D. G. Survey of the year 2007 commercial optical biosensor literature. J Mol Recognit. 21 (6), 355-400 (2008).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved