Method Article
PyDesigner v1.0: מימוש פיתוני של צינור DESIGNER עבור דימות תהודה מגנטית דיפוזיה
* These authors contributed equally
In This Article
Summary
כאן אנו מציגים את PyDesigner, צינור עיבוד הדמיית תהודה מגנטית דיפוזיה (dMRI) מבוסס Python המסוגל לתקן חפצי dMRI טיפוסיים ולייצר הדמיית טנזור דיפוזיה (DTI), הדמיית קורטוזיס דיפוזיה (DKI), הדמיית כדור סיבים (FBI), מידול מיקרו-מבנה (שלמות החומר הלבן [WMTI] וחומר לבן כדור סיבים [FBWM]), ויציאות טרקטוגרפיה.
Abstract
PyDesigner היא חבילת תוכנה מבוססת-Python המבוססת על פרמטר Diffusion המקורי EStImation עם Gibbs and NoisE Removal (DESIGNER) pipeline (Dv1) עבור עיבוד מקדים של dMRI והערכת טנזור. תוכנה זו מסופקת בגלוי למחקר לא מסחרי ואין להשתמש בה לטיפול קליני. PyDesigner משלב כלים מ-FSL ו-MRtrix3 כדי לבצע דה-נוז, תיקון צלצול גיבס, תיקון תנועה של זרם מערבולת, מיסוך מוח, החלקת תמונה ותיקון הטיה ריציאנית כדי לייעל את ההערכה של מדדי דיפוזיה מרובים. ניתן להשתמש בו בפלטפורמות שונות ב-Windows, Mac ו-Linux כדי להפיק במדויק מדדים נפוצים ממערכי נתונים של DKI, DTI, WMTI, FBI ו-FBWM, כמו גם קבצי ODF ו-.fib של טרקטוגרפיה. הוא גם אגנוסטי לפורמט קובץ, ומקבל קלט בצורה של פורמט .nii, .nii.gz, .mif, ו-dicom. תוכנה זו, ידידותית למשתמש וקלה להתקנה, מוציאה גם מדדי בקרת איכות הממחישים גרפים של יחס אות לרעש, ווקסלים חריגים ותנועת ראש כדי להעריך את תקינות הנתונים. בנוסף, צינור עיבוד dMRI זה תומך בעיבוד מערכי נתונים מרובים בזמן הד וכולל התאמה אישית של צינור, המאפשר למשתמש לציין אילו תהליכים מופעלים ואילו פלטים מיוצרים כדי לענות על מגוון צרכי המשתמש.
Introduction
דיפוזיה MRI (dMRI) מיושמת באופן נרחב למחקר לא פולשני של תכונות מיקרו-מבניות במוח. בעוד שהוצעו שיטות dMRI רבות, שתיים נפוצות הן הדמיית טנזור דיפוזיה (DTI) והדמיית קורטוזיס דיפוזיה (DKI). טכניקות אלו קשורות קשר הדוק, כאשר DKI הוא הרחבה של DTI הכוללת כימות של דיפוזיה שאינה גאוסית1. שניהם מספקים מגוון אמצעי דיפוזיה סקלרית ומאפשרים בניית טרקטוגרפיה של סיבי חומר לבן. יתרון חשוב של DTI ו-DKI הוא שיש להם בסיס איתן בפיזיקת דיפוזיה כך שתוקף שלהם אינו מסתמך על הנחות מפורטות לגבי מיקרו-מבנה רקמות 2,3. זה מאפשר ליישם DTI ו-DKI בכל המוח והגוף עבור משתתפים בכל גיל ובכל מצב מחלה.
מכיוון שתמונות משוקללות דיפוזיה גולמיות (DWIs) מושפלות על ידי מספר גורמים, כולל רעש אות, תנועה, צלצול גיבס ועיוות זרם מערבולת, יש להשתמש בעיבוד מקדים לפני חישוב כמויות דיפוזיה כלשהן4. עיבוד מקדים של DWIs הוא תחום מחקר פעיל שכיום מפותח מאוד. עם זאת, שילוב שלבי העיבוד הדרושים לקו צינור יחיד שנותן תוצאות עקביות הוא מאתגר מכיוון שיש להתאים מספר הגדרות המוגדרות על ידי המשתמש בהתאם לפרטי רכישת ה-dMRI ומכיוון שהסדר שבו מבוצעים שלבי העיבוד המקדים משפיע על התוצאה. מסיבה זו, צינור פרמטר Diffusion EStImation עם Gibbs and NoisE Removal (DESIGNER, GitHub: NYU-DiffusionMRI/DESIGNER) הוצע לראשונה בשנת 2016 כדי לייעל, לתקנן ולייעל את העיבוד המקדים עבור DWIs5. DESIGNER Dv1 מסתמך על כלי תוכנה המוטמעים ב-FSL, MRtrix3, MATLAB ו-Python כדי ליצור תהליך DWI חלק ושלם - כזה המקיף תיקון תמונה באמצעות עיבוד מקדים והערכת טנזור דיפוזיה/קורטוזיס5. עם דגלי בקרה להפעלה או כיבוי של שלבי עיבוד מקדים, ניתן לבצע תיקוני DWI באופן סלקטיבי. DESIGNER תהליכים מקדימים בסדר מסוים - (i) ניתוח רכיבים עיקריים של Marchenko-Pastur (MP-PCA) הפחתת רעשים6, (ii) תיקון צלצול גיבס7, (iii) תיקון עיוות הדמיה אקו-מישורית (EPI)8, תיקון זרם מערבולת9, תיקון תנועה10 והחלפת חריג11, (iv) תיקון שדה הטיה B1, (v) יצירת מסכת מוח, (vi) החלקה, (vii) תיקון הטיית רעש ריציאנית, ו- (viii) נורמליזציה b0. עיבוד מקדים בסדר ספציפי זה משפר הן את הדיוק והן את יחס האות לרעש האפקטיבי (SNR)5. ראוי לציין שכל שלב ב-PyDesigner הוא אופציונלי וניתן להשתמש בו או לדלג עליו על סמך העדפת המשתמש. החלקה, למשל, עשויה שלא להיות שלב הכרחי לעיבוד מקדים עבור מערכי נתונים מסוימים. למרות שזה עוזר להפחית שגיאות סינון, ייתכן שלא יהיה צורך בו עבור מערכי נתונים באיכות גבוהה במיוחד. ככזה, משתמשים יכולים לבחור להשתמש רק בשלבים הדרושים עבור הנתונים שלהם.
הטמעת DESIGNER בפלטפורמות שונות היא מאתגרת עקב הבדלים במערכות ההפעלה ובהגדרות הסביבה. בפרט, העובדה ש-DESIGNER Dv1 נכתב בעיקר ב-MATLAB יוצרת בעיות ניידות משמעותיות הנובעות מדרישות תצורה מסובכות הדרושות כדי לאפשר ממשק Python-MATLAB. יתר על כן, שילובים שונים של גרסאות MATLAB, Python ותלות מסכנים את יכולת השחזור של צינור זה. מסיבה זו, פיתחנו את PyDesigner, שמבוסס כולו על Python. לא רק שצינור עיבוד dMRI זה מאפשר עיבוד מקדים חלק, אלא הוא גם מאפשר לשלב את הצינור במיכל Docker, מה שמשפר מאוד את הניידות והשחזור. בנוסף, על ידי החלפת קוד MATLAB, PyDesigner חוסך את כל דמי הרישוי ומשפר את הנגישות.
צינור עיבוד dMRI זה מגדיל את גישת הדיבורית שהוצגה על ידי DESIGNER, מוסיף מספר תכונות חדשות ומשלב כלים מ-FSL ו-MRtrix3 לביצוע עיבוד מקדים. ספריות Python מתמטיות סטנדרטיות כגון Numpy12, SciPy13 ו-CVXPY 14,15 שימשו להחלפת חלקי MATLAB של DESIGNER בקוד Python. תוכנה זו מסופקת באופן גלוי וזמינה ב-Github16. מטרת מאמר זה אינה לאמת או להשוות את התוכנה שלנו לתוכנות דומות, אלא לתת למשתמשים מדריך צעד אחר צעד לעיבוד הנתונים שלהם עם PyDesigner אם הם בוחרים לעשות זאת.
Protocol
כל הנתונים ששימשו לפיתוח ובדיקת התוכנה נאספו תחת פרוטוקול מחקר שאושר על ידי מועצת הביקורת המוסדית.
הערה: PyDesigner נבנה ב-MacOS 10.14 ודורש מערכת משנה של Windows עבור Linux (WSL) כדי לפעול ב-Windows. ניתן להשתמש באותן פקודות עבור מערכות לינוקס/מק.
1. תאימות מערכת הפעלה
- עבור מערכות Windows, עקוב אחר הקישור כדי להתקין את WSL17 ולאחר מכן המשך לשלב 2.6.
- עבור מערכות מבוססות יוניקס (כגון לינוקס ו-Mac) המשך לסעיף 2.
הערה: מערכות מבוססות יוניקס יכולות להתקין באופן מקורי את PyDesigner ואת יחסי התלות שלו.
2. התקן FSL
הערה: כל הבדיקות של PyDesigner בוצעו עם FSL גרסה 6.0.2, ומומלץ להשתמש בגרסה 6.0.2 ומעלה.
- גשו ל־FSL wiki18 ובחרו הורדה/התקנה. עבור משתמשי Windows, עבור לשלב 2.6.
- בחר הורד FSL תחת הכותרת התקנת FSL. לאחר מכן בחר את רישיון התוכנה להתקנת FSL, קרא את ההסכם ובחר מסכים.
- מלא את השדות הנדרשים ובחר הירשם.
- בחר את הקישור הורד FSL (הקישור הראשון בדף). לאחר מכן בחר את קישור הוראות ההתקנה (קישור שני בדף). עקוב אחר הוראות אלה כדי להוריד FSL עבור כל מערכת הפעלה.
הערה: התקנת FSL אורכת כ-10 דקות. - כדי לוודא התקנה מוצלחת, יש להזין את הפרטים הבאים בחלון מסוף חדש:
$ flirt -version. פעולה זו אמורה להחזיר את מספר הגירסה. המשך לשלב 3. - עבור משתמשי Windows, עקוב אחר מדריך ההתקנה של WSL עבור FSL19.
3. צור סביבת קונדה
- התקן חבילת conda לניהול סביבות Python. משתמשים יכולים לבחור בין Miniconda או Anaconda.
- הורד את Miniconda/Anaconda20. משתמשי Mac ו-Linux ממשיכים לשלב 3.2.
- עבור Windows, הגדר conda בתוך WSL21.
הערה: ההוראות הבאות מיועדות ל-Miniconda, אך אותן פקודות פועלות עבור Anaconda. למרות ש-conda כבר לא תומכת ב-Python 3.7, עדיין ניתן ליצור סביבת conda עם Python 3.7, המאפשרת ליישם פרוטוקול זה בדיוק כפי שתואר.
- הזן את הדברים הבאים בטרמינל כדי ליצור את סביבת הקונדה:
$ conda create -n mri python=3.7. השתמש בסביבת conda זו עבור כל הפעילויות הקשורות ל- PyDesigner.- כאשר תתבקש להשתמש ב
Proceed ([y]/n)?- Entery.
הערה: דגל -n: השתמש באפשרות זו כדי לציין את שם סביבת הקונדה. בסרטון, סביבת הקונדה נקראת mri.
הערה:python=גרסה 3.7 מומלצת, מכיוון ש-PyDesigner נבנה ונבדק על גרסה זו.
- כאשר תתבקש להשתמש ב
4. התקן את MRtrix3
- הפעל את סביבת conda על ידי הזנת הפקודה הבאה:
$ conda activate mri - פתח את דף ההורדה של MRtrix322 ופעל לפי הוראות ההורדה של מערכת ההפעלה המתאימה. קוד ההתקנה יבקש אישור של יחסי תלות מסוימים. היכנס
yכדי להמשיך בתהליך ההורדה.- הדפס את תיעוד MRtrix3 כדי לאשר את ההתקנה על ידי הזנת הדברים הבאים במסוף:
$ mrinfo -h
- הדפס את תיעוד MRtrix3 כדי לאשר את ההתקנה על ידי הזנת הדברים הבאים במסוף:
5. התקן את PyDesigner
- פתח חלון מסוף חדש והפעל את סביבת הקונדה על ידי הזנת הדברים הבאים לטרמינל:
$ conda activate mri - הפעל את הפקודה:
$ pip install PyDesigner-DWI - כדי לוודא התקנה מוצלחת, הדפס את מספר הגרסה על ידי הזנת הדברים הבאים בטרמינל:
$ pydesigner -v- אם השגיאה "המודול לא נמצא" מופיעה, עיין בסעיף פתרון בעיות בסוף הפרוטוקול (סעיף 10)
הערה: $ pydesigner -h ידפיס תיעוד עזרה המסביר את הפקודות והתחביר עבור התוכנה. ניתן להוריד את PyDesigner גם דרך Github. ראה את הוראות ההתקנה באתר23.
- אם השגיאה "המודול לא נמצא" מופיעה, עיין בסעיף פתרון בעיות בסוף הפרוטוקול (סעיף 10)
6. הכנת נתונים
הערה: PyDesigner עובד עם סוגי קבצים מרובים כגון .nii , .nii.gz , .mif ו- DICOM. בסרטון, DICOMs ממוינים ומומרים ל-NIfTI לפני השימוש בצינור עיבוד dMRI זה. קל יותר לעבוד עם קבצי NIfTI מכיוון שהם לא מזוהים, ויש קובץ אחד לכל רצף.
- כדי למיין את רכיבי DICOM, השתמש בכלי DicomSort24.
הערה: כל כלי מיון DICOM אחר זמין שמבצע את אותה משימה מותר. - לאחר מיון ה-DICOMs, המר אותם לקבצי NIfTI באמצעות dcm2niix25.
- התקן dcm2niix על ידי הזנת הפקודה הבאה לטרמינל:
$ sudo apt installdcm2niix - לאחר ההורדה, הקלד את הדברים הבאים במסוף כדי לאמת את ההתקנה על ידי הדפסת התיעוד עבור dcm2nii:
$ dcm2niix -h
- התקן dcm2niix על ידי הזנת הפקודה הבאה לטרמינל:
- כדי להפעיל את dcm2niix, הזן את הפקודה הבאה לטרמינל:
$ dcm2niix -f %s_%p_%d -o pathtosavefolder/ pathtorawdicoms/
הערה: -f: כדי לציין את מבנה שם הקובץ עבור קובץ הפלט, עקוב אחר תג זה עם כל מבנה שם קובץ רצוי. הסרטון משתמש: -f %s_%p_%d . %s, %p ו-%d הם סימונים לסדרה, מספר פרוטוקול ותיאור, בהתאמה. כל מבנה קובץ יעבוד. הפלטים הצפויים מ-dcm2niix הם קבצי .bval, .bvec, .json ו-.nii. אם נרכשו נתוני TOPUP, יהיו קבצי .json ו- .nii TOPUP.
7. שימוש בסיסי ב-PyDesigner
הערה: PyDesigner עשוי להימשך עד 1.5 שעות עבור ערכת נתונים בודדת, בהתאם לפרמטרי העיבוד וליכולות המחשב.
- כדי להפעיל עיבוד רגיל, השתמש בדגל
-sאו-standardב- . פקודה זו תפעיל את צינור העיבוד המקדים המומלץ בסדר הבא: הפחתת רעשים6, תיקון צלצול גיבס7, תיקון עיוות EPI8, תיקון תנועת זרם מערבולת9, יצירת מסכות מוח ונוזל מוחי (CSF), החלקה, תיקון הטיה ריציאנית, זיהוי חריג11, הערכת טנזור ויצירת מפות פרמטריות. - כדי להפעיל את הפקודה הבסיסית של PyDesigner, הפעל תחילה את סביבת conda על ידי הזנת הפקודה הבאה לטרמינל:
$ conda activate mri - הדפס את תיעוד העזרה עבור התוכנה על-ידי הזנת הפקודה הבאה:
$ pydesigner -h. תיעוד זה מסביר את כל הפקודות והתחביר האפשריים עבור PyDesigner. - השתמש בדוגמה הבאה כדי לבנות את הפקודה.
$ pydesigner -s --verbose --rpe_pairs 1 -o ./output_folder/ ./input_folder/input1.nii ./input_folder/input2.nii - שימו לב לדגלים החשובים הבאים:
--verboseהשתמש באפשרות זו כדי להציג את מסוף PyDesigner בזמן שהוא מעבד נתונים (לא נדרש).:--rpe pairs #השתמש בזה כדי לאפשר למשתמשים עם רצף TOPUP להאיץ את תהליך תיקון העיוות של EPI8 באמצעות TOPUP.: # מתייחס לכמה אמצעי אחסון B0 מרצף TOPUP ישמשו. שימוש ב-#=1 מונע הערכת יתר של שדה העיוות ומצמצם את הזמן הדרוש ליצירת שדה זה. משתמשים השתמשו באופן שגרתי ב-#=1 עם תוצאות אמינות. בעת שימוש ב-#=1 PyDesigner יקבל כברירת מחדל את אמצעי האחסון הראשון.
8. שימוש מתקדם ב-PyDesigner: התאמת התוכנה לצרכים האישיים של המשתמשים
הערה: בעת התאמת PyDesigner, השמט את -s הדגל; דגל זה הוא הפקודה לעיבוד מקדים אוטומטי.
- הפעל את סביבת הקונדה על ידי הזנת הפקודה הבאה לטרמינל:
$ conda activate mri - עיין באתר התוכנה23 לקבלת רשימה של דגלים; כל דגל מאפשר למשתמש להריץ כל חלק של PyDesigner בנפרד.
- בנה את הפקודה. התחל עם הדגלים עבור כל רצף שהמשתמש רוצה לבצע. לאחר מכן, הפנה את PyDesigner לקבצי הקלט והפלט. לדוגמה:
$ pydesigner flag1 flag2 flag3 -o ./output_folder/ ./input_folder/input1.nii - דוגמה 1: הפעל את התאמת הטנזור לבד (ללא עיבוד מקדים נוסף).
- הפעל את סביבת הקונדה על ידי הזנת הפקודה הבאה לטרמינל:
$ conda activate mri - עיין באתר PyDesigner23 לקבלת רשימת דגלים. דוגמה זו משתמשת ב-
-m. -mהוסף דגל זה כדי להגביל את התאמת הטנזור לווקסלים במוח בלבד.: זה מאיץ את התאמת הטנזור + זיהוי חריג.- השתמש בדוגמה הבאה כדי לבנות את הפקודה:
$ pydesigner -m -o ./output_folder/ ./input_folder/input1.nii
הערה: הקלט להתאמת טנזור הוא קובץ .nii מעובד מראש.
- הפעל את סביבת הקונדה על ידי הזנת הפקודה הבאה לטרמינל:
- דוגמה 2: הפעל הפחתת רעשים לבד (ללא עיבוד מקדים נוסף).
- הפעל את סביבת הקונדה על ידי הזנת הדברים הבאים לטרמינל:
$ conda activate mri - עיין באתר PyDesigner23 לקבלת רשימת דגלים. דוגמה זו משתמשת ב-
-n. - -n: הוסף דגל זה כדי להשלים את שלב הפחתת הרעשים. הזן את קובץ ה-DWI שעובד מראש בעבר שכותרתו DWI_preproccessed.nii, שנמצא בתיקיית הפלט של כל נושא.
- השתמש בדוגמה הבאה כדי לבנות את הפקודה:
$ pydesigner -n -o ./output_folder/ ./input_folder/input1.nii
- הפעל את סביבת הקונדה על ידי הזנת הדברים הבאים לטרמינל:
9. עדכונים עתידיים של PyDesigner
הערה: עדכונים ניתן למצוא באתר23.
- כדי לעדכן את PyDesigner, הזן את הדברים הבאים בחלון מסוף חדש:
$ pip install --upgrade PyDesigner-DWI
הערה: אם PyDesigner הותקן דרך Github16, יש לעדכן אותו גם באמצעות git.
10. פתרון בעיות בהתקנה
- בעת הורדת PyDesigner, קוד השגיאה "מודול לא נמצא" יופיע אם חסר מודול Python.
- תקן מודול חסר על ידי הזנת הדברים הבאים לטרמינל:
$ pip install nameofmodule - לאחר מכן חזור על פקודת ההתקנה הראשונה על ידי הזנת הדברים הבאים לטרמינל :
$ pip install PyDesigner-DWI - PyDesigner אמור להיות מותקן כעת במלואו. אנא חזור לשלב 5.3. אם מתרחשת אותה שגיאה, חזור על תהליך זה עד להתקנת כל מודולי Python החסרים.
- תקן מודול חסר על ידי הזנת הדברים הבאים לטרמינל:
11. פתרון בעיות שגיאות פלט
- אם מתרחשות בעיות כלשהן, סקור את קובץ log_command.json כדי לזהות שגיאות עיבוד מקדים. סקור את קבצי הביניים כדי לקבוע את השגיאה הספציפית.
- לקבלת עזרה בפתרון בעיות, שלח בקשה בעמוד16 של GitHub.
12. הפעלת PyDesigner עם מערך נתונים לדוגמה
- הורד את מערך הנתונים לדוגמה מ-OpenNeuro כאן26.
הערה: מערך נתונים לדוגמה זה מכיל נתונים גולמיים ומעובדים מראש. הנתונים הגולמיים מיועדים כקלט לעיבוד לדוגמה של PyDesigner. הנתונים המעובדים מראש מורכבים מהפלטים שנוצרו לאחר עיבוד הנתונים הגולמיים באמצעות צינור עיבוד ה-dMRI ב-iMac OS v12.4. משתמשים יכולים להוריד את הנתונים הגולמיים, לעבד נתונים אלה באמצעות PyDesigner, ולהשוות את הפלטים שלהם לפלטים לדוגמה שסופקו כדי להבטיח ש-PyDesigner פועל כהלכה. נתונים גולמיים כלולים בתיקייה הראשית (/PyDesigner Example Dataset/sub-01) והנתונים המעובדים מראש כלולים בתיקיית הנגזרות (/PyDesigner Example Dataset/derivatives/sub-01). אלא אם צוין אחרת, מערך הנתונים של OpenNeuro יורד לתיקיית המשתמש המוגדרת כברירת מחדל. - פתח מסוף חדש ב- Mac / Linux או ב- WSL ב- Windows.
הערה: כדי להפעיל את קו הצינור הרגיל של PyDesigner, בצע את מבנה הפקודות לדוגמה להלן. הזן את הפקודות הבאות למטה בטרמינל/WSL. בדוגמה זו, כאשר " ./user_download_folder/" מציין את הנתיב עבור תיקיית הנתונים לדוגמה. נתיב זה יהיה ספציפי למקום שבו מאוחסנים הנתונים לדוגמה במחשב המשתמשים. - כדי ליצור תיקיה חדשה לפלטים, הזן את הדברים הבאים בטרמינל:
$ mkdir ./user_download_folder/ ds004945-download /PyDesigner_Outputs - כדי להפעיל את סביבת הקונדה, הזן את הדברים הבאים בטרמינל:
$ conda activate mri - כדי להפעיל את PyDesigner במערך הנתונים לדוגמה, הזן את הדברים הבאים לטרמינל:
$ pydesigner - o ./user_download_folder/ds004945-download/PyDesigner_Outputs -s ./user_download_folder/ds004945-download/sub-01/dwi/sub-01_dwi.nii
הערה: בדוגמה זו, ./user_download_folder/ מציין את הנתיב עבור תיקיית הנתונים לדוגמה. נתיב זה יהיה ספציפי למחשב של כל משתמש. הפקודות בשלבים 12.3 עד 12.5 עוקבות אחר המבנה של שלב 8.3. פקודה זו משתמשת בדגל -s, דגל העיבוד הסטנדרטי של PyDesigner, המפעיל את שלבי העיבוד הבאים בסדר זה: הפחתת רעש6, תיקון צלצול גיבס7, ביטול עיוות 8,9, מיסוך מוח, החלקה ותיקון הטיה ריציאנית. - אם התוכנה מותקנת כהלכה, כל היציאות יאוכלסו בתיקיית PyDesigner_Outputs החדשה שנוצרה. השווה את קבצי העיבוד המקדים שהופקו על ידי PyDesigner עם התיקיה המעובדת מראש של מערך הנתונים לדוגמה שנמצאת בתיקיית "נגזרות" שהורדה מ-OpenNeuro.
- עיין בסעיף התוצאות המייצגות כדי להשוות את המפות המטריות שלהם לאלו שבתוצאות מערך הנתונים לדוגמה.
תוצאות
תוכנת PyDesigner מחילה שלבים מרובים לתיקון תמונה על נתוני דיפוזיה גולמיים ומייצרת פלטים המשמשים לשיפור הדיוק בקבצים הגולמיים בעת ביצוע ניתוח. כל שלב זמין בצנרת אומת בעבר באמצעות פרסומים שעברו ביקורת עמיתים 5,6,7,8,9,10,11 כפי שנדון בהקדמה. ניתן להשתמש בתפוקות מהתוכנה בניתוחים כגון פרופילי טרקטוגרפיה, מטריצות קישוריות, ניתוחים מבחינת ווקסל, ניתוחי החזר ROI, ניתוחי fODF, TBSS וניתוחים מבוססי פיקסל.
אתר התוכנה23 מפרט את כל קבצי הפלט שנוצרו במהלך צינור העיבוד המקדים. לאחר הפעלת כל בקשה, המסוף יפיק תיאור של כל התהליכים שהושלמו. ישנם 3 סוגים של קבצי פלט: עיבוד קבצים, מדדים ובקרת איכות. מבנה ספריית הפלט מוצג באיור 1. קבצים אלה זמינים בעת שימוש בעיבוד מקדים סטנדרטי (עיין בסעיף 7 של הפרוטוקול). אם המשתמש זקוק לשימוש מתקדם יותר (עיין בסעיף 8 של הפרוטוקול), קבצי הפלט הזמינים יהיו תלויים בתהליכים שהושלמו.
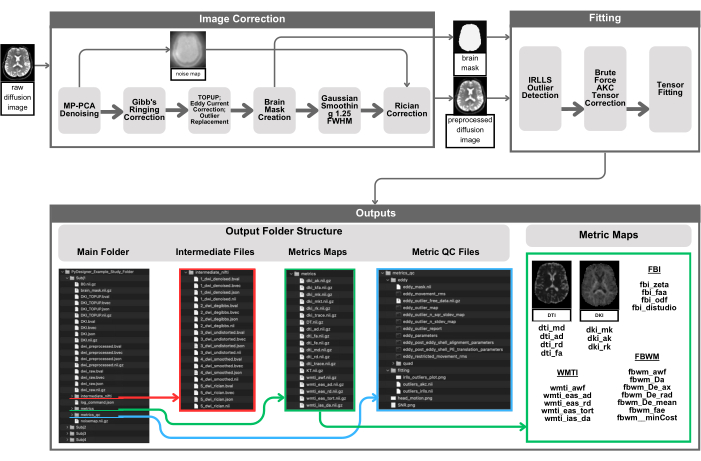
איור 1: ייצוג חזותי של צינור PyDesigner. עיבוד מקדים מתחיל במתן קלט 4D DWI ל-PyDesigner (משמאל למעלה), אשר לאחר מכן עובר הפחתת רעשים MP-PCA כדי להפיק DWI 4D נטול רעש ומפת רעש תלת מימדית. לאחר מכן, ה-4D DWI המנוטרל עובר תיקון צלצול גיבס, תיקון הטיה ריציאנית, TOPUP, תיקון זרם מערבולת ותיקון חריג. לאחר מכן מחושבת מסכת מוח עבור השלבים הבאים, זיהוי חריג והתאמת טנזור כדי להאיץ את החישובים על ידי ביצועם בתוך מסכת המוח בלבד. ניתן למצוא פלטים בספריות משנה בתיקיית עיבוד הנושאים הראשית - intermediate_nifti, מדדים ו-metrics_qc. שימו לב שצילומי המסך של פלטי PyDesigner לא נועדו להוות רשימה ממצה של כל הפלטים האפשריים אלא לספק דוגמה ויזואלית סכמטית למה שמשתמשים יכולים לצפות. הפלטים, הן הפלטים הסופיים והן קבצי הביניים, ישתנו בהתאם לנתוני קלט המשתמש ודגלי העיבוד המופעלים. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
עיבוד
קבצי עיבוד משמשים במהלך קו הצינור של PyDesigner ומאוחסנים בספריית הפלט הבסיסית. עבור כל שלב עיבוד מקדים, קובצי DWI ביניים נשמרים בתיקיית הפלט "intermediate_nifti" כפי שמוצג באיור 1. יש להתייחס לקבצים אלה בעת טיפול בבעיות כלשהן בעיבוד או בתפוקות כדי להעריך כל שלב בצנרת בנפרד.
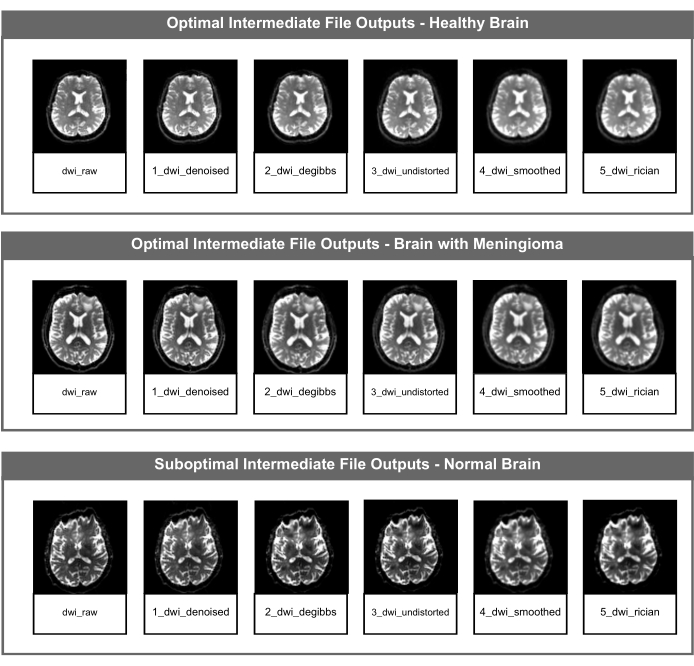
איור 2: קבצי DWI NifTI ביניים אופטימליים ותת-אופטימליים. האיור מציג את קובץ ה-NIfTI הבינוני עבור כל שלב תיקון תמונה של קו הצינור של PyDesigner. השורה העליונה היא דוגמה לפלטי קבצי ביניים אופטימליים באמצעות נתונים ממוח בוגר בריא, השורה האמצעית היא דוגמה לפלטי קבצי ביניים אופטימליים באמצעות נתונים ממוח פתולוגי (מנינגיומה), והשורה התחתונה מציגה פלטי קבצי ביניים לא אופטימליים באמצעות נתונים ממוח מבוגר בריא עם חפץ רגיש שאינו קשור למבנה המוח או לבריאותו. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
מדדי
תיקיה זו מכילה את כל המפות הפרמטריות ש-PyDesigner מחשב (ראה איור 1). זה כולל מפות פרמטריות עבור DTI/DKI, הדמיית כדור סיבים (FBI) / חומר לבן כדורי סיבים (FBWM) ומדדי שלמות דרכי החומר הלבן (WMTI) (טבלה 1)16.

טבלה 1: טווח הערכים הצפוי עבור מדדי DTI/DKI ו-FBI/FBWM. הטבלה כוללת רשימה של מדדי DTI, DKI, FBI ו-FBWM חזקים שנוצרו על ידי PyDesigner וטווחי הערך הצפויים שלהם. הרצפים וערכי b הנדרשים (s/mm2) כדי לגזור כל מדד מפורטים גם הם. מדדי ה-DTI המפורטים הם FA, MD, AD ו-RD. מדדי ה-DKI המפורטים הם MK, AK, RK ו-KFA. מדד ה-FBI הרשום הוא FAA. מדדי ה-FBWM המפורטים הם AWF, DA, DE_AX, DE_RAD ו-FAE.
משתמשים יכולים לבצע בקרת איכות חזותית וערכית (QC) של מדדי הדיפוזיביות הממוצעת (MD), האניזוטרופיה החלקית (FA) והקורטוזיס הממוצע (MK) כדי לזהות תוצאות לא אופטימליות. אם מדדים אלה אינם אופטימליים לפי הסטנדרטים המתוארים להלן, על המשתמשים להסתכל על כל קובץ ביניים המתואר באיור 2 כדי לקבוע איזה שלב עיבוד מקדים לא הצליח.
QC חזותי משמש לזיהוי תוצאות לא אופטימליות (למשל, בעיות התאמת טנזור וחפצים). אנו ממליצים להשתמש ב-ImageJ עבור QC חזותי כדי להבטיח שלא מתבצעת מניפולציה בתמונה באמצעות ברירות מחדל של תוכנה. השורה העליונה של איור 3 מציגה מפות מטריות טיפוסיות של MD, FA ו-MK תוך שימוש בסף סביר מבחינה ביולוגית של 0-3 מיקרומטר2/ms, 0-1 מיקרומטר2/ms ו-0-2 מיקרומטר2/ms, בהתאמה (איור 3 [שורה עליונה]). מפת MD צריכה להכיל את הערכים הגבוהים ביותר בחדרים וערכים גבוהים בחומר האפור בקליפת המוח (איור 3A [שורה עליונה]). מפת FA צריכה להכיל מסלולים של חומר לבן שברורים בכל המוח (איור 3B [שורה עליונה]). למפת MK צריכים להיות ערכים גבוהים בזיכרון העבודה וערכים נמוכים יותר בחומר האפור וב-CSF (איור 3C [שורה עליונה]). איור תלת מימד [שורה עליונה] הוא דוגמה למפה מטרית עם בעיות התאמת טנזור, מה שמביא לאשכולות של ווקסלים בעלי ערך אפס. אם מתרחשות בעיות כלשהן, סקור את קובץ log_command.json כדי למצוא שגיאות עיבוד מקדים. סקור את קבצי הביניים כדי לקבוע את השגיאה הספציפית. לקבלת עזרה בפתרון בעיות, שלח בקשה בדף GitHub של PyDesigner.
בקרת איכות ערך משמשת כדי לזהות אם הווקסלים של מפה מטרית נתונה אחידים יחסית בין הנבדקים עבור כל מערך נתונים. טווח הערכים הצפוי עבור כל מפה ומערך נתונים תלוי בנתונים ובפרמטרים של PyDesigner. במערך נתוני הבדיקה שלנו, היו לנו שיאים עקביים בטווחים של 8000 עד 10,000, 2500 עד 4000 ו-5000 עד 13,000 עבור MD, FA ו-MK, בהתאמה, תוך שימוש בשילוב היסטוגרמה ברירת מחדל של FSLeyes. השורה התחתונה של איור 3 מספקת דוגמאות לשונות היסטוגרמה. טבלה 1 מכילה את ערכי ציר ה-x הצפויים עבור מדדים אלה. ווקסלים גבוהים או נמוכים יותר אופייניים למערך הנתונים או מצביעים על חפץ או בעיית עיבוד מקדים (איור תלת מימד [שורה תחתונה]).
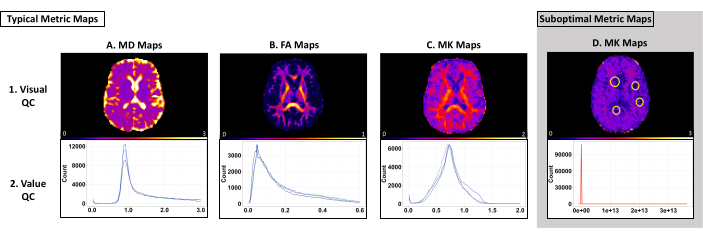
איור 3: דוגמה למפות מטריות והיסטוגרמות מבית PyDesigner עם תוצאות אופטימליות ותת-אופטימליות. השורה העליונה מציגה דוגמאות של מפות MD, FA ו-MK של נושא יחיד המשמשות ל-QC חזותי. השורה התחתונה מציגה היסטוגרמות מרובות נושאים המשמשות לערך QC. (א-ג) דוגמאות אופייניות למפות מטריות והיסטוגרמות שעוברות בקרת איכות חזותית וערכית. כל קו כחול בהיסטוגרמות עבור כל סוג מדד מייצג מערך נתונים בודד. שימו לב שכל מערך נתונים עוקב אחר עקומה דומה ונופל בטווח דומה. (ד) דוגמה למפה מטרית שאינה עוברת בקרת איכות חזותית או ערכית. שימו לב כיצד הקו האדום בהיסטוגרמה מציג עקומה שונה מזו של A-C. האפס ווקסלים המוקפים במפה מטרית זו נובעים מבעיות התאמת טנזור במהלך העיבוד המקדים (פאנל D, שורה עליונה). היסטוגרמה זו היא דוגמה לווקסלים גבוהים או נמוכים בדרך כלל במערך נתונים מהצפוי (פאנל D, שורה תחתונה). אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
בקרת איכות
לאחר ש-PyDesigner עיבד את הנתונים, יש להשתמש בתיקיית metrics_qc (ראה איור 1) לזיהוי מערכי נתונים לא אופטימליים. עבור כל מערך נתונים, PyDesigner מוציא שלוש עלילות המשמשות לבקרת איכות.
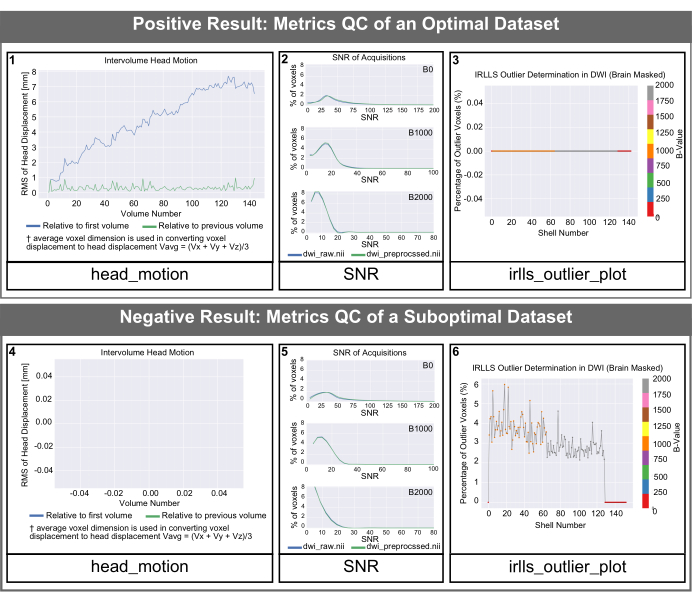
איור 4: היסטוגרמות QC שנוצרו על ידי PyDesigner עבור מערכי נתונים אופטימליים ותת-אופטימליים. SNR, תנועת ראש בין-נפחית והיסטוגרמות חריגות שנוצרו על ידי PyDesigner. שתי השורות מייצגות נתונים ממוח בוגר בריא. השורה העליונה היא דוגמה להיסטוגרמות בקרת איכות עבור מערך נתונים אופטימלי. השורה התחתונה מציגה פלטי בקרת איכות של מערך נתונים לא אופטימלי עם חפץ רגישות שאינו קשור למבנה המוח או לבריאותו. שימו לב שגודל הטקסט של התוויות בפלטי ברירת המחדל של PyDesigner קטן יותר ממה שהן יופיעו באיור זה. הגדלנו את גודל הטקסט באיור זה לקריאות. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
הגרפים head_motion באיור 4 מראים את תזוזה של הראש ביחס לכרך הראשון ולכרך הקודם. כפי שניתן לראות באיור 4 (פאנל 1), תזוזת הראש היא בדרך כלל קטנה ו- PyDesigner מתאים את עצמו עבור חפצי תנועה אלה בצנרת העיבוד הסטנדרטית באמצעות תוכנית FSL Eddy יחד עם TOPUP עבור תיקון תנועה וזרם מערבולת9. עבור מערכי נתונים לא אופטימליים, גרף head_motion עשוי להיראות ריק, כפי שמוצג באיור 4 (פאנל 4). זה מצביע על כך שתיקון תנועת זרם המערבולת לא הצליח ולכן ה-PyDesigner לא הצליח להפיק גרף. ניתן למצוא את יומן התיקון הנוכחי של Eddy files בתיקיית המשנה eddy בתוך תיקיית metrics_qc (ראה איור 1). גרף יחס האות לרעש (SNR) מציג 3 חלקות. כל תרשים מיועד לערך b אחר ומציג הן את הנתונים המעובדים מראש והן את הנתונים הגולמיים. עבור מערך נתונים אופטימלי, שיא ה-SNR של הנתונים הגולמיים צריך להיות ≥5 (איור 4 [פאנל 2]). מערכי נתונים תת-אופטימליים יהיו בעלי שיא SNR של נתונים גולמיים של ≤3 (איור 4 [פאנל 5]). באופן אידיאלי, המשתמשים צריכים לראות ששיא ה-SNR עבור כל ערכי b עולה מעט אך לא באופן דרמטי. התרשים החריג נמצא בתיקיית ההתאמה בתוך metrics_qc ומציג את אחוז החריגים במערך הנתונים (איור 4 [פאנל 3 ו-6]). מערך נתונים טוב צריך להיות בעל אחוז נמוך של חריגים, בדרך כלל פחות מ-5% (איור 4 [פאנל 3]). למערך נתונים לא אופטימלי יהיה אחוז גדול של חריגים, כפי שמוצג באיור 4 (פאנל 6).
תוצאות מערך נתונים לדוגמה
לאחר ש-PyDesigner סיים לעבד את מערך הנתונים לדוגמה, כל הפלטים צריכים להיות כלולים בתיקיית "PyDesigner_Outputs". ניתן להשוות את הפלטים הללו לאלה שנמצאים בתיקיית "נגזרות" ארוזה עם מערך הנתונים לדוגמה שהורד מ-OpenNeuro (מעובד ב-MacOS 12.4). אם התוכנה פועלת כהלכה, מבנה הקבצים של "PyDesigner_Outputs" ו"נגזרות" יהיה זהה לחלוטין. באופן דומה, ה-SNR, תנועת הראש והעלילות החריגות שנמצאות בתיקיית המשנה "metrics_qc" צריכות להתאים לאלה באיור 5A. ניתן להשוות מפות מטריות (שנמצאות בתיקיית המשנה "מדדים") באמצעות תוכנות הדמיה כגון FSLeyes, MRIcron, ImageJ וכו'. ניתן לראות היסטוגרמות של ערכי FA, MD ו-MK ממדדים מעובדים מראש/מדדים באיור 5B. שימו לב שכל ההיסטוגרמות המטריות המוצגות באיור 5B מותאמות על סמך סולמות הערכים המטריים המוצעים בטבלה 1.
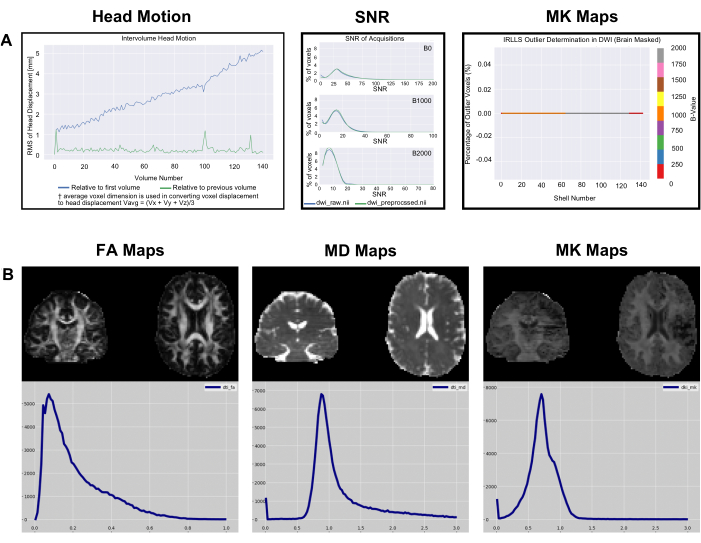
איור 5: דוגמה להיסטוגרמות QC מטריות ומטריות של נתונים. (A) SNR, תנועת ראש בין-נפחית והיסטוגרמות חריגות שנוצרו על ידי PyDesigner עבור הנתונים לדוגמה שהורדו מ-OpenNeuro. שים לב שגודל הטקסט הוגדל בעלילות באיור זה לצורך קריאות. (B) מטרי נושא יחיד ממפה היסטוגרמות של ספירת ערכי ווקסל עבור FA, MD ו-MK עבור אותו מערך נתונים, המוצג באמצעות FSLeyes v6.0. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
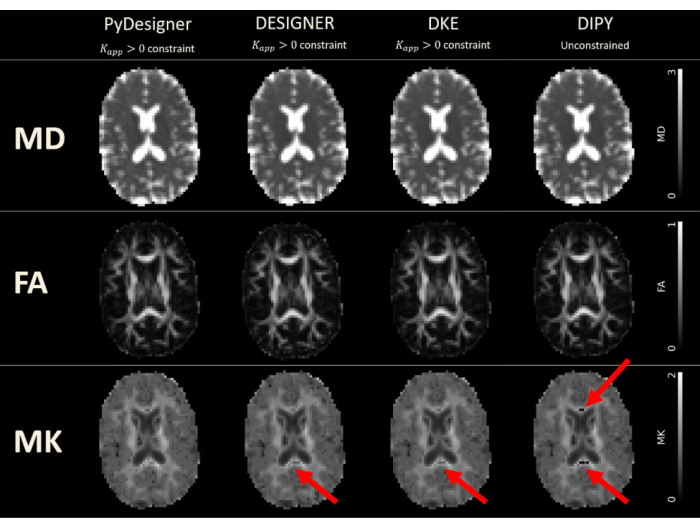
איור 6: מפות DTI ו-DKI נגזרות מ-PyDesigner, DESIGNER, DKE ו-DIPY. התאמת טנזור בוצעה עם אילוץ Kapp > 0 ב-PyDesigner, DESIGNER5 ו-Diffusional Kurtosis Estimator (DKE)27, בעוד שהתאמה לא מוגבלת שימשה ב-Diffusion Imaging in Python (DIPY)28 עקב מגבלות תוכנה. היחידות עבור MD הן מיקרומטר מרובע לאלפית שנייה (מיקרומטר2/ms), בעוד שהמדדים האחרים הם חסרי מימד. ניתן לראות נשירה עקב בעיה גנרית מתיקון הצלצול של גיבס במפות MK שהופקו על ידי DESIGNER, DKE ו-DIPY. נתון זה שוחזר באישור של Dhiman et al.29. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
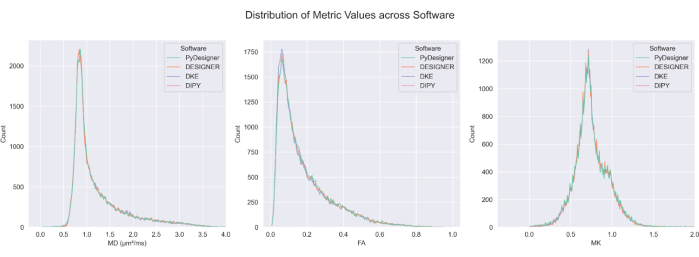
איור 7: השוואה בין FA, MD ו-MK על פני צינורות. התפלגות הערכים המחושבים עבור FA, MD ו-MK מ-PyDesigner, DESIGNER5, DKE27 ו-DIPY28 במוחות שאינם נכללים ב-CSF דומה ברוב הווקסלים. חישוב מפה מטרית בין שיטות ניתן להשוואה. נתון זה שוחזר באישור של Dhiman et al.29. אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
Discussion
המוטיבציה העיקרית לפיתוח PyDesigner הייתה ליישם את מרכיבי המפתח של DESIGNER תוך החלפת כל קוד MATLAB ב-Python, ובכך לאפשר ניידות ונגישות רבה יותר. PyDesigner ו-DESIGNER Dv1 מניבים תפוקות כמעט זהות29. עם זאת, ישנן כמה אפשרויות נוספות, הגדרות ברירת מחדל ותיקוני באגים קלים הכלולים ב-PyDesigner. התיעוד המקוון של PyDesigner16 מתאר זאת בפירוט.
PyDesigner גם מניב תוצאות דומות לכלי ניתוח Diffusional Kurtosis Estimator (DKE)27 ו-Diffusion Imaging in Python (DIPY)28 DKIכלי ניתוח 29 (ראה איור 6 ואיור 7), אך המפות שנוצרו על סמך dMRI מעובד מראש של DESIGNER נחשבות למדויקות יותר בשל אלגוריתם ההתאמה שלהן כפי שהודגם על ידי Ades-Aron et al.5. ראה איור 6 להשוואה בין המפות המטריות MD, FA ו-MK מ-PyDesigner, DESIGNER5, DKE27 ו-DIPY28. איור 7 מציג את ההשוואה בין היסטוגרמות MD, FA ו-MK מכל צינור. שילוב של התאמת טנזור מוגבלת, גילוי חריג ותיקון מקדם קורטוזיס לכאורה מניב התאמת טנזור חזקה ומדויקת יותר, כפי שניתן לראות ב-PyDesignerו-DESIGNER 5.
היתרון של PyDesigner על פני DESIGNER Dv1 הוא שהוא זמין דרך מיכל NeuroDock Docker30, מה שמשפר מאוד את הניידות ומפשט את ההתקנה. מיכל זה פועל בכל פלטפורמות מערכת ההפעלה העיקריות התואמות ל-Docker, כולל Windows, Mac OS והפצות לינוקס שונות. בהתבסס על משוב משתמשים, PyDesigner v2.0 יכלול עדכונים למיכל PyDesigner Docker. השיפורים שהוצגו על ידי הגרסה החדשה של PyDesigner והכנסת Dockerfile רב-שלבי יפתרו את כל הבעיות הקיימות שעמן מתמודדים המשתמשים. אם למשתמשים יש בעיות בהתקנה, אנו ממליצים לשלוח שאלות לדף השיחה של PyDesigner16. טכנולוגיית הקונטיינרים של Docker מאפשרת גם פריסה פשוטה לאשכולות בעלי ביצועים גבוהים (HPC) לעיבוד אצווה של DWIs במהירות באשכולות מקומיים תואמי Docker.
PyDesigner כולל גם חישובי מידול מיקרו-מבני החורגים מ-DKI, כולל WMTI3, FBI ו-FBWM. עבור WMTI, מערך נתונים סטנדרטי של DKI מספיק, והפרמטרים המיקרו-מבניים הקשורים מחושבים כברירת מחדל. עם זאת, יש להדגיש כי התוקף של WMTI מוגבל לאזורי חומר לבן עם FA גבוה (כלומר, FA ≥ 0.4). לחלק ממדדי WMTI יש דיוק מוגבל בשל ההנחה של יישור מקביל של אקסונים בכל ווקסל31 נתון. FBI 32,33,34 היא שיטת dMRI מובהקת המיושמת בכל החומר הלבן במוח, הדורשת ערך b גבוה (כלומר, b ≥ 4000 s/mm 2) ונתוני dMRI שנדגמו עם מינימום של 64 כיווני קידוד דיפוזיה (יחד עם נתונים עבור b = 0). התפוקות העיקריות של ה-FBI הן פונקציית צפיפות כיוון הסיבים (fODF) עבור כל ווקסל חומר לבן, שיכולה לשמש לטרקטוגרפיה של חומר לבן ומשמשת כקלט ל-FBWM, כמו גם לאניזוטרופיה חלקית תוך-אקסונלית (FAA). FBWM משתמש בנתוני ה-dMRI הן מ-DKI והן מה-FBI כדי להעריך את אותם פרמטרים כמו WMTI אך עם דיוק משופר, וניתן ליישם אותם בכל החומר הלבן ללא קשר לערך ה-FA. לפיכך, אם נתונים נוספים אלה זמינים, אז הערכות FBWM עדיפות על פני אלה של WMTI35. כמו ב-FBI, FBWM אומת רק בחומר לבן מוחי במבוגרים.
בנוסף למשתנים הסיבוביים המסופקים על ידי PyDesigner (כיוון RAS), קבצי .fib ספציפיים ל-DSIstudio (כיוון LPS) מיוצרים בנפרד עבור DTI, DKI ו-FBI. קבצי ה-.fib מכילים מידע כיווני ODF ליצירת פרופילי טרקטוגרפיה עבור כל שיטה. ניתן להשתמש בפרופילי הטרקטוגרפיה המתקבלים ליצירת מטריצות קישוריות ברמת הנושא והקבוצה. קבצי ה-.fib של DKI ו-FBI מכילים מידע רב-כיווני המעביר מידע על סיבים חוצים שאינו זמין עם טרקטוגרפיה של DTI. יתר על כן, בתוך כל קובץ .fib נכללו המשתנים הסיבובים השונים של כל שיטה, שניתן להשתמש בהם בשילובים שונים כקריטריונים לזריעה, ביצוע ועצירת הטרקטוגרפיה. פרטים נוספים על DSIstudio ניתן למצוא באתר האינטרנט שלהם36.
תכונה בולטת נוספת של PyDesigner היא קלט מרובה קבצים, המאפשר לו לטפל בכניסות קבצים שונות - NifTi (.nii), NifTi דחוס (.nii.gz), DICOM (.dcm) ופורמט קובץ MRtrix (.mif). PyDesigner יכול לזהות באופן אוטומטי מידע רכישה ממטא נתונים של כותרת ללא קשר לפורמט הקלט ולבצע תיקונים בהתאם, ובכך לתמוך בגישה ללא ידיים. ללא קשר להבדלים בפרוטוקולים, ניתן להשתמש באותה פקודה (ראה לעיל) לעיבוד מגוון רחב של DWIs. תוכנה זו חוסכת זמן ומאמץ על ידי מזעור שלבי עיבוד מקדים ופקודות ידניות. בעדכון שפורסם לאחרונה (v1.0-RC10), זה שופר על ידי הצגת תאימות למספר מערכי נתונים של זמן הד (multi-TE). זה מאפשר ל- PyDesigner להריץ שלבי עיבוד מקדים של תמונה, שאינם תלויים ברובם ב- TE, על DWI מרובה TE כדי להפיק תמונה עם רעש וחפצים מינימליים. חישובי טנזור תלויי TE מבוצעים לאחר מכן על כל TE בנפרד כדי לייצר מדדי דיפוזיה או קורטוזיס.
נציין כי PyDesigner v1.0 אינו מייצג נקודת קצה בעיצוב צינורות עיבוד מקדים של תמונה. פיתוח ותיקוף של כלי עיבוד תמונה הוא תחום מחקר פעיל. בפרט, במהלך הכנת כתב היד הזה, הוצעו פיתוחים חדשים בהפחתת רעשי תמונה והסרת צלצולים של גיבס, וכתוצאה מכך שוחרר צינור מתוקן של Designer, Designer Dv237, הכולל הפחתת רעשים ותיקון משופרים של חפצי גיבס עבור dMRI שנרכשו עם רכישות פורייה חלקיות35, וזמין ב-DESIGNER Github38. כמו כן, תאימות ארגונית ל-BIDS39 היא היבט חשוב של כל צינור ניתוח MRI. פורמט BIDS מאפשר שיתוף נתונים יעיל יותר והטמעת צנרת על ידי יצירת מבנה מוגדר ספציפי לכל סוגי הנתונים של MRI. למרות ש-PyDesigner אינו תואם כרגע ל-BIDS, גרסה קרובה של PyDesigner תכלול פלטים תואמי BIDS. בנוסף, PyDesigner נכתב במקור באמצעות Python גרסה 3.7. בזמן פרסום זה, Python 3.7 נחשב כעת למיושן. עם זאת, המידע הכלול בכתב יד זה אינו תלוי בגרסת פייתון. כלומר, ההליכים המתוארים כאן שהם פונקציות עיבוד מקדים חשובות (למשל, דה-נוז, תיקון צלצול גיבס וכו') יעקבו אחר אותה זרימת עבודה תפעולית וקונספטואלית ללא קשר לשינויים בהליכי ההתקנה. חשוב לציין, כמו בכל התוכנות המבוססות על Python, גרסאות עתידיות של PyDesigner יתעדכנו לגרסה חדשה של Python. אנו שואפים לאמת ולשלב התפתחויות כאלה לאורך זמן.
לקבלת הגרסה העדכנית ביותר של PyDesigner, כולל כל תיעוד חדש הרלוונטי לעדכוני גרסת Python, הקוראים מוזמנים להתייעץ עם האתר לפני תחילת ניתוח חדש ולהשתתף בפורום הדיונים שבו ניתן להגיש שאלות בנוגע ל-PyDesigner16. יישום Docker לניידות נקרא NeuroDock30, המכיל את PyDesigner והתלות שלו כדי לאפשר עיבוד על פני מגוון רחב של פלטפורמות.
Disclosures
ללא.
Acknowledgements
אנו אסירי תודה על הדיונים המועילים עם אוליביה הורן, דניאל לנץ' וגרהם וורנר.
המחקר המדווח בפרסום זה נתמך, בין השאר, על ידי מענקי המכונים הלאומיים לבריאות R01AG054159, R01AG057602, R01AG055132, R01DC014021, R01NS110347, R21DA050085, F31NS108623, P20GM109040, P50DC000422, T32GM008716 ו-T32DC014435. התוכן הוא באחריות המחברים בלבד ואינו מייצג בהכרח את העמדות הרשמיות של המכונים הלאומיים לבריאות. מימון נוסף ניתן על ידי קרן ליטוין.
Materials
| Name | Company | Catalog Number | Comments |
| Python version 3.7 or above | Python Software Foundation | https://www.python.org/ | |
| FMRIB Software Library (FSL) verison 6.0.2 or above | University of Oxford Centre for Integrative Neuroimaging | https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/ | |
| MRtrix3 version 3.0_RC3 or above | numerous contributors | https://www.mrtrix.org/ | |
| Anaconda | Anaconda | https://anaconda.org/ | |
| Computer | Apple | Mac OS 10.14 | Built on Mac OS 10.14; tested on Mac OS 12.4, Mac OS 13, Windows 11 via WSL |
References
- Jensen, J. H., Helpern, J. A. MRI quantification of non-Gaussian water diffusion by kurtosis analysis. NMR Biomed. 23 (7), 698-710 (2010).
- Basser, P. J. Relationships between diffusion tensor and q-space MRI. Magn Reson Med. 47 (2), 392-397 (2002).
- Fieremans, E., Jensen, J. H., Helpern, J. A. White matter characterization with diffusional kurtosis imaging. Neuroimage. 58 (1), 177-188 (2011).
- Le Bihan, D., Poupon, C., Amadon, A., Lethimonnier, F. Artifacts and pitfalls in diffusion MRI. J Magn Reson Imaging. 24 (3), 478-488 (2006).
- Ades-Aron, B., et al. Evaluation of the accuracy and precision of the diffusion parameter EStImation with Gibbs and NoisE removal pipeline. Neuroimage. 183, 532-543 (2018).
- Veraart, J., Novikov, D. S., Christiaens, D., Ades-aron, B., Sijbers, J., Fieremans, E. Denoising of diffusion MRI using random matrix theory. Neuroimage. 142, 394-406 (2016).
- Kellner, E., Dhital, B., Kiselev, V. G., Reisert, M. Gibbs-ringing artifact removal based on local subvoxel-shifts. Magn Reson Med. 76 (5), 1574-1581 (2016).
- Holland, D., Kuperman, J. M., Dale, A. M. Efficient correction of inhomogeneous static magnetic field-induced distortion in Echo Planar Imaging. Neuroimage. 50 (1), 175-183 (2010).
- Andersson, J. L. R., Sotiropoulos, S. N. An integrated approach to correction for off-resonance effects and subject movement in diffusion MR imaging. Neuroimage. 125, 1063-1078 (2016).
- Godenschweger, F., et al. Motion correction in MRI of the brain. Phys Med Biol. 61 (5), 32-56 (2016).
- Andersson, J. L. R., Graham, M. S., Zsoldos, E., Sotiropoulos, S. N. Incorporating outlier detection and replacement into a non-parametric framework for movement and distortion correction of diffusion MR images. Neuroimage. 141, 556-572 (2016).
- Harris, C. R., et al. Array programming with NumPy. Nature. 585 (7825), 357-362 (2020).
- Virtanen, P., et al. SciPy 1.0: fundamental algorithms for scientific computing in Python. Nat Methods. 17 (3), 261-272 (2020).
- Agrawal, A., Verschueren, R., Diamond, S., Boyd, S. A rewriting system for convex optimization problems. J Control Decis. 5 (1), 42-60 (2018).
- Diamond, S., Boyd, S. CVXPY: A Python-embedded modeling language for convex optimization. , Available from: http://arxiv.org/abs/1603.00943 (2016).
- PyDesigner. , Available from: https://github.com/muscbridge/PyDesigner (2023).
- How to install Linux on Windows with WSL. , Available from: https://learn.microsoft.com/en-us/windows/wsl/install (2023).
- FSL Wiki. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki (2024).
- Fsl Instillation. , Available from: https://fsl.fmrib.ox.ac.uk/fsl/fslwiki/FslInstallation/Windows (2024).
- Installing conda. , Available from: https://conda.io/projects/conda/en/stable/user-guide/install/download.html (2024).
- Steps to install Anaconda on Windows Ubuntu terminal. , Available from: https://gist.github.com/kauffmanes/5e74916617f9993bc3479f401dfec7da (2024).
- Install MRtrix3. , Available from: https://www.mrtrix.org (2024).
- PyDesigner Read the Docs. , Available from: https://pydesigner.readthedocs.io/en/latest/ (2024).
- dicomSort. , Available from: https://github.com/muscbridgelab/dicomSort (2024).
- dcm2niix. , Available from: https://github.com/rordenlab/dcm2niix (2024).
- PyDesigner Example Dataset. , Available from: https://openneuro.org/datasets/ds004945 (2024).
- Tabesh, A., Jensen, J. H., Ardekani, B. A., Helpern, J. A. Estimation of tensors and tensor-derived measures in diffusional kurtosis imaging. Magn Reson Med. 65 (3), 823-836 (2011).
- Garyfallidis, E., et al. Dipy, a library for the analysis of diffusion MRI data. Front Neuroinform. 8, 8(2014).
- Dhiman, S., et al. PyDesigner: A Pythonic implementation of the DESIGNER pipeline for diffusion tensor and diffusional Kurtosis imaging. bioRxiv. , (2021).
- Neurodock. , Available from: https://hub.docker.com/r/dmri/neurodock (2024).
- Jelescu, I. O., et al. One diffusion acquisition and different white matter models: How does microstructure change in human early development based on WMTI and NODDI. Neuroimage. 107, 242-256 (2015).
- Jensen, J. H., Russell Glenn, G., Helpern, J. A. Fiber ball imaging. Neuroimage. 124, 824-833 (2016).
- Moss, H. G., Jensen, J. H. High fidelity fiber orientation density functions from fiber ball imaging. NMR Biomed. 35 (1), e4613(2022).
- Moss, H. G., McKinnon, E. T., Glenn, G. R., Helpern, J. A., Jensen, J. H. Optimization of data acquisition and analysis for fiber ball imaging. Neuroimage. 200, 690-703 (2019).
- McKinnon, E. T., Helpern, J. A., Jensen, J. H. Modeling white matter microstructure with fiber ball imaging. Neuroimage. 176, 11-21 (2018).
- DSIstudio. , Available from: https://dsi-studio.labsolver.org (2024).
- Chen, J., et al. Optimization and validation of the DESIGNER dMRI preprocessing pipeline in white matter aging. ArXiv. , arXiv:2305.14445v2 (2024).
- DESIGNER. , Available from: https://github.com/NYU-DiffusionMRI/DESIGNER (2024).
- Gorgolewski, K. J., et al. The brain imaging data structure, a format for organizing and describing outputs of neuroimaging experiments. Sci Data. 3, 160044(2016).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved