A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
יישום של ניתוח גורמים רב-אומיים ללא פיקוח כדי לחשוף דפוסים של שונות ותהליכים מולקולריים הקשורים למחלות לב וכלי דם
In This Article
Summary
אנו מציגים זרימת עבודה גמישה וניתנת להרחבה המבוססת על מעבדת יופיטר לניתוח ללא פיקוח של מערכי נתונים מורכבים של מולטי-אומיקס, המשלבת שלבים שונים של עיבוד מקדים, הערכה של מודל ניתוח הגורמים הרב-אומיקס ומספר ניתוחים במורד הזרם.
Abstract
מנגנוני מחלה הם בדרך כלל מורכבים ונשלטים על ידי אינטראקציה של מספר תהליכים מולקולריים שונים. מערכי נתונים מורכבים ורב-ממדיים הם משאב רב ערך ליצירת תובנות נוספות לגבי תהליכים אלה, אך הניתוח של מערכי נתונים כאלה יכול להיות מאתגר בשל המימדיות הגבוהה הנובעת למשל ממצבי מחלה שונים, נקודות זמן ואומיקה הלוכדת את התהליך ברזולוציות שונות.
כאן, אנו מציגים גישה לנתח ולחקור מערך נתונים מורכב כזה של מולטיומיקה באופן בלתי מפוקח על ידי יישום ניתוח גורמים מולטי-אומיים (MOFA) על מערך נתונים שנוצר מדגימות דם הלוכדות את התגובה החיסונית בתסמונות כליליות חריפות וכרוניות. מערך הנתונים מורכב ממספר בדיקות ברזולוציות שונות, כולל נתוני ציטוקינים ברמת הדגימה, פלזמה-פרוטאומיקה ונויטרופילים ראשוניים-seq, ונתוני RNA-seq חד-תאי (scRNA-seq). מורכבות נוספת מתווספת על ידי כך שנמדדו מספר נקודות זמן שונות לכל מטופל וכמה תת-קבוצות של מטופלים.
זרימת העבודה של הניתוח מתארת כיצד לשלב ולנתח את הנתונים במספר שלבים: (1) עיבוד מראש של נתונים והרמוניזציה, (2) הערכה של מודל MOFA, (3) ניתוח במורד הזרם. שלב 1 מתאר כיצד לעבד את התכונות של סוגי הנתונים השונים, לסנן תכונות באיכות נמוכה ולנרמל אותן כדי ליצור הרמוניה בין ההפצות שלהן לניתוח נוסף. שלב 2 מראה כיצד ליישם את מודל MOFA ולחקור את מקורות השונות העיקריים בתוך מערך הנתונים בכל האומיקה והתכונות. שלב 3 מציג מספר אסטרטגיות לניתוח במורד הזרם של הדפוסים שנלכדו, ומקשר אותם לתנאי המחלה ולתהליכים המולקולריים הפוטנציאליים השולטים בתנאים אלה.
בסך הכל, אנו מציגים זרימת עבודה לחקירת נתונים ללא פיקוח של מערכי נתונים מולטי-אומיים מורכבים כדי לאפשר זיהוי של צירי וריאציה עיקריים המורכבים מתכונות מולקולריות שונות שניתן ליישם גם בהקשרים אחרים ובמערכי נתונים רב-אומיים (כולל בדיקות אחרות כפי שהוצגו במקרה השימוש לדוגמה).
Introduction
מנגנוני מחלה הם בדרך כלל מורכבים ונשלטים על ידי אינטראקציה של מספר תהליכים מולקולריים שונים. פענוח המנגנונים המולקולריים המורכבים המובילים למחלות ספציפיות או שולטים באבולוציה של מחלה היא משימה בעלת רלוונטיות רפואית גבוהה מכיוון שהיא עשויה לחשוף תובנות חדשות להבנה וטיפול במחלות.
ההתקדמות הטכנולוגית האחרונה מאפשרת למדוד תהליכים אלה ברזולוציה גבוהה יותר (למשל, ברמת התא הבודד) ובשכבות ביולוגיות שונות (למשל, DNA, mRNA, נגישות כרומטין, מתילציה של DNA, פרוטאומיקה) בו זמנית. זה מוביל לדור הולך וגדל של מערכי נתונים ביולוגיים רב-ממדיים גדולים, שניתן לנתח במשותף כדי להפיק תובנות נוספות לגבי התהליכים הבסיסיים. יחד עם זאת, שילוב וניתוח מקורות הנתונים השונים באופן בעל משמעות ביולוגית נותר משימה מאתגרת1.
מגבלות טכנולוגיות, רעשים וטווחי שונות שונים בין אומיקה שונים מציבים אתגר אחד. לדוגמה, נתוני ריצוף RNA חד-תאי (scRNA-seq) הם דלילים מאוד ומושפעים לעתים קרובות מהשפעות טכניות או אצווה גדולות. בנוסף, מרחב התכונות הוא לעתים קרובות גדול מאוד, ונע על פני כמה אלפי גנים או חלבונים שנמדדו, בעוד שגודל הדגימה מוגבל. זה מסובך עוד יותר על ידי תכנונים מורכבים, שעשויים לכלול מספר מצבי מחלה, גורמים מבלבלים, נקודות זמן ורזולוציות. לדוגמה, במקרה השימוש המוצג, סוגי נתונים שונים היו זמינים ברמת תא בודד או מדגם (בתפזורת). מלבד זאת, הנתונים עשויים להיות חלקיים, ולא כל המדידות עשויות להיות זמינות עבור כל הנבדקים שנותחו.
בשל אתגרים אלה, אומיקה שונה ותכונות כלולות עדיין מנותחות לעתים קרובות רק בנפרד2 למרות שביצוע ניתוח משולב אינו יכול רק לספק תמונה מלאה של התהליך, אלא רעשים ביולוגיים וטכניים מאומיקה אחת עשויים להיות מפוצים גם על ידי אומיקה אחרת 3,4. מספר שיטות שונות הוצעו לביצוע ניתוח משולב של נתונים מולטי-אומיקס, כולל שיטות בייסיאניות, שיטות מבוססות רשת 5,6, למידה עמוקה רב-מודאלית7 ושיטות הפחתת ממדיות באמצעות פקטורליזציה של מטריצה 8,9. עבור האחרון, התוצאות של מחקר השוואת ביצועים גדול10 הראו את שיטת MOFA9 (ניתוח גורמים multi-omic) כאחד הכלים המתאימים יותר כאשר הנתונים צריכים להיות מקושרים ביאורים קליניים.
במיוחד בסביבות מורכבות, שיטות פירוק לגורמים של מטריצה ללא פיקוח הן גישה שימושית להפחתת המורכבות ולחילוץ אותות משותפים ומשלימים ממקורות נתונים ותכונות שונות. על ידי פירוק המרחב המרוכב לייצוגים סמויים מדרגה נמוכה יותר, ניתן לחקור במהירות את מקורות השונות העיקריים בתוך הנתונים ולקשר אותם לקובריאטים ידועים. במקרה שאותו דפוס של שונות משותף על פני תכונות מרובות (למשל, גנים או חלבונים), זה עשוי להיות מצטבר למספר גורמים בזמן שהרעש מופחת. ניתן להשתמש ברגולציה כדי להגדיל את הדלילות של מקדמי המודל, מה שהופך את הגישה למתאימה היטב בהגדרות שבהן מרחב התכונות גדול בעוד שמספר הדגימות מוגבל9.
פרוטוקול זה מציג זרימת עבודה גמישה של ניתוח המשתמש במודל MOFA כדי להציג כיצד לחקור במהירות מערך נתונים מורכב של מולטי-אומיקס ולזקק את דפוסי השונות העיקריים המאפיינים מערך נתונים זה. זרימת העבודה מורכבת משלושה שלבים עיקריים. בשלב הראשון, עיבוד מראש של נתונים והרמוניזציה, מוצגות אסטרטגיות שונות לעיבוד מקדים של נתונים המבוססים על סוגי נתוני קלט שונים (scRNA-seq, פרוטאומיקה, ציטוקינים, נתונים קליניים). הפרוטוקול מפרט כיצד לעבד את התכונות של מערכי נתוני הקלט השונים, לסנן תכונות באיכות נמוכה ולנרמל אותן כדי ליצור הרמוניה בין ההתפלגויות שלהן. אנו גם מראים כיצד החלטות קדם-עיבוד אלה עשויות להשפיע על התוצאות במורד הזרם. בשלב השני, מודל MOFA מוחל על הנתונים, וניתן להשתמש בפירוק השונות המתקבל כדי להעריך את השילוב של מערכי הנתונים השונים. השלב השלישי מראה כיצד לקשר את הגורמים שנלכדו למשתנים משותפים ולחשוף את התוכניות המולקולריות המגדירות גורמים אלה. בעזרת זרימת העבודה שהוצגה, הצלחנו לחלץ מספר גורמים סמויים הקשורים לקו-משתנים קליניים במערך נתונים של חולים הסובלים מתסמונות כליליות ולזהות תוכניות חיסון רב-תאיות פוטנציאליות מפרויקט קודם11. אנו נשתמש במערך נתונים זה כאן, אך ניתן להחיל את הפרוטוקול בקלות על הקשרים אחרים, כולל omics אחרים.
מערך הנתונים מורכב מדגימות ממטופלים עם תסמונות כליליות כרוניות יציבות (CCS), תסמונות כליליות חריפות (ACS) וקבוצת ביקורת עם מחלות לב כליליות בריאות (שאינן CCS) (איור 1). ACS נגרמת על ידי קרע פלאק ב- CCS קיים, המוביל להפרעה חריפה של זרימת הדם לשריר הלב ופגיעה איסכמית של הלב לאחר מכן. פגיעה זו גורמת לתגובה דלקתית של מערכת החיסון ולאחריה שלב מתקן, שנמשך עד מספר ימים לאחר האירוע החריף12. כדי לאפיין תגובה חיסונית זו עבור חולי ACS, דגימות דם נלקחו בארבע נקודות זמן שונות: אקוטי (TP1); לאחר חידוש (14 [± 8] ח) (TP2); 60 [± 12] שעה מאוחר יותר (TP3); לפני השחרור (6.5 [±1.5] ימים) (TP4) (איור 1A). עבור CCS וחולים עם מחלות לב כליליות בריאות, רק נקודת זמן אחת הייתה זמינה- (TP0). עבור כל החולים ונקודות הזמן נמדדו בדיקות שונות המבוססות על דגימות הדם: סמנים קליניים של דלקת (קריאטין-קינאז (CK), CK-MB, טרופונין, חלבון מגיב C (CRP)), scRNA-seq של תאי דם חד-גרעיניים היקפיים (PBMCs), ניתוח ציטוקינים, פרוטאומיקה פלזמה ונתוני Prime-seq13 של נויטרופילים.
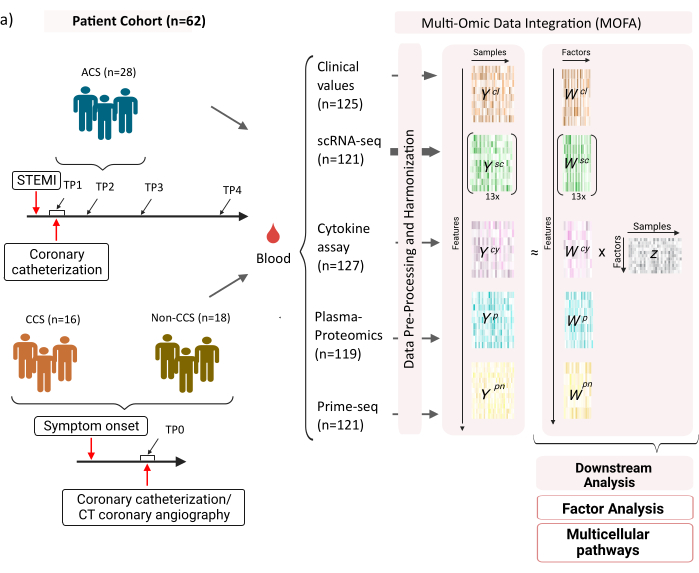
איור 1: ערכת נתונים של קלט מולטי-אומי של אוטם שריר הלב. ערכת נתוני קלט: הנתונים המנותחים כוללים דגימות דם מחולים (n = 62) עם תסמונת כלילית חריפה (ACS), תסמונות כליליות כרוניות (CCS) וחולים עם כליליים בריאים (שאינם CCS). עבור חולי ACS דגימות דם נכללו בארבע נקודות זמן שונות (TP1-4), עבור CCS וחולים שאינם CCS בנקודת זמן אחת (TP0). כל שילוב של מטופל ונקודת זמן מטופל כמדגם נפרד בניתוח. בדיקות OMIC שונות נמדדו על הדגימות: בדיקות דם קליניות (n = 125), scRNA-seq (n = 121), פלזמה-פרוטאומיקה (n = 119), בדיקת ציטוקינים (n = 127) ונויטרופילים prime-seq (n = 121). לאחר מכן, הפרוטוקול המתואר יושם כדי לשלב את הנתונים בכל האומיקה ולחקור אותם באמצעות מודל MOFA וניתוח במורד הזרם (ניתוח גורמים, העשרת מסלולים). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
כקלט עבור זרימת העבודה כפי שמוצג כאן, אנו לוקחים ספירות גולמיות מנתוני scRNA-seq לאחר עיבוד עם cellranger ובקרת איכות (QC) כמתואר לדוגמה, במדריך העיבוד מראש של scanpy14 . עבור ביאור מסוג תא, השתמשנו בצינור Azimuth15 האוטומטי. לאחר מכן הספירות נצברות ברמת המדגם עבור כל סוג תא על-ידי לקיחת הממוצע על פני כל התאים עבור כל דגימה וסוג תא (צבירה פסאודו-בתפזורת). פלזמה-פרוטאומיקה נכללת כעוצמות מנורמלות וממוקדות חציון, ועבור נויטרופילים, אנו לוקחים את ספירת האקסון של המזהה המולקולרי הייחודי של UMI (UMI) מהפריים-סק. על ציטוקינים וערכים קליניים, לא יושם עיבוד מקדים קודם. פרטים נוספים על הפקת הנתונים (הניסיוניים) מפורטים בכתב היד המקביל11. מכיוון שהתוצאות המוצגות כאן מבוססות על שימוש בביאור האזימוט האוטומטי עבור סוגי תאים בנתוני scRNA-seq בהשוואה לאסטרטגיה מבוססת הסמן ששימשה בפרסום המוזכר, התוצאות המוצגות כאן דומות אך לא בדיוק זהות לאלה שהוצגו בפרסום. בכתב היד ניתן להראות כי אסטרטגיית הביאור מסוג התא אינה משנה את הדפוסים העיקריים ואת הפרשנויות הביולוגיות של הניתוח, אך שינויים קטנים בערכים המדויקים הנובעים מהמודל עשויים להשתנות. בסך הכל, נתוני הקלט היו מערך נתונים רב-ממדי מורכב הכולל נקודות זמן שונות ורמות מדידה שונות (תאים בודדים לעומת תפזורת) של יותר מ-10,000 תכונות שונות (גנים, חלבונים, ערכים קליניים). אסטרטגיה קפדנית של עיבוד מקדים והרמוניזציה של נתונים ואחריה ניתוח MOFA הוכחה ככלי שימושי ומהיר לחקר הנתונים ולחילוץ תוכנית חיסונית רלוונטית. כל נקודת זמן ושילוב מטופלים מטופלים כמדגם עצמאי בניתוח MOFA. כל סוג נתונים וסוג תא נחשבים לתצוגה נפרדת בניתוח MOFA.
פרוטוקול זה מספק הוראות להכנת נתוני הקלט עבור זרימת העבודה, ביצוע שלבי זרימת העבודה השונים, התאמה אישית של תצורות, פירוש הנתונים המתקבלים והתאמה איטרטיבית של התצורות בהתבסס על הפרשנויות. סקירה כללית של השלבים השונים של הפרוטוקול, ערכות נתוני הקלט הנדרשות בכל שלב, והנתונים ומערכי הנתונים המתקבלים ניתנת על-ידי סקירת זרימת העבודה הטכנית (איור 2).
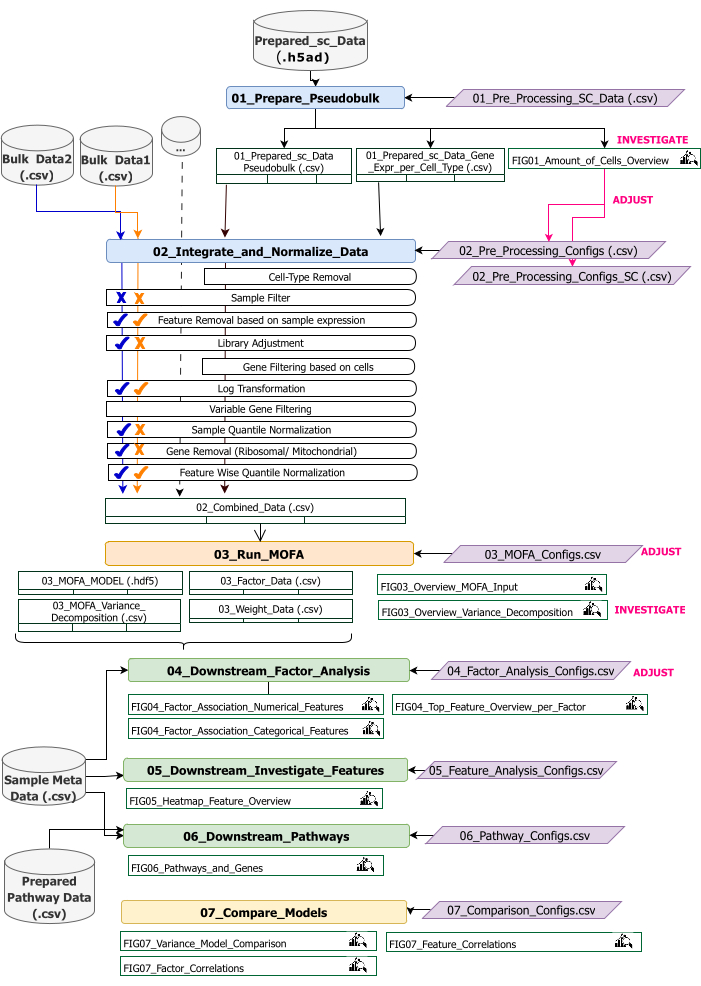
איור 2: מבט כולל על זרימת עבודה טכנית. מתווה זרימת העבודה לניתוח ערכת הנתונים multi-omics. האלמנטים השונים מודגשים על ידי צבעים וסמלים שונים. מחברות Jupyter השייכות לשלב עיבוד מראש והרמוניזציה של נתונים (1) צבועות בכחול. מחברות יופיטר השייכות לשלב 'מודל MOFA' (2) צבועות בכתום. מחברות יופיטר השייכות לשלב 'ניתוח במורד הזרם' (3) צבועות בירוק. מחברת יופיטר אחת שתשמש להשוואת התוצאות צבועה בצהוב. קבצי תצורה שבהם ניתן לשנות פרמטרים לביצוע זרימת העבודה מודגשים בסגול. ערכות נתונים של קלט הדרושות להפעלת זרימת העבודה מסומנות על-ידי סמל ערכת הנתונים ומודגשות באפור. כל פלטי האיור הנוצרים במהלך ביצוע זרימת העבודה מסומנים על-ידי סמל זכוכית המגדלת. ערכות נתונים שנוצרו במהלך ביצוע זרימת עבודה מסומנות כטבלאות. באופן כללי, זרימת העבודה מבוצעת ברצף: (1) עיבוד מקדים והרמוניזציה של נתונים מורכב משני שלבים: הדור הראשון של טבלה פסאודו-תפזורת המבוססת על נתוני הקלט scRNA-seq (01_Prepare_Pseudobulk) ולאחר מכן אינטגרציה ונורמליזציה של נתונים אלה יחד עם כל הקלטים האחרים ברמת המדגם (בתפזורת) (02_Integrate_and_Normalize_Data). במסגרת שלב זה באמצעות קבצי התצורה, ניתן להגדיר עבור כל ערכת נתונים בנפרד איזה משלבי העיבוד מראש והנורמליזציה שצוינו (לדוגמה, מסנן לדוגמה) יש ליישם. (2) 'מודל MOFA': מפעיל את מודל MOFA על הקלט שנוצר של השלב הראשון עם התצורות שצוינו בקובץ התצורה (03_MOFA_configs.csv) (3) 'ניתוח במורד הזרם': מורכב משלוש מחברות שונות שניתן להריץ בנפרד זו מזו כדי להפיק תובנות לגבי תוצאות MOFA שנוצרו ולשייך אותן למטא-נתונים לדוגמה (קובריאטים) שסופקו כקלט באמצעות קובץ 'מטא Data.csv לדוגמה'. (4) 'השוואת מודלים': הוא שלב נפרד קטן שניתן להשתמש בו כדי להשוות מודלים שונים שנוצרו בשלב 2. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
תהליך העבודה מורכב ממספר מחברות Jupyter שנכתבו ב-R וב-Python (ידע בשפת R ו-Python אינו נדרש להפעלת זרימת העבודה, אך עשוי להיות שימושי במקרה של הופעת שגיאות). בשלבים שונים של הפרוטוקול, פרמטרים משתנים באמצעות קבצי תצורה (קבצי '.csv' המכילים את הקידומת '_Configs' בשם). בתוך הפרוטוקול, אנו מתארים רק את הפרמטרים שיש לשנות החל מתצורת ברירת המחדל.
מספר פרמטרים אחרים עשויים להשתנות גם, למשל כדי להתאים אישית את העיבוד מראש. תיעוד של פרמטרים והסברים אלה ניתן בקובץ 'Documentation_Config_Parameter', הכלול במאגר שהורדת.
Protocol
1. הכנות: התקנה והתקנה טכניות
הערה: כדי להפעיל תוכנית זו, יש להתקין מראש את wget , git ו- Apptainer בהתקן. מדריך להתקנת Apptainer במערכות שונות (Linux, Windows, Mac) ניתן כאן: https://apptainer.org/docs/admin/main/installation.html. מידע התקנה על git ניתן למצוא כאן: https://git-scm.com/book/en/v2/Getting-Started-Installing-Git. בהתאם לגודל של ערכות נתוני הקלט השונות, מומלץ להפעיל את זרימת העבודה במחשב מתאים (16 CPU, 64GB זיכרון). ניתן לבצע בדיקת עשן עם הנתונים לדוגמה שסופקו במחשב המקומי. הוראות ופלטים צפויים מהפעלת הפרוטוקול על הנתונים לדוגמה ניתנים בקובץ משלים 1. עיין בקובץ וידאו משלים 1 לקבלת השלבים החשובים של הפרוטוקול המבוצעים במערך הנתונים המתואר לעיל.
- פתח את הקונסולה ובחר או צור תיקיה שבה יאוחסנו כל קוד הניתוח והפלטים. נווט אל התיקיה על-ידי הקלדת הפקודה: תקליטור path_to_folder במסוף.
- הורד או שכפל את מאגר הקוד מ- Github (https://github.com/heiniglab/mofa_workflow) או על ידי הקלדת https://github.com/heiniglab/mofa_workflow.git שיבוט git בחלון המסוף.
- הורד את התמונה המכילה את כל ההתקנות הנדרשות מ- Zenodo על ידי הקלדת wget https://zenodo.org/records/11192947/files/mofa_image.sif בחלון הטרמינל.
- צור תיקיה שבה יאוחסנו כל נתוני התוצאה על ידי הקלדת תוצאות mkdir בחלון המסוף.
- צור תיקיה שבה כל נתוני הקלט שישמשו בניתוח יתווספו על ידי הקלדת mkdir input_data בחלון המסוף.
- הפעל את הגורם המכיל שיתחיל הפעלת JupyterLab על-ידי הקלדת הפקודה הבאה במסוף: apptainer run mofa_image.sif. העתק את כתובת האתר המוחזרת על ידי הפקודה לדפדפן, אשר תפתח הפעלה של Jupyter-lab (מידע נוסף על Jupyter-lab ניתן למצוא בתיעוד התוכנה16).
הערה: כאשר זרימת העבודה מבוצעת באופן מקומי במחשב נישא, מומלץ להשתמש בפקודה apptainer exec mofa_image.sif jupyter-lab במקום זאת, אשר תחזיר ישירות כתובת מארח מקומי. במקרה שהקונטיינר מבוצע בסביבת מחשוב מקובצת, ייתכן שיהיה צורך להגדיר העברת יציאות, שניתן לבצע באמצעות ssh.
2. אתחול והכנת נתונים
- בהפעלה Jupyter-Lab, השתמש בתפריט הניווט בצד שמאל. נווט אל התיקיה input_data על ידי לחיצה כפולה על input_data.
- העתק את כל ערכות הנתונים שישמשו כקלט עבור הניתוח לספריית input_data באמצעות Drag&Drop. גרור את הקובץ מהתיקייה שבה הוא נמצא כעת ושחרר אותו בסשן Jupyter-lab באזור שמתחת לתיקייה input_data.
הערה: כל ערכות הנתונים צריכות להיות בתבנית .csv או .h5ad (במקרה של נתונים חד-תאיים). כל הקבצים .csv חייבים להכיל עמודת sample_id תואמת (יש להשתמש במזהים זהים בכל ערכות הנתונים). כל שאר העמודות ישמשו כתכונות. בתוך הקובץ h5ad, ביאור התא חייב להכיל שני מזהים המציינים את sample_id ואת cluster_id. אלה ישמשו לצבירה והתאמה. יש להמיר ערכות נתונים Omic בתבניות אחרות לתבנית .csv שצוינה לפני השימוש (איור 3). ערכות נתונים scRNA-seq שניתנו בתבנית .h5seurat עשויות להיות מומרות ל- .h5ad המפעיל את מחברת Jupyter: 00_Data_Conversion.ipynb. - נווט אל תיקיית התצורות על-ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על mofa_workflow התיקיות, קבצי ה- Script והתצורות. בתוך התיקייה, פתח את Data_configs.csv הקובץ על ידי לחיצה כפולה עליו.
- בעמודת הערך , הוסף את הנתיבים לתיקיות של התיקיות input_data (data_path) והתוצאות (result_path). הוסף שם שיתווסף כסיומת קובץ לכל הקבצים השמורים בעמודת הערך עבור configuration_name (פרוטוקול זה משמש MI_v1 [גירסה אוטם שריר הלב]) (איור 4).
- שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV בתפריט בחלק העליון.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית הסקריפטים על ידי לחיצה על סקריפטים. פתח את מחברת האתחול על-ידי לחיצה כפולה על 00_Configuration_Update.ipynb. הפעל את הסקריפט על-ידי לחיצה על לחצן הפעל מחדש את הליבה והפעל את כל התאים בחלק העליון, ולחיצה על הפעל מחדש בחלון המוקפץ (איור 5).
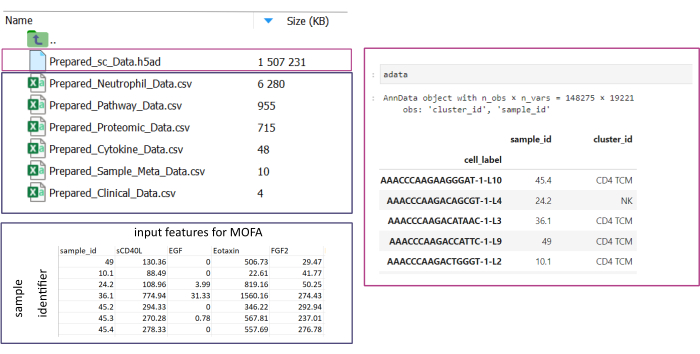
איור 3: קלט נתונים והגדרתם. לצורך ביצוע זרימת העבודה, יש לאחסן את כל הנתונים בתיקיית input_data שצוינה. עבור כל ערכת נתוני קלט יש לספק קובץ נפרד. נתונים של תא בודד צריכים להינתן כ- .h5ad המכיל ביאור תאים ב- cluster_id (הנובע למשל משלבי ביאור קודמים מסוג תא) ועמודת sample_id (המזהה באופן ייחודי כל מדגם נפרד שיש לנתח). כל שאר מערכי נתוני הקלט צריכים להינתן בתבנית '.csv', כולל עמודה אחת המציינת את sample_id (התאמה לעמודה המתאימה של הנתונים בתא בודד) ותכונות שישמשו בניתוח MOFA בכל העמודות האחרות. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
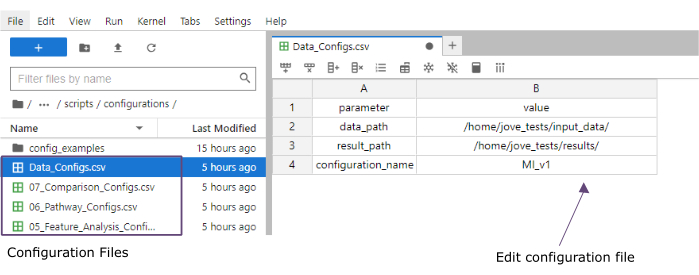
איור 4: קובצי תצורה של Jupyter-lab. במהלך ביצוע זרימת העבודה, שינויים בפרמטרים (למשל, התאמת אפשרויות סינון וכו ') מוגדרים באמצעות קבצי תצורה '.csv'. בתוך המאגר המשוכפל, נכללים קבצי תצורה המוגדרים כברירת מחדל עבור כל שלב. ניתן לערוך אותם ישירות במסוף מעבדת יופיטר, בדומה לגיליון אלקטרוני. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
איור 5: סקריפטים של Jupyter-notebooks. זרימת העבודה המלאה מורכבת מסדרה של מחברות Jupyter שיבוצעו ברצף לאחר שינוי קבצי התצורה המתאימים. על ידי לחיצה כפולה על מחברת יופיטר בצד שמאל, הקובץ המתאים ייפתח בצד ימין. ניתן להתחיל את הביצוע המלא של הקובץ עם הלחצן המודגש בחלק העליון. אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
3. עיבוד מקדים והרמוניזציה של נתונים
- עיבוד מקדים - המרת נתוני sc לפסאודו-תפזורת.
הערה: יש לבצע שלב זה רק אם נעשה שימוש בנתונים של תא בודד בניתוח.- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית התצורות על-ידי לחיצה כפולה על התצורה. פתח את 01_Preprocessing_SC_Data.csv הקובץ על-ידי לחיצה כפולה. בדוק את הערכים שמולאו באופן אוטומטי בקובץ, ובמידת הצורך, התאם את הערכים בעמודה data_name כך שיתאימו לשמות הקבצים של ערכות הנתונים של התא היחיד בתיקיית input_data שישמשו לניתוח.
הערה: כברירת מחדל, כל השמות של קבצי .h5ad בתיקיית נתוני הקלט יתווספו לקובץ התצורה בסקריפט האתחול. אם חלק ממערכי הנתונים לא ישמשו לניתוח, ניתן להסיר אותם כאן. - שמור שינויים שבוצעו על ידי לחיצה על קובץ > שמור קובץ CSV בתפריט בחלק העליון.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית הסקריפטים על ידי לחיצה על סקריפטים. פתח את המחברת 01_Prepare_Pseudobulk.ipynb על-ידי לחיצה כפולה עליה. הפעל את הסקריפט על ידי לחיצה על הפעל מחדש את הקרנל והפעל את כל התאים כפתור בחלק העליון, ולחיצה על הפעל מחדש בחלון המוקפץ.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית האיורים על-ידי לחיצה כפולה תחילה על איורים ולאחר מכן על 01_figures. פתח את העלילה החדשה שנוצרה FIG01_Amount_of_Cells_overview על ידי לחיצה כפולה עליה.
הערה: ביצוע המחברת עשוי להימשך מספר דקות. לאחר ביצוע מוצלח של המחברת, יופיע חלון מוקפץ וקובץ FIG01_Amount_of_Cells_Overview יעודכן על-ידי המחברת או ייווצר מחדש. העמודה השתנה לאחרונה יכולה לציין מתי הקובץ נוצר כדי להעריך אם זהו קובץ חדש או ישן. - חקור את העלילה כדי לזהות צבירי תאים עם מספר נמוך מאוד של תאים לדגימה. רשמו את שמות cluster_ids אלה כדי לא לכלול אותם בשלבים הבאים (איור 6).
- השתמש בתפריט הניווט בצד שמאל כדי לנווט חזרה לתיקיית התצורות על ידי לחיצה על ... ולאחר מכן לחיצה כפולה על תצורות. פתח את 02_Preprocessing_Configs_SC.csv הקובץ על ידי לחיצה כפולה עליו.
- בדוק את הערכים בעמודות configuration_name ו- data_name והתאם אותם במידת הצורך.
הערה: בתוך סקריפט האתחול, ערכים אלה ממולאים מראש עם כל השמות של קבצי .h5ad בתיקיית נתוני הקלט וערכי configuration_name שהוגדרו בקובץ Data_Configs.csv קודם לכן. במקרה שיש להוציא קבצים מהניתוח או להשתמש בסיומת אחרת לשמות קבצים, ניתן להתאים זאת כאן. - התאם את הערך בעמודה cell_type_exclusion והוסף את כל cluster_id שזוהו כדי לא לכלול בשלב הקודם כשהם מופרדים על-ידי ','.
- שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV בסרגל הניווט בחלק העליון.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית התצורות על-ידי לחיצה כפולה על התצורה. פתח את 01_Preprocessing_SC_Data.csv הקובץ על-ידי לחיצה כפולה. בדוק את הערכים שמולאו באופן אוטומטי בקובץ, ובמידת הצורך, התאם את הערכים בעמודה data_name כך שיתאימו לשמות הקבצים של ערכות הנתונים של התא היחיד בתיקיית input_data שישמשו לניתוח.
- עיבוד מקדים - הרמוניה ושילוב מקורות נתונים אחרים של omics.
- פתח את 02_Preprocessing_Configs.csv הקובץ על ידי לחיצה כפולה עליו והתאם את תצורת העיבוד מראש עבור כל אחד ממערכי הנתונים שייכללו ומאוחסנים בתיקיית data_input (שורה אחת לכל ערכת נתונים).
- בדוק את הערכים בעמודות configuration_name ו- data_name והתאם אותם במידת הצורך.
- התאם את שאר הפרמטרים בעמודות בהתאם, בהתאם לשלבי העיבוד מראש שיש ליישם.
הערה: ערכי ברירת מחדל נוספים עבור כל ערכת נתונים שנמצאת בתוך תיקיית input_dataset, אך אינה ספציפית לסוגי הנתונים הבודדים של הנתונים. לכן, יהיה צורך בהתאמות. תיעוד מפורט של פרמטרים ניתן בקובץ Documentation_Config_Parameter.doc . - שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית הסקריפטים על ידי לחיצה על סקריפטים. פתח את המחברת 02_Integrate_and_Normalized_Data_Sources.ipynb על-ידי לחיצה כפולה עליה. הפעל את הסקריפט על ידי לחיצה על הפעל מחדש את הקרנל והפעל את כל התאים כפתור בחלק העליון, ולחיצה על הפעל מחדש בחלון המוקפץ.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית 02_results שנוצרה על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על תוצאות ו - 02_results. בדוק אם הוא כולל את הקובץ 02_Combined_data_'configuration_name'_Integrated.csv המכיל את קובץ קלט הנתונים המשולב שעובד מראש.
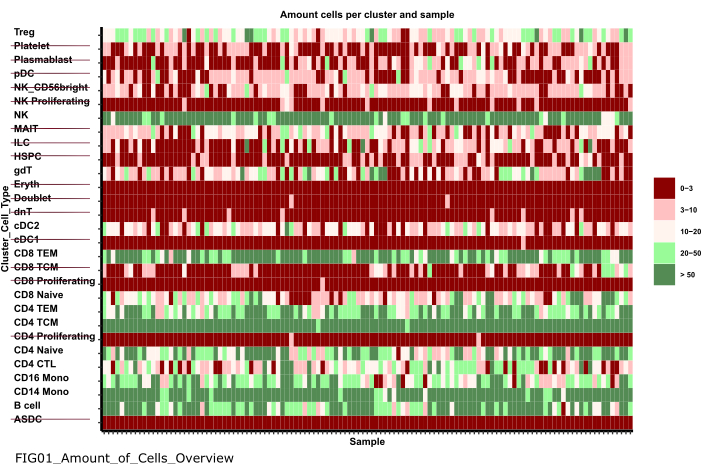
איור 6: עיבוד מקדים והרמוניזציה של נתונים. אחד התוצרים של שלב '01_Prepare_Pseudobulk' הוא העלילה 'Fig01_Amount_of_Cells_Overview'. כאן, עבור כל cluster_id (ציר y המציין את סוג התא משלבי ביאור קודמים של סוג תא), ניתן מספר התאים לכל דגימה ('sample_id'). בתוך התוצאות המוצגות, סוגי תאים עם כמות נמוכה של תאים לכל מדגם אינם נכללים בניתוח הבא (מסומן על ידי קו חוצה). אנא לחץ כאן כדי להציג גרסה גדולה יותר של איור זה.
4. הפעלת MOFA
- ב- Jupyter-Lab, השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית התצורות על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על mofa_workflow, ולאחר מכן לחיצה כפולה על סקריפטים ותצורות. פתח את 03_MOFA_Configs.csv הקובץ על ידי לחיצה כפולה עליו.
- בדוק את הערכים עבור העמודות configuration_name ו- mofa_result_name והתאם את הערכים אם יש להשתמש בשמות חלופיים.
הערה: mofa_result_name תצורף כסיומת קובץ לכל קובצי התוצאה שנוצרו בהתבסס על MOFA. ערך זה עשוי להיות שונה מערך configuration_name מכיוון שהגדרות MOFA שונות עשויות להיות מופעלות עם אותם נתוני קלט (פרוטוקול זה משתמש ב- MI_v1_MOFA). - הזן את כמות הגורמים שיש להעריך במודל MOFA (עמודה amount_of_factors ) והגדר אם יש להחיל שקלול ושינוי קנה מידה (עמודות weighting_of_views ו- scale_views ) באמצעות התאמת הערכים בקובץ.
- שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית הסקריפטים על ידי לחיצה על 'סקריפטים'. פתח את המחברת 03_Run_MOFA.ipynb על-ידי לחיצה כפולה על הקובץ. הפעל את הסקריפט על ידי לחיצה על הפעל מחדש ליבה והפעל את כל התאים כפתור בחלק העליון ולחיצה על הפעל מחדש בחלון המוקפץ.
- נווט אל התיקיה 03_figures על-ידי לחיצה כפולה על איורים ולאחר מכן 03_figures. פתחו את התרשים שנוצר FIG03_Overview_Variance_Decomposition_'mofa_result_name וחקרו את תוצאת המודל (איור 7A).
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית 03_results שנוצרה על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על התוצאות וה 03_results. בדוק אם הוא כולל את קובץ ערכי גורם המדגם 03_Factor_Data_'mofa_result_name'.csv ואת קובץ משקל גורם התכונה 03_Weight_Data_'mofa_result_name'.csv.
5. ניתוח במורד הזרם
- פרשנות גורמים.
- השתמש בתפריט ניווט בצד שמאל כדי לנווט לתיקיית input_data על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על input_data.
- הכן קובץ .csv (Prepared_Sample_Meta_Data.csv) המכיל את כל המטא-נתונים (קובריאטים) של הדגימות שינותחו בשיתוף עם הגורמים שנוצרו. העתק את הקובץ לתיקיית input_data באמצעות Drag&Drop שחרור הקובץ בסקירה הכללית של התיקיה input_data .
הערה: עליו להכיל את עמודת sample_id לצורך התאמתה לנתונים שהיו בשימוש בעבר ועמודות נוספות עבור כל תכונה שיש לנתח. - ב- Jupyter-Lab, השתמש בתפריט הניווט משמאל כדי לנווט חזרה לתיקיית התצורות על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על mofa_workflow, ולאחר מכן סקריפטים ותצורה. פתח את 04_Factor_Analysis.csv הקובץ על ידי לחיצה כפולה עליו.
- ודא שהערכים עבור configuration_name ו- mofa_result_name מכילים את שמות התצורה ותוצאות MOFA שינותחו בסקריפט והתאם אותם במידת הצורך.
- בעמודה numeric_covariates, הוסף את השם של כל העמודות המספריות בקובץ Prepared_Sample_Meta_Data.csv שייחקרו ביחס לגורמי MOFA המופרדים בפסיק (פרוטוקול זה משתמש ב- CRP,CK).
- בעמודה categorical_covariates', הוסף את השם של כל העמודות הקטגוריאליות בקובץ Prepared_Sample_Meta_Data.csv שייחקרו ביחס לגורמי MOFA המופרדים בפסיק (פרוטוקול זה משתמש במדידה).
- שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית 'סקריפטים' על ידי לחיצה על סקריפטים. פתח את המחברת 04_Downstream_Factor_Analysis.ipynb על-ידי לחיצה כפולה עליה. הפעל את הסקריפט על ידי לחיצה על הפעל מחדש ליבה והפעל את כל התאים כפתור בחלק העליון ולחיצה על הפעל מחדש בחלון הקופץ.
- השתמש בתפריט הניווט משמאל כדי לנווט לתיקייה 04_figures על-ידי לחיצה כפולה על איורים ולאחר מכן 04_figures. פתח את החלקות שנוצרו על ידי לחיצה כפולה עליהן ולחקור את הגורמים לדפוסים ואסוציאציות מעניינות: FIG04_Factor_Association_with_numeric_features_
'mofa_result_name.pdf (איור 7B). FIG04_Factor_Association_
with_categorical_features_'mofa_result_name.pdf (איור 7C). FIG04_Top_Feature_Overview_per_Factor _'mofa_result_name.pdf (איור 8A).
- ניתוח תכונות
- השתמש בתפריט הניווט בצד שמאל כדי לנווט חזרה לתיקיית התצורות על ידי לחיצה על ... ולאחר מכן לחיצה כפולה על תצורות. פתח את 05_Feature_Analysis_Configs.csv הקובץ על ידי לחיצה כפולה עליו.
- בדוק שהערכים עבור העמודות configuration_name ו- mofa_result_name תואמים לשמות התצורה ותוצאת MOFA שנוצרה שתשמש לניתוח במורד הזרם והתאם אותם במידת הצורך.
- בעמודה גורם , הוסף את הגורם שעבורו התכונות המובילות יותוו בסקריפט הבא.
- faceting_variable העמודה, הוסף שם עמודה של עמודה קטגורית ב- Prepared_Sample_Meta_Data.csv שישמש לקיבוץ הדגימות בהתוויית העניין (פרוטוקול זה משתמש במדידה)
- שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית הסקריפטים על ידי לחיצה על סקריפטים. פתח את המחברת 05_Downstream_Investigate_Features_Heatmap.ipynb על-ידי לחיצה כפולה עליה. הפעל את הסקריפט על ידי לחיצה על הפעל מחדש את הקרנל והפעל את כל התאים כפתור בחלק העליון, ולחיצה על הפעל מחדש בחלון המוקפץ.
- השתמש בתפריט הניווט משמאל כדי לנווט לתיקיה 05_figures על-ידי לחיצה כפולה תחילה על איורים ולאחר מכן על 05_figures. לפתוח ולחקור את העלילה שנוצרה FIG05_Heatmap_Feature_Overview__ 'mofa_result_name'.PDF על-ידי לחיצה כפולה על הקובץ (איור 8B).
הערה: בהתאם לכמות התכונות שיוצגו בעלילה, ייתכן שיהיה צורך להתאים את הפרמטרים plot_width ו- plot_height בתוך 05_Feature_Analysis_Configs.csv ולהפעיל מחדש את הסקריפט כדי לוודא שהכל מתאים לעלילה.
- ניתוח נתיבים
- השתמש בתפריט ניווט בצד שמאל כדי לנווט לתיקיית input_data על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על input_data.
- הכינו קובץ .csv (Prepared_Pathway_Data.csv) המכיל רשימת מסלולים שייבדקו להעשרה. העתק את הקובץ לתיקייה input_data באמצעות Drag&Drop שחרור הקובץ בסקירה הכללית של התיקיה input_data.
הערה: הוא צריך להכיל שלוש עמודות: מזהה ( מזהה ייחודי של המסלול), גן (הגנים שניתנו על ידי שם הגן שלהם (SYMBOL) השייכים למסלול, שורה אחת לכל גן), pathway_name (שם/תיאור טקסטואלי של המסלולים). - בהפעלת Jupyter-Lab, השתמש בתפריט הניווט משמאל כדי לנווט לתיקיית התצורות על ידי לחיצה על ... ולאחר מכן לחיצה כפולה על תצורות. פתח את 06_Pathway_Configs.csv הקובץ על ידי לחיצה כפולה עליו.
- בדוק את הערך עבור עמודת mofa_result_name וודא שהוא תואם לשם תוצאת MOFA שתיווצר שתשמש לחישוב העשרת המסלול.
- בדוק את הערך בעמודה סוגים והסר את הערכים בעמודה סוגים שאינם מכילים תכונות התואמות לעמודת הגן בקובץ Prepared_Pathway_Data.csv .
הערה: כברירת מחדל, כל התצוגות השונות שנעשה בהן שימוש במודל MOFA מתווספות לקובץ זה במהלך הפעלת זרימת העבודה. במקרה שיש תצוגות שאינן מכילות תכונות התואמות למסלול אחד לפחות, יש להסיר אותן; אחרת, הביצוע ייכשל. דוגמה לכך היא שקובץ המסלול מכיל רק ביאורי מסלול עבור גנים, אך קיימת תצוגה המכילה שמות חלבונים. - שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט הניווט כדי לנווט לתיקיית הסקריפטים על-ידי לחיצה על סקריפטים. פתח את המחברת 06_Downstream_Pathways.ipynb על-ידי לחיצה כפולה עליה. הפעל את הסקריפט על ידי לחיצה על הפעל מחדש ליבה והפעל את כל התאים כפתור בחלק העליון, ולחיצה על הפעל מחדש בחלון המוקפץ.
- השתמש בתפריט הניווט משמאל כדי לנווט לתיקייה 06_figures על-ידי לחיצה כפולה תחילה על איורים ולאחר מכן 06_figures. פתחו את העלילה שנוצרה FIG06_Pathways_and_Genes_'mofa_result_name על-ידי לחיצה כפולה עליה וחקרו את המסלולים החזותיים (איור 8C).
הערה: ניתן להגדיר את אופן בחירת הנתיבים החזותיים באמצעות קובץ התצורה. לפרטים נוספים, עיין בתיעוד הפרמטרים. - השתמש בתפריט הניווט בצד שמאל כדי לנווט לתיקיית 06_results שנוצרה על ידי לחיצה על סמל התיקיה ולאחר מכן לחיצה כפולה על התוצאות ו - 06_results. בדוק אם הוא כולל את הקובץ כולל המסלולים המועשרים 06_Pathway_enrichment__'mofa_result_name'.
6. השוואת תצורות וגרסאות שונות (איור משלים 1, תרשים משלים 2, תרשים משלים 3, תרשים משלים 4)
- כדי להשוות את ההשפעה של שימוש בפרמטרים/תצורות שונות לאורך זרימת העבודה, הפעל מחדש את מקטעים 3-5, שינוי הפרמטרים בקבצי התצורה ושימוש במזהי configuration_name ו-mofa_result_name שונים.
הערה: תוצאות חדשות יאוחסנו עם שמות אלה שישמשו להשוואה בין ריצות שונות. - ב- Jupyter-Lab, השתמש בתפריט הניווט משמאל כדי לנווט לתיקיית התצורות . פתח את 07_Comparison_Configs.csv הקובץ על ידי לחיצה כפולה עליו.
- בעמודה mofa_result_name , הוסף את השמות של כל ריצות MOFA הקודמות שיושוו (שורה אחת לכל שם/תצורה, לדוגמה, MI_v1_MOFA, MI_v2_MOFA).
- בעמודה compare_factors , הוסף את הגורמים שיושוו בין הדגמים. כברירת מחדל, הוא Factor1,Factor2,Factor3. (תרשים משלים 2A).
הערה: בסקריפט זה יושוו ערכי התכונות והגורמים של המודלים השונים על-ידי התאמה ביניהם. זה עובד רק עבור מודלים המבוססים על אותן דגימות (מסומנות על ידי sample_id) ועל אותה קבוצה של תכונות. במקרה שדוגמאות או תכונות אינן תואמות בין הגרסאות המושוות, הן לא ייכללו בהשוואה. - שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט בצד שמאל כדי לנווט לתיקיית הסקריפטים על ידי לחיצה על סקריפטים. פתח את המחברת 07_Compare_Models.ipynb על-ידי לחיצה כפולה עליה. הפעל את הסקריפט על ידי לחיצה על הליבה הפעל מחדש והפעל את כל התאים כפתור ולחיצה על הפעל מחדש בחלון המוקפץ.
- השתמש בתפריט מימין כדי לנווט אל התיקיה 06_figures על-ידי לחיצה כפולה תחילה על איורים ולאחר מכן 06_figures. פתח את החלקות שנוצרו על ידי לחיצה כפולה על הקבצים כדי לנתח את הדמיון בין הגרסאות השונות:
FIG07_Variance_Model_Comparison.pdf (איור משלים 2B)
FIG07_Factor_Correlations.pdf
(איור משלים 2C)
FIG07_Feature_Correlations.pdf
(איור משלים 3C)
7. הרחבת זרימת העבודה: הוספת פרמטרים ותצורות אחרות
הערה: מלבד הפרמטרים הניתנים כעת להגדרה בקבצי התצורה, ייתכן שייכללו התאמות אחרות בקוד או בפרמטרים אחרים. לדוגמה, מודל MOFA עצמו מציע מספר פרמטרי אימון אחרים17 שניתן לשנות ישירות בקוד או לבצע כוונון באמצעות קבצי התצורה. החלק הבא של הפרוטוקול יתאר דוגמה כיצד לעשות זאת עבור פרמטרים נוספים של אימון מודל MOFA. עבור חלק זה, נדרש ידע בתכנות R.
- ב- Jupyter-Lab, השתמש בתפריט הניווט משמאל כדי לנווט לתיקיית הסקריפטים . פתח את המחברת 03_Run_MOFA.ipynb על-ידי לחיצה כפולה עליה.
- לחץ על תוכן העניינים הכרטיסייה בצד שמאל ולאחר מכן נווט לתת-סעיף 4.3 הגדר אפשרויות אימון MOFA והפעל את אימון המודל על ידי לחיצה עליו. גלול מטה כדי לראות את הפלט המודפס של מודל MOFA של פרמטרים הניתנים להגדרה במחברת.
- בתוך הלולאה R for בקוד מתחת לכותרת, כל הנתונים, המודל ואפשרויות ההדרכה של MOFA מוגדרים. מתחת לשורה model_opts$num_factors = mofa_configs$amount_of_factors[i], הוסף שורה נוספת עם הקוד למטה
model_opts$likelihoods['data_type'] = 'פואסון'.
הערה: פעולה זו תשנה את ההתפלגות שהמודל מקבל כקלט עבור התצוגה שצוינה על-ידי השם data_type עבור כל הפעלות MOFA. בעת ציון פואסון עבור סוג נתונים, המודל יפעל רק כאשר התכונות עבור סוג נתונים זה הן מספרים שלמים (לדוגמה, ספירות קריאה מ- RNA-seq). כדי לקבל מידע נוסף על נתוני MOFA, הדרכה ואפשרויות מודל, ניתן גם לעיין בהדרכות ובתיעוד של MOFA17. - שמור את השינויים במחברת על ידי לחיצה על להציל כפתור בחלק העליון.
- כדי למסור פרמטרים חדשים באמצעות קבצי התצורה .csv , השתמש בניווט בצד שמאל כדי לנווט לתיקיית התצורות על ידי לחיצה כפולה על תצורות ופתח את 03_MOFA_Configs.csv הקובץ על ידי לחיצה כפולה.
- הוסף עמודה חדשה המציינת את שם הפרמטר, לדוגמה, number_iterations והזן ערך, לדוגמה, 1000. שמור את השינויים על ידי לחיצה על קובץ > שמור קובץ CSV.
- השתמש בתפריט הניווט כדי לנווט בתיקיית הסקריפטים על-ידי לחיצה על סקריפטים. פתח את המחברת 03_Run_Mofa.ipynb על-ידי לחיצה כפולה עליה. לחץ על תוכן העניינים הכרטיסייה בצד שמאל ולאחר מכן נווט לתת-סעיף 4.3 הגדר אפשרויות אימון MOFA והפעל את אימון המודל על ידי לחיצה עליו.
- החלף את השורה train_opts$maxiter = 50000 ב- train_opts$maxiter = mofa_configs$column_name[i] (כאשר שם העמודה שנוספה הוא number_of_iterations הוא train_opts$maxiter = mofa_configs$number_of_iterations[i])).
הערה: 03_MOFA_Configs.csv קובץ התצורה במחברת זו נקרא בתחילת המחברת (תת-סעיף: דרישות מוקדמות, תצורות ופרמטרים) כ- data.frame mofa_config בהפעלה ולכן, בשורת קוד זו, אובייקט זה והעמודה המתאימה שנוצרה לאחרונה מופנים. מכיוון שניתן להפעיל תצורות מרובות בו-זמנית, i מזהה את השורה של data.frame כאשר הערכת המודל מופעלת בלולאה עבור לאורך כל השורות השונות בקובץ .csv . עקרון הקריאה בקובץ התצורה בתחילת המחברת בסעיף 'תנאים מוקדמים, תצורות ופרמטרים' זהה עבור כל המחברות, וניתן לבצע שינויים נוספים כך. - שמור את השינויים במחברת על ידי לחיצה על להציל לחצן.
תוצאות
לאחר ביצוע מוצלח של זרימת העבודה, נוצרות מספר טבלאות ואיורים כפי שמצוין באיור 2. האיורים ימוקמו בתיקייה /figures (איור 6, איור 7, איור 8, איור משלים 1, איור משלים 2, איור משלים 3, <...
Discussion
עם הפרוטוקול המתואר, מוצגת זרימת עבודה מודולרית וניתנת להרחבה המבוססת על מחברת Jupyter, שניתן להשתמש בה כדי לחקור במהירות מערך נתונים רב-אומי מורכב. החלקים העיקריים של זרימת העבודה כוללים את החלק של עיבוד מראש והרמוניזציה של נתונים (המציע שלבים סטנדרטיים שונים לסינון ונורמ...
Disclosures
המחברים מצהירים כי אין ניגוד עניינים.
Acknowledgements
C.L. נתמך על ידי אגודת הלמהולץ תחת בית הספר למחקר משותף "Munich School for Data Science - MUDS".
Materials
| Name | Company | Catalog Number | Comments |
| Apptainer | NA | NA | https://apptainer.org/docs/admin/main/installation.html |
| Compute server or workstation or cloud (Linux, Mac or Windows environment). Depending on the size of the different input datasets we recommend running the workflow on a suitable machine (in our setting we use: 16 CPU, 64GB Memory) | Any manufacturer | 16 CPU, 64GB Memory | Large Memory is only required for the processing of the raw single cell data. After preprocessing the later analysis steps can also be performed on regular desktop or laptop computers |
| git | NA | NA | https://git-scm.com/book/en/v2/Getting-Started-Installing-Git |
| GitHub | GitHub | NA | https://github.com/heiniglab/mofa_workflow |
References
- Lähnemann, D., et al. Eleven grand challenges in single-cell data science. Genome Biol. 21 (1), 31 (2020).
- Colomé-Tatché, M., Theis, F. J. Statistical single cell multi-omics integration. Curr Opin Syst Biol. 7, 54-59 (2018).
- Hawe, J., Theis, F., Heinig, M. Inferring interaction networks from multi-omics data. Front Genet. 10, 535 (2019).
- Hawe, J. S., et al. Network reconstruction for trans acting genetic loci using multi-omics data and prior information. Genome Med. 14 (1), 125 (2022).
- Koh, H. W. L., Fermin, D., Vogel, C., Choi, K. P., Ewing, R. M., Choi, H. iOmicsPASS: network-based integration of multiomics data for predictive subnetwork discovery. NPJ Syst Biol Appl. 5, 22 (2019).
- Ogris, C., Hu, Y., Arloth, J., Müller, N. S. Versatile knowledge guided network inference method for prioritizing key regulatory factors in multi-omics data. Sci Rep. 11, 6806 (2021).
- Lee, C., vander Schaar, M. A variational information bottleneck approach to multi-omics data integration. Proceedings of The 24th International Conference on Artificial Intelligence and Statistics. 130, 1513-1521 (2021).
- Singh, A., et al. DIABLO: an integrative approach for identifying key molecular drivers from multi-omics assays. Bioinformatics. 35 (17), 3055-3062 (2019).
- Argelaguet, R., et al. Multi-omics factor analysis-a framework for unsupervised integration of multi-omics data sets. Mol Syst Biol. 14 (6), e8124 (2018).
- Cantini, L., et al. Benchmarking joint multi-omics dimensionality reduction approaches for the study of cancer. Nature Commun. 12 (1), 124 (2021).
- Pekayvaz, K., et al. Multiomic analyses uncover immunological signatures in acute and chronic coronary syndromes. Nature Medicine. 30 (6), 1696-1710 (2024).
- Swirski, F. K., Nahrendorf, M. Cardioimmunology: the immune system in cardiac homeostasis and disease. Nat Rev Immunol. 18 (12), 733-744 (2018).
- Janjic, A., et al. Prime-seq, efficient and powerful bulk RNA sequencing. Genome Biol. 23 (1), 88 (2022).
- Wolf, F. A., Angerer, P., Theis, F. J. SCANPY: large-scale single-cell gene expression data analysis. Genome Biol. 19 (1), 15 (2018).
- Cao, Y., et al. Integrated analysis of multimodal single-cell data with structural similarity. Nucleic Acids Res. 50 (21), e121 (2022).
- . Get Started - JupyterLab 4.1.0a4 documentation Available from: https://jupyterlab.readthedocs.io/en/latest/getting_started/overview.html (2024)
- . MOFA2: training a model in R Available from: https://raw.githack.com/bioFAM/MOFA2_tutorials/master/R_tutorials/getting_started_R.html (2020)
- De Silva, D., et al. Robust T cell activation requires an eIF3-driven burst in T cell receptor translation. eLife. 10, e74272 (2021).
- Li, G., Liang, X., Lotze, M. HMGB1: The central cytokine for all lymphoid cells. Front Immunol. 4, 68 (2013).
- Jassal, B., et al. The reactome pathway knowledgebase. Nucleic Acids Res. 48 (D1), D498-D503 (2020).
- Argelaguet, R., et al. MOFA+: a statistical framework for comprehensive integration of multimodal single-cell data. Genome Biol. 21 (1), 111 (2020).
- Velten, B., et al. Identifying temporal and spatial patterns of variation from multimodal data using MEFISTO. Nat Methods. 19 (2), 179-186 (2022).
- Qoku, A., Buettner, F. Encoding domain knowledge in multi-view latent variable models: A Bayesian approach with structured sparsity. Proceedings of The 26th International Conference on Artificial Intelligence and Statistics. 206, 11545-11562 (2022).
- Multi-Omics Factor Analysis. MOFA Available from: https://biofam.github.io/MOFA2/ (2024)
- Mitchel, J., et al. Tensor decomposition reveals coordinated multicellular patterns of transcriptional variation that distinguish and stratify disease individuals. bioRxiv. , (2023).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionThis article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved