A subscription to JoVE is required to view this content. Sign in or start your free trial.
Method Article
מיצוי RNA חד-חיה יחיד להשוואה של רמות תעתיק יחסיות באמצעות qRT-PCR בטרדיגראד Hypsibius exemplaris
In This Article
Summary
עבודה זו מציגה מיצוי RNA מהיר ושיטת השוואת רמת תעתיק לניתוח ביטוי גנים ב- Hypsibius exemplaris. באמצעות ליזה פיזית, שיטה זו בעלת תפוקה גבוהה דורשת טרדיגרד יחיד כחומר המוצא ומביאה לייצור חזק של cDNA לתגובת שרשרת פולימראז שעתוק הפוך כמותי (qRT-PCR).
Abstract
ה-Hypsibius exemplaris הוא אורגניזם מודל מתפתח הידוע ביכולתו לשרוד סביבות קיצוניות. כדי לחקור את המנגנונים המולקולריים והבסיס הגנטי של סובלנות קיצונית כזו, מחקרים רבים מסתמכים על ריצוף RNA (RNA-seq), שניתן לבצע על אוכלוסיות החל מקבוצות גדולות ועד לבעלי חיים בודדים. תגובת שרשרת פולימראז שעתוק הפוך (RT-PCR) והפרעות RNA (RNAi) משמשות לאחר מכן לאישור ממצאי RNA-seq ולהערכת הדרישות הגנטיות לגנים מועמדים, בהתאמה. מחקרים כאלה דורשים שיטה יעילה, מדויקת ומשתלמת למיצוי RNA ומדידת רמות תעתיק יחסיות על ידי RT-PCR כמותי (qRT-PCR). עבודה זו מציגה שיטת מיצוי RNA יעילה של צינור יחיד (STST) שלא רק מבודדת באופן אמין RNA מטרדיגראדים בודדים אלא גם מפחיתה את הזמן והעלות הנדרשים עבור כל מיצוי. שיטת מיצוי RNA זו מניבה כמויות של cDNA שניתן להשתמש בהן כדי להגביר ולזהות תעתיקים מרובים על ידי PCR כמותי (qRT-PCR). השיטה מאומתת על ידי ניתוח שינויים דינמיים בביטוי הגנים המקודדים שני חלבונים מווסתים הלם חום, חלבון הלם חום 70 β2 (HSP70 β2) וחלבון הלם חום 90α (HSP90α), מה שמאפשר להעריך את רמות הביטוי היחסיות שלהם אצל אנשים שנחשפו לחום באמצעות qRT-PCR. STST משלימה ביעילות את שיטות מיצוי ה-RNA הקיימות בתפזורת ובטרדיגראד יחיד, ומאפשרת בדיקה מהירה ובמחיר סביר של רמות שעתוק טרדיגראד בודדות על ידי qRT-PCR.
Introduction
טרדיגראדים הם בעלי חיים רב-תאיים קטנים הידועים ביכולתם לשרוד בתנאים קיצוניים שהם קטלניים לרוב צורות החיים האחרות1. לדוגמה, בעלי חיים אלה יכולים לשרוד כמעט פי 1000 ממינון הקרינה המייננת הקטלנית לבני אדם 2,3,4,5,6,7,8,9,10, ייבוש כמעט מוחלט 11,12,13,14,15, קפוא בהיעדר תוספת קריופרוטקטנטים 16,17,18, ובמצבם היבש, אפילו הריק של החלל 19,20. בשל יכולתם הייחודית לשרוד בסביבות קיצוניות, בעלי חיים אלה הפכו למודלים בסיסיים להבנת סובלנות קיצונית באורגניזמים מורכבים ורב-תאיים 1,21,22,23.
מניפולציה גנטית יציבה של בעלי חיים יוצאי דופן אלה, כולל טרנסגנזה ושינוי גנטי בקו הנבט, נותרה חמקמקה עד לאחרונה24,25. ככזה, רוב הניסויים לחשיפת מנגנונים מולקולריים של סובלנות קיצונית מבוצעים באמצעות פרופיל שעתוק באמצעות ריצוף RNA. קיימים מערכי נתונים רבים ואינפורמטיביים של ריצוף RNA עבור טרדיגראדים בתנאים קיצוניים שונים, החל מקרינה 8,9,26,27,28, מתח חום29, מתח הקפאה12 וייבוש 27,30,31,32,33 . חלק מהמחקרים הללו השתמשו בשיטות מיצוי וטיהור RNA בתפזורת כדי להאיר את ההבנה המולקולרית שלנו לגבי סובלנות קיצונית. עם זאת, מיצוי בתפזורת של תעתיקי RNA מבעלי חיים רבים מונע ניתוח של שונות בביטוי גנים בין פרטים, ובכך מפספס את העושר הפוטנציאלי של מערכי נתונים מעודנים יותר. חשוב לציין שמחקרים אלה מנתחים לעתים קרובות אוכלוסיות הטרוגניות של בעלי חיים הכוללים גם חיות ששורדות גורמי עקה סביבתיים וגם כאלה שלא. ככאלה, מחקרים אלה מבולבלים על ידי ממוצע נתוני ביטוי ממצבי תגובה מרובים ועשויים להיות שונים באופן דרמטי. כדי לטפל בבעיה זו, Arakawa et al., 201634 פיתחו צינור RNA-seq אלגנטי בעל קלט נמוך המיישם ערכת מיצוי RNA ואחריה שלב הגברה PCR ליניארי תוך שימוש ב-34,35,36 יחיד אומספר 30,37,38 בעלי חיים כקלט. מחקרים אלה היו בסיסיים להבנתנו את הסבילות לאקסטרים טרדיגראד22. מעניין לציין שפרוטוקול זה יושם גם על qRT-PCR תוך שימוש בשבע חיות כחומר התחלתי24.
ברוב אורגניזמי המודל, לאחר שזיהו מטרות פוטנציאליות באמצעות RNA-seq, qRT-PCR מבוצע לאחר מכן כדי לאשר שינויים בשעתוק שזוהו על ידי RNA-seq ולהעריך את מהלך זמן הביטוי של גנים מועמדים בצורה ברזולוציה גבוהה. כדי לבחון את תפקודם של גנים מזוהים, מחקרים כאלה מלווים לעתים קרובות בהפלה בתיווך RNAi של מטרות מולקולריות 39,40 וניתוח יכולת סובלנות קיצונית12,41. היעילות של כל נוקדאון RNAi מאושרת בדרך כלל על ידי qRT-PCR על ידי ניטור ישיר של הירידה בשפע התעתיק. עם זאת, RNAi הוא תהליך עתיר עבודה בטרדיגראדים שכן כל dsRNA חייב להיות מועבר באמצעות מיקרו-הזרקה ידנית שלאנשים 39,40. בשל אופי התפוקה הנמוך של אסטרטגיה זו, שיטת מיצוי RNA מהירה ובעלות נמוכה המותאמת ל-qRT-PCR מבעלי חיים בודדים תהיה בעלת ערך רב למחקר טרדיגראד. למרות שפותחו שיטות קודמות לחילוץ RNA מטרדיגראדים בודדים, פרוטוקולים אלה לא שילבו את מיצוי שלהם עם qRT-PCR, במקום זאת הסתמכו על שיטות מבוססות צפיפות אופטית 12,40,41. מונעים על ידי אתגרים אלה, ביקשנו לפתח פרוטוקול שמניב באופן אמין RNA בכמות ובאיכות שניתן להשתמש בו עבור qRT-PCR מ-H. exemplaris יחיד.
מותאם מפרוטוקול מיצוי RNA של בעל חיים יחיד שפותח עבור Caenorhabditis elegans42, STST מותאם ל-H. exemplaris. שיטת המיצוי מורכבת משישה שלבי הקפאה-הפשרה מהירים, המשבשים פיזית את הציפורן, ומאפשרים מיצוי RNA וסינתזת cDNA לאחר מכן. שיטת STST מפחיתה את זמן המיצוי ביותר מפי 24 בהשוואה לשיטות מיצוי RNA בתפזורת, כפי שתוארו על ידי Boothby, 201843, וב-30% בהשוואה לערכות מיצוי RNA בודדות, כפי שתוארו על ידי Arakawa et al., 201634. יתר על כן, מספר האינטראקציות בין דגימה לנסיין ירד מ-5 ל-1 בלבד בהשוואה לתכשירי ערכת מיצוי RNA, ובכך מפחית את הסיכון לזיהום על ידי ריבונוקלאזים אקסוגניים. בעת שאילתות עבור גנים בעלי ביטוי גבוה, שיטת STST מייצרת מספיק cDNA עבור 25 תגובות RT-PCR כמותיות לכל טרדיגראד בודד, הדורשת רק 1 מיקרוליטר מתוך נפח ה-cDNA הכולל של 25 מיקרוליטר לכל תגובה. עם זאת, יש לקבוע אמפירית את ריכוזי התבניות עבור תעתיקי שפע נמוכים יותר.
היעילות של שיטת STST לניתוח שינויים דינמיים בביטוי גנים הוערכה על ידי חקירת הביטוי הדיפרנציאלי של הגנים המקודדים חלבון הלם חום-90α (HSP90α) וחלבון הלם חום 70β2 (HSP70β2) בתגובה להלם חום לטווח קצר ב-35 מעלות צלזיוס למשך 20 דקות. גם HSP70β2 וגם HSP90α ברוב האורגניזמים האיקריוטיים מווסתים במהירות בעקבות חשיפה קצרת טווח להלם חום (20 דקות)42. ניתוח ב-H. exemplaris גילה כי גם ה-RNA המקודד HSP70β2 וגם HSP90α שהופקו מטרדיגרדים בודדים שטופלו בחום הראו עלייה מובהקת סטטיסטית בביטוי לאחר חשיפה לחום לטווח קצר. ממצאים אלה מדגימים כי ניתן להשתמש בפרוטוקול STST כדי לנתח שינויים דינמיים בביטוי גנים בבעלי חיים בודדים לאורך זמן.
שיטת מיצוי STST צריכה להשלים שיטות ניסיוניות קיימות כגון RNA-seq על ידי הקלה על מיצוי RNA מהיר וזול והשוואה לאחר מכן של רמות התעתיק על ידי qRT-PCR. שיטה זו תהיה בעלת ערך גם להערכת היעילות והחדירה של RNAi באנשים המוזרקים ידנית בצורה כמותית יותר מאשר צפיפות אופטית בלבד. לבסוף, בשל המבנים הקוטיקולריים והמאפיינים הפיזיים הדומים שלהם, סביר להניח ששיטה זו תהיה יעילה גם לניתוח ביטוי גנים במינים אחרים של טרדיגראד44.
Protocol
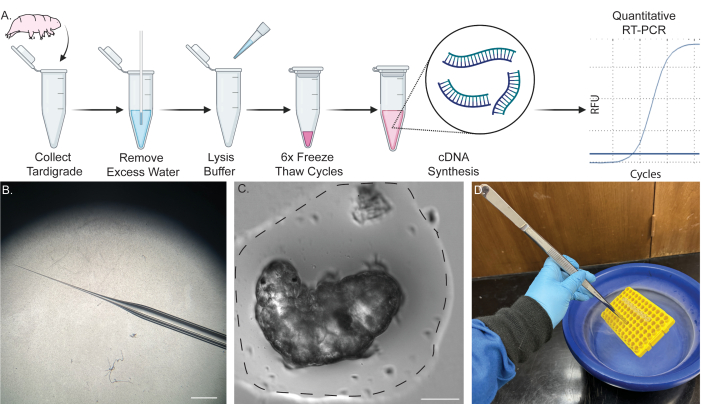
איור 1: צינור חד-שפופרת עבור מיצוי RNA מטרדיגראד יחיד. (A) סכמה המציגה את הפרוטוקול עבור מיצוי RNA מטרדיגראד יחיד, כולל שישה מחזורי הקפאה-הפשרה וסינתזת cDNA לאחר מכן. לאחר מכן ניתן להשתמש בדגימות עבור RT-PCR ו-qRT-PCR. (B) תמונה של מיקרו-פיפטה מתחדדת המשמשת לסילוק מים. סרגל קנה מידה: 2 מ"מ. (C) תמונת שדה בהיר של טרדיגראד בנפח קטן של מים (קו מקווקו). הסרת רוב המים במידה המוצגת נדרשת למיצוי מוצלח ומונעת דילול של מאגר הליזיס. סרגל קנה מידה: 50 מיקרומטר. (D) תמונה שמראה טבילה של דגימות בחנקן נוזלי באמצעות מלקחיים ארוכים כדי להקפיא ולהפשיר במהירות את הדגימות בבטחה. חלק מהתוכן נוצר ב-BioRender. קירק, מ. (2022) BioRender.com/d93s511 אנא לחץ כאן לצפייה בגרסה גדולה יותר של איור זה.
הערה: איור 1A מציג סכימה של ההליך. לנהלים מפורטים של טרדיגראד ותרבות אצות, עיין בדוחות שפורסמו בעבר 45,46,47.
1. עיקור מי מעיין
- יוצקים 2 ליטר מי מעיין מכד מים בנפח 5 ליטר (ראה טבלת חומרים לפרטים) לבקבוק זכוכית 2 ליטר בטוח לחיטוי.
- הניחו את הפקק על הבקבוק הבטוח לחיטוי ואטמו עם כמות קטנה של סרט חיטוי. אין להדק את הבקבוק; הניחו את הכובע מעל.
- חיטוי מי המעיין למשך 50 דקות במחזור רטוב ללא שלב ייבוש.
- הניחו למים להגיע לטמפרטורת החדר (RT), ואטמו היטב את המכסה לפני אחסונם ב-RT.
2. משיכת מיקרופיפטה מזכוכית (עם מושך פיפטה)
- אבטח מיקרופיפטה מזכוכית (OD: 1 מ"מ, תעודת זהות: 0.58 מ"מ, אורך: 10 ס"מ) על חולץ מיקרופיפטה. הימנע ממגע עם חוט החימום, מכיוון שהדבר ישנה את צורת הפיפטה ויפגע בחוט.
- קבע את משיכת הפיפטה באופן אמפירי עבור כל חוט נימה ומושך פיפטה. עם זאת, כדי לשמש כנקודת התחלה לאופטימיזציה, השתמש ב-78 מעלות צלזיוס ובצעד משיכה יחיד של משקל משיכה של 182.2 גרם.
- אפשרו לחוט להתחמם וכוח הכבידה להפריד את המיקרופיפטה מזכוכית לשתי מיקרופיפטות זכוכית עם נקודות חדות (איור 1B).
- אחסן את המיקרופיפטות הזכוכית המשוכות הללו בצלחת פטרי סגורה בגודל 100 מ"מ עם שעווה או חימר כדי להחזיק אותן במקומם ולמנוע מהקצוות החדים להישבר.
3. משיכת מיקרופיפטה מזכוכית (ללא מושך פיפטה)
- הדליקו מבער Bunsen או מקור להבה מבוקר אחר על הגדרה נמוכה.
- קח מיקרופיפטה מזכוכית עם קצה אחד בכל יד.
- החזק את מרכז המיקרופיפטה מזכוכית מעל הלהבה עד שהכוס מתחילה להימס. לאחר מכן, משוך במהירות את שני הקצוות זה מזה. זה ייצור שני טיפים חדים עדינים מאוד.
- שוברים קלות את הקצה בעזרת זוג מלקחיים עדינים סטריליים.
- אחסן את המיקרופיפטות הזכוכית המשוכות הללו בצלחת פטרי סגורה בגודל 100 מ"מ עם שעווה או חימר כדי להחזיק אותן במקומם ולמנוע מהקצוות החדים להישבר.
4. מיצוי RNA
- השג 0.5 ליטר חנקן נוזלי במיכל בטוח לקריו.
זהירות: חנקן נוזלי הוא קריוגני ועלול לגרום לכוויות אם הוא נחשף לעור או לעיניים. בעת הטיפול, השתמש בביגוד מגן, משקפי התזה, כפפות ניטריל, כפפות קריו, מעיל מעבדה ונעליים סגורות. ודא שהמיכל בטוח לחנקן נוזלי לפני הובלת הנוזל. שימוש באמבטיית קרח יבש באתנול לשלב זה עשוי להיות אפשרי גם כן. - הכינו תערובת מאסטר לסינתזת cDNA: תמיסה של 10 מיקרוליטר המכילה 1 מיקרוליטר של פריימר הקסמר אקראי, 2 מיקרוליטר של DNase, 4 מיקרוליטר של מאגר 5x RT, 1 מיקרוליטר תערובת אנזימים, 1 מיקרוליטר של H2O ו-1 מיקרוליטר של 10 מ"מ dNTPs. אחסן תמיסה זו על קרח.
- הכן מאגר ליזה טרדיגרד (5 מ"מ טריס (pH = 8), 0.5% (v/v) חומר ניקוי 1, 0.5% (v/v) חומר ניקוי 2, 0.25 מ"מ EDTA במים סטריליים ללא נוקלאז).
הערה: ניתן לאחסן תמיסה זו על הספסל למשך 6 חודשים. עם זאת, שמור על סטריליות והימנע ממקורות מזהמים RNAse פוטנציאליים. - יש מספיק מאגר ליזה למיצויים (2 מיקרוליטר / טרדיגראד).
- הוסף מעכב RNAse לתמיסת מאגר הליזיס של טרדיגרד לריכוז סופי של 4 U/μL.
- מערבולת וסובב את התמיסה ב-RT על צנטריפוגה על ספסל במהירות של 2000 x g למשך 5 שניות לפני אחסון התמיסה על קרח.
- הסר כמה טרדיגרדים הדרושים לניסוי מהתרבית באמצעות פיפטה P1000 סטרילית עם קצה מסנן והנח אותם בצלחת פטרי סטרילית 35 מ"מ.
הערה: ניתן לעבד כל מספר של טרדיגרדים בדרך זו. בדרך כלל, שלושה טרדיגרדים לכל מצב מעובדים למיצוי. - שטפו את הטרדיגרדים שלוש פעמים, באמצעות 1 מ"ל של מי מעיין סטריליים שעברו חיטוי ופיפטה P1000 סטרילית עם קצה מסנן. פיפטינג איטי שלהם למעלה ולמטה עוזר להסיר מזהמי אצות.
- באמצעות מיקרוסקופ מנתח בהגדלה של פי 25 עד פי 50, העבירו טרדיגראד בודד מתרבית שטיפה זו לצלחת פטרי סטרילית חדשה בגודל 35 מ"מ באמצעות פיפטה P10 סטרילית עם קצה מסנן.
- השתמש בפיפטה P200 סטרילית עם קצה מסנן כדי לשטוף את הטרדיגראד הבודד ב-100 מיקרוליטר של מים סטריליים נטולי נוקלאז.
הערה: שלב כביסה זה משמש להסרה נוספת של מזהמים, כולל ריבונוקלאזים. - העבירו את הטרדיגרד השטוף לתחתית צינור PCR נקי וסטרילי ב-1-2 מיקרוליטר של מים סטריליים נטולי נוקלאז באמצעות פיפטת P10 סטרילית עם קצה מסנן, תוך הקפדה על כך שהטרדיגראד לא נדבק לצד הקצה.
- דמיין את הטרדיגראד תחת מיקרוסקופ מנתח בהגדלה של פי 25.
- כדי להקל על פינוי מים של מים, שברו קלות את קצה המיקרופיפטה הזכוכית הנמשכת מחוץ לצינור. ודא שהקדח גדול מספיק כדי למשוך את המים אך לא את הטרדיגראד.
- באמצעות פעולה נימית של מיקרופיפטת זכוכית משוכה, הסר מים עד שהחיה מוקפת בבועת מים קטנה בקוטר של כשני אורכי טרדיגראד.
- עקוב אחר תהליך פינוי המים באמצעות היקף החיתוך כדי לוודא שמפלס המים מתאים והטרדיגראד נשאר לחות.
הערה: איור 1C מציע דוגמה לכמות המים שיש להסיר. זהו צעד קריטי. בועת מים קטנה תקיף את הטרדיגראד כדי למנוע את התייבשותו, אך יש להסיר כמה שיותר עודפי מים כדי למנוע דילול של מאגר הליזיס. לדוגמא של מפלסי המים הנותרים, עיין באיור 1C. - מיד לאחר הסרת המים, הוסף 2 מיקרוליטר של מאגר ליזה טרדיגרד לתחתית הצינור, מערבולת קצרה, וצנטריפוגה את הצינור ב-RT למשך 5 שניות ב-2000 x גרם על צנטריפוגה על השולחן.
- הנח מיד את הדגימות המכילות את הטרדיגרדים לתוך מתלה צינור PCR וודא שהן מוחזקות היטב על ידי המתלה.
- אחזו במדף באמצעות זוג מלקחיים ארוכים וגסים וטבלו בעדינות את המדף המכיל את הדגימות לתוך החנקן הנוזלי עד להקפאה מלאה (איור 1D).
- הסר את המתלה מהחנקן הנוזלי והניח אותו מיד על קרח. אפשר לדגימה להפשיר (לוקח ~45 שניות עד דקה אחת). עקוב אחר הדגימה כל 15 שניות על ידי הוצאתה מהקרח ובדיקתה באופן גלוי. לאחר שהדגימה שקופה לעין, עברו לשלב הבא.
- חזור על שלבים 4.18-4.19 חמש פעמים נוספות. בסך הכל נדרשים שישה מחזורי הקפאה-הפשרה לליזה ומיצוי מקסימליים (איור 2A,B).
- לאחר השלמת ההקפאה-הפשרה, הניחו דגימות על קרח והתקדמו מיד לשלב הבא. אין להקפיא את הדגימות בשלב זה לאחסון, מכיוון שהדבר יפחית את ה-RNA הזמין להכנת cDNA.
5. סינתזת cDNA
- הוסף 2 מיקרוליטר של תערובת מאסטר לסינתזת cDNA לצינור ה-PCR המכיל ליזאט טרדיגראד. העבירו בקצרה את הצינור וסובבו אותו ב-RT ב-2000 x גרם למשך 5 שניות עם צנטריפוגה שולחנית לפני החלפת הדגימות על קרח.
- מניחים את הדגימות בתרמו-סייקלר ודוגרים בטמפרטורה של 25 מעלות צלזיוס למשך 10 דקות כדי לחסל פריימרים, בטמפרטורה של 55 מעלות צלזיוס למשך 30 דקות לביצוע שעתוק הפוך, ולבסוף, אנזימים משביתי חום בטמפרטורה של 85 מעלות צלזיוס למשך 5 דקות.
- לאחר הדגירה, הניחו מיד את הצינור על קרח ודללו את הדגימה לנפח כולל של 25 מיקרוליטר על ידי הוספת 21 מיקרוליטר של מים סטריליים ללא נוקלאז. עבור תמלילים עם מספר עותק נמוך, שנה את שלב הדילול הזה כפי שנקבע אמפירית.
6. qPCR
- קבע את טמפרטורת החישול של ערכת הפריימר באמצעות סך ה-RNA שהוכן מכמויות גדולות יותר של טרדיגרדים, למשל, שיטת המיצוי בתפזורת שהוצגה ב-Boothby, 201843.
- הפעל שיפוע טמפרטורת PCR כדי לקבוע את טמפרטורת החישול האופטימלית לפני הפעלת qRT-PCR (עבור כל הגדרות ה-PCR המשמשות בפרוטוקול זה, עיין בטבלה 1 ובטבלה 2).
- להפשיר צינור אחד של צבע מחוון סופר לערבב על קרח ולבודד מהאור. הניחו צלחת qPCR של 96 בארות על קרח והניחו 5 מיקרוליטר של תערובת סופר, 2 מיקרוליטר מים, 1 מיקרוליטר מכל פריימר (10 מיקרומטר) ו-1 מיקרוליטר של תוצר cDNA במספר הבארות הרצויות.
- אטום את לוחית ה-PCR עם אטם צלחת והפעל את ה-qRT-PCR באמצעות טמפרטורת חישול המתאימה לערכת הפריימר (לכל הגדרות qRT-PCR המשמשות במאמר זה, עיין בטבלה 3).
7. כימות ופרשנות תוצאות
- השווה את התוצאות כמותית לגן ביקורת אחד או יותר, שביטויו צפוי להיות קבוע בתנאים שנכפו. במחקר זה נעשה שימוש בגן האקטין.
- השג והשווה את ערכי Ct או סף המחזור עבור כל באר לערכי Ct של תגובות הגן של משק הבית הבקרה. חשב את שינוי הקיפול בביטוי הגנים באמצעות המשוואה הבאה:
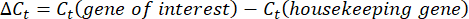
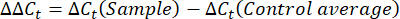
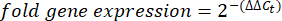
הערה: ביטוי גן קיפול משורטט עבור כל תעתיק וטרדיגראד כ-2-(ΔΔCt)48. - כדי לקבל הערכה גסה של מספר התעתיק מערךC t, השתמש במשוואה הבאה:
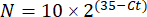
כאשר N הוא מספר התעתיקים, ו-2 הוא יעילות ה-PCR המשוערת או עליית הקיפול בקרינה למחזור של PCR48.
תוצאות
פיתוח ואופטימיזציה של מיצוי RNA סינגל-טרדיגראד
תוך התאמת הפרוטוקול מ-Ly et al., 201542 עבור מיצוי RNA בטרדיגראדים, מערכת STST ממוטבת כדי למקסם את כמות ואיכות התכשיר (איור 1A). RT-PCR בוצע עבור תמלילי אקטין, וכימת תפוקת התעתיק על ידי הגברת אזור של 527 ...
Discussion
מחקר זה מציג שיטה יעילה למיצוי RNA עבור qRT-PCR חד-טרדיגראד. השוואה ישירה של מתודולוגיית STST לערכת מיצוי RNA בודדת קיימת גילתה כי מיצוי RNA STST מניב כמויות גבוהות פי >200 של תעתיקי RNA אקטין מפחית את העלות לפחות מדולר אחד לדגימה, ומפחית את הזמן הנדרש למיצוי ב-30%. כדי ליישם STST על שאלה ביו?...
Disclosures
המחברים מצהירים שאין ניגודי אינטרסים לחשוף.
Acknowledgements
אנו רוצים להכיר במלגת NIH רות קירשטיין # 5F32AG081056-02 ובמלגת הפוסט-דוקטורט של ארט פישר, שתמכה בד"ר מולי ג'יי קירק, במלגת משפחת קרואו, שתמכה בצ'אומינג שו, ובמענק הסנאט האקדמי של אוניברסיטת קליפורניה, סנטה ברברה, ומענקי NIH #R01GM143771 ו-#2R01HD081266, שתמכו במאמצי המחקר הללו. המחברים מכירים גם בשימוש במעבדה לננו-מבנים ביולוגיים במכון הננו-מערכות של קליפורניה, הנתמכת על ידי אוניברסיטת קליפורניה, סנטה ברברה, ואוניברסיטת קליפורניה, משרד הנשיא.
Materials
| Name | Company | Catalog Number | Comments |
| 10 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-401 | Refered to as Sterile Filter-Tipped P 10 Pipette Tips |
| 1000 µL Premium Pipet Tips, Low Binding, Racked, Sterile | Genesee Scientific | 23-165RS | Refered to as Sterile Filter-Tipped P 1000 Pipette Tips |
| 200 µL Premium Barrier Tips Low Binding, Racked, Sterile | Genesee Scientific | 23-412 | Refered to as Sterile Filter-Tipped P 200 Pipette Tips |
| 4 Star Straight Strong Medium Point Tweezer | Excelta | 00-SA-DC | Refered to as Long forceps |
| 96-Well PCR Rack with Lid Assorted, 5 Racks/Unit | Genesee Scientific | 27-202A | Refered to as PCR Rack |
| Andwin Scientific 3M LEAD FREE AUTOCLAVE TAPE 1" | Thermo Fisher Scientific | NC0802040 | Refered to as Autoclave Tape |
| Autoclave Tape | Thermo Fisher Scientific | AB1170 | Refered to as PCR Plate Seals |
| Benchling v8 | Benchling | N/A | Refered to as Benchling |
| BioRadHard-Shell 96-Well PCR Plate | BioRad | HSS9641 | Refered to as PCR Plate |
| BULWARK FR Lab Coat: | Grainger | 26CF64 | Refered to as Lab Coat |
| C1000 Touch Bio-rad Thermocycler | BioRad | 1851148 | Refered to as Thermocycler |
| C1000 Touch Bio-rad Thermocycler with CFX Optics Module | BioRad | 1845097 | Refered to as qPCR thermocycler |
| Chloroccoccum hypnosporum. | Carolina | 152091 | Refered to as Algae |
| Corning PYREX Reusable Media Storage Bottles | Thermo Fisher Scientific | 06-414-1E | Refered to as 2 L Autoclave-safe Glass Bottle |
| Daigger & Company Vortex-Genie 2 Laboratory Mixer | Thermo Fisher Scientific | 3030A | Refered to as Vortexer |
| Direct-zol Micro Prep | Zymo Research | R2060 | Refered to as RNA extraction kit |
| Dumont 5 Biology Tweezers | Fine Science Tools | 11254-20 | Refered to as Fine Forceps |
| EDTA | Fisher Scientific | S311-500 | Refered to as EDTA |
| FIJI v 2.14.0/1.54f | ImageJ, | N/A | Refered to as FIJI/ImageJ |
| Filament for pippette Puller | Tritech Research | PC-10H | Refered to as Filament |
| Fisherbrand Economy Impact Goggles | Fisher Scientific | 19-181-501 | Refered to as Splash Goggles |
| Glass Micropipette O.D. 1mm ID 0.58, Length 10 cm | TriTech Research | GD-1 | Reffered to as glass micropipette |
| Hypsibius exemplaris Z151 Strain | Carolina | 133960 | Refered to as Tardigrades or H. exemplaris |
| Liquid Nitrogen Dewar 1 L | Agar Scientific | AGB7475 | Refered to as Cryo-safe container |
| Maxima H Minus First Strand cDNA Synthesis Kit | Thermo Fisher Scientific | K1651 | Refered to as cDNA Synthesis Master Mix |
| Narishige Dual-Stage Glass Micropipette Puller | Tritech Research | PC-10 | Refered to as micropipette puller |
| Nitrile Gloves | Fisher Scientific | 17-000-314 | Refered to as Nitrile Gloves |
| PETRI DISH, PS, 35/10 mm, WITH VENTS | Grenier | 627102 | Refered to as 35 mm Petri dish |
| PIPETMAN P10, 1–10 µL, Metal Ejector | Gilson | F144055M | Refered to as P 10 Pipette |
| PIPETMAN P1000, 100–1000 µL, Metal Ejector | Gilson | F144059M | Refered to as P 1000 Pipette |
| PIPETMAN P200, 20–200 µL, Metal Ejector | Gilson | F144058M | Refered to as P 200 Pipette |
| Pound This 4-Color Modeling Clay | American Science Surplus | 96517P001 | Refered to as Clay |
| Prism v10.0 | GraphPad | N/A | Refered to a Prism |
| RNAse-Free, 8 Strip 0.2 mL PCR Tubes with caps | Invitrogen | AM12230 | Refered to as Sterile PCR Tube |
| RNasin Ribonuclease Inhibitor | Promega | N2111 | Refered to as RNAse inhibitor |
| Spring water | Nestle Pure Life | 44221229 | Refered to as Spring Water |
| SsoAdvanced Universal SYBR Green Supermix | BIO RAD | 1725271 | Refered to as Indicator Dye Super mix |
| Stereo-Microscope System w/optics and illumination | TriTech Research | SMT1 | Refered to as Dissecting Microscope |
| Supertek Scientific Tirrill Burners | Thermo Fisher Scientific | S09572B | Refered to as Bunsen Burner |
| Table Top Centrifuge | Qualitron | DW-41-115-NEW | Refered to as Table Top Centrifuge |
| Tempshield Cryo-Gloves | Fisher Scientific | 11-394-305 | Refered to as Cryo Gloves |
| Thermo Scientific Nunc Petri Dishes | Thermo Fisher Scientific | 08-757-099 | Refered to as 100 mm Petri dish |
| Tris base | Fisher Scientific | T395-500 | Refered to as Tris or Tris Base |
| Triton X-100 | Fluka | 93443 | Refered to as Detergent 1 |
| TWEEN 20 | Sigma aldrich | P1379-500 | Refered to as Detergent 2 |
| Water - PCR/RT-PCR certified, nuclease-free | Growcells | PCPW-0500 | Refered to as Sterile Nuclease Free Water |
References
- Møbjerg, N., Neves, R. C. New insights into survival strategies of tardigrades. Comp Biochem Physiol Part A Mol Integr Physiol. 254, 110890 (2021).
- Jönsson, K. I., Harms-Ringdahl, M., Torudd, J. Radiation tolerance in the eutardigrade Richtersius coronifer. Int J Radiat Biol. 81 (9), 649-656 (2005).
- Horikawa, D. D., et al. Radiation tolerance in the tardigrade Milnesium tardigradum. Int J Radiat Biol. 82 (12), 843-848 (2006).
- Bruckbauer, S. T., Cox, M. M. Experimental evolution of extremophile resistance to ionizing radiation. Trends Genet. 37 (9), 830-845 (2021).
- Jönsson, K. I., Hygum, T. L., Andersen, K. N., Clausen, L. K. B., Møbjerg, N. Tolerance to gamma radiation in the marine heterotardigrade, Echiniscoides sigismundi. PLoS One. 11 (12), e0168884 (2016).
- Jönsson, K. I. Radiation tolerance in tardigrades: Current knowledge and potential applications in medicine. Cancers (Basel). 11 (9), 1333 (2019).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Clark-Hachtel, C. M., et al. The tardigrade Hypsibius exemplaris dramatically upregulates DNA repair pathway genes in response to ionizing radiation. Curr Biol. 34 (9), 1819-1830.e6 (2024).
- Anoud, M., et al. Comparative transcriptomics reveal a novel tardigrade specific DNA binding protein induced in response to ionizing radiation. Elife. 13, RP92621 (2024).
- Jönsson, K. I., Schill, R. O. Induction of Hsp70 by desiccation, ionising radiation and heat-shock in the eutardigrade Richtersius coronifer. Comp Biochem Physiol B Biochem Mol Biol. 146 (4), 456-460 (2007).
- Boothby, T. C. Desiccation of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 871-873 (2018).
- Boothby, T. C., et al. Tardigrades use intrinsically disordered proteins to survive desiccation. Mol Cell. 65 (6), 975-984.e5 (2017).
- Horikawa, D. D., Higashi, S. Desiccation tolerance of the tardigrade Milnesium tardigradum collected in Sapporo, Japan, and Bogor, Indonesia. Zoolog Sci. 21 (8), 813-816 (2004).
- Halberg, K. A., Jørgensen, A., Møbjerg, N. Desiccation tolerance in the tardigrade Richtersius coronifer relies on muscle mediated structural reorganization. PLoS One. 8 (12), e3330 (2013).
- Sørensen-Hygum, T. L., Stuart, R. M., Jørgensen, A., Møbjerg, N. Modelling extreme desiccation tolerance in a marine tardigrade. Sci Rep. 8 (1), 11495 (2018).
- Lyons, A. M., Roberts, K. T., Williams, C. M. Survival of tardigrades (Hypsibius exemplaris) to subzero temperatures depends on exposure intensity, duration, and ice-nucleation - as shown by large-scale mortality dye-based assays. bioRxiv. , (2024).
- Møbjerg, A., et al. Extreme freeze-tolerance in cryophilic tardigrades relies on controlled ice formation but does not involve significant change in transcription. Comp Biochem Physiol Part A Mol Integr Physiol. 271, 111245 (2022).
- Tsujimoto, M., Imura, S., Kanda, H. Recovery and reproduction of an Antarctic tardigrade retrieved from a moss sample frozen for over 30 years. Cryobiology. 72 (1), 78-81 (2016).
- Jönsson, K. I. Tardigrades as a potential model organism in space research. Astrobiology. 7 (5), 757-766 (2007).
- Jönsson, K. I., Rabbow, E., Schill, R. O., Harms-Ringdahl, M., Rettberg, P. Tardigrades survive exposure to space in low Earth orbit. Curr Biol. 18 (17), R729-R731 (2008).
- Kasianchuk, N., Rzymski, P., Kaczmarek, &. #. 3. 2. 1. ;. The biomedical potential of tardigrade proteins: A review. Biomed Pharmacother. 158, 113983 (2023).
- Arakawa, K. Examples of extreme survival: Tardigrade genomics and molecular anhydrobiology. Annu Rev Anim Biosci. 10 (1), 519-542 (2022).
- Hvidepil, L. K. B., Møbjerg, N. New insights into osmobiosis and chemobiosis in tardigrades. Front Physiol. 14, 1274522 (2023).
- Tanaka, S., Aoki, K., Arakawa, K. In vivo expression vector derived from anhydrobiotic tardigrade genome enables live imaging in Eutardigrada. Proc Natl Acad Sci U S A. 120 (5), e2216739120 (2023).
- Kondo, K., Tanaka, A., Kunieda, T. Single-step generation of homozygous knockout/knock-in individuals in an extremotolerant parthenogenetic tardigrade using DIPA-CRISPR. PloS Genet. 20 (6), e1011298 (2024).
- Yoshida, Y., Hirayama, A., Arakawa, K. Transcriptome analysis of the tardigrade Hypsibius exemplaris exposed to the DNA-damaging agent bleomycin. bioRxiv. , (2024).
- Yoshida, Y., et al. Time-series transcriptomic screening of factors contributing to the cross-tolerance to UV radiation and anhydrobiosis in tardigrades. BMC Genomics. 23 (1), 405 (2022).
- Yoshida, Y., et al. RNA sequencing data for gamma radiation response in the extremotolerant tardigrade Ramazzottius varieornatus. Data Brief. 36, 107111 (2021).
- Neves, R. C., et al. Differential expression profiling of heat stressed tardigrades reveals major shift in the transcriptome. Comp Biochem Physiol Part A Mol Integr Physiol. 267, 111143 (2022).
- Yoshida, Y., et al. Comparative genomics of the tardigrades Hypsibius dujardini and Ramazzottius varieornatus. PLoS Biol. 15 (7), e2002266 (2017).
- Wang, C., Grohme, M. A., Mali, B., Schill, R. O., Frohme, M. Towards decrypting cryptobiosis - analyzing anhydrobiosis in the tardigrade Milnesium tardigradum using transcriptome sequencing. PLoS One. 9 (3), e92663 (2014).
- Mali, B., et al. Transcriptome survey of the anhydrobiotic tardigrade Milnesium tardigradum in comparison with Hypsibius dujardini and Richtersius coronifer. BMC Genomics. 11 (1), 168 (2010).
- Förster, F., et al. Transcriptome analysis in tardigrade species reveals specific molecular pathways for stress adaptations. Bioinform Biol Insights. 6, 69-96 (2012).
- Arakawa, K., Yoshida, Y., Tomita, M. Genome sequencing of a single tardigrade Hypsibius dujardini individual. Sci Data. 3 (1), 160063 (2016).
- Arakawa, K. Transcriptome assembly of Richtersius coronifer with annotated BLAST result against Ramazzottius varieornatus. Figshare. Dataset. , (2019).
- Yoshida, Y., Konno, S., Nishino, R., Murai, Y., Tomita, M., Arakawa, K. Ultralow input genome sequencing library preparation from a single tardigrade specimen. J Vis Exp. (137), (2018).
- Murai, Y., et al. Multiomics study of a heterotardigrade, Echiniscus testudo, suggests convergent evolution of anhydrobiosis-related proteins in Tardigrada. bioRxiv. , (2020).
- Yoshida, Y., Sugiura, K., Tomita, M., Matsumoto, M., Arakawa, K. Comparison of the transcriptomes of two tardigrades with different hatching coordination. BMC Dev Biol. 19 (1), 24 (2019).
- Tenlen, J. R. Microinjection of dsRNA in tardigrades. Cold Spring Harb Protoc. 2018 (11), (2018).
- Tenlen, J. R., McCaskill, S., Goldstein, B. RNA interference can be used to disrupt gene function in tardigrades. Dev Genes Evol. 223 (3), 171-181 (2013).
- Giovannini, I., et al. Production of reactive oxygen species and involvement of bioprotectants during anhydrobiosis in the tardigrade Paramacrobiotus spatialis. Sci Rep. 12 (1), 15888 (2022).
- Ly, K., Reid, S. J., Snell, R. G. Rapid RNA analysis of individual Caenorhabditis elegans. MethodsX. 2, 59-63 (2015).
- Boothby, T. C. Total RNA extraction from tardigrades. Cold Spring Harb Protoc. 2018 (11), 905-907 (2018).
- Czerneková, M., Vinopal, S. The tardigrade cuticle. Limnol Rev. 21 (3), 127-146 (2021).
- McNuff, R. Laboratory culture of Hypsibius exemplaris. Cold Spring Harb Protoc. 2018 (11), 867-870 (2018).
- Gabriel, W. N., et al. The tardigrade Hypsibius dujardini, a new model for studying the evolution of development. Dev Biol. 312 (2), 545-559 (2007).
- Ruiz-Villalba, A., Ruijter, J. M., van den Hoff, M. J. B. Use and misuse of cq in qPCR data analysis and reporting. Life (Basel). 11 (6), 508 (2021).
- Antonov, J., et al. Reliable gene expression measurements from degraded RNA by quantitative real-time PCR depend on short amplicons and a proper normalization. Lab Invest. 85 (8), 1040-1050 (2005).
- Toussaint, J., et al. Improvement of the clinical applicability of the genomic grade index through a qRT-PCR test performed on frozen and formalin-fixed paraffin-embedded tissues. BMC Genomics. 10, 424 (2009).
Reprints and Permissions
Request permission to reuse the text or figures of this JoVE article
Request PermissionExplore More Articles
This article has been published
Video Coming Soon
Copyright © 2025 MyJoVE Corporation. All rights reserved