Method Article
Cella singola Profiling trascrizionale dei cardiomiociti adulti mouse
In questo articolo
Riepilogo
Singoli profili di espressione delle cellule consente la dettagliata analisi di espressione genica di cellule singole. Noi descrivere i metodi per l'isolamento dei cardiomiociti, e preparare il lisati risultante sia per microarray intero trascrittoma o qPCR di obiettivi specifici.
Abstract
Mentre numerosi studi hanno esaminato i cambiamenti di espressione genica da omogenati di tessuto cardiaco, questo impedisce studiando la variazione inerente stocastica tra le cellule all'interno di un tessuto. Isolamento delle popolazioni di cardiomiociti pura attraverso una perfusione collagenasi dei cuori del mouse facilita la generazione di microarray singola cella per tutta l'espressione genica trascrittoma, o qPCR di obiettivi specifici utilizzando gli array nanofluidic. Descriviamo qui una procedura per esaminare le singole cellule profili di espressione genica dei cardiomiociti isolati dal cuore. Questo paradigma permette per la valutazione delle metriche di interesse che non dipendono dalla media (per esempio la varianza tra le cellule all'interno di un tessuto) che non è possibile quando si utilizza tessuti tradizionali flussi di lavoro intero per la valutazione dell'espressione genica (Figura 1). Abbiamo raggiunto l'amplificazione robusta del trascrittoma microgrammi singola cellula cedimento di cDNA doppio filamento che facilita l'uso di microarray su icellule ndividual. Nella procedura viene descritto l'uso di array NimbleGen che sono stati selezionati per la loro facilità d'uso e possibilità di personalizzare il loro design. In alternativa, un trascrittasi inversa - amplificazione del target specifico (RT-STA) di reazione, permette di qPCR di centinaia di obiettivi da parte nanofluidic PCR. Utilizzando uno di questi due approcci, è possibile esaminare la variabilità di espressione tra le cellule, così come l'esame di profili di espressione di tipi di cellule rare all'interno di un tessuto. Nel complesso, il singolo gene approccio cellula espressione permette la generazione di dati che sono potenzialmente in grado di identificare profili di espressione idiosincratica che sono tipicamente mediati in sede di esame l'espressione di milioni di cellule da omogenati tipici generati dai tessuti interi.
Protocollo
1. Fase 1 - Isolamento Cardiomyocyte
- Preparare il tampone di digestione come segue:
10 x Digestion Buffer (made in ultra-puri H 2 O)Conc. finale. g / L g/500 ml 10x soluzione g / L NaCl 130 mm 7,597 3,3 75,97 KCl 5 mM 0,4 0,2 4.0 L'acido piruvico 3 nm 0,33 0,165 3,30 HEPES 25 mM 5,96 2,85 59,6 MgCl 2 0,5 mM 0,101 0,05 1,01 NaH 2PO 4monobasic 0,33 mm 0,04 0,02 0,40 Destrosio 22 mm 3,96 1,98 39,6
pH a 7,4 con NaOH - Utilizzando il Buffer 10x digestione, effettuare le seguenti quattro soluzioni per ogni cuore che verranno perfusi (made in ultra-puri H 2 O):
Digestion Buffer A (Fai tre aliquote di questa soluzione)
- 50 ml - 1x Digestion Buffer contenente:
- 75 microlitri - soluzione EGTA (100 mM EGTA)
Digestione del buffer B (fare uno di questi)
- 25 mL - Digestion Buffer 1x contenente:
- 1,25 microlitri - CaCl 2 Soluzione (1 M)
- 1 mg - Proteasi
- 25 mg - Collagenasi (can utilizzare anche il 75% di tale importo = 18,75 mg) **
Collezione Buffer (Fai uno di questi)
- 2,5 ml - Digestion Buffer 1x contenente:
- 1,25 microlitri - CaCl 2 Soluzione (1 M)
- 0,5 mg - proteasi
- 15 mg - Collagenasi (75% = 11,25 mg) **
- 125 mg - BSA
Neutralizzazione tampone di lavaggio (fare uno di questi)
- 25 mL - Digestion Buffer 1x contenente:
- 6,25 microlitri - CaCl 2 Soluzione (1 M)
- 250 mg di BSA
** A seconda della sorgente e lotto di collagenasi, l'attività degli enzimi può variare. Potrebbe essere necessario ottimizzare la quantità di enzima aggiunto a questo buffer per evitare sovra-digestione.
- Impostare il sistema di isolamento come descritto in precedenza in detail 1,2 con tre bicchieri 15 mL di Digestion Buffer A su ghiaccio, un tubo da 50 ml di tampone di digestione A e la digestione 25 B ml tampone pronto per perfusione (Digestion Buffer A deve essere lavata attraverso la linea, come minimo di 5 ml eseguire attraverso il sistema), Buffer Collection scaldato a bagnomaria per aumentare la temperatura a 37 ° C, e il tampone di lavaggio di neutralizzazione a temperatura ambiente.
- Riempire la cannula di perfusione e il piatto dissezione dove il cuore sarà ripulita e legato sulla punta perfusione con A. freddo Digestion Buffer Liberamente nodo alcuni sutura (4-O a 6-O dimensioni) intorno alla porzione superiore della cannula. Questo verrà successivamente utilizzato per contenere l'aorta per evitare che il cuore scivolare la cannula.
- Terminali sedare il mouse con una iniezione di anestetico IP agente (0,25 mL Pentobarbital soluzione di sodio) e di garantire il mouse è inconscia e non risponde agli stimoli del dolore. Taglio aperto nella cavità toracica con cura lungo il margine del torace tagliando il diaframma(Figura 2). Tagliato superiormente verso l'alto e riflette la gabbia toracica anteriore (queste incisioni sono designati B e A, rispettivamente in Figura 2) Critical -. Il mouse deve essere ancora respirare prima di queste incisioni.
- Usando una pipetta di plastica tagliata a misura, succhiare dolcemente il cuore nella pipetta. Tagliare il cuore fuori della cavità toracica, facendo attenzione a lasciare abbastanza dell'aorta sul cuore di identificare chiaramente il ramo dell'aorta (Figura 3). Una pipetta viene utilizzato in quanto provoca danni minimi al cuore.
- Cadere il cuore in una coppa di Digestion Buffer ghiaccio freddo A e lavare via il sangue. Dopo 15-45 sec luogo del cuore nel secondo becher di Digestion Buffer ghiacciata A per sciacquare brevemente di nuovo. Trasferire il cuore nella capsula di Petri anche pieno di A. freddo Digestion Buffer
- Visualizza il cuore nel piatto di Digestion Buffer Un nell'ambito di applicazione dissezione e con attenzione isolare l'aorta ei suoi rami. Rifilare con cura appena inferiore agli ultimi Branch (Figura 4).
- Inserire la cannula nell'aorta e legare in posizione con il 4-O a 6-O sutura utilizzando la fase di setup al microscopio di dissezione. Assicurarsi che la cannula non sedersi troppo in basso all'interno della aorta e rimane sopra le arterie coronarie (Figura 4).
- Con Digestion Buffer A che scorre attraverso il setup perfusione, posizionare la punta perfusione al setup, e cominciare a perfusione del cuore fino a quando la maggior parte del sangue viene lavato dal cuore e fluido lasciando il cuore è chiaro. Critica - la temperatura del liquido di perfusione deve essere il più vicino possibile a 37 ° C quando si esce dal catetere. Se la soluzione è troppo caldo o troppo freddo allora la perfusione ucciderà il tessuto. Il tempo di perfusione del cuore è anche critico. La finestra temporale deve essere inferiore a 5-10 minuti dalla rimozione del cuore fino a quando inizia la perfusione. Il cuore deve essere rimosso sotto anestesia, non CO 2 apposizione o dislocazione cervicale.
- Una volta che il sangue è statohed dal cuore, passare alla B Digestion Buffer da A. Digestion Buffer Assicurarsi che la B Digestion Buffer è ora che passa attraverso il cuore. La soluzione inizierà ad avere una tinta gialla. Attendere 2-3 minuti fino a quando il cuore presenta segni di perdere la sua forma e inizia a guardare arrotondata che è un'indicazione che la digestione è in funzione. Miocardio dovrebbe apparire più chiaro con il procedere della perfusione.
- Tagliare i ventricoli con forbici e posto in una provetta da 50 ml conica contenente 15 ml di tampone di raccolta a 37 ° C. Delicatamente tagliare il ventricolo in piccoli pezzi durante l'inserimento del tessuto nel buffer Collection. Mescolare delicatamente con una pipetta di plastica per sciogliere la cellula-cellula adesioni e fare una singola miscela cardiomiociti delle cellule.
- Quando la maggior parte del tessuto è dissolto, (meno di 1 minuto) permettono i pezzi di tessuto più grandi si depositano sul fondo. Rimuovere e conservare il surnatante in una provetta da 15 ml. Lascia la gravità tirare una pallina da surnatante di oltre 10 a 20 min (15min preferito) a temperatura ambiente.
- Rimuovere e scartare il surnatante. Lavare a pellet aggiungendo 5 ml di tampone di lavaggio di neutralizzazione per il pellet e mescolare delicatamente per risospendere le cellule di loro. A questo punto le cellule sono subito pronti per essere selezionati per l'analisi singola cella (passare al punto 2).
2. Fase 2 - isolamento di singole cellule
- Preparare un micromanipolatore utilizzando una fiamma e un tubo capillare di vetro, che saranno utilizzati per manipolare le cellule. Il micromanipolatore è semplicemente un tubo molto sottile capillare che viene poi attaccato ad un lungo pezzo di tubo flessibile che può spostare o raccogliere le cellule quando l'aspirazione è applicato.
- Immediatamente procedere alla selezione delle singole cellule al microscopio (Nikon Eclipse TS100, 20x) utilizzando un micromanipolatore che è stato creato con una pipetta tirato e una camera di compensazione per evitare che il materiale venga trasferito al singolo campione campioni di cellule. Assicurarsi che la popolazione cellulare è sano(70% vitali) e non danneggiato dal processo di isolamento.
- Diluire le cellule verso il basso in una piccola scatola di Petri in modo che possano essere selezionati singolarmente. Quando le cellule raccolta essere sicuri di catturare le cellule in un piccolo volume (meno di 2 mL). Selezionare solo le cellule sane che hanno la particolare morfologia dei cardiomiociti normale (Figura 5).
- Luogo singole cellule nella parte inferiore dei singoli tubi PCR e congelare immediatamente in ghiaccio secco. Queste cellule vengono poi conservati a -80 ° C fino a che non devono essere utilizzati per l'analisi di espressione genica.
3. Fase 3A - Singola cellula qPCR espressione genica
Per eseguire qPCR su singole cellule utilizzano un protocollo adattato da quello sviluppato per la singola espressione genica su cellule di Fluidigm Corporation per il loro BioMark nanofluidic array qPCR (Fluidigm - Protocollo per lo sviluppo Advance # 30) 3,4.
- Preparazione della trascrizione inversa - Amplificazione target specifico (RT-STA). Per esseregin, risospendere tutte le coppie di primer (che abbiamo usato fino a 125 coppie di primer con successo) per i geni di interesse a 100 mM in tampone 1x sospensione del DNA (10 mM Tris, pH 8.0, 0.1 mM EDTA). Combina e indebolirà ulteriormente le coppie di primer forward e reverse a 20 mM per ciascun test. Infine, diluire tutte le coppie di primer in un'unica soluzione con una concentrazione finale di 200 Nm per ogni test che verrà amplificato nella reazione RT-STA. Per fare questo, prendere ogni coppia di primer e diluire li 1:100. Per esempio, se si hanno 30 coppie di test iniettore a 20 micron, è necessario aggiungere 1 ml di ciascuna coppia (30 in totale mL), e poi compongono il resto del volume con 70 ml di tampone del DNA sospensione per raggiungere la finale il volume di diluizione di 100 ul.
- Ora che le soluzioni di fondo sono preparati, assemblare la RT-STA master mix di reazione. Preparare abbastanza master mix per ciascuna delle singole cellule che si desidera amplificare.
Tabella 1. RT-STA Master MixReagente Volume in microlitri (per reazione) CellsDirect 2x mix di reazione 5.0 SuperScript III RT Plantinum Taq miscelare 0,2 RT-STA mix Primer (200 nM ogni test) 2,5 Nucleasi gratuito H 2 O (o 1x buffer di sospensione del DNA) 1,3 Totale 9.0 - Rimuovere immediatamente cellule congelate e centrifugare a RCF massimo per 30 secondi per assicurare che le cellule sono posizionati al fondo della provetta. Aggiungere 9 ml di RT-STA mix al tubo e procedere alla reazione in un termociclatore.
- 50 ° C - 15 minuti
- 95 ° C - 2 minuti
- 18 cicli di
- 95 ° C per 15 secondi
- 60° C per 4 minuti
- Mantenere a 4 ° C
- Rimuovere i tubi dalla macchina PCR e aggiungere 4 ml di ExoSAP-IT per la reazione. ExoSAP-IT è un reagente di pulizia PCR che rimuoverà primer in eccesso e degli enzimi dalla RT-STA reazione. Eseguire i tubi attraverso la seguente reazione in una macchina PCR.
- 37 ° C - 15 minuti
- 80 ° C - 15 minuti
- Mantenere a 4 ° C
- Quando il ExoSAP-IT trattamento è completa; diluire il campione 1:05 con il tampone di sospensione del DNA. I campioni sono ora pronti per l'analisi qPCR. Usiamo questo materiale in congiunzione con le matrici Fluidigm espressione genica qPCR (48,48 o 96,96 piastre). E 'anche possibile utilizzare questo approccio con più qPCR strumentazione tradizionale (96 o 384 formati ben bene). Tuttavia, questi strumenti in genere richiedono input di esempio molto più che limita il numero di test che possono essere performed da una singola cellula.
3. Punto 3B - amplificazione intero trascrittoma e microarray di singole cellule
Estrazione e amplificazione
- Usa Sigma WTA2 Kit (Cat # WTA2) per amplificare il cDNA doppio filamento sia da pellet e singole cellule. Singole cellule vengono "estratti" via la prima fase del protocollo WTA2 Sigma secondo le istruzioni del produttore. Durante questa prima fase, l'incubazione con tampone biblioteca di sintesi a 37 ° C per 5 minuti distrugge la membrana delle singole cellule e "estrae" il messaggio per l'amplificazione.
- Amplificare singole cellule in due turni, con le indicazioni da kit WTA2 la Sigma-Aldrich. Eseguire il passaggio di preparazione biblioteca secondo il protocollo del produttore, quindi aggiungere 1 / 5 del campione biblioteca a 70 microlitri della amplificazione WTA2
Master Mix. Amplificare i campioni per 25 cicli utilizzando i parametri di ciclismo raccomandata. - Purificare l'USI campioning Qiagen è QIAquick PCR Purification Kit (Cat # 28104) e di processo su Qiacube Qiagen utilizzando il "Cleanup QIAquick reazioni PCR di amplificazione standard V4" protocollo.
- Concentrare i campioni purificati a 5 microlitri, con un vuoto di velocità, e sottoposto a un secondo turno di amplificazione per 17 cicli (WTA2 protocollo di amplificazione e parametri ciclismo ripetute).
- Purificare i campioni utilizzando la Purificazione QIAquick PCR Kit e controllo di qualità utilizzando uno spettrofotometro NanoDrop e Agilent Bioanalyzer DNA 7500 kit in base al protocollo produttori '.
Etichettatura e NimbleGen array di espressione genica
- Etichetta 2 mg di doppia elica del DNA da ciascun campione di cellule amplificato utilizzando NimbleGen One Kit di etichettatura a colori (Cat # 05223555001) secondo il protocollo del produttore.
- Ibridare 5 mcg di Cy3 campione etichettati in modo da Mus musculus 12x135k, NimbleGen array di espressione genica (Cat # 05543797001) secondoording il protocollo del produttore. Dopo l'ibridazione dei campioni per gli array, i vetrini devono essere lavati ed asciugati prima di essere ripreso su uno scanner laser.
- Gli array di espressione genica di scansione utilizzando uno scanner laser. Qui, usiamo un Molecular Devices GenePix Scanner 4200A con impostazioni di 100POW, e 300-350PMT. Quindi generare i file coppia per ogni array utilizzando il software NimbleScan NimbleGen e, successivamente, di analizzare per intero l'espressione trascrittoma da ciascuna matrice singola cellula (una tavola di tipico trascrizioni stabilmente espresso cardiomiociti è incluso come supplemento; S1 tabella).
Se la perfusione cardiaca lavorato bene ci dovrebbe essere una percentuale di cellule del cuore sano che conservano la loro morfologia tipica forma rettangolare su di isolamento. Se la perfusione non ha proceduto bene allora ci sarà una grande percentuale di cellule morte (vedi immagini in Figura 5). Se le cellule sono correttamente amplificato utilizzando i metodi WTA2 o RT-STA l'n il prodotto finale dovrebbe passare le successive prove di controllo di qualità per la qualità dei campioni di mRNA da NanoDrop e BioAnalyser (Figura 6). Per il flusso di lavoro microarray, questo è completata dopo l'amplificazione WTA in cui viene testata l'mRNA sia NanoDrop e Bioanalyzer. Rappresentante risultati positivi per questa analisi sono mostrati in figura 6. Il WTA2 campioni dovrebbe mostrare un robusto amplificazione sia con la lettura dello spettrofotometro NanoDrop, così come la lettura elettroferogramma dal (BA) Chip Bioanalyzer (Figura 6). Una tabella di geni che sono stati rilevati in modo stabile tramite microarray di singole cellule è incluso come un esempio (Tabella S1). Per il processo qPCR, la qualità dei dati può essere valutata eseguendo un passo di fusione alla fine della reazione PCR per garantire che il set di primer sono amplificando il prodotto desiderato (Figura 7). Se è corretta, questo passo si fondono dovrebbe generare un picco specifico sciogliere. Per illustrare questo punto, un sottoinsieme di dati nanofluidic qPCR è esposta in una heatmap picco di fondere temperatura (Figura 8) 8. E 'possibile valutare la qualità di un prodotto di PCR per la sua temperatura di fusione per garantire l'assenza di prodotti non specifici 5. Ogni riga di questa mappa rappresenta un campione delle cellule (S1-S40) testati in 43 saggi qPCR separato (A1-A43). In questa figura, è chiaro che alcuni test sono molto variabili e sono probabilmente meno affidabili i test più stabile con una maggiore specificità della PCR.
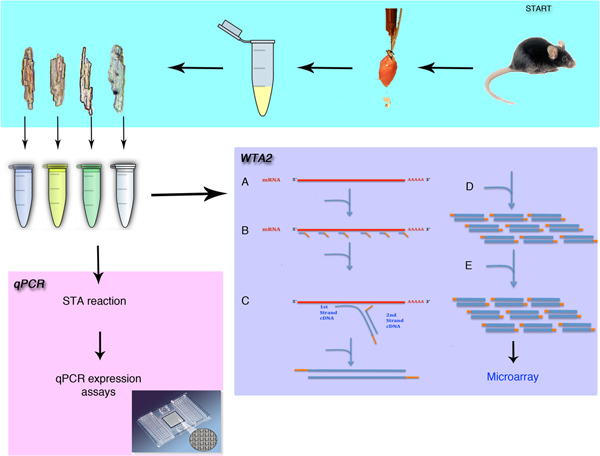
Figura 1. Panoramica della singola cella di isolamento e analisi di espressione genica. La procedura dimostrerà la rimozione chirurgica del cuore mouse e l'isolamento dei singoli cardiomiociti. I metodi discussi in questa procedura sono i metodi sia per qPCR o analisi microarray dopo amplificazione intero trascrittoma (WTA). La procedura WTA inizia con la lisi (A), quindi il legame del primer Sigma universale (B) per l'mRNApiscina. L'estensione di questi primer (C) e la (D) Fase di amplificazione creare microgrammi di materiale amplificato. Questa amplificazione viene quindi ripetuto (E) per generare abbastanza materiale per la procedura di microarray.

Figura 2. Rappresentazione della posizione delle incisioni per rimuovere il cuore dal mouse. Tagliare lungo la parete laterale della gabbia toracica (A), quindi tagliare lungo il margine inferiore della gabbia toracica (B). Taglio i vasi sopra e sotto il cuore consentire la rimozione pur lasciando abbastanza dell'aorta cannulate per il cuore. Immagini adattato da MJ Cook 6.
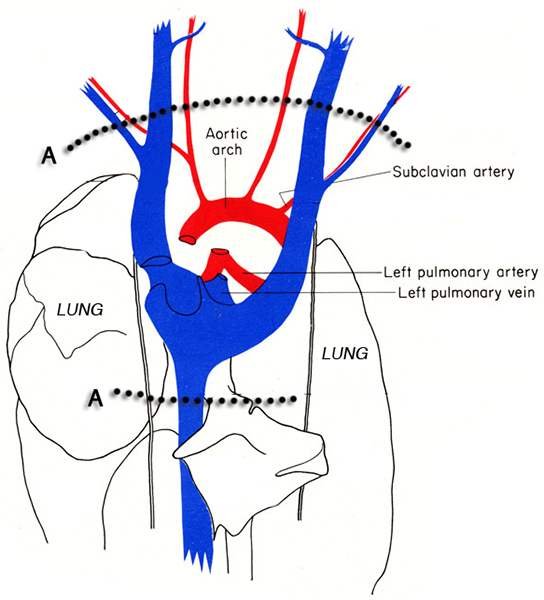
Figura 3. Schema del torace del mouse. Le linee tratteggiate indicano le posizioni delle incisioni (A) per asportare il cuore dalla cavità toracica senza danneggiare la h eart tessuti. Immagini adattato da MJ Cook 6.
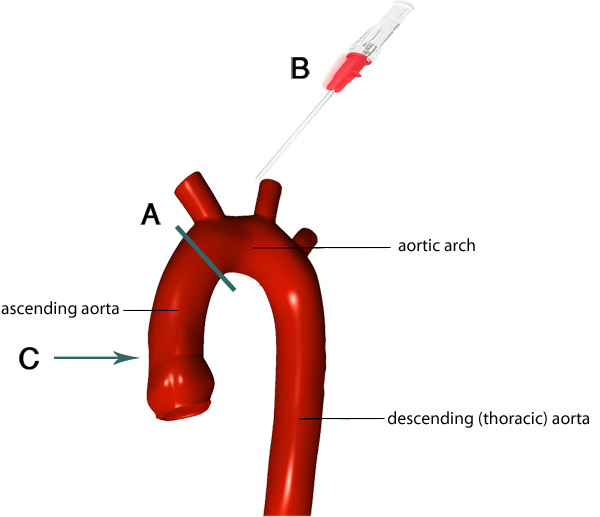
Figura 4. Immagine dell'arco aortico e dei suoi rami. La linea grigia indica il luogo ideale dove tagliare l'aorta (A). L'aorta che rimangono aderenti al cuore è cannulata modo che la punta della cannula (B) va al livello appropriato all'interno dell'aorta (C). Immagini adattato da F. Gaillard 7.

Figura 5. Selezione delle singole cellule per l'analisi di espressione genica. Ogni pannello mostra cardiomiociti fotografato al microscopio ottico che mostra morfologia tipica dei cardiomiociti. Cardiomiociti sano sono indicate dalle frecce verdi. Cellule che sono morti o morenti sono indicati con frecce rosse. Queste cellule morte non deve essere utilizzato per l'analisi.
iles/ftp_upload/3302/3302fig6.jpg "alt =" Figura 6 "/>
Figura 6. Controllo della qualità dei risultati WTA per l'amplificazione singola cella. (A) Immagine di una lettura spettrofotometro NanoDrop che è appropriato per le trascrizioni amplificato dalla reazione WTA. (B) La lettura dal chip Bioanalyzer mostra i risultati di tre cellule amplificate.
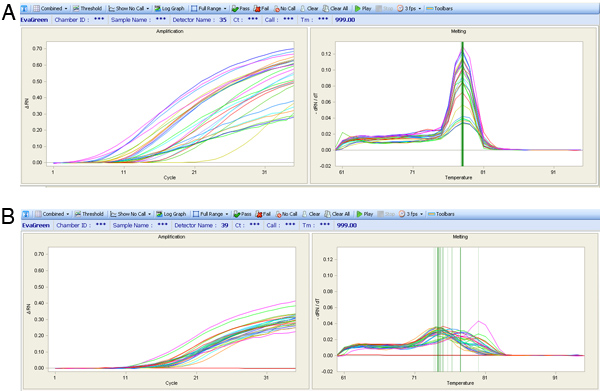
Figura 7. Curve di amplificazione qPCR (grafico a sinistra) e il picco si fondono curve di temperatura (grafico a destra). (A) L'espressione di un test di qualità vengono visualizzati con un unico picco sciogliere, nonostante le curve di amplificazione diverse. (B) Sciogliere le curve per un set fondo poveri, che ha picchi di sciogliersi molto variabile, che non è l'ideale per l'analisi di espressione.
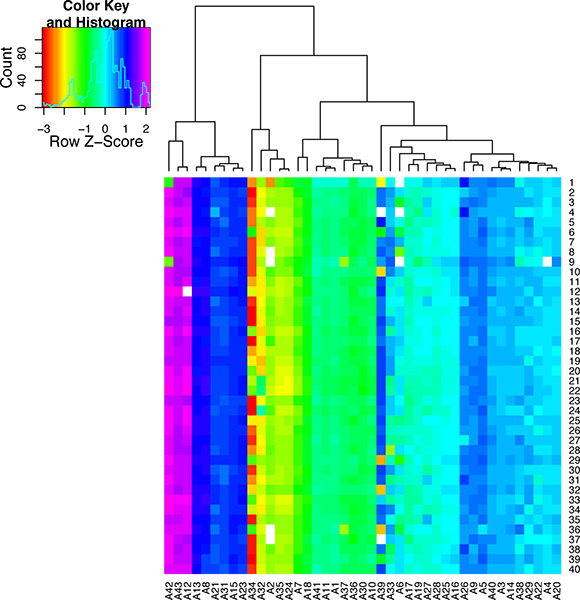
Figura 8. Controllo dei risultati di qualità per la singola cellula qPCR reagireioni. Questa mappa di calore è stato creato per visualizzare la temperatura del fuso come una scala di colori. La temperatura di fusione di picco per ogni dosaggio qPCR (A1-A43) è indicato per 40 celle singole (S1-40). I saggi sono stati raggruppati secondo la loro temperatura di fusione. Guardando verso il basso la colonna per ogni test è possibile vedere la variazione di fondere in tutti i campioni. Questo dato dimostra che alcuni saggi qPCR sono molto specifici, mentre altri sono molto variabili e quindi non sono adatti per l'analisi singola cella.
S1 tavolo. tabella supplementare dei dati di microarray. Questi geni sono elencati in base alla robustezza in cui vengono rilevati in cardiomiociti solo attraverso un set di dati di grandi dimensioni. Questa lista gene indica la sensibilità di rilevazione per una serie di geni che sono di solito espresse in cardiomiociti singolo.
Discussione
Questo metodo ha la possibilità di generare cardiomiociti per una serie di funzionali e studi di espressione genica. L'esame di singole cellule è una zona fiorente in analisi di espressione genica. Il vantaggio di esaminare i livelli di trascrizione a livello della singola cellula è che permette l'esame di una popolazione di cellule pure, che non è possibile dal preparazioni tessuto intero. Inoltre, l'analisi singola cella consente per l'esame di variazione stocastica dei livelli di mRNA nelle cellule individuali per definire popolazioni di cellule che in precedenza erano ritenuti omogenei 8,9. Oltre a identificare i geni che sono potenzialmente stocastici nella loro espressione genica 10,11,12, il metodo consente anche per l'identificazione delle popolazioni cellulari rari definito dai profili gene loro espressione.
Mentre questo metodo fornisce una serie di entusiasmanti possibilità di utilizzo in analisi di espressione genica, ci sono somavvertimenti e considerazioni e nell'uso di singole cellule. Il limite principale ad analisi singola cella, è la raccolta di cellule sufficiente per l'esperimento di raggiungere una significatività statistica per quanto riguarda la metrica di interesse, per esempio varianza. Nei casi in cui il numero di cellule isolate dal tessuto di interessi non è una limitazione, si può esaminare centinaia di cellule per gruppo, utilizzando approcci come sequenziamento di prossima generazione, o array nanofluidic. Tuttavia, in alcuni casi, ci possono essere difficoltà a ottenere cellule sufficienti dal tessuto di interesse. Questo può in parte essere causato da procedure momento idiosincratico intensivo per la raccolta del tipo di cellula bersaglio. Tuttavia, un'attenta pianificazione e la preparazione prima di procedere con la raccolta singola cellula può ancora consentire una rigorosa analisi singola cellula con limitato errore tecnico. Quando l'esame dei risultati deve essere considerato, che anche se questa procedura per le celle di isolamento è stato utilizzato in numerose Studies (tra cui microscopia, elettrofisiologia, ecc), lo stesso isolamento potrebbe potenzialmente effetto di espressione genica. Come per qualsiasi metodo che manipola i campioni biologici, i risultati delle indagini svolte deve essere attentamente convalidato per garantire l'espressione singola cella è rappresentativo del tessuto stesso e pregiudizi non tecnico. Metodi come ibridazione in situ può risultare utile per verificare questi risultati nel tessuto intatto. Infine, è fondamentale per garantire che i dati generati viene accuratamente controllato per il controllo qualità. I dati riportati in figura 8 dimostra che i test qPCR può essere molto robusti nella loro specificità, come il saggio # 13 (A13) o hanno un elevato livello di variabilità che possono portare a variazioni tecniche come test # 34 (A34).
Divulgazioni
Accesso libero e la produzione di questo di questo articolo è sponsorizzato Sigma-Aldrich.
Riconoscimenti
Gli autori desiderano ringraziare per l'assistenza tecnica di C. Zambataro durante le riprese di questo protocollo. Noi riconosciamo con gratitudine il sostegno della Fondazione Glenn for Medical Research (SM), La fondazione Hillblom, e il National Institutes of Health per un premio Centro Shock Nathan (P30AG025708) e PO1AG025901. JMF è stato sostenuto da T32AG000266 assegnato al Buck Institute for Research on Aging.
Materiali
| Name | Company | Catalog Number | Comments |
| Nome del reagente | Azienda | Numero di catalogo | Commenti |
| NaCl | Sigma-Aldrich | 71378 | |
| KCl | Sigma-Aldrich | 60128 | |
| L'acido piruvico | Sigma-Aldrich | P4562 | |
| Hepes | Sigma-Aldrich | 54457 | |
| MgCl 2 | Sigma-Aldrich | M1020 | |
| NaH 2 PO 4 monobasico | Sigma-Aldrich | P9791 | |
| Destrosio | Sigma-Aldrich | G6721 | |
| NaOH | Sigma-Aldrich | 72068 | |
| EGTA | Sigma-Aldrich | O3777 | |
| CaCl 2 | Sigma-Aldrich | 21115 | |
| Proteasi, Tipo XIV | Sigma-Aldrich | P5147 | |
| Collagenase, Tipo I | Worthington | C9891 | |
| 1x Sospensione tampone del DNA (10 mM Tris, pH 8.0, 0.1 mM EDTA) | TEKnova, | PN T0221 | |
| BSA | Sigma-Aldrich | B8667 | |
| Sodio pentobarbital | Henry Schein Vet. Alimentazione * | Contattare il fornitore per l'ordinazione | * Deve essere acquistato attraverso un laboratorio veterinario o animale professionale |
| ExoSAP IT | Affymetrix / USB | 78201 | |
| CellsDirect 2x Mix RXN | Invitrogen | 11737-030 | |
| SuperScript III RT Platinum Taq Mix | Invitrogen | 10928-042 | |
| RT-STA Primer Mix | IDT | Costume | |
| Nucleasi acqua gratis | Sigma-Aldrich | W4502 | |
| WTA2 | Sigma-Aldrich | WTA2-50rxn | |
| QIAquick PCR kit di purificazione | Qiagen | 28104 | |
| Qiacube | Qiagen | 9001292 | |
| NanoDrop Spettrofotometro | NanoDrop | 2000c | |
| Bioanalyzer DNA 7500 Kit | Agilent | 7500 kit | |
| Un colore kit etichettatura | Roche-NimbleGen | 5223555001 | |
| Mus musculus 12x135k serie | Roche-NimbleGen | 5543797001 | |
| GenePix 4200A Scanner | Molecular Devices | 4200A | |
| TransPlex Completa intero kit di amplificazione trascrittoma (WTA2 Kit) | Sigma-Aldrich | WTA2-50RXN |
Riferimenti
- Wolska, B. M., Solaro, R. J. Method for isolation of adult mouse cardiac myocytes for studies of contraction and microfluorimetry. Am. J. Physiol. 271 (3 Pt 2), H1250-H1255 (1996).
- Shioya, T. A simple technique for isolating healthy heart cells from mouse models. J. Physiol. Sci. 57 (6), 327-335 (2007).
- Guo, G., Huss, M., Tong, G. Q., Wang, C., Li Sun, L., Clarke, N. D., Robson, P. Resolution of cell fate decisions revealed by single-cell gene expression analysis from zygote to blastocyst. Dev. Cell. 18 (4), 675-685 (2010).
- Narsinh, K. H., Sun, N., Sanchez-Freire, V., Lee, A. S., Almeida, P., Hu, S., Jan, T., Wilson, K. D., Leong, D., Rosenberg, J., Yao, M., Robbins, R. C., Wu, J. C. Single cell transcriptional profiling reveals heterogeneity of human induced pluripotent stem cells. J. Clin. Invest. 121 (3), 1217-1221 (2011).
- D'haene, B., Vandesompele, J., Hellemans, J. Accurate and objective copy number profiling using real-time quantitative PCR. Methods. 50 (4), 262-270 (2010).
- Cook, M. J. The Anatomy of the Laboratory Mouse. Informatics. , (1965).
- Gaillard, F. Aorta. Radiopaedia. , (2008).
- Ståhlberg, A., Andersson, D., Aurelius, J., Faiz, M., Pekna, M., Kubista, M., Pekny, M. Defining cell populations with single-cell gene expression profiling: correlations and identification of astrocyte subpopulations. Nucleic Acids Res. 39 (4), e24-e24 (2011).
- Zhong, J. F., Chen, Y., Marcus, J. S., Scherer, A., Quake, S. R., Taylor, C. R., Weiner, L. P. A microfluidic processor for gene expression profiling of single human embryonic stem cells. Lab Chip. 8 (1), 68-74 (2008).
- Bahar, R., Hartmann, C. H., Rodriguez, K. A., Denny, A. D., Busuttil, R. A., Dollé, M. E., Calder, R. B., Chisholm, G. B., Pollock, B. H., Klein, C. A., Vijg, J. Increased cell-to-cell variation in gene expression in ageing mouse heart. Nature. 441 (7096), 1011-1014 (2006).
- Raj, A., van Oudenaarden, A. Nature, nurture, or chance: stochastic gene expression and its consequences. Cell. 135 (2), 216-226 (2008).
- Elowitz, M., Levine, A., Siggia, E., Swain, P. Stochastic gene expression in a single cell. Science. 297 (5584), 1183-1186 (2002).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon