È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Sviluppo di Sulfidogenic fanghi da Marine Sedimenti e Tricloroetilene Riduzione in una coperta reattore Upflow anaerobica dei fanghi
In questo articolo
Riepilogo
Microbial sulfate reduction is a process of great importance in environmental biotechnology. The success of the sulfidogenic reactors depends among other factors on the microbial composition of the sludge. Here, we present a protocol to develop sulfidogenic sludge from hydrothermal vents sediments in a UASB reactor for reductive dechlorination purposes.
Abstract
The importance of microbial sulfate reduction relies on the various applications that it offers in environmental biotechnology. Engineered sulfate reduction is used in industrial wastewater treatment to remove large concentrations of sulfate along with the chemical oxygen demand (COD) and heavy metals. The most common approach to the process is with anaerobic bioreactors in which sulfidogenic sludge is obtained through adaptation of predominantly methanogenic granular sludge to sulfidogenesis. This process may take a long time and does not always eliminate the competition for substrate due to the presence of methanogens in the sludge. In this work, we propose a novel approach to obtain sulfidogenic sludge in which hydrothermal vents sediments are the original source of microorganisms. The microbial community developed in the presence of sulfate and volatile fatty acids is wide enough to sustain sulfate reduction over a long period of time without exhibiting inhibition due to sulfide.
This protocol describes the procedure to generate the sludge from the sediments in an upflow anaerobic sludge blanket (UASB) type of reactor. Furthermore, the protocol presents the procedure to demonstrate the capability of the sludge to remove by reductive dechlorination a model of a highly toxic organic pollutant such as trichloroethylene (TCE). The protocol is divided in three stages: (1) the formation of the sludge and the determination of its sulfate reducing activity in the UASB, (2) the experiment to remove the TCE by the sludge, and (3) the identification of microorganisms in the sludge after the TCE reduction. Although in this case the sediments were taken from a site located in Mexico, the generation of a sulfidogenic sludge by using this procedure may work if a different source of sediments is taken since marine sediments are a natural pool of microorganisms that may be enriched in sulfate reducing bacteria.
Introduzione
Uno dei contributi più importanti alla biotecnologia ambientale è stata la progettazione di bioreattori in cui il fanghi utilizzati (inoculo) era in grado di eseguire in condizioni solfato riducendo. Riduzione solfato (SR) consente il trattamento dei flussi di acque reflue che contengono elevate concentrazioni di solfato oltre alla rimozione simultanea di COD, metalli pesanti e inquinanti organici, un fatto che rende una caratteristica desiderabile SR dei fanghi 1. Alcuni esempi di effluenti contaminati con solfato provengono da conceria, carta, farmaceutico e manifatturiero industrie chimiche 1. Tuttavia, la maggior parte della letteratura si riferisce a sulfidogenic fanghi quando metanogenica fango granulare è stata adattata per sulfidogenesis 2. Questo adattamento è comunemente ottenuto manipolando il COD / SO 4 2- rapporto nel bioreattore e aggiunta di prodotti chimici per inibire methanogens del 2,3 fanghi. In aggiunta al tempo che may richiedere la formazione di granuli sulfidogenic, la competizione tra methanogens e riduttori di solfato e la tolleranza del fango ad alte concentrazioni di solfuro sono alcuni dei problemi principali che possono insorgere se l'fanghi sulfidogenic utilizzato nel bioreattore è ottenuto dall'adattamento di prevalentemente fanghi metanogenica di solfato condizioni riducenti. In questo lavoro, si descrive la procedura per ottenere un fango prevalentemente sulfidogenic da sorgenti idrotermali sedimenti (Punta Mita, Nayarit, Messico) in un anaerobico reattore fanghi coperta flusso verso l'alto (UASB), poi si valuta il suo solfato riducendo l'attività nel tempo e condurre un esperimento per valutare la sua applicazione su declorazione riduttiva. La posizione dei sedimenti è stato scelto perché è stato riferito che in quel sito vi è formazione di solfuri dovuta all'attività solfato riducendo esibito dalla comunità microbica che abitano quel determinato posto 4.
Ci sono severAl vantaggi nell'ottenere questo fanghi sulfidogenic da sedimenti più di adattarsi metanogenica fango granulare a sulfidogenesis. Alcuni di questi vantaggi sono: (1) non è necessario formare granuli per il bioreattore di operare, (2) i fanghi tollera concentrazioni relativamente elevate di solfuro rispetto ad altri UASB che operano con adattato fanghi methanogenic, e (3) non viene competizione per substrato con methanogens anche se acetato è utilizzato nella miscela di acidi grassi volatili che è incluso nel mezzo di coltura per promuovere la formazione di fanghi.
Questa procedura è stata seguita per promuovere sulfidogenesis perché sedimenti marini sono una piscina naturale di una grande varietà di microrganismi come batteri solfato riduzione, fermentazione batteri e dealogenante batteri solo per citarne alcuni 5,6. Il tipo di consorzio sviluppato da sedimenti marini utilizzando questo protocollo può presentare efficienza nella riduzione solfato e quindi, alti s ulfate riducendo l'attività nel tempo e maggiore tolleranza al solfuro a concentrazioni superiori al segnalato come tossici per methanogens e solfato riducendo batteri. D'altro canto, è probabile che la capacità dealogenante è anche mostrato nei sedimenti seguendo il protocollo qui proposto ma può dipendere dalla comunità microbica originale. Questa ipotesi viene fatto sulla base del fatto che la declorazione riduttiva può avvenire sia con la respirazione o co-metabolismo, entrambe le condizioni che possono essere promossi nella comunità microbica marina 7. La coltivazione dei sedimenti per ottenere i fanghi è stata condotta utilizzando una miscela di acetato, propionato e butirrato come substrato perché questi acidi grassi volatili sono utilizzati da diversi ceppi di batteri solfato-riduzione. Questi acidi sono anche il tipo di composti di carbonio spesso si trovano nei sedimenti marini, secondo diversi rapporti in letteratura sul materiale carbonioso in sedimenti marini 5,6.
content "> Infine, alcuni dei composti più tossici che si trovano in corpi idrici e altri acqua in tutto il mondo sono i solventi clorurati come il tricloroetilene (TCE) o percloroetilene (PCE). Questi composti sono tossici, non solo per l'essere umano, ma anche a microrganismi, in particolare TCE, che è ancora considerato un inquinante prioritario dall'Agenzia di Protezione Ambientale degli Stati Uniti 8. In questo lavoro abbiamo proposto un esperimento in cui i fanghi sulfidogenic è testato su sua capacità di ridurre TCE a concentrazioni che sono nella gamma riportato per composti clorurati biodegradazione in condizioni methanogenic 9,10. Vale la pena ricordare che la maggior parte della ricerca sulla biodegradazione di composti clorurati è stata condotta in condizioni methanogenic 9,10. Riteniamo che l'esperimento con TCE proposto in questo protocollo è un buon esempio delle potenziali applicazioni del fango. L'obiettivo di questo esperimento era di evalutare la tolleranza del fango al TCE e l'effetto TCE sul solfato riduzione dell'attività. Tenendo conto che la maggior parte della ricerca sulla biodegradazione di composti clorurati è effettuata in condizioni methanogenic, questo protocollo suggerisce la formazione di un fango può essere usato per simultaneamente: (1) rimuovere solfato, (2) rimuovere COD e (3) rimuovere composti clorurati. Un ulteriore passo potrebbe essere quello di valutare l'fanghi sulla rimozione simultanea del TCE e metalli pesanti (in aggiunta a solfato e COD), due condizioni che non possono essere valutati in condizioni methanogenic.Protocollo

Figura 1. Schema per le fasi del protocollo. Cliccate qui per vedere una versione più grande di questa figura.
1. Raccogliere marine sedimenti per la Formazione dei fanghi
- Identificare un'area sottomarina accessibile sia vicino a sorgenti idrotermali (a causa della presenza di solfuri, che può indicare una maggiore solfato riducendo l'attività) o ad una zona in cui i residui della sostanza organica sono rilevabili.
- Ai fini di questo lavoro, richiede circa 3 o 4 kg di sedimenti e drenare l'acqua fuori i campioni. Mettere i campioni in sacchetti di plastica scuri. Non è necessaria alcuna refrigerazione.
- Una volta in laboratorio, tenere le borse con i campioni in frigorifero, se non sono in corso per essere utilizzata immediatamente. Ai fini di questo lavoro, SAMPles può essere in frigorifero per settimane o mesi prima di utilizzarli.
- Prendere una grande porzione del campione di sedimento (cioè, 1 o 2 kg) e usare una rete appropriata (0,2 cm) per eliminare dai sedimenti grande detriti di materiale carbonioso che possono essere trovati o alcune rocce che possono essere presenti.
Nota: In questo caso una maglia di 0.20 cm di diametro (0,0767 in) è stato utilizzato, ma può essere di dimensioni diverse a seconda delle dimensioni delle particelle nel campione.- Dopo aver superato il sedimento attraverso la maglia, mescolare la porzione selezionata di promuovere che la porzione è omogenea.
- Prendere campioni più piccoli separati (cioè, da 2 a 3 g) determinare i solidi sospesi volatili (VSS) Opere seguendo i metodi standard 11.
Nota: Vedere la Figura 2 per i passaggi 1.2 a 1.4.

Figura 2. Le fotografie dei campioni di sedimento.Campioni (A) del sedimento appena dopo essere prese. Campione (B) del sedimento dopo il passaggio attraverso la rete. (C) Campione prelevato per la pesatura prima solidi sospesi volatili (VSS) determinazione. La piastra di Petri non ha bisogno di essere sterilizzati. Clicca qui per vedere una versione più grande di questa figura.
2. Bioreattore Set Up
- Ai fini di questo lavoro, utilizzare un reattore in vetro UASB con un volume totale di lavoro 3 L. In alternativa, utilizzare un reattore di vetro del volume 1 o 2 L.
- Sulla base del contenuto VSS dei sedimenti calcolare la quantità di sedimenti da utilizzare come inoculo per ottenere 5 g di VSS in 1 L.
- Tener conto che se la quantità di sedimenti dopo il calcolo è troppo grande, quindi circa il 25% al 30% del volume del bioreattore dovrebbe essere occupato da sedimenti invece.
- Registrare il contenuto VSS in quantocambierà quando la comunità microbica si arricchisce nel bioreattore. Il contenuto VSS è necessario per i calcoli di solfato di ridurre l'attività nel bioreattore.
- Assicurare che la concentrazione finale della soluzione tampone medio basale nel bioreattore è simile al riportato da Guerrero-Barajas et al. (2014) 12.
- Assicurarsi che i volumi finali dei sedimenti, medio basale, soluzione tampone e acidi grassi volatili sono pari al volume finale di lavoro del reattore. Il basale medio ricetta 12 contiene le concentrazioni adeguate per la metalli in tracce e vitamine soluzione.
- Preparare una soluzione stock di soluzione tampone terreno di base e in una concentrazione appropriata per il volume di lavoro del reattore utilizzato (ad esempio, 2, 3 o 4 volte più concentrato di quello riportato al punto 2.4) per garantire che quando viene diluito è alla concentrazione riportata da Guerrero-Barajas et al. (2014)12).
Nota: La soluzione madre per mezzo base è sempre necessario, tuttavia, la soluzione tampone è necessaria solo all'avviamento. Non è necessario aggiungere soluzione tampone dopo questo tempo. - Preparare una soluzione stock di acidi grassi volatili: acetato, propionato e butirrato in 2.5: 1: 1 COD proporzione. Prendere in considerazione per il calcolo del sodio acetato incluso nel terreno di base. La concentrazione finale COD nel reattore deve essere 2,7 g / L.
Attenzione: Preparare questa soluzione in una cappa aspirante. Indossare guanti di nitrile e occhiali per la preparazione di questa soluzione. Considerare la stechiometria delle reazioni di solfato con acidi grassi volatili che viene mostrato in figura 3. - Preparare una soluzione stock di solfato di sodio (Na 2 SO 4) in una concentrazione appropriata per fornire al reattore una concentrazione finale di 4,000 mg / l di ione solfato (SO 4 2-). In alternativa, includere °e quantità di solfato richiesto nel terreno di base invece di aggiungere da una soluzione madre finché il solfato finale (SO 4 2-) concentrazione è giusto.
- Posizionare i sedimenti nel reattore mescolato con una parte del terreno di base per assicurarsi che raggiungano il fondo del reattore.
- Aggiungere il resto della soluzione tampone basale medio e mescolato con la soluzione di acidi grassi volatili e la soluzione di solfato. Assicurarsi che la soluzione di acidi grassi volatili viene versato nel liquido. Nota: Effettuare questa operazione in una cappa aspirante.
- Impostare le connessioni e tubazioni del reattore per la pompa di riciclo. Impostare la portata di riciclaggio a 60 ml / min. Impostare il bioreattore nella camera di temperatura a 34 ° C. Regolarmente controllare che le variazioni di temperatura sono piccole (ad esempio, 34 ± 1,7 ° C)
- Impostare i collegamenti alla colonna di spostamento del gas.
Nota: La Figura 4 passi da 2.1 a 2.5.
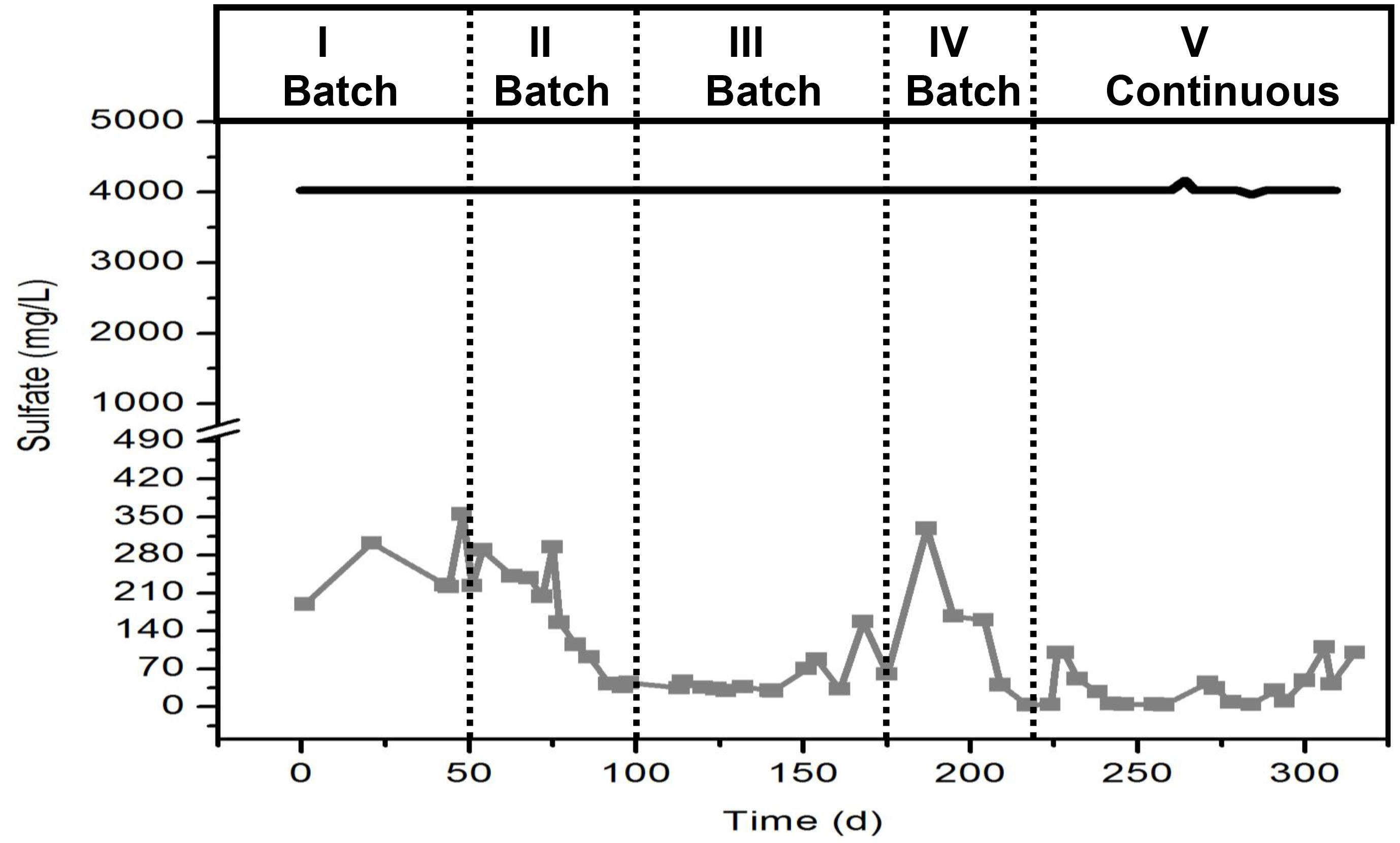
Figura 3. Stechiometria di riduzione solfato con VFA (acetato, propionato e butirrato). Cliccate qui per vedere una versione più grande di questa figura.
Figura 4. UASB reattore. (A) tempo iniziale. (B) il regime continua dopo 300 giorni di funzionamento. Cliccate qui per vedere una versione più grande di questa figura.3. Il funzionamento del reattore per promuovere Sulfidogenesis e crescita dei microrganismi
Nota: Consenti per l'inoculo di consumare la volatile acidi grassi e solfato. A questo scopo, attendere una settimana per effettuare la prima analisi solfato, solfuro e consumi COD.
- Dopo una settimana di incubazione prelevare un campione di 5 a 7 ml di liquido per condurre un'analisi per COD, solfato e solfuro contenuti e pH seguenti metodi standard 11, 13.
- Analizzare solfuro nella spettrofotometricamente liquido (ad una lunghezza d'onda (λ) di 670 nm) seguendo il metodo di metilene blu 13.
- Porre 5 ml di una soluzione di acetato di zinco (2% w / w) in un matraccio da 25 ml, aggiungere 200 ml di rapidamente il campione alla soluzione di acetato di zinco.
- Aggiungere 2,5 ml di N, N -dimethyl- p-fenilendiamina ossalato soluzione (DMP) (0,2% w / w in 20% H 2 SO 4) e 125 ml di ferro (III) soluzione di solfato di ammonio (10% w / w 2% in H 2 SO 4) e terminare con acqua distillata 25 ml in matraccio tarato. Attendere 30 minuti per la reazionea verificarsi, momento in cui il colore blu è stabilizzato. 13.
Nota: Attendere almeno 15 minuti, ma non più di 60 minuti per testare i campioni nello spettrofotometro. Effettuare la lettura della soluzione finale blu nello spettrofotometro.
- Analizzare solfato secondo metodi standard 11. Qui, come quantificare solfato di bario solfato utilizzando un metodo turbidimetrico.
- Mettere 5 ml di una soluzione di condizionamento (acido cloridrico HCl 1: 1) in un pallone tarato da 25 ml, aggiungere 1 ml di campione precedentemente centrifugato (a 11.320 xg), completare l'25 ml del pallone tarato con acqua distillata e aggiungere 1 g di cloruro di bario.
- Miscelare la soluzione per 1 minuto in un vortice. Attendere 4 min per il solfato di bario per formare e leggere il campione nel spettrofotometro a (λ) di 420 nm 11.
- Analizzare COD secondo metodi standard 11. In alternativa, utilizzare un COD DeterminaKit zione.
- Prima della determinazione COD, centrifugare il campione a fondo (a 11.320 xg) per rimuovere il solfuro restando che potrebbero interferire nella determinazione. Se necessario, centrifughi due volte: la prima volta immediatamente dopo il prelievo del campione e la seconda volta attendere 6 o 8 ore e quindi effettuare l'analisi COD.
- Aggiungere 2 ml di campione in una fiala reazione del kit di determinazione del COD, sigillare il flacone e omogeneizzare la miscela agitando con prudenza. Preparare un vuoto aggiungendo 2 ml di acqua distillata in un altro flacone di reazione e omogeneizzare la miscela.
- Posizionare le fiale nel reattore digestione a 150 ° C per 2 ore. Rimuovere le fiale e lasciateli raffreddare nel buio. Prendere le letture dei flaconi nella spettrofotometro ad una lunghezza d'onda di 620 nm.
- Ottenere il volume di gas dalla colonna spostamento gas.
- Analizzare solfuro nella spettrofotometricamente liquido (ad una lunghezza d'onda (λ) di 670 nm) seguendo il metodo di metilene blu 13.
- Attendere fino a un altro 5 a 7 giorni fino a quando il solfato è consumato. Solfato e COD devono essere consumati in unpproximately 85% al 90% prima che sia avviato un nuovo lotto alimentato.
- Una volta solfato (e COD) sono consumati, completamente ripetere il punto 2.4. Fornire un mezzo fresco e nuove sostanze nutritive per ciascun lotto.
- Ripetere i punti 3.1 e 3.2. A questo punto ogni partita dovrebbe durare tra i 7 ei 10 giorni.
- Quando sono state completate da 3 a 4 lotti, ripetere il passaggio 2.4 ma aumentare la concentrazione di COD 4 g / L.
- Ripetere il passaggio 3.1 e punto 3.2.
- Ripetere il punto 3.3, ma aumentare la concentrazione di COD 6 g / L.
- Ripetere 3.6 e 3.6.1 aumentando gradualmente concentrazione COD finché è 10 g / L.
Nota: rendere il grafico che presenta la concentrazione di solfato (mg / L) rispetto al tempo (d).
- Quando il consumo solfato è superiore all'80% in meno di 24 ore e questo avviene per più di una settimana, commutare il funzionamento del reattore a modalità continua. Per il funzionamento continuo impostare il tempo di ritenzione idraulica (HRT) a 24 ore e mantenere la concentrazione di solfato a 4 g / L e il CODa 10 g / L.
Nota: Nel corso del tempo il consumo di solfato dovrebbe essere veloce.
4. Solfato Ridurre test Attività
- Prima di questo test assicurarsi che il bioreattore in regime continuo presenta variazione inferiore al 10% della concentrazione di solfato rimanente.
- In un dato giorno, fermare il reattore dopo un ciclo di terapia ormonale sostitutiva e condotta passo 2.4. Per passo 2.4.3 utilizzare una concentrazione COD di 10 g / L.
- Una volta che il bioreattore è alimentato, prendere 5 a 7 ml di campioni di liquido e per eseguire analisi COD, solfato, solfuro (passo 3.1) e pH ogni ora. Registrare il volume di gas prodotto.
- Calcolare il solfato ridurre l'attività secondo la letteratura 14.

SRA = solfato riducendo l'attività (mg COD-H 2 S) / gVSS * d
m H 2 S = concentrazione di solfuro espressa come mg COD-H 2 S
VSS = volatili concentrazione di solidi sospesi
t = tempo (d o ore)
- Fare le corrispondenti grafici che mostrano la percentuale di consumo di solfato in funzione della concentrazione di solfuro nel tempo in mg / L. Rendere i grafici che mostrano percentuale del consumo COD nel tempo. Rendere i grafici che mostrano la variazione di pH nel tempo.
5. tricloroetilene (TCE) Riduzione di prova
- Prima di questo test assicurarsi che il bioreattore lavora in regime continuo e presenta variazione inferiore al 10% della concentrazione di solfato rimanente. Non iniziare il test se riduzione solfato nel bioreattore è inferiore al 90%.
- Preparare una soluzione stock di tricloroetilene (TCE) tenendo conto che la concentrazione finale di questo composto nella fase liquida del bioreattore deve essere di 300 micron. Si consideri il partitioning del composto allo spazio di testa utilizzando la costante adimensionale legge Henry's (H') per TCE a 34 ° C. H'at 34 ° C per il TCE è 0,4722.
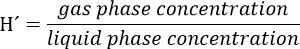
Attenzione: Preparare questa soluzione in un fumehood e indossare guanti e occhiali protettivi.- Ad esempio, per una soluzione madre 5.000 micron, calcolare come seguito:
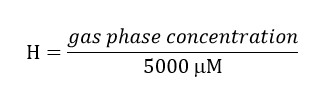
Concentrazione TCE fase gassosa = (0,4722) * (5000) = 2.139 micron. Includere questa concentrazione nella preparazione della soluzione di riserva poiché questa quantità di TCE sarà nello spazio di testa.
Poi nel liquido (acqua) della soluzione madre, la concentrazione effettiva TCE sarà: 5,000 + 2,139 = 7.139 pM. Densità TCE = 1,43 g / ml. Convertire il 7139 pM a mg e poi in base alla densità del TCE calcolare il volume del TCE per soluzione madre.
Nota: La concentrazione della soluzione madre TCE may essere inferiore a 5.000 micron, cioè, 3000 o 1000 pM, questo dipende dalla quantità di volume di tale soluzione può essere consegnato al bioreattore secondo il volume fase liquida.
- Ad esempio, per una soluzione madre 5.000 micron, calcolare come seguito:
- Preparare curve standard nel gascromatografo per TCE, cis--1,2 dicloroetilene, trans -1,2-dicloroetilene, cloruro di vinile e etene. Preparare i cis -1,2-dicloroetilene e trans -1,2-DICLOROETILENE curve standard da una soluzione madre di questi composti seguendo la stessa procedura descritta per la soluzione 5.2 TCE magazzino. Preparare le curve standard per il cloruro di vinile e etilene diluendo la concentrazione di ciascun gas dagli standard (cilindri gas).
- Preparare le curve standard di questi composti in una gamma da 20 a 300 micron. Utilizzare il metodo riportato da Guerrero-Barajas et al. (2011) 15 per l'analisi di questi composti nel gascromatografo.
Attenzione: Preparare questi standLe soluzioni ard in un fumehood e indossare guanti e occhiali protettivi.
- Preparare le curve standard di questi composti in una gamma da 20 a 300 micron. Utilizzare il metodo riportato da Guerrero-Barajas et al. (2011) 15 per l'analisi di questi composti nel gascromatografo.
- In un dato giorno, fermare il reattore dopo un ciclo di terapia ormonale sostitutiva e condotta passo 2.4. Per passo 2.4.3 utilizzare una concentrazione COD di 10 g / L.
- Una volta che il bioreattore viene alimentato, aggiungere il TCE direttamente al liquido nel bioreattore dalla soluzione madre preparata in 5.2, la concentrazione finale TCE nella fase liquida del bioreattore deve essere di 300 pM. Impostare la HRT di 12 ore.
- Alla fine di un ciclo HRT prelevare campioni di liquido (500 a 1,000 microlitri) e l'analisi condotta per COD, solfato e solfuro (passi 3.1.1, 3.1.2 e 3.1.3). Prelevare campioni di spazio di testa (100 a 250 microlitri) e condurre analisi per TCE, cis -1,2-dicloroetilene, trans -1,2-dicloroetilene, cloruro di vinile e etilene nel gascromatografo.
- Ripetere il punto 2.4. Per passo 2.4.3 utilizzare una concentrazione COD di 10 g / L.
- Non ripetere qualsiasi test riduzione TCE fino al bioreattore presenta oltre 9Riduzione 0% solfato e meno del 10% di variazione in entrambi, riduzione solfato e solfato rimanente nel bioreattore.
- Ripetere 5.4, 5.5 e 5.6 due o tre volte di più.
- Dai campioni di sedimento (0,5 g) per condurre l'identificazione dei microrganismi subito dopo un test di riduzione TCE è terminata. Fare questo dopo 2 o 3 test di riduzione del TCE.
6. Solfato Ridurre test attività dopo TCE Riduzione Experiment
- Ripetere il punto 4 completamente.
7. Individuazione dei Microrganismi
- Prelevare campioni di fanghi di circa 0,5 g ciascuno e condurre l'estrazione di RNA totale secondo il metodo standard 12.
- Amplificare il gene 16S rRNA con la trascrizione inversa e condurre la reazione a catena della polimerasi (RT-PCR) Rinforzo un passo 12.
- Progettare i primer per amplificare o usare come un primo approccio quelli proposti nella letteratura 11. Seguire la amplifProcedura icazione suggerito in letteratura 12.
- Costruire le librerie 16S rRNA. Ampliconi della PCR possono essere clonati utilizzando un kit di clonazione-11. Tipicamente, 10 colonie da ciascuna piastra (ogni colonia rappresenta un prodotto PCR) possono essere clonati. Preparare il DNA plasmidico per sequenziamento secondo quanto suggerito in letteratura 12.
- Condurre il sequenziamento dei frammenti. Re-amplificare circa 1.400 bp dei prodotti esterni PCR con il protocollo per l'amplificazione PCR precedentemente descritto (passo 7.4) e clone secondo quanto suggerito in letteratura 12. Isolare il plasmide ricombinante da E. colonie coli come suggerito in letteratura 12. Non condurre la gara parziale per il sequenziamento con M13 primer universali 12.
- Effettuare l'analisi di sequenze. Allineare le sequenze nucleotidiche tramite Clustal X e regolare manualmente nell'editor di testo. Effettuare ricerche BLAST delle databas NCBIe. (http://www.ncbi.nlm.nih.gov/BLAST/Blast.cgi) 12.
- Ottenere i numeri di accesso sequenza nucleotidica. Depositare le sequenze nucleotidiche dei cloni individuati nel database sequenza nucleotidica EMBL (Gen-Bank / EMBL / DDBJ) sotto i corrispondenti numeri di accesso (ad esempio, per le sequenze da JQ713915eJQ713925 ampliconi) 12.
Risultati
Un comportamento tipico della riduzione solfato nel bioreattore è mostrato in Figura 5. E 'importante notare che durante le prime settimane di riduzione solfato funzionamento sarà lenta. Tuttavia lento, il consumo di oltre il 90% di solfato nel tempo indica che l'inoculo sta sviluppando una comunità microbica in grado di ridurre solfato e quindi arricchito in solforiduttori batteri. I diversi periodi della cifra indicano che la riduzione del solfato aumentava la sua velocità...
Discussione
Ci sono diverse applicazioni di sulfidogenesis della biotecnologia ambientale, una delle applicazioni più utilizzate del metabolismo dei batteri solfato riducenti in consorzi con fermentazione batteri è in trattamento delle acque reflue. Reattori UASB sono tra i principali approcci di ingegneria al trattamento delle acque reflue industriali con alte concentrazioni di solfato. In questo lavoro, vi presentiamo un protocollo per ottenere fanghi sulfidogenic da sedimenti marini in un reattore UASB. Le fasi critiche nel pr...
Divulgazioni
The authors Selene Montserrat García-Solares, Claudio Garibay-Orijel and Claudia Guerrero Barajas submitted (as inventors) in 2012 the application to obtain a patent entitled: “Process for the anaerobic treatment of industrial wastewater with high content of sulfate and chlorinated compounds”. This submission has been approved to the stage of the formatting. The submission has Instituto Politécnico Nacional as the rights owner.
Riconoscimenti
The authors are grateful for the financial support provided by Instituto Politécnico Nacional grants 20120110, 20130399 and 20140239 SIP and also by Instituto de Ciencia y Tecnología del Distrito Federal Mexico (PICS 08-79, ICYT-DF, 2009-2012). Thanks also to CONACYT – Mexico for the graduate scholarship (225806) awarded to Selene Montserrat García-Solares and for the financial support provided by grant 82627.
Materiali
| Name | Company | Catalog Number | Comments |
| trichloroethylene | sigma Aldrich | 251402 | |
| cis-1,2-dichlorotehylene | sigma Aldrich | ||
| trans-1,2-dichloroethylene | sigma Aldrich | D-62209 | |
| vinyl chloride scotty standard | supelco | 1,000 ppm v/v in nitrogen | |
| ethene scotty standard | supelco | 99% purity | |
| pump | Masterflex | Model 7553-75 | |
| spectrophotometer | any | ||
| microcentrifuge | any | ||
| gas tight syringes | any | 100 and 200 microliters | |
| UASB glass reactor | any | under design | |
| gas chromatograph | any | FID detector | |
| capillary column SPB-624 | supelco | ||
| pH meter | any | ||
| viton tubing | Masterflex | ||
| basal medium reagents | any | ||
| trace metals reagents | any | ||
| vitamins solution reagents | any | ||
| sodium sulfate | any | ||
| volatile fatty acids | any | ||
| COD determination kit | HACH | range 0-15,000 mg/L | |
| TOPO-TA cloning kit pCR®4.0 | Invitrogen, US | ||
| S.N.A.P. TM Miniprep Kit | Invitrogen, UK | ||
| Pure link TM Quick Plasmid Miniprep kit | Invitrogen |
Riferimenti
- Lens, P., Esposito, M. V. G., Zandvoort, M. Perspectives of sulfate reducing bioreactors in environmental biotechnology. ReViews Environmental Science and Biotechnology. 1 (4), 311-325 (2002).
- Omil, F., Lens, P., Hulshoff, P., Lettinga, G. Characterization of biomass from a sulfidogenic, volatile fatty acid-degrading granular sludge reactor. Enzyme and MicrobialTechnology. 20, 229-236 (1997).
- Lopes, S. I. C., Wang, X., Capela, M. I., Lens, P. N. L. Sulfate reduction during the acidification of sucrose at pH 5 under thermophilic (55 °C) conditions.II: Effect of sulfide and COD/SO4-2 ratio. Bioresource Technology. 101, 4278-4284 (2010).
- Alfonso, P., Prol-Ledesma, R. M., Canet, C., Melgarejo, J. C., Fallick, A. E. Sulfur isotope geochemistry of the submarine hydrothermal coastal vents of Punta Mita, Mexico. Journal of Geochemical Exploration. 78-79, 301-304 (2003).
- Valdemarsen, T., Kristensen, E. Degradation of dissolved organic monomers and short chain fatty acids in sandy marine sediment by fermentation and sulfate reduction. Geochimica et Cosmochimica Acta. 74, 1593-1605 (2010).
- Quistad, S. D., Valentine, D. L. Anaerobic propane oxidation in marine hydrocarbon seep sediments. Geochimica et Cosmochimica Acta. 75, 2159-2169 (2011).
- Futagami, T., Morono, Y., Terada, T., Kaksonen, A. H., Inagaki, F. Dehalogenation activities and distribution of reductive dehalogenase homologous genes in marine subsurface sediments. Applied and Environmental Microbiology. 75 (21), 6905-6909 (2009).
- U.S. Environmental Protection Agency. List of priority pollutants. Clean Water Methods. , (2014).
- Ozdemir, C., Dursun, S., Karatas, M., Sen, N., Sahinkaya, S. Removal of trichloroethylene (TCE) in upFlow anaerobic sludge blanket reactors (UASB). Biotechnology and Biotechnological Equipment. 21 (1), 107-112 (2007).
- Zhang, Y., Wang, X., Hu, M., Li, P. Effect of hydraulic retention time (HRT) on the biodegradation of trichloroethylene wastewater and anaerobic bacterial community in the UASB reactor. Applied Microbiology and Biotechnology. 99, 1977-1987 (2015).
- . . Standard Methods for the Examination of Water and Wastewater. , (1998).
- Guerrero-Barajas, C., et al. Enhanced sulfate reduction and trichloroethylene (TCE) biodegradation in a UASB reactor operated with sludge developed from hydrothermal vents sediments: process and microbial ecology. International Biodeterioration and Biodegradation. 94, 182-191 (2014).
- Trüper, H. G., Schlegel, H. G. Sulphur metabolism in Thiorhodaceae I. Quantitative measurements on growing cells of Chromatium okenii. Antoine van Leeuwenhoek. 30, 225-238 (1964).
- Gallegos-García, M. G. . Biological processes of sulfate reduction in biofilms for metals precipitation [Ph D thesis]. , (2009).
- Guerrero-Barajas, C., Garibay-Orijel, C., Rosas-Rocha, L. E. Sulfate reduction and trichloroethylene biodegradation by a marine microbial community from hydrothermal vents sediments. International Biodeterioration and Biodegradation. 65, 116-123 (2011).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon