Method Article
L'incorporazione specifici residuo di non canonici amminoacidi in proteine modello utilizzando un
In questo articolo
Riepilogo
An easy-to-use, cell-free expression protocol for the residue-specific incorporation of noncanonical amino acid analogs into proteins, including downstream analysis, is presented for medical, pharmaceutic, structural and functional studies.
Abstract
L'insieme canonica di aminoacidi porta ad una vastissima gamma di funzionalità della proteina. Tuttavia, l'insieme di residui impone ancora limitazioni potenziali applicazioni proteine. L'incorporazione di aminoacidi non canonici può allargare questo ambito. Ci sono due approcci complementari per l'incorporazione di aminoacidi non canonici. Per incorporazione sito-specifica, oltre ai endogeni macchinari traslazionali canonici, una coppia-tRNA-sintetasi-tRNA ortogonali deve essere previsto che non interagisce con i canonici. Di conseguenza, un codone che non è assegnato ad un aminoacido canonica, di solito un codone di stop, è anche necessario. Questa espansione codice genetico consente l'incorporazione di un amminoacido non canonica in un unico, determinato sito all'interno della proteina. Il lavoro qui presentato descrive incorporazione specifici residui in cui il codice genetico viene riassegnato all'interno del sistema di traslazione endogeno. Il meccanismo di traduzione di unccepts l'aminoacido non canonica come surrogato per incorporarlo in luoghi canonicamente previsti, vale a dire, tutte le occorrenze di un amminoacido nella proteina canonica sono sostituite da quella canonica. L'incorporazione di amminoacidi non canonici può modificare la struttura della proteina, causando considerevolmente modificate le proprietà fisiche e chimiche. Non canonici analoghi aminoacidi spesso agiscono come inibitori della crescita delle cellule per gli host di espressione in quanto modificano proteine endogene, limitante nella produzione di proteine vivo. In vivo incorporazione di aminoacidi non canonici tossici in proteine rimane particolarmente impegnativo. Qui, un approccio senza cella per una sostituzione completa di L-arginina dal noncanonical aminoacido L-canavanina è presentato. Si aggira le difficoltà insite di espressione in vivo. Inoltre, un protocollo per preparare proteine bersaglio per l'analisi spettrale di massa è inclusa. E 'dimostrato che la L-lisina può essere sostituito da L-idrossi-lisina,anche se con minore efficienza. In linea di principio, qualsiasi analogico amminoacido noncanonical può essere incorporato con il metodo presentato finché il sistema in vitro traduzione endogena riconosce.
Introduzione
Il codice genetico è universale alla biosfera. Codici per una serie di 20 aminoacidi canonici, che a volte è prorogato di selenocisteina 1 o 2 pirrolisina. È ribosoma che traduce il codice genetico con l'aiuto di tRNA in catene di amminoacidi che si piegano in proteine. I gruppi funzionali degli amminoacidi canonici, in combinazione con modificazioni post-traduzionali, contribuiscono ad una vastissima gamma di funzione proteica 3,4. In linea di massima, limitazioni funzionali a causa del numero limitato di aminoacidi canoniche possono essere superate incorporando ulteriormente, aminoacidi non canonici (NCAAs) che consentono nuove chimiche e nuove funzionalità 3,4.
Ci sono due approcci complementari per l'incorporazione di NCAAs: il sito-specifica o l'incorporazione residui. Il primo metodo comporta notevoli difficoltà tecniche, dal momento che la serie canonica-tRNA-synthetases (RAA) e tRNA devono essere ampliati da un paio ortogonali RAA-tRNA che non devono interagire con le macchine di traduzione endogena. Sulla base di un'attenta progettazione, questo approccio incorpora le NCAAs come mutazioni puntiformi singole nei siti proteina desiderata. Site-specific incorporazione di NCAAs è geneticamente codificato da un codone che non è assegnato ad un acido amino canonica (CAA), di solito un codone di stop 5-9. Questo metodo comporta cambiamenti nella funzione in un determinato sito, piuttosto che su tutta la proteina 10-13.
Al contrario, l'incorporazione specifico residuo si basa sul riconoscimento errato dell'amminoacido noncanonical dalla macchina di traduzione canonica. L'incorporazione verifica a causa della mancanza di specificità di substrato delle RAA. L'incorporazione specifici residuo di NCAAs, costruita sul lavoro di Cohen e colleghi 14, ha portato a importanti applicazioni 3,10, tra i quali bio-ortogonali etichettatura 15-17 di proteineo la struttura delle proteine delucidazione in cristallografia a raggi X 18.
Come AARS naturali in genere preferiscono il loro aminoacido cognate su un NCAA isostructural, efficiente incorporazione vivo specifici residui di solito richiede un host espressione auxotrophic non in grado di sintetizzare l'analogo canonica della NCAA. Le cellule ospiti vengono coltivate in terreno di crescita che fornisce solo una bassa concentrazione del CAA analogo. Il suo esaurimento in combinazione con l'integrazione consecutiva con la NCAA costringe l'host espressione di incorporare la NCAA nella proteina modello a più siti, canonicamente prescritti. In contrasto con l'approccio sito-specifica, questo ha generalmente un profondo impatto su tutta la struttura proteica, determinando modificato notevolmente le proprietà fisiche e chimiche delle proteine 19,20. Tuttavia, la maggior parte del NCAAs sono inibitori della crescita per l'espressione ospitante 3, in cui sono incorporati in molti altri proteins oltre a quelli di interesse durante l'espressione del gene ricombinante. Questo limita chiaramente l'approccio in vivo. L'incorporazione in vivo di aminoacidi che sono tossici o che hanno una forte influenza sulla struttura delle proteine rimane particolarmente impegnativo. Tuttavia, queste molecole sono tra i più promettenti per progettare proteine con funzioni straordinarie.
Un esempio è il, noncanonical, naturale L-canavanina tossica (Can), un analogo di L-arginina (Arg). Essa colpisce e blocca ARG percorsi associati reazione normativi e catalitiche, e la sua presenza nella cellula vivente può portare a morte immediata 3,21-23. La sua incorporazione in proteine in posizioni di arginina può ridurre la stabilità proteica 21-23. A causa della tossicità risultante, espressione di canavanina contenente proteine in Escherichia coli (E. coli) e altri host di espressione comune rimane una sfida. Per queste ragioni, completa in vivo incorporation di Can in tutte le posizioni Arg è stata adeguatamente confermata solo una volta 24, utilizzando un sistema di produzione elaborato singola proteina. Tuttavia, Can è stato proposto come un agente anti-cancro 25-27, e come stimolatore per le malattie autoimmuni nell'uomo 28. Inoltre, è oggetto di diversi studi sulla sua anti-metabolico, antibatterica, antimicotica e proprietà antivirali 25. Queste proprietà sollevano una domanda di efficiente e facile da eseguire metodi per esprimere Può contenenti proteine per farmacia, mediche e studi funzionali.
Anche se molti problemi che sono connessi alla produzione in vivo possono essere aggirate utilizzando sistemi di espressione cell-free, negli approcci vitro specifici residui sono realtà poco esplorata. Sono stati segnalati L'incorporazione privo di cellule specifiche del residuo di un analogo L-triptofano 29 e multipla NCAAs 30. Questi metodi si basano sul altamente efficiente T7 RNA polimerasi. La polimerasi T7 RNA comporta trascrizione batteriofago-like, riducendo in tal modo la funzionalità genetica rispetto alla trascrizione endogeno.
L'incorporazione completa specifica per i residui di Can in una proteina modello in tutte le posizioni Arg è stato recentemente riportato 31, utilizzando un sistema di espressione cell-free 32. Una leggera modifica dello stesso sistema ha permesso l'incorporazione site-specific di diversi analoghi pirrolisina in una proteina modello tramite codone di stop soppressione 33. Il sistema non cellulare impiegato 31 - 33 si basa su un all E. sistema di trascrizione-traduzione coli. Tuttavia, consente l'espressione della proteina nel modo più efficiente in sistemi batteriofago correnti (0,5 - 1 mg / ml di proteina ricombinante) 32, pur mantenendo buona parte della modularità trascrizione-traduzione originale.
In questo lavoro, un protocollo dettagliato è fornito come il residspecifico ue incorporazione di NCAAs può essere realizzato utilizzando questo tutto E. sistema non cellulare coli 32. Inoltre, vengono proposti ulteriori passi per preparare le proteine espresse per la valutazione del caso tramite spettroscopia di massa HPLC-ESI. Per espandere le proprietà di questo sistema privo di cellule, questo lavoro non si riferisce solo alla incorporazione pubblicato Can 31, ma presenta anche nuovi dati relativi al noncanonical L-lisina analogico L-idrossi-lisina.
Il seguente protocollo per l'inserimento specifico per residuo di NCAAs è un adattamento di un protocollo recentemente pubblicato in JoVE 34. Quest'ultimo protocollo descrive come eseguire altamente efficiente libera espressione-cella con amminoacidi standard. Inoltre, esso presenta la preparazione dell'estratto libera cellulari grezzi, la soluzione di aminoacidi, la soluzione di energia stock e il buffer energia utilizzata in questo approccio. Il seguente protocollo concentra su passi modificati rispetto alla precedente pROTOCOLLO al fine di consentire l'integrazione specifica per residuo di NCAAs. pipette calibrate, consigli bassi vincolante pipetta e tubi micro-centrifuga sono raccomandati per la preparazione. Nel seguito, vengono utilizzate le abbreviazioni IUPAC per gli aminoacidi.
Protocollo
Attenzione! Si prega di consultare tutte le schede di sicurezza materiale pertinente (MSDS) prima dell'uso. Molte delle sostanze chimiche utilizzate sono tossicità acuta. Dispositivi di protezione individuale è richiesto (Eyeshield, maschera antipolvere, guanti, camice, pantaloni a figura intera, chiuso-toe scarpe) così come lavorare in una cappa aspirante.
1. Amino Acid Solution Preparazione
- La preparazione della soluzione della NCAA (168 mm)
NOTA: La preparazione della soluzione del NCAA è descritto per l'analogico Arg Can come un esempio. Di conseguenza adattare i valori per gli altri NCAAs.- Posizionare un tubo di 1,5 ml di reazione su una microbilancia. Pesare 46,1 mg di Can all'interno del tubo di reazione per la preparazione di 1 ml di una soluzione 168 mM. Utilizzare un microspatula sterile. Per una miscela racemica di NCAA, raddoppiare la concentrazione della soluzione di riserva.
- Aggiungere 977 ml di DDH sterili 2 O. Completamente vortex fino Can è idissoluzione completa n.
NOTA: Per un volume della soluzione totale di 1 ml, il volume fisico dell'amminoacido disciolto deve essere compensata. Per qualsiasi degli amminoacidi, stima la metà della massa solida in mg come l'aumento di volume corrispondente microlitri (100 mg di solido avrà un volume di 50 microlitri della soluzione) 35. Maggior parte degli amminoacidi possono essere sciolti a questa concentrazione. In caso contrario, ridurre la concentrazione fino a completa dissoluzione. - Direttamente utilizzare la soluzione madre NCAA per la preparazione delle soluzioni di aminoacidi nella sezione 1.2 o Flash congelare in azoto liquido e conservarlo a -20 ° C. ATTENZIONE! Per motivi di sicurezza, indossare un Eyeshield e crio-guanti di essere protetti da liquido spruzzi di azoto.
- Preparazione delle soluzioni di aminoacidi
NOTA: Per la preparazione delle soluzioni di aminoacidi, utilizzare il campionatore aminoacido fornendo gli L-isomeri dei 20 CAAS in magazzino separatasoluzioni (1,5 ml, tamponato con HEPES / KOH, <0,1% NaN3, pH 7,5), ciascuno in concentrazione di 168 mM, fatta eccezione per L-Leucina (140 mM). Per una preparazione fatta in casa di queste soluzioni madre (tamponata con KOH), seguire questo protocollo 35.- Scongelare le soluzioni madre delle 20 CAAS (campionatore aminoacido o preparati secondo a 35) e della NCAA (preparato nella sezione 1.1) a temperatura ambiente.
- Dopo lo scongelamento, spesso vortice le soluzioni madre di riportare in soluzione eventuali aminoacidi precipitati. Come alcuni aminoacidi sono più difficili da sciogliere, incubare su una piastra riscaldante a 37 ° C fino a completa dissoluzione. Cys può non completamente dissolversi. Mettere tutti gli aminoacidi sul ghiaccio, ad eccezione di Asn, Phe e Cys - mantenere questi a RT per evitare la precipitazione.
- Utilizzare i seguenti valori per usare un settimo del kit completo.
NOTA: Scala verso il basso in modo appropriato a lavorare con i volumi più piccoli e per salvare le parti del kit per ulteriori esperimenti. Scala per incorporateviione di NCAAs in proteine modello su larga scala. Per evitare scongelamento frequente, che possa ridurre la stabilità degli aminoacidi, un'aliquota le singole soluzioni madre aminoacidi in volumi di 200 microlitri. Questo volume dell'aliquota 200 pl e dei volumi aliquota prelevata per passo 1.2.4.1 conto delle perdite dovute alla dispensazione. - In primo luogo, preparare una soluzione di master mix di aminoacidi che sarà diviso in fase 1.2.4.3 per finalizzare la preparazione di 3 soluzioni di aminoacidi diversamente composte (sezioni 1.2.5 - 1.2.7). In queste soluzioni, concentrare tutte aminoacidi a 6 mM, tranne Leu (5 mM).
- Trasferimento 1.4 ml di DDH 2 O sterile in una provetta da centrifuga da 15 ml. Mettilo sul ghiaccio. Aggiungere 175 ml di ogni soluzione di aminoacidi magazzino. Aggiungere uno dopo l'altro, fatta eccezione per la soluzione stock di CAA (ad esempio, Arg) per essere sostituito da NCAA (ad esempio, Can). Completamente vortice dopo ogni aggiunta e mettere la soluzione di nuovo sul ghiaccio.
NOTA:Leu è a 5 mM in 3 soluzioni aminoacidi diversamente composte, rispetto al 6 mM per altri amminoacidi. La concentrazione ridotta non riduce l'efficienza di espressione. Scaling fino a 6 mm è adatto pure. - Trasferire le soluzioni di aminoacidi azionari nel seguente ordine di evitare la precipitazione: Ala, Arg, Asn, Asp, Gln, Glu, Gly, il suo, Ile, Lys, Met, Phe, Pro, Ser, Thr, Val, Trp, Tyr, Leu, e Cys. Ricordarsi di non aggiungere la soluzione di riserva del CAA (ad esempio, Arg), che è analoga alla NCAA (ad esempio, può). Infine, a fondo vortice. Incubare a 37 ° C per rendere la soluzione più chiara possibile.
- Split questa soluzione master mix di aminoacidi in tre volumi uguali di 1,35 ml. Trasferire ciascuno dei volumi divisi in provette da 1,5 ml. Tenerli in ghiaccio.
- Trasferimento 1.4 ml di DDH 2 O sterile in una provetta da centrifuga da 15 ml. Mettilo sul ghiaccio. Aggiungere 175 ml di ogni soluzione di aminoacidi magazzino. Aggiungere uno dopo l'altro, fatta eccezione per la soluzione stock di CAA (ad esempio, Arg) per essere sostituito da NCAA (ad esempio, Can). Completamente vortice dopo ogni aggiunta e mettere la soluzione di nuovo sul ghiaccio.
- Preparare una soluzione di aminoacidi che consiste di tutte le 20 CAAS ad una concentrazione di 6 mM ciascuna, tranne Leu che è 5 mM. Per il primo volume of 1,35 ml, a seguito della scissione in fase 1.2.4.3, aggiungere 50 ml di soluzione madre mM 168 del CAA (ad esempio, Arg), che è analoga alla NCAA (ad esempio, Can). Completamente vortice.
- Mettere di nuovo sul ghiaccio. Un'aliquota questo 1,4 ml di soluzione in volumi di 16 ml in provette di reazione. Si noti che questo volume soluzione porta circa a 85 aliquote. Etichettare queste aliquote "+ Caa" (ad esempio, + Arg).
- Flash congelare le aliquote in azoto liquido e conservare a -80 ° C. ATTENZIONE! Per motivi di sicurezza, indossare un Eyeshield e crio-guanti per essere protetti dagli spruzzi di azoto liquido.
- Preparare una soluzione di aminoacidi che si compone di 19 Caas eccezione CAA (ad esempio, Arg) che è analogo al ncaa (ad esempio, Can). Aggiungere ogni amminoacido ad una concentrazione di 6 mM, tranne Leu (5 mM).
- Aggiungere 50 ml di DDH 2 O sterile al secondo volume di 1,35 ml, come risultato della scissioneal punto 1.2.4.3. Completamente vortice e mettere di nuovo sul ghiaccio. Un'aliquota questo 1,4 ml di soluzione in volumi di 16 ml in provette di reazione. Si noti che questo volume soluzione porta circa a 85 aliquote. Etichettare queste aliquote "- Caa" (ad esempio, - Arg).
- Flash congelare le aliquote in azoto liquido e conservare a -80 ° C. ATTENZIONE! Per motivi di sicurezza, indossare un Eyeshield e crio-guanti per essere protetti da spruzzi di azoto.
- Preparare una miscela di aminoacidi che contiene 19 CAAS e la NCAA (ad esempio, Can) che sostituisce quella canonica (per esempio, Arg). Aggiungere ogni amminoacido ad una concentrazione di 6 mM, tranne Leu (5 mM). Per l'ultimo volume 1,35 ml, come risultato della divisione nella fase 1.2.4.3, aggiungere 50 ml di soluzione madre mM di NCAA 168 (ad esempio, Can). Etichettarlo "+ NCAA" (ad esempio, + Can). Completamente vortice e mettere di nuovo sul ghiaccio.
- Un'aliquota questo 1,4 ml di soluzione in volumi di 16 & #181; l in tubi di reazione. Si noti che questo volume soluzione porta circa a 85 aliquote. Etichettare queste aliquote "+ NCAA" (ad esempio, + CAN).
- Flash congelare le aliquote in azoto liquido e conservare a -80 ° C. ATTENZIONE! Per motivi di sicurezza, indossare un Eyeshield e crio-guanti per essere protetti da spruzzi di azoto.
NOTA: I 16 volumi aliquote microlitri utilizzati nelle fasi 1.2.5.1, 1.2.6.1 e 1.2.7.1 sono leggermente superiori rispetto a quelle richieste per tenere conto delle perdite dovute alla dispensazione.
2. Buffer Preparazione Energia
NOTA: Ogni lotto di greggio estratto è unica e richiede concentrazioni di Mg e K-glutammato 34 ottimizzato. Il volume dell'aliquota estratto grezzo dipende dalla concentrazione proteica 34. Utilizzare il modello di calcolo previsto (Supplemental Materiale 1) per diversi valori. Trova ulteriori istruzioni in Supplemental Materiale 1 figura leggenda, a fornire i chiarimentivorazio come impiegare questo modello.
- Preparare e conservare a -80 ° C la soluzione 14x energia e aliquote estratto grezzo secondo il protocollo non modificato 34. Calibrare il estratto grezzo a seconda delle concentrazioni di Mg e K-glutammato per ottimizzare l'efficienza di espressione 34.
NOTA: La composizione finale del 14x soluzione di energia è: 700 mm HEPES (pH 8), 21 mm ATP, 21 mM GTP, 12,6 millimetri CTP, 12,6 millimetri UTP, 2,8 mg / ml tRNA, 3,64 mM CoA, 4,62 mM NAD, 10.5 mM cAMP, 0,95 mM acido folinico, 14 mm spermidina, 420 mm a 3-PGA. - Scongelare su ghiaccio la soluzione 100 mM Mg-glutammato magazzino, soluzione madre 3 M K-glutammato, soluzione 14x di energia e il 40% PEG-8000 per preparare il master mix. Tenerli in ghiaccio.
- Mescolare 9.18 ml di soluzione madre 100 mM Mg-glutammato, 3,06 ml di soluzione madre 3 M K-glutammato, 21.86 ml di 14x soluzione di energia, 15,3 ml di 40% PEG-8000 e 1,6 ml sterili DDH 2 O in un tubo di reazione. Completamente vortice questo alberoer mescolare dopo ogni aggiunta, e tenerlo in ghiaccio.
- Aliquota del master mix (51 ml) in volumi di 16 ml (3 aliquote) in tubi di reazione. vortice Spesso il master mix durante la suddivisione in aliquote. Flash congelare le aliquote in azoto liquido.
NOTA: Il volume aliquota 16 microlitri così come il volume master mix sono leggermente superiori rispetto a quelle richieste per tenere conto delle perdite dovute alla dispensazione. - Utilizzare un filtro per raccogliere i tubi del buffer di energia. Conservare le provette a -80 ° C. ATTENZIONE! Indossare un Eyeshield e crio-guanti per essere protetti da spruzzi di azoto.
3. preparazione ed esecuzione delle reazioni cell-free per incorporazione senza residui specifici di NCAAs
- In primo luogo, preparare la soluzione di DNA vettore in DDH 2 O.
- Per l'espressione della proteina altamente efficiente, utilizzare il vettore di espressione pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 32. Clonare il gene che codifica per la proteina modello in questo vettore 36,37 </ Sup>.
NOTA: In alternativa, utilizzare altri promotori che sono riconosciuti da σ 70 pure, ma è da notare che l'efficienza di espressione può essere ridotto. - Trasformare 37,38 il vettore in E. coli ceppo KL 740 32 (Yale CGCS #: 4382), Purificare amplificato vettore DNA 37,39 e quantificare la concentrazione della soluzione di DNA 40-42. Conservare la soluzione di DNA a -20 ° C o direttamente usarlo per la preparazione reazione delle cellule-tassa (passi 3.4.1, 3.4.2 e 3.4.3).
- Per l'espressione della proteina altamente efficiente, utilizzare il vettore di espressione pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 32. Clonare il gene che codifica per la proteina modello in questo vettore 36,37 </ Sup>.
- Calibrare l'efficienza libera espressione di cellule a seconda della concentrazione vettore costrutto utilizzato secondo il protocollo non modificato 34. Utilizzare la concentrazione ottimale che porta a più alti rendimenti di proteine per la preparazione reazione acellulare (passi 3.4.1, 3.4.2 e 3.4.3).
NOTA: La preparazione di reazioni acellulari è esemplificata utilizzando una soluzione di DNA vettore stock 90 nM che conduce ad una concentrazione vettoriale finale di 10 nM nel libero cellulereazione e segue i valori ottimali sopra di Mg e K-glutammato e il volume dell'estratto aliquota. Utilizzare il modello di calcolo per valori diversi. - Scongelare su ghiaccio 3 aliquote greggio estratto ciascuno dei 30 volumi microlitri (preparato secondo il protocollo non modificato 34), 1 amino soluzione di acido aliquota etichettato "+ Caa" (ad esempio, + Arg), 1 amino soluzione di acido aliquota etichetta "- Caa" (ad esempio, , - Arg) e 1 amino soluzione di acido aliquota etichettato "+ NCAA" (ad esempio, + CAN) (preparato in sezioni 1.2.5 - 1.2.7), 3 tamponi di energia aliquote (preparato in sezione 2) e la soluzione di DNA vettore ( preparato in sezione 3.1).
NOTA: L'estratto grezzo è leggermente viscosa e può contenere bolle d'aria. Rimuovere le bolle d'aria per centrifugazione a 10.000 xg per 30 sec a 4 ° C. Mettere aliquote estratto grezzo di nuovo sul ghiaccio. - Preparare 3 reazioni privi di cellule diversamente composte (ogni 90 ml di volume finale) mescolando estratto grezzo (33,33%), l'energiatampone (16,67%), 1 dei 3 aliquote diversamente composti aminoacidi soluzione (16,67%) e soluzione di DNA vettoriale. Facoltativamente, aggiungere ulteriori biomolecole (DNA, proteine, tRNA, ecc), ma in modo appropriato ridurre il volume di DDH 2 O.
- Preparare la reazione senza cella di riferimento (90 ml) che esprime la proteina modello non modificato.
- Aggiungere 15 ml di buffer di energia, 15 ml di soluzione di acido amino aliquota etichettato "+ Caa" (ad esempio, + Arg), 10 ml di 90 soluzione di DNA vettore Nm e 20 ml di DDH sterili 2 O ai 30 ml di greggio estratto. Mescolare pipettando su e giù e delicatamente vortex dopo l'aggiunta di ciascun ingrediente.
- Aliquota del 90 microlitri di reazione cell-free in 15 volumi uguali di 6 ml. Trasferire ciascuno dei 15 volumi in un tubo di reazione separata. Chiudere i tubi e rimetterli sul ghiaccio. Etichettare tutti i tubi di reazione come "CFR (+ CAA)" (ad esempio, CFR (+ Arg)).
- Preparare la reazione negativa di controllo cell-free (90 ml), in cui né la Caa (ad esempio, Arg), né l'analogico non canonica (per esempio, Can) vengono aggiunti.
- Aggiungere 15 ml di tampone di energia, 15 ml di soluzione di un'aliquota di aminoacidi etichettati "- Caa" (ad esempio, - Arg), 10 ml di 90 soluzione di DNA vettore Nm e 20 ml di DDH sterile 2 O ai 30 ml di greggio estratto. Mescolare pipettando su e giù e delicatamente vortex dopo l'aggiunta di ciascun ingrediente.
- Aliquota del 90 microlitri di reazione cell-free in 15 volumi uguali di 6 ml. Trasferire ciascuno dei 15 volumi in un tubo di reazione separata. Chiudere i tubi e rimetterli sul ghiaccio. Etichettare tutti i tubi di reazione come "CFR (-cAA)" (ad esempio, CFR (-Arg)).
- Preparare la reazione acellulare (90 ml), che si suppone residuo specificamente incorporare la NCAA (ad esempio, Can) nella proteina modello di espresso.
- Aggiungere il 15microlitri di tampone di energia, 15 microlitri della soluzione di aminoacidi aliquota etichettato "+ NCAA" (ad esempio, + Can), 10 ml di soluzione di DNA 90 nM e 20 ml di DDH 2 O sterile ai 30 ml di estratto grezzo. Mescolare pipettando su e giù e delicatamente vortex dopo l'aggiunta di ciascun ingrediente.
NOTA: L'efficienza espressione può essere ridotta rispetto all'espressione con amminoacidi standard. Se necessario, semplicemente scalare il volume di reazione acellulare. - Aliquota del 90 microlitri di reazione cell-free in 15 volumi uguali di 6 ml. Trasferire ciascuno dei 15 volumi in un tubo di reazione separata. Chiudere i tubi e rimetterli sul ghiaccio. Per un aumento dei volumi di reazione, di conseguenza aliquota in ulteriori volumi di 6 ml. Etichettare tutti i tubi di reazione come "CFR (+ NCAA)" (ad esempio, CFR (+ Can)).
- Aggiungere il 15microlitri di tampone di energia, 15 microlitri della soluzione di aminoacidi aliquota etichettato "+ NCAA" (ad esempio, + Can), 10 ml di soluzione di DNA 90 nM e 20 ml di DDH 2 O sterile ai 30 ml di estratto grezzo. Mescolare pipettando su e giù e delicatamente vortex dopo l'aggiunta di ciascun ingrediente.
- Preparare la reazione senza cella di riferimento (90 ml) che esprime la proteina modello non modificato.
- Incubare tutte le provette a 29 ° CO / N.
NOTA: volumi di reazione solo piccole consentono adeguati diff ossigenousione nella reazione che è essenziale per l'espressione di proteine altamente efficiente. Volumi di reazione superiori a 10 ml richiedono l'ossigenazione attiva attraverso l'agitazione 34. Per grandi volumi, dividere la reazione in volumi di dimensioni inferiori a 15 ml. - Dopo espressione cell-free, piscina tutto identico composto reazioni senza Dividi cella di 6 ml. In primo luogo, piscina tutti i 15 reazioni senza Dividi cella di 6 ml etichettate "CFR (+ CAA)" (ad esempio, CFR (+ Arg)). Poi, piscina tutti i 15 reazioni senza Dividi cella di 6 ml etichettate "CFR (-cAA)" (ad esempio, CFR (-Arg)). Infine, piscina tutti i 15 reazioni senza Dividi cella di 6 ml etichettate "CFR (+ NCAA)" (ad esempio, CFR (+ Can)). Per un aumento dei volumi di reazione, di conseguenza piscina ulteriori volumi di 6 ml.
- Controllare il livello di espressione della proteina modello in tutti e tre pool, diversamente composto reazioni privi di cellule eseguendo denaturanti SDS-PAGE 43,44 (punto 4.1).
NOTA: Utilizzare questa metho d per una valutazione preliminare dell'esperimento incorporazione. - Preparare le proteine modello espresse cell-free per un'analisi appropriata tramite spettroscopia di massa HPLC-ESI (paragrafo 4.4). In primo luogo, purificarli 45 (paragrafo 4.2). Infine, lo scambio di buffer (paragrafo 4.3) per evitare alto rumore di fondo durante la spettroscopia di massa.
NOTA: Le sezioni 3.4.1, 3.4.2 e 3.4.3 portare a condizioni di reazione cell-free tipici 34: 8,9-9,9 mg / ml di proteine (dal greggio estratto), 4,5-10,5 mM Mg-glutammato, 40-160 mM K-glutammato, 1 mM di ciascun amminoacido tranne leucina, 0,83 mM leucina, 50 mM HEPES, 1,5 mM ATP e GTP, 0,9 mM CTP e UTP, 0,2 mg / ml tRNA, 0,26 mM CoA, 0,33 mM NAD, 0,75 mM cAMP , 0,068 mM acido folinico, 1 mM spermidina, 30 mm 3-PGA, 2% PEG-8000 e 10 nM pBEST-OR2-OR1-Pr-UTR1-gene_of_model_protein-T500. Se lo si desidera, una diversa procedura di preparazione reazione privo di cellule può essere condotto che conduce alle condizioni di reazione di cui sopra.
- SDS-PAGE delle reazioni cell-free
NOTA: Eseguire denaturanti SDS-PAGE per un'analisi rapida e preliminare delle proteine espresse, senza alcuna ulteriore purificazione o di estrazione, ed eseguire le seguenti operazioni.- Scongelare lo standard di proteine in ghiaccio. Assicurarsi che consiste di proteine con pesi molecolari simili alle proteine modello espressa per la sua localizzazione sul gel (fase 4.1.13).
NOTA: Qui, lo standard utilizzato fornisce proteine su una vasta gamma di pesi molecolari (miosina: 212 kDa, maltosio-binding-protein-β-galattosidasi: 158 kDa, β-galattosidasi: 116 kDa, fosforilasi b: 97 kDa, albumina sierica : 66 kDa, deidrogenasi glutammico: 56 kDa, maltosio-binding proteine: 43 kDa, tioredossina reduttasi: 35 kDa, isomerase triosephosphate: 27 kDa, inibitore della tripsina: 20 kDa, lysozyme: 14 kDa, aprotinina: 7 kDa, l'insulina A: 3 kDa, catena B: 2 kDa). - Dal momento che le reazioni privi di cellule sono leggermente viscoso a causa della elevata concentrazione di proteine, diluirli con DDH sterile 2 O 5 - 10 volte prima di SDS-PAGE per garantire una migrazione gel adeguata e per evitare la saturazione dopo la colorazione. Al fine di non perdere troppo campione, diluire 1 ml di reazione priva di cella in 4 ml di DDH sterili 2 O. Preparare la diluizione in provette, fondo vortice e poco far girare il liquido verso il basso con un mini centrifugare per 2 - 3 sec a 2.000 x g.
- Aggiungere 5 ml di tampone 2x carico di 5 ml di reazione privo di cellule diluito. Completamente vortice e spin down con un mini centrifugare per 2 - 3 sec a 2.000 x g.
NOTA: Qui, dopo l'aggiunta, le biomolecole vengono sciolti in 62.5 mM Tris / Cl -, 10% glicerolo, 2% SDS, e 0,0025% blu di bromofenolo a pH 6,8, una composizione tipica. L'uso di altri coloranti di carico che portano a leggermente diversa diluizione colli operativi possono essere adatti pure. - Accuratamente vortice standard proteine e trasferire 15 microlitri di in un tubo di reazione.
NOTA: A seconda delle dimensioni gel e per altri standard di proteine, il volume consigliato possono differire da quanto sopra. - Mettere le provette di reazione in un blocco di riscaldamento. Controllare se i coperchi dei tubi siano chiusi. Calore a 95 - 100 ° C per 3 - 5 min. Fate questo per denaturare le proteine e per consentire SDS-wrapping intorno alla spina dorsale delle proteine.
NOTA: Alcuni standard di proteine non deve essere riscaldata, vedere le istruzioni del fornitore. - Nel frattempo, trasferire il tampone di corsa nella camera di elettroforesi su gel. Il contenuto di 1x tampone di corsa è di 25 mM Tris, 192 mM glicina, 0,1% SDS a pH 8,3 (con HCl).
- Fissare la prefabbricazione 4 - 20% gradiente Tris-glicina-SDS gel (10 cm x 10 cm x 1 mm) alla camera di elettroforesi. Rimuovere il pettine e lavare i pozzetti con una siringa carica di tampone di corsa.
NOTA: La concentrazione del gel strongly dipende dalle dimensioni e la natura della proteina modello di espresso. La concentrazione superiore separa la E. proteine estratto grezzo coli e bassa molecolare proteine modello peso. gel auto-cast sono adatti pure. - Rimuovere i tubi dal blocco di riscaldamento. Spin il liquido verso il basso con un mini centrifugare per 2 - 3 sec a 2.000 x g. Poco vortice e ripetere filatura premuto per 2-3 secondi a 2.000 x g.
- Trasferire i 15 ml di standard di proteine (24 - proteina 48 mg) e 10 ml (11-22 mg di proteina) di ciascun campione nei pozzetti e iniziare l'elettroforesi. Qui, SDS-PAGE è effettuata a 125 V e 20 - 40 mA per circa 90 min, un protocollo tipico.
- estrarre delicatamente il gel dalla cassetta. Trasferire nella soluzione di fissaggio (50% metanolo, acido acetico 10%, 40% DDH 2 O) per 30 min. ATTENZIONE metanolo è tossico per inalazione e contatto con la pelle. Indossare guanti protettivi e di lavoro sotto una cappa aspirante.
- Trasferire il gel in soluzione colorante (0,025% Coomassie Brilliant Blue G-250, il 10% di acido acetico, il 90% DDH 2 O). Macchia per 60 min.
- Trasferire il gel in soluzione decolorante (acido acetico al 10%, 90% DDH 2 O) 60 - 120 min.
NOTA: fissaggio, colorazione e decolorazione fortemente dipendono dalle dimensioni e la natura della proteina espressa. Questo protocollo si applica a una vasta gamma di proteine piuttosto piccoli pesi molecolari 46. - Spostare il gel su un foglio trasparente opaco su sfondo bianco che genera un contrasto appropriato per le bande proteiche macchiati.
- Scongelare lo standard di proteine in ghiaccio. Assicurarsi che consiste di proteine con pesi molecolari simili alle proteine modello espressa per la sua localizzazione sul gel (fase 4.1.13).
- Purificazione dei suoi tag-45 proteine modello dalle reazioni cell-free
NOTA: per la purificazione di proteine, esistono diversi metodi che forniscono risultati simili. Questo protocollo purifica proteine modello espresse privi di cellule che hanno un polyhistidine-tag C-terminale (His-tag). Esso utilizza un kit di purificazione adatto per proteine che sono EXPressed nei piccoli volumi di reazione di espressione proteica cell-free. È identica per la purificazione di proteine native o modificati modello. Così, è generalmente descritto sulla base di un'unica reazione privo di cellule.- Per un adeguato HPLC-ESI analisi spettroscopica di massa, estrarre e purificare le proteine modello dalla reazione acellulare ed eseguire le seguenti operazioni.
- Miscelare volumi uguali di 90 - 150 ml di His-Binding Buffer e 90 - 150 ml di reazione cell-free. Pipetta su e giù, seguita da vortexando gentilmente.
NOTA: Usando il suo vincolante tampone è raccomandato. Tuttavia, mezzo di reazione acellulare può essere un materiale di partenza finché la proteina modello è solubile pH è tra 7,5 - 8, imidazolo / sua concentrazione è <10 mM, concentrazione di forti agenti riducenti è <15 mM e non metallurgica agenti chelanti sono presenti. Se il volume di miscela supera i 300 ml, dividerlo in volumi uguali e aliquota tesi volumi in diverse rTubi eaction per le seguenti fasi di purificazione. - Preparare il sistema a colonna. Accuratamente vortice soluzione di riserva del gel sua affinità fino a quando la resina gel è completamente sciolto. Trasferire 250 ml di resina gel nella colonna. Utilizzare una punta della pipetta 1 ml per la resina gel viscoso. Porre la colonna in un tubo di raccolta.
- Centrifuga tubo colonna / collezione per 5 - 10 secondi a 13.000 - 15.000 x g. Assicurarsi che la resina del gel è completamente scarica. Se no, prolungare il tempo di centrifugazione per ulteriori 5 - 10 sec, ma attenzione che il gel di affinità non è over-essiccato. Di conseguenza prolungare il tempo di centrifugazione a passi 4.2.1.5, 4.2.1.7 e 4.2.1.9.
NOTA: La resina gel è completamente scarica, se diventa rigida e non surnatante resta in cima. - Trasferire 150 - 300 ml di tampone di reazione gratuita Cell-/ His-legame alla colonna. Se il volume della miscela supera il volume consigliato, dividere su più colonne di spin. Risospendere la resina gel da frEquent maschiatura e vortexando gentilmente durante un tempo di incubazione di almeno 2 min. Per i volumi superiori a 200 ml, incubare per ulteriori 1 - 2 min.
NOTA: tempo di incubazione sufficiente è essenziale per il legame delle proteine modello alla resina gel. - Centrifuga tubo colonna / collezione per 5 - 10 secondi a 13.000 - 15.000 x g. Gettare il flow-through e posizionare la colonna indietro nel tubo di raccolta.
- Aggiungere 250 ml di His-tampone di lavaggio. Risospendere la resina gel toccando e gentile vortex.
- Centrifuga tubo colonna / collezione per 5 - 10 secondi a 13.000 - 15.000 x g. Gettare il flow-through e posizionare la colonna indietro nel tubo di raccolta. Ripetere il passaggio 4.2.1.6 e centrifugare di nuovo per 5 - 10 secondi a 13.000 - 15.000 x g.
- Porre la colonna in un tubo di reazione da 1,5 ml standard. Aggiungere 150 ml di tampone di eluizione e risospendere la resina gel toccando e gentile vortex. Ridurre il volume di 100 l per una maggiore modello purificata proteine concentrazioni dopo eluizione nella fase successiva 4.2.1.9.
NOTA: L'abbondanza totale di proteine modello purificata può essere ridotta a causa di possibili eluizione incompleta di tutte le proteine resina-bound gel, se il volume tampone di eluizione viene ridotto. - Centrifuga tubo della colonna / reazione per 5 - 10 sec a 13.000 - 15.000 xg per diluire la proteina purificata nel tampone di eluizione. Se dividere prima, in comune tutte le soluzioni in un'unica soluzione madre.
- Prima di eseguire lo scambio di buffer, determinare la concentrazione di proteine usando metodi standard, ad esempio, il dosaggio Bradford 47,48.
NOTA: Per un adeguato spettroscopia di massa HPLC-ESI (paragrafo 4.4), usare concentrazioni di proteine ottimali che di solito sono circa 0,5 mg / ml con volumi richiesti di 20 - 50 ml. Se la concentrazione è inferiore a 0,2 mg / ml, concentrare la soluzione proteica dopo lo scambio buffer utilizzando un concentratore a vuoto filatura. In questo caso, prima di leggere la sezione 4.3.8 per adattare in modo appropriato il buff scambioER.
- Miscelare volumi uguali di 90 - 150 ml di His-Binding Buffer e 90 - 150 ml di reazione cell-free. Pipetta su e giù, seguita da vortexando gentilmente.
- Per un adeguato HPLC-ESI analisi spettroscopica di massa, estrarre e purificare le proteine modello dalla reazione acellulare ed eseguire le seguenti operazioni.
- Cambio buffer per la spettroscopia di massa HPLC-ESI
NOTA: Sostituire tampone di eluizione His-tag per evitare elevato rumore di fondo in analisi spettroscopica di massa a causa dell'alta concentrazione di imidazolo (> 150 mM) e altri sali come NaH 2 PO 4 (> 300 mM) o NaCl (> 50 mM) 49. Il seguente protocollo di scambio di buffer utilizza un sistema di colonne di spin gel filtrazione prehydrated. E 'identico eseguito per proteine native o modificate modello. Così, è generalmente descritto sulla base di un'unica reazione privo di cellule. Si noti che ci sono altri metodi di scambio di buffer facili da eseguire, ad esempio, cartucce mini-dialisi.- Preparare 100 ml di tampone di conservazione proteine (50 mM Tris-Cl pH 8, 100 mM NaCl e 10% glicerolo). Pesare in un autoclave da 100 ml 605,7 mg Tris-Base, 584,4 mg di NaCl e aggiungere 10 ml di glicerolo. Riempire fino a circa 80 ml e regolare il pH a 8 con unggiunta NaOH. controlla costantemente il valore del pH con un pH-metro. Infine, riempire fino al segno 100 ml.
NOTA: utilizzare questo buffer per stabilizzare una vasta gamma di proteine modello e non disturbare analisi spettroscopica di massa, purché HPLC viene eseguita prima della misura spettroscopica di massa. Tuttavia, a seconda della natura della proteina espressa modello, utilizzare altri buffer che potrebbe essere meglio adatti. Se la proteina bersaglio non è stabile a pH 8, regolare il pH di conseguenza. Non utilizzare concentrazioni più elevate di glicerolo. - Riscaldare le colonne di rotazione di filtrazione di gel per almeno 15 minuti a temperatura ambiente.
- Risospendere il gel prehydrated picchiettando leggermente o vortex e rimuovere le bolle d'aria.
- In primo luogo togliere il tappo di fondo, e poi prendere il tappo superiore di distanza. Porre la colonna in un tubo di lavaggio (almeno 2 ml). Trasferire il tubo della colonna / lavaggio nella centrifuga. Ogni colonna possiede un segno di orientamento. Centrifugare a 1000 xg per 2 minuti per rimuovere il tampone di conservazione gel. Discoard flow-through.
NOTA: Prestare attenzione a procedere in questo modo. Assicurarsi che la colonna ha lo stesso orientamento in tutte le ulteriori fasi di centrifugazione. - Aggiungere fino a 400 ml di tampone di stoccaggio proteine. Centrifugare per 2 min a 1000 x g. Ripetere questo per caricare completamente il buffer di memorizzazione delle proteine in gel. Aggiungere con cautela fino a 100 microlitri della soluzione della proteina purificata modello. Pipettare direttamente sul centro del letto gel.
NOTA: volumi delle soluzioni di proteine purificate, in particolare di proteine modificate, potrebbe essere superiore. Split tali soluzioni su più colonne di scambio di buffer. Le proteine devono avere pesi molecolari superiori a 5 kDa per questo tipo di scambio buffer. - Porre la colonna in un tubo di raccolta e centrifugare per 2 min a 1000 x g.
NOTA: Questo passaggio conduce alla diluizione della proteina purificata modello nel buffer di memorizzazione proteine. Se dividere prima, in comune tutte le soluzioni in un'unica soluzione madre. - esimo Flash freezesoluzione proteica e in azoto liquido e conservare a -80 ° C o direttamente analizzare tramite HPLC-ESI spettroscopia di massa 50 (paragrafo 4.4). ATTENZIONE! Indossare un Eyeshield e crio-guanti per essere protetti da spruzzi di azoto.
- Eseguire i seguenti passaggi del protocollo di concentrare la soluzione proteica utilizzando un concentratore a vuoto di filatura che evapora con attenzione il solvente 51.
NOTA: Questi passaggi sono necessari se la concentrazione di proteine è troppo bassa per la spettroscopia di massa HPLC-ESI (<0,2 mg / ml). La concentrazione di proteine volume della soluzione e il modello è più o meno la stessa prima e dopo lo scambio di buffer. Il processo di concentrazione è descritto per mezzo del seguente esempio: Dopo purificazione His-tag, la proteina modello è disciolto in 100 microlitri suo tampone di eluizione (passo 4.2.1.9) e ha una concentrazione di 0,07 mg / ml (step 4.2.1.10) .- Per garantire che la concentrazione di proteine e il volume della soluzione sono dopo evaporazione aalmeno 0,2 mg / ml e 20 ml, rispettivamente diluire, prima scambio di buffer, il buffer di memorizzazione proteine preparato al punto 4.3.1 almeno tre volte, ma al massimo di cinque volte. Altrettanto evaporare seguente almeno due terzi (66,67 ml) o al massimo quattro quinti (80 microlitri).
NOTA: La diluizione del tampone di conservazione proteine prima dello scambio buffer è essenziale tener conto che i valori di concentrazione di questo buffer (fase 4.3.1) sono ancora realizzati nonostante evaporazione. Dopo la diluizione del buffer di stoccaggio di proteine, prima di eseguire lo scambio di buffer (passi 4.3.2 - 4.3.6) prima di eseguire il seguente passi 4.3.8.2 - 4.3.8.4. - Dopo lo scambio di buffer, mettere i completi 100 ml di soluzione proteica modello in un tubo aperto nel concentratore di vuoto di filatura.
NOTA: La proteina modello è disciolto nel tampone di conservazione proteine diluito. Inclinare verso il centro di rotazione per evitare liquido perdita durante la rotazione. Assicurarsi che un vuoto aperto dello stesso volume diH 2 O è perfettamente simmetrico per equilibrare il sistema di filatura. - Chiudere il coperchio del concentratore e inizia la rotazione. Per assicurare la stabilità della proteina, si concentra a RT oa basse temperature fino a 30 ° C.
NOTA: Il concentratore utilizzato accelera automaticamente a 170 xg e stabilisce il vuoto. Nella seguente accelera la sua velocità massima di 240 x g. - Spesso interrompe il processo di concentrazione di rilevare il volume del liquido. Fermare la concentrazione, quando il volume soluzione rimanente è compreso tra 20 ml e 33 ml.
NOTA: Di conseguenza adattare le misure 4.3.8.1 - 4.3.8.4 per altri volumi di soluzione (step 4.2.1.9) e altre concentrazioni di proteine (step 4.2.1.10). Flash congelare la soluzione proteica concentrata in azoto liquido e conservare a -80 ° C, o direttamente analizzarlo tramite HPLC-ESI spettroscopia di massa 50. ATTENZIONE! Indossare un eyeshield e crio-guanti per essere protetti da spruzzi di azoto.
- Per garantire che la concentrazione di proteine e il volume della soluzione sono dopo evaporazione aalmeno 0,2 mg / ml e 20 ml, rispettivamente diluire, prima scambio di buffer, il buffer di memorizzazione proteine preparato al punto 4.3.1 almeno tre volte, ma al massimo di cinque volte. Altrettanto evaporare seguente almeno due terzi (66,67 ml) o al massimo quattro quinti (80 microlitri).
- Preparare 100 ml di tampone di conservazione proteine (50 mM Tris-Cl pH 8, 100 mM NaCl e 10% glicerolo). Pesare in un autoclave da 100 ml 605,7 mg Tris-Base, 584,4 mg di NaCl e aggiungere 10 ml di glicerolo. Riempire fino a circa 80 ml e regolare il pH a 8 con unggiunta NaOH. controlla costantemente il valore del pH con un pH-metro. Infine, riempire fino al segno 100 ml.
- HPLC-ESI analisi di spettrometria di massa 50 di proteine modello
- Eseguire la separazione HPLC 5 - soluzione 15 microlitri proteina (preparato nella sezione 4.3) su una colonna C5 fase inversa (3 um, 100 x 2,1 mm) con un gradiente di 20% - 90% acetonitrile e 0,1% di acido formico over 25 min e successiva spettrometria di massa ESI con rilevamento su un analysator di massa a tempo di volo la portata di 300 - 3.000 m / z.
NOTA: A seconda della natura della proteina espressa modello, usare altri solventi o colonne che potrebbero essere meglio adatti. - Deconvolute misurato spettri di massa utilizzando il software appropriato 52 per calcolare masse zero carica.
- Eseguire la separazione HPLC 5 - soluzione 15 microlitri proteina (preparato nella sezione 4.3) su una colonna C5 fase inversa (3 um, 100 x 2,1 mm) con un gradiente di 20% - 90% acetonitrile e 0,1% di acido formico over 25 min e successiva spettrometria di massa ESI con rilevamento su un analysator di massa a tempo di volo la portata di 300 - 3.000 m / z.
Risultati
Questo protocollo guida attraverso l'incorporazione specifici residuo privo di cellule di NCAAs in proteine modello. Essa propone SDS-PAGE per una valutazione preliminare dell'esperimento costituzione e ulteriori passi per preparare le proteine modello per un adeguato HPLC-ESI analisi spettroscopica di massa.
Qui, risultati rappresentativi dell'incorporazione specifico residuo privo di cellule del analogico Arg Can, nonché la Lys analogico L-idrossi-lisina (Hyl) sono presentati. Le diverse soluzioni di aminoacidi, il buffer di energia, il DNA codificante vettoriale per il modello proteine e reazioni privi di cellule sono preparati come descritto sopra. La reazione libera-cella di riferimento è fornito con la soluzione di aminoacidi costituito dai 20 CAAS. Per ogni esperimento, una reazione cell-free controllo negativo viene fornito con la soluzione di aminoacidi che manca l'analogo canonica della NCAA in questione. Per ogni approach, una reazione cell-free esprime la proteina modello in presenza della soluzione di aminoacidi, in cui la CAA è sostituito dalla analogico non canonica. His-tag purificazione, lo scambio di buffer e HPLC-ESI analisi di spettrometria di massa vengono eseguiti secondo il protocollo sopra descritto.
La proteina modello è il C-terminale His-tagged deGFP 32, una versione troncata di EGFP 53. La sua una lettera sequenza aminoacidica in cui si trovano (materiale supplementare 2). Questa proteina modello contiene 6 Arg e 18 posizioni Lys, rispettivamente. Il vettore di espressione è pBEST-OR2-OR1-Pr-UTR1-deGFP-T500.
Nel caso di completa incorporazione della NCAA, si può supporre che la reazione di controllo negativo non esprime deGFP, dal momento che uno dei 20 CAAS è mancante. Al contrario, deGFP deve essere rilevabili negli altri due reazioni: quello nativo nel refeRence reazione cell-free e la proteina modificata nella reazione priva di cellule che viene fornito con la NCAA.
La figura 1A mostra la valutazione preliminare SDS-PAGE dell'esperimento incorporazione Can. La reazione di riferimento-cella libera ha il più alto livello di espressione deGFP. Nella reazione priva di cellule che viene fornito con Can, deGFP è espressa a concentrazioni leggermente inferiori. Nessuna espressione deGFP può essere rilevata nel controllo negativo. Questo risultato SDS-PAGE è una buona indicazione per un inserimento di successo di Can nel deGFP proteina bersaglio.
Per dimostrare l'incorporazione completa ipotizzato di Can in deGFP, entrambe le proteine purificate modello, visualizzati nella figura 1B, sono analizzati tramite spettroscopia di massa HPLC-ESI. Figura 1C mostra gli spettri di massa deconvoluzione delle molecole deGFP purificate. La massa deconvoluzione di °? P che si esprime nella reazione priva di cella di riferimento è 26,192.8 Da. Per deGFP espresso nella Can contenente reazione priva di cella appare una massa di 26202,5 Da. Le masse attesi per il nativo deGFP6Arg e la deGFP6Can modificato con Arg essere completamente sostituiti da Can sono 26.193 e 26.204 Da Da, rispettivamente. La differenza di massa di 1,5 Da per deGFP6Can si trova l'errore dello spettro deconvoluzione. Così, è confermata la piena integrazione di Can in deGFP in tutte le 6 posizioni Arg.
I due picchi di intensità ridotta corrispondono alla deGFP6Arg nativo e deGFP6Can modificati che non ha raggiunto il fluoroforo maturo. Il fluoroforo viene autocatalytically generato dalla eliminazione di una molecola di H 2 O, seguita da ossidazione. Questo porta ad una massa maggiore del 20 Da se questo processo non procede.
pg "/>
Figura 1. valutazione SDS-PAGE del esperimento può costituzione e spettroscopia di massa HPLC-ESI dei cell-free molecole deGFP espressi e purificati. (A) la valutazione preliminare della esperimento può incorporazione mediante SDS-PAGE. Da sinistra a destra: standard di proteine, reazione priva di cella di riferimento, controllo negativo e la reazione cell-free che fornisce Can invece di Arg. (B) SDS-PAGE dopo His-tag purificazione e buffer di scambio delle molecole deGFP espresse. Da sinistra a destra: standard di proteine, deGFP purificata dalla reazione di riferimento, purificato deGFP dalla reazione può che contiene. (C) La conferma della piena incorporazione di Can mediante spettroscopia di massa HPLC-ESI. Le masse attesi del nativo deGFP e la deGFP modificato con Arg completamente sostituite da Can sono 26.193 e 26.204 Da Da, rispettivamente. Ogni spettro è normalizzata alla sua massima intensità (conteggi). posizioni di picco sono indicatein Da. Per scopi di visualizzazione, le corsie del gel vengono estratti dalle immagini gel, uniti insieme, sono convertiti in formato scala di grigi, formato è ottimizzato e contrasto e luminosità vengono migliorate. Corsie del gel originali sono presentati in materiale supplementare 3. Cliccate qui per vedere una versione più grande di questa figura.
La figura 2A mostra la valutazione preliminare SDS-PAGE dell'esperimento incorporazione Hyl. Nella reazione priva di cellule che viene fornito con Hyl, deGFP si esprime. Contrariamente al primo esperimento, una banda deGFP debole può essere osservato nella reazione di controllo negativo. Questo potrebbe essere dovuto a residui di Lys nelle reazioni cell-free. Ciò consente un'espressione deGFP debole nella reazione di controllo negativo, in cui vengono aggiunti né Lys né Hyl.
Per H Spettroscopia di massa PLC-ESI, le molecole deGFP della reazione privo di cellule dotate Hyl sono purificati e buffer vengono scambiati (Figura 2B).
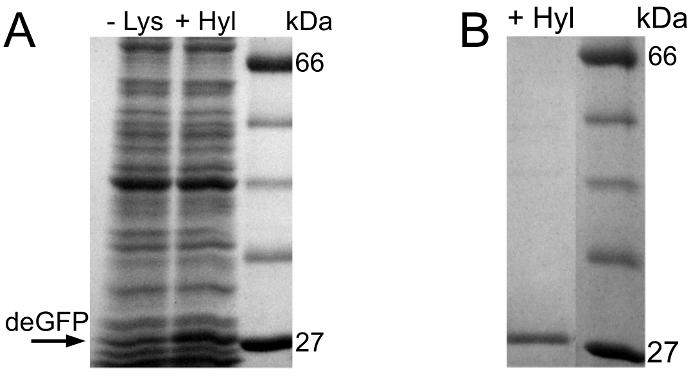
Figura 2. valutazione SDS-PAGE dell'esperimento incorporazione Hyl (A) Da sinistra a destra:. Reazione priva di cellule di controllo negativo, la reazione senza cella contenente Hyl e standard di proteine. (B) SDS-PAGE dopo His-tag purificazione e buffer di scambio delle molecole deGFP espresse. Da sinistra a destra: purificata deGFP dal Hyl contenente reazione e standard di proteine. Per scopi di visualizzazione, le corsie del gel vengono estratti dalle immagini gel, uniti insieme, sono convertiti in formato scala di grigi, formato è ottimizzato e contrasto e luminosità vengono migliorate. corsie del gel originali sono presentati in materiale supplementare 3.f = "https://www.jove.com/files/ftp_upload/54273/54273fig2large.jpg" target = "_ blank"> Clicca qui per vedere una versione più grande di questa figura.
La figura 3 mostra lo spettro di massa deconvoluzione di molecole deGFP purificati. Figura 3A conferma l'ipotesi del già presenti residui Lys nelle reazioni acellulari. Il picco predominante dello spettro corrisponde al nativo (massa prevista: 26.193 Da) deGFP. Anche in questo caso, deGFP molecole di un 20 Da massa superiore che non hanno sviluppato la loro fluoroforo possono essere rilevati. I residui Lys sono preferenzialmente caricati sul tRNA Lys da lysyl-tRNA-sintetasi portando ad un alto livello di espressione dei deGFP18Lys nativi.
La differenza di massa tra Hyl e Lys è di 16 bis. A causa della presenza di Hyl che è in concorrenza ai residui Lys tutte le specie deGFP possibili sono generate (deGFP18Hyl,deGFP17Hly + 1Lys, ..., deGFP16Hyl + 2Lys) (Figura 3B). Certamente, il picco di deGFP1Hyl + 17Lys sovrappone con il picco del deGFP nativa che non producono sua fluoroforo (Figura 3A) e la massa di alcuni picchi differisce più di 2 Da dalla massa prevista. Queste differenze di massa possono essere attribuiti al rumore elevato a causa di basse quantità di specie deGFP. Tuttavia, Hyl è generalmente costituita dal sistema privo di cellule. Ulteriori miglioramenti devono essere fatte per abolire i residui di Lys nelle reazioni cell-free.
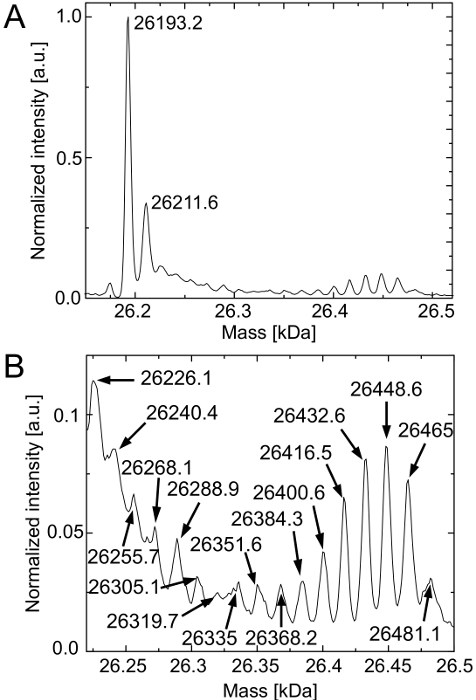
. Figura 3. HPLC-ESI spettroscopia di massa delle molecole deGFP purificate della reazione priva di cella che contiene Hyl (A) I deGFP18Lys nativi è prevalentemente rilevata (masse attese: con fluoroforo 26.193 Da, senza fluoroforo maturo 26.213 Da). (B)Ingrandimento rivela l'esistenza di tutte le possibili specie deGFP (deGFP18Hyl, deGFP17Hly + 1Lys, deGFP16Hyl + 2Lys, ..., deGFP1Hyl + 17Lys). Le loro masse sono attesi 26.193 Da + N x 16 Da (N = 1, ..., 18). Lo spettro è normalizzata alla sua massima intensità (conteggi). Posizioni di picco sono indicate in Da. Cliccate qui per vedere una versione più grande di questa figura.

Supplemental materiale 1. modello di calcolo. Nella sezione 2, la preparazione del master mix buffer di energia è esemplificato con aliquote greggio estratto di 30 ml e ottimale Mg e le concentrazioni di K-glutammato rispettivamente 3 mm e 30 mm. Questo esempio conduce ad un volume master mix che produce 3 aliquote tampone energia. nel seczione 3, la preparazione della reazione acellulare è esemplificato utilizzando una soluzione a 90 nm il DNA stock portando ad una concentrazione ottimale di DNA vettore di 10 Nm nella reazione acellulare. Clicca qui per scaricare questo file.
Per un'adeguata tracciabilità, l'uso del modello di calcolo è esemplificato mediante inserimento di questi valori tipici che differiscono dall'esempio precedente: Crude volumi estratto: 28 microlitri, ottimale Mg-glutammato: 2 mM, ottimale K-glutammato: 40 mm, N. di aliquote tampone desiderati: 100, concentrazione ottimale vettore in reazione cell-free: 8 nm, DNA vettore stock soluzione: 150 nm.
Prima immettere 28 microlitri come volume estratto grezzo nel campo arancione della prima sezione del modello. Poi, entrare nella seconda sezione del modello da 2 mm und 40 mm come ottimale Mg e le concentrazioni di K-glutammato nei campi di colore arancione. Tenendo conto della ottimale Mg e K concentrazioni, la composizione di un buffer di energia 15 microlitri, nonché un corrispondente, scalati aliquota 16 microlitri viene calcolato. Qui di seguito, di conseguenza immettere 100 come numero desiderato di aliquote di buffer di energia (16 ml). Il modello si adatta ai volumi dei diversi componenti del tampone per il master mix 1.700 ml come segue: 204 ml di soluzione madre 100 mM Mg-glutammato, 136 ml di soluzione madre 3 M K-glutammato, 728.73 ml di 14x soluzione di energia, 510 microlitri del 40% PEG-8000 e 121.27 ul sterile DDH 2 O. Infine, nella terza sezione del modello, inserire 8 nm e 150 nm come concentrazione ottimale vettore nella reazione privo di cellule e, rispettivamente, il DNA vettore concentrazione della soluzione. Il modello adatta i volumi dei diversi componenti che devono essere aggiunti ai 28 ml di estratto grezzo per finalizzare la preparazione di un 90 microlitrireazione cell-free come segue: 15 microlitri di tampone di energia, 15 ml di una delle 3 soluzioni aminoacidi diversamente composte, 4,80 ml di soluzione di DNA vettoriale (150 nM) e 27.20 ml di DDH sterile 2 O.
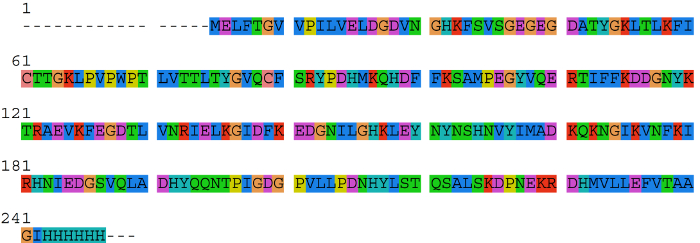
Supplemental Materiale 2. Una lettera sequenza aminoacidica della proteina modello deGFP. Questa proteina modello contiene 6 Arg e 18 posizioni Lys. Clicca qui per vedere una versione più grande di questa figura.
Materiale supplementare 3. lunghezza e corsie del gel non modificati che corrispondono alle tabelle gel presentato Figura Figura 1 e 2. Le corsie del gel presentato in ogni individuo s ubfigure sono estratti dal medesimo gel di poliacrilammide SDS. Nella figura 1 e 2 queste corsie sono state riunite insieme per scopi di presentazione. pesi molecolari di bande proteiche di serie sono indicate accanto alle figure. (A.1) corsie del gel non tagliate di figura 1A. Da sinistra a destra: standard di proteine, reazione priva di cella di riferimento, controllo negativo e la reazione cell-free che fornisce Can invece di Arg. (A.2) corsie del gel non tagliate di Figura 1B. Da sinistra a destra: standard di proteine, deGFP purificata dalla reazione di riferimento, purificato deGFP dalla reazione può che contiene. (B.1) corsie del gel non tagliate di figura 2A. Da sinistra a destra: la reazione priva di cellule di controllo negativo, la reazione senza cella contenente Hyl e standard di proteine. (B.2) corsie del gel non tagliate di figura 2B. Da sinistra a destra: purificata deGFP dal Hyl contenente reazione e standard di proteine.d / 54273 / 54273supfig3large.jpg "target =" _ blank "> Clicca qui per vedere una versione più grande di questa figura.
Discussione
Un-facile da usare sistema di espressione cell-free come una strategia praticabile per residuo specificamente incorporare NCAAs in proteine, viene presentato. A tal fine, l'estratto grezzo è completato con DNA codificante vettoriale per la proteina di interesse, il buffer di energia e gli amminoacidi corrispondenti. Si noti che il volume dell'aliquota estratto grezzo dipende dalla concentrazione proteica estratto grezzo 34. L'efficienza di espressione acellulare è ottimizzato in funzione della concentrazione di vettore di costruzione di DNA. I volumi dei componenti del tampone di energia variano in funzione della ottimizzato Mg e concentrazioni K-glutammato per consentire alte rese della proteina espressa modello cell-free.
Una valutazione preliminare dell'esperimento incorporazione può essere ottenuta eseguendo SDS-PAGE del mezzo di reazione privo di cellule non purificate. Per un'analisi più dettagliata, la spettroscopia di massa HPLC-ESI si propone come un mezzo per verificare la presenza di incor completi e specifici residuiporation della NCAA. Come preparazione per questi ultimi, i sistemi di rotazione delle colonne vengono utilizzate per consentire la purificazione His-tag e buffer di scambio con i piccoli volumi che usiamo in questo protocollo.
Tra cui la spettroscopia di massa HPLC-ESI, l'intero protocollo può essere eseguita entro 2 giorni. Non include tutte le misure di particolare criticità. Tuttavia, ottimizzazioni concentrazione di Mg e K-glutammato e del vettore di DNA sono cruciali per esprimere rese elevate della proteina modello. L'uso del altamente efficiente vettore di espressione pBEST-OR2-OR1-Pr-UTR1-gene_of_model_protein-T500 è fortemente raccomandato. Eluizione di His-tagged proteine è di solito a causa dell'alta concentrazione di imidazolo (> 150 mm) e altri sali, quali NaH 2 PO 4 (> 300 mm) o NaCl (> 50 mm) che generano alto rumore di fondo in analisi spettroscopica di massa 49 . Scambio di tali buffer di eluizione con un buffer di memorizzazione proteina adatto stabilizza il modello protein e riduce drasticamente il rumore di fondo durante l'analisi spettroscopica di massa.
Come risultato, può sostituisce Arg in ogni sei posizioni all'interno della proteina modello. Nel sistema di espressione, senza residui Arg possono essere rilevati. Questo semplifica l'incorporazione specifici residui degli analoghi Arg rispetto ad altri sistemi di espressione che richiedono ulteriori strategie esaurimento 29,30. L'approccio cell-free presentato aggira i limiti intrinseci di vivo approcci che sono dovuti alla tossicità può, o la forte dipendenza sequenza di mRNA a singola proteina strategie di produzione 24,31. Contrariamente a quanto impiegato nel sistema vitro, in vivo scissione di Can a omoserina e hydroxyguanidine si verifica 31.
Tuttavia, il sistema non cellulare mantiene una quantità sufficiente di Lys per competere con analoghi quali Hyl. L'analisi HPLC-ESI spettroscopia di massa mostra che la proteina Modello contenga sia la canonical così come l'analogo noncanonical in proporzioni diverse. L'incorporazione specifico residuo di Lys è possibile in generale, ma per sostituzione completa, altre strategie di esaurimento, o RAA appositamente progettati e tRNA ottimizzato per il riconoscimento di NCAAs necessario sviluppare.
Abbiamo ottenuto ottime rese di cellule libere espressi, proteine modello modificati aggiungendo NCAA alla stessa concentrazione di quelli canonici. L'efficacia di incorporazione dipende dalla natura del NCAA essere incorporati. Anche i rendimenti più elevati potrebbero essere ancora realizzabile ottimizzando la concentrazione del NCAA.
I risultati presentati dimostrano l'applicabilità del sistema utilizzato per la costituzione specifica per residuo di NCAAs fintanto che sono accettate dal sistema di traslazione endogena canonica. Per l'inserimento specifico per residuo di NCAAs specifici, un ulteriore bisogno di controllare se i residui del distur CAA analogob il sistema di espressione.
Sistemi di trascrizione-traduzione privi di cellule possono essere progettati da organismi diversi per rispondere alle diverse esigenze 54. Il tutto E. coli macchinari trascrizione-traduzione del sistema acellulare qui presentati consentono l'uso di batteriofago ed E. promotori coli, e possono agire in parallelo o consecutivamente in cascata 55. L'applicabilità generale e usabilità rendono il metodo un potente strumento per ulteriori ricerche nella tossicità aminoacidi e applicazioni terapeutiche.
Divulgazioni
The authors have nothing to disclose.
Riconoscimenti
E.G. Worst and A. Ott acknowledge financial support by the Deutsche Forschungsgemeinschaft (DFG) within the collaborative research center SFB 1027 as well as Saarland University. E.G. Worst, A. Ott and V. Noireaux further acknowledge financial aid by the Human Frontiers Science Program Organization (HFSPO). The authors thank Tobias Baumann and Stefan Oehm (Institute of Chemistry, Technische Universität Berlin) for critical reading.
Materiali
| Name | Company | Catalog Number | Comments |
| Protective eyewear | Sigma-Aldrich, St. Louis, USA | Z758841 | |
| Nitrile gloves (size S) | Sigma-Aldrich, St. Louis, USA | Z768960 | Catalog numbers other sizes: Z768979 for M, Z768987 for L and Z768995 for XL |
| Eppendorf Safe-Lock Tube 1.5 ml (PCR clean) | Eppendorf, Hamburg, Germany | 30123.328 | |
| Microbalance Discovery DV114CM | Ohaus, Greifensee, Switzerland | 80104140 | |
| Microspatula (L 6 5/8 in., stainless steel, rod diam. 0.09 in.) | Sigma-Aldrich, St. Louis, USA | Z243213 | |
| L-Canavanine | Sigma-Aldrich, St. Louis, USA | C9758 | Acute toxicity: wear eyeshields, dust mask, protective gloves |
| Hydroxylysine (racemic mixture) | Sigma-Aldrich, St. Louis, USA | H0377 | |
| Cryo-gloves (size S, water resistent) | Sigma-Aldrich, St. Louis, USA | Z183490 | Catalog numbers other sizes: Z183512 for M, Z183520 for L and Z183539 for XL |
| RTS Amino Acid Sampler | Biotechrabbit, Hennigsdorf, Germany | BR1401801 | For homemade preparation of amino acid stock solutions, follow this protocol35 and use the solid amino acid kit LAA21-1KT, L-proline (81709-25G), L-cysteine (30089-25G), L-histidine (53319-25G) and L-lysine (L5501-5G) (all from Sigma-Aldrich, St. Louis, USA) |
| HEPES | Sigma-Aldrich, St. Louis, USA | H6147 | |
| ATP | Sigma-Aldrich, St. Louis, USA | A8937 | |
| CTP | Affymetrix, Santa Clara, USA | 14121 | |
| GTP | Affymetrix, Santa Clara, USA | 16800 | |
| UTP | Affymetrix, Santa Clara, USA | 23160 | |
| tRNA (from E. coli, pack size 100 mg) | Sigma-Aldrich, St. Louis, USA | 10109541001 | Catalog number for pack size of 500 mg is 10109550001 |
| CoA | Sigma-Aldrich, St. Louis, USA | C4282 | |
| NAD (from yeast ) | Sigma-Aldrich, St. Louis, USA | N6522 | |
| cAMP | Sigma-Aldrich, St. Louis, USA | A9501 | |
| Folinic acid | Sigma-Aldrich, St. Louis, USA | F7878 | |
| 3-PGA | Sigma-Aldrich, St. Louis, USA | P8877 | |
| Mg-glutamate | Sigma-Aldrich, St. Louis, USA | 49605 | |
| K-glutamate | Sigma-Aldrich, St. Louis, USA | G1149 | |
| pBEST-OR2-OR1-Pr-UTR1-deGFP-T500 | Addgene, Cambridge, USA | Plasmid #40019 | |
| 4-20% precast Tris-Glycine Gels (10 cm x 10 cm x 1 mm, 10 courses) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 81610 | |
| SDS running buffer (10 x concentrate, 5,000 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 50001 | |
| SDS loading buffer (2 x concentrate, 50 ml) | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | TG 05002 | |
| Unstained protein marker, broad range (2-212 kDa) | New England Biolabs, Ipswich, USA | P7702S | |
| Methanol | Merck, Darmstadt, Germany | 1060091011 | Toxic by inhalation, in contact with skin and if swallowed: wear protective gloves and work under fume hood |
| Acetic acid (99.8%) | VWR International, Darmstadt, Germany | 20104.447 | |
| Coomassie Blue G-250 (10 g) | Biozym Scientific, Hessisch Oldendorf, Germany | 902120 | |
| His-Spin Protein Miniprep kit | Zymo Research Europe, Freiburg, Germany | P2002 | Product also distributed by Zymo Research Corporation, Irvine, USA |
| Trizma Base | Sigma-Aldrich, St. Louis, USA | T1503 | |
| Hydrochloric acid | Sigma-Aldrich, St. Louis, USA | H1758 | |
| Glycerol, 99% | VWR International, Darmstadt, Germany | 24397.296DB | |
| CentriPure Z25 mini spin columns | Genaxxon bioscience, Ulm, Germany | CP-0205-Z100 | |
| Sodium chloride | Sigma-Aldrich, St. Louis, USA | S9888 | |
| Concentrator 5301 | Eppendorf, Hamburg, Germany | 5301 000.210 | |
| 2xYT | MP biomedicals, Santa Ana, USA | 113012032 | |
| Bacto-Agar | BD Diagnostics, Franklin Lakes, USA | 214010 | |
| Bead-beating tubes (polypropylene microvials) | Biospec, Bartlesville, USA | 522S | |
| Beads, 0.1 mm dia. | Biospec, Bartlesville, USA | 11079101 | |
| BL21 Rosetta 2 E. coli strain | Merck, Darmstadt, Germany | 71402 | |
| Bradford BSA protein assay Kit | Bio-Rad, München, Germany | 500-0201 | |
| Chloramphenicol | Sigma-Aldrich, St. Louis, USA | C1919 | |
| Cuvettes, 1.5 ml | Thermo Fisher Scientific, Waltham, USA | 14-955-127 | |
| DTT | Sigma-Aldrich, St. Louis, USA | D0632 | |
| Micro Bio-Spin Chromatography Columns | Bio-Rad, München, Germany | 732-6204 | |
| Nunc 384-well optical bottom plates | Thermo Fisher Scientific, Waltham, USA | 142761 | |
| Nunc sealing tape | Thermo Fisher Scientific, Waltham, USA | 232701 | |
| PEG-8000 | Promega, Madison, USA | V3011 | |
| Potassium phosphate dibasic solution | Sigma-Aldrich, St. Louis, USA | P8584 | |
| Potassium phosphate monobasic solution | Sigma-Aldrich, St. Louis, USA | P8709 | |
| Slide-A-Lyzer dialysis cassettes, 10k MWCO (Kit) | Thermo Fisher Scientific, Waltham, USA | 66382 | |
| Spermidine | Sigma-Aldrich, St. Louis, USA | 85558 | |
| 1 L centrifuge bottle | Beckman-Coulter, Brea, USA | A98813 | |
| 4 L Erlenmeyer flask | Kimble Chase, Vineland (NJ), USA | 26500-4000 | |
| Avanti J-26XP centrifuge | Beckman-Coulter, Brea, USA | 393127 | Or equivalent centrifuge able to centrifuge 1 L bottles. |
| Forma 480 orbital shaker | Thermo Fisher Scientific, Waltham, USA | 480 | Or equivalent shaker able to shake chest-size 6 x 4 L . |
| JLA-8.1000 rotor | Beckman-Coulter, Brea, USA | 363688 | Or equivalent 5,000 x g rotor for the centrifuge above, able to centrifuge 1 L bottles. |
| Mini-Beadbeater-1 | Biospec, Bartlesville, USA | 3110BX | |
| Microfuge 22R refrigerated microcentrifuge | Beckman-Coulter, Brea, USA | 368831 | Or equivalent centrifuge able to centrifuge 2 ml reaction tubes. |
| Heating block HLC HBT 130 | Labexchange, Burladingen, Germany | 24465 | Or equivalent heating block able to heat samples in reaction tubes up to 100 °C. |
| Eppendorf MiniSpin centrifuge | Eppendorf, Hamburg, Germany | 5452000018 | Or equivalent centrifuge able to centrifuge 2 ml reaction tubes. |
| IKA Vortex 3 (4 mm orbital shaker diameter, 0 - 2,500 rpm) | Sigma-Aldrich, St. Louis, USA | Z654760 | Or equivalent vortex. |
| Scotsman AF103 ice flaker machine | Kälte-Berlin, Berlin, Germany | AF103 | Or equivalent ice flaker machine. |
| MyTemp mini digital incubator | Sigma-Aldrich, St. Louis, USA | Z763314 | Or equivalent incubator able to heat samples at 29 °C. |
| EcoCell electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12005 | Or equivalent electrophoresis chamber able to perform vertical gel electrophoresis with the above precast gels or other gels used. |
| Power-phor power supply for electrophoresis cell / chamber | Anamed Elektrophorese, Groß-Bieberau / Rodau, Germany | AN12001 | Or equivalent power supply able to supply above used electrophoresis cell / chamber with power |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 1 | VWR International, Darmstadt, Germany | 613-5278 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| VWR Signature Ergonomic High Performance Single-Channel Variable Volume Pipettors Starter Kit 2 | VWR International, Darmstadt, Germany | 613-5279 | Or equivalent micropipettes enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D10ST (0.1 - 10 µl) | Gilson, Middleton, USA | F171101 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D200ST (2 - 200 µl) | Gilson, Middleton, USA | F171301 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| Pipetman Tips Diamond D1000ST (100 - 1,000 µl) | Gilson, Middleton, USA | F171501 | Or equivalent low-binding pipette tips enabling to pipette similar volumes with the same precision |
| 50 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 50-0156 | |
| 15 ml centrifuge tubes with screw caps (sterile) | VWR International, Darmstadt, Germany | 525-0150 | |
| 14 ml polypropylene tubes (round bottom, two-position vent stopper, sterile) | Greiner Bio-One, Frickenhausen, Germany | 187262 | |
| Discovery BIO Wide Pore C5 HPLC Column (3 µm particle size, L x I.D. 10 cm x 2.1 mm) | Sigma-Aldrich, St. Louis, USA | 567227-U | |
| Agilent 1260 HPLC machine | Agilent Technologies, Santa Clara, USA | G1312B | |
| 6500 Series Accurate-Mass Quadrupole Time-of-Flight (Q-TOF) LC/MS | Agilent Technologies, Santa Clara, USA | G6530BA | |
| Acetonitrile | Sigma-Aldrich, St. Louis, USA | 270717 | |
| FLUOstar Omega microplate reader | BMG Labtech, Ortenberg, Germany | 415-101 | Or equivalent microplate reader able to measure the fluorescence of the expressed model protein |
| Hanna Checker pH meter | Sigma-Aldrich, St. Louis, USA | Z351091 | |
| Formic acid eluent additive for LC-MS | Sigma-Aldrich, St. Louis, USA | 56302 |
Riferimenti
- Böck, A., et al. Selenocysteine: the 21st amino acid. Mol. Microbiol. 5 (3), 515-520 (1991).
- Srinivasan, G., James, C. M., Krzycki, J. A. Pyrrolysine Encoded by UAG in Archaea: Charging of a UAG-Decoding Specialized tRNA. Science. 296 (5572), 1459-1462 (2002).
- Budisa, N. Prolegomena to Future Experimental Efforts on Genetic Code Engineering by Expanding Its Amino Acid Repertoire. Angew. Chem. Int. Ed. Engl. 43, 6426-6463 (2004).
- Wang, L., Xie, J., Schultz, P. G. Expanding the Genetic Code. Annu. Rev. Biophys. Biomol. Struct. 35, 225-249 (2006).
- Chin, J. W. Expanding and Reprogramming the Genetic Code of Cells and Animals. Annu. Rev. Biochem. 83, 379-408 (2014).
- Neumann, H. Rewiring translation - Genetic code expansion and its applications. FEBS Lett. 586 (15), 2057-2064 (2012).
- Liu, C. C., Schultz, P. G. Adding New Chemistries to the Genetic Code. Annu. Rev. Biochem. 79, 413-444 (2010).
- Goerke, A. R., Swartz, J. R. High-Level Cell-Free Synthesis Yields of Proteins Containing Site-Specific Non-Natural Amino Acids. Biotechnol. Bioeng. 102 (2), 400-416 (2009).
- Albayrak, C., Swartz, J. R. Cell-free co-production of an orthogonal transfer RNA activates efficient site-specific non-natural amino acid incorporation. Nucleic Acids Res. 41 (11), 5949-5963 (2013).
- Johnson, J. A., Lu, Y. Y., Van Deventer, J. A., Tirrell, D. A. Residue-specific incorporation of non-canonical amino acids into proteins: recent developments and applications. Curr. Opin. Chem. Biol. 14 (6), 774-780 (2010).
- Xiu, X., Puskar, N. L., Shanata, J. A. P., Lester, H. A., Dougherty, D. A. Nicotine binding to brain receptors requires a strong cation-pi interaction. Nature. 458 (7237), 534-537 (2009).
- Grünewald, J., et al. Mechanistic studies of the immunochemical termination of self-tolerance with unnatural amino acids. Proc. Natl. Acad. Sci. USA. 106 (11), 4337-4342 (2009).
- Nikić, I., Lemke, E. A. Genetic code expansion enabled site-specific dual-color protein labeling: superresolution microscopy and beyond. Curr. Opin. Chem. Bio. 28, 164-173 (2015).
- Munier, R., Cohen, G. N. Incorporation d'analogues structuraux d'aminoacides dans les protéines bactériennes. Biochim. Biophys. Acta. 21 (3), 592-593 (1956).
- Lepthien, S., Merkel, L., Budisa, N. In Vivo Double and Triple Labeling of Proteins Using Synthetic Amino Acids. Angew. Chem. Int. Ed. 49 (32), 5446-5450 (2010).
- Dieterich, D. C., Link, A. J., Graumann, J., Tirrell, D. A., Schuman, E. M. Selective identification of newly synthesized proteins in mammalian cells using bioorthogonal noncanonical amino acid tagging (BONCAT). Proc. Natl. Acad. Sci. USA. 103 (25), 9482-9487 (2006).
- Dieterich, D. C., et al. In situ visualization and dynamics of newly synthesized proteins in rat hippocampal neurons. Nat. Neurosci. 13 (7), 897-905 (2011).
- Hendrickson, W. A., Horton, J. R., LeMaster, D. M. Selenomethionyl proteins produced for analysis by multiwavelength anomalous diffraction (MAD): a vehicle for direct determination of three-dimensional structure. EMBO J. 9 (5), 1665-1672 (1990).
- Hoesl, M. G., et al. Lipase Congeners Designed by Genetic Code Engineering. ChemCatChem. 3 (1), 213-221 (2011).
- Bae, J. H., et al. Expansion of the Genetic Code Enables Design of a Novel "Gold" Class of Green Fluorescent Proteins. J. Mol. Biol. 328 (5), 1071-1081 (2003).
- Schachtele, C. F., Rogers, P. Canavanine death in Escherichia coli. J. Mol. Biol. 14 (2), 474-489 (1965).
- Rosenthal, G. A. The biological effects and mode of action of L-canavanine, a structural analogue of L-arginine. Q. Rev. Biol. 52 (2), 155-178 (1977).
- Rosenthal, G. A., Dahlman, D. L. Incorporation of L-Canavanine into Proteins and the Expression of Its Antimetabolic Effects. J. Agric. Food Chem. 39 (5), 987-990 (1991).
- Ishida, Y., Park, J. H., Mao, L., Yamaguchi, Y., Inouye, M. Replacement of All Arginine Residues with Canavanine in MazF-bs mRNA Interferase Changes Its Specificity. J. Biol. Chem. 288 (11), 7564-7571 (2013).
- Thomas, D. A., Rosenthal, G. A., Gold, D. V., Dickey, K. Growth Inhibition of a Rat Colon Tumor by L-Canavanine. Cancer Res. 46 (6), 2898-2903 (1986).
- Bence, A. K., Worthen, D. R., Adams, V. R., Crooks, P. A. The antiproliferative and immunotoxic effects of L-canavanine and L-canaline. Anticancer Drugs. 13 (3), 313-320 (2002).
- Bence, A. K., Crooks, P. A. The Mechanism of L-Canavanine Cytotoxicity: Arginyl tRNA Synthetase as a Novel Target for Anticancer Drug Discovery. J. Enzyme Inhib. Med. Chem. 18 (5), 383-394 (2003).
- Akaogi, J., et al. Role of non-protein amino acid L-canavanine in autoimmunity. Autoimmun. Rev. 5 (6), 429-435 (2006).
- Singh-Blom, A., Hughes, R. A., Ellington, A. D. An amino acid depleted cell-free protein synthesis system for the incorporation of non-canonical amino acid analogs into proteins. J. Biotechnol. 178, 12-22 (2014).
- Oh, S. -. J., Lee, K. -. H., Kim, H. -. C., Catherine, C., Yun, H., Kim, D. -. M. Translational Incorporation of Multiple Unnatural Amino Acids in a Cell-free Protein Synthesis System. Bioprocess. Eng. 19 (3), 426-432 (2014).
- Worst, E. G., Exner, M. P., De Simone, A., Schenkelberger, M., Noireaux, V., Budisa, N., Ott, A. Cell-free expression with the toxic amino acid canavanine. Bioorg. Med. Chem. Lett. 25 (17), 3658-3660 (2015).
- Shin, J., Noireaux, V. Efficient cell-free expression with the endogenous E. Coli RNA polymerase and sigma factor 70. J. Biol. Eng. 4 (8), (2010).
- Chemla, Y., Ozer, E., Schlesinger, O., Noireaux, V., Alfonta, L. Genetically expanded cell-free protein synthesis using endogenous pyrrolysyl orthogonal translation system. Biotechnol. Bioeng. 112 (8), 1663-1672 (2015).
- Sun, Z. Z., Hayes, C. A., Shin, J., Caschera, F., Murray, R. M., Noireaux, V. Protocols for Implementing an Escherichia coli Based TX-TL Cell-Free Expression System for Synthetic Biology. J. Vis. Exp. (79), e50762 (2013).
- Caschera, F., Noireaux, V. Preparation of amino acid mixtures for cell-free expression systems. Biotechniques. 58 (1), 40-43 (2015).
- Green, M. R., Sambrook, J. . Molecular Cloning: A Laboratory Manual. , (2012).
- Froger, A., Hall, J. E. Transformation of Plasmid DNA into E. coli Using the Heat Shock Method. J. Vis. Exp. (6), e253 (2007).
- Zhang, S., Cahalan, M. D. Purifying Plasmid DNA from Bacterial Colonies Using the Qiagen Miniprep Kit. J. Vis. Exp. (6), e247 (2007).
- Moreno, L. A., Cox, K. L. Quantification of dsDNA using the Hitachi F-7000 Fluorescence Spectrophotometer and PicoGreen Dye. J. Vis. Exp. (45), e2465 (2010).
- Desjardins, P., Conklin, D. NanoDrop Microvolume Quantitation of Nucleic Acids. J. Vis. Exp. (45), e2699 (2010).
- Sukumaran, S. Concentration Determination of Nucleic Acids and Proteins Using the Micro-volume Bio-spec Nano Spectrophotometer. J. Vis. Exp. (48), e2699 (2011).
- Laemmli, U. K. Cleavage of Structural Proteins during the Assembly of the Head of Bacteriophage T4. Nature. 227 (5259), 680-685 (1970).
- Hochuli, E., Bannwarth, W., Döbeli, H., Gentz, R., Stüber, D. Genetic Approach to Facilitate Purification of Recombinant Proteins with a Novel Metal Chelate Adsorbent. Nature Biotechnology. 6 (11), 1321-1325 (1988).
- Schägger, H., Aquila, H., Von Jagow, G. Coomassie blue-sodium dodecyl sulfate-polyacrylamide gel electrophoresis for direct visualization of polypeptides during electrophoresis. Anal. Biochem. 173 (1), 201-205 (1988).
- Bradford, M. M. A Rapid Method for the Quantitation of Microgram Quantities of Protein Utilizing the Principle of Protein-Dye Binding. Anal Biochem. 72, 248-254 (1976).
- Ernst, O., Zor, T. Linearization of the Bradford Protein Assay. J. Vis. Exp. (38), e1918 (2010).
- Banerjee, S., Mazumdar, S. Electrospray Ionization Mass Spectrometry: A Technique to Access the Information beyond the Molecular Weight of the Analyte. Int. J. Anal. Chem. 2012, 1-40 (2012).
- Fujiwara, K., Nomura, S. M. Condensation of an additive-free cell extract to mimic the conditions of live cells. PLoS One. 8 (1), e54155 (2013).
- Zhang, Z., Marshall, A. G. A universal algorithm for fast and automated charge state deconvolution of electrospray mass-to-charge ratio spectra. J. Am. Soc. Mass. Spectrom. 9 (3), 225-233 (1998).
- Li, X., Zhang, G., Ngo, N., Zhao, X., Kain, S. R., Huang, C. C. Deletions of the Aequorea victoria Green Fluorescent Protein Define the Minimal Domain Required for Fluorescence. J. Biol. Chem. 272 (45), 28545-28549 (1997).
- Gagoski, D., Polinkovsky, M. E., Mureev, S., Kunert, A., Johnston, W., Gambin, Y., Alexandrov, K. Performance benchmarking of four cell-free protein expression systems. Biotechnol. Bioeng. 113 (2), 292-300 (2016).
- Shin, J., Noireaux, V. An E. coli Cell-Free Expression Toolbox: Application to Synthetic Gene Circuits and Artificial Cells. ACS Synth. Biol. 1 (1), 29-41 (2012).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon