È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Method Article
Misurazione di ceppi tissutali locali nei tendini tramite correlazione di immagini digitali open source
In questo articolo
Riepilogo
Questo documento descrive un algoritmo di correlazione delle immagini digitali open source per misurare ceppi tissutali 2D locali all'interno di espianti di tendini. L'accuratezza della tecnica è stata convalidata utilizzando più tecniche ed è disponibile per uso pubblico.
Abstract
C'è un notevole interesse scientifico nella comprensione dei ceppi che le cellule tendinee sperimentano in situ e di come questi ceppi influenzano il rimodellamento dei tessuti. Sulla base di questo interesse, sono state sviluppate diverse tecniche analitiche per misurare i ceppi tissutali locali all'interno degli espianti di tendini durante il carico. Tuttavia, in diversi casi, l'accuratezza e la sensibilità di queste tecniche non sono state riportate e nessuno degli algoritmi è disponibile pubblicamente. Ciò ha reso difficile la misurazione più diffusa dei ceppi tissutali locali negli espianti di tendini. Pertanto, l'obiettivo di questo documento era quello di creare uno strumento di analisi convalidato per misurare ceppi tissutali locali negli espianti di tendini che fosse prontamente disponibile e facile da usare. In particolare, un algoritmo di correlazione digitale lagrangiana aumentata (ALDIC) disponibile al pubblico è stato adattato per misurare i ceppi 2D monitorando gli spostamenti dei nuclei cellulari all'interno dei tendini di Achille del topo sotto tensione uniassiale. Inoltre, l'accuratezza delle deformazioni calcolate è stata convalidata analizzando immagini trasformate digitalmente e confrontando le deformazioni con valori determinati da una tecnica indipendente (cioè linee fotosbiancate). Infine, nell'algoritmo è stata incorporata una tecnica per ricostruire l'immagine di riferimento utilizzando il campo di spostamento calcolato, che può essere utilizzato per valutare l'accuratezza dell'algoritmo in assenza di valori di deformazione noti o di una tecnica di misurazione secondaria. L'algoritmo è in grado di misurare deformazioni fino a 0,1 con una precisione di 0,00015. La tecnica per confrontare un'immagine di riferimento ricostruita con l'immagine di riferimento effettiva ha identificato con successo campioni che avevano dati errati e ha indicato che, nei campioni con dati validi, circa l'85% del campo di spostamento era accurato. Infine, i ceppi misurati nei tendini di Achille del topo erano coerenti con la letteratura precedente. Pertanto, questo algoritmo è uno strumento molto utile e adattabile per misurare con precisione i ceppi tissutali locali nei tendini.
Introduzione
I tendini sono tessuti meccanosensibili che si adattano e degenerano in risposta al carico meccanico 1,2,3,4. A causa del ruolo che gli stimoli meccanici svolgono nella biologia delle cellule tendinee, c'è un grande interesse nella comprensione dei ceppi che le cellule tendinee sperimentano nell'ambiente tissutale nativo durante il carico. Sono state sviluppate diverse tecniche sperimentali e analitiche per misurare i ceppi tissutali locali nei tendini. Questi includono analisi di correlazione digitale delle immagini (DIC) 2D / 3D di ceppi superficiali utilizzando modelli di macchie o linee fotosbiancate (PBL)5,6,7,8, misurazione dei cambiamenti nella distanza centroide-centroide dei singoli nuclei all'interno del tessuto 9,10 e un recente metodo DIC 3D a campo completo che considera il movimento fuori dal piano e le deformazioni 3D 11 . Tuttavia, l'accuratezza e la sensibilità di queste tecniche sono state riportate solo in pochi casi e nessuna di queste tecniche è stata resa disponibile al pubblico, il che rende difficile l'adozione e l'utilizzo diffuso di queste tecniche.
L'obiettivo di questo lavoro era quello di creare uno strumento di analisi convalidato per misurare i ceppi tissutali locali negli espianti di tendini che fosse prontamente disponibile e facile da usare. Il metodo scelto si basa su un algoritmo di correlazione digitale delle immagini agrangiane (ALDIC) disponibile pubblicamente e scritto in MATLAB sviluppato da Yang e Bhattacharya12. Questo algoritmo è stato adattato per analizzare campioni di tendini e convalidato applicandolo a immagini trasformate digitalmente e confrontando le tensioni misurate in campioni di tendini reali con i risultati ottenuti da linee fotosbiancate. Inoltre, nell'algoritmo sono state implementate funzionalità aggiuntive per confermare l'accuratezza del campo di spostamento calcolato anche in assenza di valori di deformazione noti o di una tecnica di misurazione secondaria. Pertanto, questo algoritmo è uno strumento altamente utile e adattabile per misurare con precisione i ceppi tissutali 2D locali nei tendini.
Protocollo
Questo studio è stato approvato dal Pennsylvania State University Institutional Animal Care and Use Committee.
1. Preparazione dei tessuti
- Per questo protocollo, raccogliere i tendini di Achille da topi maschi C57BL / 6 di 2-4 mesi.
NOTA: potrebbero essere utilizzati anche diversi tendini o legamenti di topi o altri piccoli animali.- Fare un'incisione sulla pelle superficiale al tendine di Achille per esporre il tendine plantare e il tessuto connettivo circostante. Quindi, rimuoverli usando una lama chirurgica.
- Separare i muscoli soleo e gastrocnemio esposti dall'arto posteriore e raschiarli accuratamente dal tendine di Achille con la lama chirurgica
- Separare il calcagno dal resto del piede con un attacco della ruota di taglio su uno strumento rotante.
- Colorare il tessuto in 1,5 mL di una soluzione da 5 μg/mL di 5-(4,6-diclorotriazinil) amminofluoresceina (DTAF) e tampone di bicarbonato di sodio 0,1 M per 20 minuti su un miscelatore rotante a temperatura ambiente. Questa soluzione colora le proteine (ad esempio, matrice extracellulare) nel tessuto.
NOTA: durante questo periodo di 20 minuti, il passaggio 1.3 deve essere completato. - Preparare una soluzione 1:1.000 di DRAQ5 in soluzione salina tamponata con fosfato (PBS) per colorare i nuclei. Utilizzare un miscelatore a vortice per omogeneizzare la soluzione.
- Dopo il periodo di incubazione di 20 minuti nella fase 1.2, trasferire il tessuto dalla soluzione DTAF alla soluzione DRAQ5 e incubare in uno spazio buio per 10 minuti a temperatura ambiente.
2. Carico del tendine e acquisizione delle immagini
NOTA: Questo protocollo richiede un dispositivo di trazione che può essere montato sopra un microscopio confocale. Per questo studio, è stato utilizzato il dispositivo di microtrazione descritto da Peterson e Szczesny13 .
- Posizionare il tendine nelle impugnature del dispositivo di carico della trazione. Prima di montare le impugnature nel dispositivo di caricamento, utilizzare calibri digitali per misurare la distanza tra l'attacco del calcagno e l'impugnatura opposta. Questa distanza è la lunghezza del tendine.
- In alternativa, montare le impugnature nel dispositivo di caricamento prima di inserire il tendine e spingere in contatto per definire la posizione del motore a spostamento zero. Lo spostamento dei motori dopo l'inserimento del tendine potrebbe fornire una lunghezza del manometro grip-to-grip potenzialmente più accurata.
- Montare le impugnature nel dispositivo di caricamento, che contiene PBS per mantenere l'idratazione dei tessuti. Allineare il tendine nel miglior modo possibile con l'asse x o y delle immagini del microscopio in modo che le uscite della deformazione x e della deformazione y dell'algoritmo corrispondano agli assi del tendine.
NOTA: In questo studio, i tendini erano allineati con l'asse x. Se non è possibile allineare perfettamente il tendine con gli assi dell'immagine, allora le uscite x-strain e y-strain dell'algoritmo possono essere trasformate per allinearsi con gli assi longitudinali/perpendicolari del tendine usando le equazioni standard di trasformazione della deformazione14. - Precaricare il tendine con 1 g di tensione e, se lo si desidera, applicare il carico ciclico per precondizionare il campione. In questo protocollo, non è stato utilizzato alcun precondizionamento poiché l'obiettivo dello studio era quello di convalidare i ceppi tissutali locali misurati piuttosto che misurare le proprietà del materiale tissutale. Se c'è interesse a misurare le proprietà del materiale su macroscala, che dipendono dalla cronologia di caricamento, si consiglia il precondizionamento. Dopo il precondizionamento e il recupero, riapplicare un precarico di 1 g.
- Se lo si desidera, fotocandeggiare una serie di quattro linee distanziate di 80 μm nella regione centrale del tessuto (vedere Peterson e Szczesny13 per maggiori dettagli).
NOTA: Le linee fotosbiancate sono state utilizzate per convalidare le misurazioni dell'algoritmo ALDIC e non sono necessarie per eseguire l'ALDIC stesso. Il numero e la spaziatura delle linee possono essere regolati e la posizione delle linee deve essere scelta per evitare artefatti nel campione che ridurrebbero la chiarezza della linea. - Ripetere la procedura di fotosbiancamento sugli estremi sinistro e destro del tessuto vicino alle impugnature.
- Utilizzando il microscopio confocale, acquisire immagini volumetriche (x,y: 1,25 μm/pixel, z: 2,5 μm/pixel) della fluorescenza DTAF e DRAQ5 a 1 g di precarico.
- Eseguire una rampa di deformazione dallo 0,5% / s al 2%. Si noti che la velocità di deformazione e l'entità della deformazione incrementale possono essere regolate.
- Lasciare che il tessuto si rilassi per 10 minuti.
NOTA: La durata del rilassamento dello stress deve essere scelta in modo tale che il campione sia sottoposto a un carico approssimativamente quasistatico durante l'acquisizione dell'immagine. Per determinare se la durata del rilassamento dello stress è accettabile, determinare la pendenza della curva forza-tempo durante l'ultimo minuto di rilassamento dello stress (Figura supplementare 1) e moltiplicare questa pendenza per la durata totale dell'immagine. In questo studio, la forza applicata al massimo incremento di deformazione non è mai cambiata di oltre il 5%. - Prendi un'altra immagine volumetrica del tessuto dopo la deformazione.
- Ripetere i passaggi 2.7-2.9 fino a raggiungere la deformazione finale desiderata. In questo documento, è stato scelto un valore finale di deformazione del 12%.
3. Elaborazione delle immagini
- Utilizzare ImageJ o Fiji per creare proiezioni z massime di ogni immagine volumetrica del canale DRAQ5 (nucleare). Questo servirà come immagini macchiate 2D per l'ALDIC.
- Salvare le proiezioni z di intensità massima come file .tiff e denominarle in base alla seguente convenzione di denominazione.
- Utilizzare un numero come primo carattere del nome dell'immagine.
- Fare in modo che il numero corrisponda all'ordine in cui le immagini saranno considerate durante l'analisi della deformazione. Ad esempio, la prima immagine dovrebbe iniziare con una e la seconda immagine dovrebbe iniziare con due. È possibile scegliere numeri diversi, ma devono aumentare in sequenza. Un esempio di convenzione di denominazione è il seguente: "0_Experiment1_MaxZProjection".
- Salvate tutte le proiezioni z di intensità massima rinominate in una cartella.
4. Installazione e applicazione del codice di analisi della linea fotosbiancata
NOTA: questi passaggi sono necessari solo se si desidera confermare l'accuratezza dell'algoritmo ALDIC utilizzando linee fotosbiancate. Il codice calcola il ceppo tissutale locale come la variazione media normalizzata della distanza tra ciascuna linea fotosbiancata all'interno del set di linee fotosbiancate. In questo studio, i valori locali medi sono stati quindi mediati tra tutti i set di linee fotosbiancate (cioè alle estremità centrale e sinistra / destra) per determinare un singolo valore medio di ceppo tissutale locale per ciascun campione. Questo valore è stato quindi utilizzato per stimare l'accuratezza dell'algoritmo ALDIC.
- Scarica la cartella "PBL Code" da GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) e sposta tutto il contenuto nella directory di lavoro in MATLAB.
- Apri lo script MATLAB "Micro_Mech_Template.m".
- Premere Esegui e selezionare uno dei file di immagine contenenti le immagini volumetriche. Le immagini volumetriche possono essere uno dei seguenti tipi di file: .lsm, .tiff, .nd2.
- Il software caricherà automaticamente tutte le immagini nella cartella e visualizzerà un'immagine proiettata dell'immagine volumetrica di riferimento. Quando richiesto, fare clic con il pulsante sinistro del mouse per creare linee multipunto che tracciano le estremità sinistra e destra del campione. Fare clic con il pulsante destro del mouse per terminare una riga. Una volta elaborato l'input, se i bordi sono corretti, premere OK per accettare il risultato.
- Quando richiesto, disegnate una linea diagonale casuale sul campione come linea di riferimento.
- Immettere il numero di linee fotosbiancate create e tracciare le linee sbiancate con più punti.
- Se il risultato è accettabile, accettalo. Se il risultato è errato, regolarlo e rielaborarlo.
- Ripetere il passaggio 4.2 per tutte le immagini e spostare tutte le immagini delle linee tracciate in un'unica cartella.
- Aprire lo script "Micro_Mech_Strain.m".
- Premere Esegui per eseguire il codice e selezionare una delle immagini salvate in cui vengono tracciate le linee photobleached.
- Verificare che le immagini di accompagnamento selezionate siano corrette una volta selezionata l'immagine premendo Ok.
5. Creazione di immagini trasformate digitalmente
NOTA: questi passaggi sono necessari solo se si desidera confermare l'accuratezza dell'algoritmo ALDIC utilizzando immagini trasformate digitalmente. Queste immagini simulano campi di deformazione 2D omogenei di una grandezza nota trasformando artificialmente l'immagine di riferimento.
- Scarica il codice "Digital_strain.m" da GitHub (https://github.com/Szczesnytendon/TendonStrainCalc).
- Aprire ed eseguire il codice.
- Quando richiesto, inserire i valori desiderati per la deformazione massima applicata, l'incremento della deformazione applicata e il rapporto di Poisson. Premere OK.
NOTA: Per questo esperimento, la deformazione massima applicata è stata di 0,1 (10%), l'incremento della deformazione applicata è stato di 0,02 (2%) ed è stato utilizzato un rapporto di Poisson di 1, che è coerente con i dati sperimentali della prova di trazione tendinea15,16. Il codice utilizza la funzione incorporata MATLAB imwarp e i valori di input (ad esempio, incrementi di deformazione, rapporto di Poisson) per creare le immagini trasformate digitalmente. - Quando richiesto, selezionate l'immagine di riferimento non deformata.
- Per ogni incremento di deformazione, viene visualizzata una sovrapposizione dell'immagine di riferimento e dell'immagine trasformata. L'immagine trasformata verrà salvata nella directory sotto il titolo "DigitallyTransformedX%Strain", dove X è l'incremento di deformazione.
6. Installazione e applicazione del codice di calcolo della deformazione e convalida
- Scarica la cartella "Strain Calculation and Validation Code" da GitHub (https://github.com/Szczesnytendon/TendonStrainCalc) e sposta tutto il contenuto nella directory di lavoro di MATLAB
- Installare un compilatore mex C / C ++ secondo Yang e Bhattacharya12. I passaggi sono riepilogati di seguito.
- Controlla MATLAB per vedere se è stato installato un compilatore mex C/C++ digitando "mex -setup" nella finestra di comando di MATLAB e premendo Invio.
- Se viene visualizzato un errore che indica che un compilatore non è supportato o non presente, procedere ai passaggi 6.3 e 6.4.
- Se non è presente alcun errore, procedere al passaggio 6.5
- Per scaricare un compilatore mex C/C++, vai su "https:/tdm-gcc.tdragon.net/" e scegli il compilatore TDM-gcc.
- Installare il compilatore scaricato in un percorso noto.
- Torna alla finestra di comando di MATLAB e digita: "setenv("MW_MINGW64_LOC","[Digita qui il tuo percorso di installazione]")". Ad esempio, potrebbe essere "setenv("MW_MINGW64_LOC","C:\TDM-GCC-64")". Se questo comando viene eseguito correttamente, il compilatore mex è installato correttamente.
- Immettere lo script della funzione "main_aldic.m" e modificare la riga 22 in modo che corrisponda al comando eseguito nel passaggio 6.5.
- Aprire lo script "Strain_calc_and_validate.m".
- Premere Esegui per iniziare l'analisi dell'immagine.
- Quando richiesto, modificare i valori per i parametri ALDIC come desiderato.
NOTA: la dimensione della finestra deve essere da 0,25 a 1 volte la dimensione del sottoinsieme. Per ulteriori informazioni sulle scelte dei parametri, consultare il manuale utente in linea: (https://www.researchgate.net/publication/344796296_Augmented_Lagrangian_Digital
_Image_Correlation_AL-DIC_Code_Manual).- In questo studio sono stati utilizzati i seguenti valori:
Dimensione del sottoinsieme (pixel): 20
Dimensioni finestra (pixel): 10
Metodo per risolvere ALDIC: differenza finita (1)
Il calcolo parallelo non è stato utilizzato (1)
Metodo per calcolare l'ipotesi iniziale: ricerca multigrid basata sulla piramide delle immagini (0)
- In questo studio sono stati utilizzati i seguenti valori:
- Quando richiesto, selezionare la casella di controllo "Sì" per fare in modo che l'algoritmo salvi automaticamente il valore medio, la deviazione standard e le mappe 2D per la raccolta desiderata di variabili (ad esempio, deformazione x, deformazione y, deformazione di taglio, regioni danneggiate, ecc.). Selezionare le variabili da salvare e premere OK.
- Quando richiesto, modificare i parametri come desiderato.
- In questo esperimento sono stati utilizzati i seguenti valori:
Punti circostanti per calcolare la deformazione (numP): 12
Coefficiente di correlazione per l'identificazione di regioni non valide (corr_threshold): 0,5
Dimensione sottoregione (pixel) per l'analisi della regione danneggiata (Sottodimensione): 32
- In questo esperimento sono stati utilizzati i seguenti valori:
- Quando richiesto, selezionare la cartella contenente le proiezioni z di intensità massima rinominate. Si noti che il software esegue automaticamente ALDIC incrementali per determinare i campi di deformazione delle immagini deformate. Cioè, ogni immagine deformata funge da nuova immagine "di riferimento" per la successiva immagine deformata. Ciò migliora l'accuratezza dei risultati (Figura supplementare 2) rispetto all'esecuzione di ALDIC cumulativo, in cui ogni immagine deformata viene confrontata con l'immagine di riferimento originale (sforzo 0%). Per eseguire un'analisi cumulativa, caricare le immagini ma selezionare solo l'immagine di riferimento originale e l'immagine deformata di interesse.
NOTA: La tensione normale è calcolata come λ - 1, dove λ è l'allungamento del tessuto. L'allungamento del tessuto è calcolato secondo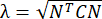 , dove N = [1 0]T o [0 1]T per la direzione x e la direzione y, rispettivamente, e C = F T F, dove F è il gradiente di deformazione calcolato utilizzando i punti "numP" che circondano ciascun punto dati emesso dall'algoritmo ALDIC. La deformazione di taglio è calcolata come
, dove N = [1 0]T o [0 1]T per la direzione x e la direzione y, rispettivamente, e C = F T F, dove F è il gradiente di deformazione calcolato utilizzando i punti "numP" che circondano ciascun punto dati emesso dall'algoritmo ALDIC. La deformazione di taglio è calcolata come 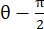 , dove
, dove 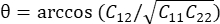 .
. - Quando richiesto, fare clic con il pulsante sinistro del mouse per creare un poligono a quattro punti per definire la regione di interesse per la misurazione delle deformazioni. Inizia con il punto nell'angolo in alto a sinistra e assegna i punti successivi in senso orario.
NOTA: La variabile "Storage" salvata nell'area di lavoro MATLAB contiene tutti i valori per la deformazione x media, la deviazione standard della deformazione x, la deformazione y media, la deviazione standard della deformazione y, la deformazione media di taglio, la deviazione standard della deformazione di taglio e la percentuale delle regioni danneggiate. Le regioni danneggiate sono definite in base all'analisi del coefficiente di correlazione all'interno della regione di interesse selezionata nella fase 6.13. La cartella "NuclearTrackingResults" (che può essere rinominata regolando le righe 555 e 556) memorizza tutti i grafici specificati nel passaggio 6.10. Questa cartella contiene anche un file di foglio di calcolo con il nome "Risultati", che memorizza tutte le medie e le deviazioni standard specificate nel passaggio 6.10.
Risultati
Prima di analizzare i campi di deformazione in campioni di tessuto reali, il protocollo ALDIC è stato convalidato utilizzando immagini tese / trasformate digitalmente di nuclei all'interno dei tendini di Achille del topo. In particolare, le immagini sono state trasformate per produrre digitalmente ceppi uniformi nella direzione x del 2%, 4%, 6%, 8% e 10% di deformazione con un rapporto di Poisson simulato di 115,16. L'accuratezza dell'algoritmo ALDIC è stata qu...
Discussione
L'obiettivo di questo documento era fornire un metodo open source convalidato per misurare i campi di deformazione 2D nei tendini sotto carico di trazione. La base del software era basata su un algoritmo ALDIC disponibile al pubblico12. Questo algoritmo è stato incorporato in un codice MATLAB più grande con l'aggiunta della funzionalità di analisi incrementale (rispetto a cumulativa) della deformazione. Questo algoritmo adattato è stato quindi applicato alla prova di trazione dei tendini e la ...
Divulgazioni
Tutti gli autori non hanno conflitti di interesse da rivelare.
Riconoscimenti
Questo lavoro è stato finanziato dal National Institutes of Health (R21 AR079095) e dalla National Science Foundation (2142627).
Materiali
| Name | Company | Catalog Number | Comments |
| 5-DTAF (5-(4,6-Dichlorotriazinyl) Aminofluorescein), single isomer | ThermoFisher | D16 | |
| Calipers | Mitutoyo | 500-196-30 | |
| Confocal Microscope | Nikon | A1R HD | |
| Corning LSE Vortex Mixer | Coning | 6775 | |
| DRAQ5 Fluorescent Probe Solution (5 mM) | ThermoFisher | 62554 | |
| MATLAB | MathWorks | R2022b | |
| Tensile Loading Device | N/A | N/A | Tensile loading device described in Peterson et al, 2020. (ref 13) |
| Tube Revolver Rotator | ThermoFisher | 88881001 |
Riferimenti
- Devkota, A. C. Distributing a fixed amount of cyclic loading to tendon explants over longer periods induces greater cellular and mechanical responses. Journal of Orthopaedic Research. 11 (4), 1609-1612 (2007).
- Sun, H. B., et al. Cycle-dependent matrix remodeling gene expression response in fatigue-loaded rat patellar tendons. Journal of Orthopaedic Research. 28 (10), 1380-1386 (2010).
- Shepherd, J. H., Screen, H. R. C. Fatigue loading of tendon. International Journal of Experimental Pathology. 94 (4), 260-270 (2013).
- Paschall, L., Pedaprolu, K., Carrozzi, S., Dhawan, A., Szczesny, S. Mechanical stimulation as both the cause and the cure of tendon and ligament injuries. Regenerative Rehabilitation: From Basic Science to the Clinic. , 359-386 (2022).
- Andarawis-Puri, N., Ricchetti, E. T., Soslowsky, L. J. Rotator cuff tendon strain correlates with tear propagation. Journal of Biomechanics. 42 (2), 158-163 (2009).
- Cheng, V. W. T., Screen, H. R. C. The micro-structural strain response of tendon. Journal of Materials Science. 42 (21), 8957-8965 (2007).
- Luyckx, T., et al. Digital image correlation as a tool for three-dimensional strain analysis in human tendon tissue. Journal of Experimental Orthopaedics. 1 (1), 7 (2014).
- Duncan, N. A., Bruehlmann, S. B., Hunter, C. J., Shao, X., Kelly, E. J. In situ cell-matrix mechanics in tendon fascicles and seeded collagen gels: Implications for the multiscale design of biomaterials. Computer Methods in Biomechanics and Biomedical Engineering. 17 (1), 39-47 (2014).
- Arnoczky, S. P., Lavagnino, M., Whallon, J. H., Hoonjan, A. In situ cell nucleus deformation in tendons under tensile load; A morphological analysis using confocal laser microscopy. Journal of Orthopaedic Research. 20 (1), 29-35 (2002).
- Screen, H. R. C., Bader, D. L., Lee, D. A., Shelton, J. C. Local strain measurement within tendon. Strain. 40 (4), 157-163 (2004).
- Fung, A. K., Paredes, J. J., Andarawis-Puri, N. Novel image analysis methods for quantification of in situ 3-D tendon cell and matrix strain. Journal of Biomechanics. 67, 184-189 (2018).
- Yang, J., Bhattacharya, K. Augmented Lagrangian digital image correlation. Experimental Mechanics. 59 (2), 187-205 (2019).
- Peterson, B. E., Szczesny, S. E. Dependence of tendon multiscale mechanics on sample gauge length is consistent with discontinuous collagen fibrils. Acta Biomaterialia. 117, 302-309 (2020).
- Humphrey, J. D., O'Rourke, S. L. . An Introduction to Biomechanics. , (2015).
- Reese, S. P., Weiss, J. A. Tendon fascicles exhibit a linear correlation between Poisson's ratio and force during uniaxial stress relaxation. Journal of Biomechanical Engineering. 135 (3), 34501 (2013).
- Ahmadzadeh, H., Freedman, B. R., Connizzo, B. K., Soslowsky, L. J., Shenoy, V. B. Micromechanical poroelastic finite element and shear-lag models of tendon predict large strain dependent Poisson's ratios and fluid expulsion under tensile loading. Acta Biomaterialia. 22, 83-91 (2015).
- Szczesny, S. E., Elliott, D. M. Interfibrillar shear stress is the loading mechanism of collagen fibrils in tendon. Acta Biomaterialia. 10 (6), 2582-2590 (2014).
- Han, W. M., et al. Macro- to microscale strain transfer in fibrous tissues is heterogeneous and tissue-specific. Biophysical Journal. 105 (3), 807-817 (2013).
- Pedaprolu, K., Szczesny, S. E. A novel, open-source, low-cost bioreactor for load-controlled cyclic loading of tendon explants. Journal of Biomechanical Engineering. 144 (8), 084505 (2022).
- Gatt, R., et al. Negative Poisson's ratios in tendons: An unexpected mechanical response. Acta Biomaterialia. 24, 201-208 (2015).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon