Analisi dell'ereditarietà epigenetica transgenerazionale in C. elegans utilizzando un reporter fluorescente e l'immunoprecipitazione della cromatina (ChIP)
* Questi autori hanno contribuito in egual misura
In questo articolo
Riepilogo
Questo protocollo descrive un test di interferenza dell'RNA e ChIP per studiare l'ereditarietà epigenetica del silenziamento indotto dall'RNAi e le modificazioni della cromatina associate in C. elegans.
Abstract
L'eredità epigenetica transgenerazionale (TEI) consente la trasmissione di informazioni attraverso la linea germinale senza modificare la sequenza del genoma, attraverso fattori come gli RNA non codificanti e le modificazioni della cromatina. Il fenomeno dell'ereditarietà dell'interferenza dell'RNA (RNAi) nel nematode Caenorhabditis elegans è un modello efficace per studiare la TEI che sfrutta il breve ciclo di vita, l'autopropagazione e la trasparenza di questo organismo modello. Nell'ereditarietà dell'RNAi, l'esposizione degli animali all'RNAi porta al silenziamento genico e all'alterazione delle firme della cromatina nel locus bersaglio che persistono per più generazioni in assenza dell'innesco iniziale. Questo protocollo descrive l'analisi dell'ereditarietà dell'RNAi in C. elegans utilizzando un reporter della proteina fluorescente verde nucleare (GFP) espressa dalla linea germinale. Il silenziamento del reporter viene avviato alimentando gli animali con batteri che esprimono RNA a doppio filamento che hanno come bersaglio la GFP. Ad ogni generazione, gli animali vengono sottoposti a uno sviluppo sincronizzato e il silenziamento del gene reporter viene determinato al microscopio. A generazioni selezionate, le popolazioni vengono raccolte e processate per l'immunoprecipitazione della cromatina (ChIP)-reazione a catena quantitativa della polimerasi (qPCR) per misurare l'arricchimento della modificazione istonica nel locus reporter della GFP. Questo protocollo per lo studio dell'ereditarietà dell'RNAi può essere facilmente modificato e combinato con altre analisi per indagare ulteriormente i fattori TEI nelle piccole vie dell'RNA e della cromatina.
Introduzione
L'eredità epigenetica consente la trasmissione di informazioni di regolazione genica attraverso le generazioni e può quindi consentire all'ambiente o alle esperienze dei genitori di influenzare la loro progenie. In C. elegans, il silenziamento genico germinale iniziato dall'RNA esogeno a doppio filamento (dsRNA) può essere ereditato per più generazioni in progenie non esposta al trigger originale 1,2,3,4. Questo processo, chiamato ereditarietà dell'interferenza dell'RNA (RNAi), è uno dei numerosi fenomeni di silenziamento epigenetico correlati in C. elegans, tra cui il silenziamento multigenerazionale 2,5 avviato da piRNA, la paramutazione/RNAe (silenziamento epigenetico indotto da RNA)6,7,8 e il silenziamento 9 avviato da array multicopia, che hanno requisiti sovrapposti ma distinti per piccoli meccanismi di regolazione dell'RNA e della cromatina . Nell'RNAi esogeno di C. elegans, il dsRNA viene processato in piccoli RNA interferenti (siRNA) che agiscono in un complesso con le proteine primarie Argonaute per riconoscere il loro mRNA bersaglio. Questo targeting porta all'amplificazione di siRNA secondari che si associano agli Argonauti secondari per silenziare l'mRNA bersaglio attraverso le vie di silenziamento citoplasmatico e nucleare. Per i bersagli RNAi espressi dalla linea germinale, l'Argonaute HRDE-1 secondario nucleare e altri fattori RNAi nucleari prendono di mira i trascritti nascenti, con conseguente repressione trascrizionale e reclutamento di metiltransferasi istoniche per depositare marcature cromatiniche repressive, tra cui H3K9me310. L'istone H3K9me3 promuove l'instaurazione di un silenziamento ereditario dei transgeni della proteina fluorescente verde (GFP) espressa dalla linea germinale mediante l'ereditarietà dell'RNAi11,12.
L'obiettivo di questo protocollo è quello di utilizzare un transgene che esprima una proteina di fusione GFP-istone nella linea germinale come reporter per l'ereditarietà dell'RNAi e di saggiare i cambiamenti nelle modificazioni istoniche nel locus del transgene reporter utilizzando l'immunoprecipitazione della cromatina e la reazione a catena della polimerasi quantitativa (ChIP-qPCR). Questo protocollo descrive un approccio standardizzato di alimentazione dell'RNAi basato su piastre per avviare il silenziamento del reporter. Fornisce inoltre una cronologia dettagliata per il passaggio di animali da una generazione all'altra isolando embrioni in utero da adulti gravidi mediante trattamento con ipoclorito alcalino ("sbiancamento"). Vengono inoltre descritti metodi e dati rappresentativi per il monitoraggio della frequenza del silenziamento della GFP in un sottogruppo della popolazione mediante microscopia a fluorescenza e per la ChIP-qPCR dell'istone H3K9me3. I saggi di ereditarietà dell'RNAi basati su reporter forniscono un sistema altamente trattabile per sezionare funzionalmente i ruoli dei fattori genetici e ambientali nella regolazione epigenetica 13,14 e gli screening genetici che utilizzano tali reporter hanno identificato sia i geni che sononecessari per 2,3,15 che i geni che regolano negativamente 16,17 la durata dell'eredità epigenetica transgenerazionale.
Protocollo
NOTA: Nella Figura 1 è fornita una sequenza temporale del saggio.
1. Preparazione di piastre per la crescita di nematodi RNAi (RNAi-NGM)
- Eliminare E. coli HT115(DE3) contenente GFP o vettori RNAi di controllo da stock di glicerolo su piastre di agar Luria-Bertani (LB) integrate con 100 μg/mL di ampicillina. Incubare i batteri per una notte a 37 °C.
- Il giorno seguente, preparare le piastre RNAi-NGM (1,7% [p/v] agar, 0,3% [p/v] NaCl, 0,25% [p/v] peptone, 1 mM CaCl2, 5 μg/mL di colesterolo, 25 mM tampone fosfato di potassio [pH 6,0], 1 mM MgSO4, 25 μg/mL di carbenicillina e 5 mM isopropilico β-d-1-tiogalattopiranoside [IPTG]) secondo il protocollo standard18.
- Per ciascun ceppo del saggio, preparare un minimo di sei piastre RNAi-NGM seminate con GFP RNAi (due piastre per ciascuna delle tre repliche biologiche) e due piastre RNAi-NGM seminate con RNAi di controllo (una replica biologica).
- Copri le piastre con un foglio di alluminio per proteggerle dalla luce e lasciale asciugare per una notte a temperatura ambiente.
- Lo stesso giorno del versamento della piastra RNAi-NGM, preparare 4 mL di colture liquide di batteri RNAi.
- In condizioni sterili, aliquota di 4 mL di brodo LB integrato con 10 μg/mL di tetraciclina e 100 μg/mL di ampicillina in provette di coltura. Prelevare singole colonie dalle piastre di agar LB in ciascuna provetta e incubare per una notte agitando per circa 16 ore a 37 °C.
- Seminare i batteri RNAi sulle piastre RNAi-NGM.
- Dopo la crescita notturna, misurare la densità ottica (OD600) delle colture utilizzando una diluizione 1:5 dei batteri con brodo LB.
- Diluire i batteri RNAi a un OD relativo di 600 di 2 utilizzando brodo LB integrato con 10 μg/mL di tetraciclina e100 μg/mL di ampicillina.
- In condizioni sterili, aggiungere 150 μL di batteri RNAi di controllo o GFP su ciascuna piastra RNAi-NGM.
- Coprire le piastre con un foglio di alluminio e lasciare crescere i batteri per almeno 24 ore a temperatura ambiente prima dell'uso.
NOTA: Le piastre RNAi-NGM non utilizzate possono essere conservate capovolte per un massimo di 1 settimana a 4 °C in un contenitore sigillato al buio.
2. Avvio del saggio di ereditarietà dell'RNAi: sbiancamento e placcatura di embrioni per la generazione di P0
NOTA: Prima di iniziare il saggio di ereditarietà dell'RNAi, i vermi contenenti il reporter GFP mjIs134 [mex-5p::gfp-h2b::tbb-2 3'UTR]7 devono essere mantenuti senza fame a 21 °C per almeno due generazioni.
- A 4 giorni (96 ore) prima dell'inizio del test di ereditarietà dell'RNAi (Figura 1), prelevare tre o quattro adulti gravidi su ciascuna piastra NGM standard da 35 mm seminata con batteri OP50-1.
NOTA: Sono sufficienti tre piastre NGM per ceppo. Per i ceppi con fertilità compromessa, possono essere necessari più di quattro animali per piastra. - Dopo 4 giorni, assicurarsi che la popolazione adulta abbia iniziato a deporre gli embrioni sulla piastra. Lavare i vermi dalle piastre in provette da 1,5 mL utilizzando 800 μL di tampone M9 integrato con Triton X-100 (TX-100) (22 mM KH 2 PO 4, 42 mM Na2HPO 4, 86 mM NaCl, 1 mM MgSO 4, 0,01% (v/v) TX-100). Ripetere il lavaggio e il pooling nello stesso tubo.
- Isolare gli embrioni dalla popolazione mediante sbiancamento come segue.
- Preparare una soluzione sbiancante con candeggina (ipoclorito di sodio al 6%) e 10 N NaOH in un rapporto di volume di 1,5:1. Preparare una quantità sufficiente in modo tale da poter utilizzare 250 μL per ciascun campione.
- Centrifugare le provette da 1,5 mL con i vermi a 1.000 x g per 1,5-2 min.
- Aspirare il surnatante, lasciando 100 μL senza disturbare il "pellet" del verme. In alternativa, i tubi possono essere lasciati per circa 2 minuti per consentire ai vermi di depositarsi prima di aspirare.
- Aggiungere 650 μL di ddH2O a ciascuna provetta per ottenere un volume finale di 750 μL.
NOTA: I seguenti passaggi sono sensibili al fattore tempo. - Aggiungere 250 μL di soluzione sbiancante in ciascuna provetta e avviare un timer.
- Ogni 1-2 minuti, agitare accuratamente i tubi. Dopo 5 minuti, controllare i vermi sotto lo stereoscopio per monitorare la degradazione degli adulti.
- Continuare a vorticare fino a quando i vermi non si sono completamente sciolti e rimangono solo embrioni. Centrifugare immediatamente le provette a 1.000 x g per 1,5 min.
NOTA: Lo sbiancamento è generalmente completo entro 6-7 minuti, ma deve essere monitorato con uno stereoscopio con un ingrandimento sufficiente per osservare gli embrioni rilasciati (40x). Non lasciare i vermi nella candeggina per un periodo prolungato, poiché ciò comprometterebbe gli embrioni. - Dopo la centrifugazione, aspirare il surnatante e lasciare circa 50-100 μL. Aggiungere 1 mL di tampone M9 con TX-100 per lavare e miscelare mediante vortex. Centrifugare di nuovo a 1.000 x g per 1,5-2 minuti e lavare almeno altre due volte.
- Aspirare il surnatante dal lavaggio finale e lasciare circa 100 μL nella provetta. Miscelare mediante vortex. Pipettare due volte 2 μL da ciascuna provetta su un vetrino etichettato. Contare gli embrioni utilizzando un contatore per stimare la concentrazione di embrioni per microlitro.
NOTA: I vetrini con più pozzetti smerigliati possono essere utilizzati qui per contare le repliche di più ceppi. I vetrini di conteggio possono essere lavati e riutilizzati.
- Per avviare una crescita semi-sincronizzata della popolazione, miscelare e pipettare un volume contenente 250 embrioni su ciascuna piastra RNAi-NGM. Utilizzare due piastre per ogni replica. Lasciare assorbire il liquido.
- Per la generazione P0 trattata con RNAi, incubare a 21 °C per 4 giorni (96 h) (dagli embrioni all'età adulta). I tempi possono variare a seconda del genotipo.
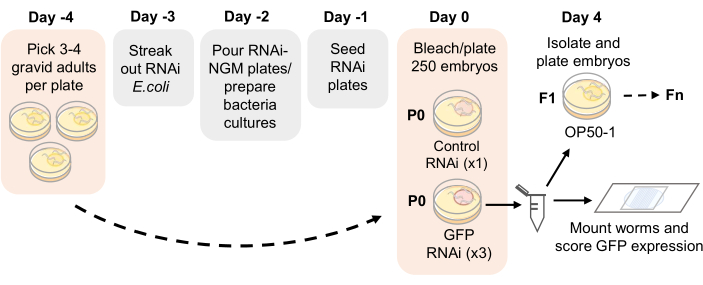
Figura 1: Schema del saggio di ereditarietà dell'RNAi. Tempistica proposta per la preparazione della piastra RNAi-NGM e l'impostazione del saggio di ereditarietà RNAi. Gli adulti gravidi vengono raccolti il giorno -4 su piastre NGM seminate con OP50-1. Dopo 4 giorni, la progenie adulta viene sbiancata e gli embrioni vengono placcati su piastre RNAi-NGM. La generazione di P0 è esposta all'RNAi per 4 giorni a 21 °C. Una volta che i vermi raggiungono l'età adulta, le repliche vengono passate per sbiancamento e valutate per l'espressione germinale della GFP ad ogni generazione. Fare clic qui per visualizzare una versione più grande di questa figura.
3. Passare e assegnare un punteggio a ciascuna generazione per l'espressione germinale della GFP
NOTA: Per facilitare l'incisione, utilizzare un disco in vinile per creare cuscinetti di agarosio con creste lineari per allineare i vermi. Questo metodo è stato adattato da Rivera Gomez e Schvarzstein19.
- Preparare l'agarosio all'1% (p/v) sciogliendo l'agarosio in ddH2O in un matraccio riscaldandolo in microonde. Aggiungere una barra per mescolare e coprire con un foglio. In caso di rifusione, scaldare l'agarosio su una piastra a 200 °C mescolando. Una volta sciolto, abbassate la fiamma a 80 °C.
- Lavare via i vermi dalle piastre NGM utilizzando 800 μL di tampone M9 con TX-100 in una provetta da 1,5 mL. Lavare le piastre due volte per rimuovere tutti gli adulti gravidi. Controllare le piastre sotto lo stereoscopio per confermare che gli animali sono stati raccolti in modo efficiente.
- Centrifugare le provette a 1.000 x g per 1,5-2 minuti o lasciare che i vermi si depositino per 2 minuti. Aspirare il surnatante e lasciare 100 μL senza disturbare il "pellet" del verme.
- Preparare i cuscinetti di agarosio per il montaggio dei vermi come segue.
- Mettere una goccia di agarosio fuso all'1% (p/v) sul disco in vinile (descritto in precedenza19) usando una pipetta Pasteur di vetro (con l'estremità stretta rotta).
- Orientare un vetrino in modo che le linee sul disco siano orizzontali o verticali e posizionare rapidamente il vetrino sulla goccia di agarosio. Attendere circa 30 secondi prima di rimuovere il vetrino dal disco.
NOTA: Se si segnano più genotipi, può essere utile fare un tampone di agarosio più grande su un vetrino e tagliarlo a metà usando una lama.
- Montare i vermi sui cuscinetti di agarosio.
- Sotto lo stereoscopio, aggiungere circa 5 μL di levamisolo 5 mM al cuscinetto di agarosio. La diluizione con levamisolo deve essere preparata fresca ogni settimana.
- Muovere delicatamente i tubi di vermi per mescolare e trasferire 5-10 μL di vermi sul tampone di agarosio. Stimare il numero di vermi a occhio mentre vengono aggiunti, in modo che ci siano circa 40 vermi per replica.
- Allinea i vermi in file usando un plettro per ciglia per facilitare l'incisione. Aggiungi un vetrino coprioggetti.
NOTA: I vetrini con animali montati in questo modo possono durare diverse ore prima di asciugarsi.
- Isolare gli embrioni dagli animali rimanenti utilizzando il protocollo di sbiancamento (fare riferimento al punto 2.3) prima di assegnare un punteggio ai vetrini. Placcare 250 embrioni su piastre NGM da 35 mm seminate con OP50-120. Una volta assorbito il liquido, capovolgere le piastre e rimetterle nell'incubatore a 21 °C.
- Utilizzare uno stereoscopio a fluorescenza con un set di filtri GFP. Contare e registrare il numero di vermi GFP positivi e GFP negativi sui vetrini per ogni replica utilizzando un contatore di conteggio.
NOTA: I vermi GFP positivi avranno un'espressione nucleare di GFP nella loro linea germinale e negli embrioni in utero. Le linee germinali dei vermi GFP negativi non saranno fluorescenti, tuttavia, quando la GFP inizia a riaccendersi nelle generazioni successive, la fluorescenza potrebbe essere fioca. Può essere utile montare ulteriori ceppi di vermi non transgenici per tenere conto dell'eventuale autofluorescenza osservata. - Passaggio della popolazione per sbiancamento, come sopra descritto, ogni 4 giorni circa (96 h) a 21 °C. In alternativa, il passaggio può essere effettuato con un programma alternato di 3 giorni/4 giorni per comodità. Impiattare ~50 embrioni in più per le generazioni di passaggio più brevi se alternati, poiché potrebbe esserci una resa inferiore di embrioni dopo 72 ore.
NOTA: Mantenere costante il tempo di passaggio della generazione P0 a 4 giorni dopo la placcatura degli embrioni.
4. Raccolta di animali per ChIP
NOTA: Il numero di animali e la tempistica dipendono dal ceppo, dallo stadio di sviluppo, dall'epitopo e dal numero di bersagli di immunoprecipitazione (IP). Nell'esempio seguente, raccogliere gli animali per tre IP: H3K9me3, istone H3 e controllo IgG. Il protocollo ChIP è stato adattato da Askjaer et al.21.
- Prima della raccolta del campione ChIP, espandere la generazione precedente del saggio di ereditarietà dell'RNAi di altre tre piastre NGM (ad almeno quattro piastre per replica). Coltivare per 4 giorni a 21 °C.
- Sbiancare gli adulti gravidi per isolare gli embrioni (fare riferimento al punto 2.3).
- Per ogni ceppo, impiattare circa 3.500 embrioni su 14 piastre (250 per piastra) e far crescere per 3 giorni a 21 °C fino allo stadio di giovane adulto.
- Lavare gli animali in provette da 1,5 mL con soluzione salina tamponata con fosfato (PBS) contenente lo 0,01% (v/v) di TX-100 (PBS/TX). Centrifugare a 1.000 x g per 2 min. Aspirare e raggruppare gli animali di un ceppo in una provetta e lavare altre tre volte con almeno 1 mL di PBS/TX.
- Aspirare a 1.000 μL, capovolgere per miscelare e contare il numero di animali in 3 μL per tre volte (fare riferimento al punto 2.3.9).
- Calcolare la concentrazione di animali e trasferire un volume corrispondente a 3.000 animali in una nuova provetta per la reticolazione della formaldeide.
- Assicurarsi che il volume totale sia almeno 10 volte il volume del pellet di vite senza fine.
5. Reticolazione della formaldeide
ATTENZIONE: Lavorare con la formaldeide in una cappa aspirante per prevenire l'esposizione al vapore.
- Aggiungere formaldeide (concentrazione finale dell'1,8%) nella provetta contenente i vermi. Ruotare a temperatura ambiente per 6 min.
- Congelare immediatamente in azoto liquido. In questa fase l'esperimento può essere messo in pausa e i campioni possono essere conservati a -80 °C.
- Scongelare il campione reticolato a bagnomaria a temperatura ambiente per 3 minuti e ruotare per 16 minuti a temperatura ambiente.
- Aggiungere 1,25 M di glicina a una concentrazione finale di 125 mM e ruotare per 5 minuti.
NOTA: Fino all'eluizione delle microsfere magnetiche, conservare i campioni su ghiaccio o a 4 °C e utilizzare tamponi ghiacciati. - Centrifugare il campione a 1.000 x g per 3 minuti. Lavare tre volte con 1 mL di PBS/TX ogni volta. Lavare due volte con 1 mL di tampone di risospensione (150 mM NaCl, 50 mM HEPES-KOH [pH 7,5], 1 mM di acido etilendiamminotetraacetico [EDTA], 0,01% TX-100, inibitore della proteasi [una compressa per 5 mL]).
- Dopo l'ultimo lavaggio, lasciare una quantità sufficiente di tampone per garantire che il volume totale sia almeno tre volte il volume del pellet e un minimo di ~100 μL.
6. Sonicazione
NOTA: I parametri di sonicazione dipendono dal tipo e dal modello del sonicatore e dello stadio animale. Parametri come il volume e la concentrazione del campione, gli intervalli di accensione/spegnimento, il numero di cicli e l'impostazione della potenza devono essere ottimizzati empiricamente. Ad esempio, nel corso di un periodo di sonicazione, monitorare la lisi dei vermi utilizzando un saggio proteico e determinare quando la concentrazione raggiunge un plateau. Inoltre, monitorare quando la dimensione media di taglio del DNA genomico è di circa 200-1.000 bp mediante elettroforesi del DNA, purificato dopo l'inversione della reticolazione, su un gel di agarosio/tris-acetato-EDTA (TAE) all'1,5%.
- Misurare il volume dei campioni del passaggio precedente. Miscelare e aliquotare 90-120 μL in un tubo di sonicazione in polistirolo.
- Aggiungere un volume uguale di tampone di risospensione contenente 2 detergenti (150 mM NaCl, 50 mM HEPES-KOH [pH 7,5], 1 mM EDTA, 0,2% desossicolato di sodio, 0,7% sarkosyl).
- Sonicare in un sonicatore a bagnomaria al 50% della potenza per 7 min (20 s acceso/40 s spento) a 4 °C. Miscelare delicatamente mediante pipettaggio. Ripetere la sonicazione per altri 7 minuti.
- Trasferire il lisato sonicato in una provetta da 1,5 mL. Aggiungere 0,5 volumi di tampone di risospensione senza detergenti (150 mM NaCl, 50 mM HEPES-KOH [pH 7,5], 1 mM EDTA) (ad esempio, aggiungere 100 μL di tampone a 200 μL di campione).
- Centrifugare a 13.000 x g per 15 minuti a 4 °C. Conservare il surnatante del lisato e trasferirlo in una nuova provetta.
- Facoltativamente, eseguire un test proteico per determinare le concentrazioni di ciascun lisato. Questo passaggio può essere utile per grandi quantità di campione o confronti tra saggi. Tuttavia, la standardizzazione del numero di animali come descritto sopra funziona bene per le quantità di campione qui descritte, poiché esiste una migliore correlazione con la resa di cromatina/DNA determinata dalla qPCR.
7. Immunoprecipitazione
NOTA: Adattare la quantità di anticorpi e microsfere magnetiche al volume e alla concentrazione del lisato
- Dividere il surnatante del lisato in quattro porzioni: tre volumi uguali per ogni IP e il 10% del volume di un IP come ingresso (ad esempio, 100 μL di IP #1, 100 μL di IP #2, 100 μL di IP #3, 10 μL di ingresso). Trasferire il lisato IP in una provetta PCR da 200 μL e conservare il lisato di ingresso a -20 °C in una provetta da 1,5 mL.
- Aggiungere 0,5 μg di anticorpo anti-H3K9me3, anticorpo anti-istone H3 o IgG al campione IP appropriato. Incubare a 4 °C per una notte con rotazione.
- Il giorno seguente, aliquotare 9 μL di microsfere magnetiche rivestite di proteina G per IP in una singola provetta da 1,5 mL. Lavare le perle due volte con 1 mL di FA-150 (150 mM NaCl, 50 mM HEPES-KOH [pH 7,5], 1 mM EDTA, 1% TX-100, 0,1% desossicolato di sodio).
- Risospendere le perle magnetiche nel tampone FA-150 al volume originale prelevato dal magazzino nel passaggio 7.3 di cui sopra. Aggiungere 7,5 μL a ciascun IP. Incubare a 4 °C per 2 h con rotazione.
8. Lavaggi ed eluizione
NOTA: Per garantire che le microsfere magnetiche non si asciughino, aggiungere rapidamente ogni lavaggio o tampone di eluizione dopo aver aspirato il lavaggio precedente.
- Utilizzare ogni volta 0,2-1 mL delle seguenti soluzioni tampone per lavare le perle. Raccogli le perline su un supporto magnetico per aspirare i lavaggi. Incubare ogni lavaggio a 4 °C per 5 minuti con rotazione. Segui l'ordine come di seguito.
- Lavare due volte con FA-150.
- Lavare una volta con FA-1M (FA-150 con 1 M NaCl).
- Lavare una volta con FA-0.5M (FA-150 con 0.5 M NaCl). Trasferire in una nuova provetta PCR.
- Lavare una volta con TE-LiCl (250 mM LiCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 1% IGEPAL CA-630, 1% desossicolato di sodio).
- Lavare due volte con TE+ (50 mM NaCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 0,005% IGEPAL CA-630).
NOTA: Continuare l'elaborazione del campione a temperatura ambiente, se non diversamente indicato.
- Aspirare l'ultimo tampone di lavaggio. Risospendere le biglie magnetiche con 50 μL di tampone di eluizione ChIP (200 mM NaCl, 10 mM Tris-Cl [pH 8,0], 1 mM EDTA, 1% sodio dodecil solfato [SDS]) e trasferire in una provetta da 1,5 mL.
- Eluire a 65 °C per 15 minuti in termomiscelatore con miscelazione a 1.000 giri/min per 5 secondi al minuto.
- Raccogli le perline su un supporto magnetico e trasferisci il surnatante in una nuova provetta.
- Ripetere l'eluizione con altri 50 μL di tampone di eluizione ChIP. Raggruppare i surnatanti per un totale di 100 μL. Procedere alla reticolazione inversa e all'eluizione del DNA, come descritto di seguito.
9. Reticolazione inversa ed eluizione del DNA
- Scongelare il campione di lisato in ingresso. Rabboccare fino a 100 μL con tampone di eluizione ChIP.
- Aggiungere 16,5 μg di RNasi A a ciascun campione IP e di ingresso. Incubare a 37 °C per 1 h.
- Aggiungere 40 μg di proteinasi K e incubare a 55 °C per 2 ore. Quindi, incubare a 65 °C per una notte.
- Raffreddare i campioni a temperatura ambiente e purificare il DNA con un kit di colonna rotante.
10. Impostazione ed esecuzione della reazione qPCR
NOTA: Il primer, la configurazione della reazione e i parametri del termociclatore devono essere modificati in modo che corrispondano alle raccomandazioni del produttore per la miscela di reazione qPCR in uso.
- Preparare set di primer mirati al reporter GFP RNAi e alle regioni di controllo dell'arricchimento positivo e negativo H3K9me3. La temperatura di fusione del primer è di 60 °C. Vedere la tabella dei materiali per le sequenze di primer.
- Per il DNA in ingresso, effettuare quattro diluizioni seriali quadruple (ad esempio, 1:5, 1:20, 1:80, 1:320).
NOTA: L'IP DNA può essere utilizzato direttamente o diluito (ad esempio, 1:2 o 1:3) per consentire più reazioni. L'idoneità della diluizione deve essere determinata empiricamente, poiché le prestazioni della qPCR possono essere influenzate. - Organizzare tutte le reazioni corrispondenti a un set di primer su una piastra PCR. Impostare le reazioni tecniche duplicate per ogni diluizione del DNA in ingresso e per ogni campione di DNA IP. Ogni reazione da 10 μL contiene 1,5 μL di DNA in ingresso o IP (o tampone di eluizione come controllo), miscela master qPCR (1x concentrazione finale), primer diretto e primer inverso (concentrazione finale di 400 nM per ciascun primer).
- Su un termociclatore in tempo reale, eseguire il seguente programma: denaturazione iniziale: 95 °C per 4 min; amplificazione e rivelazione di fluorescenza: 40 cicli di 95 °C per 10 s e 60 °C per 30 s con lettura su piastra; prolungamento finale: 60 °C per 5 min; curva di fusione: da 60 °C a 90 °C con incrementi di 0,5 °C, 5 s per passo.
11. Determinazione dell'efficienza di amplificazione e verifica della specificità del prodotto
- Per ogni serie di diluizioni del DNA in ingresso, tracciare i quattro punti dati con [log10(1/diluizione)] sull'asse x e [Input Cq] sull'asse y. Determinare la pendenza della linea di adattamento.
- Calcola l'efficienza di amplificazione. I set di primer ideali dovrebbero mostrare costantemente un'efficienza del 95%-100%.
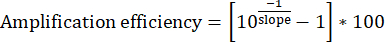
- Verificare che tutte le reazioni abbiano un picco acuto della curva di fusione e che le reazioni con lo stesso primer impostato abbiano la stessa temperatura di fusione. Picchi multipli o diverse temperature di fusione possono indicare un'amplificazione non specifica.
- Facoltativamente, eseguire le reazioni su un gel standard di agarosio/TAE al 2% a temperatura ambiente per verificare le dimensioni della banda del prodotto.
12. Calcolo della percentuale di input
- Calcolare il fattore di diluizione in ingresso. Poiché il 10% del volume di lisato IP è stato salvato come input, il fattore di diluizione per la diluizione 1:5 del DNA in ingresso è 50.
- Determinare il fattore di diluizione IP. Se il DNA IP non viene diluito prima della qPCR, il fattore di diluizione è 1.
- Calcolare la differenza Cq tra l'IP e l'ingresso regolato per i fattori di diluizione.
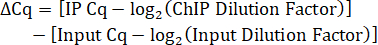
- Calcola la percentuale di input.
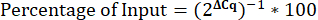
Risultati Rappresentativi
Gli animali portatori del reporter H2B [mex-5p::gfp-h2b::tbb-2 3'UTR]7 espresso dalla linea germinale (Figura 2A) sono stati esposti a GFP RNAi o RNAi di controllo attraverso l'alimentazione e sono passati come descritto nel protocollo e nella Figura 1. Il segnale nucleare GFP nella linea germinale è stato valutato manualmente utilizzando un microscopio da dissezione a fluorescenza per un campione della popolazione ad ogni generazione. Il silenziamento del transgene è risultato completamente penetrante negli animali con punteggio P0 e F1 trattati con GFP RNAi (Figura 2B). Nella generazione F2, la percentuale della popolazione che mostrava un'ereditarietà del silenziamento GFP era di circa il 50%. Dalla generazione F5, la maggior parte della popolazione non ha mostrato ereditarietà del silenziamento e dalla generazione F10 non è stata rilevata alcuna ereditarietà, poiché tutti gli animali hanno espresso GFP.
Per determinare la variazione dell'arricchimento dell'istone H3K9me3 corrispondente al silenziamento indotto dall'RNAi, la ChIP-qPCR è stata eseguita su animali della generazione F1 dopo il trattamento con GFP RNAi o RNAi di controllo. Come previsto, la popolazione trattata con GFP RNAi ha mostrato livelli più elevati di istone H3K9me3 al bersaglio GFP e nella regione a valle di 1,3 kb rispetto agli animali di controllo trattati con RNAi (Figura 2C). La specificità dell'istone H3K9me3 ChIP è supportata dall'arricchimento in un locus di controllo positivo (clec-18) noto per essere arricchito in questo mark, ma non in un locus di controllo negativo vicino (hrp-2). L'arricchimento dell'istone H3 e l'arricchimento near-background nelle immunoprecipitazioni di controllo delle IgG sono stati rilevati anche in tutti i loci qPCR, come previsto. Quando l'arricchimento di ChIP al reporter è normalizzato al locus di controllo positivo clec-18 , viene mostrato un maggiore arricchimento dell'istone H3K9me3 su GFP RNAi, mentre l'arricchimento dell'istone H3 è simile tra il controllo e i trattamenti con GFP RNAi (Figura 2D). Poiché non si prevede che l'RNAi GFP influenzi l'istone H3K9me3 o l'occupazione totale dell'istone H3 nel locus clec-18 , questa normalizzazione mitiga le variazioni tecniche, come le differenze nell'efficienza ChIP tra l'RNAi GFP e i campioni di RNAi di controllo. Il fold-change dei livelli di istone H3K9me3 e H3 tra i trattamenti con RNAi mostra l'arricchimento dell'istone H3K9me3 specifico per il reporter GFP, indipendentemente dall'occupazione istonica, in seguito al silenziamento indotto da GFP RNAi (Figura 2E).
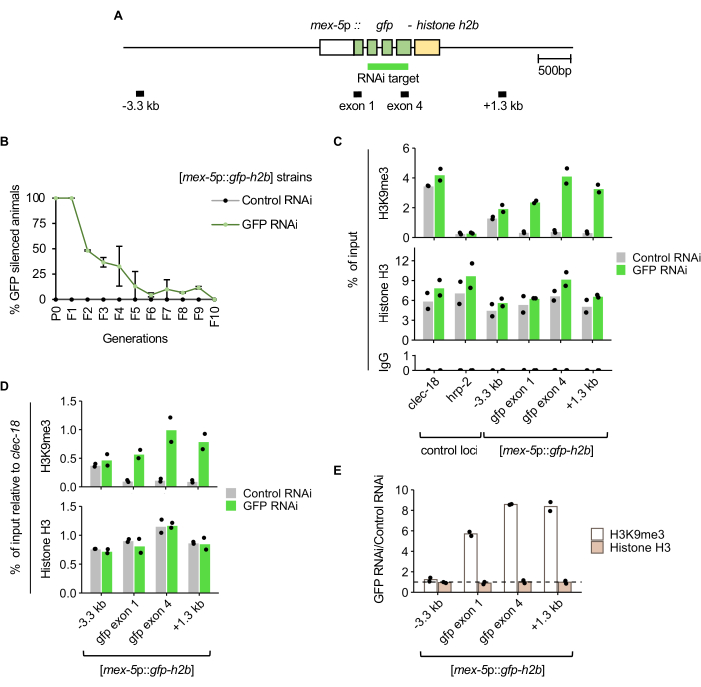
Figura 2: Il silenziamento indotto da GFP RNAi corrisponde a un elevato arricchimento di H3K9me3 nel bersaglio dell'RNAi. (A) Diagramma del reporter dell'RNAi GFP espresso dalla linea germinale mjIs134[mex-5p::gfp-h2b::tbb-2 3'UTR] con regioni di ampliconi qPCR marcate. (B) L'espressione della GFP è stata valutata attraverso le generazioni dopo trattamenti con RNAi a 21 °C. Le barre di errore rappresentano la deviazione standard di due repliche biologiche. (C) ChIP-qPCR di H3K9me3, istone H3 e controllo IgG in giovani adulti F1 da due repliche biologiche. clec-18 e hrp-2 sono rispettivamente i loci di controllo positivo e negativo per l'arricchimento di H3K9me3. (D) L'arricchimento di H3K9me3 e dell'istone H3 al reporter GFP RNAi normalizzato al locus di controllo positivo clec-18. (E) Variazione dell'arricchimento di H3K9me3 e dell'istone H3 tra gli animali trattati con GFP RNAi e RNAi di controllo, con normalizzazione a clec-18. La linea tratteggiata rappresenta un cambio di piegatura pari a 1. Fare clic qui per visualizzare una versione più grande di questa figura.
Discussione
In questo protocollo, il dsRNA viene introdotto attraverso l'alimentazione, che è diventato un metodo standard in C. elegans18. Per i saggi di ereditarietà dell'RNAi, l'approccio di alimentazione fornisce un metodo semplice per ottenere un'ampia popolazione P0 2,11,12,22,23,24,25. Tuttavia, la tempistica e la durata dell'esposizione all'RNAi influenzano l'efficacia del silenziamento transgenico26 e la concentrazione dei batteri RNAi influisce sulla persistenza del silenziamento ereditario dell'RNAi1. Pertanto, la crescita standardizzata dei batteri RNAi e dei vermi è importante per ottenere un livello costante di silenziamento della GFP e una durata dell'ereditarietà. Qui, gli embrioni vengono placcati su piastre RNAi in modo che gli animali P0 siano esposti ai batteri RNAi dalla schiusa. Approcci alternativi hanno placcato animali sincronizzati in stadio L1 24,27 oL4 25 su piastre RNAi-NGM. Inoltre, poiché la fame e altri stress influenzano il mantenimento dell'ereditarietà dell'RNAi13, le piastre devono essere monitorate per prevenire il sovraffollamento e l'esaurimento delle scorte di cibo. In alternativa all'inizio dell'RNAi attraverso l'alimentazione, alcuni dei pionieri dell'ereditarietà dell'RNAi di C. elegans hanno indotto l'RNAi attraverso l'iniezione di gonadi, che fornisce un maggiore controllo della concentrazione di dsRNA 1,28.
In questo protocollo, ogni generazione viene fatta passare come una popolazione con trattamento con ipoclorito alcalino, come descritto in precedenza 16,22,23,24. Il trattamento con ipoclorito garantisce che la generazione F1 non venga contaminata da batteri RNAi provenienti dall'ambiente parentale22 e previene potenziali colli di bottiglia indesiderati nella popolazione. Tuttavia, il passaggio di massa può anche essere un limite, poiché i singoli animali possono avere modelli di ereditarietà distinti 1,29. Un metodo alternativo per stabilire ogni generazione è quello di selezionare i singoli animali 1,2,9,11,25. Questo approccio consente di tracciare i fenotipi all'interno dei lignaggi e l'incorporazione di incroci genetici. L'analisi del lignaggio può essere vantaggiosa anche per i fenotipi a bassa penetranza12.
L'espressione della proteina fluorescente è una lettura potente e conveniente del silenziamentoindotto dall'RNAi 2,3,4,12,14,15,16,25,30. Come descritto in questo protocollo, l'espressione GFP può essere valutata manualmente come ON o OFF 11,12,15,16,25. Il punteggio manuale può essere ulteriormente perfezionato assegnando livelli di intensità qualitativa 3,4,14. In alternativa, il punteggio automatico dell'intensità delle immagini al microscopio 11,12,14,25,27 o la misurazione della fluorescenza con citometria a flussodi animali vivi 2 possono fornire una lettura quantitativa e ad alto rendimento. Tuttavia, poiché l'espressione reporter è limitata alla linea germinale, un avvertimento degli approcci automatizzati è che devono anche differenziare tra animali con espressione GFP silenziata rispetto ad animali senza linea germinale, specialmente se lo studio incorpora mutanti con sviluppo germinale anormale. In alternativa alla fluorescenza GFP, la trascrizione inversa (RT)-qPCR può essere utilizzata per quantificare i livelli di pre-mRNA e mRNAtarget RNAi 2,16,23,24. Questo approccio fornisce una lettura più diretta del silenziamento, che prende di mira l'RNA, ed è particolarmente utile per altri bersagli RNAi in cui il silenziamento non produce un fenotipo visibile. Un limite dell'uso di reporter GFP artificiali è che le sequenze esogene ed endogene sono regolate in modo differenziale nell'RNAitransgenerazionale 11. Gli studi con bersagli endogeni, come l'allele letale embrionale sensibile alla temperatura oma-1(zu405)1,11,16,25, dovrebbero quindi essere considerati come un approccio complementare ai reporter transgenici fluorescenti.
L'analisi della ChIP nel contesto dei saggi di ereditarietà dell'RNAi richiede il confronto tra trattamenti e repliche. Innanzitutto, per tenere conto delle differenze nel materiale di partenza tra i campioni, il segnale ChIP viene normalizzato al segnale di ingresso nello stesso luogo della "percentuale di ingresso". L'elaborazione parallela di un ChIP H3 dell'istone aiuterà a determinare se le alterazioni della modificazione dell'istone corrispondono a variazioni nella densità del nucleosoma. Inoltre, poiché ChIP è un processo a più fasi, l'efficienza può variare da un campione all'altro. La selezione di appropriati loci di controllo positivi e negativi è utile per valutare e confrontare il rapporto segnale/rumore tra campioni ed esperimenti. Inoltre, per facilitare i confronti tra i campioni, i valori del ciclo di soglia qPCR del DNA ChIP sul bersaglio RNAi sono spesso normalizzati in un locusdi controllo 3,11,15,23,30,31. Per valutare gli effetti del trattamento con RNAi, viene anche confrontato il rapporto tra il segnale ChIP nell'RNAi di trattamento e le condizioni di controllo o di assenza di RNAi. Un limite dell'approccio attuale è che la ChIP viene eseguita con animali interi, mentre la risposta al trattamento con RNAi può essere unica per la linea germinale. Un approccio per superare questo avvertimento consiste nell'eseguire ChIP utilizzando nuclei germinali isolati. Ulteriori considerazioni tecniche per l'ottimizzazione di ChIP sono state ampiamente discusse altrove32,33.
Nel complesso, questo protocollo di ereditarietà dell'RNAi e ChIP fornisce una base dettagliata e facile da adattare che può essere integrata con altre tecniche per esplorare ulteriormente la regolazione epigenetica transgenerazionale. Ad esempio, è possibile costruire librerie di sequenziamento ad alto rendimento a partire dal DNA ChIP (ChIP-seq) per una visione più dettagliata del panorama della cromatina sia prossimale al bersaglio RNAi che su scala genome-wide.
Divulgazioni
Tutti gli autori confermano di non avere conflitti da rivelare.
Riconoscimenti
Desideriamo ringraziare i laboratori della comunità di C. elegans che hanno sviluppato e condiviso gli strumenti e il cui lavoro è citato in questo manoscritto. Alcuni ceppi sono stati forniti dal CGC, che è finanziato dall'Ufficio NIH dei programmi di infrastruttura di ricerca (P40 OD010440). Questo lavoro è stato sostenuto da una sovvenzione del progetto Canadian Institutes of Health Research (CIHR) ad A.L.S. (PJT-175245). CL è supportato da una borsa di studio post-laurea (PGS-D) del Natural Sciences and Engineering Research Council of Canada (NSERC).
Materiali
| Name | Company | Catalog Number | Comments |
| Agarose | Bioshop | AGA002 | |
| Ampicillin | Bioshop | AMP201 | Make a 100 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Anti-H3K9me3 Rabbit Polyclonal Antibody | Abcam | ab8898 | Concentration is batch-dependent (0.9 - 1 mg/mL). |
| Anti-Histone H3 Rabbit Polyclonal Antibody | Abcam | ab1791 | Concentration is batch-dependent (0.7 - 1 mg/mL). |
| Bleach (6% Sodium hypochlorite) | Lavo | 02358107 | |
| C. elegans strain with GFP RNAi Reporter | NA | SX1263 | Sapetschnig et al. 2015 (ref. 7). A gift from E. Miska lab, University of Cambridge. |
| Carbenicillin | BioShop | CAR544 | Make a 25 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| Dynabeads Protein G Magnetic Beads | Invitrogen | 10003D | |
| E. coli strain HT115(DE3) | Caenorhabditis Genetics Center (CGC) | HT115(DE3) | |
| E. coli strain OP50-1 | Caenorhabditis Genetics Center (CGC) | OP50-1 | |
| EDTA (0.5 M, pH 8.0) | Invitrogen | 15575020 | |
| Fluorescence Stereoscope | Zeiss | Axio Zoom.V16 | |
| Formaldehyde (37%) | Sigma | F8775 | |
| Glycine | Sigma | 50046 | Make a 1.25 M solution and store at 4 °C. |
| HEPES-KOH (1 M, pH 7.5) | Teknova | H1035 | |
| Hydrophobic Printed Slides, 10 wells | VWR | 100488-904 | |
| IGEPAL CA-630 (Octylphenol ethoxylate) | BioShop | NON999 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
| IPTG (Isopropyl-β-D-thiogalactoside) | BioShop | IPT001 | Make a 0.2 g/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| iTaq Universal SYBR Green Supermix | Bio-Rad | 1725122 | |
| LB Agar Plates supplemented with 100 µg/mL Ampicillin | NA | NA | Standard lab recipe. |
| Levamisole (Tetramisole hydrochloride) | Sigma | L9756 | Make a 200 mM solution in ultrapure water. Store at -20 °C. |
| LiCl (8 M) | Sigma | L7026 | |
| M9 Buffer | NA | NA | 22 mM KH2PO4, 42 mM Na2HPO4, 86 mM NaCl, 1 mM MgSO4. |
| Magnetic Separator (1.5 mL tubes) | Applied Biosystems | A13346 | |
| Magnetic Separator (0.2 mL tubes) | Permagen | MSR812 | |
| Microscope Cover Glass | Fisher Scientific | 12541B | |
| Microscope Slide | Technologist Choice | LAB-037 | |
| NaCl (5 M) | Promega | V4221 | For ChIP buffers. |
| NaOH | Sigma | S5881 | Make a 10 M solution and store at room temperature. |
| NGM Plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 μg/mL cholesterol, 25 mM Potassium phosphate pH 6.0, 1 mM MgSO4, 50 µg/mL streptomycin. |
| Normal Rabbit IgG (1 mg/mL) | Cell Signaling Technology | 2729 | |
| Petri Dishes (35 mm x 10 mm) | Sarstedt | 82.1135.500 | |
| Phosphate Buffered Saline (10X) | Fisher BioReagents | BP3991 | |
| Plasmid - Control RNAi | Addgene | L4440 (Plasmid #1654) | |
| Plasmid - GFP-targetting RNAi | Addgene | L4417 (Plasmid #1649) | Note, alternative L4440-derived plasmids targeting GFP can be used. |
| Primer pair [-3.3 kb upstream of gfp] | Integrated DNA Technologies | NA | F: AAACCAAAGGACGAGAGATTCA, R: GGCTCGATCAAGTAAAATTTCG |
| Primer pair [+1.3 kb downstream of gfp] | Integrated DNA Technologies | NA | F: TCGACCAGTTCTAAAGTCACCG, R: ACGTGCGGGATCATTTCTTACT |
| Primer pair [clec-18] | Integrated DNA Technologies | NA | F: TGCTCCATGACCTCAACAACA, R: AGTACAGTTCACCGATCCAGA |
| Primer pair [gfp exon 1] | Integrated DNA Technologies | NA | F: CTGGAGTTGTCCCAATTCTTGT, R: GGGTAAGTTTTCCGTATGTTGC |
| Primer pair [gfp exon 4] | Integrated DNA Technologies | NA | F: GATGGCCCTGTCCTTTTACCA, R: ATGCCATGTGTAATCCCAGCA |
| Primer pair [hrp-2] | Integrated DNA Technologies | NA | F: CGTCAACAGGGAGCAGCTG, R: CCTCCGAACTTTCTCTGTCCA |
| Protease Inhibitor Cocktail Tablet | Roche | 11836170001 | |
| Proteinase K | Bioline | BIO-37084 | |
| QIAquick PCR Purification Kit | Qiagen | 28104 | |
| Real-Time PCR Detection System | Bio-Rad | CFX96 | |
| RNAi-NGM plates | NA | NA | 1.7% (w/v) agar, 0.3% (w/v) NaCl, 0.25% (w/v) peptone, 1 mM CaCl2, 5 µg/mL cholesterol, 25 mM Potassium phosphate buffer pH 6.0, 1 mM MgSO4, 25 µg/mL carbenicillin and 5 mM IPTG. |
| RNase A | Sigma | R4642 | |
| Sarkosyl (N-Lauroylsarcosine sodium salt) | Sigma | L5777 | Make a 10% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| SDS | Sigma | 74255 | Make a 10% (w/v) solution and store at room temperature. |
| Sodium deoxycholate | Sigma | 30970 | Make a 5% (w/v) solution and store at room temperature protected from light for a maximum of 1 month. |
| Sonication Tube | Evergreen | 214-3721-010 | |
| Sonication Tube Cap | Evergreen | 300-2911-020 | |
| Sonicator | Qsonica | Q800R3-110 | |
| Streptomycin sulfate | Bioshop | STP101 | Make a 50 mg/mL solution in ultrapure water. Filter-sterilize and store at -20 °C. |
| TAE buffer (1X) | NA | NA | 40 mM Tris, 20 mM acetate, 1 mM EDTA |
| Tally counter clicker | Uline | H-7350 | |
| Tetracycline | Bioshop | TET701 | Make a 5 mg/mL solution in ethanol and store at -20 °C. |
| Thermomixer | Eppendorf | 05-400-205 | |
| Tris-HCl (1 M, pH 8.0) | Invitrogen | 15568025 | |
| Triton X-100 | Sigma | T8787 | Make a 10% (v/v) solution in ultrapure water and store at room temperature. |
Riferimenti
- Alcazar, R. M., Lin, R., Fire, A. Z. Transmission dynamics of heritable silencing induced by double-stranded RNA in Caenorhabditis elegans. Genetics. 180 (3), 1275-1288 (2008).
- Ashe, A., et al. piRNAs can trigger a multigenerational epigenetic memory in the germline of C. elegans. Cell. 150 (1), 88-99 (2012).
- Buckley, B. A., et al. A nuclear Argonaute promotes multigenerational epigenetic inheritance and germline immortality. Nature. 489 (7416), 447-451 (2012).
- Vastenhouw, N. L., et al. Long-term gene silencing by RNAi. Nature. 442 (7105), 882 (2006).
- Lee, H. -. C., et al. C. elegans piRNAs mediate the genome-wide surveillance of germline transcripts. Cell. 150 (1), 78-87 (2012).
- Luteijn, M. J., et al. Extremely stable Piwi-induced gene silencing in Caenorhabditis elegans. The EMBO Journal. 31 (16), 3422-3430 (2012).
- Sapetschnig, A., Sarkies, P., Lehrbach, N. J., Miska, E. A. Tertiary siRNAs mediate paramutation in C. elegans. PLoS Genetics. 11 (3), e1005078 (2015).
- Shirayama, M., et al. piRNAs initiate an epigenetic memory of nonself RNA in the C. elegans germline. Cell. 150 (1), 65-77 (2012).
- Minkina, O., Hunter, C. P. Stable heritable germline silencing directs somatic silencing at an endogenous locus. Molecular Cell. 65 (4), 659-670 (2017).
- Seroussi, U., et al. Mechanisms of epigenetic regulation by C. elegans nuclear RNA interference pathways. Seminars in Cell & Developmental Biology. 127, 142-154 (2022).
- Lev, I., Gingold, H., Rechavi, O. H3K9me3 is required for inheritance of small RNAs that target a unique subset of newly evolved genes. eLife. 8, e40448 (2019).
- Woodhouse, R. M., et al. Chromatin modifiers SET-25 and SET-32 are required for establishment but not long-term maintenance of transgenerational epigenetic inheritance. Cell Reports. 25 (8), 2259-2272 (2018).
- Houri-Zeevi, L., Teichman, G., Gingold, H., Rechavi, O. Stress resets ancestral heritable small RNA responses. eLife. 10, e65797 (2021).
- Houri-Ze'evi, L., et al. A tunable mechanism determines the duration of the transgenerational small RNA inheritance in C. elegans. Cell. 165 (1), 88-99 (2016).
- Spracklin, G., et al. The RNAi inheritance machinery of Caenorhabditis elegans. Genetics. 206 (3), 1403-1416 (2017).
- Perales, R., et al. Transgenerational epigenetic inheritance is negatively regulated by the HERI-1 chromodomain protein. Genetics. 210 (4), 1287-1299 (2018).
- Shukla, A., Perales, R., Kennedy, S. piRNAs coordinate poly(UG) tailing to prevent aberrant and perpetual gene silencing. Current Biology. 31 (20), 4473-4485 (2021).
- Ahringer, J. Reverse GeneticsWormBook: the Online Review of C. elegans Biology. WormBook. , (2006).
- Rivera Gomez, K., Schvarzstein, M. Immobilization of nematodes for live imaging using an agarose pad produced with a Vinyl Record. microPublication Biology. 2018, (2018).
- Stiernagle, T. Maintenance of C. elegans. WormBook: The Online Review of C. Elegans Biology. , 1-11 (2006).
- Askjaer, P., Ercan, S., Meister, P. Modern techniques for the analysis of chromatin and nuclear organization in C. elegans. WormBook: the Online Review of C. elegans Biology. , 1-35 (2014).
- Gu, S. G., et al. Amplification of siRNA in Caenorhabditis elegans generates a transgenerational sequence-targeted histone H3 lysine 9 methylation footprint. Nature Genetics. 44 (2), 157-164 (2012).
- Kalinava, N., Ni, J. Z., Peterman, K., Chen, E., Gu, S. G. Decoupling the downstream effects of germline nuclear RNAi reveals that H3K9me3 is dispensable for heritable RNAi and the maintenance of endogenous siRNA-mediated transcriptional silencing in Caenorhabditis elegans. Epigenetics & Chromatin. 10, 6 (2017).
- Kalinava, N., et al. elegans heterochromatin factor SET-32 plays an essential role in transgenerational establishment of nuclear RNAi-mediated epigenetic silencing. Cell Reports. 25 (8), 2273-2284 (2018).
- Lev, I., et al. MET-2-dependent H3K9 methylation suppresses transgenerational small RNA inheritance. Current Biology. 27 (8), 1138-1147 (2017).
- Kamath, R. S., Martinez-Campos, M., Zipperlen, P., Fraser, A. G., Ahringer, J. Effectiveness of specific RNA-mediated interference through ingested double-stranded RNA in Caenorhabditis elegans. Genome Biology. 2 (1), (2001).
- Xu, F., et al. A cytoplasmic Argonaute protein promotes the inheritance of RNAi. Cell Reports. 23 (8), 2482-2494 (2018).
- Grishok, A., Tabara, H., Mello, C. C. Genetic requirements for inheritance of RNAi in C. elegans. Science. 287 (5462), 2494-2497 (2000).
- Houri-Zeevi, L., Korem Kohanim, Y., Antonova, O., Rechavi, O. Three rules explain transgenerational small RNA inheritance in C. elegans. Cell. 182 (5), 1186-1197 (2020).
- Burton, N. O., Burkhart, K. B., Kennedy, S. Nuclear RNAi maintains heritable gene silencing in Caenorhabditis elegans. Proceedings of the National Academy of Sciences. 108 (49), 19683-19688 (2011).
- Mao, H., et al. The Nrde pathway mediates small-RNA-directed histone H3 lysine 27 trimethylation in Caenorhabditis elegans. Current Biology. 25 (18), 2398-2403 (2015).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22 (9), 1813-1831 (2012).
- Mukhopadhyay, A., Deplancke, B., Walhout, A. J. M., Tissenbaum, H. A. Chromatin immunoprecipitation (ChIP) coupled to detection by quantitative real-time PCR to study transcription factor binding to DNA in Caenorhabditis elegans. Nature Protocols. 3 (4), 698-709 (2008).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneEsplora altri articoli
This article has been published
Video Coming Soon