È necessario avere un abbonamento a JoVE per visualizzare questo. Accedi o inizia la tua prova gratuita.
Generazione e analisi di immagini istologiche ad alto parametro con la citometria a flusso istodinamico
In questo articolo
Riepilogo
Qui descritto è un metodo che può essere utilizzato per visualizzare cinque o più parametri fluorescenti mediante microscopia immunofluorescente. Viene delineata una pipeline di analisi per estrarre singole cellule da queste immagini e condurre analisi di singole cellule attraverso strategie di gating simili alla citometria a flusso, in grado di identificare sottogruppi di cellule in sezioni di tessuto.
Abstract
L'uso dell'istologia per studiare la diversità delle cellule immunitarie in sezioni di tessuto, come quelle derivate dal sistema nervoso centrale (SNC), è limitato in modo critico dal numero di parametri fluorescenti che possono essere visualizzati contemporaneamente. La maggior parte dei sottogruppi di cellule immunitarie è stata definita utilizzando la citometria a flusso utilizzando combinazioni complesse di marcatori proteici, che spesso richiedono quattro o più parametri per essere identificati in modo definitivo, il che va oltre le capacità della maggior parte dei microscopi convenzionali. Poiché la citometria a flusso dissocia i tessuti e perde informazioni spaziali, sono necessarie tecniche in grado di conservare le informazioni spaziali mentre interrogano i ruoli dei tipi di cellule complesse. Questi problemi sono affrontati qui creando un metodo per espandere il numero di parametri fluorescenti che possono essere visualizzati raccogliendo i segnali dei fluorofori spettralmente sovrapposti e utilizzando l'unmixing spettrale per separare i segnali di ogni singolo fluoroforo. Queste immagini vengono quindi elaborate utilizzando una pipeline di analisi per acquisire immagini istologiche ad alto parametro ed estrarre singole cellule da queste immagini in modo che le proprietà fluorescenti uniche di ciascuna cellula possano essere analizzate a livello di singola cellula. Utilizzando strategie di gating simili alla citometria a flusso, le cellule possono quindi essere profilate in sottogruppi e mappate nuovamente sulle sezioni istologiche non solo per quantificare la loro abbondanza, ma anche per stabilire come interagiscono con l'ambiente tissutale. Nel complesso, è stata dimostrata la semplicità e il potenziale dell'utilizzo della citometria a flusso istologico per studiare popolazioni immunitarie complesse nelle sezioni istologiche.
Introduzione
L'infiammazione guidata dalle cellule del sistema immunitario e dalle cellule gliali può contribuire a disturbi cronici del SNC in cui ogni popolazione può promuovere l'attività dell'altra 1,2,3. Capire come il sistema immunitario interagisce con questi elementi del SNC per promuovere l'infiammazione del SNC è attualmente un argomento di grande interesse ed è stato notevolmente facilitato da tecniche ad alto parametro come il sequenziamento dell'RNA a singola cellula. Attraverso il sequenziamento dell'RNA a singola cellula, abbiamo scoperto che esiste un'ampia comunicazione tra le cellule gliali e il sistema immunitario in diversi disturbi del SNC 4,5,6. Capire come queste interazioni stanno influenzando questi disturbi sarà fondamentale per chiarire la biologia di queste malattie.
Un problema con le analisi di sequenziamento di singole cellule è che queste tecniche richiedono che il tessuto venga distrutto per ottenere singole cellule o nuclei, con conseguente perdita completa di informazioni spaziali. Sapere dove si trova una cellula in un tessuto è fondamentale per comprendere il ruolo della cellula nel guidare l'infiammazione. Ad esempio, le cellule immunitarie come le cellule B possono concentrarsi nel SNC durante la neuroinfiammazione; tuttavia, raramente entrano nel parenchima del SNC e si concentrano invece nelle barriereSNC 7. Data la loro localizzazione, è improbabile che queste cellule contribuiscano all'infiammazione del SNC interagendo fisicamente con le cellule gliali nel parenchima del SNC, suggerendo che qualsiasi interazione che potrebbero avere con le cellule gliali si verificherebbe attraverso fattori secrenti. Inoltre, la patologia che si verifica nei disturbi del SNC ha spesso una struttura 8,9 tale che la localizzazione di una cellula nel tessuto potrebbe determinare in modo critico se sta contribuendo attivamente al disturbo o è uno spettatore. Pertanto, l'uso dell'orientamento spaziale per valutare il ruolo di una cellula nella patologia è essenziale.
Lo studio delle cellule nei tessuti è stato in genere realizzato utilizzando l'immunoistochimica o la microscopia a fluorescenza immunitaria. Un problema con queste tecniche è che in genere possono visualizzare solo fino a quattro parametri contemporaneamente. Questo è un grosso limite a queste tecniche, poiché sappiamo dalla citometria a flusso e dalle analisi di sequenziamento dell'RNA a singola cellula che molte popolazioni cellulari richiedono due o più parametri per la loro identificazione; Inoltre, il numero di parametri richiesti aumenta in genere quando si cercano sottoinsiemi specifici di un tipo di cella10. Pertanto, non è pratico utilizzare tecniche di imaging standard per studiare come sottogruppi di cellule possono interagire all'interno di un tessuto.
Questo problema è stato parzialmente superato attraverso nuovi metodi ad alto parametro in grado di conservare le informazioni spaziali, come il sequenziamento spaziale dell'RNA11 e la citometria di massa per imaging12. Sebbene queste tecniche siano preziose, presentano diversi problemi, come la non essere ampiamente disponibili, la riduzione dei dati tridimensionali in due dimensioni e la richiesta di una notevole esperienza per l'esecuzione. Un'altra tecnica nota come colorazione sequenziale, in cui i tessuti vengono colorati con una serie di anticorpi seguita dall'inattivazione della precedente serie di anticorpi prima di colorare con un'altra serie di anticorpi, può ottenere un'istologia ad alto parametro senza la necessità di attrezzature specializzate o competenze 13,14,15. Tuttavia, la colorazione sequenziale può essere estremamente laboriosa e richiede una grande quantità di tempo di microscopia, il che può essere poco pratico per i laboratori che non possiedono un microscopio personale. Pertanto, c'è bisogno di tecniche in grado di espandere il numero di parametri fluorescenti che possono essere visualizzati contemporaneamente su microscopi ampiamente disponibili e in modo tempestivo.
Una volta acquisiti i dati ad alto parametro, sorge un altro problema: è improbabile che i metodi convenzionali di analisi delle immagini riescano ad analizzare con successo i dati. Tecniche come il conteggio manuale o la soglia sono praticabili solo se l'analisi consiste in un singolo parametro o se più marcatori hanno la stessa localizzazione in cui vengono contati solo i segnali sovrapposti. Questa limitazione rende l'analisi tradizionale inadeguata per lavorare con set di dati ad alto parametro. L'analisi di successo di questi set di dati è stata ottenuta segmentando singole cellule da immagini istologiche e quindi conducendo strategie di gating simili alla citometria a flusso per identificare i tipi di cellule16,17. Tuttavia, un altro problema che influisce su queste analisi è che funzionano solo per set di dati in cui tutte le cellule di interesse sono fisicamente separate l'una dall'altra, poiché queste tecniche non impiegano metodi in grado di separare accuratamente le cellule che sono in contatto fisico. Pertanto, è necessario un metodo più recente in grado di condurre analisi di singole cellule su sezioni istologiche anche se le cellule sono in contatto fisico.
In questo articolo viene descritto un semplice protocollo chiamato citometria a istoflusso che è stato precedentemente introdotto18 che espande il numero di parametri fluorescenti che possono essere visualizzati contemporaneamente utilizzando microscopi ampiamente disponibili. Questo protocollo funziona colorando i tessuti con coloranti spettralmente sovrapposti e quindi utilizzando la compensazione spettrale per rimuovere il sanguinamento dai canali sovrapposti per ottenere singole colorazioni chiare. Per facilitare l'analisi di immagini istologiche ad alto parametro, viene descritta una pipeline di analisi dettagliata che estrae singole cellule da sezioni di tessuto allo scopo di smistare le cellule in popolazioni distinte utilizzando strategie di gating simili alla citometria a flusso. Questo protocollo funziona nei tessuti in cui le cellule sono diffusamente presenti e nei tessuti in cui le cellule sono strettamente compattate insieme, rendendo questa tecnica versatile per lo studio di tessuti come il SNC sia nell'omeostasi che nella neuroinfiammazione. La citometria a istoflusso è, quindi, una tecnica utile per studiare le interazioni tra tipi di cellule complesse che richiedono più marcatori cellulari per definire le cellule mantenendo le informazioni spaziali.
Protocollo
Questo protocollo non copre il sezionamento dei tessuti per l'istologia; si veda Jain et al.18 o19 per le descrizioni di come sezionare i tessuti per l'istologia. Questo protocollo può essere utilizzato con qualsiasi tessuto sezionato su vetrini. Questo articolo utilizza linfonodi inguinali isolati da un animale immunizzato come descritto in precedenza18. La procedura e la tempistica di questo protocollo sono riassunte nella Figura 1. I dettagli dei reagenti e delle attrezzature utilizzate in questo studio sono elencati nella tabella dei materiali.
1. Colorazione di sezioni di tessuto
NOTA: Oltre a colorare i tessuti con tutte le etichette degli anticorpi a cui il ricercatore è interessato, il ricercatore dovrebbe anche preparare controlli a singolo colore (uno per ogni parametro fluorescente che si intende utilizzare) su vetrini di tessuto adiacenti o comunque identici in cui ogni singolo controllo del colore è colorato con i reagenti necessari per produrre il segnale di un parametro fluorescente.
- Preparare la permanente e il tampone di blocco e la permanente e il tampone di colorazione (vedere la Tabella 1 per la composizione).
- Scongelare le sezioni di tessuto sui vetrini a temperatura ambiente e rimuovere la condensa con un panno. Usa un pennarello idrofobico per delineare il bordo dei tessuti che devono essere colorati e lascia asciugare i segni.
- Reidratare i tessuti immergendo le sezioni di tessuto in soluzione salina tamponata con fosfato (PBS) per 1 minuto. Scrollarsi di dosso il PBS e utilizzare una salvietta per rimuovere il PBS in eccesso senza toccare direttamente il fazzoletto.
- Fissare i tessuti aggiungendo abbastanza 4% di paraformaldeide (PFA) per coprire completamente il tessuto. Incubare a temperatura ambiente per 20 min.
ATTENZIONE: Il PFA può essere tossico e deve essere maneggiato all'interno di una cappa aspirante.
NOTA: Questo passaggio è necessario solo per i tessuti freschi congelati. Se il tessuto è già fissato, ignora questo passaggio. La fissazione del PFA è descritta qui, ma il tessuto può essere fissato utilizzando altri metodi a seconda delle esigenze dell'esperimento. - Lavare i fazzoletti in un bagno PBS per 1 minuto con un leggero dondolo, quindi rimuovere lo scivolo dalla vasca. Sostituire il PBS e ripetere il lavaggio altre 3 volte.
- Scrollarsi di dosso il PBS in eccesso e utilizzare una salvietta per rimuovere il PBS in eccesso senza toccare direttamente il fazzoletto.
- Determinare quali anticorpi secondari devono essere utilizzati nella fase 1.12 del protocollo e le specie di animali da cui derivano gli anticorpi secondari. Fortificare la permanente e il tampone bloccante aggiungendo siero di questi animali alla permanente e tampone bloccante a una concentrazione finale del 5% per ogni specie.
NOTA: L'uso di anticorpi secondari aiuta ad amplificare l'intensità del segnale. Pertanto, si raccomanda che gli anticorpi primari che producono segnali deboli siano accoppiati con anticorpi secondari per amplificare i loro segnali. Se tutti i bersagli anticorpali che vengono colorati sono sufficientemente luminosi da non richiedere anticorpi secondari, i passaggi 1.7-1.12 e 1.15 possono essere saltati. - Bloccare il legame non specifico degli anticorpi aggiungendo una quantità sufficiente di permanente fortificata e tampone bloccante per coprire ogni tessuto. Incubare i vetrini per 1-8 ore a temperatura ambiente in una camera umidificata al riparo dalla luce.
- Aggiungere anticorpi primari non coniugati al tampone di perm e di colorazione a diluizioni determinate sperimentalmente (le specie o gli isotipi di immunoglobuline degli anticorpi primari dovrebbero essere tutti diversi e distinguibili dagli anticorpi secondari che il ricercatore intende utilizzare nel passaggio 1.12).
- Scaricare la permanente e il tampone bloccante e utilizzare una salvietta per rimuovere il tampone in eccesso senza toccare direttamente il tessuto. Aggiungere una quantità sufficiente di permanente e tampone di colorazione con anticorpi primari al tessuto per coprirlo e incubare il vetrino a 4 °C per 12-48 ore.
NOTA: Il ricercatore dovrà determinare il momento ottimale per l'incubazione degli anticorpi. - Preparare il tampone di blocco e il tampone di colorazione (Tabella 1).
- Aggiungere gli anticorpi secondari al tampone di colorazione preparato al punto 1.11 a diluizioni determinate sperimentalmente. Gli anticorpi secondari utilizzati devono avere come bersaglio le stesse specie o gli stessi isotipi di anticorpi utilizzati nella fase 1.9.
- Preparare il tampone di tempra (come nella Tabella 1).
NOTA: Questo passaggio aiuta a ridurre l'autofluorescenza dal tessuto. Se ciò non è necessario, i passaggi 1.13 e 1.14 possono essere saltati. - Lavare i fazzoletti come descritto nei passaggi 1.5-1.6. Quindi, aggiungere una quantità di tampone di tempra sufficiente a coprire i tessuti sui vetrini e incubare i vetrini a temperatura ambiente per 1-2 minuti.
- Lavare il fazzoletto come descritto nei passaggi 1.5-1.6. Quindi, aggiungere ai tessuti gli anticorpi secondari diluiti nel tampone di colorazione preparato al punto 1.12 in modo che i tessuti siano completamente ricoperti dal tampone di colorazione. Incubare i vetrini a 4 °C per 2-8 ore.
- Preparare la soluzione bloccante aggiungendo 50 μL di siero normale di qualsiasi specie di anticorpi che verrà utilizzata nella fase 1.18 (5% di ciascuna specie) al tampone bloccante preparato nella fase 1.11.
- Lavare i fazzoletti come descritto nei passaggi 1.5-1.6. Quindi, aggiungere la soluzione bloccante con il normale siero preparato al punto 1.16 ai tessuti in modo che siano completamente ricoperti da tampone colorante. Incubare i vetrini a temperatura ambiente per 1-8 ore.
- Preparare la soluzione colorante aggiungendo anticorpi primari coniugati con fluoroforo a diluizioni determinate sperimentalmente al tampone di colorazione preparato al punto 1.11. Inoltre, aggiungere una colorazione nucleare, come il giallo nucleare, alla soluzione colorante a una diluizione determinata sperimentalmente.
NOTA: I passaggi 1.18-1.19 sono necessari solo se il ricercatore deve utilizzare etichette di anticorpi aggiuntive che non richiedono anticorpi secondari per l'amplificazione del segnale. Se non si utilizzano anticorpi coniugati primari, le colorazioni dei nuclei possono essere aggiunte al passaggio 1.12. - Scuotere il tampone bloccante e utilizzare una salvietta per rimuovere il tampone in eccesso senza toccare direttamente il tessuto. Aggiungere al tessuto una quantità sufficiente di soluzione colorante preparata al punto 1.18 per coprirlo e incubare a 4 °C per 12-48 ore.
- Lavare i fazzoletti come descritto nei passaggi 1.5-1.6. Aggiungere 5 gocce di montante ad ogni vetrino senza introdurre bolle e rovesciare delicatamente e lentamente un vetrino coprioggetti sul fazzoletto per coprire completamente il tessuto di montante ed escludere tutte le bolle d'aria. Conservare i vetrini a 4 °C al buio fino a quando non sono pronti per essere ripresi.
2. Imaging delle sezioni di tessuto
NOTA: In questa fase, le sezioni di tessuto vengono colorate con tutti gli anticorpi di interesse per tutte le condizioni che si desidera visualizzare e i controlli monocolore vengono colorati con un parametro fluorescente su ciascuna sezione. L'imaging dovrebbe essere eseguito su un microscopio dotato di più rivelatori che possono essere sintonizzati per rilevare specifiche gamme di luce e, idealmente, dovrebbe avere accesso al maggior numero possibile di linee laser per l'eccitazione.
- Inserire uno dei vetrini completamente colorati nel microscopio e impostare il microscopio per visualizzare la sezione utilizzando l'obiettivo che si intende utilizzare per la raccolta dei dati.
- Sulla base dei fluorofori con cui viene colorato il tessuto, identificare le linee laser di eccitazione necessarie per stimolare tali fluorofori e identificare gli intervalli di emissione per ciascun fluoroforo che contengono la maggior quantità di segnale.
NOTA: Gli strumenti di visualizzazione degli spettri di fluorescenza online (ad esempio, vedere la Tabella dei materiali) possono essere utili per identificare le lunghezze d'onda di eccitazione ottimali dei fluorofori e identificare gli intervalli ottimali per il rilevamento dei fluorofori. I fluorofori utilizzati per la citometria a istoflusso devono essere scelti in modo tale che ogni fluoroforo differisca nella linea laser ottimale da cui il fluoroforo è stimolato o differisca per l'intervallo ottimale di lunghezze d'onda di emissione. I fluorofori che hanno lunghezze d'onda di eccitazione/emissione più vicine saranno più difficili da separare. I fluorofori utilizzati per questo studio sono dettagliati nella sezione dei risultati. - Aprire il Software 1 (vedere la Tabella dei materiali) in un microscopio confocale e selezionare la scheda di configurazione in alto. Quindi selezionare il pulsante hardware sul lato sinistro e modificare la profondità di bit a 16 bit.
NOTA: Si consiglia di utilizzare profondità di bit più elevate, come dati a 16 bit, per la raccolta dei dati, in quanto ciò migliorerà l'accuratezza della compensazione spettrale. Inoltre, densità di pixel più elevate, numeri maggiori per le medie delle linee e più passaggi nello z-stack miglioreranno la qualità della separazione spettrale. - Seleziona la scheda Acquisisci nella parte superiore dello schermo e inserisci l'impostazione del formato per includere la densità di pixel dell'immagine (in genere, risoluzioni più elevate producono risultati migliori).
- Se disponibile, selezionare l'acquisizione X bidirezionale per accelerare il tempo di imaging, a condizione che questa funzione sia stata adeguatamente calibrata.
- Scegliere un valore stenopeico per il microscopio confocale (in genere tra 400 e 600 nm) in cui valori più bassi produrranno in genere risultati di citometria a flusso istoforico di qualità superiore, anche se valori stenopeici più piccoli renderanno più difficile l'acquisizione delle immagini e potrebbero richiedere un tempo di imaging aggiuntivo o intensità di eccitazione più elevate per produrre immagini.
- Seleziona Mostra pannello di scansione sequenziale nella parte superiore del menu a sinistra per aprire l'opzione di scansione sequenziale. Utilizzare il segno '+' per impostare sequenze aggiuntive, in cui ogni sequenza conterrà una lunghezza d'onda di eccitazione decisa nel passaggio 2.2.
- Accendi tutti i laser necessari facendo clic sui pulsanti ON associati ai laser di interesse al centro dello schermo per trasformarli in uno stato ON. Entra in ogni sequenza specifica e aumenta la potenza del laser al centro dello schermo fino al valore desiderato, dove ogni sequenza avrà una lunghezza d'onda di eccitazione utilizzata.
- Accendi tutti i rilevatori necessari nella parte inferiore dello schermo in ogni sequenza per visualizzare tutti i fluorofori che verranno ripresi su quella linea laser. Regolare le gamme dei rilevatori facendo doppio clic sulle loro gamme e inserendo le gamme superiore e inferiore delle lunghezze d'onda che devono essere raccolte.
NOTA: Si raccomanda di utilizzare rilevatori ad alta sensibilità ove possibile e, idealmente, tutti i canali dovrebbero essere raccolti sugli stessi tipi di rilevatori, sebbene ciò non sia richiesto. - Premere il pulsante live, quindi concentrarsi sul campione. Utilizzare il pulsante Scala automatica a sinistra dell'immagine nel software per valutare se i canali sono adeguatamente saturi di segnale. L'utente deve regolare i valori di guadagno e le intensità laser in ogni sequenza fino a quando l'autoscaling non raggiunge il valore massimo (65535 per i dati a 16 bit) ma è molto vicino alla saturazione per tutti i canali.
NOTA: Poiché diversi fluorofori possono differire nella loro luminosità e diversi bersagli anticorpali possono differire nella loro abbondanza, la sensibilità dei rivelatori dovrà essere regolata in modo che sia di bassa sensibilità per i segnali luminosi e alta sensibilità per i segnali deboli se entrambi i fluorofori sono stimolati dallo stesso laser (l'intensità del laser non può cambiare in questo scenario poiché entrambi i fluorofori ne risentirebbero). Per ottenere i migliori risultati, il segnale di fluorescenza da ciascun canale nell'esperimento dovrebbe essere bilanciato in modo che ogni canale abbia quantità di segnale più o meno equivalenti, il che ridurrà il bleed-through spettrale in altri canali e si tradurrà in segnali più forti. Poiché campioni diversi possono differire nell'abbondanza dei marcatori di interesse da colorare, si raccomanda di testare le impostazioni del microscopio su più condizioni sperimentali per confermare che lo sbiancamento non si verifichi in nessuna delle condizioni sperimentali. - Una volta finalizzate le impostazioni del microscopio, caricare il microscopio con i vetrini di controllo del colore singolo e trovare la colorazione rappresentativa per ciascun fluoroforo. Imposta uno Z-stack mettendo a fuoco sotto il campione e premendo il pulsante di inizio nel menu z stack, quindi mettendo a fuoco sopra il campione e premendo il pulsante di fine .
- Scegli il numero di passaggi e la distanza tra i passaggi (e utilizza lo stesso numero di passaggi e parametri come la densità dei pixel e la profondità di bit che verranno utilizzati per visualizzare le diapositive completamente colorate), quindi premi il pulsante di avvio.
NOTA: Idealmente, queste immagini dovrebbero includere i segnali più luminosi provenienti da una colorazione reale che si possono trovare sul vetrino. - Dopo aver acquisito l'immagine dei singoli controlli del colore, procedere all'utilizzo delle stesse impostazioni del microscopio per visualizzare tutti i campioni completamente colorati.
3. Compensazione della fluorescenza
- Apri i file .lif nel Software 1 e fai clic sulla scheda del processo.
- Scegliere il modulo Separazione colorante , quindi scegliere l'opzione Separazione automatica del colorante . Selezionare il metodo Manuale per la separazione del colorante e scegliere di non riscalare.
- Aprire una delle immagini di controllo a colore singolo. Ispeziona manualmente i controlli dei singoli colori per verificare la presenza di smarginature spettrali da un canale all'altro osservando le finestre in scala di grigi per ciascun canale per verificare la presenza di un segnale in canali inappropriati. Utilizzare il pulsante Scala automatica se necessario per vedere una debole sbavatura.
- Iniziare a rimuovere il sanguinamento inserendo manualmente i numeri (in genere 0-1, dove 0 rappresenta l'assenza di sanguinamento e 1 rappresenta il 100% di sanguinamento) nella matrice sullo schermo, quindi premendo applica per verificare se il sanguinamento è stato rimosso adeguatamente. Questa operazione deve essere ripetuta fino a quando i segnali in tutti gli altri canali, ad eccezione del canale di interesse, sono stati ridotti ai livelli di fondo.
NOTA: La sovracompensazione porterà a risultati imprecisi, quindi la fluorescenza deve essere ridotta ai livelli di fondo ma non al di sotto di essi. I dati sovracompensati appariranno come un canale che ha un segnale e altri canali che sembrano avere buchi neri dove si trova il segnale di interesse (Figura 2). - Una volta completato uno dei controlli a colore singolo, registrare i valori nella matrice, quindi reimpostare la matrice e passare al controllo a colore singolo successivo. Ripetere questo processo per ogni singolo controllo colore.
NOTA: Il primo colorante si troverà nella riga del colorante 1 e la quantità di fluorescenza che deve essere rimossa dal colorante 1 si troverà nelle colonne adiacenti. - Assemblare tutti i valori acquisiti da ciascun controllo in un'unica matrice e applicare questa matrice a un campione completamente colorato. Se la separazione sembra accurata, applica le impostazioni a tutte le immagini dell'esperimento, altrimenti rivaluta i singoli controlli del colore.
NOTA: Una compensazione efficace dovrebbe rimuovere la maggior parte del sanguinamento attraverso i canali, come riassunto e mostrato nella Figura 3. - Salva tutti i file compensati in un file .lif separato.
4. Identificazione di nuclei e cellule utilizzando ilastik
- Apri i file .lif compensati in FIJI20. Abilita la visualizzazione delle pile in un hyperstack e seleziona un'immagine.
- (Facoltativo) Utilizzare uno strumento di selezione, ad esempio lo strumento rettangolo, per includere le parti dell'immagine che verranno analizzate ed escludere le aree che non verranno analizzate. Ritagliare l'immagine utilizzando la funzione di ritaglio > immagine.
NOTA: La riduzione delle dimensioni dell'immagine è utile nei passaggi successivi per ridurre il consumo di memoria ad accesso casuale (RAM) e aumentare la velocità di elaborazione. - Salva l'immagine come file .tif. Inoltre, salva l'immagine come file hdf5 utilizzando il plug-in ilastik per FIJI facendo clic su Plug-in > ilastik > Esporta hdf5. Salva il file come "nome a scelta".h5 con compressione 0.
- Apri Ilastik21 e crea un progetto di classificazione dei pixel. Utilizza la funzione Aggiungi nuovo > Aggiungi immagini separate per aggiungere almeno 3 immagini rappresentative andando ad aggiungere nuovi dati di input.
- Fare clic con il pulsante destro del mouse su una delle immagini, selezionare Modifica proprietà e modificare la modalità di visualizzazione in scala di grigi.
- A sinistra dello schermo, seleziona la scheda di selezione delle funzionalità. Fare clic su Seleziona funzioni e selezionare tutte le funzioni fino al valore sigma più alto che il programma ti consentirà di utilizzare, quindi premere OK.
- Ora seleziona la scheda Allenamento. Utilizzare lo strumento pennello (mentre è selezionata l'etichetta uno ) per evidenziare le singole cellule (Figura 4) di interesse in modo che l'interno (citoplasma e nucleo) e la membrana cellulare siano inclusi nell'evidenziazione.
- Ripetere l'operazione su tutti i campioni di addestramento in cui vengono selezionati tutti i tipi di cellule di interesse e da ciascun campione devono essere selezionate più cellule di diversa intensità, morfologia e localizzazione nel tessuto.
NOTA: A seconda dei marcatori scelti per una singola serie di colorazioni, possono esserci o meno colorazioni intracellulari o colorazioni della membrana cellulare per le cellule di interesse. Qualunque sia il marcatore utilizzato, assicurarsi che l'evidenziazione includa la colorazione più esterna sulle cellule di interesse e includa l'interno della cellula indipendentemente dal fatto che venga utilizzata una colorazione intracellulare. - Quindi, usa l'etichetta due per evidenziare tutto ciò che non è le celle di interesse (Figura 4). Ciò può includere spazio vuoto, nuclei non marcati o tessuto che non ha marcatori di interesse. Assicurarsi di includere lo spazio immediatamente adiacente alle celle di interesse per promuovere l'apprendimento dei confini esatti delle celle.
- Utilizza la funzione Live Update per valutare la qualità della formazione sulle immagini di interesse. Se la formazione è inadeguata, continuare la formazione nei passaggi da 4.7 a 4.9. In caso contrario, se il risultato è soddisfacente, fare clic sulla scheda Esportazione previsione .
- Imposta la fonte in modo che sia probabilità. Fare clic sul pulsante Scegli impostazioni immagine di esportazione , quindi impostare il parametro c su Esporta 0-1 (supponendo che l'etichetta 1 evidenzi i parametri di interesse).
- Converti il tipo di dati in un numero intero a 16 bit ed esporta come file hdf5 nella directory di interesse con il nome di interesse (si consiglia di utilizzare {nickname} mask.h5), quindi premi OK.
- Fare clic sulla scheda Elaborazione batch . Premere il pulsante Seleziona file di dati grezzi , selezionare tutti i file .h5 di interesse che richiedono l'elaborazione, quindi premere Apri. Fare clic su Elabora tutti i file.
- Ripetere i passaggi da 4.4 a 4.13, tranne per il fatto che invece di identificare le cellule, ora identifica i nuclei ed esclude tutto il resto (Figura 4). Esportalo come previsione separata per identificare i nuclei cellulari.
- Apri Ilastik e crea un nuovo progetto di conversione dati. Aggiungere un file hdf5 rappresentativo nella scheda Dati di input utilizzando la funzione Aggiungi nuovo > Aggiungi immagine separata .
- Fare clic sulla scheda Esportazione dati e scegliere il pulsante Scegli impostazioni immagine di esportazione con la sorgente impostata su input. Cambia il formato in tiff multipagina, scegli una posizione di salvataggio con la funzione di selezione e imposta il nome del file su {nickname}. Premere OK.
- Fare clic sulla scheda Elaborazione batch e selezionare tutti i file hdf5 utilizzando il pulsante Seleziona file di dati di input e premere il pulsante Elabora tutti i file .
- Aprire Software 2 (vedere Tabella dei materiali) e aggiungere tutti i file tif/tiff generati nei passaggi 4.3 e 4.17 utilizzando il pulsante Aggiungi file . Scegli una posizione per gli output dei file e premi il pulsante Avvia tutto per convertire tutti i file in file .ims. I file sono ora pronti per l'analisi nel Software 3 (vedere la Tabella dei materiali).
5. Analisi nel software 3
- Apri una delle immagini contenenti tutte le macchie fluorescenti. Seleziona Modifica-> Aggiungi canali e aggiungi i file .ims corrispondenti alle maschere realizzate su nuclei e corpi cellulari. Ripetere l'operazione per tutti i campioni.
- Seleziona il pulsante Aggiungi nuove celle . Selezionare l'opzione Rileva nucleo e cella e procedere in avanti.
- Selezionare la maschera sui nuclei come canale sorgente per il rilevamento dei nuclei e selezionare l'opzione avanzata per dividere i nuclei in base ai punti di partenza. Scegliere un valore per il diametro del nucleo) e procedere in avanti.
NOTA: Il valore ottimale per il diametro del nucleo dovrà essere testato eseguendo la simulazione e valutando manualmente se il programma non sta dividendo abbastanza nuclei (più nuclei sono raggruppati insieme) o se c'è una divisione eccessiva (un nucleo è diviso in più di uno). - Scegliere un valore per la valutazione della qualità, quindi procedere. In genere, è meglio includere tutti i nuclei, poiché l'omissione di nuclei può portare alla fusione di più cellule in una.
- Scegliere un valore di soglia per la creazione di superfici nucleari e quindi procedere. Assicurarsi che tutti i nuclei, anche quelli più deboli, siano ancora inclusi nella soglia. Non abbassare troppo la soglia, in quanto ciò potrebbe sovrastimare le dimensioni dei nuclei o potenzialmente includere detriti come colorazione nucleare.
- Selezionare un valore per il numero minimo di Voxel per nucleo e procedere. In genere, è meglio conservare tutto, a meno che non si possano escludere detriti o non nuclei utilizzando questo parametro.
- Scegli l'opzione Confine cellulare e citoplasma , seleziona la maschera sulle cellule come canale di origine per rilevare i corpi cellulari, quindi procedi.
- Scegliere una soglia di cella che acquisisca con precisione tutte le celle di interesse, ma non sia così bassa da includere lo sfondo o qualsiasi rilevamento non specifico delle celle. Selezionare l'opzione per dividere le cellule toccanti in modo tale che ci sia un nucleo per cellula e siano separate dalla distanza dai nuclei. Inoltre, scegli di espandere le cellule sui nuclei e poi procedi.
- Seleziona un valore per il numero minimo di voxel per cella e procedi. In genere, è meglio conservare tutto, a meno che non sia possibile escludere detriti o non celle utilizzando questo parametro.
- Valuta se le superfici create sulle celle sono accurate osservando se più celle sono state fuse insieme o se mancano celle. Se le superfici sono inadeguate, tornare al passaggio 5.2 e provare nuovi parametri fino a determinare le impostazioni ottimali.
- Una volta stabilite le impostazioni migliori, fare clic sull'oggetto celle create nel menu a sinistra, fare clic sulla scheda di creazione e fare clic sul pulsante Memorizza parametri per batch per salvare le impostazioni utilizzate.
- Apri più finestre del Software 3 e apri immagini separate in ciascuna finestra. Premi il pulsante nuove celle in ogni finestra, scegli le impostazioni salvate nel menu Parametri di creazione preferiti e chiedi a ciascuna finestra di funzionare fino al completamento. Ripetere l'operazione tutte le volte necessarie fino a quando tutti i campioni non sono stati analizzati.
NOTA: l'esecuzione di più istanze del software 3 renderà l'analisi più veloce, ma il numero di finestre che possono essere eseguite in parallelo dipenderà dalla velocità di elaborazione dell'unità di elaborazione centrale (CPU) e dalla quantità di RAM su cui viene eseguita l'analisi. - Una volta completati tutti i campioni, fare clic sull'oggetto celle create nel menu a sinistra di ogni immagine e fare clic sulla scheda Statistiche. Fare clic sul pulsante Configura elenco di valori statistici visibili, deselezionare tutte le statistiche tranne il valore 'Media intensità cella', quindi premere OK.
- Per ogni immagine che deve essere analizzata, fare clic sul pulsante Esporta tutte le statistiche su file nella scheda delle statistiche e salvare i file in una posizione.
6. Analisi citometria a istoflusso e mappatura delle popolazioni su sezioni di tessuto
- Apri Anaconda e JupyterLab all'interno di Anaconda. Quindi, apri lo script fornito in File di codifica supplementare 1. Esegui il codice utilizzando il menu Esegui e seleziona Esegui tutte le celle oppure esegui le celle singolarmente nell'ordine selezionando Esegui celle selezionate.
- Quando viene richiesto di inserire il file di origine, inserire la directory del file per i valori fluorescenti esportati generati nel passaggio 5.14. La directory del file può essere ottenuta osservando le proprietà di uno dei file di .csv esportati e copiando il percorso del file nel prompt.
- Successivamente, il programma chiederà una posizione di output per i file elaborati. Immettere la directory dei file per qualsiasi posizione adatta per il salvataggio dei file elaborati. Il programma rileverà automaticamente il numero di canali fluorescenti contenuti nella cartella.
- Eseguire la sezione successiva del codice per annotare i dati con i nomi di ogni canale. Questo codice può essere modificato in base alle esigenze per modificare il parametro "numberOfChannels" in modo che corrisponda al numero di canali rilevati nel passaggio 6.3. Inoltre, i nomi dei canali possono essere modificati per tutti i canali necessari ripetendo la linea 'ChX':'Marker' tutte le volte che è necessario e modificando i nomi dei marcatori.
- L'esecuzione della riga di codice successiva creerà un file di .csv nella posizione specificata che viene annotato e contiene tutti i parametri fluorescenti nel set di dati. Ripetere i passaggi 6.1-6.5 per ogni set di esportazioni del software 3 per creare file .csv per tutti i dati.
- Converti i file .csv in file .fcs aprendo il Software 4 (vedi Tabella dei materiali) e trascinando i file .csv nella finestra del programma. Il software 4 inizierà automaticamente a convertire i file in file .fcs.
- Fare doppio clic su uno degli esempi nel Software 4 per aprire una finestra su quell'esempio. Utilizza gli strumenti di gating nel Software 4 per creare una strategia di gating. Si consiglia di iniziare con il gating su un marcatore che definisce il lignaggio contenente tutte le cellule di interesse, quindi procedere con i gate che rimuovono prima i sottoinsiemi più rari e quindi terminare con i tipi di cellule più abbondanti.
- Una volta stabilita una strategia di gating, verifica l'accuratezza del gating facendo clic con il pulsante destro del mouse su quella popolazione nel menu e scegliendo Esporta. Esporta tutti i parametri come file .csv con intestazioni incluse.
- Apri JupyterLab in Anaconda. Quindi, apri lo script fornito nel file di codifica supplementare 2 ed esegui il codice. Quando viene richiesto di inserire la posizione del file Software 4, immettere la directory del file per il file .csv esportato generato nel passaggio precedente. La directory del file può essere ottenuta esaminando le proprietà del file di .csv esportato e copiando il percorso del file nel prompt.
- Quando viene richiesto di aggiungere la posizione di output, scegliere una directory di file a scelta e assicurarsi che la directory di file includa il nome del file alla fine ('directory di file'\filename.txt). Eseguire il resto del codice.
- Copiare il testo nel file .txt e aprire il file .ims corrispondente nel software 3 per lo stesso campione su cui si basano i dati esportati. Fai clic sull'oggetto Cella nel file, passa alla scheda delle statistiche, incolla il testo nella barra di ricerca e avvia la ricerca. Verranno evidenziate tutte le celle di interesse.
- Ispeziona manualmente se tutte le celle di interesse sono state acquisite utilizzando la strategia di gating. Se il numero di celle è sottostimato o sovrastimato (esempi mostrati nella Figura 5), tornare al passaggio 6.7, regolare il cancello e rivalutare se le celle vengono acquisite con precisione con il nuovo cancello. Ripetere l'operazione con tutti i gate nella strategia di gating e valutare il gating su più campioni.
- Quando tutte le porte sono state convalidate, esportare i dati dal software 4 utilizzando la funzione di editor di tabelle e analizzare i dati utilizzando il metodo di analisi preferito.
NOTA: Le popolazioni cellulari possono essere mappate nuovamente sulle sezioni di tessuto nel Software 3 identificando la popolazione gated come nei passaggi 6.8-6.11 e quindi creando una superficie basata sulle cellule selezionate. Questo può essere fatto per ogni popolazione recintata, come mostrato nella Figura 6 e nella Figura 7.
Risultati
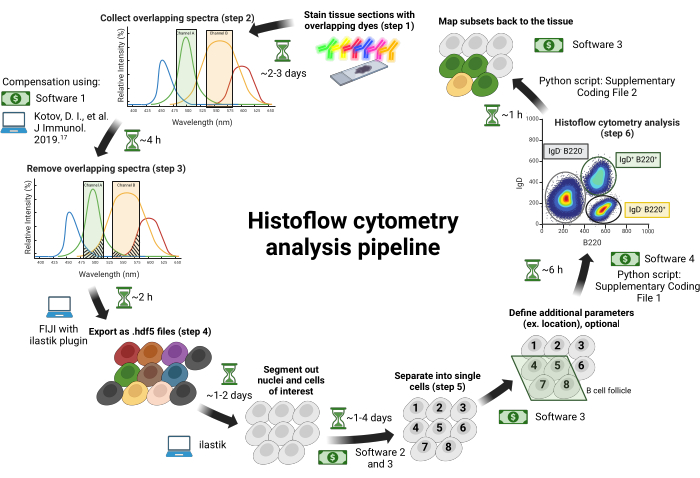
Figura 1: Flusso di lavoro della citometria a istoflusso. Le sezioni di tessuto vengono colorate con coloranti spettralmente sovrapposti (fase 1). Le immagini vengono raccolte attraverso singoli laser di eccitazione abbinati a filtri passa-banda regolabili per ridurre al minimo il bleed-through spettrale tra i fluorofori (passaggio 2). Il sanguiname...
Discussione
Qui viene descritto l'uso della citometria a istoflusso, una tecnica che è stata convalidata in precedenza18. È dimostrato che quando si colorano sezioni di tessuto con coloranti spettralmente sovrapposti, il sanguinamento attraverso i canali può essere rimosso utilizzando la compensazione spettrale, con conseguente risoluzione chiara di un numero maggiore di parametri fluorescenti rispetto a quanto sarebbe normalmente possibile con i metodi convenzionali. Poiché le immagini istologiche ad alt...
Divulgazioni
Gli autori non hanno conflitti di interesse finanziari.
Riconoscimenti
Ringraziamo l'Hotchkiss Brain Institute Advanced Microscopy Platform per l'infrastruttura di imaging e l'esperienza. RWJ è stato supportato da borse di studio post-dottorato del programma Eyes High dell'Università di Calgary e da una Multiple Sclerosis Society of Canada e da una borsa di studio educativa illimitata Roche Canada. VWY ha ricevuto un sostegno salariale dal programma Canada Research Chair Tier 1. Questo lavoro è stato sostenuto da fondi operativi del Canadian Institutes of Health Research Grant 1049959, della Multiple Sclerosis Society of Canada Grant 3236 e del Dipartimento della Difesa degli Stati Uniti del Programma di ricerca sulla sclerosi multipla diretto dal Congresso. La Figura 1 viene creata con BioRender.com. Le cifre adattate in questa pubblicazione sono state originariamente pubblicate su The Journal of Immunology. Rajiv W. Jain, David A. Elliott e V. Wee Yong. 2023. Analisi di singole cellule di immagini istologiche ad alto parametro mediante citometria a istoflusso. J. Immunol. 210: 2038-2049. Diritto d'autore © [2023]. L'Associazione Americana degli Immunologi, Inc.
Materiali
| Name | Company | Catalog Number | Comments |
| 100% Ethanol | Sigma | 676829-1L | |
| 4% PFA | Electron Microscopy Sciences | 157-4 | |
| Anaconda | N/A | N/A | https://www.anaconda.com/download |
| Bovine Serum Albumin | Sigma | A4503-50G | |
| Cold fish stain gelatin | Sigma | G7765 | |
| Collating multichannel data from Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Convert FlowJo output to txt file for Cell selection in Imaris.ipynb script | N/A | N/A | https://github.com/elliottcalgary/Histoflow-Cytometry-Analysis- |
| Donkey anti-rat Alexa Fluor 647 | JacksonImmunoResearch | 712-605-153 | 1:300 concentration |
| Donkey anti-rat DyLight 405 | Jackson ImmunoResearch | 712-475-153 | 1:200 concentration |
| Donkey Serum | JacksonImmunoResearch | 017-000-001 | |
| F(ab')2-Goat anti-Mouse IgG PerCP-eFluor 710 | Thermofisher | 46-4010-82 | 1:25 concentration |
| FIJI | N/A | N/A | https://imagej.net/software/fiji/ |
| FlowJo | FlowJo LLC | Software 4 | |
| Fluorescence spectraviewer | https://www.thermofisher.com/order/fluorescence-spectraviewer/#!/ | ||
| Fluoromount-G | Southern Biotech | 0100-01 | |
| Fresh frozen human tonsil sections | amsbio | HF-707 | |
| Glass coverslip | VWR | 48393 106 | |
| Goat anti-human IgA Alexa Fluor 488 | JacksonImmunoResearch | 109-546-011 | 1:400 concentration |
| Goat anti-human IgG Cy3 | JacksonImmunoResearch | 709-166-098 | 1:400 concentration |
| Goat anti-human IgM Dylight 405 | JacksonImmunoResearch | 109-476-129 | 1:300 concentration |
| Goat anti-rabbit A546 | Thermo Fisher Scientific | A-11035 | 1:250 concentration |
| Goat anti-rabbit IgG PE-Alexa Fluor 610 | Thermofisher | A-20981 | 1:250 concentration |
| Horse Serum | Sigma | H1138 | |
| Ilastik | N/A | N/A | https://www.ilastik.org/ |
| Ilastik FIJI plugin | N/A | N/A | https://www.ilastik.org/documentation/fiji_export/plugin |
| Imaris File Converter | Oxford Instruments | Software 2 | |
| Imaris with cell module | Oxford Instruments | Software 3 | |
| kimwipe | Kimtech | 34155 | |
| LasX Life Science software | Leica | Software 1 | |
| Mouse anti-human CD20 | VWR | CA95024-322 | 1:40 concentration |
| Mouse anti-human CD38 APC-R700 | BD Biosciences | 564980 | 1:20 concentration |
| Normal Goat Serum | JacksonImmunoResearch | 005-000-001 | |
| Normal Mouse Serum | JacksonImmunoResearch | 015-000-001 | |
| Normal Rabbit Serum | JacksonImmunoResearch | 011-000-001 | |
| Normal Rat Serum | JacksonImmunoResearch | 012-000-120 | |
| Nuclear Yellow | Abcam | ab138903 | Dissolve in DMSO at a concentration of 2 mg/ml and store at 4°C in the dark |
| PAP pen | Cedarlane | MU22 | |
| PBS | Gibco | 10010-023 | |
| Rabbit anti-human Ki67 | Abcam | ab15580 | 1:500 concentration |
| Rabbit anti-mouse Iba1 | Wako | 019-19741 | 1:500 concentration |
| Rat anti-human Blimp1 | Thermofisher | 14-5963-82 | 1:40 concentration |
| Rat anti-mouse B220 Alexa Fluor 647 | BioLegend | 103226 | 1:250 concentration |
| Rat anti-mouse CD138 | Biolegend | 142502 | 1:200 concentration |
| Rat anti-mouse CD3 PE-eFluor 610 | Thermo Fisher Scientific | 61-0032-82 | 1:40 concentration |
| Rat anti-mouse CD4 Alexa Fluor 488 | BioLegend | 100529 | 1:200 concentration |
| Rat anti-mouse CD45 allophycocyanin-R700 | BD Biosciences | 565478 | 1:50 concentration |
| Rat anti-mouse IgD PerCP-eFluor 710 | Thermo Fisher Scientific | 46-5993-82 | 1:50 concentration |
| SP8 Confocal microscope | Leica | ||
| Triton X-100 | Sigma | X100-500ml | |
| Trueblack | Biotium | 23007 | |
| Tween-20 | Sigma | P7949-500ml | |
| Ultracomp ebeads | Thermofisher | 01-2222-42 |
Riferimenti
- Bar-Or, A., Li, R. Cellular immunology of relapsing multiple sclerosis: interactions, checks, and balances. Lancet Neurol. 20 (6), 470-483 (2021).
- Borst, K., Dumas, A. A., Prinz, M. Microglia: Immune and non-immune functions. Immunity. 54 (10), 2194-2208 (2021).
- Han, R. T., Kim, R. D., Molofsky, A. V., Liddelow, S. A. Astrocyte-immune cell interactions in physiology and pathology. Immunity. 54 (2), 211-224 (2021).
- Absinta, M., et al. A lymphocyte-microglia-astrocyte axis in chronic active multiple sclerosis. Nature. 597 (7878), 709-714 (2021).
- Piwecka, M., Rajewsky, N., Rybak-Wolf, A. Single-cell and spatial transcriptomics: Deciphering brain complexity in health and disease. Nat Rev Neurol. 19 (6), 346-362 (2023).
- Schirmer, L., et al. Neuronal vulnerability and multilineage diversity in multiple sclerosis. Nature. 573 (7772), 75-82 (2019).
- Jain, R. W., Yong, V. W. B cells in central nervous system disease: Diversity, locations and pathophysiology. Nat Rev Immunol. 22 (8), 513-524 (2022).
- Lassmann, H. Multiple sclerosis pathology. Cold Spring Harb Perspect Med. 8 (3), (2018).
- Yong, V. W. Microglia in multiple sclerosis: Protectors turn destroyers. Neuron. 110 (21), 3534-3548 (2022).
- Sharma, S., Boyer, J., Teyton, L. A practitioner's view of spectral flow cytometry. Nat Methods. 21 (5), 740-743 (2024).
- Longo, S. K., Guo, M. G., Ji, A. L., Khavari, P. A. Integrating single-cell and spatial transcriptomics to elucidate intercellular tissue dynamics. Nat Rev Genet. 22 (10), 627-644 (2021).
- Ramaglia, V., et al. Multiplexed imaging of immune cells in staged multiple sclerosis lesions by mass cytometry. Elife. 8, e48051 (2019).
- Bolognesi, M. M., et al. Multiplex staining by sequential immunostaining and antibody removal on routine tissue sections. J Histochem Cytochem. 65 (8), 431-444 (2017).
- Radtke, A. J., et al. IBEX: An iterative immunolabeling and chemical bleaching method for high-content imaging of diverse tissues. Nat Protoc. 17 (2), 378-401 (2022).
- Radtke, A. J., et al. IBEX: A versatile multiplex optical imaging approach for deep phenotyping and spatial analysis of cells in complex tissues. Proc Natl Acad Sci U S A. 117 (52), 33455-33465 (2020).
- Gerner, M. Y., Kastenmuller, W., Ifrim, I., Kabat, J., Germain, R. N. Histo-cytometry: A method for highly multiplex quantitative tissue imaging analysis applied to dendritic cell subset microanatomy in lymph nodes. Immunity. 37 (2), 364-376 (2012).
- Kotov, D. I., Pengo, T., Mitchell, J. S., Gastinger, M. J., Jenkins, M. K. Chrysalis: A new method for high-throughput histo-cytometry analysis of images and movies. J Immunol. 202 (1), 300-308 (2019).
- Jain, R. W., Elliott, D. A., Yong, V. W. Single-cell analysis of high-parameter histology images using histoflow cytometry. J Immunol. 210 (12), 2038-2049 (2023).
- JoVE Science Education Database. General Laboratory Techniques. Histological Sample Preparation for Light Microscopy. JoVE. , (2023).
- Schindelin, J., et al. Fiji: An open-source platform for biological-image analysis. Nat Methods. 9 (7), 676-682 (2012).
- Berg, S., et al. ilastik: Interactive machine learning for (bio)image analysis. Nat Methods. 16 (12), 1226-1232 (2019).
- Reichard, A., Asosingh, K. Best practices for preparing a single cell suspension from solid tissues for flow cytometry. Cytometry A. 95 (2), 219-226 (2019).
- Ruhlandt, D., et al. Absolute quantum yield measurements of fluorescent proteins using a plasmonic nanocavity. Commun Biol. 3 (1), 627 (2020).
- Combs, C. A., Shroff, H. Fluorescence microscopy: A concise guide to current imaging methods. Curr Protoc Neurosci. 79, 1-25 (2017).
- Elliott, A. D. Confocal microscopy: Principles and modern practices. Curr Protoc Cytom. 92 (1), e68 (2020).
- Jonkman, J., Brown, C. M., Wright, G. D., Anderson, K. I., North, A. J. Tutorial: Guidance for quantitative confocal microscopy. Nat Protoc. 15 (5), 1585-1611 (2020).
- McNally, J. G., Karpova, T., Cooper, J., Conchello, J. A. Three-dimensional imaging by deconvolution microscopy. Methods. 19 (3), 373-385 (1999).
- Stringer, C., Wang, T., Michaelos, M., Pachitariu, M. Cellpose: A generalist algorithm for cellular segmentation. Nat Methods. 18 (1), 100-106 (2021).
Ristampe e Autorizzazioni
Richiedi autorizzazione per utilizzare il testo o le figure di questo articolo JoVE
Richiedi AutorizzazioneThis article has been published
Video Coming Soon