このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
繊維状タンパク質をストレッチし、測定する新しい重力分光器の使用を実証
要約
これは、目的、操作、そして斬新な重力の分光計からの代表的な結果を示すステップバイステップガイドです。
要約
巨大分子構造の研究は、分子機構と機能の解明に重要になっています。タンパク質の構造的特徴の力依存性をテストできるいくつかの限られた、しかし重要なbioinstrumentsがあります。規模は、研究者がそのような核酸、酵素、および生命維持の作業を実行するモータータンパク質などの分子のナノの世界を覗きできるか正確に上に制限するパラメータとなっています。原子間力顕微鏡(AFM)はよく電子顕微鏡と同等の距離分解能で繊維状のタンパク質のネイティブ構造を決定するために調整されています。しかし、AFMの力の研究で、軍は通常、単一の分子よりもはるかに高くなっています1、2を発生する可能性があります。光学トラップ(OT)がトラップされたビーズとの間の相対距離を判断するに非常に優れていると、彼らは非常に小さな力3を付与することができる。しかし、彼らが検討されて分子の正確な絶対的な長さを得ることはありません。分子シミュレーションは、実験に協力的な情報を提供しますが、同じ大規模な分子サイズ、長いタイムフレームを、処理し、他の証拠2、4の存在下でいくつかの研究者を説得する能力に制限されています。
重力分光計(GFS)は、能力のユニークな組み合わせを提供することにより、研究者の武器庫での重要なニッチを埋める。この楽器は、femtonewtonの範囲からnanonewtonの範囲に98%以上の精度で一般的に力を生成することができます。距離の測定は、現在、光トラップと同様の精度で5ナノメートル、そして相対的なビーズペアの分離距離に絶対的な分子の長さを解決する能力があります。また、GFSは、ストレッチや力が平衡に近い場所uncoilingを決定する、または任意の測定の構造変化に対して並置する段階的な力を提供することができます。それは生理的な力の負荷2でイベントをuncoilingに関与しているか、多くのアミノ酸残基を決定することも可能です。あらゆるアッセイを先行しなければならない大きな力のキャリブレーションが存在する他の方法とは異なり、GFSはそのような力のキャリブレーション5を必要としません。他の方法の強みを補完することで、GFSは重要な蛋白質と他の高分子のナノメカニクスを理解する上でのギャップをブリッジします。
プロトコル
新規GFSの構成の概要
定期的な光学顕微鏡、赤道儀、カメラ、およびコンピュータ[図1]:GFSは、いくつかの不可欠なコンポーネントで構成されています。サンプルを保持する密閉型フローセルのチャンバーは、GFSのデザインに応じて不可欠である。範囲は宇宙の別の方向に回転させることができるので、光学顕微鏡は、赤道儀に装着される。この能力は、重力の静的なベクトルは重力の力が試料にpiconewtonレンジの力の負荷を与えることができるので、サンプルはベクトルに関連して動的に配向させることができるように悪用されることができます。それはサンプルの向きの変化を記録できるようにカメラは、光学顕微鏡の接眼レンズに置き換えられます。この生データをデジタル化し、実際の力と距離の測定にデータを解釈するコンピュータによって操作されます。密封されたフローチャンバーは、試料を失うことなく空間中の全自由度をできるように設計されています。チャンバー内チャンバーの表面に接着され、"アンカー"ビーズに一方の末端近くにつながれているサンプルの分子が存在する。反対側の末端は、チャンバーの表面から自由である"モバイル"ビーズにつながれている。それは、それによってこのような低荷重負荷時のテザー分子を伸ばし重力によって処理することができるアッセイバッファー内の空きこのモバイルビーズである[図2]。それは、両方のビーズは、フローチャンバーの表面の上に座っているペアから分子への付着によって結合良好な使用可能なペアを識別するためにいくつかの経験を取るいませんが、サンプルは、単純に、顕微鏡下でマイクロスフェアのペアのように見えます。システムに対する1つの変更は、GFSを保持し、バネによって中断されているフローティングプラットフォームの追加です。この構成では、一度サンプルでは重力がサンプルに作用することができる位置に回転している、プラットフォームとそのすべてのコンポーネントは、バネ定数に対してドロップすることができます。自由落下の近くに、モバイルビーズに作用する力はゼロに近く、温泉"最大拡張時に、重力が2倍と同じくらいで乗算されます。このように、段階的な力/距離応答は別の力の負荷での単一分子の挙動を測定するためにグラフ化することができます。
1。ミクロスフェアの調製
- 0.04パーセント2分間3 - アミノプロピルトリエトキシシラン(アセトンでカット)のガラス又はシリカビーズ10mgを約水没。
- 蒸留水の二つの変更ですすぎ、5分間、2,000 xgでペレットに遠心。上清を捨てる。
- カップリングバッファー(0.01 Mピリジンは再蒸留水で切断してpHを6.0に調整)5mLを追加し、精力的にこの混合物を振る。上記のよう心する。このステップを3回繰り返します。
- ビーズのウェットケーキに、5%グルタルアルデヒド溶液(カップリングバッファーとグルタルアルデヒドをカット)2 mLを加える。精力的に振る。
- フードの下で、室温で3時間ビーズ/グルタルアルデヒドの混合物を回転させる。
- 上記のように遠心し上清を吸引除去する。
- 精力的にそれらを揺することによってカップリング緩衝液5mLにビーズを洗浄し、上清を遠心分離し、吸引除去する。このあと3回繰り返します。
- ビーズに所望の抗体の約15μLを追加し、積極的に振る。ビーズは、16〜24時間のために回転する必要があります。
- ボンネットの下に、1 Mグリシンクエンチング液(蒸留水でグリシンをカットし、pHを7.0に調整)5 mLを加える。精力的混合物を振とうし、30分間回転させます。
- 遠心し上清を吸引除去する。
- 洗浄バッファー(; 0.1%アジ化ナトリウム、0.1%BSA、0.15 M塩化ナトリウム、及び0.001 M EDTA、0.01Mトリス、pH 7.0)5 mLを追加します。精力的にこれを振って、遠心し上清を吸引除去する。このステップをさらに3回を繰り返します。
- 低塩緩衝液(0.02 Mイミダゾール、5 mMのMgCl 2、pH7.0に調整し、0.1MのKCl)にバッファを変更します。 3回繰り返します。
2。ミクロスフェアへのサンプルの添付ファイル
- 準備ビーズを少量取り、それらを追加する(バッチ処理の間に直径の大きな食い違いがある場合は、しかし、ビーズの各ケーキから約2μLを、それは小さなビーズの大の8:1比率の周りに使用することが有利である)アッセイバッファー付きマイクロ遠心チューブ。アッセイバッファーを使用して、約5μMにあなたの蛋白質の濃度を減少させる。バッファー、蛋白質、そしてビーズを含む400μLの少なくとも合計ボリュームを準備します。
- 3時間約1回転でこの混合物を回転させる(蛋白質は、それが集約し、無用になると、あまりにも興奮している場合)。
3。スライドのチャンバーの準備
- コート0.01%ニトロセルロース(酢酸アミルで)と厚い顕微鏡用スライド。約10分間このスライドは、乾かします。
- チャンバーとなるように鋭いガラスカッターで、カバースライドをカット。これは、ガラスの4つのストリップを必要とします。
- 事実を使用してくださいエッジの両側に真空グリースの塗抹標本でガラスと熊手それのORYエッジ。
- 乾燥ニトロセルロース上にプレスグリースコーティングストリップはスライドの表面上にボックスを作成するスライドをカバー。
- ピペットボックスの内側にガラスの表面にピペッターをタップしてビーズ/タンパク質混合物の約2μL。
- チャンバーの大きさに応じて、低塩緩衝液の20から400まで約μLを加える。
- すでに密封と、バッファ室を完了するために真空グリースでコーティングされているボックスの上にカバースリップを押す。
- ビーズはニトロセルロースに自分自身を固定するために十分な時間があるので、スライドのレベルの場所に座ってみましょう。
4。 GFSデータ収集
- GFSステージ上にマウントスライド
- GFSのカメラは、小さなマイクロスフェアが大きい微小球の赤道付近に添付されている正当な"ビーズのペア、"のための記録と検索が何であるか監視する。
- 潜在的なビーズのペアが検出されると、"モバイル"ビーズはスライドの表面上に載っていないかどうかを判断するために焦点の深さを通過。
- 一度、適切なペアが識別され、メモ角度モバイルビーズは、D MAXから離れていること(D MAX =結合微小球の重心間の最大距離が)。ビデオを取得する位置に範囲を移動します。
- GFSの旅行の角度は、分子の長さに応じて25〜90度のために呼び出す可能性のあるD 分、D MAX、およびdの分を記録するために十分なはずです。
- ペアとしてRECORDは、d minからD MAXにして戻ってD minに移動。
- それは分析のために後で引くことができるようにも背景の動画を撮影することをお勧めします。
- GFSのドロップを実行した場合、D MAXに戻って範囲を移動し、毎秒少なくとも60フレームのドロップを記録。重要な部分は、最初の発振だが、長い録音時間は、動的な試験にも使用することができます。
5。 GFSデータ解析
- デジタル"閾値"のイメージに生の映像を変換し、ビデオの各フレームにおける各ビーズの重心位置を決定するためのImageJのマクロを実行します。これはドロップのビデオにもなります。
- ExcelにImageJのからX、Yおよび地域のデータをダンプしてポイントをプロットする。
- 適切なビーズペアがビデオに買収された場合、グラフに顕著なこぶは、(d minは)彼らの最も近いにいるビーズを示していると、グラフの頂点にd maxの位置です。
- このデータを使用して、各ビーズの半径は、ImageJので正確に決定されるべきであるとすべてのこの情報は、ネストされた式に配置されます。
D = [(G罪α)2 +(G COSα+ D MAX - G)2] 0.5
G = [d minは2 + R B 2 - (R A + R B)2] 0.5 = R B罪をβ
C = D MAX - R - G
重心間(D =距離、重力のG =力、平行対物レンズの度数で、α=角度、軸から添付ファイルのβ=角度、モバイルビーズのR B =半径、固定されたビーズのR =半径固定されたビーズの赤道。 - 溶剤の浮力が減算された後も、そのボリュームをもたらすモバイルビーズのフィット半径を使用して、そしてビーズの密度を与え、モバイルビーズが分子に与える力はピコニュートンに計算することができます。このメソッドは、ピコニュートンのテザー分子に働く力を測定し、ナノメートルで抗体の添付ファイルとの間の絶対的な分子の長さを計算します。ガラス微小球のF = V(DB)A(F =力、V =体積、D =密度、=加速重力のために、B =避難水の密度)。
6。代表的な結果:
ビーズの準備が適切に行われた場合、まだ時折ビーズ塊があるかもしれないが、最低限のビーズ凝集が存在します。スコープで見たときは、チャンバー内でペアになっているかどうかビーズの合理的な分布があるはずです。
できるだけ多くの振動を最小限に抑えることが重要であり、このどちらかのエアテーブルを行うために、特別なショックEQマウントを保持している三脚の脚の吸収、またはばねが振動絶縁のために使用することができます利用するシステムに。
密封されたフローチャンバーに係る別の有用なヒントは、それは全面的に構築された後、それはレベルのテーブルの上に約5分間静置することです。これは、任意のアタッチされていない大きなビーズはニトロセルロースの層でバッファし、残りを下にフロートすることができます。スライドではなく完了時に直接マウントされていた場合、調査官は、継続的に文字通り視野を飛んでビーズに対処する必要があります、これはビデオの取り込み中に起これば、それは目を破損する可能性が電子実験。これが適切に行われている場合は、ビーズの便が大幅に最小化され、クリーンなビデオの結果さ。
潜在的なビーズのペアがGFS演算子によって識別されると、それはペアの動作を監視するための予備回転を介して置くと便利です。まれに、大きなビーズはしっかりとスライドに貼付されていません。この問題が発生した場合、それは買収の期間を通じて固定された位置に大きなアンカービーズ滞在することが重要ですので、ペアを利用するには使用はありません。ペアが安定していると示していない場合、"アンカービーズロールを、"それは実験に適しています。
重力からの相対角度の変化に対するビーズ分離距離のプロットは、取得したデータを評価するために使用することができます。良い代表的な結果は、高原にはほとんど分離を表しているだろうし、モバイルビーズのために重力の影響の固定されたビーズから自身を解放すると、グラフはより多くの分離が表示されます。これは素敵なピークはD MAXと呼ばれるに達するまで継続され、曲線が再びベースラインに戻って下向きに始まるd minは[図1]。理想的な条件下では、この署名は対称的です。非代表の結果は異なるD MIN - D MAX - D 分のパターンを示していない支離滅裂なパターンを持つグラフを表しているだろう、またはそれが間塵の部分があったかもしれないことを示す一つの分子に対して高すぎる桁違いな分離を示すだろうモバイルビーズは単純にすべてに添付されていないことを多分ビーズ、または。 、ペアを見つけ、それを撮影、それを処理し、そしてそれを分析のプロセスは、不適切なビーズのペアがグレーに淘汰される多くのストップギャップを持っています。そのため、全体のプロセスの最後に到達して、前の結果と一致している分子の長さを持っている場合、にオリジナルのビーズペアが代表的であるとの結果に含めることのできることを確信できます。良い日には、ビーズのペアショットの約半分は、正当なデータポイントになることを通して撮影することができます。例えば、MF20とMF30抗体との間のミオシンのコイルドコイルの長さは、EMのデータおよびAFM、100nmの2,7,8,9に近づくために、データに基づいて知られています。結果はこの長さは数倍である場合、サンプルが集約しています。ここに示された結果は[図2] MF30の測定と密接に同意する(ミオシンのサブフラグメント-2のN末端に結合する)、標準的なGFSローテーションの実験からのものであるし、96 nmの± 5nmの距離を示し、MF20文献値から推定されたミオシン上(ライトメロミオシンに結合する)抗体の分離距離[図3]。
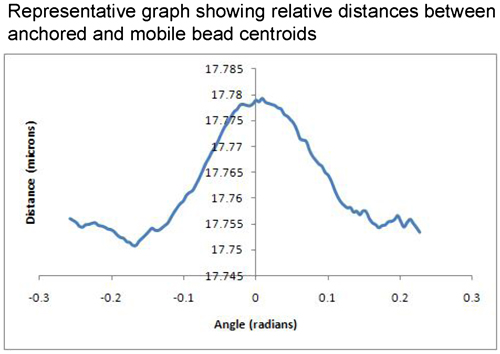
図1。GFSの構成。 GFSの主要部分が標識されている。
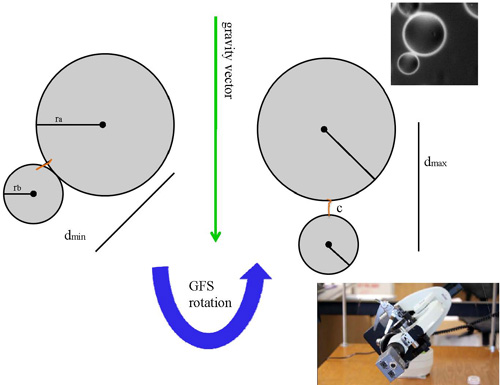
図2。GFSの原理の模式図。左側は固定されたビーズの重心からの最小距離でモバイルビーズの重心を示しています。 GFSが回転するように、モバイルビーズはまた、テザー分子の軸と平行に実行されている重力のベクトルに合わせて配置されます。この位置では、モバイルとアンカービーズの重心間の距離が最大になります。右上には、GFSの映画から代表的なスライスを示しています。右下には、GFS受けて回転のイメージです。
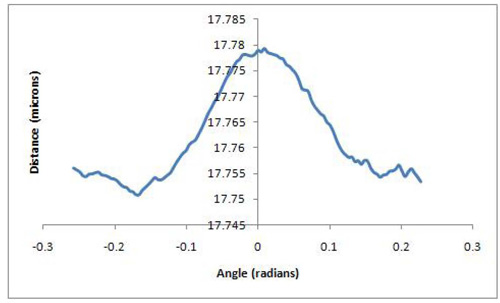
図3代表のグラフの左側にあるDの分を示す結果、17.78ミクロンの相対的な分離でD MAX、とビーズの間の相対的な分離の17.75ミクロンのベースラインの周りのD 分に復帰。
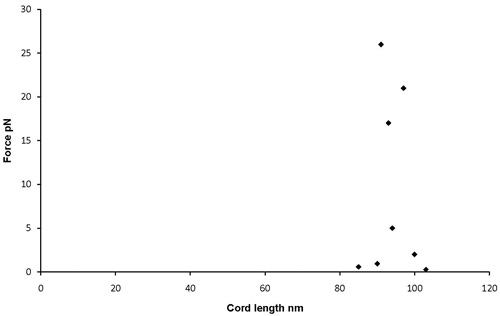
図4。MF20とMF30抗体平均96 nmの間の距離を示すGFSローテーションの実験の代表的な結果。これは、S2のおおよその長さを表します。
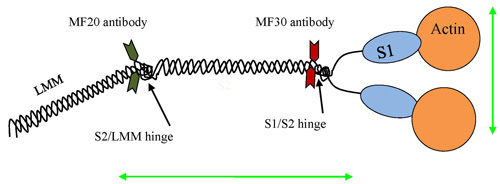
図5は、。ミオシンII二量体は、抗体および/ またはアクチンの添付ファイルを含むGFSで使用するために可能な添付ファイルを表示するために使用。別の添付ファイルの可能性別のGFSの戦略が異なる領域を測定するために、または力の垂直またはミオシンの二量体の棒のドメインへの並列を適用することが可能になります。
ディスカッション
デジタル閾値表現にムービーを変換する場合、それはビデオの各フレームで同じ領域を維持するために閾値画像のための非常に重要です。ビーズペアのビーズは、互いに独立して移動するので、閾値領域でのドリフトも、大きな誤差を漂流して紹介するビーズの重心間の相対距離を引き起こす可能性があります。しきい値領域を制御することダウンは5nm〜26 nmの距離の測定値の5倍の誤差を減?...
開示事項
謝辞
この材料は、グラント番号0842736の下で全米科学財団によってサポートされている作業に基づいています。
資料
| Name | Company | Catalog Number | Comments |
| 3-Aminopropyltriethoxysilane | Polysciences, Inc. | 919-30-2 | |
| Acetone | Fisher Scientific | A18P-4 | |
| Pyridine | Sigma-Aldrich | 110-86-1 | |
| Glutaraldehyde | Fisher Scientific | G7776 | |
| Glycine | Research Organics | BP381-1 | |
| Tris | Sigma-Aldrich | 9682T | |
| Sodium azide | Amresco | 71289 | |
| BSA | Sigma-Aldrich | AMR-0332-100G | |
| NaCl | Sigma-Aldrich | S7653 | |
| EDTA | MSI | E9884 | |
| Nitrocellulose | Sigma-Aldrich | 60443 | |
| N-N Dimethyl Formamide | Extracted from Large New | D4254 | |
| Rabbit skeletal myosin II | Zealand White Rabbits (7-8) | NA | |
| MF30 antibody (9-10) | Developmental Studies Hybridoma Bank | MF30 | |
| MF20 antibody (6) | Hybridoma Bank | MF20 | |
| Lab microscope | Boreal | WW57905M00 | |
| Equatorial mount | Celestron | CG-5 | |
| Digital video cam | Sony Corporation | XCDV60 | |
| Caliper release | Cabelas | IA-415482 | |
| Compression spring | Jones Spring Co. | 723 | |
| Extension spring | Jones Spring Co. | 770 | |
| ImageJ | National Institutes of Health | NA | |
| Fire-i drivers & application | Unibrain | 3.80 | |
| Excel | Microsoft | NA |
参考文献
- Schwaiger, I., Sattler, C., Hostetter, D. R., Rief, M. The myosin coiled-coil is a truly elastic protein structure. Nat. Mater. 1, 232-235 (2002).
- Root, D. D., Yadavalli, V. M., Forbes, J. G., Wang, K. Coiled-coil nanomechanics and uncoiling and unfolding of the superhelix and alpha-helices of myosin. Biophysical Journal. 90, 2852-2866 (2006).
- Nishizaka, T., Miyata, H., Yoshikawa, H., Ishiwata, S., Kinosita, K. Unbinding force of a single motor molecule of muscle measured using optical tweezers. Nature. 377, 251-254 (1995).
- Gawalapu, R. K., Root, D. D. Fluorescence labeling and computational analysis of the strut of myosin's 50 kDa cleft. Arch. Biochem. Biophys. 456, 102-111 (2006).
- Kellermayer, M. S. Z. Visualizing and manipulating individual protein. Molecules Physiol. Meas. 26, R119-R153 (2005).
- Shimizu, T., Dennis, J. E., Masaki, T., Fischman, D. A. Axial arrangement of the myosin rod in vertebrate thick filaments: immunoelectron microscopy with a monoclonal antibody to light meromyosin. J. Cell Biol. 101, 1115-1123 (1985).
- Godfrey, J. E., Harrington, W. F. Self-association in the myosin system at high ionic strength. I. Sensitivity of the interaction to pH and ionic environment. Biochemistry. 9, 886-893 (1970).
- Root, D. D., Stewart, S., Xu, J. Dynamic docking of myosin and actin observed with resonance energy transfer. Biochemistry. 41, 1786-1794 (2002).
- Xu, J., Root, D. D. Conformational Selection during Weak Binding at the Actin and Myosin Interface. Biophys. J. 79, 1498-1510 (2000).
- Sattin, B. D., Pelling, A. E., Goh, M. C. DNA base pair resolution by single molecule force spectroscopy. Nucleic Acids Res. 32, 4876-4883 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved