このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
バイオマットから高分子量DNAの抽出
要約
我々は、塩性微生物マットから高分子量のDNAを抽出するための改良されたプロトコルを提供しています。微生物細胞は、DNAの抽出と精製の前にマットのマトリックスから分離されています。これは、濃度、品質、およびDNAの大きさを高めます。プロトコルは、他の難治性のサンプルに使用することができる。
要約
メタゲノムデータの成功したと正確な分析と解釈は、質の高い、高分子量(HMW)のコミュニティDNAの効率的な抽出に依存しています。しかし、環境マットのサンプルは、しばしば高品質の、HMWのDNAの大規模な濃度を得るために困難をもたらす。塩性の微生物マットは高い細胞外高分子物質の量(EPS)1、抽出されたDNAの下流のアプリケーションを阻害する塩が含まれています。ダイレクトと厳しい方法は、しばしば難治性の試料からのDNA抽出に使用されています。マット、接着剤マトリックス、のEPSは直接溶解中にDNA 2,3を結合するので、これらのメソッドは一般的に使用されています。厳しい抽出方法の結果として、DNAは、小さいサイズ4,5,6に分割になります。
DNAは、このように大規模挿入ベクトルのクローニングには不適切となります。これらの制限を回避するために、我々は塩性微生物マットから良い質と量のHMW DNAを抽出するために改良された方法論を報告する。我々はブレンドと分画遠心法を通じて、背景のマットマトリックスからの微生物細胞の分離を伴う間接的な方法を採用。機械的および化学的手順の組み合わせは、抽出された微生物細胞からDNAを抽出精製するために使用された。我々のプロトコルは1.6の280分の260比で、マットのサンプルのグラム当たりHMW DNA(35〜50キロバイト)の約2μgを得られます。また、16S rRNA遺伝子7の増幅は、プロトコルが汚染物質のいずれかの阻害効果を最小化または排除することができることを示唆している。我々の結果は、機能的なメタゲノム研究のためのバイオマットからHMWのDNAの抽出のための適切な方法論を提供し、DNA抽出が難しいされているから、他の環境試料にも適用可能である。
プロトコル
1。微生物細胞の抽出:
- 徹底的に混合することにより滅菌粉砕乳棒でバイオマットをホモジナイズする。場所は約全30ワーリングブレンダーの滅菌容器に均質化したマットの材料のg(湿重量)、1MのNaCl(または使用中のサンプルに特定の濃度)の約100mLを追加し、1分間、中程度の速度で三回をブレンド1分間-20℃の冷凍庫で断続的に冷却。 250mLの遠心ボトルにスラリーを移し、1 M NaClで、残りの空のボリュームを埋める。オートクレーブのDI水にNaCl溶液を準備し、それを殺菌するフィルタ。
- さらに30分間室温でボルテックス(渦魔神R 2)を用いて(150 rpm)し振とうすることにより、マトリックスからの微生物細胞を取り除く。
- 4℃で15分間のための低速で遠心(500分の1グラム)ゆっくりと堆積物への最小限の障害でフラスコに上清を移す。
- ペレット堆積物を使用して、ステップ1.3の末尾に、各堆積物ペレットに新鮮なNaClの添加でブレンドし、手順1.2と1.3はさらに4回繰り返します。各NaClの抽出からの上清を新しいフラスコに転送されます。
- 4℃で15分間高速(25,000 × g)で200mLのアリコートに5セルの抽出と遠心の上清を組み合わせる℃に上清を廃棄し、2%ヘキサメタリン酸ナトリウム10mlに各細胞ペレットを再懸濁し、ペレットを組み合わせ、2%ヘキサメタリン酸ナトリウムを加えて200mlにボリュームを構成しています。 30分間室温(150 rpm)で振とうして細胞を洗浄。このステップの最後には、次のステップに進むために、細胞の唯一のチューブが必要です。
- 4℃で15分間のための高速で遠心分離(25,000 × g)でTE(50mMのEDTA)200mLに上清を、再懸細胞ペレットを捨て、そして室温で10分間(150回転)振とうして細胞を洗浄。
- 4℃で15分間、高速(25,000 × g)で遠心℃で、15 mLのTE(10mMのEDTA)に再懸濁し、上清を、破棄、および-80℃ストア200μL分注し° CまでのDNA抽出のために必要。
2。 DNAの抽出と精製:
- 200μLの微生物細胞のアリコートに200μLの5 M NaCl、200μL10%SDS、および100μL14.3 Mβ-メルカプトエタノールを追加。反転4-6回で静かに混和。
- 5分間65℃の水浴で融解することによって続いて2分間液体窒素に浸して凍結融解の3ラウンドに従う混合物。 10分に解凍最後の拡張。
- 200μLの5 M酢酸カリウム(pH5.5)に、10分間氷上に場所を追加します。 4℃で10分間、10,000 xgで遠心℃、ワイドボアピペットチップを用いて上清を新しいチューブに移す。 1時間のためにRNaseを37℃でインキュベート℃の3μLを加える。
- 等量のクロロホルムを追加し、反転が簡単に振る、そして室温で10分間、15,000 xgで遠心。
- 慎重にワイドボアピペットチップを使用して、新しいチューブに上清の最上層を移す。上部と下部層の間にある白い界面層をピペッティングは避けてください。クロロホルム抽出の手順を繰り返します。
- DNAを沈殿に30分間氷上で上清とインキュベートするとチルドの等量が(-20℃)、イソプロパノール追加。
- 4℃で10分間ペレットDNAにCの最大速度(20800 × g)で遠心。
- 上清を破棄し、チルド1mLでDNAペレットを洗浄は、70%エタノール(-20℃時)。
- で10分間4℃で最大スピード(20800 × g)で遠心機は、70%エタノールで洗浄し、10分間空気乾燥DNAペレットを繰り返し、上清を捨てる。 70%エタノールで洗浄を繰り返します。
- 25μLのTE(10mMのEDTA)に再懸濁し、DNA、65℃暖かい℃で5分間は、複数のチューブが使用されている場合は、1つのチューブに結合し、そしてTEで500μLにボリュームをメイク。
- 追加500μLの氷冷(または4℃)20%ポリエチレングリコール(PEG)(1.2 M NaClに調製)。氷上で10分間インキュベート反転、、、4℃で10分間ペレットDNAにCの最大速度で遠心分離により穏やかに混合する。取り外して廃棄上清を、10分間空気乾燥、冷却した70%エタノール1mLでペレットDNAを洗浄、30〜50μlのTEまたは分子等級水に再懸濁します。 65温暖化によるDNAの再懸濁を促進〜80時5分とストアのDNAのためのC℃に
3。 DNAの純度、濃度とサイズの決定:
- 280分の260と光度計1000分光光度またはその他の適切な計測機器を使用して230分の260の比率を測定することにより、DNAの品質を決定します。
- メーカーの指示に従ってクワント- ITのdsDNAのアッセイキットを用いてDNA濃度を決定する。
- パルスフィールドゲル電気泳動を用いたDNAのサイズを決定します。 0.5 × TBEで1%アガロースゲルを準備し、以下のCHEF MapperのXAシステム上でパラメータ使用、0.35 sの最初のスイッチ時間、7.67秒の最後のスイッチ時間、および120℃の時6.0 V / cmのグラデーションの角度を含め線形R係数をリアンプ。 30分間1 × SYBRグリーンIを使用してゲルを染色する。ゲル上700-1000 ngのDNAの間にロードします。図を参照してください。完全なプロトコルの圧縮された形式3。
代表的な結果:
細胞の抽出:
シーケンシャル微生物の細胞抽出物は、(上清)抽出物の濁度は、抽出数が増えるにつれて減少することが示されている。これは、連続したセルの抽出後の細胞数の減少を示唆している。ここで注意すべき重要な追加の各セルの抽出工程は、汚染物質の導入のための機会を提供するので、細胞の抽出数は、最終細胞ペレットは、全体的な微生物群集の代表となるように最小化されるべきであるということです。汚染の他の情報源は、適切な実験室無菌技術を採用することによって制御されていました。例えば、ソリューションは、オートクレーブ純水で調製し、滅菌フィルター。容器は、アルコールで滅菌オートクレーブ、紫外線や紫外線架橋剤で処理した。
DNAの濃度と品質決定:
プロトコルは、マットの1.6の280分の260比を持つ試料、および0.7の230分の260の比(表1)のグラムあたりHMW DNAの約2μgの(35〜50キロバイト)(図4)を得た。 230分の260の比率が低いように見えるが、そのような16S rRNA遺伝子のPCR -増幅のような下流分子ベースのアプリケーションの阻害は別の研究7で観察されなかった。 EPSと塩;塩性マットからそのDNAコンタミネーションの2つの主な情報源を持っていることに注意することが重要です。それは、これらの汚染物質の痕跡がダウンストリームのアプリケーションに与える影響を低減するために多大な努力にもかかわらず、230分の260の比率に影響を与えることができることが可能となる。
DNAサイズの決定:
図3に示すように、パルスフィールドゲル電気泳動は、DNAの塗抹標本で、その結果、断続的な方向のスイッチと脈動電流の流れを採用しています。我々のプロトコルは、約35〜50キロバイトのHMW DNAが得られた。いくつかのDNAサイズが30 KBより小さいかもしれないが、それは、フォスミドクローン作成は、約40 kbのDNAを挿入し、より大きなDNA断片は、無傷の生合成経路へのより大きなアクセスを提供する必要があることを考えると35キロバイト上記の塗抹標本の一部を持つことが重要です。我々の研究では、35キロバイト上記のDNAスミアは大インサートベクトルのクローニングと他の分子のアプリケーション用に切り出し、精製した。

エリューセラ、バハマにある図1。塩性の微生物マットのサンプリングサイト(ビッグ池)。

図2本研究で使用した塩性微生物マットの断面。微生物マットはエリューセラ、バハマにある塩性池から入手した。
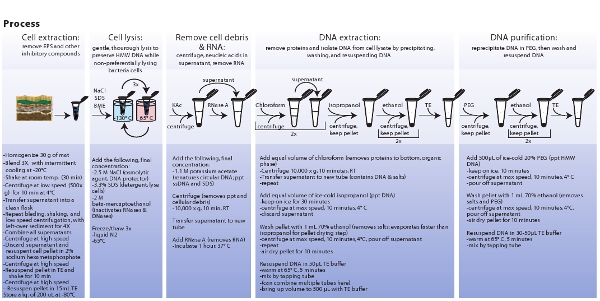
図3。微生物細胞の抽出、細胞溶解、抽出、およびメタゲノムDNAの精製に関連する手順の模式図は。 拡大画像を表示するにはここをクリックしてください。

図4パルスフィールドゲル電気泳動を用いて抽出したDNAの分子量特性評価。レーンMは、HMWマーカーであり、レーン1と2は、上記のプロトコルを使用してエリューセラ塩性のマットから抽出したメタゲノムDNAの複製です。
| 抽出方法 | 濃度ng / gの | 280分の260 | 230分の260 | |
| PEG | 担当者1 | 1806.1 | 1.64 | 0.76 |
| 担当者2 | 2010.8 | 1.61 | 0.75 |
表1。微生物塩性マットから抽出したDNAの濃度と品質の測定。
ディスカッション
複雑かつ高度に多様な微生物マット試料からの全細胞の除去が実用的ではないことを考えると、主な関心事は、抽出された細胞は、全体的な微生物マットのコミュニティを表す方法もあります。以前の研究で、微生物の16S rRNA遺伝子のPCR - DGGE解析では、このプロトコルで使用する5つの細胞の除去の手順は、全体的な微生物マットのコミュニティ7の代表的な細胞を抽出することを示し...
開示事項
謝辞
この作品は、全米科学財団環境ゲノミクスプログラム(グラント番号EF - 0723707)により賄われていた。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | コメント |
|---|---|---|---|
| β-メルカプトエタノール | シグマアルドリッチ | M3148 | |
| ポリエチレングリコール8000 | プロメガ | V3011 | 1.2 M NaClを約20% |
| カリウム酢酸 | フィッシャーサイエンティフィック | フィッシャーサイエンティフィック | |
| クワント- IT dsDNAをアッセイキット | インビトロジェン | Q33130 | |
| RNaseの | EPICENTRE | MRNA092 | |
| 塩化ナトリウム | BDHケミカルズ | BDH8014 | 適切なコンク。 |
| 硫酸ドデシルナトリウム | フィッシャーサイエンティフィック | 03-500-509 | 水の10% |
| ヘキサメタリン酸ソーダ | EMDケミカルズ | SX0583 - 3 | 水の2% |
| TBE | フィッシャーサイエンティフィック | BP1333 - 1 | |
| CHEF MapperのXAシステム | バイオラッドラボラトリーズ | 170-3670 | |
| 光度計1000分光 | サーモサイエンティフィック | ND - 1000 | |
| ボルテックス | サイエンティフィックインダストリーズ株式会社 | ||
| 紫外線架橋剤 | UVP | ||
| ワーリングブレンダー | ワーリング実験室 | LB10S |
参考文献
- Decho, A. W. Microbial biofilms in intertidal systems: an overview. Cont. Shelf Res. 20, 1257-1273 (2000).
- Dupraz, C., Visscher, P. T. Microbial lithification in marine stromatolites and hypersaline mats. Trends Microbiol. 13, 429-438 (2005).
- Steffan, R. J., Goksoyr, J., Boj, A. K., Atlas, R. M. Recovery of DNA from soils and sediments. Appl. Environ. Microbiol. 54, 2908-2915 (1988).
- Lee, Y. K., Kim, H. W., Liu, C. L., Lee, H. K. A simple method for DNA extraction from marine bacteria that produce extracellular materials. J. Microbiol. Methods. 52, 245-250 (2003).
- Roose-Amsaleg, C. L., Garnier-Sillam, E., Harry, M. Extraction and Purification of Microbial DNA from Soil and Sediment Samples. Appl. Soil Ecol. 18, 47-60 (2001).
- de Lipthay, J. R., Enzinger, C., Johnsen, K., Aamand, J., Sørensen, S. J. Impact of DNA extraction method on bacterial community composition measured by denaturing gradient gel electrophoresis. Soil Biol. Biochem. 36, 1607-1614 (2004).
- Bey, B. S., Fichot, E. B., Dayama, G., Decho, A. W., Norman, R. S. Extraction of High Molecular Weight DNA from Microbial Mats. Biotechniques. 49, 631-640 (2010).
- Kakirde, S. K., Parsley, L. C., Liles, M. R. Size does matter: Application-driven approaches for soil metagenomics. Soil Biology & Biochemistry. 42, 1911-1923 (2010).
- Rodon, M. R., August, P. R., Bettermann, A. D., Brady, S. F., Grossman, T. H., Liles, M. R., Loiacono, K. A., Lynch, B. A., MacNeil, I. A., Minor, C., Tiong, C. L., Gilman, M., Osburne, M. S., Clardy, J., Handelsman, J., Goodman, R. M. Cloning the Soil Metagenomic: A Strategy for Accessing the Genetic and Functional Diversity of Uncultured Microorganisms. Appl. Environ. Microbiol. 66, 2541-2547 (2000).
- Beja, O., Aravind, L., Koonin, E. V., Suzuki, M. T., Hadd, A., Nguyen, L. P., Jovanovich, S. B., Gates, C. M., Feldman, R. A., Spudich, J. L., Spudich, E. N., DeLong, E. F. Bacterial Rhodopsin: Evidence for a New Type of Phototrophy in the Sea. Science. 289, 1902-1906 (2000).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved