このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
モデリングとイメージング、3次元の集団細胞浸潤
要約
三次元細胞外マトリックスへの腫瘍細胞の浸潤のモデルより反映 in vivoで状況。蛍光標識細胞の共焦点イメージングと組み合わせたマトリックス浸潤アッセイを使用して、侵略モードと主要な対後の細胞の明確な寄与に関する詳細な情報を得ることができる。
要約
癌の悪性腫瘍の特徴は、浸潤転移1です。いくつかの癌(E. グラム 。神経膠腫2)で、健康な組織を周囲に局所浸潤は、病気や死亡の根本的な原因です。他のがんの場合(E. グラム 。乳房、肺、 等 。)、それは腫瘍細胞は、原発腫瘍塊から移動遠位部位を植民地化し、最終的に臓器不全に関与する、転移のプロセスであり、それは最終的に罹患率につながると死亡3。それは、浸潤転移は、がんによる死亡4の90%に責任があると推定されている。その結果、改善の診断と治療5の目的のために浸潤転移の分子プロセスと重要なタンパク質のメディエーターを特定するのに強い関心が集まっている。
癌の科学者にとっての課題は、十分にin vivoの状況に似ている浸潤アッセイを開発することである正確な病気のモデリング6を有効にする。二次元の細胞の運動性アッセイは、侵略の一つの側面についてのみ有益であり、また重要な要素になっているアカウント細胞外マトリックス(ECM)タンパク質のリモデリングは考慮されていません。最近、研究では、腫瘍細胞の浸潤の我々の理解を精緻化し、個々の細胞は細長いまたは丸みのモード7で動く可能性があることを明らかにした。さらに、癌8の普及に、特に上皮特性を維持する高度に差別化された腫瘍の細胞はストランド、シートやサーバークラスタ内に侵入した集団的侵略の貢献のより深い理解を、、、があった。
私たちは、集団的侵略10に候補タンパク質の寄与を調べるための洗練された方法9を提示する。特に、異なる蛍光タンパク質を発現する細胞の工学的に個別のプールで、それは分子的に活動してproteiを分析することが可能です。NSは、以下の細胞で必要とされる対リードする細胞に必要。 RNAiの使用は、実験的に個々の細胞侵入だけでなく、集団の侵略の異なる位置に関連するプロセスを分解するための分子ツールを提供しています。この手順では、蛍光標識した細胞の混合物が以前にマトリゲルECM蛋白質で満たされたトランスウェルインサートの底に播種され、その後、フィルターを通過し、マトリゲルに"上向き"に侵入することができました。 3次元表現への共焦点イメージングで得られたZシリーズの画像スタック、、の再建は、総称してストランドとリードに対する次のような位置で蛍光標識した細胞の表現の分析を侵入の可視化が可能になります。
プロトコル
1。蛍光タンパク質と細胞のレトロウイルスラベリング
- 0.25 × 10 6細胞におけるウェルあたり6ウェルの外プレートレトロウイルスパッケージング細胞( 例えばフェニックス) 10%ウシ胎児血清(FBS)/ DMEMで料理。
- レトロウイルスDNAとトランスフェクション細胞を48時間後に細胞はメーカーの指示に従ってEffecteneを使用。
- 後で培地で2回24時間の井戸を洗い流した後、ウェルあたり10%FBS / DMEM 1.5 mlを加える。
- 2 mlのマイクロチューブにピペッティングし、転送することにより、48時間後に組織培養の培地にパッケージ化ウイルスを収集する。
- ペレット任意のセルに5分間1600 rpmで遠心する。
- -80きれいなチューブに上清と格納、削除℃の
- レトロウイルス形質導入される細胞を6ウェルディッシュのウェル当たり1.5 × 10 5細胞で行わ播種し、37℃で一晩加湿インキュベータ内に配置されます。
- 翌日、細胞や広告からメディアを削除するウイルスストックのD 1mlを各ウェルに4μlのポリブレン(濃度2μg/ mL)で補った。インキュベーター内でプレートを交換してください。
- 5〜6時間以下のインキュベーション、各ウェルプレートに2 mlの10%FBS / DMEMを追加するには° C inbubatorは一晩37に置き換えられます。
- 翌日、適切な選択培地を追加する前にメディアを交換し、細胞を24時間可能です。
- ときに(成長細胞の割合、およびレトロウイルス形質導入の効率性に依存するが、通常は4から14日から行われる)コンフルエントに、基準として非形質導入細胞を用いた蛍光用セルとソートをトリプシン処理、収集し、プールの細胞と蛍光-80℃、非形質導入細胞と凍結のアリコート℃を超えて将来の使用のために。
2。逆マトリゲル浸潤アッセイ
- ゆっくりと氷(図1A)の完全なマトリゲル(すなわち、増殖因子を含む)のアリコートを解凍。
- 氷冷PBSで一度解凍、希釈マトリゲル1:1(その他の追加と共に前の希釈にPBSで2倍の濃度でのトリートメント、図1B)。それは容易に重合する前にマトリゲルを処理するようにするには、すべてのプラスチック製品(例えばチップ、チューブなど)の氷冷する必要があります。
- コーティングされていない多くの8ミクロンの細孔6.5ミリメートルの直径は、24ウェル組織培養プレートのウェルに必要なTranswellsとして挿入し、慎重に各ウエルに希釈したマトリゲルの100μlをピペットで37〜30分間インキュベートしておく℃で固化する(図1C)。
- この時間の間に、彼らの正常な成長に各前処理条件の細胞株に依存しミリリットルあたり4〜1の間× 10 5細胞、、(E. グラム 。siRNAは、薬物治療)で1つまたは複数の蛍光標識した細胞懸濁液を準備する培地(図1D)。
- マトリゲルが固化したときに、フィルタ(図1E)の上向き下側にTranswellsと細胞懸濁液の100μL添加して反転。
- 慎重に24ウェル組織の基盤とTranswellsをカバー培養プレート、細胞懸濁液(図1F)の各液滴と接触させる。
- 細胞接着性(図1G)を可能にするために4時間倒立状態でプレートをインキュベートする。
- この時間の後、プレートを右サイドアップにして、2 × 1 ml血清フリー培地(図2A)に順次浸漬して、各トランスウェルを洗う。
- (図2B)も必要な薬や治療法を含む3番目にTranswellsを残す。
- ピペット固化マトリゲル/ PBS混合物の上にトランスウェルに10%FBS / DMEMを加えた誘引物質(E. グラム 。25 ng / mlの時EGF)の100μlを、37℃で3-5日のために蓋とインキュベートを交換してください° Cで5%CO 2(図2C)。
3。染色と可視化
- 蛍光タンパク質を発現する細胞は、前の共焦点顕微鏡に以下の手順をスキップできます。
- 新鮮な24ウェルディッシュと4μMのピペット1mlのマトリゲル、場所のTranswellsに侵入する画像非蛍光細胞へカルセインは、側面を波及し、上部と下部から染色するので、それぞれのマトリゲルプラグの上に染色液を午前。カルセインAM(カルセインのアセトキシメチルエステル)は、その染色細胞全体緑生細胞の色素であり、ない固定を必要としません。
- 37℃で1時間インキュベート·ポイントの細胞が完全に染色し共焦点顕微鏡によって撮像される準備ができている時、5%CO 2加湿雰囲気、でCを。
- また、マトリゲルに侵入する非蛍光細胞を固定し、3.9に3.5で後述するように染色されることがあります。
- 新鮮な24穴プレートに各トランスウェルを転送する。オーバーレイ4パーセントpara-formaldehyde/0.2%トリトンX - 100を1ml。
- 0.5時間室温でインキュベートする。
- 固定液を取り出して、1mlのPBSで3回洗浄する。
- 100μg/ mlのRNaseを使用して30分RNase処理で細胞質RNAを削除します。 PBSで2回洗浄する。
- 可視化のためにPBSで希釈した0.01 mg / mlのヨウ化プロピジウム(PI)を追加し、DAで、室温で残す0.5時間RK。 PBSで3回洗浄する。この段階では、PIは、トランスウェルは、少なくとも1ヶ月、暗所で室温で保存することができる染色。
- 共焦点顕微鏡による精密なイメージング法は、利用可能なリソースに依存しています。非浸漬20 X客観的および光学的切片をマトリゲルプラグの底から10〜15μmの捕捉以上の大規模なカバースリップ上に残存する少量のPBS(気泡が存在しないことを確認)で倒立共焦点顕微鏡、場所のトランスウェルを使用する。個々の光のスライスは、侵略の範囲(図3A)を定量化するかVolocity(図3B)などの適切なソフトウェアを使用して3次元のオブジェクトに組み込むために使用することができます。
4。代表的な結果
光学スライスのZシリーズの例を図3Aに示されています。このインスタンスでは、細胞は、カルセインAMで染色し、フィルタから浸潤細胞の数は、距離とともに減少する見ることができます。での定量化vasionは、それぞれの間隔で、または上記に詳述固定/染色法を用いて、各位置でのPI陽性核をカウントすることで負のピクセルに正のピクセル回答カルセインの比率を分析することによって行うことができます。カルセインの利点の一つである染色午前細胞の浸潤のその3次元再構成は、侵略のモード(図3B)の視覚的描写を与えて、そのようなVolocityとしてソフトウェアを使用して組み立てることができます。細胞を蛍光タンパク質の発現によってラベル付けされている場合は、各色のセルの位置はどちらの側(図3C)から、または再構築(図3D)を介してスライスを行うことで閲覧、3次元再構成で視覚化することができます。
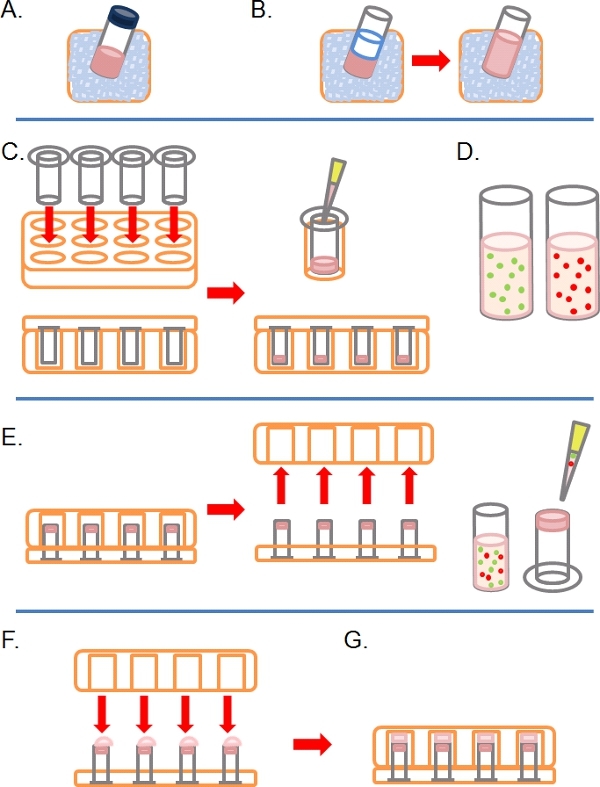
図1逆浸潤アッセイのセットアップに必要な手順の模式図。 A)マトリゲルECMは、氷上で解凍。 B)マトリゲルをPBSで二回最終濃度で、任意の薬物治療を含むで1:1に希釈される。 C)トランスウェルインサートは、マルチウェルプレートに入れ、およびマトリゲルは、それぞれにピペットされています。 D)細胞懸濁液は、所望の濃度でなさ。 E)マトリゲルを設定した後、プレートを反転して削除され、細胞はトランスウェルインサートの底面フィルター上にプレーティングされています。 F)倒立位置では、多層板を慎重に細胞懸濁液と接触すること、トランスウェルインサート上に置かれる。 G)細胞は4時間のためにフィルタに付着させています。
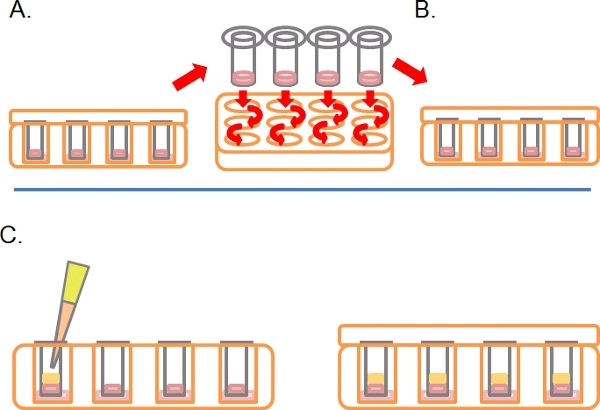
図2逆浸潤アッセイの模式図の継続。細胞が付着している)したら、二回バラセルを削除するには、無血清培地に各トランスウェルを浸す。 B)場所は、必要に応じて最終的なも含まれているメディアに加え、治療にトランスウェルを洗浄した。 C)必要に応じて治療法と走化性因子(例えば10%ウシ胎児血清)を含むメディアは、慎重にマトリゲル上に重層されています。
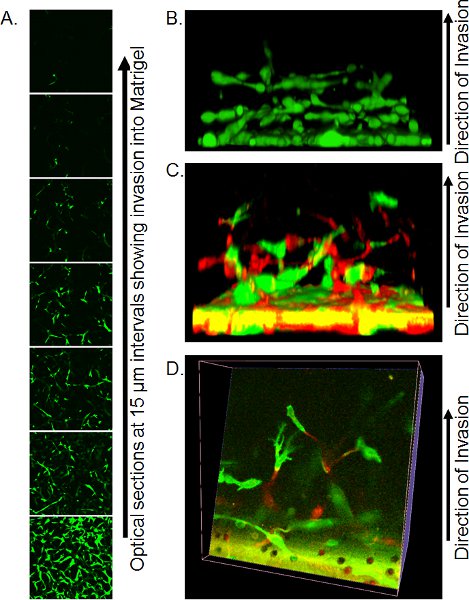
図3。逆浸潤アッセイの結果の代表的な画像。 A)カルセインで染色した細胞の光学セクションでは、共焦点顕微鏡で15μmの間隔で撮影された、マトリゲルに侵入しています。側から見た焦点Zシリーズの画像のスタックからの細胞浸潤の3次元再構築のB)再建、。参考文献10から転載。側から見たマトリゲルに侵入GFPおよびRFP標識細胞のC)の三次元再構築。 GFPおよびRFP標識細胞の3次元再構成を介してD)光学スライス。参考文献10から転載。
ディスカッション
マトリゲル浸潤アッセイは、従来、化学誘引物質誘発性運動性に向かってとし、下部のフィルタを通して細胞外マトリックス蛋白質の層の上に置かれた細胞を使用して設定されています。侵襲性がフィルターの下側にカウントできるセルの数の関数として採点した。上記の"逆"浸潤アッセイでほとんど差が実質的にあるが、彼らはマトリゲルを通って移動して浸潤細胞を可視化するこ?...
開示事項
著者らは、開示することは何もない。
謝辞
この研究のための資金は、癌研究英国からのものです。
資料
| Name | Company | Catalog Number | Comments |
| 試薬の名前 | 会社 | カタログ番号 | |
|---|---|---|---|
| DMEM(ダルベッコ変法イーグル培地) | GIBCO | 21969 | |
| ウシ胎仔血清 | PAA | A15 - 101 | |
| ペニシリンストレプトマイシン | GIBCO | 15140 | |
| 200mMのL -グルタミン(100倍) | GIBCO | 25050-032 | |
| ピューロマイシン | シグマアルドリッチ | P8833 | |
| 0.05%トリプシンEDTA | GIBCO | 25300 | |
| ポリブレン | シグマアルドリッチ | AL - 118 | |
| リポフェクトアミン2000試薬 | インビトロジェン | 11668019 | |
| 6.5ミリメートルTranswells、8.0μmの孔径 | コーニング | 3422 | |
| 完全なマトリ | BDバイオサイエンス | 354234 | |
| カルセインAM | インビトロジェン | C1430 | |
| RNaseの | キアゲン | 19101 | |
| ヨウ化プロピジウム | シグマアルドリッチ | P4864 | |
| 共焦点microcope | ライカ | SP2MP |
参考文献
- Olson, M. F., Sahai, E. The actin cytoskeleton in cancer cell motility. Clin. Exp. Metastasis. 26, 273-287 (2008).
- Hoelzinger, D. B., Demuth, T., Berens, M. E. Autocrine factors that sustain glioma invasion and paracrine biology in the brain microenvironment. J. Natl. Cancer. Inst. 99, 1583-1593 (2007).
- Chaffer, C. L., Weinberg, R. A. A perspective on cancer cell metastasis. Science. 331, 1559-1564 (2011).
- Hanahan, D., Weinberg, R. A. The Hallmarks of Cancer. Cell. 100, 57-70 (2000).
- Francia, G., Cruz-Munoz, W., Man, S., Xu, P., Kerbel, R. S. Mouse models of advanced spontaneous metastasis for experimental therapeutics. Nat. Rev. Cancer. 11, 135-141 (2011).
- Hooper, S., Marshall, J. F., Sahai, E. Tumor cell migration in three dimensions. Methods. Enzymol. 406, 625-643 (2006).
- Croft, D. R., Olson, M. F. Regulating the conversion between rounded and elongated modes of cancer cell movement. Cancer Cell. 14, 349-351 (2008).
- Wolf, K. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell. Biol. 9, 893-904 (2007).
- Hennigan, R. F., Hawker, K. L., Ozanne, B. W. Fos-transformation activates genes associated with invasion. Oncogene. 9, 3591-3600 (1994).
- Scott, R. W. LIM kinases are required for invasive path generation by tumor and tumor-associated stromal cells. J. Cell. Biol. 191, 169-185 (2010).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved