Method Article
インフルエンザポリメラーゼPB2サブユニットの遺伝子の構造決定のためのマルチターゲット並列処理アプローチ
* これらの著者は同等に貢献しました
要約
構造に基づいたドラッグデザインは、薬物開発において重要な役割を果たしている。並行して複数のターゲットを追求すると、大幅にリード発見のための成功の可能性が高くなります。次の記事では、感染症のためにシアトル構造ゲノム科学センターでは、PB2インフルエンザサブユニットの遺伝子対構造決定のためのマルチターゲットアプローチを利用してどのように強調しています。
要約
強毒性インフルエンザのパンデミック発生は、世界中の人間の集団における広範な罹患率と死亡率を引き起こす可能性があります。米国だけでも、41,400人が死亡し、186万入院の平均は、毎年1インフルエンザウイルス感染によって引き起こされます。ポリメラーゼ塩基性タンパク質2サブユニット(PB2)における点突然変異はヒトの2におけるウイルス感染の適応にリンクされている。このような研究から得られた知見は、このように抗ウイルス薬ターゲットとしての可能性を強調し、病原性因子としてPB2の生物学的意義を明らかにした。
アレルギー感染症研究所(NIAID)で出す構造ゲノミクスプログラムはエメラルドバイオと一緒に感染症(SSGCID)がシアトル構造ゲノム科学センターを構成する他の3つの太平洋岸北西部機関に資金を提供しています。 SSGCIDはTHREと科学コミュニティを提供することに専念してNIAIDカテゴリAC病原体の電子次元タンパク質構造。科学コミュニティが利用できるような構造情報を作ることは構造に基づく薬物設計を加速するのに役立つ。
構造に基づいたドラッグデザインは、薬物開発において重要な役割を果たしている。並行して複数のターゲットを追求すると、大幅に経路または全体のタンパク質ファミリーをターゲットにして、新しいリード発見のための成功の可能性が高くなります。エメラルドバイオコンソーシアムをサポートするための遺伝子の構造決定のための高スループット、マルチターゲット並列処理パイプライン(MTPP)を開発した。ここでは、4つの異なるインフルエンザ株からPB2サブユニットの構造を決定するために使用されるプロトコルを説明する。
プロトコル
プロトコルの概要を図1に示す。
分子生物学
1。デザインを構築
タンパク質構築とコドン設計合成遺伝子配列を設計する遺伝子Composerソフトウェアを使用してください。ジーンComposerソフトウェアの使用は、他の場所で3詳細に提供されています。
- アライメントビューアモジュールを使用し、タンパク質配列アラインメントを比較し、タンパク質構造を定義するためにデザインモジュールを構築します。 ( 図2)可能であれば、タンパク質構造データバンク(PDB)に同族からの一次および3次元構造要素の両方にターゲットのアミノ酸配列の位置を合わせます。
- 一次構造の保全と同族の立体構造に基づいて、新しい末端を選択することによって、構造誘導構造の設計を行うためにアライメント情報を使用します。
- デザイン挿入PCR(iPCRを)とベクトルPCR(VPCR)アンプリマー(端末プライマー)。
- ジーンCを使用したomposerのタンパク質対DNAアルゴリズムは、コドン設計された核酸配列内に構築されるアミノ酸配列を逆変換する。
- E.での発現のための配列を最適化するための適切なコドン使用テーブル(CUT)を使用大腸菌 。
- 実質的にN末端6×ヒスチジンタグおよび容易な精製を可能にするSmt3/SUMO融合タンパク質を組み込むように修正pET28ベクターにインサートをクローニングする。
- DNA 2.0と統合されたDNAの技術からの注文プライマーを用いて合成遺伝子の順序を置くこと。
2。ポリメラーゼ不完全なプライマー伸長(PIPE)クローニング
- プライマーおよび遺伝子を準備
- 1分間1,000 rpmでプライマーを含むベンダー提供のプレートを遠心分離します。
- 100μMにプライマー濃度を持参し、50μlのTEバッファーを追加します。
- 96ウェルV底プレートに脱イオン(DI)水で10μMにプライマーを希釈します。
- 1分間1,300 rpmで1.5 mlのチューブにベンダー提供の遺伝子を遠心分離します。
- TEバッファーを使用して、50 ngの/μlに各チューブのDNA濃度をもたらす。
- 1.5 mlのチューブに、10 ngの/μlに各プライマーの希釈液を作る。
- 使用しないときは-20℃で保存プライマーと遺伝子。
- PCR(iPCRを)を挿入準備
- 氷の上でPfuのマスターミックスのバイアルを解凍、室温で遺伝子とプライマーを保つ。
- プライマーセットにプレートマップ割り当てる井戸を作成し、構築します。
- 96ウェルPCRプレートの各ウェルに、DI水の13μLを加える。
- 各ウェルの間にヒントを変更するために確保し、プレートマップに従って96ウェルプレートの各反応を楽しみに5μlのとリバースプライマーの5μLを加える。
- ウェルプレートマップに従ってその適切に各全長遺伝子の2μLを加える。
- 各ウェルの間にヒントを変更するために確保し、各ウェルにPfuのマスターミックス25μLを加える。
- 以下のPCR条件を用いて反応サイクル:
- 95℃で2分
- 95℃30秒
- 50°C 45秒
- 68℃で3分
- 4℃∞
- 25サイクルのステップBDを繰り返します。
- 新たな96ウェルPCRプレートに各iPCRを反応液10μlを移す。
- 各サンプルに6X負荷色素の3μLを加える。
- フラグメントの増幅を確認するために、100〜500 bpsのDNAラダーの隣に110 Vで1%TAE EtBrをアガロースゲル上で各サンプルを分離する。
- -20°Cで保存iPCRを製品使用しないときは(凍結融解を極力避けるため)。
3。ベクトルPCR(VPCR)を準備
- 形質転換の一晩培養を開始プラスミドpET28ベクターを大腸菌 。
- 50μgの/ mlのカナマイシンを2-YTブロス2個の5 mlチューブに接種。
- 220rpmで37℃で一晩培養°シェーカーでCを育てる。
- 15分間3,000 rpmで遠心分離することによって一晩成長後の文化をスピンダウン。
- キアゲンQIAを使用プレップスピンミニプレップキットは、製造元の指示に従って、細菌ペレットからpET28ベクトルを抽出する。
- 抽出pET28プラスミドのセットアップ制限酵素消化。
- pET28ベクトルの20μlに10倍のBamHIバッファー2.2μL及びBamHIとHindIIIを1μlを追加します。
- 37℃で1時間反応をインキュベート℃で
- ゲル上の別の消化産物。
- 2.2.10手順を参照してください。
- ゲルからベクトル帯域をカットし、製造元の指示に従ってをQIAquickゲル抽出キットを用いて精製する。
- 光度計を用いて、DNA濃度を定量化する。
- は10ng /μlにベクトルをカット希薄。 -20°C使用しないときに保管してください。
- VPCRプライマーを準備します。
- 1,300 rpmで1分間IDT供給oligonucliotidesを遠心分離。
- DI水で100μMの濃度を持参。
- 1.5mlチューブの両方のフォワードおよびリバースプライマーの10μMの希釈を準備します。
- -20℃で保存プライマーおよびプライマー希釈
- 室温で氷の融解テンプレートとプライマーでPfuのマスターミックスを解凍。
- 96ウェルPCRプレートのセットアップVPCR反応:
- 96ウェルプレートの最初の行では、順方向および逆方向の両方VPCRプライマー60μlの消化pET28テンプレート(10 ngの/μL)の24μLを兼ね備えています。
- 12チップマルチチャンネルピペットを用いて、プレートのウェル残りの各プライマーとテンプレートマスターミックス12μLを移す。これは、プレートの各ウェルにおけるプライマーとテンプレートマスターミックス12μlのになるはず。
- 各ウェルにDI水13μLを加える。
- 各ウェルにPfuのマスターミックス25μlのを追加します。
- ステップ2.2.7で使用したPCR条件を通じサイクルが反応。
- 15ミリリットルファルコンチューブにVPCR反応のプールすべて。
- 消化pET28ベクトルのゲル(予想される長さにプールされたPCR産物10μlのを分離することによってフラグメント増幅を確認約6 KB)です。
- 2.2.10手順を参照してください。
- プレートをマージする準備をします。
- 96ウェルV字底プレートの各ウェルにVPCR産物のアリコート3μlの。
- -20で保存プレート°iPCRを製品とマージされるまでC。
4。 iPCRをとVPCR製品をマージ
- 解凍iPCRを製品や室温での事前分注しVPCR 96ウェルマージプレート。
- マージプレートの、それぞれのウェルに各iPCRを製品の3μLを加える。
- トップテン化学的にコンピテント細胞にプレートをマージ変換。
- ベンダー提供のケミカルコンピテント細胞の単一50μlをチューブに各マージ反応2μlのを追加し、製造業者の供給するプロトコルを進める。
- 変換プレートから各構築のために一晩培養を準備します。
- ディープウェルブロックの各ウェルに25ミリリットル滅菌容器からアリコートを5ml TBブロス(50μgの/ mlのカナマイシンを含む)。
- 殺菌を使用したル技術は、各変換プレートから単離したコロニーを選択し、ディープウェルブロックの適切な十分に接種。
- Airporeカバー付きブロックをカバーしています。
- 37℃で一晩220 rpmでブロックを横に振る。
- 4,000 rpmで30分間ブロックを遠心分離により細胞をペレット化。
- 上清を捨て、ペーパータオルで乾燥したブロックの上部をなでる。
- ミニプレップは、製造業者の指示に従ってキアゲン96ウェル真空装置を用いた。
5。に成功クローン構築のグリセロールストックを準備する
- 正常に製造業者の指示に従ってBL21(DE3)コンピテント細胞に化学的に配列検証DNAをクローニングし、トランスフォーム。
- 各構築物については、2 - YTブロス1mlの(50μgの/ mlのカナマイシンを含む)にBL21(DE3)形質転換および接種から単一の孤立したコロニーを選択します。
- 37℃で3-4時間、220rpmで文化を振る℃に
- 1.5ミリリットルにラベルを付けユニークな構造の識別番号、細胞株、および日付とキャップチューブをねじ込みます。 50%グリセロール500μlの細胞培養液500μlを加え、数回転倒。直ちにドライアイス上または-80℃の冷凍庫中のグリセロールストックを格納します。
6。式のテスト
| 溶解Buffe R | バッファを洗う | 溶出緩衝液 |
| 50mMののNaH 2 PO 4、pH8.0で 300mMのNaClを 10 mMイミダゾール 1%のTween 20 2 mMのMgCl 2を 0.1μL/ mlのベンゾナーゼ 1 mg / mlのリゾチーム | 50mMののNaH 2 PO 4、pH8.0で 300mMのNaClを 20 mMイミダゾール 0.05%のTween 20 | 25mMトリス、pH8.0の 300mMのNaClを 250 mMイミダゾール 0.05%のTween 20 |
*追加ベンゾナーゼ、リゾチーム、溶解直前とプロテアーゼ阻害剤。
- ストリークはカナマイシン選択寒天の上にグリセロールストックからのサンプルと37℃で一晩インキュベート℃、
- 96ウェル丸底ブロックで非誘導前培養を開始し、新たに成長し、E.、0.5%グルコースを補っ接種1.2ミリリットルのTBブロス(50 mg / mlのカナマイシンを含む) 大腸菌は分離します。一晩37℃で220 rpmで振盪育つ
- 一晩増殖させた後、前培養の40μLでNovagen社一晩Expressシステム1(製造業者のプロトコールに従って)を補充TBブロス1.2mlのを(50 mg / mlのカナマイシンを含む)接種することによって誘導培養を開始します。
- 220 rpmで振盪、48時間20℃で小規模誘導文化を育てる。
- 15分間4,000 rpmで遠心分離によって収穫細胞は、処理前に少なくとも1時間-20℃で上清と店舗°Cをオフに注ぐ。
- 96ウェルブロックに、300μlの溶解緩衝液中で細胞ペレットを再懸濁する。
- 激しく室温で30分間振盪することによって機械的溶解、続いて30分間室温で溶解緩衝液で細胞をインキュベートする。
- 4℃で4,000 rpmで30分間遠心分離により粗溶解物を明確にする
- 96ウェル平底トレー(キアゲン)に明らかライセート(可溶性画分)を200μlを転送するために、マルチチャンネルピペットを使用してください。サンプルウェルを含むそれぞれについて、40μlのニッケル-NTA磁気ビーズ(キアゲン)を追加します。
- ゆっくりと16℃で1時間のためにロッカーにプレートを攪拌
- 磁性支柱プレート(キアゲン)上にプレートを置き、結合していない可溶性画分を削除します。のNi-NTAビーズのいずれかをピペットではないように注意してください。
- ポストプレートからプレートを取り外し、ゆっくり200μlの洗浄バッファーでビーズを再懸濁した。ペッティング上下30秒間、その後ポストプレートにバックプレートを配置。
- 洗浄バッファーを外し、ステップ6.12を繰り返します。
- ポストプレートからプレートを取り外し、TAをバインドのNi-NTAを溶出5分間50μlの溶出緩衝液で洗浄することによりrgetタンパク質である。
- 磁性支柱プレートに平らな底板を返し、新鮮な96ウェルV底プレートに溶出を転送します。
- 新鮮な96ウェルV底プレートに溶出液20μlを移し、1μlのULP1プロテアーゼと反応する。
- 製造業者のプロトコルに従って、ラボチップ90を用いてキャピラリー電気泳動によって溶出し、溶出+ Ulp1画分を分析する。
- あるいは、発現試験のすべての画分をSDS-PAGEにより分析することができる。
7。大規模発酵
- グリセロールストックからスクレープを取得するために滅菌ピペットチップを使用し、100ミリリットルのTBブロス(50 mg / mlのカナマイシンと)を接種し、一晩成長。 220rpmで振ると37℃
- 一晩増殖させた後、10mlの2 Lバッフル付きフラスコにEMDの自己誘導ソリューション(製造業者のプロトコールを参照)(50 mg / mlのカナマイシンを含む)とTBブロスの1 Lを接種し前培養を拡大前培養(1:100希釈)。
- 37℃で発泡1 L培養物を振る°C; 20に振盪インキュベーターの温度を変化°C 0.6(OD 600)の光学密度に達したとき。
- 一晩増殖させた後、式のテストのために各構築物の代表を10mlのアリコートを取る。
- 収穫セルは15分間5,000 rpmで遠心分離することにより貼り付け、上澄み捨てる。
- -80℃で貼り付ける凍結細胞℃、
タンパク質精製
バッファ:
| 溶解バッファー | (平衡)バッファ | バッファーB(溶出) | サイジングカラム緩衝液 |
| 25mMトリス、pH8.0の 200mMのNaClを 0.5%グリセロール 0.02%CHAPS 10 mMイミダゾール 1 mMのTCEP 50mMのアルギニン 5μlのベンゾナーゼ 100メートルGリゾチーム 3プロテアーゼ阻害剤錠(EDTAフリー) | 25mMトリス、pH8.0の 200mMのNaClを 10 mMイミダゾール 1 mMのTCEP 50mMのアルギニン 0.25%グリセロール | 25mMトリス、pH8.0の 200mMのNaClを 200 mMイミダゾール 1 mMのTCEP | 25mMトリス、pH8.0の 200mMのNaClを 1%グリセロール 1 mMのTCEP |
*直ちに溶解前に各150ミリリットルサンプルにベンゾナーゼ、リゾチーム、およびプロテアーゼ阻害剤錠を追加します。
8。細胞溶解
- 溶解緩衝液2 Lを作り、リゾチーム、プロテアーゼ阻害剤、錠剤またはベンゾナーゼ(各試料を溶解緩衝液150mlに溶解し、別途説明する)を追加しない。
- 融解と再懸濁細胞は1:5質量で溶解バッファーに貼り付ける:体積比を精力的に4で30分間攪拌℃とすることによりきれいなスパチュラを用いてビーカーの側面から緩いチャンクを破る。この間の期間は、Niと日記を準備SISバッファ
- 氷の上で、Misonix超音波処理(3分で70%の電力、2秒オン/ 1秒オフパルス)と過熱を防ぐため優しくスワールコンテナを用いて細胞を溶解する。将来の分析のための粗溶解物の小(200μl)をアリコートを保存します。
- 、4℃で35分間、18,000×gで遠心分離により粗溶解°Cを明確に上清を収集し、将来の分析のために小(200μl)をアリコートを保存します。それが確認されるまで4で保存ペレット°Cタンパク質が可溶性画分に溶解されています。
9。プリランプロテインメーカーのセットアップ
- プロテインメーカーでオンとソフトウェアが開き、機器を初期化します。
- 一度初期化された、サンプルの各々のためのガントリーの別の行に1 5.0ミリリットルGEヘルスケアHisTrap FFニッケルキレートカラム(ニッケルカラム)を添付してください。
- 各カラムを通して平衡化バッファーの3-4カラム容量(CV)を実行します。
- 総理平衡および溶出バッファーライン。
- Equilibr一度カラムを通してバッファを吸引して、列を食べました。
10。ニッケル1(NI1)カラム
- ストレージ·バッファを削除するには、20ミリリットルのMilli-Q水で各カラムを洗浄。 5ミリリットル緩衝液Bと平衡化のために25ミリリットルバッファを実行します。
- 2ミリリットル/分の速度でカラムに可溶化されたタンパク質を含むライセートをロードし、バッファAと15ミリリットルの洗浄でフォロー
- バッファ丁重に以下の比率でAとBとの段階勾配で結合タンパク質を溶出:5ミリリットル95:5、5ミリリットル60:40、10ミリリットル0:100。個別に各溶出画分を収集します。
- 分析:溶出画分、粗溶解物、明らかライセート、フロースルーをSDS-PAGEで。プール画分を、タンパク質を含有し、ほぼ存在するタンパク質の量を決定するために280を測定するための光度計を使用する。
11。 ULP1切断
- その後のゲル分析用NI1列プールの小アリコート(250μlの)を保つ。残りを持参10ミリリットルにNI1プールおよびHis-SMT親和性タグを削除するには、1μL/総タンパク5mgのでユビキチン様プロテアーゼ1(ULP1)を追加します。
- 4℃で撹拌プレート上で10 kDaの分子量カットオフ(MWCO)に4℃で4時間のためのバッファの2 Lに対してNI1プール+ ULP1を透析
- 透析後、NI1プールとNI1プールのSDS-PAGEを実行する+ ULP1 ULP1切断が成功したかどうかを判断する。
12。ニッケル2(をNi2)カラム
- 同じニッケルカラムに切断されたタンパク質をロードし、1ml /分の減少流量でステップ9.3を繰り返します。タグは、カラムに結合され、タグなし、標的タンパク質は、今では流れます切断。新鮮な容器にフロースルー収集。
- 3ミリリットルバッファーでニッケルカラムを洗浄すべてのHisタグと非特異的に結合したタンパク質を溶出するために緩衝液Bを5mlが続く。個別に各画分を収集します。
- をNi2のSDS-PAGE洗う、フロースルーとをNi2溶出画分ULP1裂を確認すると、その実行PRoteinは、フロースルー中に存在する。ほぼタンパク質の存在を決定するために280を測定するための光度計を使用する。
13。集中
- をNi2フロースルー(とをNi2溶出タンパク質が存在する場合)5ミリリットルにアミコンウルトラの10kDa MWCOの遠心チューブを集中。 4℃で4,000 rpmで10分間隔でスピン膜に沿って過集中からタンパク質を防ぐために各スピンの間にピペットで混ぜる。
14。サイズ排除クロマトグラフィー(SEC)
- 0.5ミリリットル/ AKTApurifierシステム(GEヘルスケア)に分の流速で200ミリリットルSECバッファーで平衡化することによりセファクリルS-100 10月30日GLカラム(GEヘルスケア)を設定します。
- 製造元の指示に従って、SECカラムで使用するための10ml superloopsを準備します。
- 5ミリリットル注射器を用いて、superloopsにサンプルをロードし、SECの実行を開始します。
- 少量FRを収集しながら、280nmでのUV吸光度トレースを監視するアクション。
- SDS-PAGE経由SEC画分を実行します。
- 最高強度のバンドを示すSEC画分をプール。
- プールされたSEC画分を集中。 13.1のステップを参照してください。
- 100μlのサンプルを、-80℃で液体窒素とストアのフラッシュ凍結℃までにアリコートタンパク質
晶析
15。タンパク質結晶化
- 選択肢の結晶画面80μlの(エメラルドバイオ)で96ウェルコンパクトジュニア結晶化プレートの各貯水池(エメラルドバイオ)を事前記入してください。
- 氷の上に2-20 mg / mlのとストアにバッファのサイジングでタンパク質を希釈します。
- 96ウェルのそれぞれにタンパク質と結晶化画面の0.4μlの0.4μLを分注し、クリスタルクリアなシールテープ(マンコ)でカバーしています。
- 解剖顕微鏡下で、今後数週間にわたって定期的にタンパク質結晶化のために確認しながら、16℃でプレートを保管してください。
16。クリスタル収穫
- 母液とエチレングリコールから凍結防止剤を作成します。標的タンパク質の結晶とよくをカバーする明確なテープをカットします。よく空には、対応する結晶化条件の1.6μlを添加し、20%エチレングリコールと80%の結晶化条件の最終濃度が得られるエチレングリコールの0.4μlのと組み合わせる。注:グリセリン、オイル、低分子量ポリエチレングリコール、および/または凍結保護剤の割合を変えることで、次のような別の凍結保護剤を試して結晶回折を最適化する。
- ふたが付いている液体窒素とカバーでいっぱいデュワーにおけるALS-スタイルパッククールダウン収穫前。
- マグネティッククリスタルワンド(ハンプトンリサーチ)よく溶液から直接すくうの結晶の大きさに一致する内径CryoLoopを配置することにより、結晶を収穫。
- すぐにCを凍結フラッシュするALS-スタイルパックに凍結防止剤その後水没に収穫されたクリスタルでCryoLoopを浸しrystal。結晶は、所望の数のために繰り返します。
17。クリスタルスクリーニング/データ収集
- 一度収穫ALSパックに磁気冷凍パックの蓋を置くためにパックの杖を使って完了です。曲がったトングと、パックを逆さまに反転させます。
- リガクACTORデュワーにパックを移し、パックにパックプッシャーをねじ込むと、表向きピンとデュワーでそれを残して蓋をオフにパンチ。
- JDirectorソフトウェアを使用して、次のパラメータの下画面の各クリスタル:0.5度に設定されたビームスリット50 mmとし、検出器の距離は、70度にイメージステップと、露光の長さは30秒に設定されています。
- あなたは最高の結晶と戦略は、データ収集のために何であるかを決定するためにJDirectorで撮影したテスト画像でMOSFLMを実行します。
- MOSFLMからあなたの結果に基づいて、完全なデータセットを収集します。
18。データ処理/構造決定
- データセットを処理するためにXDS / XSCALE 4を実行します。
- CCP4 Suiteソフトウェアを開きます。
- 利用可能な場合、高い相同性検索モデルを用いた分子置換ソリューションを計算するフェイザー5を実行します。このケースでは、検索モデル6としてPDBID 3CW4を使用していました。
- データセット内の収集、観測反射照らし合わせて分子モデルを絞り込むことREFMAC 7を実行します。最終的な解像度が高いシェルのオフに基づいて、以下のパラメータによって決定されるべきである:Rファクター> 50%、I /シグマ> 2、および完全性> 90%。
- 分子グラフィックソフトウェアCOOT 8で3次元電子密度モデルを構築します。
- PDB構造を堆積する前に構造の質が堆積するのに適していることを確認するMolProbity 9ソフトウェアでそれを検証します。
結果
次の結果が記述されたプロトコルの期待される結果を示しており、PB2、観測された結果の場合には
遺伝子Composerを用いて、インフルエンザウイルスポリメラーゼサブユニットPB2の5の全長標的アミノ酸配列( 図2)を設計した。 PB2シーケンスはバック翻訳多くの工学のステップ3を行い、E.での発現のために最適化されたコドン調和シーケンスになりましコリ。iPCRを製品( 図3b)、三〇から四構築物の総加入に成功し、図3aに示すように、PIPEクローニング3を用いたHis-Smtを融合タグはN末端6×有する変性pET28ベクター系10にクローニングした。クローニングワークフローの概要は図4に示されている。
クローニングの成功後、各構築物のマイクロスケールのタンパク質発現は、BL21(DE3) 大腸菌で試験した大腸菌細胞。細胞を220 rpmで振盪インキュベーター·セットに20℃で48時間Novagen社物エクスプレス1媒体(製造業者のプロトコルに従って)を補充したTB培地中で増殖させた。成長後、細胞をラボチップキャリパー90がキャピラリ電気泳動を用いて可溶性タンパク質の発現のために採取し、試験した。可溶性の標的タンパク質につながったと三〇から四PB2構文の14は、大規模な発酵に入った。各構築物の大規模な培養物を製造業者のプロトコルに従ってNovagen社のエクスプレス物1媒体を補充TB培地中で増殖させた。成長後、細胞を遠心分離によって回収し、-80℃で保存した。各培養物の大規模タンパク質の発現は、大規模精製を開始する前に、SDS-PAGE分析( 図5)を介して確認した。
タンパク質メーカーは14 PB2構築物の並列精製を実施するために使用された。アルの明確化ライセートL 14構築物は、ニッケルキレートカラムに通した。 SDS-PAGEによる標的タンパク質を含有する画分を決定した後、対応する画分を各試料についてプールし、それぞれの濃度は、280読み取りにより測定した。 6×彼-SMTタグの除去は、一晩透析し、第二ニッケルカラム、続いてULP1を添加することによって行った。彼-SMTタグ除去の確認は、SDS-PAGE( 図6)により行い、各サンプルは10 kDaのアミコンウルトラ遠心管で濃縮した。アミコンウルトラ遠心管を使用して濃縮した後、各サンプルは結晶純度を達成するためのサイジングカラムに実行されました。第2の濃度は、結晶化に必要なレベルにタンパク質濃度を増加させるために実施した。すべての14の構築物を成功裏に精製し、結晶化試験に入った。
結晶化は、以前にFRを解凍することによって開始されましたozenタンパク質。結晶化は16で気候制御室で行われた℃のシッティングドロップ蒸気拡散( 図7)のために特別に設計されたプレート(エメラルドバイオ)を使用。最初のスクリーニングは、4つのスパースマトリックススクリーニングを行った。JCSG +、パクト、ウィザードフル、および拡張ニューマン戦略に続くCryoFull(エメラルドバイオ)、。タンパク質溶液の0.4μlを、次に、96ウェルコンパクトジュニア結晶プレート(エメラルドバイオ社製)を用いて、対応するリザーバからcrystallantの0.4液(またはリザーバー溶液)と混合した。そのうち9 14精製試料の回折研究( 図8)に適した結晶を得た。社内回折データセットはオスミウムバリマックスHF光学系とサターン944 + CCD検出器を備えたリガクsuperbrightのFR-E +回転陽極X線発生装置を用いてCuのKα波長で結晶9構築物5( 図9に回収した)。各データセットは、XDS / XSCALE 4で処理した< SUP />、最終解像度にスケーリング。分子置換により構造を解決するための試みがCCP4スイート7からフェーザー 5を用いて行った。最終モデルはREFMAC 7の洗練とオオバン 11と手動再構築後に得られた。構造はMolProbity 9とジオメトリとフィットネスのために評価し、修正されました。PB2サブユニットの4構造の合計が決定されます( 図10)、PDBに堆積した。 図11は MTPPパイプラインの各段階での全体的な結果を示しています。

図1。エメラルドバイオでマルチターゲット並列処理SSGCID遺伝子ツー構造経路の概要。
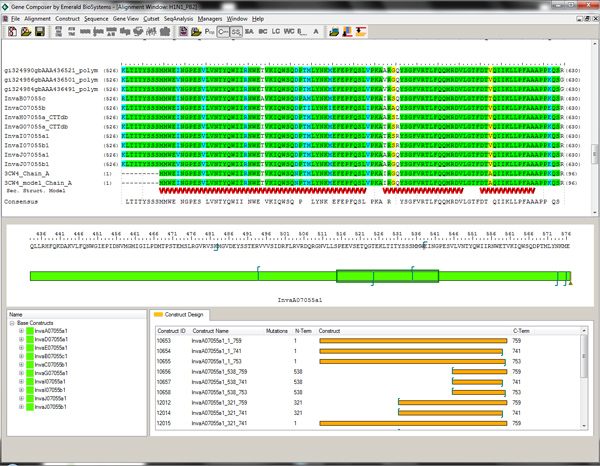
図2。アラインメントビューアとプロテイン遺伝子Composerソフトウェアの設計モジュールを構築します。アミノ酸ベースでは、ターゲットの構築緑色で示されています(中央のウィンドウ)と代替構文の構造誘導トランケーションは、金(下のウィンドウ)に表示されます。複数のインフルエンザウイルスのPB2配列のアラインメントは、配列および二次構造要素に比べて表示されますPDBID 3CW4からC末端ドメインである。ドメイン構造と二次構造要素の知識は、N末端 短縮が所望のアミノ酸残基を右クリックすることで、遺伝子作曲·デザイン·モジュール内で選択することができます。 大きい数字を表示するには、ここをクリックしてください 。
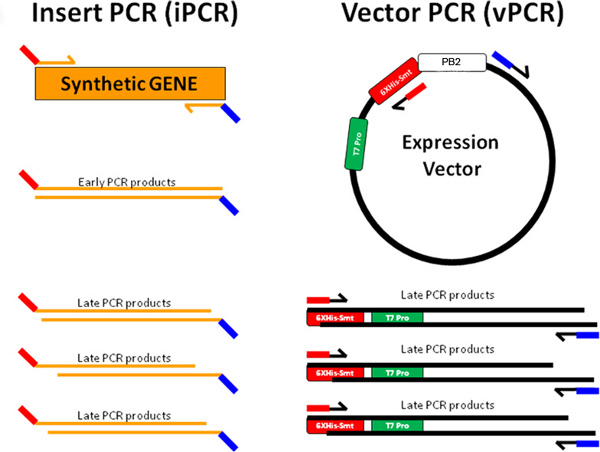
図3a。 PIPEクローニングは、合成遺伝子挿入(オレンジ)がインサートPCR材料を生成するように前方に設計された(赤オレンジ色の線)および逆方向(オレンジ青線)プライマーにより増幅される前記示されている。発現ベクターを逆(赤-黒線で増幅される)、前方(ブルーブラックライン)プライマーベクトルPCR材を生成する。末端配列のiPCRを製品はVPCR製品の末端配列(iPCRをの赤iPCRをVPCRの青の赤を補完VPCRの青を補完)に相補的である。これはiPCRをとVPCR製品はホストBL21(DE3)化学的にコンピに転換によって複製されたプラスミドを形成するためにアニールすることができます大腸菌細胞 。

図3b。 iPCRを生産のアガロースゲル分析成功iPCRを製品は堅牢なバンドで表現されながら、PB2サブユニットからTS。iPCRを障害が、かすかまたはべたつくバンドとして見られるかもしれない。 iPCRを製品の品質は、一般に、クローニングの成功と相関させることができる。分子量マーカーはキロダルトンである。図は、レイモンドら、2011年12から再生される。

図4。対象の遺伝子工学的工程PB2タンパク質は遺伝子Composerソフトウェアを用いて行った。操作された核酸配列は、ターゲットごとに確立された後、6-7代替タンパク質構築物は、それぞれのために設計された。遺伝子の設計およびクローニングの初期段階で、マルチターゲット並列処理はEの可溶性タンパク質を生産し実行可能な目標であった14そのうち34コンストラクトをもたらし大腸菌 。
再5 "SRC =" / files/ftp_upload/4225/4225fig5.jpg "/>
図5。堅牢なタンパク質発現(25.76 kDaで予想されるサイズ)、(レーン7)溶出されたタンパク質から6×彼-SMTタグの切断約50%の可溶性(レーン4)および約50%を示した大規模な発酵の代表的なSDS-PAGE分析。
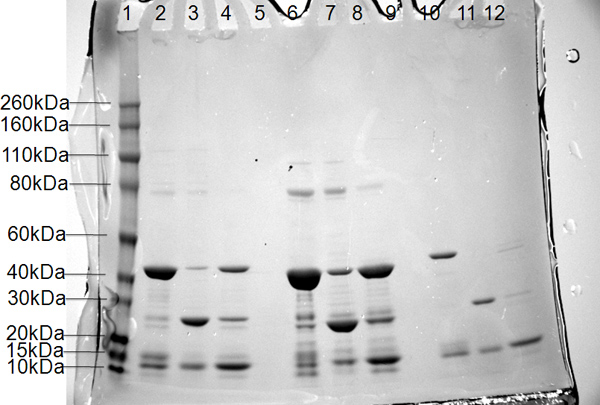
図6。ポリメラーゼPB2サブユニットの三構築物のためにSDS-PAGEの結果レーン1、分子量マーカー(kDaの左側に標識された);レーン2、図6、10、ニッケル1カラムからのプールされたタンパク質、レーン3,7、そして11、ニッケル2から切断された緩衝液中のタンパク質のフロースルー、レーン4,8、および12、ニッケル2からバッファBで6倍彼-SMTタグを除去した。
d/4225/4225fig7.jpg "/>
図7。シッティングドロップ法による蒸気拡散の概略。シッティングドロップ法タンパク質結晶化のためには、蒸気拡散に該当。この方法は、高濃度で同様の条件を含む大規模な貯水池と平衡するタンパク質と沈殿剤の精製されたサンプルを必要とする。水はタンパク質試料及び振替からリザーバーに気化したように、沈殿剤濃度は、タンパク質結晶化のための最適なレベルに増加します。

図8。インフルエンザウイルス株に由来するポリメラーゼPB2サブユニットのタンパク質結晶。

図9。からポリメラーゼPB2サブユニットのX線回折像インフルエンザウイルスの菌株。

図10。 4 PB2の構造の結晶学的非対称単位中の分子のリボン図。対応するPDBコードを持つ虹のパターンで色の二次構造(A)3K2V(A/Yokohama/2017/2003/H3N2)(b)の 3KHW(/メキシコ/ InDRE4487/2009/H1N1)(c)は3KC6(A/Vietnam/1203/2004/H5N1)(d)の 3L56(A/Vietnam/1203/2004/H5N1)。

図11。記載されている方法によってインフルエンザPB2ターゲットに対する結果分析。structur電子決定パイプラインは5つのステップで説明します。クローニング、溶解、精製、結晶化や構造決定を。
ディスカッション
マルチターゲット並列処理
構造に基づいたドラッグデザインは、創薬に重要な役割を果たしている。 SSGCIDはNIAIDカテゴリAC病原体から3次元タンパク質構造と科学界に提供することに専念している。広く利用可能な構造情報を作ることは、最終的に構造ベースの薬物設計を加速するのに役立つでしょう。
MTPPアプローチの最初の重要なステップは、構造設計です。各標的タンパク質の複数の構築物は、成功した構造決定を増加させ、ターンアラウンドの確率を増加させる。これは、いくつかのタンパク質構造は、パイプラインの各ステージ中に失敗することは避けられません。 PIPEクローニング法を実装すると、労働集約的な精製工程なしで96ウェルフォーマットの多くの構成要素の生成を可能にすることによってMTPP方法をサポートしています。同じ96ウェルフォーマット(キャリパーラボにおけるタンパク質発現を分析する能力をPIPEクローニングのペアリングチップ90)はさらに、全体の流れを迅速に。これらの方法の組み合わせは、大規模タンパク質産生および精製の成功を保証する可溶性タンパク質を生産する構築物の迅速な同定を可能にする。
MTPP高スループットの成功に重要な側面は、プロテインメーカー(米国特許第6818060、エメラルドバイオ)機器です。プロテインメーカーは、高スループットのタンパク質生産および関連構造ゲノムパイプラインの研究アプリケーションの効率性を高めるために特別に開発された24チャンネル並列液体クロマトグラフィーシステムです。タンパク質メーカーについて以前に記載されたプロトコルを使用して、利点は、単一のラインFPLCシステムと比較して明らかである。一人は、8時間の期間内に並列に48のターゲットにまで浄化することができる。対照的に、単一のラインFPLCシステムを使用して一人は、同じ期間内の最大4つのターゲットを浄化することができる。各ターゲットの純度の高いレベルプロテインメーカーで達成構造解析のために成長しているタンパク質結晶の後の成功の重要な要因である。
制限事項およびトラブルシューティング
X線結晶学によって三次元構造を解決する可溶性標的タンパク質を大量に得ることができないことである一つは多くの課題を有する多段努力である。溶解度の問題を克服するために実施することができる1つの戦略はE.などの代替の発現宿主の使用である大腸菌細胞は、いくつかの重要な真核翻訳後修飾を行うことができない。様々な酵母での発現は、これらの翻訳後修飾は、しばしば適切な代替である実行することができる昆虫および哺乳類細胞株。標的タンパク質は、時には発現が、標準的な溶解状態で完全に不溶性である。プロテインメーカーは、代替細胞溶解条件の迅速なテストのための貴重なリソースとなることができますとしてSmith らで説明。 2011 13。この戦略は、多くの場合、ターゲットは、パイプラインを介して動き続けることが必要である。任意の構造ゲノミクスのパイプラインでは、標準化されたプロトコルは、パイプラインと目標は、個々の最適化が必要になることがあり伝わってくるすべての目標のために適していないかもしれません。例えば、我々は、すべての凍結防止剤、20%エチレングリコールを使用することを選択した。この条件は適していないことのケースでは、代替の凍結保護剤又は濃度が試験される必要があるかもしれません。
個々のタンパク質標的のユニークな性質により、律速と構造を決定するステップは、予測不可能な結晶である。初期の疎行列の画面から最適化タンパク質結晶化のMTPPパイプラインオフセットは、一般的に低い成功率。市販のスパース行列の画面からヒット各初期結晶はさらにE-スクリーン·ビルダー(エメラルドバイオ)で最適化されています。最適化画面はARに設計されています緩衝液、塩、および添加剤の濃度を変える、初期結晶ヒットの状態をoundここ。成功した最適化画面は回折研究と構造決定に適した結晶を得ることができます。
アレルギー感染症研究所(NIAID)で出す構造ゲノミクスプログラムはエメラルドバイオと一緒にSSGCID(エメラルドバイオ、SeattleBiomed、ワシントン大学、パシフィックノースウェスト国立研究所)は他の3つの太平洋岸北西部機関に資金を提供しています。コンソーシアムの各メンバーは、NIAID構造ゲノミクスプログラムの目標を達成するために必要な最先端の技術を適用することで彼らの専門知識のために選ばれた。現在までに、SSGCIDは世界第7位貢献としてPDBランキングそれに461の構造を堆積しており、2011年に、最も生産。 SSGCIDのプロトコルと方法論は恩恵を受けてのつもりで用意されてい科学界と感染症の研究を永続。
開示事項
著者はエメラルドバイオ社の従業員である
謝辞
著者はSSGCIDコンソーシアムのすべてのメンバーに感謝したいと思います。 SSGCIDの目標の達成は、エメラルドバイオのすべてのチームメンバーの多大な努力によって実現されています。本研究では、アレルギーと国立感染症研究所からの連邦契約番号HHSN272200700057C、健康と保健社会福祉省の国立研究所で賄われていた。
資料
| Name | Company | Catalog Number | Comments |
| Primers | IDT | ||
| Genes | DNA 2.0 | ||
| TE buffer | Qiagen | provided in kit | |
| 96-well half skirt PCR plates | VWR | 10011-248 | |
| PFU Master Mix | |||
| 6X Orange Loading Dye | Fermentas | R0631 | |
| 10X TAE | Teknova | T0280 | |
| Agarose | Sigma-Aldrich | A9414-10G | |
| pET28 vector | |||
| 2-YT Broth | VWR | 101446-848 | |
| Kanamycin | Teknova | K2151 | |
| Restriction Enzymes | Fermentas | ||
| QIAquick Gel Extraction Kit | Qiagen | 28704 | |
| Top 10 chemically comp cells | Invitrogen | C4040-06 | |
| Disposable Troughs (Sterile, 25 ml) | VWR | 89094-662 | |
| Airpore covers (Rayon films for bio cultures) | VWR | 60941-086 | |
| 24-well blocks | VWR | 13503-188 | |
| QIAvac 96 | Qiagen | 19504 | |
| BL21(DE3) cells chemcomp (phageR) | NEB | C2527H | |
| 50% Glycerol | VWR | 100217-622 | |
| TB Media | Teknova | T7060 | |
| IPTG | Sigma-Aldrich | ||
| 1 M Tris pH 8.0 | Mediatech | 46-031-CM | |
| 5 M NaCl | Teknova | S0251 | |
| Glycerol | Aldrich | G7893-4L | |
| CHAPS | JT Baker | 4145-01 | |
| Imidazole | Sigma | 56749-1KG | |
| TCEP | Amresco | K831-10G | |
| L-arginine | Amresco | 0877-500G | |
| Benzonase | EMD | 70746-3 | |
| Lysozyme | USB | 1864525GM | |
| 10 kDa MWCO dialysis tubing | Thermo | 68100 | |
| Amicon Ultra 10 ka MWCO concentrators | Millipore | UFC901024 | |
| HisTrap FF columns | GE | 17-5255-01 | |
| HiTrap Chelating columns | GE | 17/0408-01 | |
| Compact Jr crystallization plates | Emerald Bio | EBS-XJR | |
| Crystalization screens | Emerald Bio | ||
| Ethylene Glycol 100% | Emerald Bio | EBS-250-EGLY | |
| Crystal Wand Magnetic Straight | Hampton Research | HR4-729 | |
| Mounted CryoLoop 0.1-0.2 mm | Hampton Research | HR4-955 | |
| ALS style puck | |||
| Puck Wand | |||
| Bent Tongs | |||
| Puck Pusher |
参考文献
- Lowen, A. C., Mubareka, S., Steel, J., Palese, P. Influenza virus transmission is dependent on relative humidity and temperature. PLoS Pathogens. 3 (10), 1470-1476 (2007).
- Yamada, S., et al. Biological and structural characterization of a host-adapting amino acid in influenza virus. PLoS Pathog. 6, e1001034 (2010).
- Lorimer, D., Raymond, A., Walchli, J., Mixon, M., Barrow, A., Wallace, E., Grice, R., Burgin, A., Gene Stewart, L. Composer: database software for protein construct design, codon engineering, and gene synthesis. BMC Biotechnol. 9, 36 (2009).
- Kabsch, W. Integration, scaling, space-group assignment and post-refinement. Acta Cryst. D. 66, 125-132 (2010).
- McCoy, A. J., Grosse-Kunstleve, R. W., Adams, P. D., Winn, M. D., Storoni, L. C., Read, R. J. J. Phaser crystallographic software. Appl. Cryst. 40, 658-674 (2007).
- Kuzuhara, T., Kise, D., et al. Structural basis of the influenza A virus RNA polymerase PB2 RNA-binding domain containing the pathogenicity-determinant lysine 627 residue. J. Biol. Chem. 284, 6855-6860 (2009).
- Murshudov, G. N., Skubàk, P., Lebedev, A. A., Pannu, N. S., Steiner, R. A., Nicholls, R. A., Winn, M. D., Long, F., Vagin, A. A. REFMAC5 for the refinement of macromolecular crystal structures. Acta Cryst. D. 67, 355-367 (2011).
- Cowtan, K. Recent developments in classical density modification. Acta Cryst. D. 66, 470-478 (2010).
- Chen, V. B., Arendall, W. B., Headd, J. J., Keedy, D. A., Immormino, R. M., Kapral, G. J., Murray, L. W., Richardson, J. S., Richardson, D. C. MolProbity: all-atom structure validation for macromolecular crystallography. Acta Cryst. D. 66, 12-21 (2010).
- Mossessova, E., Lima, C. D. Ulp1-SUMO crystal structure and genetic analysis reveal conserved interactions and a regulatory element essential for cell growth in yeast. Mol. Cell. 5, 865-876 (2000).
- Emsley, P., Lohkamp, B., Scott, W. G., Cowtan, K. Features and development of Coot. Acta Cryst. D. 66, 486-501 (2010).
- Raymond, A. C., Haffner, T. E., Ng, N., Lorimer, D., Staker, B. L., Stewart, L. J. Gene design, cloning and protein-expression methods for high-value targets at the Seattle Structural Genomics Center for Infectious Disease. Acta Cryst. F. 67, 992-997 (2011).
- Smith, E. R., Begley, D. W., Anderson, V., Raymond, A. C., Haffner, T. E., Robinson, J. I., Edwards, T. E., Duncan, N., Gerdts, C. J., Mixon, M. B., Nollert, P., Staker, B. L., Stewart, L. J. The Protein Maker: an automated system for high-throughput parallel purification. Acta Cryst. F. 67, 1015-1021 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved