このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ハイスループットSHAPEを使用したRNA二次構造予測
要約
プライマー伸長により解析し、高スループット選択2 'ヒドロキシルアシル化(SHAPE)数百から一塩基の解像度で数千ヌクレオチドRNAの構造を決定するための技術が、逆転写、キャピラリー電気泳動および二次構造予測ソフトウェアをプロービングする新規化学物質を利用しています。
要約
生物学的プロセスに関与してRNAの機能を理解することは、RNA構造の徹底的な知識が必要です。この終わりに向かって、またはSHAPE "プライマー伸長により解析のハイスループット選択2 'ヒドロキシルアシル化"と呼ばれる方法論は、一塩基分解能を持つRNAの二次構造を予測することができます。このアプローチは、優先的に水溶液中でのRNAの一本鎖またはフレキシブル領域をアシル化する化学プローブエージェントを利用しています。化学修飾部位が修飾されたRNAの逆転写によって検出され、この反応の生成物は、自動化されたキャピラリー電気泳動(CE)によって分画されている。逆転写酵素は、SHAPE試薬によって変更それらのRNAヌクレオチドで一時停止しているため、得られたcDNAライブラリーは、間接的に、単一の折られたRNAの文脈で足止めされているそれらのリボヌクレオチドをマッピング。 ShapeFinderソフトウェアを使用して、自動化されたCEによって生成された電気泳動図はNUに加工と変換される自身がRNAStructure(V5.3)予測アルゴリズムで使用される擬似エネルギー制約に変換されcleotide反応テーブル。 シリコ RNAの二次構造予測でプロービングSHAPEを組み合わせることにより得られる二次元RNA構造は単独の方法を用いて得られた構造体よりもはるかに正確であることが見出されている。
概要
スプライシング、翻訳、ウイルス複製および癌の調節に関与する触媒と非コーディングRNAの機能を理解するために、RNAの構造の詳細な知識が必要である1,2。残念なことに、RNA折りたたみの正確な予測は手ごわい課題を提示。クラシックプロービングエージェントは実験当たり100-150ヌクレオチドに限定され、このような毒性など多くの欠点から不完全なヌクレオチドカバレッジおよび/またはスループットに苦しむ。肉眼二次構造予測アルゴリズムは、効果的に精力的に類似した構造を区別することができないことに起因する不正確さに起因し、同様に不利である。特に大規模なRNAは、また、それらのコンフォメーションの柔軟性と、これらの技術に必要な高純度の試料を大量に起因するX線結晶構造解析と核磁気共鳴(NMR)分光法、などの3D構造決定の方法にしばしば難治性である。
HIGHスループットのSHAPEは、単一ヌクレオチド解像度で大RNAの構造をプロービングする効果的な、単純なアプローチを提供することによってこれらの問題の多くを解決する。また、SHAPEに使用される試薬は、取り扱いが安全で容易であり、試薬をプロービング他のほとんどの化学物質とは対照的に、すべての4つのリボヌクレオチドと反応する。これらの試薬 はまた、 インビボ可能コンテキスト (複数可)3 でその中にRNAを精査すること、細胞膜を貫通することができる。もともと週実験室で開発された4、SHAPEはRNAの多種多様〜9キロバイトHIV-1 RNAゲノムの5の完全な二次構造の決定が最も顕著な例を分析するために使用されている。 SHAPEを使用して他の注目すべき成果はビリオン関連HIV-1 RNAの3中のタンパク質結合部位を同定するだけでなく、感染ウイロイド6、人間の長い非コードRNA 7、酵母リボソーム8、リボスイッチ9の構造の解明を含む。 WHイルSHAPEプロトコルの元と高スループットの変化が他の場所で10-12公開されている、本研究は、蛍光オリゴヌクレオチドを用いてハイスループットSHAPEによるRNAの二次構造の決定の詳細な説明は、ベックマン·コールターCEQ 8000遺伝分析装置を提供し、 SHAPEfinderとRNAStructure(V5.3)ソフトウェア。以前に未発表の技術的な詳細とトラブルシューティングに関するアドバイスも含まれています。
SHAPEのバリエーション
SHAPEとそのバリエーションの本質は選択的に修正のサイトでかさばる付加物を生産し、2'-水酸基(2'-OH)リボースグループをアシル求電子無水物に水溶液中のRNAの露出です。この化学反応は、ベースが対になっている間、一本鎖ヌクレオチドは、これらの試薬による求電子攻撃を助長するコンホメーションを採用することが、より傾向があるように、地元のRNA構造力学を尋問の手段として、またはアーキテクチャコンストラクタainedヌクレオチドは10以下または非反応性である。付加体形成の部位が修飾されたRNA( "(+)"プライマー伸長反応)上の特定の部位にハイブリダイズし、蛍光または放射性標識されたプライマーから開始する逆転写によって検出される。逆転写酵素(RT)でアシル化リボヌクレオチドを通過しなかった場合には、cDNA産物のプールは、その長さ修飾部位と一致製造される。制御"( - )"試薬にさらされていないRNAを用いたプライマー伸長反応は、構造、非特異的なRNA鎖の破損等を RNAにそのDNA合成の早期終了( すなわち 、 "止まり")が原因ように制御されるかもしれない。化学修飾によって生成された一時停止と区別する。最後に、同じプライマーから開始する2ジデオキシ配列決定反応は電気泳動後のRNA一次配列と反応ヌクレオチドを相関させるためのマーカーとして使用される。
SHAPの元のアプリケーションで( - )、および2つ配列決定反応E、同一の32 P-末端標識プライマー(+)のために利用される。これらの反応の生成物は、5〜8%ポリアクリルアミドスラブゲルに隣接するウェルにロードし、ポリアクリルアミドゲル電気泳動(PAGE; 図1)変性により分画している。従来のSHAPEによって生成されたゲル画像の定量的分析は、SAFA、半自動フット解析ソフトウェア13を用いて行うことができる。
対照的に、高スループットSHAPEは、蛍光標識プライマーおよび自動化されたキャピラリー電気泳動を用いる。具体的には、調査中のRNAのそれぞれの地域のために、一般的な配列を有する4 DNAプライマーのセットが、異なる5 '蛍光標識は、合成または購入する必要があります。これらの異なる標識オリゴヌクレオチドは、プライム2 SHAPE反応と2配列決定反応、プールし、分留/自動キャピラリー電気泳動(CE)によって検出されたそのうちの製品に役立つ。 WHEREASのRNAの100〜150ヌクレオチドの反応性プロフィールは、独創的なアプローチを使用して、4つの一連の反応から得ることができる、高スループットSHAPEは、単一のプールされた試料300から600 3ヌクレオチドの分解能を可能にする。できるだけ多くの96としてサンプルが12連続したCEの実行( 図2)の過程で分別のために準備ができますが、同時に、分別することがあり、最大の反応8セットまで。また、SHAPEfinderソフトウェアは、プロセスに開発され、CEQや他の遺伝的分析装置から出てくるデータを分析し、より自動化されており、SAFA 13または他のゲル解析パッケージよりもはるかに少ないユーザーの介入を必要とする。
より高度な高スループットの方法論は、最近取得した次世代シーケンシング技術と一緒に構造特異的酵素ではなく、アルキル化試薬を使用PARS(RNA構造の並列解析)14とFragの-SEQ(断片シークエンシング)15、などの浮上しているINFORMATIORNA構造に関するnである。これらの技術の魅力にもかかわらず、プロービングヌクレアーゼに固有の多くの制限はまだ16のままです。これらの問題は、次世代のシーケンシングは、従来の形状で実施したのと同様の化学修飾およびRNAの逆転写によって先行されるSHAPEシークエンシング(配列SHAPE-)17プロトコルで回避することができる。これらのメソッドは、RNAの構造決定の未来を表すことが、次世代シーケンシングは非常に高価であり、多くの研究室が利用できないままであることを覚えておくことが重要です。
形状データ解析
遺伝分析装置に生成されたデータは、移動時間のインデックスに対してプロットされているキャピラリ検出器に流れる試料の蛍光強度(複数可)を含み、電気泳動の形で提示される。このプロットは、4つの蛍光チャネルに対応する重複のトレースの形をとるsが異なる蛍光を検出するために使用され、各トレースは、個々のcDNAの配列決定または製品に対応するピークで構成される。電気泳動データは、タブ区切りのテキストファイルとして遺伝アナライザからエクスポートしShapeFinder変換と解析ソフトウェア18にインポートされます。
ShapeFinderは、最初の移行時間とピークのボリュームを正確それぞれ、反応生成物のアイデンティティと数量を反映することを保証するためにデータを数学的な一連の変換を実行するために使用される。ピークは次いで、位置合わせて一体化し、その結果、一次RNA配列と一緒にまとめられている。 RNAの当該セグメントの "反応性プロフィールは、"(+)RNAヌクレオチドそれぞれに関連付けられた値からの制御値を減算し、後述するように、データを正規化することによって得られる。このプロファイルは、正規化された反応性valを変換しRNAstructure(V5.3)ソフトウェア19,20、にインポートされRNAの二次構造の折りたたみアルゴリズムに組み込まれている擬似エネルギー制約へのUE。このようにアルゴリズムをプロービングや製本化学物質を組み合わせることで大幅にどちらかの方法だけでは12,21に比べて構造予測の精度を向上させます。 RNAstructureの出力(V5.3)は、最低エネルギーの画像を含むRNA二次構造テキストドットブラケット表記でSHAPE反応性プロファイル(S)だけでなく、同じ構造を色分け。後者はその後、そのようなヴァルナ22およびPseudoViewer 23としてRNA二次構造のグラフィカル表示に専用のソフトウェアにエクスポートすることもできる。

図1。 SHAPE 4,10経由RNAの構造決定のフローチャート(A)RNA MAYは、生物学的試料から、またはインビトロ転写によって得られる。(B)ソースに応じて、RNAは、折り畳まれたまたは他の方法で処理され、SHAPE試薬で修飾されている。(C)逆転写反応は、蛍光または放射性標識プライマーを用いた(D)cDNA産物であるどちらキャピラリーまたはスラブゲルベースの電気泳動経由画し。(E)フラグメント分析。(F)のRNA構造予測。 より大きい数字を表示するには、ここをクリックしてください。
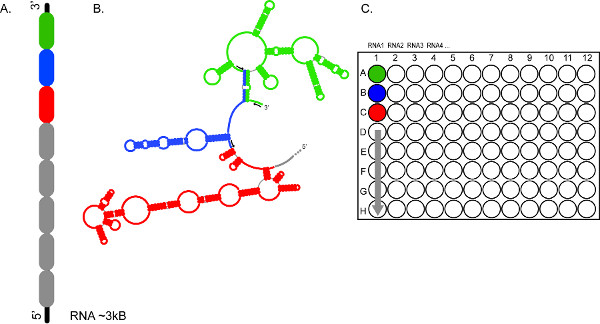
図2。 CEベースのSHAPEの高スループットの文字が急速複数RNAの解析、および/ または、同じRNAの複数のセグメントを許可します。() は(緑、青と赤のカラーコード化)(B)RNAの切片を蛍光プライマーの異なるセット(黒矢印)(C)を用いて独立して、プローブされたRNAは、300〜600ヌクレオチドの区間に分割することができる方法のセットを表し反応は〜3キロバイトRNA1のための完全なカバレッジを提供し、それぞれのウェルA1、B1、C1、 等にプールされ、ロードされています。 RNAは2、3、4等からの反応生成物は、同様に連続した電気泳動の実行で分画に調製することができる。 より大きい数字を表示するには、ここをクリックしてください。
プロトコル
RNAの3 '末端のプライマーの設計と拡張
ハイスループットSHAPEにより長いRNAを分析するために、プライマーハイブリダイゼーション部位一連のは、それらが(i)は〜300ヌクレオチドによって分離されるように選択されるべきであること、(ii)長さ20〜30ヌクレオチドであり、および(iii)そのRNA /これらのサイトにDNAをアニーリングにより生成さDNAハイブリッドは> 50℃での予想される融解温度を有するこのような判定を行うことがしばしば利用できないRNA構造のいくつかの予知を必要とするが、また、高度に構造化されると予測されるRNAのセグメントは、避けるべきである。これらの部位にハイブリダイズするDNAプライマーはそれらが安定した二量体又は鎖内の二次構造を形成することが期待されないように注意しながら、設計されるべきである。
一度設計し、プライマーセットのいずれかをご購入された(統合DNAテクノロジー、エイムズ、アイオワ州からなど )または24,25を合成する必要があります。プライマー5'-Cy5標識、Cy5.5のと、WellRedD2(ベックマン·コールター)とIRDye800(Lycor)/ WellRedD1(ベックマン·コールター)は最高のクロストークを最小限に抑えながら、優れた信号強度を提供し、ベックマン·コールター8000 CEQに適しています。標識されたオリゴヌクレオチドは、-20℃で小、10μMのアリコートで無期限に保存することができる、繰り返し凍結/融解サイクルを避ける。
このように設計したプライマーを用いることにより、任意の長さのほぼ全体RNAための形状データを得ることができる。プライマーは4をハイブリダイズさせることへのターミナルの拡張子( "構造カセット" など )しかし、3またはその付近の配列'RNAは3含むように操作されない限り、RNAの末端は、常に形にアクセスできません'を。
キャピラリー電気泳動を通じてRNA調製
生体試料からのRNAは、ハイスループットSHAPEのために利用することができるが、ここで与えられたプロトコルは、in vitro転写 RNAによって生成のために最適化されている。商業TRAこのようなMegaClear RNA精製カラム(Ambion社)と組み合わせて使用MegaShortScript(Ambion社)としてnscriptionキットはよく純粋なRNAを大量に生成するのに適している。 RNAは、-20℃〜-80℃の間のTEバッファに格納されるべきである最良の結果を得るには、RNAは両方変性および非変性ポリアクリルアミドゲル電気泳動によって均質で表示されるはずです。
1。 RNA折りたたみ
- 0.5ミリリットルのマイクロチューブに、水で18μlに、RNAの12ピコモルを希釈し、10Xの再生緩衝液2μlのを追加します。よく混ぜる。
- 85ヒート°C 4〜その後クール1分、℃で0.1の割合で℃/秒。
- 水を100μlと5Xフォールディングバッファー30μlのを追加します。
- 折り畳まれたRNAに応じて30〜60分間、37℃でインキュベートする。一般的に、長く、より構造化されたRNAのの Mg 2 +依存性の折り畳みは、より長いインキュベーション時間を必要とする。
- 2の0.5mlマイクロチューブのそれぞれに72μlのアリコートを移す:MODIFIED(+)とコントロール( - )。
2。 RNAの化学修飾
十分に特徴付け、求電子試薬は、SHAPEイサト酸無水物(IA)、N-methylisatoic無水物(NMIA)、1 -メチル-7 -ニトロイサト酸無水物(1M7)26、及びベンゾイルシアン(BzCN)27を含む。これらのうち、最も一般的に高スループットSHAPEに使用は1M7とNMIAあり、唯一後者(Life Technologies社)市販されている。試薬を変更して、最終濃度"は、単一のヒット"変形速度を得るために各RNAのために最適化する必要があり、溶液中で最もRNAは11分析されるRNAの領域に一度変更された状態、即ち 。この最適濃度は、試薬の濃度は、以下の2.1節の表に示される範囲(複数可)を横切って変化させた複数の反応を行うことによって決定することができる。容易に検出信号を生成する試薬の濃度を使用しながら、分長期および短期のDNA合成生成物( 例えば、 図3)との間の信号強度の差をimizing。
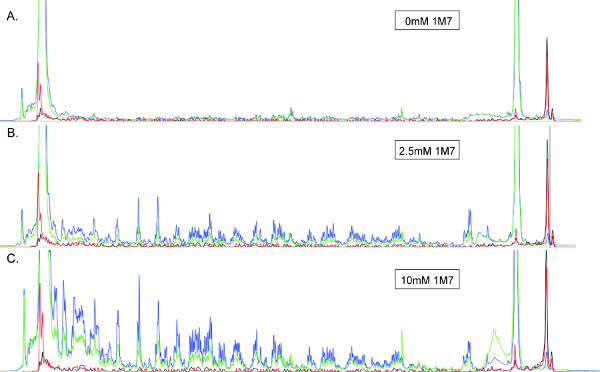
図3。 (A)は0(B)又は2.5mMの(C)10mMの1M7で処理し〜360ヌクレオチドRNAから製造SHAPEの電気泳動図。全ての電気泳動図は、同じスケールで表示されている。青、緑、赤と黒の痕跡反応生成物(+)に対応している(Cy5で)、( - )は、それぞれ反応生成物(Cy5.5の)、および2つの配列決定はしご(WellRed D2とIRDye800)。画像(B)を製造するために使用されるRNAは、トレース全体の最小の信号減衰(左)と、良好なピーク分解能と強度を示す、1M7の最適量で処理した。長さを読み、これらの条件の下で最大になる。対照的に、培地int型の不在ensity、うまく解決されたピークに()は1M7のサブ最適濃度を示唆している。逆に、(C)における明らかな信号減衰は、単一のヒット速度を示しているが観察されず、RNAオーバー変性である。このような場合には、RTはRNA鋳型の5 '末端に遭遇すると予想されない場合は特に、長さがリード最適ではないであろう。
- SHAPE試薬(NMIAまたは1M7)の10倍の株式を準備します。これが最良次いで所望の濃度を達成するためにDMSOを添加し、1.5mlマイクロ遠心チューブに試薬を少量添加することによって達成される重要:RNAと混合されるまでSHAPE試薬溶液は無水でなければなりません。室温で保存しデシケーター中でDMSO、及び周囲の水蒸気への曝露を最小限にするために、使用する直前にストック溶液を調製する。
試薬 最適な10倍濃度(DMSO中) までの時間試薬27の完全な分解 NMIA 10-100 mMの 〜20分 1M7 10-50 mMの 70秒
表1。電子試薬は、RNA修飾に使用されます。 - 修正された(+)とコントロールに8μlの10倍NMIA/1M7又は無水DMSOを追加( - )は、それぞれ、ミックス注:2.5 mMのにかかわらず、RNAの分析されて、NMIAと1M7両方に有効開始濃度であることが証明された。
- 50分(NMIA)または必要に応じて5分(1M7)、37℃でインキュベートする。
- 3 MのNaOAc液(pH 5.2)の8μL(0.1倍量)を追加することにより、沈殿RNA、8μlの100 mMのEDTA、冷エタノール及び1μlの10 mg / mlのグリコーゲンの240μL(3巻)。 4℃で30分間14,000 xgで遠心分離後、2時間冷蔵と冷たい70%エタノールで2回ペレットを洗浄注意:これは、することが重要ですこの電気泳動中に悪影響ピーク分解能に影響を与えることができるように、塩の共沈殿を最小限に抑えるために、冷蔵時、遠心時間と速度を最小限に抑える。
- マイクロピペットで上清を除去し、空気乾燥したペレットを室温で5分間。
- 10μlのTE緩衝液に沈殿したRNAを溶解し、室温で5分間インキュベートする。これは、十分に後進2転写反応のためのRNAを溶解させる。 -20℃ 注意で使用されていない部分を保管する:ペレットの機械的再懸濁は、通常は必要ありませんし、RNAを損傷する恐れがあります。
3。逆転写
このステップは、間接的RNAのヌクレオチドはSHAPE試薬によって変更された程度を識別するために使用される蛍光標識されたcDNA産物を生成する。形状は、スーパースクリプトIII(Invitrogen社)RTの性能は、他のすべてのテストされたのRTよりも優れていた、とこれに使用するために選択された酵素であるプロトコル。それぞれ、反応( - )とCy5のCy5.5ので標識されたオリゴヌクレオチドは、プライム(+)とするために使用されます。短いRNAのために、プライマーは、末端4 '3についての情報を得るために天然のRNAの末端伸長( 例えば 、 "構造カセット")'を3にハイブリダイズされる注目:この時点からCEを介して、サンプルをから保護されるべきである光。
- 調製(+)と( - )の0.5mlマイクロチューブ内の逆転写のためのサンプル。 、 - ( - )RT反応、5μlのコントロールRNAを混合するための()、(+)RT反応、修飾されたRNA(+)、6μlの水と1μlのCy5で標識したプライマー(10μM)を5μlを混合するための6μlの水、および1μlのCy5で標識したプライマー(10μM) 注意:ザルスタットPCRチューブ(REF 72.735.002)、このアプリケーションのために推奨されています。
- 場所は、サーマルサイクラーにチューブ、そして次のプログラム適用することにより、RNAと逆転写のために準備するプライマーアニーリング:85℃、1分、60℃、5分;35℃、5分、50℃、保持します。
- アニーリングステップ中に、実行される反応の数、プラス50%( - )反応、スケール4.5倍2(+)と2(用など )のために十分な2.5X RTミックスを調製する。 4μlの5X RTバッファー、1μlの100mMのDTT、1.5μlの水、1μLの10mMのdNTP、0.5μlのスーパースクリプトIII RT:一つの反応は、以下のように8μLを、必要とします。氷の上に保つ注意:5X RTバッファーとの100mM DTTはスーパースクリプトIII RTで提供されています。
- ( - )一度アニーリングミックスの温度は℃、(+)とに2.5X RTミックスの8μlの50に到達した反応を推奨:37〜RTミックスウォーム℃で5分間反応に追加する前に、 。
- 50℃で50分間インキュベートし、次にC、4℃まで冷却し、および/ または氷の上の場所を注:50分よりも長いためにRT反応のインキュベーションは、異常なcDNA産物になることがあります。
- 95℃に1μlの4 M NaOHおよび加熱を追加することで、RNAを加水分解 3分間。氷とその後にクール反応は2 M HClを2μLを追加することにより、それらを中和する重要:cDNA産物の悪い質分離のこのステップの結果の省略。
- 冷エタノール及び10 mg / mlのグリコーゲンを1μlの100mMのEDTA、1.5倍量の容量反応と3 MのNaOAcの0.1容量を追加することで、cDNAのを沈殿、0.1 - 組み合わせ(+)と()。 2時間冷蔵し、4℃で30分間14,000 xgで遠心冷たい70%エタノールで2回ペレットを洗浄注意:。遠心高いレートで、またはペレット(複数可)を再懸濁困難で長い期間の結果を得るために。
- 30分以上激しくボルテックス続いて10分間のために65℃に加熱することにより脱イオン化ホルムアミド40μlのでペレット化したcDNAを再懸濁注意:。ペレットは見えないかもしれません。信号または弱い信号は、次の電気泳動の欠如は、十分にこの段階でペレットを溶解するための失敗の結果であるかもしれません。
配列決定はしごは、データ処理中にヌクレオチド位置を決定するためのマーカーとして役立つ。これらは、USBサイクルシークエンシングキット(#78500)、RNAが検討されているのと同じ配列を有するDNA、およびWellRed D2またはD1/Lycor 800で標識したプライマーを用いて生成される。典型的には、この反応に用いられるDNAは、問題のRNAの転写のためのテンプレートとして使用することであろう。反応プロトコルがここに提示されていますが密接にキットのメーカーが推奨する、反応が数倍にスケールアップしていることに似ています。 DDAとDDTは、後述する反応において連鎖停止剤として使用されているが、ターミネーターの任意のペアを配列決定梯子を生成するために使用することができる。
- DDA終端ミックス40μlの、DNAテンプレートの5ピコモル、10Xシークエナーゼバッファ、WellRed D2標識プライマー、82μlに総量をもたらすシーケナーゼと水4.6μlの液10μlの4.6μLを混ぜる。最後シーケナーゼを追加します。 PREPA代わりにDDTとLICOR IR800標識プライマーを利用して、同様に第配列決定反応を再度。
- USB推奨条件を用いてPCR増幅に進み注意:鉱物油の添加は必要でも、プロトコル/加熱蓋を利用してサーマルサイクラーには推奨されません。
- 1 1.5ミリリットルマイクロチューブ(〜164μlの合計)に配列決定反応DDAとDDTを兼ね備えています。
- 次のように沈殿DNA:16μlの3 MのNaOAc液(pH 5.2)、16μlの100 mMのEDTA、1μlの10 mg / mlのグリコーゲン、および480μlの95%エタノールを追加します。よく混ぜ、4℃でインキュベート℃で30分間遠心分離し、30分間14,000 xgで4℃で
- 少なくとも30分間激しくボルテックス続いて10分間のために65℃に加熱することにより脱イオン化ホルムアミド100μlにペレット化したcDNAを再懸濁します。
5。キャピラリー電気泳動法による反応生成物の分画
キャピラリー電気泳動は、同時ができます単一のサンプルにプール4からのcDNA合成反応の生成物の分離。限り多くのサンプルが96としてシングルラン( 図2)の間に分画したかもしれないが8つのサンプルは、同時に画してもよい。
- 10プールされたシーケンシングはしごのμL、および96ウェルサンプルプレートへの転送と共にプールSHAPEサンプル40μlのを混在させる注意:これは、LPA-Iゲル含むベックマン·コールター試薬およびプレート(、バッファ、ミネラルオイル、サンプルを実行することが不可欠ですローディング溶液と試料とバッファプレートは)ベックマン·コールターCEQ 8000遺伝分析装置で使用される。
- プログラムおよびキャピラリー電気泳動装置を準備し、製造業者の指示に従って実行開始注:サンプルの最高の解像度については、以前に公開されたCAFAメソッドパラメータ28を使用しています。
すべての4つの電気泳動さtの各ピークのために理想的には、プライマーと強いワンストップピークの外側に、信号レースは直線範囲内でなければなりません。信号の緩やかなドロップオフが許容されます。しかし、時には、大きなピーク(停留所)コントロール反応においても明らかであり、これらは、後続のデータ処理を妨げることができる。これらのピークを生じさせる切り捨てられたcDNAは、天然の逆転写の間に障害物( 例えば、RNAの二次構造)、又はRNA分解の結果とすることができる。前者の場合では、そのようなベタインなどの添加剤は、RTの処理能力を向上させ、RTは/尚早終了を一時低下する可能性があります。
データ処理
ShapeFinderソフトウェアは、ユーザーが視覚化し変換CEトレースをし、SHAPE反応性プロファイル18にそれらを変換することができます。反応値が集計されると、それらは正規化され、二次構造モデルを生成し、洗練するRNAStructure(V5.3)にインポートされます。
6。 ShapeFinderソフトウェア
BaseFinderトレース処理プラットフォームの拡張フォーム29は 、ShapeFinderの公開されたバージョンは、非商業的な使用のために18自由に利用可能です。 ShapeFinderでデータ処理のための詳細な手順については、ソフトウェアのマニュアルに付属しています。
- 電気泳動は、それらが(i)の蛍光バックグラウンドを補正するために調整されShapeFinder、、にCEQから輸入されています(II)の蛍光強度の蛍光チャネル間のスペクトルの重なり、(iii)の移動度が異なるタグを付けたプライマーによって付与シフトは、(ⅳ)の違い逆転写の早期終結に起因する共通の異なる蛍光で標識された製品、および(v)信号減衰。
- ShapeFinderで "に合わせ、統合"ツールの "設定"機能が自動的に個々のピークにIDを割り当て、ユーザの入力と2シークエンスラダーによって定義されたRNA配列にこれを相関させる。初期割り当てが一般不完全ですが、エラーはの "変更"機能を使用して手動で補正することができ同じツール。最後に、 "フィット"機能、整列(+)と下の領域を計算します( - )反応のピークが、タブ区切りのテキストファイルに対応するヌクレオチド番号とともにこれらの反応値を集計。
注:データの分析は、SHAPEの正確性が重要である、いくつかの考慮事項を含む、この解析中に非常に重要です:
- 信号対雑音:信号対雑音比は、個々のピークは低反応性とポジションであっても容易に識別可能であるべきであるようなものでなければならない。 ShapeFinderは、データスムージングオプションが用意されていますが、この代替は、それがスキューその後の分析ができるように、非常に慎重に使用する必要があります。
- 分析領域:通常、信頼性の高いデータは、バックグラウンドノイズと区別することが困難なレベルの信号減衰としてプライマーの3 '末端から削除地域40-80 NTで開始および終了、cDNAを300-600 NT長いから入手することができます。マルチの使用PLEプライマーセットは、RNAの長いストレッチを分析するために必要とされる。この場合には、プライマーセットとの間の信頼できる信号の重複は30〜50ヌクレオチドの範囲であることが推奨される。逆転写酵素は、頻繁にRNAテンプレートの最後に到達する短いRNAは、上、注意がその信号対雑音比がDNA合成の強力な停止によって影響されるそれらのピークを除外するように注意しなければなりません。
- 信号減衰:信号減衰は実験と同様にRTの不完全な処理能力の間にRNA修飾の程度に関連しています。理想的には、分析対象のRNAの地域に比べて、シングルヒット動態は、長さを読んで最大化するために達成されるべきである。 Shapefinderは、信号減衰を補正するのに有効であるツールが含まれ、これは、解析にエラーを導入する傾向があるためしかし、 -単一のヒット動態が観察されていない場合は特に、信号減衰が最小である場合には、最良の( すなわち 、使用される場合の分布ピークはシングルヒット雌牛と一致しているチック)。最近では、信号の信号減衰を変換するための改良されたアルゴリズムは、30公開されていると、信号減衰が特定の実験で特別な関心事である場合、調査すべきである。
- 信号スケーリング。 ( - )トレース等しい間違いなく、制御プロファイルは最小限反応(+)との間でピーク強度がように形状データ処理における最も任意のステップにスケーリングされるべきである。あまりにも大幅に制御トレースをスケーリングする(下記データの正規化を参照)最初の四分位に負の反応値の豊富さになります。このイベントでは、スケールファクタはそれに応じて減少し、データが再統合されるべきである。
- ピークス割り当て。一般的には、ピークの割り当ての自動化されたバージョンではうまく動作します。プロセスが失敗した場合、しかしながら、ユーザは信号対雑音比が低い場合は特に、すべてのピークはソフトウェアによって認識されていることを確認することが肝要である。肩ピークは、例えば、常に検出されないと、G-リッチそれquencesは頻繁に圧縮されます。
7。データ正規化
RNAStructure(V5.3)ソフトウェアで使用される二次構造のアルゴリズムにヌクレオチド反応性プロファイルを組み込むこと、および/ または密接に関連RNAのプロファイルを比較するために、形状データが標準化された方法12に正規化する必要があります。これは、 "効果的な最大"反応性( すなわち 、反応値の最高8%の平均は、外れ値を除く)を決定するその後の計算から外れ値、(ⅱ)を除く(i)が含まれ、そして、すべての反応値を割ること(ⅲ)ノーマライゼーション次のように "効果的な最大値"、:
- アライメントと統合した後に生成されたタブ区切りのテキストファイルを開いて、Excelスプレッドシートにその内容をコピーします。このファイルの右端の列(RX.area-BG.area)はRNAの各ヌクレオチドのための計算された絶対的なSHAPEの反応値が含まれています。一番左の列は、RNA sequにこの反応を関連付けるリファレンス。
- 最初と三分の四分位( すなわち 、25日と75パーセンタイル)の値(RX.area-BG.area) "= QUARTILE( 配列 、 クォート )" Excel関数を使用して計算し、保存
- 計算して格納四分位の違いを"= QUARTILE( 配列 、3) - QUARTILE( 配列 、1)"
- 式"=(QUARTILE、 配列 、3)+1.5 *を((QUARTILE( 配列 、3) - QUARTILE( 配列 、1))"を使って"外れ値のカットオフ値"を計算し、格納します。すべての反応は、この値よりも大きくされている値その後の計算から除外する。
- (RX.area-BG.area)からの反応値をコピーして、隣接した、空の列に貼り付け、その後、最大の列の一番上にあるように、これらの値をソートします。
- 新しく作成された "並べ替え値欄"は、外れ値のカットオフ値以上の値を削除します。
- "並べ替え値coluに残っ反応値の最大8%の平均値を計算して格納MN "。この値は、"有効な最大 "反応。
- "正規化された反応度値"を取得するために、 "効果的な最大の"反応度値で(外れ値を含む)の各ヌクレオチドの(RX.area-BG.area)分別を分割。左に空の列を残して、空の列で、これらを保管してください。次に、テーブルの左側に塩基番号をコピーして、直接 "正規化された反応度値"の左に空の列に貼り付けます。
- テキストエディタにヌクレオチド位置正規化反応値のペアをコピーして貼り付ける。
- これらの可能性が高いRTはテンプレートの化学修飾以外の理由でcDNA合成時に一時停止の結果であるとして、-0.09( すなわち 、スペースを空白のままにします)以下の値を排除します。また、強力な一時停止が(として "合わせ、統合" ShapeFinderプロファイルの目視検査によって決定される)変更されていないテンプレートに観察されるヌクレオチドのための任意の反応性の値は、除外する必要があります。
- SARNAstructure(V5.3)ソフトウェアと構造解析で使用するため。形状拡張子を持つファイルをもらっ。
8。データのモデル化
RNAstructure(V5.3)ソフトウェアはSHAPE分析19に由来する擬似自由エネルギー制約を使用して実験的にサポートされているRNAの二次構造(複数可)を予測するために使用される。ソフトウェアは、最低エネルギー2D RNA構造のグラフィカルな表現と同様、ドットブラケット表記でこれらの構造のテキスト表現を提供します。後者は、出版品質の画像を生成するために、ユーザの嗜好、 例えば Pseudoviewer 23またはヴァルナ22のRNA構造ビューアにインポートすることができます。
注:RNAstructure(V5.3)ソフトウェアによって生成された構造を検討する際には注意が必要です。例えば、ソフトウェアは、シュードノットとキスループなどの三次相互作用を解決できない、またの不足かどうかを区別することができます特定の地域での反応性は、結合したタンパク質によってbasepairingまたは立体保護によるものです。決定的な構造モデルを提示したときに結果として、これらの要因は、個々の構造のために報告されたエネルギーと一緒に、考慮しなければならない。
結果
RNAは存在下でHIV-1回転応答エレメント(RRE)および3 '末端構造カセット4は 、それが 37℃で加熱、冷却、およびインキュベーションによって折られた後に、in vitro転写により線状プラスミドから調製した°Cを含有するのMgCl 2。 RNAはNMIAに露出した後、3 '末端構造カセットにハイブリダイズした5'-末端標識DNAプライマーから逆転写。得られたcDNAライブラリ...
ディスカッション
我々はここで高スループットSHAPEの詳細なプロトコルがあるどの規模のRNAのための一塩基の解像度に二次構造の決定を可能にする手法を提示する。また、二次構造の予測アルゴリズムを実験SHAPEデータを結合するだけではどちらの方法で可能であるよりも高精度にRNA二次元モデルの生成を容易にする。蛍光標識されたプライマーと自動CEの組み合わせは、単一の実験で長いRNA配列の解決を促進?...
開示事項
利害の衝突は宣言されていない。
謝辞
S. Lusvarghi、J. Sztuba-Solinska、KJ Purzycka、JWラウシュとSFJル·グライスは、国立癌研究所、国立衛生研究所、米国の学内研究プログラムによってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| REAGENTS | |||
| N-methylisatoic anhydride (NMIA) | Life technologies | M25 | Dissolve in anhydrous DMSO |
| 1-methyl-t-nitroisatoic anhydride (1M7) | see ref. 22 | ||
| Superscript III Reverse Transcriptase | Life technologies | 18080044 | 10,000 units |
| Thermo sequenase cycle sequencing kit | Affymetrix | 78500 | |
| Materials provided by the user | |||
| RNA of interest | 6 pmol per reaction (the limit of detection will be determined by the instrument) | ||
| Sets of four 5' labeled primers (Cy5, Cy5.5, WellRed D2 and WellRed D1/Licor IR800) | Primers are complementary to the RNA and are used in reverse transcription and sequencing reactions. The listed fluorophores are optimal for the Beckman Coulter 8000 CEQ. Primers may be purchased or synthesized in house. | ||
| DNA template | DNA is used for sequencing reactions, and must contain the sequence of the RNA being studied - including any 3'terminal extension, if present. Where applicable, it is often convenient to use the RNA transcription template. | ||
| Buffers | |||
| 10x RNA renaturation buffer | 100 mM Tris-HCl pH 8.0, 1 M KCl, 1 mM EDTA | ||
| 5X RNA folding buffer | 200 mM Tris-HCl pH 8.0, 25 mM MgCl2, 2.5 mM EDTA, 650 mM KCl. (This buffer might be changed depending on the case (e.g. pH, EDTA, Mg, RNase inhibitor) | ||
| 2.5X RT mix | 4 μl 5X buffer, 1 μl 100 mM DTT, 1.5 μl water,1 μl 10 mM dNTPs, 0.5 μl SuperScript III. Note that the 5X buffer and 100 mM DTT are provided with purchase of SuperScript III (Invitrogen). | ||
| GenomeLab Sample Loading Solution (Beckman Coulter) | Attention: Avoid multiple freeze-thaw cycles | ||
| EQUIPMENT | |||
| Capillary electrophoresis | Beckman | CEQ8000 | |
| Thermocycler | varies | ||
参考文献
- Scott, W. G., Martick, M., Chi, Y. I. Structure and function of regulatory RNA elements: ribozymes that regulate gene expression. Biochim. Biophys. Acta. 1789, 634-641 (2009).
- Moore, P. B., Steitz, T. A. The roles of RNA in the synthesis of protein. Cold Spring Harb. Perspect. Biol. 3, a003780 (2011).
- Wilkinson, K. A., et al. High-throughput SHAPE analysis reveals structures in HIV-1 genomic RNA strongly conserved across distinct biological states. Plos Biol. 6, 883-899 (2008).
- Merino, E. J., Wilkinson, K. A., Coughlan, J. L., Weeks, K. M. RNA structure analysis at single nucleotide resolution by selective 2 '-hydroxyl acylation and primer extension (SHAPE). J. Am. Chem. Soc. 127, 4223-4231 (2005).
- Watts, J. M., et al. Architecture and secondary structure of an entire HIV-1 RNA genome. Nature. 460, 711-716 (2009).
- Xu, W., Bolduc, F., Hong, N., Perreault, J. P. The use of a combination of computer-assisted structure prediction and SHAPE probing to elucidate the secondary structures of five viroids. Mol. Plant Pathol. , (2012).
- Novikova, I. V., Hennelly, S. P., Sanbonmatsu, K. Y. Structural architecture of the human long non-coding RNA, steroid receptor RNA activator. Nucleic Acids Res. 40, 5034-5051 (2012).
- Leshin, J. A., Heselpoth, R., Belew, A. T., Dinman, J. High-throughput structural analysis of yeast ribosomes using hSHAPE. RNA Biol. 8, 478-487 (2011).
- Souliere, M. F., Haller, A., Rieder, R., Micura, R. A powerful approach for the selection of 2-aminopurine substitution sites to investigate RNA folding. J. Am. Chem. Soc. 133, 16161-16167 (2011).
- Wilkinson, K. A., Merino, E. J., Weeks, K. M. Selective 2 '-hydroxyl acylation analyzed by primer extension (SHAPE): quantitative RNA structure analysis at single nucleotide resolution. Nat. Protoc. 1, 1610-1616 (2006).
- McGinnis, J. L., Duncan, C. D. S., Weeks, K. M. High-Throughput Shape and Hydroxyl Radical Analysis of Rna Structure and Ribonucleoprotein Assembly. Method Enzymol. 468, 67-89 (2009).
- Low, J. T., Weeks, K. M. SHAPE-directed RNA secondary structure prediction. Methods. 52, 150-158 (2010).
- Das, R., Laederach, A., Pearlman, S. M., Herschlag, D., Altman, R. B. S. A. F. A. Semi-automated footprinting analysis software for high-throughput quantification of nucleic acid footprinting experiments. Rna-a Publication of the Rna Society. 11, 344-354 (2005).
- Kertesz, M., et al. Genome-wide measurement of RNA secondary structure in yeast. Nature. 467, 103-107 (2010).
- Underwood, J. G., et al. FragSeq: transcriptome-wide RNA structure probing using high-throughput sequencing. Nat. Methods. 7, 995-1001 (2010).
- Mauger, D. M., Weeks, K. M. Toward global RNA structure analysis. Nat. Biotechnol. 28, 1178-1179 (2010).
- Lucks, J. B., et al. Multiplexed RNA structure characterization with selective 2'-hydroxyl acylation analyzed by primer extension sequencing (SHAPE-Seq). Proc. Natl. Acad. Sci. USA. 108, 11063-11068 (2011).
- Vasa, S. M., Guex, N., Wilkinson, K. A., Weeks, K. M., Giddings, M. C. ShapeFinder: a software system for high-throughput quantitative analysis of nucleic acid reactivity information resolved by capillary electrophoresis. RNA. 14, 1979-1990 (2008).
- Reuter, J. S., Mathews, D. H. RNAstructure: software for RNA secondary structure prediction and analysis. BMC Bioinformatics. 11, 129 (2010).
- Pang, P. S., Elazar, M., Pham, E. A., Glenn, J. S. Simplified RNA secondary structure mapping by automation of SHAPE data analysis. Nucleic Acids Res. 39, e151 (2011).
- Deigan, K. E., Li, T. W., Mathews, D. H., Weeks, K. M. Accurate SHAPE-directed RNA structure determination. Proc. Natl. Acad. Sci. USA. 106, 97-102 (2009).
- Darty, K., Denise, A., Ponty, Y. VARNA: Interactive drawing and editing of the RNA secondary structure. Bioinformatics. 25, 1974-1975 (2009).
- Byun, Y., Han, K. PseudoViewer: web application and web service for visualizing RNA pseudoknots and secondary structures. Nucleic Acids Res. 34, 416-422 (2006).
- Brown, T., Brown, D. J. S., Eckstein, F. . Oligonucleotides and Analogues - A Practical Approach. , 20 (1990).
- Legiewicz, M., et al. The RNA Transport Element of the Murine musD Retrotransposon Requires Long-range Intramolecular Interactions for Function. J. Biol. Chem. 285, 42097-42104 (2010).
- Steen, K., Siegfried, N. A., Weeks, K. M. Syntheis of 1-methyl-8-nitroisatoic anhydride (1M7). Protocol Exchange. , (2011).
- Mortimer, S. A., Weeks, K. M. A fast-acting reagent for accurate analysis of RNA secondary and tertiary structure by SHAPE chemistry. J. Am. Chem. Soc. 129, 4144-4145 (2007).
- Mitra, S., Shcherbakova, I. V., Altman, R. B., Brenowitz, M., Laederach, A. High-throughput single-nucleotide structural mapping by capillary automated footprinting analysis. Nucleic Acids Res. 36, e63 (2008).
- Giddings, M. C., Severin, J., Westphall, M., Wu, J., Smith, L. M. A software system for data analysis in automated DNA sequencing. Genome Res. 8, 644-665 (1998).
- Aviran, S., et al. Modeling and automation of sequencing-based characterization of RNA structure. Proc. Natl. Acad. Sci. USA. 108, 11069-11074 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved