Method Article
リソースの限定された設定のための手ごろな価格の、HIV-1薬剤耐性監視方法
要約
抗レトロウイルス療法(ART)を失敗して、HIV-1感染者のための薬剤耐性検査は、将来の治療法を導き、治療成績を向上させることができます。高いHIV感染率が、リソースが制限された設定の最適化、個々の人口健康の結果は、最終的には手頃な価格でアクセス可能な薬剤耐性遺伝子型解析と解釈の方法が必要になります。
要約
HIV-1薬剤耐性は真剣に抗レトロウイルス療法(ART)の有効性および耐衝撃性を損なう可能性を秘めている。サハラ以南のアフリカのアートプログラムは拡大を続けているため、ARTを個人が密接に薬剤耐性の出現を監視する必要があります。 ARTにすでに耐性ウイルス株の伝播を追跡するため送信された薬剤耐性の監視も重要である。ジェノタイピングは高価であり、洗練された実験室およびデータ管理インフラストラクチャを必要とするため、残念ながら、薬剤耐性検査は、まだ限られたリソースの設定中に容易にアクセスすることはできません。個人を管理し、送信された薬剤耐性を評価するためのオープンアクセスの遺伝子型薬剤耐性監視方法について説明する。この方法は、薬剤耐性パターンの解釈と個々の患者のレポートの生成のための無料のオープンソースソフトウェアを使用しています。遺伝子型決定プロトコルはavを有する血漿サンプルについて、95%以上の増幅率を有しているIRALロード>千のHIV-1 RNAコピー/ mlである。感度は、ウイルス負荷<千HIV-1 RNAコピー/ mlのために著しく低下する。ここで説明する方法は、米国食品医薬品局(FDA)、Viroseqジェノタイピング法によって承認されたテスト結果にHIV-1薬剤耐性の方法と比較して検証した。ここで説明する方法の限界は、それが自動化され、それは同じパネルからサブタイプAおよびBを増幅したものの、それはまた、サンプルの検証パネルからの循環組換え型のCRF02_AGを増幅するのに失敗したことがないという事実が挙げられる。
概要
南部アフリカにおけるHIVの流行は、特に南アフリカ2、3に、抗レトロウイルス療法(ART)を個人の付随指数関数的な増加と共に急速に1を進化されています。発症4を削減し、資源の限られた設定で、平均寿命(RLS)を5増加させるのに大規模な治療プログラムの疫学的影響に関するエビデンスとして蓄積し続け、ARTのカバレッジを向上させる努力が強化されます。テスト対象の予防ツール6、7のような処理の使用に向けたガイドラインの進化とプログラムを扱うには、治療上の個人の絶対数がさらに増加することを意味します。 ARTを個人の平均寿命は、HIV感染していない人口8のそれに近づくように多数の個体は、より長期間の技術になります。 HIVの薬剤耐性の発生および伝達がALWAを有するYS ART 9月12日の業績への脅威と考えられて。したがって、より多くの個人がARTに開始される、より厳格な監視と薬剤耐性の監視が必要とされている。
遺伝子型薬剤耐性(GRT)試験は、監視のためだけでなく、ARTを受ける個体におけるHIV-1薬剤耐性の両方の監視、先進国で成功裏に使用されている。これらの設定では、GRTは、HIV-1感染者のケアの連続体に統合されました。ほとんどの国際ガイドラインはレジメン小児患者は、母子感染(PMTCT)の防止にさらされ、大人やアート(第一ラインと第2ラインの)13〜15を失敗小児患者のためにGRTをお勧めしますが、その後16を感染させ、との設定で急性感染した個体間で送信される13-15薬剤耐性の高レベル。しかし、コスト、技術、およびインフラストラクチャの要件は、インプリメンが限られているRLSにおける薬剤耐性のモニタリングと同様のアプローチテーション。
南アフリカHIV治療とモニタリングのガイドラインは、現在のファーストラインは17をレジメン失敗個人のARTの選択を導くのにGRTを使用することをお勧めしません。個人は、主にウイルス学(HIV-1 RNAウイルス量)のパラメータに基づいて切り替えられる。しかし2012年に、南部アフリカのHIV臨床医協会は、第一南部アフリカARV薬剤耐性検査の指針18を発表した。これらのガイドラインは、第一ラインと第2ラインのアートを失敗し、すべての大人のためとPMTCT 18にさらされ、感染した幼児や子供のためにGRT検査をお勧めします。アフリカ南部19-29の透過薬剤耐性の高いレベルには現在の証拠がないので、GRTは、急性感染者のための18をお勧めしません。それは、これらの勧告のいくつかは、国家TREに時間をかけて統合されることが期待される地域の様々な国のガイドラインをatmentおよび監視。すでに、2013年南アフリカの治療ガイドラインに大人のためのセカンドラインに障害が発生した時には、子どもたち30の第一選択または第二選択のPIベースのレジメンの失敗時のGRTの勧告は今そこにある。
それは南アフリカの治療ガイドラインにGRTを組み込むことは、潜在的にコスト中立的になることが示されている。真の二次治療に切り替えられる必要がある患者を同定するGRTを用いて、比較的より高価な第一選択薬よりもセカンドラインレジメン薬のコストを考慮すると、プログラムに追加のコストを生じない。また、GRTも、失敗の他の理由を特定し、治療の選択肢を節約し、新たな耐性パターン31についての情報を生成することができます。したがって、さらに、アクセス、ANケアの質を向上させるために、薬剤耐性の監視方法のコストを低減することが必要であるDの成果。
ここでは、逆転写、ポリメラーゼ連鎖反応(PCR)および配列決定した( 表1)、ならびに薬剤耐性の解釈のために主にオープンソースソフトウェアのための一般的な(オープンソースの)プライマーを使用するように設計GRT法を提案する。臨床管理のために、プロトコルは、国の治療ガイドラインに密着した研究室で薬剤耐性のレポートの専門家の解釈を包括的レビューおよび報告の方法によって補完されている。プロトコルは、4つの異なる成分に分割され、1)HIVリボ核酸(RNA)の抽出、2)逆転写およびポリメラーゼ連鎖反応(PCR)は、ウイルス標的の増幅、3)配列決定クロマトグラム、アラインメントの分析のため4)バイオインフォマティクス法キュレーションとシーケンスデータの解釈。
プロトコル
1。エチレンジアミン四酢酸(EDTA)全血処理
注:血液は直ちにのコレクションを超えない24時間以上、4℃で保存することができた後に処理することができる。
- 生物学的安全キャビネットの中で作業し、EDTA全血試料が室温に到達させる。
- 各サンプルのサンプル識別(ID)と、ラベルに十分なクライオバイアル、貯蔵材料(プラズマ)、および日付。
- 1,000倍gで10分間サンプルを遠心分離する。遠心分離機を停止するためにブレーキを使用しないでください。血小板を含む赤血球、 - 非常に薄い層 - 血漿、白血球(軟膜):これは、3つの層(上から下へ)が得られます。
- 慎重に各クライオバイアルに上清(血漿)、アリコート500ミリリットルを吸引する。細胞層(軟膜)を崩壊させるか、いずれかのセルを転送しないように注意してください。
- RNA抽出のために必要になるまで-80℃で保存するか、すぐにRNA抽出に進みます。
- 正と負のプラズマ制御を含む、抽出されるサンプルのIDを抽出ワークシートを準備します。
- 各サンプルを抽出するために、検体ID、抽出日および「RNA」、1.5 mlの滅菌マイクロチューブにラベルを付ける。また、組み立てカラムとコレクションチューブだけでなく、抽出ワークシートから対応する番号の作業溶解液を含む2ミリリットルマイクロチューブにラベルを付ける。
- バイオセーフティキャビネットで働いて、働いて溶解液を、対応する2 mlのマイクロチューブに200μlのサンプルを追加します。
- ボルテックスウェル、室温で10分間インキュベートする。
- 10分後、チューブを軽く遠心する。
- チューブの各々に無水エタノール800ミリリットルを追加します。
- パルスボルテックスし、短時間遠心して混合する。
- 対応する列/コレクションチューブアセンブリに、この溶液の600μlのを転送します。 6000 xにおける遠心分離機1分間加えた。
- 新しいコレクションチューブにカラムを移し、ろ液を含む古いコレクションチューブを捨てる。さらに2回、上記のステップ2.8(以降)は繰り返します。
- 1分間6000×gで、各列及び遠心分離機に500μlの洗浄バッファーAW1を追加します。
- ろ液とコレクションチューブを廃棄し、新しいコレクションチューブにカラムを移す。
- 500μl加え、3分間、20,000×gでバッファAW2遠心分離した。ステップ2.11を繰り返します。
- さらに2分、20,000×gで新しいコレクションチューブに遠心分離する。
- 1.5 mlのマイクロ遠心チューブにろ液と場所の列を破棄します。
- あなたは、列の側に液体を分配しないことを確実にする列の真ん中に60μlのBuffer AVE(RNaseを含まない水)を追加します。
- 1分間室温でインキュベートする。
- 2分間6000×gで遠心分離する。
- 列を破棄し、1.5 mlのマイクロチューブにキャップ。
- サンプルは現在、残響の準備ができているSE転写。
- 試験は、最大6時間、4℃で、すぐにストアを行う場合。テストが遅延される場合は、その直後に-80℃時置く。注意:凍らせないで/ 3倍より多くのサンプルを解凍する。
3。逆転写用試薬の調製
- 開始する前に、正および負の対照を含む、プラズマ処理中のサンプルの数に必要な試薬の各々の体積を計算する。また、試薬コントロールを追加します。
- (上記の)ステップ3.1から計算されたボリュームを使用して、簡単にパルスボルテックス続くきれいな、無菌200μlのPCRチューブのデオキシリボヌクレオチド三リン酸(dNTPを)プライマーミックスを調製する。各サンプルは、表2を参照して、逆方向プライマーRT21とのdNTP 0.5μlを0.5μLを持つ必要があります。
- 200μlのPCRチューブへのdNTPプライマーミックスの分量1.0μL。
- μ1を添加することによって、逆転写酵素(RT)酵素ミックスを調製する; 10倍の逆転写緩衝液のL、ボルテックスし、短時間遠心分離して、その後の滅菌チューブに0.1M DTTおよび25mMのMgCl 2を 2μLの1μL、 表3を参照してください。
- 0.5μLRNアーゼOUTとスーパースクリプトIIIは酵素ミックスチューブに逆転写酵素のそれぞれを追加し混合する優しくチューブをタップします。
- 寒いブロック上のdNTP、プライマーミックスと酵素ミックスでチューブを保持し、RNAステーションに移動します。
4。逆転写
- 簡単に混在させるボルテックス続くのdNTP-プライマーミックスチューブにRNAサンプルの6を添加する。
- RNAを添加した後、冷たいブロックや氷の上でチューブを混ぜたdNTP /プライマー/ RNAミックスおよびRT酵素の両方を用いてPCRの部屋に移動します。
- 簡単に言うと(ステップ4.2から)のdNTP /プライマー/ RNAミックスチューブを遠心分離機サーマルサイクラーに入れます。
- RNAを変性させるために5分間65℃で加熱する。
- 4℃まで急冷、2分間保持分。
- 4℃でしばらくまだサーマルサイクラーを一時停止、チューブを取り出します。
- 寒いブロックにチューブを維持しながら、迅速酵素ミックスの5μlを加える。
- チューブをタップして穏やかに混合し、簡単にチューブを遠心し、サーマルサイクラーに戻ります。
- 逆転写を停止するために5分間85℃で酵素の変性に続いて、RNAを逆転写するために60分間50℃でチューブを保持します。
- 37℃に冷却とすぐに温度が37℃に到達し、一時停止し、サーモサイからのチューブを取る。
- すぐにチューブにリボヌクレアーゼHの0.5μlを添加し、サーマルサイクラーに戻ります。
- 20分間37℃で保持した後、4℃に冷却
- 相補的DNA(cDNA)を直ちに使用することができ、または、必要になるまで-20℃より低温で保存することができる。しかしながら、cDNAの長期保存は-80℃であるべきである
5。 PCR用試薬の調製
- BefoスタートRE、処理中のサンプルおよびコントロールの数に必要な試薬のそれぞれのボリュームを計算する。 3つのコントロール(ポジティブ、ネガティブ、および試薬)に加えて、また、PCRコントロール(HIV DNA)を追加することができます。第一および第二ラウンドのPCRミックスを同時に調製することができ、必要になるまで第2マスターミックスを-20℃で保存した。混合物を約8時間保存することができる。
- 表4および渦に示すように、2.5μlの10Xバッファー、1.0μLのMgCl 2、0.5μLのdNTP、プライマーのそれぞれの0.25μLを18.4μlの水を加える。
- プラチナTaqポリメラーゼ(5U/μl)の0.1μl加え、静かにそれをタップすることで、チューブを混ぜる。
- 200μlのPCRチューブにマスターミックスのアリコート23μlの。
- 寒さのブロックまたはPCR室へアイス移動中のマスターミックスチューブ付き。
6。ネステッドPCR
- 第一回目のPCR masteの23μlに2μlのcDNAを追加Rミックス。
- 、2分間94℃で2分間、30秒、95℃で30サイクル、58℃で20秒間、および72℃:チューブの蓋を閉め、サーマルサイクラーにサンプルを入れて、以下のPCRサイクル条件を使用する図1に示すように10分間72℃で、最終的な拡張子が続く。
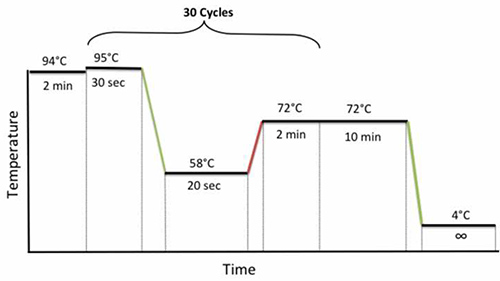
図1。ネステッドPCRサイクル条件。 拡大画像を表示するにはここをクリックしてください。
- 第二回目のPCRの段階に進むか、後の段階で必要になるまで-20℃より低温での第一回目のPCR産物を格納します。
- 第二回目のPCRのために、第二回目のPCRマスターミックスANの23μlに第一回目のPCR産物の2μLを追加図1に同じPCRプログラムを使用してDを
7。ゲル電気泳動
- ゲル製剤
- 250ミリリットルのガラスフラスコにアガロースタブレットの0.5グラムを加え、フラスコに1×TBEバッファーの50ミリリットルを追加します。
- 沸騰するまで電子レンジでの加熱、完全に可溶化するまで(約30秒毎)、頻繁に渦巻く。ホットフラスコを把握するため、シリコングリップ、シリコーンオーブン手袋を使用してください。アガロース溶液は、このプロセスを監視するように密接に非常に容易にフラスコから沸騰させることができる。
- 約10分間室温で冷却する。
- 適切なサイズの櫛を含むゲルキャスティングトレイにアガロース注ぐ、ゲルは、約20〜30分を使用する準備ができました。
- 電気室でゲルを置き、メーカーが推奨するように実行します。
- ゲル電気泳動および可視化。
- 使用前に10秒間ボルテックス小説ジュース。
- Dの5μlの小説ジュースの1μLを希釈NAのサンプルと混合します。
- 分子量マーカーの3μLを有する新規ジュースの3μLを希釈し、混合する。
- のセクション(上)7.2.2および7.2.3からミックスをロードし、PCR増幅を評価するために40分間100 Vおよび400ミリアンペアでゲルを実行します。
- 陽性増幅は1,315 bpの断片は 、 図2のようにUV光下で可視化することができる。
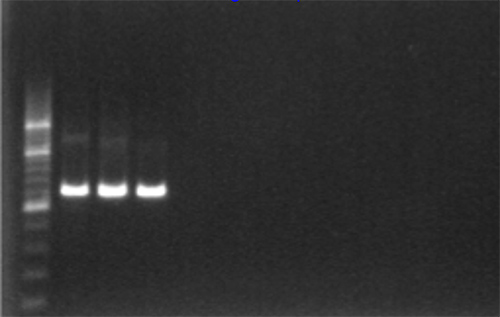
図2。 1%アガロースゲル電気泳動および200 bpラダーを用いたPCR増幅のゲル確認。 拡大画像を表示するにはここをクリックしてください。- このため、汚染がないことを示す、負電圧および試薬コントロールでは増幅があってはならない。
8。 PCR産物クリーンアップ
- シークエンシング反応の準備のために、正の第二ラウンドのPCR産物をPureLink PCR精製キットを使用してクリーンアップされる。
- PCR産物をピペットミックス20μlに結合緩衝高遮断(B3)の作業の80を添加する。
- コレクションチューブにスピンカラムに結合緩衝液と混合したサンプルを追加します。
- 1分間万×gでカラムを遠心。新しいコレクションチューブにカラムを移す。
- エタノールで洗浄バッファー650μLでカラムを洗浄します。
- 1分間万×gでカラムを遠心。新しいコレクションチューブにカラムを移す。
- 残留洗浄バッファーを除去するために2〜3分間最高速度でカラムを遠心。
- キットに付属のきれいな1.7ミリリットルの溶出チューブにスピンカラムを配置します。
- 列の中央に溶出バッファー40μlを添加し、室温TEMPERATURでカラムをインキュベート1分間のE。
- 2分(> 10,000×g)で、最高速度でカラムを遠心。
- 溶出チューブを配列決定のために準備ができてあなたの精製PCR産物が含まれています。列を破棄する。
- ナノドロップを使用したDNAの濃度と品質を決定します。
- まだ社内シーケンシング設備が利用できない場合、精製されたPCR産物は、この段階で商業的シーケンシング研究室に送ることができる。
9。シークエンシング反応
- PCR産物は、ビッグダイターミネーターキットバージョン各試料について3.1及び4のプライマー(二フォワードおよびリバース二つ)を用いて配列決定する。プライマー配列を表2に示す。そのため、シークエンシングの実行後に、各サンプルは、コンティグに組み立てられる4つのシーケンスを持つことになります。
- 4つのプライマーの各々について、表5に示されるように、配列決定反応を設定する。
- 使用前にボルテックスすることにより、配列決定バッファーおよびプライマーを混ぜる。
- ミックス大きな色素クエンシングを添加する前に水、緩衝液およびプライマー。ボルテックスで混和する。
- 優しくチューブを反転させるか、軽くタップすることで、大きな色素シングミックスを追加した後にマスターミックスを混ぜる。
- 96ウェル光学プレートにマスターミックスのアリコート9μL。
- 図3の下に示されるように、24サンプル/プレートを実行するために、プレートを設置。
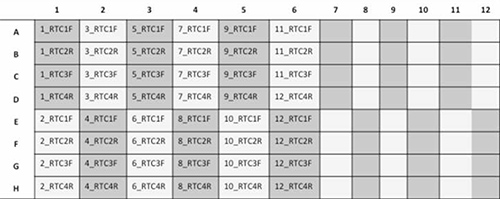
図3。 4プライマーそれぞれ(RTC1F、RTC2R、RTC3F、およびRTC4R)を用いて配列決定された12の患者サンプルを用いて96ウェルプレートの図式表現。 拡大画像を表示するにはここをクリックしてください。
- DNAサンプル(〜20〜40 NG)の1.0μl加え、プレートをプレートカバーNアルミカバーを接着した後、軽く混ぜる。
- 1分間3000×gで遠心分離する。アルミのカバーを外し、ゴム製のシールマットを追加します。
- サーモサイクラー上でプレートを配置し、 図4に示されている以下のサイクリングプログラムを実行します。
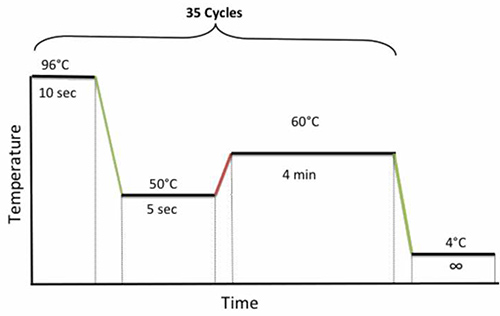
図4。シークエンシングのためのPCRサイクル条件 。 拡大画像を表示するにはここをクリックしてください。
- PCRが終了すると、すぐに配列決定産物をクリーンアップ。
10。シークエンシングのクリーンアップ
- 各配列決定反応のために、50μlの無水エタノールと5μlの3 M酢酸ナトリウムを混合します。
- マルチチャンネルピペットを使用して、55をプラス#956;各ウェルに酢酸ナトリウム/エタノール溶液をL。
- 各ウェルが適切に密閉されていることを確認して、接着剤、アルミカバーでウェルを密閉する。
- 20分間3000×gで遠心分離する。
- 20分後、カバーを取り外して(これは、ペレットを取り除くよう上清を取り除くために強打しないでください!)折り畳まれたラボの組織上に、1滑らかな動きで、プレートを反転。
- 2分間150×gで、同じ組織に反転プレートを遠心分離する。
- すぐに150μLの冷70%エタノールを加える。この段階でエタノールの添加を遅らせないでください。
- 同じ接着アルミカバーと渦とのシール。
- 5分間3000×gで遠心分離する。
- 1分間150×gで反転し、新たに折り畳ま組織や遠心分離機にプレートを反転します。
- 遠心分離した後、サーマルサイクラーで明らかになった場所と2分間50℃で、それを乾燥させます。
- プレートが乾燥したら、準備はWIを続行するまで、-20˚Cで箔とストアにラップ、接着箔カバーでそれを封印シーケンシング電気泳動番目。
- 配列に対する準備ができて、10ミリリットルのHi-ジホルムアミドで洗浄さシーケンシング製品を溶解する際に、変性させ、電気泳動用のロード。
11。バイオインフォマティクス
- シーケンスアセンブリ
- プログラムGeneiousを起動します。
- シーケンスを保存するために作業フォルダを作成します。
- インポートツールを使用して、作業フォルダに配列決定装置によって生成されたABIファイルをインポートします。 Geneiousは、インポートされた各シーケンスのためのパーセントの品質スコアを割り当てます。
- それをダブルクリックして品質スコア> 70%とのオープンシーケンス。
- 各ファイルには、新しいウィンドウで開く必要があります。ソフトウェアは、水色のバーを使用してシーケンスの質のクロマトグラムの各ヌクレオチド位置での品質を示します。ベースコールの品質が良く、バーが高い。
- カーソルを使用して、低品質で通常は両端を除外シーケンスの中間部分を、選択します。
- 良質の配列を有する領域を抽出するための抽出ボタンをクリックしてください。
- 各サンプルの4つのすべての抽出された配列を選択し、参照配列に対してそれらを組み立てる。
- 正しい読み枠内にあることを確認するために組み立て順序を点検します。 PQITLW:正しい読み枠内にある場合は、プロテアーゼの初めには、次のアミノ酸で開始する必要があります。 RTの始まりはPISPIEで始まります。
- 300番目のRTコドンにPRの先頭をカバーするコンティグ領域を抽出。このプロセスの間に、また、挿入または欠失を確認してください。
- 、抽出されたコンティグのコンセンサス配列を通過する任意の曖昧性を特定し、ベースコールの質(対称性、高さ、背景と隣接領域の肩)を検査することにより混合塩基との位置を確認します。
- コンセンサス配列を選択し、4種類のプライマーとラベからコンセンサス配列の別のファイルを作成するために、抽出ボタンをクリックしてくださいL適切に。
- コンピュータ上のバックアップ保存フォルダやネットワークフォルダにシーケンスを書き出す。
- シーケンス品質評価(HIVDB)
- でHIVDBプログラムを用いて配列を解析http://hivdb.stanford.edu 。
- 要約データ中の欠失および挿入をチェックし、シーケンスは、すべての99のプロテアーゼ(PR)のコドンと第一300 RTコドンをカバーしていることを確認してください。
- そのような終止コドン、フレームシフト、あいまいな位置や異常な残基の両方のPRおよびRT地域での品質保証(QA)の問題を強調したかどうかを確認します。
- シング品質管理
- 前回の実行からローカル配列データベースに対して新しいシーケンスを爆破。
- 新しいシーケンスは、データベース内の任意のシーケンスと同様> 97%である場合、プロトコルのすべての段階では、配列解析から始まるとバックensurにRNA抽出に行く、見直されるべきであるEなしミックスアップ(サンプルの切り替え、偽装)や汚染がなかったこと。
- 何も問題が識別されない場合は、RNA抽出段階から、新旧両方のサンプルの分析を繰り返します。
- 配列はまだ> 97%類似している場合、個人間のいずれかの疫学的連携のために評価するために患者の病歴を確認します。
- 系統発生解析
- GeneiousでのClustalWプログラムを使用して、データベースからすべての配列を整列。
- 手動で位置がずれた配列、欠失および挿入のための位置合わせを確認し、それに応じて編集します。
- PHYML、GeneiousツリービルダーまたはGeneious内の他のツリービルダーを使用して系統樹を構築します。
- 短い枝長のサンプルのための木を調べる。
- 汚染の可能性のための短い枝の長さを有するサンプルを確認してください。
12。 REGAのDB情報
- シーケンスのアップロード
- REGADにログインユニークなユーザー名とパスワードを使用して、B。
- ドロップダウンメニューで、患者IDの下で、「で始まる」を選択します。
- 患者IDを追加し、その遺伝子型アップロードする個人を選択します。
- 左側のメニューで、「ウイルス分離株」を選択します。
- ウイルス分離株の下にあるオプションから「追加」を選択します。
- サンプル日、サンプルID、シーケンス番号とシーケンスの日付を入力してください。
- 選択して「ファイルを選択」してからアップロードするシーケンスのFASTAファイルに移動します。
- アップロードするFASTAファイルを選択した後、アップロードをクリックしてください。
- アップロードされたシーケンスは、シーケンスを識別し、日付の下にヌクレオチドボックスに表示されたら、ウィンドウの右下にある[OK]ボタンをクリックします。
- ボタンタンパク質をクリックして、PRまたはRTのどちらかを選択することによって、PRおよびRTタンパク質アラインメントをチェックします。
- 抵抗ボタンをクリックして、薬剤耐性変異を確認してください。これは与えますもし3つのアルゴリズムからの抵抗プロファイル:ANRS、スタンフォードHIVDBとRegaDB。
- REGAを使用してレポートの生成
- 独自のユーザー名とパスワードを使用してRegaDBにログインします。
- ドロップダウンメニューで、患者IDの下で、「で始まる」を選択します。
- 患者IDを追加し、そのレポートを生成するか、個々に選択します。
- 右側にあるメニューで、ウイルス分離株を選択します。
- ウイルス分離株の下にあるオプションから「表示」をクリックしてください。
- レポートを作成するためのウイルス分離株をダブルクリックします。
- ウイルス分離株ウィンドウで、ウイルス分離株レポート]タブをクリックします。
- ドロップダウンメニューから遺伝子型の解釈のためのアルゴリズムを選択してから、使用するレポートテンプレートを選択します。
- アルゴリズムとテンプレートを選択したら、「生成」ボタンをクリックしてください。
- 生成されたRTFドキュメントをダウンロードしてください。
- RTFは何開くWord文書としてcument。
- 治療歴のグラフのサイズを変更。
- チャートの後には、「カルテと抵抗の解釈」を追加します。
- 抵抗テーブル及びカルテのデータを用いて、患者の治療歴から出発し、患者の耐性プロファイルの記述を追加し、ウイルス分離株が耐性である薬物れる。また、患者のウイルス量の説明と、チャートからのCD4 +細胞数のプロファイルを追加します。
- レビューと今後の患者の管理に関する推奨事項については、感染症(ID)の専門家にレポートを送信します。このプロセスは、非常に重要な品質保証ステージである。最終報告書は、患者を管理する臨床医に、すべての推奨事項と、送信される前に治療の歴史の中で遺伝子型またはinconsistencesにエラーがあると、ウイルス学的および免疫学的プロファイルが特定され、確認することができます。
結果
検証方法は、以前に報告された方法20の修正だった。 FDAによって承認されているViroseq遺伝子型決定法は、検証中の参照方法として使用した。エイズの研究とウイルス性肝炎(ANRS)フランス国立機関から取得した技能試験サンプルのパネルは2つの方法の間、一次比較に使用した。 2遺伝子型決定法は正常に両方の方法により増幅したサンプルについて、HIVDBプログラムによって解釈されるすべての臨床的に重要な薬剤耐性関連変異を同定することに100パーセント一致した。 表6に示すように、三対のヌクレオチド配列は99.5%同一であった。予測されるアミノ酸配列は100%同一であった。 5のうち一つのサンプルは正常にViroseqにより増幅することができませんでした。 Viroseqによって増幅されていない試料に加えて、社内の方法が示された第二の試料を増幅するのに失敗したViroseqによって循環組換えウイルス(CRF02_AG)されるように。両方の方法論を用いて増幅3のサンプルは、サブタイプB(2サンプル)およびサブタイプA(1サンプル)となりました。
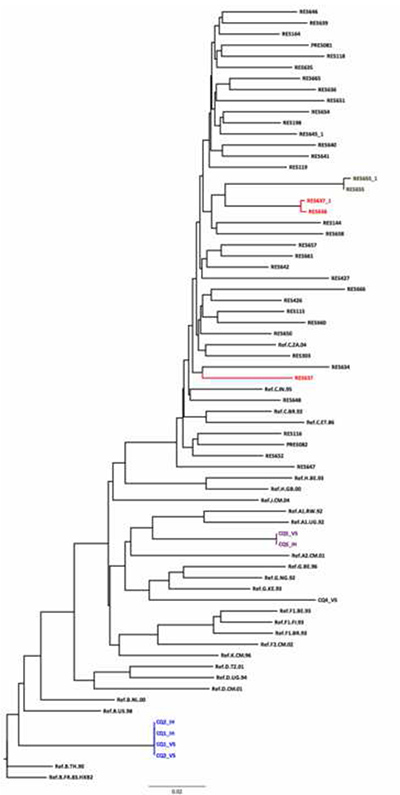
図5。シーケンスの品質保証の一環として行わHKY近隣参加ツリーの使用。非常に短い遺伝的距離との配列の4組/クラスターがあります。 RES655とRES655_1(別の日に、配列決定同じサンプル)間の遺伝距離は0.003である。それらの遺伝的距離が異なる疫学的にリンクされていない個人からのサンプルのために(0.075)が短すぎるようRES637_1/RES638ペアとの潜在的なエラーです。 RES638_1に比べて0.075の距離がツリー上の別のRES637があります。 CQ01/CQ02クラスタは、二つのサンプルを示唆しているパネルでも同様のサンプルの重複があります。彼らはREGAサブタイピングツールによって割り当てられ、サブタイプを確認するサブタイプB参照配列と一緒にクラスタ化する。 REGAのサブタイピングツールは、それぞれAとCRF02_AGとしてそれらを分類し、一方、CQ05とCQ04は、それぞれのサブタイプAおよびGとクラスター。のHIVサブタイプとの再結合のための別の有用なツールがhttp://www.datamonkey.orgで入手可能ですSCUELです。 拡大画像を表示するにはここをクリックしてください。
5つのサンプルのパネルは、社内方法の精度を評価した。十複写遺伝子型が5つの試料のそれぞれについて生成された。 16キャピラリジェネティックアナライザーを用いて、50の遺伝子型の48は同じ日に調製した24ラン、から生成した。すべての5つのサンプルについて、予測されたアミノ酸配列が反復の間で100%一致した。核酸配列は、目のためのERE> 99%対ごとの類似性があった。
このメソッドの使用の最初の2年間に、60サンプルが配列決定にRNA抽出からランダムに繰り返した。シーケンス品質スコアと複製物との間に混合塩基の数の間には統計的に有意な差は認められなかった。 60組のヌクレオチドおよびアミノ酸対比較の両方が99%以上同一であった。したがって、すべての対に対する薬剤耐性変異は100%一致した。
コスト削減
RT、PCRおよび配列決定のための反応量は、生成シーケンスの品質に妥協することなく、独自の方法20、32と比較して、少なくとも半分に短縮されました。これは、RTおよびPCR段階用の50%のコスト削減を可能にした。
新方式は、元々の配列に6配列決定プライマーで動作するように設計されましたプロテアーゼ遺伝子および逆転写酵素遺伝子20、32の最初の300コドンの全てのコドン99。同様の方法はまた、六から八プライマー33、34を使用しています。時にはseprately 35、36、プロテアーゼおよびRT遺伝子の配列を決定するが、いくつかの最近発表された方法には、6個未満のプライマーを使用している。我々は、( 図6)、六から四配列決定プライマーの数を減らすことが求められて
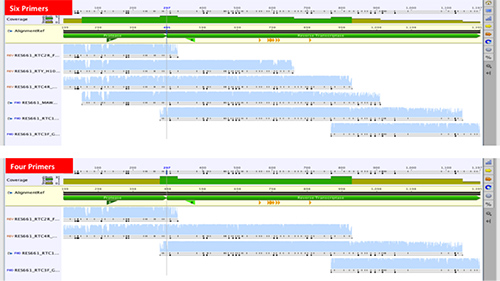
図6。全99 HIV-1プロテアーゼコドンおよび逆転写酵素遺伝子の最初の300コドンをカバーする1197 bpのpol配列の生成のための6つの4対配列決定プライマー由来の連続配列の比較。242/51242fig6highres.jpg "ターゲット=" _blank ">拡大画像を表示するにはここをクリックしてください。
17サンプルのセットからの配列は、2つのプライマー(MAW46とRTY)を除外した後に生成された配列と比較した6のプライマーから生成された。サブタイプは、シーケンスの品質スコアに有意差はなかった14のサブタイプCつのサブタイプB、一つのサブタイプA.あった。再度、核酸の17対の間の平均ペアワイズ同一性は、アミノ酸レベルで99%および100%であった。したがって、六から四配列決定プライマーを低減ほぼ3分のシーケンシングコストの低減をもたらした。
このプロトコルで使用される唯一の独自のソフトウェアツールは配列アセンブリのGeneiousであった。薬剤耐性解釈ツールならびにレポート生成ツールは、すべて無料で、オープンアクセスツールです。これは、独自のソフトウェアの使用に伴うコストをなくすことによって、コストをさらに低減する。さらに、collectivEネゴシエーションは、このプロトコルのための試薬 はライフテクノロジーズから簡単にアクセスするためのキットにパッケージ化と土星/ライフテクノロジーズは、メソッド37の遺伝子型を特定として提供されてすることができた。また、土星のメンバーは割引価格での試薬にアクセスすることができます。
臨床背景
記載されているプロトコルは、クワズールナタールの農村コミュニティの監視や薬剤耐性のサーベイランスに実装されています。 604遺伝子型の合計>千RNAコピー/ mlのウイルス量の試料の95%の増幅率で2010年12月と2013年5月の間、臨床サンプルから生成した。この臨床HIVの薬剤耐性の研究はクワズールナタール大学(参考のBF052/10)の生物医学研究倫理委員会と健康のクワズールナタール局(参照HRKM 10分の176)の健康の研究委員会によって承認された。個々の患者のレポートが生成され、診療所に送り返されました患者管理のため。
72個(72)の遺伝子型はまた、送信された薬剤耐性の研究の調査の一部として生成大規模な前向き集団ベースのHIVサーベイランス調査内にネストされた。主なサンプルは、EDTAマイクロチューブに集め、針を刺す全血だった。ジェノタイピングで79%で19の増幅率があった。調査研究からのサンプルの遺伝子型判定のための倫理承認はクワズールナタール生物医学研究倫理委員会(参考文献BE066107)の大学から入手した。
| プライマー名 | シーケンス | 長さ | 方向 | HXB2ポジション | |
| MAW-26 | TTGGAAATGTGGAAA GGAAGGAC | 23 | フォワード | 2028-2050 | 第一回目のPCR |
| RT-21 | CTGTATTTCAGCTATC AAGTCCTTTGATGGG | 31 | リバース | 3539-3509 | 第一回目のPCR |
| プロ1 | TAGAGCCAACAGCCC CACCA | 20 | フォワード | 2147-2166 | 第二ラウンドのPCR |
| RT-20 | CTGCCAATTCTAATTC TGCTTC | 22 | リバース | 3462-3441 | 第二ラウンドのPCR |
| RTC1F | ACCTACACCTGTCAA CATAATTG | 23 | フォワード | 2486-2508 | シークエンシング |
| RTC2R | TGTCAATGGCCATTG TTTAACCTTTGG | 27 | リバース | 2630-2604 | シークエンシング |
| RTC3F | CACCAGGGATTAGAT ATCAATATAATGTGC | 30 | フォワード | 2956-2994 | シークエンシング |
| RTC4R | CTAAATCAGATCCTAC ATACAAGTCATCC | 29 | リバース | 3129-3101 | シークエンシング |
| RT-Y | GTGTCTCATTGTTTAT ACTAGG | 22 | リバース | 2967-2946 | シークエンシング |
| MAW-46 | TCCCTCAGATCACTC TTTGGCAACGAC | 27 | フォワード | 2251-2277 | シークエンシング |
表1。 、、PCRを逆転写し、すべての99のHIV-1プロテアーゼコドンと逆trascriptase遺伝子の最初の300コドンをカバーする1197 bpのPOLフラグメントの生成に使用されるカスタムプライマーの配列を決定する。
| RT21(5pmol/ml) | 0.5 | 0.2 |
| のdNTP(10 mM)を | 0.5 | 0.4 |
| 合計 | 1 |
表2。のdNTP /プライマーは、逆転写反応混合する。
| 試薬 | 体積(ml)/反応 | 濃度/反応 |
| 第一鎖バッファー(10×) | 1 | 1 |
| 塩化マグネシウム(25ミリモル) | 2 | 4 |
| DTT(0.1M) | 1 | 0.008 |
| RNアーゼOUT(40単位/ ml) | 0.5 | 16 |
| 上付きIII逆転写酵素(200U/ml) | 0.5 | 8 |
| 合計 | 5 |
表3。酵素は、逆転写反応混合する。
| 試薬 | 体積(ml)/反応 | 最終濃度/反応 |
| DEPC処理水 | 18.4 | - |
| PCRバッファー(10×) | 2.5 | 1 |
| のMgCl 2(50 mM)を | 1 | 2 |
| dNTPミックス(10 mM)を | 0.5 | 0.2 |
| 偏屈なプライマー(5ピコモル/ ml)を | 0.25 | 0.05 |
| リバースプライマー(5ピコモル/ ml)を | 0.25 | 0.05 |
| プラチナTaqポリメラーゼ(5単位/ ml) | 0.1 | 0.02 |
| 小計 | 23 | - |
表4。ネステッドPCR用マスターミックス。
| 試薬 | 体積(ml)/反応 | 濃度/反応 |
| DEPC処理水 | 6.1 | |
| シークエンシングバッファー(5X) | 2 | 1 |
| プライマー(3.2ピコモル/ ml)を | 0.5 | 0.16 |
| ビッグダイターミネーターシーケンミックス | 0.4 | - |
| 合計 | 9 |
表5。シークエンシング反応用マスターミックス。
| Viroseq | 社内 | %のNa類似性 | |||||||
| 検体ID | サブタイプ | 品質スコア | PR変異 | RT変異 | サブタイプ | 品質スコア | 広報の変異 | RT変異 | |
| CQ01 | B | 99.9 | M46L、I54L、V82A、L90M | D67N、T69D、K70R、M184V、T215V、K219Q | B | 99.2 | M46L、I54L、V82、L90M | D67N、T69D、K70R、M184V、T215V、K219Q | 100 |
| CQ02 | B | 99.5 | M46L、I54L、V82A、L90M | D67N、T69D、K70R、M184V、T215V、K219Q | B | 99.5 | M46L、I54L、V82A、L90M | D67N、T69D、K70R、M184V、T215V、K219Q | 100 |
| CQ03 | NA | NA | NA | NA | NA | NA | |||
| CQ04 | CRF02_AG | 98.4 | I54V、V82F、I84V | M41L、L74I、L210W、T215Y、V108I、Y181C | NA | NA | NA | NA | NA |
| CQ05 | A | 99.7 | K103N | A | 93 | K103N | 100 | ||
表6。比較resulViroseqのジェノタイピング法とANRSによって提供されたサンプルのパネルを使用して、社内の方法との間に並列解析からのTS。
ディスカッション
いくつかの低コストのインハウスの方法は、33、34、36 HIV薬剤耐性遺伝子型決定は、より手頃な価格で提供しようとする努力に記載されている。資源が限られた設定で、抗レトロウイルス療法を受けている人々のためのケアの連続に薬剤耐性検査を統合する必要性に疑問の余地はない。しかし、報告された方法のほとんどは、集団レベルでの薬剤耐性のサーベイランスにおける薬剤耐性遺伝子型決定の適用に焦点を当てる。土星/ライフテクノロジーズの遺伝子型判定法は、薬剤耐性のサーベイランス及びモニタリングのための完全に統合されたプロトコルです。この方法は、臨床管理のための報告書の薬剤耐性と世代の解釈の大部分はオープンソースとオープンアクセスのバイオインフォマティクスのリソースを実装する手頃なプロトコルとして設計されました。
これは、ようにするViroseq遺伝子型判定法を承認したFDAとの比較により示された正常に増幅された実験室用パネルサンプルの100%において、ANRS技能試験試料のパネルからの薬剤耐性変異の同定に正確。精度もサブタイプCウイルス、南部アフリカで最も優勢なサブタイプの臨床サンプルを評価した。それは本方法はCRF02_AGが流行している世界の他の部分で使用される場合は、方法日時プライマーの改変が必要であるサブタイプAおよびBにあったような方法は、サブタイプCサンプル上で正確であったCRF02_AGを有することが示されたパネル試料の一つを増幅するのに失敗しました。あるいは、すべての基Mに感受性のプライマーの縮重セットは、サブタイプの分布が38より不均一である場合は、領域36で使用することができる、33ウイルス。
逆転写およびPCRの感度は、例えば、500ミリリットル、プラズマの高いボリュームからRNAを抽出することにより増加させることができる。プラズマはcentrifすることができますをQIAampウイルスRNA抽出ミニキットに記載されているように、プロトコルを実行する前に、ウイルス粒子を濃縮するために90分間21000×gでuged。
図示のように、新しい方法は、個々の患者管理のための包括的なレポートを生成する追加の利点を有する。これらのレポートは、RegaDBから遺伝子型の統合、免疫学的およびウイルス学的監視データだけでなく、臨床および治療の歴史である。これは、同じように、詳細な患者の病歴の見直しだけでなく、治療勧告が続く抵抗プロファイルの詳細な実験室での解釈を伴う。レポートを確認し、患者のための治療勧告を提供するために、専門の医師の使用はますますタスクシフティングのためのWHOの勧告の一部として南アフリカでARTを提供している看護実践だけでなく、経験の浅い臨床医のための待望の指導を提供します。これらの臨床レポートには、薬剤耐性管理のほとんど、またはまったく経験の臨床医のための効果的な教材であることが示されている。患者の視点から、私たちの方法は、専門家のHIVサービスにアクセスするために、集中サイトに移動する必要がなくなります。
このように、全体として説明されたプロトコルは、HIVの薬剤耐性管理がARTを失敗、HIV感染者のケアの連続に、低コストで、統合することができるような優れたプラットフォームを提供します。生成されたデータは、コミュニティ内の薬剤耐性の発生および送信を評価するために、疫学的目的に使用することができる。生成されたPOL断片の大きさは、集団レベルでの流行のよりよい理解を生成し、より複雑な系統解析には十分である。
開示事項
保健システムの強化とHIV治療失敗(HIV-:この作品は、ウェルカムトラスト(082384/Z/07/Z)、欧州連合(サンテ2007 147から790)によってサポートされていました、病気のための米国センターはCAPRISA(プロジェクトタイトルを経由して制御しますTFC))、およびスイス南アフリカ共同研究プログラム(SSJRP)と題する研究助成金「スイスのProt /南アフリカ:タンパク質バイオインフォマティクス人材育成は重要な健康関連の病原体」。RLがウェルカムトラストによってサポートされている(認可番号090999 / Z / 09 / Z)。資金提供者は、研究デザイン、データ収集と分析、公開することを決定、または原稿の準備に何の役割も持っていなかった。著者らは、競合の金融利益を宣言していません。
謝辞
著者らは、この作業を可能にしたすべての同僚、特にマヤBalamane、エリザベス·ジョンストンホワイト、シャロンSjoblom、グレッグディングZakhona Gumede、Xolile Kineri、Phindile Mabaso、Lungisa Ndwandwe、ジェームズ·ガーベイ、ギャビンコブ、Senzo Maphanga、Terusha Chettyを承認したいと思います、Keviナイドウ、アンドリューSkingsley、キャサリン·ストット、およびLungani Ndwandwe。著者はまたHlabisa、HIV治療とケアプログラムを働く保健省とアフリカセンター職員のすべての職員に感謝したいと思います。
資料
| Name | Company | Catalog Number | Comments |
| Superscript III 1st strand Synthesis kit | Life Technologies | 18080051 | Reverse Transcription |
| SATURN/LiFE Technologies Custom Primers | Life Technologies | 4473517 | PCR |
| Platinum Taq | Life Technologies | 10966026 | PCR |
| PureLink QUICK PCR Purification Kit | Life Technologies | K310002 | PCR |
| Viroseq | ABBOTT | 4J94-20 | Reverse Transcription and PCR |
| Agarose Tablets (Dnase/Rnase free) | BIOLINE | BIO-41027 | PCR |
| TBE Buffer | MERCK | 1.06177.2500 | PCR |
| O'Range Ruler 200 bp DNA Ladder | Fermentas | FE SM0633 | PCR |
| Novel Juice | GeneDireX | LD001-1000 | PCR |
| MiniBis Bioimaging System | DNR Bioimaging Systems Ltd | Gel Documentation | |
| Power Pac 300 | BIORAD | Gel Electrophoresis | |
| Big Dye Terminator Kit version 3.1 | Life Technologies | 4337456 | Sequencing |
| Arrays | Life Technolgies | 4319899 | Sequencing |
| PoP | Life Technologies | 4363785 | Sequencing |
| 10x EDTA Buffer | Life Technologies | 402824 | Sequencing |
| Formamide | Life Technologies | 4311320 | Sequencing |
| 5x Sequencing Buffer | Life Tecgnologies | 4336697 | Sequencing |
| 3130 xl Genetic Analyzer | Life Technologies | Sequencing | |
| GeneAmp PCR System 9700 | Life Technologies | RT/PCR/Sequencing | |
| Centrifuge 5804 | EPPENDORF | Sample Processing | |
| Centrifuge 5415R | EPPENDORF | RNA Extraction | |
| Centrifuge 5415R | EPPENDORF | RT and PCR | |
| Centrifuge 5415D | EPPENDORF | PCR Product Clean up | |
| Centrifuge 5810 | EPPENDORF | Sequencing Clean up | |
| Picofuge | BIORAD | C1301-230V | RT and PCR |
| Vortex Genius 3 | IKA | RNA extraction and reagent preparation | |
| Vortex mixer | IKA | Sequencing Cleanup | |
| NanoDrop 2000 UV/VIS spectrophotometer | ThermoScientific | DNA quantification | |
| 3 M Sodium Acetate | MERCK | 567422 | Sequencing Clean up |
| Absolute Ethanol | MERCK | SAAR2233540LP | Sequencing Cleanup |
| 1.5 ml SARSTEDT Tubes | BIODEX | 72.692.005 | RNA Extraction |
| 2 ml SARSTEDT Tubes | BIODEX | 72.693.005 | RNA Extraction |
| 2 ml Collection tubes | SCIENTIFIC GROUP | MCT-200-NC/S | RNA Extraction |
| Optical MicroAmp 96-well reaction plates | Life Technologies | N8010560 | Sequencing |
| 200 µl 8 Strip StarPCR Tubes with attached flat caps | STAR Lab - supplied by CELTIC | A1402-3700 | RT and PCR |
| 200 µl PCR individual tubes | Scientific Group | CR/3745 | RT and PCR |
| Geneious | Biomatters | Sequence analysis | |
| Internet Access | Preferrably high speed | ||
| Web resources | |||
| hivdb.stanford.edu | Stanford University | Drug reistance analysis | |
| http://bioafrica.mrc.ac.za:8080/regadb-ui/RegaDB | SATuRN | database | |
| http://bioafrica.mrc.ac.za/tools/pppweb.html | SATuRN | Sequence quality tool | |
参考文献
- Shao, Y., Williamson, C. The HIV-1 epidemic: low- to middle-income countries. Cold Spring Harbor Persp. Med. 2, (2012).
- Mutevedzi, P. C., et al. Scale-up of a decentralized HIV treatment programme in rural KwaZulu-Natal, South Africa: does rapid expansion affect patient outcomes. Bull. World Health Organ. 88, 593-600 (2010).
- Houlihan, C. F., et al. Cohort profile: Hlabisa HIV treatment and care programme. Int. J. Epidemiol. 40, 318-326 (2011).
- Tanser, F., Barnighausen, T., Grapsa, E., Zaidi, J., Newell, M. L. High coverage of ART associated with decline in risk of HIV acquisition in rural KwaZulu-Natal. South Africa. Science. 339, 966-971 (2013).
- Bor, J., Herbst, A. J., Newell, M. L., Barnighausen, T. Increases in adult life expectancy in rural South Africa: valuing the scale-up of HIV treatment. Science. 339, 961-965 (2013).
- Montaner, J. S., et al. The case for expanding access to highly active antiretroviral therapy to curb the growth of the HIV epidemic. Lancet. 368, 531-536 (2006).
- Granich, R. M., Gilks, C. F., Dye, C., De Cock, K. M., Williams, B. G. Universal voluntary HIV testing with immediate antiretroviral therapy as a strategy for elimination of HIV transmission: a mathematical model. Lancet. 373, 48-57 (2009).
- Johnson, L. F., et al. Life expectancies of South african adults starting antiretroviral treatment: collaborative analysis of cohort studies. PLoS Med. 10, (2013).
- Blower, S., Ma, L., Farmer, P., Koenig, S. Predicting the impact of antiretrovirals in resource-poor settings: preventing HIV infections whilst controlling drug resistance. Curr. Drug Targets. 3, 345-353 (2003).
- Geretti, A. M. Epidemiology of antiretroviral drug resistance in drug-naive persons. Curr. Opin. Infect. Dis. 20, 22-32 (2007).
- Larder, B. A., Darby, G., Richman, D. D. HIV with reduced sensitivity to zidovudine (AZT) isolated during prolonged therapy. Science. 243, 1731-1734 (1989).
- Erice, A., et al. Brief report: primary infection with zidovudine-resistant human immunodeficiency virus type 1. N. Engl. J. Med. 328, 1163-1165 (1993).
- Williams, I., et al. British HIV Association guidelines for the treatment of HIV-1-positive adults with antiretroviral therapy. HIV Med. 13 Suppl 2, 1-85 (2012).
- DHHS, . US Panel on Antiretroviral Guidelines for Adults and Adolescents. Guidelines for the use of antiretroviral agents in HIV-1-infected adults and adolescents. , (2012).
- Vandamme, A. M., et al. European recommendations for the clinical use of HIV drug resistance testing: 2011 update. AIDS Rev. 13, 77-108 (2011).
- DHHS, . US Panel on Antiretroviral Therapy and Medical Management of HIV-Infected Children. Guidelines for the Use of Antiretroviral Agents in Pediatric HIV Infection. , (2012).
- . Department of Health. Clinical guidelines for the management of HIV & AIDS in adults and adolescents. , (2010).
- Conradie, F., et al. The 2012 southern African ARV drug resistance testing guidelines. S. Afr. J.HIV Med. 13, 162-167 (2012).
- Manasa, J., et al. Primary Drug Resistance in South Africa: Data from 10 Years of Surveys. AIDS Res. Hum. Retroviruses. 28, 558-565 (2012).
- Tshabalala, M., et al. Surveillance of transmitted antiretroviral drug resistance among HIV-1 infected women attending antenatal clinics in Chitungwiza, Zimbabwe. PLoS ONE. 6, (2011).
- Bartolo, I., et al. Antiretroviral drug resistance surveillance among treatment-naive human immunodeficiency virus type 1-infected individuals in Angola: evidence for low level of transmitted drug resistance. Antimicrob. Agents Chemother. 53, 3156-3158 (2009).
- Bartolo, I., et al. HIV-1 genetic diversity and transmitted drug resistance in health care settings in Maputo, Mozambique. J. Acquir. Immune Defic. Syndr. 51, 323-331 (2009).
- Hamers, R. L., et al. HIV-1 drug resistance in antiretroviral-naive individuals in sub-Saharan Africa after rollout of antiretroviral therapy: a multicentre observational study. Lancet Infect. Dis. 11, 750-759 (2011).
- Hamers, R. L., et al. HIV-1 Drug Resistance Mutations Are Present in Six Percent of Persons Initiating Antiretroviral Therapy in Lusaka, Zambia. J. Acquir. Immune Defic. Syndr. , (2010).
- Nwobegahay, J., Selabe, G., Ndjeka, N. O., Manhaeve, C., Bessong, P. O. Low prevalence of transmitted genetic drug resistance in a cohort of HIV infected naive patients entering antiretroviral treatment programs at two sites in northern South Africa. J. Med. Virol. 84, 1839-1843 (2012).
- Iweriebor, B. C., et al. Molecular epidemiology of HIV in two highly endemic areas of northeastern South Africa. Arch. Virol. 157, 455-465 (2012).
- Parboosing, R., Naidoo, A., Gordon, M., Taylor, M., Vella, V. Resistance to antiretroviral drugs in newly diagnosed, young treatment-naive HIV-positive pregnant women in the province of KwaZulu-Natal South Africa. J. Med. Virol. 83, 1508-1513 (2011).
- Nwobegahay, J. M., et al. Prevalence of antiretroviral drug resistance mutations and HIV-I subtypes among newly-diagnosed drug-naive persons visiting a voluntary testing and counselling centre in northeastern South Africa. J. Health Popul. Nutr. 29, 303-309 (2011).
- Nwobegahay, J., et al. Prevalence of drug-resistant mutations in newly diagnosed drug-naive HIV-1-infected individuals in a treatment site in the waterberg district, limpopo province). S. Afr. Med. J. 101 (2011), 335-337 (2011).
- Rosen, S., Long, L., Sanne, I., Stevens, W. S., Fox, M. P. The net cost of incorporating resistance testing into HIV/AIDS treatment in South Africa: a Markov model with primary data. J. Int. AIDS Soc. 14, 24 (2011).
- Dalai, S. C., et al. Evolution and molecular epidemiology of subtype C HIV-1 in Zimbabwe. AIDS. 23, 2523-2532 (2009).
- Chen, J. H., et al. In-house human immunodeficiency virus-1 genotype resistance testing to determine highly active antiretroviral therapy resistance mutations in Hong Kong. Hong Kong Med. 18, 20-24 (2012).
- Lee, C. K., et al. An in-house HIV genotyping assay for the detection of drug resistance mutations in Southeast Asian patients infected with HIV-1. J. Med. Virol. 84, 394-401 (2012).
- Aitken, S. C., et al. A Pragmatic Approach to HIV-1 Drug Resistance Determination in Resource-Limited Settings by Use of a Novel Genotyping Assay Targeting the Reverse Transcriptase-Encoding Region Only. J. Clin. Microbiol. 51, 1757-1761 (2013).
- Zhou, Z., et al. Optimization of a low cost and broadly sensitive genotyping assay for HIV-1 drug resistance surveillance and monitoring in resource-limited settings. PLoS One. 6, (2011).
- , Life Technologies. Life Technologies and SATuRN Collaborate to Increase Access to HIV Testing in Africa. , (2012).
- Lihana, R. W., Ssemwanga, D., Abimiku, A., Ndembi, N. Update on HIV-1 diversity in Africa: a decade in review. AIDS Rev. 14, 83-100 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved