このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ワンナノメートルの精度で蛍光イメージング(FIONA)
要約
シングルフルオロフォアはFIONAを使用して、ナノメートルの精度で定位させることができる。ここでFIONA技術の概要が報告され、そしてどのようにFIONA実験を実施する方法について説明する。
要約
1ナノメートルの精度(FIONA)と蛍光イメージングは、xy平面上にナノメートルの精度で単一の蛍光団をローカライズするためのシンプルでありながら有用な技術である。ここでFIONA技術の概要が報告され、FIONAを使用して行われている研究の例が簡潔に記載されている。まず、 すなわち 、FIONA実験のために全反射蛍光顕微鏡(TIRFM)を、必要な機器の設定方法を、光学部品を位置合わせする方法の詳細に記載されている。次に、どのように量子ドットで標識された単一の切り捨てミオシンVaモータの36 nmのステップサイズを測定するためFIONAの使用に続いて、適切なプロトコルを使用して固定化したCy3でDNA一分子のローカライズで簡単なFIONA実験を実施するために、例示されている。最後に、厚いサンプルにFIONAのアプリケーションを拡張するための最近の取り組みが報告されている。 >(これは、水浸対物レンズを使用して、それを示され、量子ドットは、ゾル - ゲルおよびウサギの眼の角膜における深い浸し200μm)は、2〜3程度の局在化精度を達成することができる。
概要
1882年頃、エルンスト·アッベは、可視光顕微鏡の分解能は〜λ/ 2NA、または〜200nmの(λは波長であり、NAは開口数である)1,2であることがわかった。したがって、この寸法よりも小さい任意のオブジェクトは、光学顕微鏡の回折限界スポットとして現れる。しかし、非常に高い精度3であるスポットの中心、物体の位置を決定することが可能である。 1ナノメートルの精度(FIONA)と蛍光イメージングは、xy平面4ナノメートルの精度で単一の蛍光団をローカライズするためのシンプルでありながら有用な技術である。局在化の精度σμ( すなわち 、平均の標準誤差)は、収集された光子の総数に依存する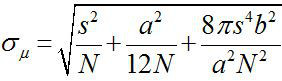 Nは、光子計数であり、sは蛍光スポットの標準偏差であり、aはピクセルイメージング検出器の大きさ、bはバックグラウンド3,4の標準偏差である。 〜万光子を放出する蛍光団のために、フィオナは〜1nmの精度4を達成することができます。
Nは、光子計数であり、sは蛍光スポットの標準偏差であり、aはピクセルイメージング検出器の大きさ、bはバックグラウンド3,4の標準偏差である。 〜万光子を放出する蛍光団のために、フィオナは〜1nmの精度4を達成することができます。
FIONA正確静止エミッタの位置、または(十分に速く取り出すことができる画像を仮定して)移動するかを決定するために用いることができる。 FIONAムービーのフレームに順次適用されるため、単一分子4〜8の動きを追跡することができます。光防護試薬は、試料が光分解しないことを保証する必要があるかもしれない。さらに、蛍光物体自体は任意のサイズであってもよい、リミット-側、例えば回折よりも小さいまたは大きいと、その膜上に分散多くの蛍光タンパク質と細胞小器官(〜1ミクロン)で構成することができる。 FIONAを使用すると、まだその平均重心の非常に正確な(ナノメートル)の平均を得ることができる。 FIONAによるローカリゼーション精度の大幅な向上はnanomeを解決することができます時間をかけてタースケールの動き。これは、分子長さスケール4〜8に顕微鏡をプッシュしています。
その発明以来、FIONAの変異体が開発されてきた。例えば1ナノメートルの精度(bFIONA)9と、明視野イメージング、FIONAのわずかな変種、画像や透過光などのメラノソームのin vivo(メラニン色素を含む暗いオブジェクト)などの高密度のオブジェクトを局在する。また、FIONAは、複数の色素を解決するために採用されている。例えば、光退色(エビ)を有する単一分子の高分解能イメージング10,11または単一分子の高解像度の共局在(SHREC)は12が約10nm内の2つの色素を解決するために開発されてきた。さらに最近では、FIONA分析はそのような確率論的光学recoのようなある種の超解像顕微鏡のローカリゼーションプロセスに貢献してきました( つまり 1が離れて、同一の染料を伝えることができますどのように正確に。、これは解像度であることに注意してください)nstruction顕微鏡(STORM)13〜15と光活性化ローカライゼーション顕微鏡(PALM)16、一時的な暗い蛍光団が励起され、その後、蛍光がローカライズされている。繰り返しエキサイティングかなり低い染料の密度(未満あたりの回折限界スポット)した後、FIONAによってそれらのそれぞれを分析し、蛍光を収集することによって、人は、高解像度のマップを構築することができます。解像度は、ちょうど光子の数によって制限され、各染料が出す、ならびに取得中に( 例えば 、顕微鏡ステージを含む)試料を静止状態に保つようなもの。
本論文では、FIONA技術の概要と簡単にFIONAが報告されている使用して行われている研究の例を記載している。まず、 すなわち 、FIONA実験のために全反射蛍光顕微鏡(TIRFM)を、必要な機器の設定方法を、光学部品を位置合わせする方法の詳細に記載されている。次に、どのように適切なプロトコルを使用して固定化されたCy3-DNAの単一分子を局在化する上で単純なFIONA実験を行って、図示されている。その後、FIONAの使用は量子ドットで標識された単一の切断型ミオシンVaモータ36 nmのステップサイズを測定することが提示されている。ミオシンVaは、アクチンフィラメントに沿って転位しながら携帯の貨物を運ぶ不可欠プロセッシブモータータンパク質である。ここで、Vaはトランケート構築ミオシンステップサイズとは無関係なドメインを除去するために使用され、およびFLAGタグがC末端に付加して、抗FLAG抗体で官能化量子ドットで標識の容易さを可能にする。この実験は、ミオシンを遅くし、フレーム毎に優れた光子数を得るために十分に長い露光時間の使用を可能にするために、低ATP下で行われる。でも十分に明るい蛍光標識は、以下のプロトコルに置き換えることができる。最後に、厚いサンプルにFIONAの適用を拡張する最近の努力が報告されている。原理証明として、量子ドットは、浸漬したゾル - ゲルおよびウサギの眼の角膜における、その後FIONAを使用して画像化し、ローカライズされた。この目的は、以前に使用100X油浸対物レンズよりも長い作動距離を有するため、撮像のためにNAと、60X浸水対物= 1.2を用いた。客観的に倍率の損失を補償するために、余分な倍率のレンズ(3.3倍または4.0X)が放出路に挿入した。また、落射蛍光(ないTIR)顕微鏡は、厚い試料の深い領域にアクセスするために使用される必要がある。これは、量子ドットは、ゾル - ゲル中の深い浸したことが示されていると、ウサギの眼の角膜(Z> 200ミクロン)に2-3 nmの精度で定位させることができる。
Access restricted. Please log in or start a trial to view this content.
プロトコル
倫理に関する声明:ウサギからの角膜組織はイリノイ大学施設内動物の管理および使用ガイドラインに従って収集した。
1 TIRFMのセットアップ
注:すべての時間レーザーの安全ゴーグルを着用する。
- 材料のリストに記載されているすべての必要な光学部品が利用可能であり、位置合わせのための準備ができていることを確認してください。必要に応じて、他の企業からの同等の機能を持つ代替品を使用しています。ミラー、レンズが使用されているレーザーと一致する反射防止(AR)コーティングを有するべきであることを確認してください。
- 顕微鏡バック口の中心の高さにすべての光学部品の高さを設定します。
- レーザは、レーザシャッターとNDフィルタ(複数可)をマウント。目に見える光を維持しながら、可能な限り低くレーザーパワーダウン減衰させるためにNDフィルターを使用してください。適切な六角キーを使用してネジを締めます。
- ビーム経路を計画し、光学テーブル上のテープまたはマーカー(ドットでマークテッド図1の青い線)。簡単にするために、光学テーブル上の穴の線に沿ってまっすぐにパスを続ける。
- 置きミラーM1( 図1)第1の直角変わり目。計画されたビーム経路の第2の直線部分に沿って二虹彩を配置します。位置やレーザーが虹彩を通過するようにM1の傾きの両方を調整します。
- 置きミラーM2( 図1)第2の直角変わり目。パスの第三の直線部分に沿って10倍のビームエキスパンダ(L1&L2、 図1)を配置します。ビームエキスパンダーは、光学テーブルと計画されたビーム経路の両方と平行になるように、その傾きを調整します。
- レーザービームエキスパンダの両方のレンズの中心を真っ直ぐに行くことをM1とM2は、反復的にそのような調整。拡大ビームのビームプロファイルがガウス非クリップされるまで、この手順を繰り返します。
- L1(Fiの上でビームを中心に、M1を調整しますグレ1)およびM2を反復L2( 図1)上のビームをセンタリングします。悪いビームプロファイルは通常、レーザーがクリップされることを意味;目でビームプロファイルをチェックするためにビームエキスパンダの後にビームを遮断するために白い紙を使用しています。高精度の分析のために、光ビームプロファイラを使用しています。
- ビームが平行化されるように、L1とL2の間の距離を調整します。必要に応じて、ステップ1.8と1.9を繰り返します。
NOTE:ビームサイズが距離によって変化しない場合には、ビームはTIRFMのセットアップのために十分コリメートされる。さらに、ビームコリメーションを改善するために、そのようなシアリング干渉計などのツールを使用することができる。膨張後の典型的なビームサイズが〜20mmである。 - レーザーシャッター。蛍光アライメントターゲットにおいて顕微鏡対物レンズとネジを外します。場所は、顕微鏡ポートにタレット内部のダイクロイックミラーに拡大されたビームを向けるためにM3とM4( 図1)ミラー。レーザービームがt跳ね返ることを確認してください彼は、ダイクロイックと天井に向かって。
- 蛍光ターゲット上のビームの最も明るい部分を中心に、M3を調整し、M4は垂直にビームチルトを調整する。
- レーザーシャッター、バックでの目標をねじ込みます。前の手順でアライメントが行われた場合だけでなく客観的に出た対称スポットがあるはずです。微調整M3及びM4の傾きは、目的外のレーザパワー及びビームプロファイルを最適化する。
- 顕微鏡にEMCCDカメラをマウントし、カメラとパソコンを接続します。カメラのソフトウェアを起動します。
- 顕微鏡で蛍光試料(蛍光体の溶液)マウントします。カメラの明るい蛍光スポットを見てください。フォーカスが変更されるようにスポットが画面上にシフトしていないことを確認してください。
- L3(約30センチ)の焦点距離に等しく、対物レンズの後焦点面からの距離でXYZ平行移動ステージ上にTIRレンズ(L3、 図1)を配置します。 L3の位置を調整しますレーザは、レンズの中心を通過するようになっている。
- ビーム視準を調整するためにビーム経路に沿って、L3を翻訳します。ビームがまだモニターを中心とした、形状が左右対称であることを確認します。
注:TIRレンズの焦点距離の減少に伴って試料面が上昇する時の照明領域。一般的には、セットアップに合うことができる最小の焦点距離を使用しています。 - 対物レンズからのビームを傾けることがビーム経路L3垂直翻訳。 TIRが達成されるようにTIRレンズを並進保つ。良いSNRを得るためにEMCCDカメラを通じて蛍光ビーズのサンプルを観察し、微調整L3。
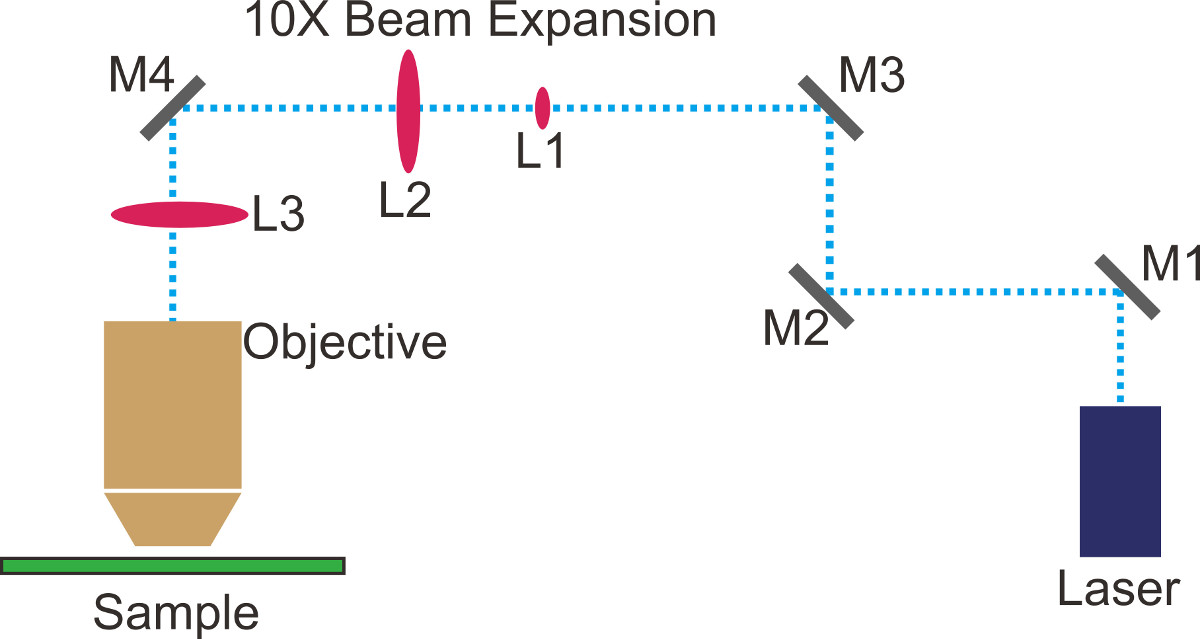
全反射蛍光顕微鏡(TIRFM)のための1の光学構成図。
Cy3でDNA上の2 FIONA
- C言語リーン顕微鏡スライドとカバーガラス:のddH 2 O及びイソプロパノールで顕微鏡スライドとカバーガラスを洗浄し、窒素ガスでそれらを乾燥;アルゴンプラズマで5分間のプラズマクリーナーにスライドやカバースリップを配置。
- ( 図2に描かれたように)サンプルチャンバを構築します。
- ベンチに組織を配置した後、組織の上にレンズ一枚の紙を置く。レンズ紙にスライドを置きます。スライドの清浄側が上向きであることを確認してください。
- 中央で3〜5程度の隙間を残して、長辺に沿ってスライド上に両面テープの2のストリップを適用します。スライドの上にきれいにカバースリップを置きます。カバースリップのクリーンサイドスライドに直面していることを確認します。
- 両面テープの上に押し下げ、ピペットチップを使用してください。テープが唯一のカバーガラスの下に残るようにスライドから余分なテープを除去するためにかみそりを使用してください。
注:チャンバの開口端は開いたままにして、入口とアウトとして機能しているてみましょう。チャンバの容積は、数マイクロリットルである。

典型的な試料室の図2。スケッチ(a)の上面図。 (b)は右からサ イドビュー。正面から(c)は側面図。
- サンプルチャンバの内面上のCy3-DNAを固定化する。
- T-50緩衝液(10mMのTris-HCl pH8.0で、50mMのNaCl)を準備。 1 mg / mlの最終濃度のT-50中のBSA-ビオチンを準備します。 10ミリグラム/ mlの最終濃度でT-50にBSAを溶解することによってT50-BSA緩衝液を調製する。
- T50-BSA緩衝液中で0.5 mg / mlのニュートラを準備します。 5-10 PMの最終濃度のビオチン化Cy3で標識したDNA T50-BSA中(のCy3-DNA)を準備します。
- ピペット10μlのBSA-ビオチン(1 mg / ml)を試料室に。 5分間待ちます。
- 40μlのT50-BSAでチャンバーを洗ってください。ピペット10μlのニュートラアビジン(0.5 mg / ml)を試料室に。 5分間インキュベートします。
- 40μlのT50-BSAでチャンバーを洗ってください。試料室にピペットで20μlのCy3でDNA。 5分間インキュベートした後、80μlのT50-BSAでチャンバーを洗浄する。
- TIRFM下の画像のCy3-DNA単一分子。
- 1μlのプロト-3,4ジオキシゲナーゼ(PCD、5μM)を混合することにより、イメージングバッファー(100μl)を準備し、4μlのプロトカテク酸(PCA、62.5 mM)を、50μlの6 - ヒドロキシ - 2,5,7,8- tetramethylchromane -2 - カルボン酸(T-50中のトロロックス、2 mM)を、45μlのT50-BSA。
- そして30μlのイメージングバッファ内のピペットは8〜10分間待ちます。
- 緑色レーザー(532nm)を装備しているTIRFM、100X油浸対物レンズ(1.45 NA)とEMCCDカメラで画像化するためのサンプルをマウントします。
- 1,0のサンプルの動画を取得し100に100から500ミリ秒と25のEMゲインへの露光時間を設定します00フレーム。
- のCy3-DNAの記録された画像にFIONAデータ解析を行います。
- 総合倍率(余分な倍率を乗じた顕微鏡対物レンズの倍率)によって(EMCCDカメラの仕様書から読み取る)物理的なピクセルサイズを分割することにより、有効画素サイズ( すなわち 、ナノメートルの画素からの変換係数)を決定します。
- CCD感度の画像取得時に使用される電子増倍管(EM)ゲインによる(CCDカメラの仕様書から読み取るA / Dカウントごと、 すなわち電子)を分周して数値を光子する画素強度から換算係数を決定します。
- コンパイルし、FIONA解析にFIONA.proを実行します。入力(ステップ2.5.1より)有効画素サイズと(ステップ2.5.2より)光子数の強度からの変換係数に、取得した画像をインポートするには、このIDLのプログラムを使用して、FIONA分析のためのスポットを選択する。
注:最後に、PROGRAm個の出力するであろう2次元ガウス関数とフィッティング結果だけでなく、総光子数およびローカリゼーション精度。典型的な結果は、代表的な結果と図4C〜4dのセクションに示されています。 - コンパイルし、光子カウントを特徴づけるためにphcount.pro実行します。 (ステップ2.5.2より)光子数の強度から取得した画像と、入力への変換係数をインポートする、光退色前の蛍光団により放射される光子の平均数を測定するために、このIDLのプログラムを使用します。
注:プログラムは、その後(手動選択はオプションです)が自動的に蛍光スポットを検出し、フレーム番号、および出力光子カウントの痕跡の関数としての光子数を計算します。- 悪いトレースを破棄し、ベースライン補正のために光退色後のフレームの範囲を指定します。最後に、プログラムは出力すべてのスポットの合計光子数のリストではなく破棄されます。そして、光子数の分布をプロットしてdistribuにフィット平均光子数を取得するために指数関数的減衰とる。典型的な結果は、代表的な結果と図4E-4Fのセクションに示されています。
3 FIONA応用は、ナノメートルスケールでのモーター( 例えば 、アクチン上のミオシン)ダイナミクスを定量化するために
- アクチンを重合FIONA実験の前に1日( すなわち 、F-アクチンを用意)。
- 一般的なアクチンバッファーで溶解を10mg /にG-アクチン(モノマー)とビオチンのG-アクチン(モノマー)を再構成する。両者が完全に溶解していることを確認し、氷の上で両方を維持するためにかき混ぜる。
- 1.5mlのマイクロチューブに1.7μlのビオチンのG-アクチンと10μlのG-アクチン(モノマー)を混ぜる。 100μlの氷冷アクチン重合バッファーを追加します。
- 4℃で一晩混合物のままにします(F-アクチンが形成された)した後、1ミリリットルの全容量のddH 2 Oを追加します。
- 実験で後で使用するために、4℃でアクチンフィラメント(F-アクチン)を保管してください。
注:フィラメントは崩壊し、短縮時間の経過が、少なくとも2週間のために使用することができるであろう。
- イメージングのためのサンプルを準備します。
- (プロトコル2.1と2.2で説明したように)試料室を作る。のddH 2 Oに1 mg / mlの時のBSAをビオチン化20μlのピペット10分間インキュベートします。 30μlののddH 2 Oですすぐ
注:このブロックは、ガラス面とアクチンフィラメントの結合にビオチンを定め。ビオチン化ポリ-L-リジン - ポリエチレングリコール(PLL-PEG)は、同じ機能を果たす可能性がある。 - 0.5 mg / mlのニュートラピペット。 2分間インキュベートし、その後30μlのM5緩衝液でチャンバーを洗浄。
- 準備されたF-アクチンでのピペットは、最終濃度〜0.004 mg / mlのために、一般的なアクチンバッファに25倍に希釈した。 10分待ってから、30μlの緩衝液でチャンバーをすすぐ。
- 最終濃度oをする(2mMのMgCl 2、25mMのKClを、1mMのEGTA、20mMのHEPES(pH7.6)中)M5緩衝液中で30倍FLAGタグとミオシンVaの希250 nMのF。 1μlの抗-FLAG-Qdot705(メーカーからの説明書に従ってQdot705抗体共役キットを用いて抗FLAG抗体とQdot705から体化〜1μM、)と1μlのミオシンを混ぜる。 10μlに埋めるために8μlのM5に追加します。アップピペット上下よく混合する。氷上で10分間インキュベートする。
注:これは〜25nMのミオシン濃度で、1モータ4への量子ドットの混合物が得られる。
- (プロトコル2.1と2.2で説明したように)試料室を作る。のddH 2 Oに1 mg / mlの時のBSAをビオチン化20μlのピペット10分間インキュベートします。 30μlののddH 2 Oですすぐ
- アクチンの上を歩いてミオシンのイメージング。
- (のddH 2 O中50μM)、2μlのDTT(のddH 2 O中の500mM)、1 ATPμL、84μlのM5-BSA(1 mg / mlのBSAをM5·バッファ)を混合することにより、イメージングバッファー(100μl)を準備する1 μlのCK(500 U / ml)を、5μlのCP(200 mM)を、1μlのPCD、4μlのPCA、1μlのミオシン-Qドット2.5 nMのミオシン濃度、および1μlのBMEにさらに10倍に希釈した後。
- 20μlのイメージングバッファ内のピペットは、チャンバをサンプリングし、8〜10分間インキュベートする。
- 画像T上のサンプル30ミリ秒露光時のIRF顕微鏡。 1,000以上のフレームを取得します。必要に応じてステップ3.3.1におけるミオシン-Qドットの音量を調整します。
- データ分析を行い、ミオシンの歩行のステップサイズを見つける。
- ImageJの17でビデオファイルを開き、移動スポットを中心にビデオをトリミング。スポットは、エッジの20ピクセル以内を取得しないことを、十分に大きな面積をトリミングし、ビデオ内の他のスポットが存在しないことを確認してください。このスポットは直線経路に移動していることを確認します。
- xとyは、ビデオの各フレーム毎にFIONA分析(ステップ2.5.3)を適用することにより、ピクセル単位で、時を座標を生成するためにビデオを介してスポットを追跡します。
- 前のセクションで説明したように、ナノメートルの画素を変換する。
- 時間の関数としての初期位置からの変位を計算します。
- ミオシン歩行のステップを得るために変位t検定を実行します。
注:t検定(step_t_test.zip)のために提供されたプログラムは、IDLや詐欺で符号化されているフォルダ内の14のサブルーチンのsists。- IDLでサブルーチンのすべてを開き、二度、すべてをコンパイルします。その後mtltyanalysis_ttest.pro実行し、単一の列のみで距離データを含むテキストファイルを選択します。出力Excelファイル、生データ、フィット感、およびステップサイズを含む、生成される。
- ステップ·サイズ欄からすべてのゼロの値を削除します。原産地またはMATLABを使用してステップサイズの分布をプロットします。ヒストグラムにガウスフィット。
注:ゼロのステップがないステップは、前のフレームからとられないことを意味するので、ゼロ値のステップサイズが削除される必要がある。 ( 図5に示すように)フィッティング36付近にピークを与える。
FIONA 4。厚い試料の調製
- ゾル - ゲル中にカプセル化された量子ドットを準備します。
- 4.5ミリリットルTMOS、1ミリリットルののddH 2 O及び100μlの塩酸(120 mm)を混ぜる。超音波洗浄器·エスピーに氷上で30分間、混合物を超音波処理材料のリスト(周波数= 40kHzで、ヒーター=オフ)にecified。ソリューションごとに10分を混ぜる。
- 1.5ミリリットルのHEPES(50mMのpH7.2)中に1.5μlのQdot605希釈する。前のステップから1.5ミリリットルのTMOSを有する溶液を混ぜる。
- ガラス底の皿に混合物を注ぐ。 1。5時間、4℃でパラフィルムや店舗のガラスボトムディッシュを密封。
- 試料皿をPBSで2ミリリットルの1%BMEを追加し、画像化の前に30分間室温でインキュベートする。
- 量子ドットで染色した角膜のサンプルを準備します。
注:ウサギの目は博士マリーナMarjanovicからの贈り物だった。- 目から角膜を分離し、3ミリメートル×3mmの小片にカット。
- 1mlのPBSに1μlのQドット605-ストレプトアビジン希釈する。 4で1 nMのQドット溶液による角膜組織をインキュベート 1時間ºC。 PBSで組織を洗浄する。
- 清浄なスライドガラスと#1.5カバースリップを取る。 、長辺に沿ってスライドガラスに両面テープの4つの層を入れてND両面テープを平行の別の4つの層は、前のテープに入れて、その間に約1cmのチャンネルにしておきます。チャネルの途中で前のステップから組織を置き、カバーガラスでそれをカバーしています。
- それはテープに固執せるカバースリップの側面に静かに押します。イメージングの前に、50μlのPBSでチャンネルを湿ら。
- ゾル - ゲル及び角膜における画像の量子ドット。
- 厚い試料におけるFIONAイメージングのために、0.27ミリメートル、または0.28ミリメートルの作動距離を持つ60X水浸対物レンズの作動距離を持つ60X水浸対物レンズを使用しています。
- 顕微鏡でサンプルをマウントします。それはエピ蛍光モードに到達するようにTIRFレンズを調整する( すなわち、レーザビームがカバーガラスに対して角度対物から出てくる)。余分な倍率レンズ(3.3倍または4.0X)を挿入します。
- 希望z位置( 例えば、>200μm)を、対物レンズの焦点面を移動します。不動の記録画像サンプル中の量子ドット。
- このプロトコルのセクション2.5で説明したようにFIONA解析を実行します。
Access restricted. Please log in or start a trial to view this content.
結果
典型的な目的型TIRFMの設定は、 図3に示されている。まず、表面に固定化されたCy3-DNA試料を画像化した。代表的な画像が図4aに示されている。画像は、EMゲイン= 50、CCD感度=カメラの12.13と、露光時間を0.5秒とした。単一のCy3-DNA分子の点広がり関数(PSF)は、 図4bに示されるカラーバーは、ピクセル強度のスケールを示す( 図4aの矢印で示すス...
Access restricted. Please log in or start a trial to view this content.
ディスカッション
フィオナは1ミリ秒4〜8までナノメートルの精度と時間分解能を有する蛍光発光体(有機フルオロフォアまたは量子ドット)の位置を局所化する技術である。十分な光子が収集されると、この技術はより正確に回折限界(〜200 nm)を超える蛍光発光体の位置を決定することができ、したがって、この技術は、従来の/従来の光学顕微鏡4で見られなかったものを観察する道を開く<...
Access restricted. Please log in or start a trial to view this content.
開示事項
著者は、彼らが競合する金融利害がないことを宣言します。
謝辞
この作品は、NIHの助成金068625、NSF助成1063188と0822613.感謝はウサギの眼の贈り物のために先端科学技術のためのベックマン研究所で博士マリーナMarjanovicに行く生きている細胞の物理学センターによってサポートされていました。
Access restricted. Please log in or start a trial to view this content.
資料
| Name | Company | Catalog Number | Comments |
| Double-sided tape | 3M | ~75 µm thick | |
| EMCCD camera | Andor Technology | DU-897E-CS0-#BV | |
| Ultrasonic cleaner | Branson | 2510 | |
| Fluorescence filter set | Chroma | 49016 | |
| Actin polymerization buffer | Cytoskeleton | BSA02 | |
| Biotin G-actin | Cytoskeleton | AB07 | |
| G-actin | Cytoskeleton | AKL95 | |
| General actin buffer | Cytoskeleton | BSA01 | |
| Laser shutter (with driver) | Electro-Optical Products Corp. | SH-10-MP | |
| IDL | Exelis Visual Information Solutions | ||
| Neutravidin | Fisher Scientific | PI-31000 | |
| Coverslip | Fisherbrand | 22X30-1.5 | 0.16-0.19 mm thick |
| Microscope slide | Gold Seal Microslides | 30103X1 | 0.93-1.05 mm thick |
| Plasma cleaner | Harrick Plasma | PDC-001 | |
| Glass bottom dish | In Vitro Scientific | D35-20-1.5-N | |
| Cy3-DNA oligos | Integrated DNA Technologies | 5'-Cy3/GCCTCGCTGCCGTCGCCA-3'Bio | |
| Fluorescent beads | Invitrogen | T-7280 | |
| Qdot 605-streptavidin | Invitrogen | Q10101MP | |
| Qdot605 | Invitrogen | Q21301MP | |
| Qdot705 | Invitrogen | Q22021MP | |
| Qdot705 Antibody Conjugation Kit | Invitrogen | Q22061MP | |
| MATLAB | MathWorks | ||
| Optical table | Newport Corp | RS4000 Series | |
| 60X Objective | Nikon | Plan Apo VC 60x WI | |
| 100X Objective | Olympus | PlanApo 100X/1.45 Oil ∞/0.17 | |
| 60X Objective | Olympus | UPlanApo 60X/1.20W | |
| Inverted microscope | Olympus | IX71/IX70/IX81 | |
| Origin | OriginLab | ||
| Anti-FLAG antibody | Sigma Aldrich | F7425-.2MG | |
| ATP | Sigma Aldrich | A7699 | |
| BME | Sigma Aldrich | 63689-25ML-F | |
| BSA | Sigma Aldrich | A7906 | |
| BSA-biotin | Sigma Aldrich | A8549-10MG | |
| CK | Sigma Aldrich | C3755 | Creatine Phosphokinase from rabbit muscle |
| CP | Sigma Aldrich | P1937 | Phosphocreatine di(tris) salt |
| DTT | Sigma Aldrich | 43815 | DL-Dithiothreitol |
| EGTA | Sigma Aldrich | E3889 | Ethylene glycol-bis(2-aminoethylether)-N,N,N′,N′-tetraacetic acid |
| HCl | Sigma Aldrich | 93363-500G | |
| HEPES | Sigma Aldrich | H0887 | |
| KCl | Sigma Aldrich | P9333 | |
| MgCl2 | Sigma Aldrich | M1028 | |
| NaCl | Sigma Aldrich | S7653 | |
| PCA | Sigma Aldrich | 03930590 | Protocatechuic acid |
| PCD | Sigma Aldrich | P8279 | Protocatechuate-3,4-dioxygenase |
| TMOS | Sigma Aldrich | 341436-25G | Tetramethyl orthosilicate |
| Tris-HCl | Sigma Aldrich | 93363 | |
| Trolox | Sigma Aldrich | 238813 | 6-hydroxy-2,5,7,8-tetramethylchromane-2-carboxylic acid |
| 1” diameter broadband dielectric mirrors with mounts | Thorlabs | BB1-E02, KM100 | Quantity: 2 |
| ½” diameter posts | Thorlabs | TR4 | Quantity ≥ 6 |
| 10X beam expander | Thorlabs | BE10M-A | |
| 2” diameter f = 300 mm lens with mount | Thorlabs | LA1256-A, LMR2 | TIR lens |
| Fluorescent alignment target | Thorlabs | VRC2SM1 | |
| Laser safety goggles | Thorlabs | LG3 | |
| ND filter(s) | Thorlabs | FW1AND | |
| Optical beam profiler | Thorlabs | BP209-VIS | |
| Post-mounted iris diaphragm | Thorlabs | ID25 | Quantity: 2 |
| Shearing interferometer | Thorlabs | SI100 | |
| XYZ translation stage, ½” travel | Thorlabs | T12XYZ | |
| Laser | World Star Technologies | TECGL-30 | 532 nm, 30 mW |
参考文献
- Abbe, E. The Relation of Aperture and Power in the Microscope. Journal of the Royal Microscopical Society. 2 (3), 300-309 (1882).
- Abbe, E. The Relation of Aperture and Power in the Microscope (continued). Journal of the Royal Microscopical Society. 2 (4), 460-473 (1882).
- Thompson, R. Precise Nanometer Localization Analysis for Individual Fluorescent Probes. Biophysical Journal. 82 (5), 2775-2783 (2002).
- Yildiz, A., et al. Myosin V Walks Hand-Over-Hand: Single Fluorophore Imaging with 1.5-nm Localization. Science. 300 (5628), 2061-2065 (2003).
- Yildiz, A., Tomishige, M., Vale, R. D., Selvin, P. R. Kinesin Walks Hand-Over-Hand. Science. 303 (5658), 676-678 (2004).
- Yildiz, A., et al. Myosin VI Steps via a Hand-over-Hand Mechanism with Its Lever Arm Undergoing Fluctuations when Attached to Actin. Journal of Biological Chemistry. 279 (36), 37223-37226 (2004).
- Yildiz, A., Selvin, P. R. Fluorescence Imaging with One Nanometer Accuracy: Application to Molecular Motors. Accounts of Chemical Research. 38 (7), 574-582 (2005).
- Toprak, E., Yildiz, A., Hoffman, M. T., Rosenfeld, S. S., Selvin, P. R. Why kinesin is so processive. Proceedings of the National Academy of Sciences. 106 (31), (2009).
- Kural, C., et al. Tracking melanosomes inside a cell to study molecular motors and their interaction. Proceedings of the National Academy of Sciences. 104 (13), 5378-5382 (2007).
- Gordon, M. P., Ha, T., Selvin, P. R. Single-molecule high-resolution imaging with photobleaching. Proceedings of the National Academy of Sciences of the United States of America. 101 (17), 6462-6465 (2004).
- Qu, X., Wu, D., Mets, L., Scherer, N. F. Nanometer-localized multiple single-molecule fluorescence microscopy. Proceedings of the National Academy of Sciences of the United States of America. 101 (31), 11298-11303 (2004).
- Churchman, L. S., Ökten, Z., Rock, R. S., Dawson, J. F., Spudich, J. A. Single molecule high-resolution colocalization of Cy3 and Cy5 attached to macromolecules measures intramolecular distances through time. Proceedings of the National Academy of Sciences of the United States of America. 102 (5), 1419-1423 (2005).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-Dimensional Super-Resolution Imaging by Stochastic Optical Reconstruction Microscopy. Science. 319 (5864), 810-813 (2008).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3 (10), 793-796 (2006).
- Bates, M., Huang, B., Dempsey, G. T., Zhuang, X. Multicolor Super-Resolution Imaging with Photo-Switchable Fluorescent Probes. Science. 317 (5845), 1749-1753 (2007).
- Betzig, E., et al. Imaging Intracellular Fluorescent Proteins at Nanometer Resolution. Science. 313 (5793), 1642-1645 (2006).
- Abramoff, M. D., Magalhães, P. J., Ram, S. J. Image processing with ImageJ. Biophotonics international. 11 (7), 36-42 (2004).
- Enderlein, J., Toprak, E., Selvin, P. R. Polarization effect on position accuracy of fluorophore localization. Optics Express. 14 (18), 8111-8120 (2006).
- Cheezum, M. K., Walker, W. F., Guilford, W. H. Quantitative comparison of algorithms for tracking single fluorescent particles. Biophysical Journal. 81 (4), 2378-2388 (2001).
- Rasnik, I., McKinney, S. A., Ha, T. Nonblinking and long-lasting single-molecule fluorescence imaging. Nature Methods. 3 (11), 891-893 (2006).
- Zhuang, X., et al. A Single-Molecule Study of RNA Catalysis and Folding. Science. 288 (5473), 2048-2051 (2000).
- Aitken, C. E., Marshall, R. A., Puglisi, J. D. An Oxygen Scavenging System for Improvement of Dye Stability in Single-Molecule Fluorescence Experiments. Biophysical Journal. 94 (5), 1826-1835 (2008).
Access restricted. Please log in or start a trial to view this content.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved