このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
メチルトランスフェラーゼと補因子類似体核酸およびタンパク質の配列特異的標識化
要約
DNA及びタンパク質は、配列特異的親和性またはDNAまたはタンパク質トランスフェラーゼおよび合成補因子類似体を用いた蛍光レポーター基で標識される。酵素の補因子特異性に応じて、アジリジンまたは二活性化補助因子アナログは、一次元または二段階標識のために用いられる。
要約
S -Adenosyl -1-メチオニンメチオニン(AdoMetまたはSAM)依存性メチルトランスフェラーゼ(MTアーゼ)、DNA、RNA、タンパク質および小生体分子の特定の位置にあるAdoMetから活性化メチル基の転移を触媒する。この天然のメチル化反応は、補因子の合成類似体を用いてアルキル化反応の多種多様に拡張することができる。アジリジン環とのAdoMetの反応性のスルホニウム中心の交換は、様々なDNAメチルトランスフェラーゼによって、DNAと結合することができる補因子につながる。これらのアジリジン補助因子は、アデニン部分の異なる位置にレポーター基を搭載し、 私は DNA(笑顔DNA) のLアベルのINGを nduced ethyltransferase- のS equence固有のMのために使用することができる。典型的な例として、我々は、DNA MTアーゼM.BseCIとアジリジン補助因子6BAzのある5'-ATCG A T-3 '配列でpBR322プラスミドDNAのビオチン化のためのプロトコルを与える1ステップ。 ctivated Gの roups(MTAG) のm個のethyltransferase指向Tの ransferのために使用されるのAdoMet類似体の別のクラスの不飽和アルキル基の結果と活性化されたメチル基の拡張。拡張された側鎖はスルホニウム中心と不飽和結合によって活性化されるので、これらの補因子は、二重活性化のAdoMet類似体と呼ばれる。これらの類似体アジリジン補因子のようなDNAメチルトランスフェラーゼのための補因子としての機能だけでなく、RNA、タンパク質および小分子MTアーゼのみならず。これらは、典型的には、第2の化学工程でレポーター基で標識されているユニークな官能基を有するMTアーゼ基質の酵素的修飾に使用される。これは、ヒストンH3タンパク質の蛍光標識のためのプロトコルに例示されている。小さなプロパルギル基はのクリックラベルに続いてヒストンH3リジン4(H3K4)MTアーゼSET7 / 9によってタンパク質に補因子アナログSeAdoYnから転送されるTAMRAアジドとアルキニル化ヒストンH3。補因子類縁体とMTアーゼ媒介ラベリングは、識別とMTアーゼ基質の機能的研究ならびにDNAジェノタイピングとメチル化検出を含む多くのエキサイティングなアプリケーションを可能にする技術である。
概要
核酸、1,2およびタンパク質3,4の特異的な標識は、機能の特徴付け、医療診断及び(ナノ)バイオテクノロジーのための主要な関心事である。ここでは、S -adenosyl-L-メチオニン(AdoMetをまたはSAM)依存性メチルトランスフェラーゼ(MTアーゼ)に基づいており、これらの生体高分子のための酵素標識法を提示する。このクラスの酵素(EC 2.1.1。)は、核酸およびタンパク質の特定の残基の中の個々の求核位置(窒素、酸素、硫黄および炭素原子)を標的と自然補因子のAdoMet( 図1A)5の活性化メチル基を転移する。また、MTアーゼは、親和性タグ、蛍光団又は他の標識( 図1B)6との特異的標識のための合成補因子類縁体を利用することができます。のAdoMet類似体の2つのクラスが開発されてきた:Sの equence固有のM ethyltransferase-ためのアジリジン補助因子を私が nduced Lのアベルる (笑顔)7とctivated のG roups のM ethyltransferase指向性T ransferのための二重活性化したのAdoMet類縁体(MTAG)8。

図1:メチルトランスフェラーゼ(MTアーゼ)によって触媒される反応は 、DNA、RNA、タンパク質および小生体分子を含む様々な基材への天然の補因子のAdoMet(SAM)からA.メチル基転移B.標識核酸およびタンパク質の/官能化(NNNNN =。 DNAのための塩基対、タンパク質についてのRNAおよびアミノ酸に対するヌクレオチド;緑色対象残基とMTアーゼのXXXXX =認識配列)合成補因子類似体。レポーター基を含むアジリジン補助因子(青球)特に標的残基に結合された配列は、(左)アデニン環にある取り付け及び二活性化のAdoMet類似体は、第二段階でバイオ直交クリック反応により標識することができる化学レポーターY(右)を担持する拡張アルキル鎖の転送につながる。 この図の拡大版をご覧になるにはこちらをクリックしてください。
アジリジン補助因子は、DNAメチルトランスフェラーゼで最適に動作する。それらは窒素原子9(またはNが 10,11 -mustard)の代わりに、スルホニウム中心の反応性基を有する3員環を含む。この窒素原子のプロトン化は、DNAと全体の補因子の共有結合につながる標的ヌクレオチドによる求核攻撃のためのアジリジン環を活性化させる。アデニン環にレポーター基を結合することにより、アジリジン補因子は、(一段階でDNAを標識するためにDNAメチルトランスフェラーゼと組み合わせて使用することができる G>左図1B)7,12。 、 バチルス·ステアロサーモフィラス (M.BseCI)16( 図2から図15(アデニン環の6位に結合したビオチンとアジリジン補助因子)およびアデニン特異的DNA MTアーゼ-これは6BAz 13によるDNAのビオチン化のために詳細に示されているアジリジン補助因子を介した DNAのワンステップのラベリング):プロトコルセクション2を参照してください。 ヘモフィルスheamolyticus(M.HhaI、5からM.BseCI(5'-ATCG A T-3 '認識配列)、 サーマス·アクアティクス (M.TaqI、5'-TCG A -3からDNA MTアーゼ')に加えて、 「-G C GC-3 ')およびスピロ (M.SssI、5'- C G-3')から正常に6BAz 17とのDNAをビオチン化するために使用されている。さらに、アジリジン補因子は、1ステップの蛍光DNA標識18,19のために使用することができる。
常に ">:" =キープtogether.withinページFO」ontent
図2:M.BseCIと6BAzとDNAの配列特異的一工程ビオチン化 DNA MTアーゼM.BseCIは、二本鎖DNA配列5'-ATCG A T-3 'を認識し、自然に二アデニンのアミノ基をメチル化のAdoMetを使用して残留物(緑色)。アジリジン補助因子6BAzとの反応のコースが変更され、M.BseCIは、ターゲットアデニンとビオチン(青)を含めた全体の補因子を結合することによって、特定のDNAビオチン化を配列決定するためにつながる。 この図の拡大版をご覧になるにはこちらをクリックしてください。
二重活性化のAdoMet類似体は、不飽和側鎖の代わりに、スルホニウム中心におけるメチル基( 図1B拡張含む、右)20。スルホニウム中心部へのβ位に不飽和二重または三重結合は、電子的に接合安定化により遷移状態の中不利な立体効果を補償します。スルホニウム中心と不飽和結合の両方が酵素転送のための側鎖を活性化するので、これらの補因子は、二重活性化のAdoMet類似体と命名した。典型的には、それらは、第二工程8,21において化学選択的標識のために、アミノ、アルキンとアジド基のようなユニークな化学基(化学レポーター)で側鎖を転送するために使用される。 RNAおよびタンパク質の追加のラベル付けを可能28 -一般的には、二重の活性化のAdoMet類似体は、RNA MTアーゼ22,23 DNA MTアーゼ8,20,21のためだけでなく、ための補因子およびタンパク質のMTアーゼ24としてだけではなく機能することができます。しかし、拡張された側鎖が立体的にメチル基より厳しいであり、タンパク質工学によってMTアーゼ活性部位を拡大することはしばしばある効率的な転送速度を得るために必要なEN。銅による化学的修飾を、以下のための酵素の転写時の遷移状態の1安定化および2反応性ハンドル:この問題に対する別の解決策は、小さなプロパルギル末端アルキンは、2つの機能を果たす基(3個の炭素)とのAdoMet類似体を使用することである触媒によるアジド - アルキン環(CuAAC)ケミストリーをクリックします。それは、結果のプロパルギルのAdoMet類縁体29は 、中性またはわずかに塩基性条件下でのみ限定された使用の非常に不安定であることが判明した。この欠点は、セレンと硫黄原子を置換することによって固定することができる。得られた補因子5 ' - [( セレン )[(3 S)-3-アミノ-3-カルボキシプロピル]プロプ-2- ynylselenonio] -5'-デオキシアデノシン(SeAdoYn、 図3)は、野生型DNAによって受け入れられ、RNA多くの場合、タンパク質工学の必要性を排除32 -とタンパク質MTアーゼ30。これは、蛍光プロ例示されるヒストンH3リジン4(H3K4)MTアーゼSET7 / 9 33(:二重に活性化補助因子を介して 2つの段階のタンパク質標識、図3、プロトコルセクション3を参照)での標識をTEIN。
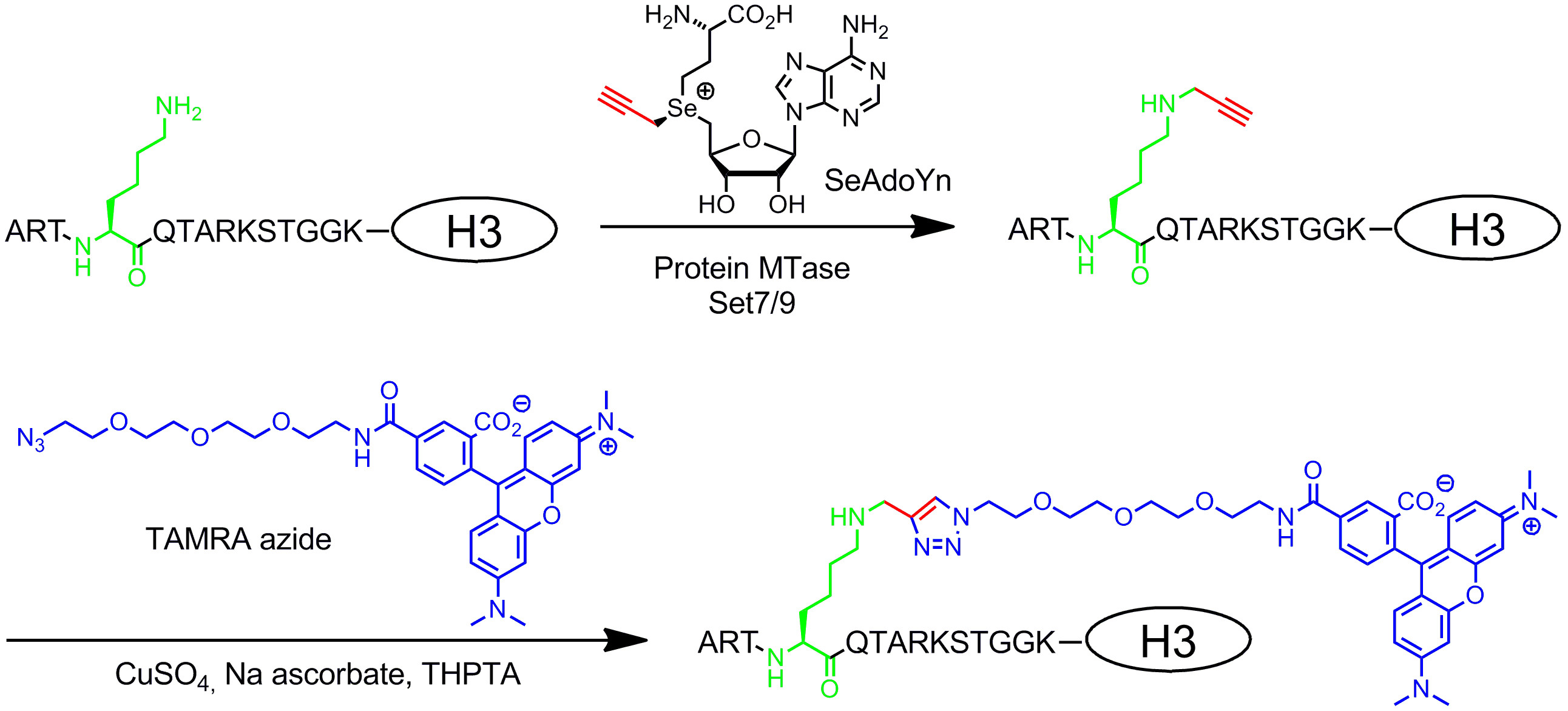
図3:SET7 / 9、SeAdoYnとTAMRAアジドとヒストンH3の配列特異的二段階蛍光標識タンパク質MTアーゼSET7 / 9が自然のAdoMetを使用して、ヒストンH3のリジン4のアミノ基(H3K4、緑)をメチル化する。。二重活性化補助因子でSeAdoYn MTアーゼは、リジン残基に小さなプロパルギル基(赤)を転送する。接続された端末三重結合は、その後選択的にアジド誘導体化TAMRA(テトラメチルローダミン、青)フルオロフォアでバイオ直交クリック反応(銅触媒アジド - アルキン付加環、CuAAC)に変更されます。ロード/ 52014 / 52014fig3highres.jpg「ターゲット= "_空白">この図の拡大版をご覧になるにはこちらをクリックしてください。
プロトコル
1.一般手順
- ストアアジリジン補助因子(DMSO中)6BAzとタンパク質MTアーゼ-80℃でSET7 / 9と二重活性化補助因子SeAdoYnとDNA MTアーゼM.BseCI -20℃(50%グリセロール)を含むすべての他の試薬。
- 脱イオン水に吸光係数ε269nmで (6BAz)= 16000センチメートル-1 M -1およびε260nmの (SeAdoYn)= 15400センチメートル-1 M -1用いたUV /可視分光法を経由して 6BAzとSeAdoYnの濃度を決定する。吸光係数は、280nmでの直接吸収を介して利用可能である場合には、ブラッドフォードアッセイによってMTアーゼの濃度を決定するか。
- 酵素活性の損失を防ぐために、集中的なペッティングまたはボルテックスにより気泡を作成しないようにしてください。その代わりに、優しく上下にピペッティングにより混合します。
- DMSO中のストック溶液からアジリジン補助因子を追加する場合はアッセイにおける最終DMSO濃度は低いことを確認してください5%未満。常にDNAとの非特異的反応を防止するアッセイ緩衝液中の10mMのマグネシウムイオンが挙げられる。
- 酸性のストック溶液から二重に活性化補助因子を追加する場合はpH変化を回避し、アッセイ溶液のpHが大きく変化しないことを確認するために小容量(高濃縮ストック溶液)を使用します。彼らは必要な銅イオンの錯体形成によってクリック反応を妨害することができるので、アッセイ緩衝液中で、チオールを避けるため、 例えば 、βメルカプトエタノールまたはジチオスレイトール(DTT)。
アジリジン補因子を介した DNAの2つのステップのラベル
- M.BseCI DNA MTアーゼおよびアジリジン補助因子6BAzプラスミドDNAの配列特異的メチルトランスフェラーゼ誘発ラベルING(笑顔)。
- 20℃における補因子ソリューションを解凍し、氷上で反応混合物を準備します。
- 検定に加えて任意の非固有の変更を可視化するために、「補因子」の制御を行う、とAN&#8220。酵素」コントロールは、MTアーゼの準備は、自然補因子のAdoMetがないことを確認します。
- (100 mMトリス-塩酸を含む、100のMgCl 2、20 mMのβメルカプトエタノール、pH7.4)で10倍の修正バッファ、pBR322の2μL(0.5μgの/μL)、10当量のアッセイミックス2μlのために。 DNA上の認識配列(pBR322の中で1認識配列)と20μlの総体積内の60μMの最終濃度にアジリジン補助因子6BAzあたりM.BseCI。最後の補因子とDNA MTアーゼを追加します。
注:βメルカプトエタノールは、毒性、腐食性、環境に有害である。 - 「補因子」制御のために代わりにM.BseCIの脱イオン水を追加し、「酵素」コントロールには6BAzの代わりに脱イオン水を追加します。
- 優しく上下にピペッティングすることによって溶液を混合。
- 1時間55℃でチューブをインキュベートします。
- チューブの底にすべての液体を回収するための遠心分離を簡単に。
- DNA修飾を確認するための制限修飾アッセイ。
- 、80μlの脱イオン水および3.3液(100mMのトリス-HCl、50mMのMgCl 2を 、1 MのNaCl、1mg / mlのウシ血清アルブミン、pHが8.0を含む)10μlの10×R.TaqI緩衝液を混合して溶液を調製サーマス·アクアティクス (R.TaqI、10 U /μL)から制限エンドヌクレアーゼ(REase)。最後のステップでREaseを追加してください。
- 各チューブに2.1.7から10倍のR.のTaqIバッファと上から溶液(2.2.1)の28μLの2μlを添加する。
- 優しく上下にピペッティングすることによって溶液を混合。
- 30分間65℃でチューブをインキュベートします。
- チューブの底にすべての液体を回収するための遠心分離を簡単に。
- ストレプトアビジンとの電気泳動移動度シフトアッセイ(EMSA)は、機能的な変更を確認します。
- 各チューブ(2.2.5)からの25μlのを削除し、ストレプトアビジンMに対するストレプトアビジン溶液(1 mMと2.4μlを添加するonomerの100mMのNa 2 HPO 4を含有するストレプトアビジン緩衝液中で、100mMのNaCl、pH7.5で、総ビオチンの4当量)。残りのチューブにストレプトアビジンバッファーの2.4を添加する。
- 1時間、37℃ですべてのチューブをインキュベートします。
- アガロースゲル電気泳動による分析。
- 各チューブに6Xローディングバッファー(0.25%ブロモフェノールブルー、30%グリセロール)の5μlを添加する。
- 優しく溶液を混合。
- 負荷(10,000倍ストック溶液から1×GelRedを含む0.5×TBE緩衝液中の1%アガロース)アガロースゲルのウェルに10μlの各サンプル。
- 約80 Vで0.5×TBE緩衝液でゲルを実行します。 1時間。
- フィルタ(540±50 nm)とを備えたCCDカメラ(312 nm)のUVテーブル上でDNAバンドを可視化する。
注:UV光が目や皮膚に損傷を与えている。
ダブル活性化補助因子を介した 3つの段階のタンパク質標識
- MethyltransfヒストンH3リジン4ラベリング(改質工程)のためのSET7 / 9と二重活性化補助因子SeAdoYnで活性化グループ(MTAG)の譲渡を消去し、指示した。
- コンポーネントを解凍し、氷上で反応混合物を準備します。注:必ずSeAdoYn劣化を避けるために冷却して続ける。
- アッセイに加えて、任意の非特異的改変を可視化するために、「補因子」制御を実行し、「酵素」制御は、蛍光プローブの非特異反応を除外する。
- 修正緩衝液(50mMのTris-HCl、5%グリセロール、pH8.5)中、10μMヒストンH3、10μMSET7 / 9及び600μMSeAdoYn(セレンの両エピマーの混合物)を含有するアッセイ溶液(20μl)を調製する。最後のステップでは、次に補因子とMTアーゼを追加します。
- 「補因子」コントロール3.1.3のようにアッセイ溶液を準備し、合成補助因子と競争する60 mMののAdoMetを追加します。 「酵素」制御のために代わりにSeAdoYnの脱イオン水を追加します。
- ゆっくり上下にピペッティングすることによって溶液を混合。 pHのストリップ( - 10 pH範囲5)の上部のフィールドに各溶液1μlを添加してpHを確認してください。
- 2時間37℃でインキュベートする。
- 1:0.1%(w / v)のAPS、0.04%の(v / v)のTEMED 12%アクリルアミド/ビスアクリルアミド37.5 357 mMのビス - トリスpHが6.5〜6.8、:一方のゲルを実行して(12%SDSポリアクリルアミドゲルを調製で;ローディングゲル:357 mMのビス - トリスpHが6.5〜6.8、0.1%APS(w / v)の、0.04%(v / v)のTEMEDと5%アクリルアミド/ビスアクリルアミド37.5:1)。
注:アクリルアミド/ビスアクリルアミドは毒性と健康に危険である。この手順の間に手袋を着用してください。
- 銅触媒アジド-アルキン環(CuAAC)(標識工程) を介してヒストンH3でalkinylatedリジン4の化学標識。
- 修飾反応の終了は、3 mMのCuSO 4を、3 mMのトリス(3-ヒドロキシプロピルトリアゾリル)アミン(THPTA)、250mMのアスコルビン酸ナトリウムおよび6 mMのTAMRAアジドを含有する5倍クリックミックスを調製する直前20μlの総容量。
- CuAACを開始し、修飾反応をクエンチするために各チューブに調製したばかりの5倍のクリックミックスの5μlを添加する。
- ピペッティングにより穏やかに混合します。
- フォアの光退色を避けるために、光からアルミホイルで全てのチューブを保護します。
- 1時間20℃でインキュベートする。
- 無料のTAMRAフルオロフォアの過剰を除去するタンパク質沈殿。
- 34 -無料のTAMRAの蛍光団の集中的なゲル内蛍光によって蛍光ラベルヒストンH3のoutshiningを回避するために、タンパク質(3.3.4 3.3.2)の沈殿によって過剰蛍光団を取り除く。
- 簡単に各添加後、各チューブとボルテックスに75μlのメタノール、18.8μlのクロロホルムと50μlの脱イオン水を追加します。 5分間16000×gで遠心分離する。タンパク質が含まれている界面層を、乱すことなく上相を除去します。
- 私は残りの相に56.3μlのメタノールを追加します。nは5分間16000×gで各チューブ、ボルテックスと遠心機は、タンパク質をペレット化する。上清を取り除きます。ペレットを洗浄するには、この手順を繰り返します。
- 糸くずの出ない組織とのオープンチューブをカバーし、15のためにそれらを乾燥させ - 30分。
- SDS PAGE による分析。
- (20μlのSDSローディング緩衝液(50mMトリス-HCl、2.5%(w / v)のSDS、10%(v / v)グリセロール、320 mMのβメルカプトエタノールおよび0.05%で3.3.4から沈殿したタンパク質を溶解/ワットv)のブロモフェノールブルー、pHは6.8)。完全にピペットでチューブの壁をすすぐことによりペレットを溶解することを確認してください。
- 10分間95℃でサンプルをインキュベートし、それらを20℃まで冷却しましょう。
- チューブの底にすべての液体を回収するための遠心分離を簡単に。
- SDSポリアクリルアミドゲル(3.1.7)のウェルに各サンプルの全量をロードします。電気泳動用のバッファを実行しているようにSDSを(w / v)の50 mMのMOPS、50mMのTris-X(Tris塩基)、5mMのEDTA、0.1%を使用してください。
- 約120 Vでゲルを実行します。 90分。
- フィルタ(50ナノメートルの波長±540)を備えたCCDカメラを用いてUVテーブル(312 nm)の上のゲル内蛍光を可視化する。
注:UV光が目や皮膚に損傷を与えている。
結果
アジリジン補因子を介したDNAのワンステップラベリング
この例では、反応は、二本鎖の5'-ATCG A T-3 '配列内の第2のアデニン残基を変更し、pBR322プラスミド( 図4A)上の1つの認識部位を持つDNA MTアーゼM.BseCI、を用いて行われる。プラスミド標識をテストするために、pBR322の制限エンドヌクレアーゼ(REase)R.TaqI(5'-TCGA-3 ')を用?...
ディスカッション
のDNA MTアーゼおよびアジリジン補助因子(笑顔DNA)を用いたDNAのワンステップ標識は堅牢な方法であるが、実験を計画する際にいくつかの側面を考慮すべきである。
アジリジン補助因子:M.BseCIとDNA標識用6BAz濃度は60であった。他のDNAメチルトランスフェラーゼを使用する場合は補因子の濃度は、20μMのがDNA MTアーゼM.TaqI 19で採用されていると低い例え?...
開示事項
The authors disclose the following competing financial interest: E.W. is inventor on related patents.
謝辞
The authors thank Kerstin Glensk for preparing the MTases M.BseCI and Set7/9 and gratefully acknowledge funding by the Excellence Initiative of the German Federal and State Governments and RWTH Aachen University. The authors are happy to provide 6BAz and SeAdoYn or other cofactor analogues for collaborative research.
資料
| Name | Company | Catalog Number | Comments |
| 6BAz | Synthesized according to Weinhold et al., Patent number US 8,129,106, published March 6, 2012. | ||
| β-Mercaptoethanol | Serva | 28625 | |
| Acetic acid | Fisher Scientific | 10304980 | |
| Acrylamide/Bis Solution, 37.5:1 | Serva | 10688 | |
| UltraPure Agarose | Invitrogen | 16500100 | |
| Ammonium persulfate (APS) | Serva | 13375 | |
| Bis-Tris | Gerbu | 1304 | |
| Boric acid | Gerbu | 1115 | |
| Bromophenol blue Na salt | Serva | 15375 | |
| Copper(II) sulfate | Aldrich | C1297 | |
| Chloroform | Fisher Scientific | 10020090 | |
| Coomassie Brilliant Blue | Serva | 17525 | |
| EDTA disodium salt | Gerbu | 1034 | |
| Ethanol | Merck | 100983 | |
| GelRed (10,000x in water) | Biotium | 41003 | |
| Glycerol (99.5%) | Gerbu | 2006 | |
| FastRuler Low Range DNA Ladder | Thermo Scientific | SM1103 | |
| Histone H3 | Expression plasmid obtained from Dr. Philipp Voigt and Prof. Danny Reinberg; expression and isolation according to T. J. Richmond et al., J. Mol. Biol. 1997, 272, 301-311. | ||
| M.BseCI | Expression plasmid obtained from Dr. Michael Kokkinidis; expression and isolation according to Kapetaniou et al., Acta Cryst. 2006, F63, 12-14. | ||
| Methanol | Fisher Scientific | 10675112 | |
| Magnesiumchloride hexahydrate | J.T. Baker | 4003 | |
| MOPS | Gerbu | 1081 | |
| Sodium chloride | Gerbu | 1112 | |
| pH strip (Neutralit) | Merck | 1,095,330,001 | |
| pBR322 | Thermo Scientific | SD0041 | |
| R.TaqI (10 u/µl) | Thermo Scientific | ER0671 | |
| SeAdoYn | Synthesized according to Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| Set7/9 | Expression plasmid obtained from Prof. Danny Reinberg, expression and isolation according to D. Reinberg et al., Genes Dev.2002, 16, 479-489. | ||
| Streptavidin | Gerbu | 3058 | |
| (+)-Sodium L-ascorbate | Sigma Life Science | A7631 | |
| SDS Granular | Gerbu | 1833 | |
| di-Sodium hydrogenphosphate | Merck | 106,586 | |
| TAMRA azide | Synthesized according to reference 30: Willnow et al., ChemBioChem 2012, 13, 1167-1173. | ||
| TaqI buffer (10x) | Thermo Scientific | B28 | |
| N,N,N',N'-Tetramethylethylenediamine (TEMED) | Acros Organics | 42058 | |
| Tris-HCl | Gerbu | 1028 | |
| Tris-X (TRIS-base) | Gerbu | 1018 | |
| Tris(3-hydroxypropyltriazolyl-methyl)amine (THPTA) | Sigma-Aldrich | 762342 |
参考文献
- Gottfried, A., Weinhold, E. Sequence-specific covalent labelling of DNA. Biochem. Soc. Trans. 39, 623-628 (2011).
- Zohar, H., Muller, S. J. Labeling DNA for single-molecule experiments: methods of labeling internal specific sequences on double-stranded DNA. Nanoscale. 3, 3027-3039 (2011).
- Hinner, M. J., Johnsson, K. How to obtain labeled proteins and what to do with them. Curr. Opin. Biotechnol. 21, 766-776 (2010).
- Wua, Y. -. W., Goody, R. S. Probing protein function by chemical modification. J. Pept. Sci. 16, 514-523 (2010).
- Struck, A. -. W., Thompson, M. L., Wong, L. S., Micklefield, J. S-Adenosyl-methionine-dependent methyltransferases: Highly versatile enzymes in biocatalysis, biosynthesis and other biotechnological applications. ChemBioChem. 13, 2642-2655 (2012).
- Klimasauskas, S., Weinhold, E. A new tool for biotechnology: AdoMet-dependent methyltransferases. Trends Biotechnol. 25, 99-104 (2007).
- Pljevaljcic, G., Schmidt, F., Weinhold, E. Sequence-specific Methyltransferase-Induced Labeling of DNA (SMILing DNA). ChemBioChem. 5, 265-269 (2004).
- Lukinavicius, G., Lapiene, V., Stasevskij, Z., Dalhoff, C., Weinhold, E., Klimasauskas, S. Targeted labeling of DNA by methyltransferase-directed Transfer of Activated Groups (mTAG). J. Am. Chem. Soc. 129, 2758-2759 (1021).
- Pignot, M., Siethoff, C., Linscheid, M., Weinhold, E. Coupling of a nucleoside with DNA by a methyltransferase. Angew. Chem. Int. Ed. 37, 2888-2891 (1998).
- Weller, R. L., Rajski, S. R. Design, synthesis, and preliminary biological evaluation of a DNA methyltransferase-directed alkylating agent. ChemBioChem. 7, 243-245 (2006).
- Du, Y., Hendrick, C. E., Frye, K. S., Comstock, L. R. Fluorescent DNA Labeling by N-Mustard Analogues of S-adenosyl-l-methionine. ChemBioChem. 13, 2225-2233 (2012).
- Pljevaljcic, G., Schmidt, F., Scheidig, A. J., Lurz, R., Weinhold, E. Quantitative labeling of long plasmid DNA with nanometer precision. ChemBioChem. 8, 1516-1519 (1002).
- Wilkinson, S., et al. Molecular scale architecture: engineered three- and four-way junctions. Bioconjugate Chem. 19, 470-475 (2008).
- Braun, G., et al. Enzyme-directed positioning of nanoparticles on large DNA templates. Bioconjugate Chem. 19, 476-479 (2008).
- Kim, S., et al. Enzymatically incorporated genomic tags for optical mapping of DNA binding proteins. Chem. Int. Ed. 51, 3578-3581 (2012).
- Rina, M., Bouriotis, V. Cloning purification and characterization of the BseCI DNA methyltransferase from Bacillus stearothermophilus. Gene. 133, 91-94 (1993).
- Weinhold, E., Meier, T., Düfel, H., Markert-Hahn, C., Schmuck, R. Sequence-specific detection of methylation in biomolecules. US Patent. , (2012).
- Pljevaljcic, G., Pignot, M., Weinhold, E. Design of a new fluorescent cofactor for DNA methyltransferases and sequence-specific labeling of DNA. J. Am. Chem. Soc. 125, 3492-3410 (2003).
- Schmidt, F. H. -. G., Hüben, M., Gider, B., Renault, F., Teulade-Fichou, M. -. P., Weinhold, E. Sequence-specific Methyltransferase-Induced Labelling (SMILing) of plasmid DNA for studying cell transfection. Bioorg. Med. Chem. 16, 40-48 (2008).
- Dalhoff, C., Lukinavicius, G., Klimasauskas, S., Weinhold, E. Direct transfer of extended groups from synthetic cofactors by DNA methyltransferases. Nat. Chem. Biol. 2, 31-32 (2006).
- Lukinavicius, G., Tomkuviene, M., Masevicius, V., Klimasauskas, S. Enhanced chemical stability of AdoMet analogues for improved methyltransferase-directed labeling of DNA. ACS Chem. Biol. 8, 1134-1139 (2013).
- Motorin, Y., et al. Expanding the chemical scope of RNA:methyltransferases to site-specific alkynylation of RNA for click labeling. Nucleic Acids Res. 39, 1943-1952 (1943).
- Schulz, D., Holstein, J. M., Rentmeister, A. A chemo-enzymatic approach for site-specific modification of the RNA cap. Angew. Chem. Int. Ed. 52, 7874-7878 (2013).
- Peters, W., et al. Enzymatic site-specific functionalization of protein methyltransferase substrates with alkynes for click labeling. Angew. Chem. Int. Ed. 49, 5170-5173 (2010).
- Islam, K., Zheng, W., Yu, H., Deng, H., Luo, M. Expanding cofactor repertoire of protein lysine methyltransferase for substrate labeling. ACS Chem. Biol. 6, 679-684 (2011).
- Wang, R., Zheng, W., Yu, H., Deng, H., Luo, M. Labeling substrates of protein arginine methyltransferase with engineered enzymes and matched S-adenosyl-l-methionine analogues. J. Am. Chem. Soc. 133, 7648-7651 (2011).
- Islam, K., et al. Bioorthogonal profiling of protein methylation using azido derivative of S-adenosyl-l-methionine. J. Am. Chem. Soc. 134, 5909-5915 (2012).
- Islam, K., et al. Defining efficient enzyme-cofactor pairs for bioorthogonal profiling of protein methylation. Proc. Natl. Acad. Sci. U.S.A. 110, 16778-16783 (2013).
- Binda, O., Boyce, M., Rush, J. S., Palaniappan, K. K., Bertozzi, C. R., Gozani, O. A chemical method for labeling lysine methyltransferase substrates. ChemBioChem. 12, 330-334 (2011).
- Willnow, S., Martin, M., Lüscher, B., Weinhold, E. A selenium-based click AdoMet analogue for versatile substrate labeling with wild-type protein methyltransferases. ChemBioChem. 13, 1167-1173 (2012).
- Bothwell, I. R., et al. Se-Adenosyl-l-selenomethionine cofactor analogue as a reporter of protein methylation. J. Am. Chem. Soc. 134, 14905-14912 (2012).
- Tomkuviene, M., Clouet-d’Orval, B., Cerniauskas, I., Weinhold, E., Klimasauskas, S. Programmable sequence-specific click-labeling of RNA using archaeal box C/D RNP methyltransferases. Nucleic Acids Res. 40, 6765-6773 (2012).
- Nishioka, K., et al. Set9, a novel histone H3 methyltransferase that facilitates transcription by precluding histone tail modifications required for heterochromatin formation. Genes Dev. 16, 479-489 (2002).
- Clark, P. M., et al. Direct in-gel fluorescence detection and cellular imaging of O-GlcNAc-modified proteins. J. Am. Chem. Soc. 130, (2008).
- Lukinavicius, G., Lapinaite, A., Urbanaviciute, G., Gerasimaite, R., Klimasauskas, S. Engineering the DNA cytosine-5 methyltransferase reaction for sequence-specific labeling of DNA. Nucleic Acids Res. 40, 11594-11602 (2012).
- Neely, R. K., Dedecker, P., Hotta, J., Urbanaviciute, G., Klimasauskas, S., Hofkens, J. DNA fluorocode: A single molecule, optical map of DNA with nanometre resolution. Chem. Sci. 1, 453-460 (2010).
- Roberts, R. J., Vincze, T., Posfai, J., Macelis, D. REBASE-a database for DNA restriction and modification: enzymes, genes and genomes. Nucleic Acids Res. 38, 234-236 (2010).
- Petrossian, T. C., Clarke, S. G. Uncovering the human methyltransferasome. Mol. Cell. Proteomics. 10, 1-12 (2011).
- Kriukiene, E., et al. DNA unmethylome profiling by covalent capture of CpG sites. Nat. Commun. 4, 2190 (2013).
- Wang, R., et al. Profiling genome-wide chromatin methylation with engineered posttranslation apparatus within living cells. J. Am. Chem. Soc. 135, 1048-1056 (2013).
- Zhang, C., Weller, R. L., Thorson, J. S., Rajski, S. R. Natural product diversification using a non-natural cofactor analogue of S-adenosyl-l-methionine. J. Am. Chem. Soc. 128, 2760-2761 (2006).
- Stecher, H., et al. Biocatalytic Fiedel-Crafts alkylation using non-natural cofactors. Angew. Chem. Int. Ed. 48, 9546-9548 (2009).
- Lee, B. W. K., Sun, H. G., Zang, T., Kim, B. J., Alfaro, J. F., Zhou, Z. S. Enzyme-catalyzed transfer of a ketone group from an S-adenosylmethionine analogue: A tool for the functional analysis of methyltransferases. J. Am. Chem. Soc. 132, 3642-3643 (2010).
- Winter, J. M., et al. Expanding the structural diversity of polyketides by exploring the cofactor tolerance of an inline methyltransferase domain. Org. Lett. 15, 3774-3777 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved