Method Article
万HeLa細胞でエピジェネティックプロファイルを生成するためのChIP-seqの実験の自動化
要約
Methods for mapping in vivo protein-DNA interactions are becoming crucial for every aspect of genomic research but they are laborious, costly, and time consuming. Here a commercially available robotic liquid handling system that automates chromatin immunoprecipitation for mapping in vivo protein-DNA interactions with limited amounts of cells is presented.
要約
Chromatin immunoprecipitation followed by next generation sequencing (ChIP-seq) is a technique of choice for studying protein-DNA interactions. ChIP-seq has been used for mapping protein-DNA interactions and allocating histones modifications. The procedure is tedious and time consuming, and one of the major limitations is the requirement for high amounts of starting material, usually millions of cells. Automation of chromatin immunoprecipitation assays is possible when the procedure is based on the use of magnetic beads. Successful automated protocols of chromatin immunoprecipitation and library preparation have been specifically designed on a commercially available robotic liquid handling system dedicated mainly to automate epigenetic assays. First, validation of automated ChIP-seq assays using antibodies directed against various histone modifications was shown, followed by optimization of the automated protocols to perform chromatin immunoprecipitation and library preparation starting with low cell numbers. The goal of these experiments is to provide a valuable tool for future epigenetic analysis of specific cell types, sub-populations, and biopsy samples.
概要
次世代シーケンシング(NGS)技術が広く普及し、より身近になってきたように、今転写因子の発見を可能にNGS検出(チップ配列)、続いてクロマチン免疫沈降されたタンパク質-DNA相互作用のゲノムワイドなマッピングのための主な方法ヒストン修飾のサイトやパターンを結合。のChIP-seqが濃縮されたDNA断片の測定によるタンパク質-DNA相互作用の定量的および定性的な分析のために使用することができる全ゲノムの高スループットのデータを提供する点で有利である。しかし、このようなシーケンシングライブラリーを作成するために十分な材料を得ることが困難であるなどの標準のChIP-seqの実験において、いくつかの欠点がある。
ChIP実験は、細胞溶解および免疫複合体の超音波処理によるクロマチン剪断、3)形成を含む2)試料調製1)架橋タンパク質のDNA結合領域を含む6つの基本的なステップに分割されている免疫複合体の4)沈殿、5)免疫複合体の洗浄、及び定量PCRとNGSによる6)濃縮された物質の溶出および分析。
良好なクロマチン調製、元のサンプル中の抗原の量、およびその同族抗原に対する抗体の特異性および親和性:ChIPアッセイの成功は、3つの主な要因に依存する。主な制限は、シーケンシングライブラリーを作成するのに十分な濃縮されたDNAを得るために、細胞数を開始大量のための要件である。そのような生検サンプルまたは細胞亜集団などの限られたサンプル量、で動作する科学者のために、チップ-seqの実験は非常に挑戦的である。セル1の量が少ない、2を操作するときに最近の研究では、のChIP-seqのアッセイを行うことができることを示した。Diagenodeのは限られた数の細胞で開始するときに、完全のChIP-seqの実験を自動化することができるロボット液体ハンドリングシステムを開発した。
オートメーション提供のChIP-seqのサンプルのマニュアル作成に比べて多くの利点は、人為的ミスを減少させるように、変動性が減少し、実験コストを低減することができる。クロマチン免疫沈降およびライブラリーを調製するための半自動化プロトコルが報告されているが、低細胞数3、4、5、6を使用するときに、これらの研究はいずれも、データを示さなかった。
本論文では、完全な自動化されたワークフローは、磁気ビーズベースの技術を使用しており、それは、プロトコル最適化における複数のパラメータに対応できるロボット液体ハンドリングシステムにおけるクロマチン免疫沈降およびライブラリの準備アッセイの両方に記載されている。ここでは、自動化されたChIP-seqの実験は成功し、簡素化の標準化、および小細胞集団におけるエピジェネティックなプロファイルを研究するために信頼性の高いソリューションを提供することを目的とした限られた数の細胞で行った。この論文に記載の自動化のChIPプロトコルは、特定のヒストン抗体および試薬Bを使用してHeLa細胞に最適化されていますワークフローの最適化実験対応する他の細胞株および抗体に適合させることができるUT。
プロトコル
1.標準ChIP実験
- 細胞収集及びDNA-タンパク質架橋。
- 80パーセント-90%の合流点にしたHeLa-S3細胞を成長させる。培養培地を除去する1×リン酸緩衝生理食塩水(PBS)10mlで二回皿を洗い、そして培養プレートをトリプシン-EDTA(1×)を加える。皿から細胞を剥離するために2分の最大インキュベートする。細胞を収集し、10ミリリットルPBSで2回洗浄します。
注:より長いインキュベーション時間は、細胞損傷につながる。 - 500×gで5分間、細胞を遠心分離し、PBS 20ml中で細胞を再懸濁する。細胞を数えるに進みます。
- 、500×gで5分間、細胞を遠心上清を破棄し、500μlのPBSを追加。固定工程のための細胞の最適数は、PBSを500μl当たり10万個の細胞である。
- 細胞懸濁液を500μlの各アリコートに新鮮な37%ホルムアルデヒドの13.5を添加する。室温で8分間、細胞を固定する。
- 1.25 Mグリシンsolutの57μLを追加固定を停止するイオン。穏やかなボルテックスにより一定に混合しながら室温で5分間インキュベートする。以降、この点から、氷上での作業。
- 4℃で5分間500×gで細胞を遠心し、細胞ペレットを乱すことなく、上清を捨てる。
- 1mlのPBSで細胞を2回洗浄する。優しく上清を捨て、氷の上で細胞ペレットを保つ。
- 80パーセント-90%の合流点にしたHeLa-S3細胞を成長させる。培養培地を除去する1×リン酸緩衝生理食塩水(PBS)10mlで二回皿を洗い、そして培養プレートをトリプシン-EDTA(1×)を加える。皿から細胞を剥離するために2分の最大インキュベートする。細胞を収集し、10ミリリットルPBSで2回洗浄します。
- 細胞溶解およびクロマチン断片
- 細胞ペレットを氷冷溶解緩衝液IL1 10mlの追加(100万個の細胞あたりの溶解緩衝液1mlを最適な比率である)。アップピペット上下に数回、穏やかに混合しながら4℃で10分間インキュベートする。
- 遠心機500×gで、4℃で5分間溶解液。上清を捨てる。
- 溶解液に氷冷溶解緩衝IL2の10ミリリットルを加え、ピペッティングにより穏やかに混合。 4℃で10分間、溶解物をインキュベートする。
- 500×gで4℃で5分間遠心し、上清を捨てる。
- PrepaIS1剪断バッファ200倍プロテアーゼ阻害剤カクテル(PIC)を加える完全な剪断バッファ再。氷上で5分間のバッファを維持し、その後、氷上で動作する。ペレット各千万細胞に完全なIS1せん断緩衝液1mlを加え、穏やかにピペッティングにより混合します。超音波処理の前に、サンプルの粘度を低下させるために10分間氷上でサンプルをインキュベートする。
- 10サイクルの各々の2から3セットのための水浴超音波処理を使用して超音波処理によりクロマチン300μlのアリコートせん断。サイクルは30秒間 "ON"と高電力設定で30秒間 "OFF"から成る。代替的に、30秒 "ON"、30秒間 "OFF"の5〜10サイクルの短い超音波処理時間でピコ超音波処理装置を使用。簡単に言うと渦と実行の間のチューブをスピン。超音波処理器の他のタイプを使用する場合は、クロマチン断片化のために、製造者の指示に対応してください。
- 10分であり、C、16,000×gで断片化したクロマチンを遠心IPステップですぐに使用される上清をollect。また、将来の使用のために最大2ヶ月間-80℃でクロマチンを保存する。
- 事前の百分の1から1.5 TAEアガロースゲルまたはbionalyzerを用いた免疫沈降工程のクロマチン断片化の効率を分析します。最適なクロマチン断片サイズは、100〜600塩基対の範囲である。
2.低細胞チップ実験
- 細胞収集及びDNA-タンパク質架橋
- 80パーセント-90%の合流点にしたHeLa-S3細胞を成長させる。培養培地を除去する1×リン酸緩衝生理食塩水(PBS)10mlで二回皿を洗い、そして培養プレートに1×トリプシン-EDTAを加える。皿から細胞を剥離するために2分の最大インキュベートする。
注:より長いインキュベーション時間は、細胞損傷につながる。 - 1ミリリットルの遠心チューブに血清を含む1mlの培地を添加して細胞を収集する。細胞を数える。
- 遠心分離機500×gで5分間、細胞。細胞数を持参固定のための培養培地1ml当たり10,000細胞である。
- 固定用の各チューブに36.5%の新鮮な準備されたホルムアルデヒドの27を添加する。チューブを2回または3回反転させ、室温で10分間インキュベートする。
- チューブを反転、サンプルに2〜3回1.25 Mグリシン溶液の115μlを加え、室温で5分間インキュベートする。以降、この点から、氷上での作業。
- 4℃で10分間300×gで細胞を遠心します。ゆっくり上清を捨てる。
- PIC(200X、最終濃度1×)で1ミリリットルの氷冷HBSSで細胞を洗浄。 4℃で10分間、300×gで細胞を遠心して再懸濁し、チューブを2回または3回反転する。優しく上清を捨て、氷の上で細胞ペレットを保つ。
- 80パーセント-90%の合流点にしたHeLa-S3細胞を成長させる。培養培地を除去する1×リン酸緩衝生理食塩水(PBS)10mlで二回皿を洗い、そして培養プレートに1×トリプシン-EDTAを加える。皿から細胞を剥離するために2分の最大インキュベートする。
- 細胞溶解およびクロマチン断片
- 10,000個の細胞あたりの完全な溶解バッファーTL1(溶解バッファーTL1 + PIC)の25μlのを追加し、細胞を再懸濁するために手動でチューブの底を攪拌する。氷上で5分間インキュベートします。
- 完全なHBSS(HBSS + PIC)の75μlの10,000個の細胞を含む各アリコートにバッファを追加します。
- 5サイクル各5セットのために超音波処理によって10,000個の細胞を100μlのアリコートせん断。サイクルは30秒間 "ON"と高電力設定で30秒間 "OFF"から成る。代替的に、30秒 "ON"、30秒間 "OFF"の5サイクルの短い超音波処理時間とともに使用ピコ超音波処理装置を使用。最適なクロマチン断片サイズは、100〜600塩基対の範囲である。クロマチン製剤、細胞型と異なる超音波処理器は、独立したせん断最適化実験が必要であることに注意してください。
- 遠心分離し、10分間14000×gで断片化したクロマチン不溶性物質を廃棄し、IPステップですぐに使用し、上清を収集する。また、将来の使用のために最大2ヶ月間-80℃でクロマチンを保存する。
- 百分の1から1.5 TAEアガロースゲルを用いて免疫沈降前工程のクロマチン断片化の効率を分析またはbionalyzer。剪断の視覚的評価を改善するために、前のアガロースゲル分析にRNアーゼを有する試料を扱う。最適なクロマチン断片サイズは、100〜600塩基対の範囲である。
3.クロマチン免疫沈降およびライブラリ準備
- 標準的な自動ChIP実験のために
- クロマチンをせん断し100μlにチップ·バッファH(チップ·バッファH + PIC)の120μlを添加する。 IPの200μlのを使用し、入力サンプルとして20μlまで2μLを保つ。
- オートメーション機器で自動化されたチップ200μlのプロトコルを選択します。プロトコルのスループットは、実行ごとに1から16のサンプルです。
- クロマチンは100万〜200万の細胞、抗H3K79me3の1-2μgのに対応し、使用して自動化ChIP実験を実行し、-H3K27me3、-H3K4me3、-H3K4me2、-H3K9ac、-H3K9 / 14ac、-H3K36me3とのH3K9me3のChIP-seqのグレードウサギポリクローナル抗体。最適な抗体量は、ヒストン修飾との親和性やスペックによって異なります対応する抗体のificity。
- アイソタイプコントロール抗体として非免疫ウサギIgGの等量を使用してください。また、コーティングされていないビーズまたはチップコントロールなどの特定のブロックされた抗体を使用しています。各反応のためのタンパク質被覆磁気ビーズの20μlのを追加。
- 抗H3K79me3と-H3K4me2ポリクローナル抗体を用いて自動化されたChIP実験を行うために自動化されたヒストンチップ-seqのキット試薬を使用してください。 、抗H3K27me3でChIP実験を行うことが理想的なのChIP-seqのキット試薬を使用し-H3K4me3、-H3K9ac、-H3K9 / 14ac、-H3K36me3と-H3K9me3。
- 自動化装置内に実装されたソフトウェアの指示に従って自動化されたChIPプロトコルを選択します。抗体コーティング工程および免疫沈降ステップのための15時間4時間までのChIP実験パラメータを設定します。逆架橋工程を4時間65℃で自動化された機器で行われる。
- 自動化されたシステム上での逆架橋DNAを精製する。 AUTを選択磁気ビーズベースのDNA精製を使用して、プロトコルまたはキットでDNA精製のためのプロトコルをomated。水25μlのDNAを溶出。
- 免疫沈降したDNAの10%を抽出することにより免疫沈降したDNAを定量化する。免疫沈降したDNAの収量は、クロマチンと抗体、細胞型および標的ヒストン修飾の品質に依存する。製造業者の説明書に従ってアッセイキットを用いてDNAを定量化する。
- 少なくとも1陽性と1陰性対照ゲノム領域のためのプライマーを用いて定量的PCRによる免疫沈降したDNAの品質を分析します。のChIP濃縮度を評価するための総免疫沈降したDNAの10%以上を使用しないでください。
- qPCR反応を準備します。 2X SyberGreen定量PCRマスターミックス10μlの、プライマーミックスを1μl、免疫沈降または入力DNAと20μlの最終反応容積に滅菌水アップの1-5を添加する。定量PCRプログラムは、95℃での初期変性ステップを含む5~10分間、Taqポリメラーゼのプロバイダによってアニール温度が選択されたプライマーに応じて設定されるべきである。
- ChIPのDNAとしてだけでなく、同じクロマチン準備から保存された入力DNAの両方を使用してライブラリを構築するために、市販のイルミナのChIP-seqのライブラリ調製試薬と互換性のある自動化ライブラリプレッププロトコルを使用してください。ライブラリを調製するための各抗体から免疫沈降したDNAの10〜20 ngのを使用してください。ランあたり16自動ライブラリまで準備します。
- ライブラリーの配列決定およびイルミナ製造元の指示に従ってクラスタを生成する。標準イルミナパイプライン、フィルター、主に次のようなバイオインフォマティクス解析(クラスターフィルタリング、ベースコーリングなど ) を実行し、エランドアライナで(現在のバージョンはGRCh38です)最新のヒトゲノムアセンブリに読み込む揃える。ピーク、呼び出しのためにSICER 7またはMACS 8を使用して、ワットピークの下流の分析を実行i番目ホーマー9、BEDTools 10又は好ましいソフトウェア。
- 自動化された低細胞数のChIP実験のために
- クロマチンをせん断し100μlに完全なチップバッファtC1の(チップ·バッファtC1の+ PIC)の120μlを添加する。 IPの200μlのを使用し、入力として20μLを保つ。
- 自動化システムで自動化されたチップ200μlのプロトコルを選択します。プロトコルのスループットは、実行ごとに1から16のサンプルです。
- 低クロマチン量で動作するように最適化された自動化されたChIPの試薬とチップグレード抗体を使用して自動化のChIP-seqの実験を実行します。 10,000個の細胞および100,000個の細胞、抗H3K27me30.5μgの、0.25μgの-H3K4me3、0.1μgの-H3K27ac、0.25μgの-H3K9me3ウサギプレミアムチップ-seqのグレードウサギポリクローナル抗体に対応したクロマチンを使用してください。最適な抗体量は、ヒストン修飾と対応する抗体の親和性および特異性に応じて異なる。
- 非免疫RAB等量のに使用しますアイソタイプコントロール抗体として、ビットのIgG。また、コーティングされていないビーズまたはチップコントロールなどの特定のブロックされた抗体を使用しています。各反応のためのタンパク質被覆磁気ビーズを10μlを追加。
- 自動化装置内に実装されたソフトウェアの指示に従って自動化されたChIPプロトコルを選択します。抗体コーティング工程および免疫沈降ステップのための15時間4時間までのChIP実験パラメータを設定します。逆架橋工程を4時間65℃で自動化された機器で行われる。
- 製造者の指示に従ってスピンカラムを用いて逆架橋されたDNAを精製し、水25μlに6μLからボリュームに溶出する。
- 市販のアッセイキットを用いてDNAを定量化する。 ChIPの品質を評価するために正と負の制御領域のためのプライマーを用いたqPCRにより結果を分析する。
- 低DNAの泉とのライブラリーを調製するために最適化されたライブラリ調製試薬でライブラリ調製キットを使用して、tities。ライブラリを調製するための30のPGとチップDNAの300 PG(それぞれ10,000 100,000細胞実験に相当)を使用します。ライブラリ調製試薬と互換性のある自動化されたプロトコルを使用してライブラリを準備します。自動ライブラリの準備のスループットは、実行ごとに1から48までのライブラリです。
- 二本鎖DNA鋳型の末端修理の後、配列決定プライマー部位を含む切断可能なステムループアダプタをライゲート。 DNA伸長工程の後に、ライブラリ調製キットのプロトコールに記載された高忠実度の増幅法でサンプルを増幅する。
- 図書館増幅後、ライブラリ調製キットのガイドライン以下のライブラリを定量化し、浄化する。精製は必要ありませんした後にそのサイズの選択に注意してください。
- ライブラリーを配列し、製造業者の指示に従ってクラスタを生成する。スタンド以下の主要なバイオインフォマティクス解析(クラスターフィルタリング、ベースコーリング、 など ) を実行ARDメーカーパイプライン、フィルタ、および揃えるエランド7アライナで(現在のバージョンはGRCh38です)最新のヒトゲノムアセンブリに読み込みます。 、BEDTools 10、または任意の好ましいソフトウェアをピーク通話用SICER 8またはMACS 9を使用し、ホーマーとピークの下流の分析を行う。

IP-スターコンパクトで自動化されたChIP実験をセットアップする方法を示すソフトウェアの図1.スクリーンショット。ソフトウェアが動作あたりのサンプル量を選択するだけでなく、重要な実験パラメータ(抗体コーティング、IPおよび洗浄を変更する柔軟性を提供)研究者のニーズに応じた。自動化された手順を並列に異なる条件( すなわち 、異なる種類および抗体の量、異なる種類の細胞の量、さらには異なるTをテストすることができますYPESと同じ実行中の磁気ビーズの量。 この図の拡大版をご覧になるにはこちらをクリックしてください。

自動化システム内のライブラリキットを使用して、次世代シーケンシングのための自動化されたライブラリの準備をセットアップする方法を示すソフトウェアの図2のスクリーンショット。 この図の拡大版をご覧になるにはこちらをクリックしてください。
結果
8つの異なるヒストンマーカーのための自動化のChIP-seqの実験の最適化
首尾よく開発し、以前に手動のChIP-配列の実験で検証された自動化されたChIPプロトコルのChIP-seqのグレード抗体を検証するために選択した(データは示さず)。抗H3K79me3、-H3K27me3、-H3K4me3、-H3K4me2、-H3K9ac、-H3K9 / 14ac、-H3K36me3と-H3K9me3:以下のChIP-seqのグレード抗体は、この研究のために選択した。全てのChIP-seqのグレードの抗体の特異性は、以前ドットブロット、ペプチドアレイおよびウェスタンブロット実験により確認した(データは示さず)。増加した抗体量のパイロットのChIP-qPCR実験は、抗体( 図3)の感度を決定した。少なくとも二つの正と負の2の制御対象と定量PCRを分析し、より高い負の目標以上の正の濃縮度を持つプロファイルは五倍シングEXPERのために修飾されているよりも、 iments。これは、断片化クロマチンの高品質のChIPとのChIP-seqの実験を行うことが重要である。この公報に示されているすべてのChIP実験は、新鮮なクロマチンを用いて行った。これは、-80℃で固定された細胞を凍結し、別の日にクロマチン準備とせん断を進めることも可能です。しかし、凍結した固定細胞から調製したクロマチンは、新たに調製したクロマチンから動作が異なる場合があり、したがって、超音波処理条件は各クロマチン調製のために最適化する必要があるかもしれません。異なる細胞型を使用する場合、別の洗剤組成物(SDS)と剪断緩衝液を使用することができる。そのような懸濁液中で増殖させた一次細胞株または細胞などの細胞型が困難な細胞が剪断およびHeLaは、低SDS濃度(0.1%)を必要とするような、剪断が容易な細胞株に対し高いSDS濃度(1%)を必要とするであるせん断バッファ。
res.jpg "/>
自動化システムを使用してChIPグレード抗体の図3.検証。チップはから断片化クロマチンに抗H3K79me3、-H3K27me3、-H3K4me3、-H3K4me2、-H3K9ac、H3K9 / 14ac、-H3K36me3と-H3K9me3ウサギポリクローナル抗体を用いて行ったヒストン修飾によっては百万のHeLa-S3細胞。 200μlの作業容積を有する自動化されたChIPプロトコールは、抗体滴定実験のための自動化装置を使用した。 1、2、5および10μgの抗体量は、ChIP実験ごとに試験したところ、2μgのIgGを各実験において陰性対照として使用した。濃縮度を定量PCRにより評価した。結果は入力の%(qPCR分析後の入力DNAに比べて免疫沈降したDNAの相対量)として示している。 この図の拡大版をご覧になるにはこちらをクリックしてください。
検証と最適条件を決定した後 ChIPグレード抗体lの量が自動化システムで使用される、自動化されたChIP-seqの実験は、各ヒストン修飾( 図4)のためのシーケンシングプロファイルを生成するために実施した。

図4.ヒストンチップ-seqのプロファイルは、自動化のChIP-seqの実験によって生成された。図はH3K4me3と、H3K9acおよびH3K9 / 14acH3K4me2 H3K79me3、及びH3K36me3のための異なるゲノム領域におけるチップ-seqのプロファイルを示す。 図4(a)は、完全なXに沿ってピーク分布を示している-chromosomeおよび4B GAPDH遺伝子を囲む75キロバイトの領域での分布。(c)は、MYT1遺伝子を囲む500キロバイト地域のH3K27me3、H3K36me3とH3K4me3とのプロファイルを示しており、4Dは ZNF12を囲む200キロバイト地域でのH3K9me3の分布を示している。large.jpg「ターゲット= "_空白">この図の拡大版をご覧になるにはこちらをクリックしてください。
遺伝子発現と関連する六つの異なるヒストン修飾のためのヒストンエピジェネティックなプロファイルが(H3K4me3と、H3K9ac、H3K9 / 14ac、H3K4me2、H3K79me3およびH3K36me3)を作成した。 図4(a)は、異なるヒストンマーカーのX染色体に沿ってのChIP-seqのプロファイルを示す。 6つの異なるヒストンプロファイルの間で観察され、高度に相関ピークは、正確で信頼性のあるデータを生成する自動化システムの能力を示している。 図4B、4Cおよび4Dは、特定のゲノム領域における異なるヒストン修飾のためのピークの分布を示す。
200セルまで自動化されたクロマチン免疫沈降実験
ChIP実験で使用することができるセルの最小量は、クロマチンの品質に依存し、specificitYと抗体とヒストン修飾やタンパク質研究の豊富さの感度。良品チップ-seqのグレード抗体を選択すると、サンプルの限られた量を使用する際に重要であり、最適な試薬および異なるキャリアの選択は、DNA回収の効率が向上し、ChIP実験の成功に貢献。特にクロマチンの少量で動作するように最適化されたChIPの試薬を使用してIP-スター自動化システムで試験した自動化のChIPプロトコルが処理できる細胞、クロマチン、抗体、磁気ビーズの異なる量の最小量を決定する。
プロトコールに記載されているように、まず、10,000個の細胞からクロマチンを超音波処理した。 ChIPの結果は、陰性対照領域における陽性対照領域と無視できる信号でH3K4me3と抗体との有意な豊富化を示す定量PCR( 図5A)により確認した。一貫性の比較および証明のため、additionaLのデータは、H3K27ac、のH3K9me3とH3K27me3抗体を用いて得られた10,000個の細胞を使用して提供される。
自動化されたChIP実験は、同一のH3K4me3と抗体を用いた細胞の少量で動作するように自動化されたシステムの機能を示すために、次に実行された。自動化されたチップは、再現性があり、手動ChIPの結果( 図5B)と高度に匹敵した10個のIP一連の反応によって実証、好調に推移しました。手動および自動化された実験が行われ、自動化されたプロトコルの利点は、可変性( 図5C)を 、実験間の低減に見られた。
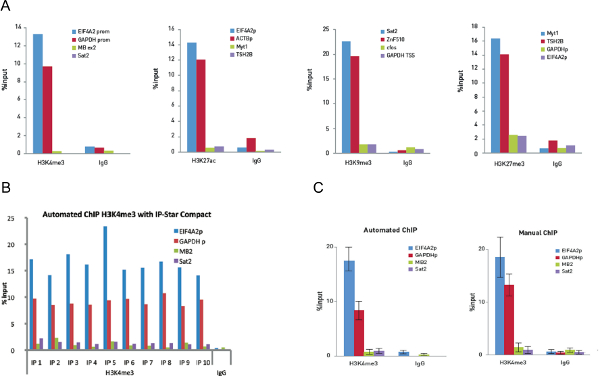
図5.手動ChIP実験は、10,000個の細胞で実施した10,000個の細胞上のチップと自動チップ実験の最適化とH3K4me3と0.25μgの、H3K27ac0.1μgの、のH3K9me3と0.20.5μgのを使用してH3K27me3抗体5μgの。ウサギIgGの同一の量を対照として用いた。定量PCRは、2正座および各ChIPアッセイのための2つの負の遺伝子座のためのプライマーを用いて行った。 図5(a)は、回復を示し、入力の割合(qPCR分析後に入力DNAに比べて免疫沈降したDNAの相対量)として表現。 図5Bショー 10 ChIPの反応は0.25μgのH3K4me3とポリクローナル抗体およびウサギIgG0.25μgのようなネガティブコントロール抗体を用いたIP-スターコンパクトで実行する。その後qPCR分析は、正座EIF4A2プロモーターおよびGAPDH TSS及び負座ミオグロビンエクソン及びSAT2のためのプライマーを用いて行った。図は、入力(qPCR分析後に入力DNAに比べて免疫沈降したDNAの相対量)のパーセントとして表され、回復を示している。 図5Cは、10自動化されたChIP実験と比較して10マニュアルChIP実験のH3K4me3とチップデータを示しています。エラーバーは、sを表す 10回の反復のそれぞれのtandard偏差。 この図の拡大版をご覧になるにはこちらをクリックしてください。
自動化されたChIPプロトコルの感度を理解するために、実験は、IP 100,000 200までの細胞の範囲であった細胞の量を用いて行った。それは非常に一般的なヒストン修飾と同様に、抗H3K27me3抗体を使用した。他のヒストンまたは非ヒストン抗体の使用は、エピトープの存在量と抗体の品質に応じて、より多くのまたはより少ない細胞を必要とし得る。実験は、定量的PCRにより確認し、そしてそれがビーズ実験における抗体のバックグラウンドの量を低減することによって、わずか200個の細胞抗体( 図6)で成功したChIP-qPCRの結果を可能に減少することが観察された。
ジル/ ftp_upload / 52150 / 52150fig6highres.jpg "/>
図6. 200細胞上のチップアッセイを自動化 。 H3K27me3に対して向けヒーラ-S3細胞および抗体。クロマチンは100万個の細胞とチップ反応ごとに使用した(10万〜200細胞相当)このクロマチンの段階希釈から剪断した。プロテインA被覆磁気ビーズの1 H3K27me3μgのと10μlのH3K27me3の0.5μgの10,000と1,000個の細胞上のビーズを10μl、及びH3K27me30.25μgのと500と200とビーズの5μlを、100,000細胞実験で使用された細胞。 100,000個の細胞をそれぞれ1,000個の細胞を用いた実験を行う際に、1μgのウサギIgGのμgの0.5は、陰性対照抗体として使用した。 図6(a)は、入力%以上でTSH2BとGAPDH遺伝子の占有を示す図6(b)は、ネガティブGAPDHコントロールに対するTSH2Bの相対的占有率を示しゲノム領域。
上のチップ-seqの結果のダウンストリーム分析10,000個の細胞
細胞の低い開始番号自動化のChIP-seqの実験の全体的品質を評価するために、自動化されたChIP-seqのアッセイ100,000のHeLa-S3細胞1万HeLa細胞およびChIP実験でH3K4me3と抗体を0.25μgを用いて実施したが、使用された実験のための陽性対照として。自動化ライブラリは、低DNA量に調製したライブラリーに適したミクロプレックスライブラリ調製キット試薬を用いて調製した。それが可能であっても、10,000未満の細胞で成功した自動化のChIP-実験を実行することに注意し、プルダウンDNAの量は、キット試薬を使用してライブラリーを調製するのに十分ではない。クラスタ生成及び配列決定は、製造者の指示に従って行った。バイオインフォマティクスは、低細胞数のChIPサンプルからの配列決定ショー優れた結果を後に分析する。出発物質の10,000個の細胞に相当する30 pgのデータセット( )、低バックグラウンドノイズと300 pgのデータセット(出発物質の100,000個の細胞に相当)と、外部参照として使用したENCODEプロジェクトのブロード研究所で生成されたH3K4me3とデータセットの両方によって確認された信頼性の高い濃縮度のピークを含む。これは、2つのデータセットの最良40%、少なくとも80%のオーバーラップがあるとを比較した場合のChIP-seqのが再現性を考慮したENCODE 11のプロジェクトで使用される標準的な方法を指すトップ40ラップ率データを、注意することが重要である重要度スコア順ピークの。 300 pgのデータセット(そのすべてのピークを考慮だけでなく、最高の40%)、およびブロード研究所データ( 表1)の両方と比較した場合、30 pgのデータセットは、これらの基準を満たす。 300 pgのデータセットは、98%のトップ40ラップ率( 図7)とブロード研究所のデータとほぼ同一のピークを示す。
ighres.jpg "/>
図7.のChIPアッセイおよびライブラリ生成10,000細胞上のチップ-seqの実験はH3K4me3と抗体(/μlの0.25μgの)を使用して、万10万のHeLa-S3細胞で生成された。 35 bpのタグがエランドアライナとヒトゲノムにマッピングされた。その後のピーク時にはSICERを呼び出すと、確実に低細胞数からだけでなく、細胞の数百万から濃縮度を識別することができる。データセットを分析し、相互にブロード研究所で生成された参照データと比較した。低い細胞サンプルが一貫して非常に高い類似性を有する。 30 pgのサンプルは満たしENCODE基準11(分。ピークのトップ40パーセントの80%が重複しなければならない)。 この図の拡大版をご覧になるにはこちらをクリックしてください。
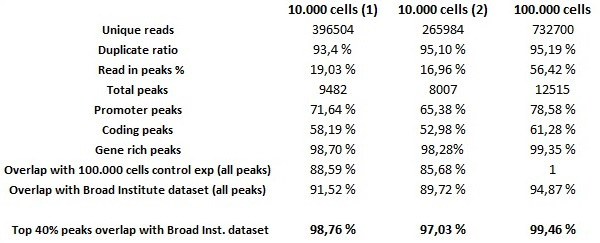
表1。
ディスカッション
シークエンシングに続いてクロマチン免疫沈降は現在、標準的な手順です。ここでは、出発物質のわずか10,000細胞を用いたクロマチンのエピジェネティックなプロファイルを生成することができ、自動化のChIP-seqのプロトコルが提供される。
ChIPのとライブラリの準備アッセイの自動化のChIP最適化手順を標準化し、実験的なばらつきを低減することができます。ここで紹介する液体ハンドリングシステムは、わずか30分の時間に手を減らすのChIPに関連したマニュアル手順の多くを排除し、サンプル損失を最小化し、ライブラリ入力のわずか数ピコグラムで正確なのChIP-seqのを可能にします。成功した自動化されたChIP-seqの実験を達成するために、それはまた、それぞれの実験系において、高品質化クロマチン調製物とのChIP-seqのグレードの抗体を使用することが重要であるが、磁気ビーズベースの技術を使用し、そのようなインキュベーションのような主要な実験パラメータを変更するための柔軟性を提供抗体のコートのための時間る免疫沈降ステップまたは研究者のChIP-seqの最適化のためのすべての必要な実験を行うことが可能な洗浄条件の変更。自動化システムはまた、各個々の細胞株および抗体のための実験条件を最適化するために並列に複数の試薬との比較を可能にし、様々なタイプ及びクロマチンの濃度を、異なる抗体および磁性であっても異なるタイプの直接比較を可能にする「オープン」なプラットフォームであるビーズ。
自動化システムの限界の一つは、200μlに5μlの範囲、ボリューム内のすべてのプロトコルを自動化する必要性である。しかし、この自動化されたプラットフォームでの実験の小型化も試薬中のコストを節約することができます。
この研究で記載されたプロトコルに加えて、システムは適用可能であるし、またそのような免疫沈降aと他の磁気ビーズに基づく様々なアプリケーションを自動化するNDメチル化DNA(MeDIPとMethylCapテクノロジ)、hydroxylmethylated DNA(hMEDIP)、シーケンシャルクロマチン免疫沈降(ReChIP)、RNA免疫沈降(RNA-IP)、亜硫酸水素変換の免疫沈降、及びDNA精製アッセイの捕獲。
開示事項
The authors of this article, at the time of its writing, are employed by Diagenode S.A. and Diagenode, Inc., the manufacturer of the automated system described.
謝辞
This work was supported by the BLUERPINT EU grant (BLUEPRINT – A BLUEPRINT of Haematopoietic Epigenomes). We also thank the Walloon Region (DG06) for its financial support.
資料
| Name | Company | Catalog Number | Comments |
| PBS | Life technologies | 14190-094 | |
| Trypsin-EDTA | Sigma | T3924-100ML | |
| Formaldehyde 37% | Sigma | F8775-25 | |
| 1.25 M Glycine Solution | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Lysis Buffer iL2 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Shearing Buffer iS1 | Diagenode | C01020010 | Component of the ideal ChIP-seq kit |
| Protease Inhibitors Mix (200x) | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| HBSS (no calcium, no magnesium, no phenol red) | Life technologies | 14175-053 | |
| Lysis Buffer tL1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ChIP Buffer tC1 | Diagenode | C01010130 | Component of the Auto True Micro ChIP kit |
| ideal ChIP-seq kit | Diagneode | C01010051 | |
| ChIP-Buffer H | Diagenode | C01010020 | Component of the Auto Histone ChIP-seq kit |
| Auto Histone ChIP-seq kit | Diagenode | C01010020 | |
| Auto True Micro ChIP kit | Diagenode | C01010130 | |
| H3K79me3 polyclonal antibody-Classic | Diagenode | C15310068 | |
| H3K27me3 polyclonal antibody-Classic | Diagenode | C15410069 | |
| H3K4me3 polyclonal antibody-Classic | Diagenode | C15410030 | |
| H3K4me2 polyclonal antibody-Classic | Diagenode | C15410035 | |
| H3K9ac polyclonal antibody-Premium | Diagenode | C15410004 | |
| H3K9/14ac polyclonal antibody-Premium | Diagenode | C15410200 | |
| H3K36me3 polyclonal antibody-Premium | Diagenode | C15410058 | |
| H3K9me3 polyclonal antibody-Premium | Diagenode | C15410193 | |
| Rabbit IgG | Diagenode | C15410206 | |
| Protein-A coated paramagnetic beads | Diagenode | C01010020 | |
| Auto IPure | Diagenode | C03010010 | |
| MicroChIP DiaPure columns | Diagenode | C03040001 | |
| Universal SyberGreenMaster Mix 1.25 ml | Diagenode | DMMLD2D100 | |
| Quant-IT dsDNA | Invitrogen | Q32854 | |
| Illumina Sample Preparation kit fro Genomic DNA | Illumina | FC-121-3001 | |
| Illumina True-seq kit ChIP library Prep kit | Illumina | IP-202-1012 | |
| MicroPlex Library Preparation Kit | Diagenode | C05010010 | |
| Agencourt AMPure XP beads | Beckman Coulter | A63881 | |
| Illumina Library prep Quantification kit | Kapa Biosystems | KK4844 | |
| IP-Star Compact Automated System | Diagenode | B03000002 | |
| Bioruptor Plus | Diagenode | B01020001 | |
| Bioruptor Pico | Diagenode | B01060001 | |
| Qubit system | Invitrogen | Q32857 | |
| Illumina Hiseq systems | Illumina |
参考文献
- Dahl, J. A., Collas, P. A rapid micro chromatin immunoprecipitation assay (microChIP). Nature Protocols. 3, 1032-1045 (2008).
- Adli, M., Bernstein, B. E. Whole-genome chromatin profiling from limited numbers of cells using nano-ChIP-seq. Nature Protocols. 6, 1656-1668 (2011).
- Blecher-Gonen, R., et al. High-throughput chromatin immunoprecipitation for genome-wide mapping of in vivo protein-DNA interactions and epigenomic states. Nature Protocols. 8, 539-554 (2013).
- Aldridge, S., et al. AHT-ChIP-seq: a completely automated robotic protocol for high-throughput chromatin immunoprecipitation. Genome Biology. 14, 124 (2013).
- Farias-Hesson, E., et al. Semi-automated library preparation for high-throughput DNA sequencing platforms. Journal of Biomedicin., & Biotechnology. 2010, (2010).
- Callejas, S., Alvarez, R., Benguria, A., Dopazo, A. AG-NGS: a powerful and user-friendly computing application for the semi-automated preparation of next-generation sequencing libraries using open liquid handling platforms. BioTechniques. 56, 28-35 (2014).
- Zang, C., et al. A clustering approach for identification of enriched domains from histone modification ChIP-Seq data. Bioinformatics. 25, 1952-1958 (2009).
- Zhang, Y., et al. Model-based analysis of ChIP-Seq (MACS). Genome Biology. 9, 137 (2008).
- Heinz, S., et al. Simple combinations of lineage-determining transcription factors prime cis-regulatory elements required for macrophage and B cell identities. Molecular Cell. 38, 576-589 (2010).
- Quinlan, A. R., Hall, I. M. BEDTools: a flexible suite of utilities for comparing genomic features. Bioinformatics (Oxford, England). 26, 841-842 (2010).
- Landt, S. G., et al. ChIP-seq guidelines and practices of the ENCODE and modENCODE consortia. Genome Research. 22, 1813-1831 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved