このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
同心円状のゲルシステムは、3D細胞移動に対するマトリックスの微小環境の生物物理役割を研究する
要約
機械的性質と細胞外マトリックスの微細構造は強く、細胞の3D移行に影響。 インビトロ方法は、集団及び個々の細胞レベルの両方での生物物理学的に可変環境における時空間細胞移動の挙動を研究するために、記載されている。
要約
遊走する細胞の能力は、腫瘍および癌転移に胚発生及び創傷治癒から一生を通じて細胞機能のさまざまな重要である。熱心な研究の努力にもかかわらず、細胞遊走の基本的な生化学的および生物物理学的な原理は、まだ完全には特に、生理学的に関連する3次元(3D)の微小環境において、理解されていない。ここでは、三次元細胞遊走挙動の定量的検査を可能にするように設計されたインビトロアッセイを記載する。この方法は、以前に占有されていない細胞外マトリックス(ECM)の中に移動する細胞のmechanosensing能力や傾向を利用する。我々は、モデル系としてコラーゲンゲルにおける高浸潤性乳癌細胞の浸潤、MDA-MB-231を使用する。培養の数週間にわたる細胞集団の広がりと個々の細胞の遊走ダイナミクス生細胞イメージングを用いてモニターし、時空間的に分解されたデータを抽出するために分析することができる。さらに、したがって、この方法は、細胞遊走に微小環境における生物物理学的因子の役割を調査するため、シンプルでありながら強力な方法を提供し、多様な細胞外マトリックスのために容易に適応可能である。
概要
細胞の遊走は、胚発生、止血、および免疫応答ならびに、血管疾患、炎症、および癌の1のような病理学的プロセスにおける様々な生理学的応答において重要な役割を果たしている。細胞遊走の基礎となる生化学的および生物物理学的な因子を解剖する細胞機能の基本的な原理を理解するだけでなく、そのような組織工学、抗転移および抗炎症薬の開発のように、様々な生物医学的用途を前進させるだけではなく、したがって基本的に重要である。 in vivoでの観察が技術的に困難であるため、多くの努力は、細胞遊走のインビトロ要約が注目されている。
細胞遊走を研究するためのインビトロ法に大きく、最も顕著なのは、2次元(2D)表面上にアッセイのために傷を設計またはアッセイ2創傷治癒されている。このようなアッセイは、簡単な実験装置を提供し、容易なの生細胞イメージング、および細胞遊走の基礎となる様々な生化学的機構に有用な洞察を提供する。しかし、これらのアッセイは、in vivoの移行で理解する上での重要な側面である細胞外マトリックス(ECM)アーキテクチャと改造、を考慮していない。最近、ますます三次元培養モデルは、多くの場合、コラーゲンベースのマトリックス3において、優れたin vivoでの状況に似ているプラットフォームを提供することが理解されている。実際に、細胞が環境4の異なる次元に特に起因する2次元表面上のものとは異なる遊走動態を示す。また、マトリックスの生物物理学的および機械的特性が敏感に腫瘍細胞の浸潤6との関連を含めて、細胞遊走5に影響を与える。
ここでは、製造条件を容易に変更することができる生物物理学的特性を有するECM内の3D細胞遊走挙動を研究するための方法を提示する。細胞である「内部ゲル」に播種しに逃げ込むと、最初は無細胞「アウターゲル」を侵略するために許可されています。この方法は、密接にセル7 mechanosensingにリンクされている外側のゲル、無細胞領域に遊走する細胞のの存在を認識する能力、及び性向に依存する。本研究では、高浸潤性乳癌細胞、MD-MBA-231に侵略されたECMコラーゲンネットワークを採用する。内部と外部の両方のゲルの機械的性質と微細構造8をチューニングし、生理学的に関連する条件を達成するために9を特徴付けることができる。セルトラックの再構築及び分析は、集団レベルおよび個々の細胞レベルの両方で、時空間移行挙動の詳細な定量的な検査を可能にする。重要なことは、同心円状のゲルシステムのセットアップは、このように重要な洞察を提供し、特に癌細胞に侵入、細胞の移行が直面している生体内組織トポロジを模倣細胞遊走および転移の物理的なメカニズム。
プロトコル
1.細胞の回収
- 37℃、5%CO 2インキュベーターからMD-MBA-231細胞を得る。 0.5%トリプシン-EDTA溶液を用いて組織培養プレートから細胞を剥離。 T25フラスコで培養した細胞のためのトリプシン-EDTA溶液1mlを使用してください。
- 4分間200×gでの遠心分離により15ミリリットルコニカルチューブ中の細胞をペレット化し、上清を吸引し、そして培養培地の5ミリリットル中に細胞を再懸濁。
- 血球計数器を使用して、ρ、細胞密度をカウントします。
注:細胞播種インナーゲルを調製するために、細胞懸濁液は、後に最終的な細胞播種密度に達するために10倍に希釈される。したがって、10倍濃縮細胞懸濁液が必要とされる。 - 10×細胞濃度を達成するために必要な媒体の体積を計算する。
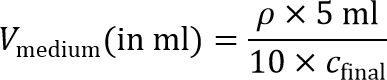
注:最終細胞播種密度を、 最後の C、約2の15; 10 6細胞/ mlのMD-MBA-231細胞に推奨されており、このプロトコルで使用されている。他の播種密度は、他の細胞型のために探索することができる。 - ペレット細胞1以上の4分間200×gでの遠心分離により15ミリリットルコニカルチューブの時間、上清を吸引除去する。
- 細胞凝集を最小限にするために十分に無血清細胞培養培地の必要量(ステップ1.3で算出したV 培地 )中で細胞を再懸濁。
注:フェノールレッドを自家蛍光であり、蛍光/反射率画像化を妨害する可能性がある。フェノールレッドを含まない培地の使用は、最高の画像品質を達成すると考えることができる。
コラーゲン溶液の調製
- 原液コラーゲン溶液、10×PBS緩衝液、ミリQ H 2 O、0.1 MのNaOH、及びいくつかの微量遠心管を得る。早期のコラーゲン重合を防止するために氷の上にすべてを保持し、無菌状態を維持する。
- 無菌のガラス - を平衡化する37℃のインキュベーター内に予め加温してボトムディッシュ。
注:このプロトコルのすべてのボリュームは、ウェル12 mmのガラスボトムディッシュに最適化されています。他の皿型が使用される場合、それに応じてボリュームを調節する。 - コラーゲンストック濃度に基づいて、インナーゲル(溶液I)が2.4 mg / mlのコラーゲン溶液50μlを調製するのに必要とされる必要な容量を計算する。
注意:インナーゲルのための他のコラーゲン濃度を使用することもできる。 - 無菌環境(通常はバイオセーフティーフード)で、ゆっくりと穏やかな旋回と(ステップ2.3で計算)コラーゲン原液の必要量に10×PBS緩衝液の5μlを添加する。気泡の形成を避けるように注意してください。
- 校正されたpHメーターを使用し、0.1M NaOHを用いて7.4に、混合物のpHを調整する。おおよその目安としては、(量がストック濃度とpHによって異なります)7.4に近いpHをもたらすために約5μLを使用しています。
注:ボリュームがこのに関与していることに注意してくださいステップは、標準pHメーターを使用するには小さすぎる。以下のトリックのいずれかを使用します。- 複数のサンプルのためのコラーゲン溶液を準備します。標準pHメーターを使用してバルクのpHを調整し、サンプル全体でコラーゲン溶液を配布。
- あるいは、より大きな体積のコラーゲン溶液のpHを調整する(すなわち、標準的pH計の使用を可能にするボリューム)。 NaOHの量は、最終的なpHにpHを必要に応じて注意してください。ボリュームを縮小し、実験のためのNaOHの適切な量を使用しています。リトマス紙を用いてpH値を確認してください。
- そうでなければ、より正確に少量のpHを調節するために、マイクロpH電極を使用する。
- H 2 Oを使用して45μLの容積に解決策を持参早期のコラーゲン重合を防止するために氷の上のすべての手順を実行します。
同心円ジェル文化の3の形成
- 予め温めておいたガラスボトムディッシュを取る目から(ステップ2.2を参照してください)Eインキュベーター。
- 溶液を十分I.再懸濁するために(ステップ1.5で調製)10倍濃縮細胞懸濁液の5μlを添加する。気泡の形成を避けるように注意してください。混合物は現在、50μlの容量を有し、( 最終 = 2×10 6細胞/ mlのc)の最終コラーゲン濃度(2.4 mg / ml)で細胞密度を含有する。
- それはドーム状の液滴( 図1A)を形成するように、私は、ウェルの中央にゆっくりと細胞含有溶液20μlを追加します。このステップでの気泡形成を避けるように注意してください。バブルフォーム場合は、慎重にしかしすぐにそれを破裂またはピペットチップを使用してそれを吸い出すするかしてみてください。静かに内側のゲルを45分間重合させるために戻ってインキュベーターで料理を置く。
- (アウター、無細胞コラーゲンゲル用)は、溶液Oこのインキュベーション工程の終了前に約15分を準備します。
注意:アウターゲル状態はコラーゲン濃度および重合のpHの条件に変更することができる異なる微細構造10とのネットワークを得る。 7.4のpHで重合を4.0mg / mlのコラーゲンゲル - このプロトコルでは、1.5に焦点を当てる。- コラーゲンストック濃度に基づいて、最終濃度のコラーゲン溶液のO200μlの準備に必要な必要量を計算する。
- ゆっくりと穏やかな旋回と(ステップ3.4で計算)コラーゲン原液の必要量に10×PBS緩衝液20μlを追加します。キャリブレーションpHメーターの使用と、0.1M NaOHを用いて、最終的なpHに混合物のpHを調整します。 pH調整に関する2.5のステップに注意してご覧ください。
- H 2 Oを使用して200μlの最終容量に解決策を持参早期のコラーゲン重合を防止するために氷の上のすべての手順を実行します。
- 内側のゲル(ステップ3.3を参照)の45分の重合後にインキュベーターから一品を取る。溶液が完全に内部を覆うように静かに、内側のゲルの上にソリューションOの180μlを添加するゲルとよく( 図1B)をいっぱいに。
- アウターゲル中の不均一な繊維配向につながる可能なソリューションを、攪拌せずに慎重にこの手順を実行します。ピペットチップで内側のゲルに触れないように注意してください、と気泡やエアポケットの形成を避けるため。バブルフォーム場合は、慎重にしかしすぐにそれを破裂またはピペットチップを使用してそれを吸い出すするかしてみてください。ゆっくりと外側のゲル重合さをできるように戻ってインキュベーターで料理を置く。
- 重合の45分後にインキュベーターから一品を取る。大体扱う場合には、それはまだ底面から切り離すことができますがゲルはすでにかなり、この時点で固化されるべきである。
- 静かに皿( 図1C)に温め、細胞培養培地2mlを注ぐ。ゲルが完全に中に沈めていることを確認します。文化の期間を通じて3日間 - ごとに2培地をリフレッシュします。
4。生細胞イメージング
- 長期生細胞イメージング機能を備えた倒立共焦点顕微鏡を用いて撮像を行う。温度(37℃)でインキュベーションチャンバーを内蔵し、CO 2(5%)の制御が含まれる。顕微鏡の電源を入れ、実験を開始する前に、ステージを少なくとも1時間のウォームアップ。
注:長作動距離対物レンズで使用する3次元ゲル内の細胞の観察および局在化を最適化する。 - 3Dシステム内のセルの正確な局在化を可能にするために30分間蛍光細胞追跡色素を5μlを含有する培地中でゲルをインキュベートする。その後、1×PBSで3回洗浄して未結合の色素を除去。その後、シャーレに細胞培養培地を加える。
- インキュベーターから料理を取り、顕微鏡ステージ( 図1D)の上に置きます。
注:ライブセルイメージングは、右アウターゲルの重合後、原則的に開始することができます。しかしこの時点では、細胞のinの内側のゲルはまだ広がっていない。アウターゲルに侵入した最初の細胞の移動を調べるために、生細胞イメージングは、培養開始後24時間(細胞型に応じて)を開始することができます。おおよその目安としては、約12 - 14日間の培養は、外側ゲルを入力するように内側のゲル中の細胞のほとんどのために必要とされる。 - インナーゲルを囲む外側のゲル中の地域で見る(VOVの)のボリュームを選択します。インキュベーションの24時間後に、細胞集団が広がっているので、内側と外側のゲルとの間の界面を越えて、外側のゲルに侵入し始めた。
- VOVのために、すぐに次の外側ゲル7の郊外に近いゲル界面、中間領域、および領域へのゲル領域を含む。可能なエッジ効果を回避するために、近い底面及び側面からだけでなく、ゲルの上部からは50μm以上の領域を除外する。各VOVは、一般的にXで(647×647×100μmの3を測定し、のz -stack5μmの間隔で、それぞれ、y、およびz方向)。
- 確実に撮影モード、チャンネル/フィルター、露光時間、及び画像の解像度が正しく選択されている。コラーゲンネットワークのラベルフリーイメージングのために、タイムラプス生細胞イメージングの間に同時に共焦点反射顕微鏡を使用しています。
- 、サンプル画像を取り、強度ヒストグラムをプロットし、十分な信号を観測し、ヒストグラムがゼロと最大強度の間にあることを保証することにより、飽和を避けるために、ゲインおよびオフセットを調整します。実験期間を通じてこれ以上、これらの設定を変更しないでください。
- 8時間(または必要に応じてより長い)のために10分、時間間隔、Δtと共に、選択したVOV年代のタイムラプス画像を撮影する。
5.細胞追跡およびデータ分析
- appropriaを使用してZ -stack画像から定量的な画像後処理を実施テ画像処理ソフトウェア。
- セグメントタイムラプス画像は、自動的に(x、y、zの、t)内の3Dセル位置を選択する。
- フレームごとに、手動でローカライズ精度を確認し、細胞のために間違われている可能性が細胞の破片と細胞の突起に起因する偽陽性を削除する。分析から増殖細胞を取り出し、個別のオブジェクトに重複または接続されたセルを分割する。
- 時系列的に各セルの位置を連結することにより、前工程で得られた細胞の座標(x、y、z、t)から3Dタイムラプス細胞トラックを生成する。
- しきい値トラック長(通常は20分)より短いトラックを除去することによって、ランダムとシステムノイズを排除します。
- 必要に応じて、トラックからの全体の正味の変位を減算することにより、サンプルのドリフトを修正してください。
- セルの変位を計算し、ジル/ ftp_upload / 52735 / 52735eq2.jpg "幅=" 80 "/>、および細胞移動距離、
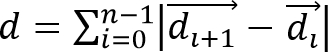 、観察細胞の軌跡から、ここで一度にセルの3次元位置を表すベクトルであり、時間点の総数である。
、観察細胞の軌跡から、ここで一度にセルの3次元位置を表すベクトルであり、時間点の総数である。 - Δtはフレーム間の時間間隔であるS = D /(nは•Δtは)、などの細胞の速度を計算する。 P =Δdを/ Dを使用して、細胞移動の方向性(または持続性)を計算します。持続性のこの単純な尺度は、P = 0のために正味の変位がゼロであり、P = 1のための軌道が直線方向線であることを意味する。
結果
ここに提示同心ゲルアッセイは高度に浸潤性乳癌細胞を用いて実施した、MDA-MB-231、2.4 mg / mlの内側のコラーゲンゲルと、一例として= 2×10 6細胞/ mlの細胞播種密度、を有する。 図2に示すように、典型的には培養の数日後に、細胞を、内外ゲル界面に違反し、外側ゲルに侵入し始めた。細胞集団は、主に半径方向外側に広がる。
外側のゲルの重合条件?...
ディスカッション
In this protocol we describe an in vitro assay to study the 3D migrational behavior of cells in matrix environments that topologically resemble ECMs encountered in vivo. There are three main strengths of this assay as compared to other currently available methods. First, this assay allows one to simultaneously examine the cell migration mechanisms at both population level and individual cell level. This opens up possibilities of studying collective cell migration13, which has to date been lar...
開示事項
著者らは、開示することは何もない。
謝辞
著者は批判的な議論のためにW.日とK·ジャンセンに感謝し、シンガポール国立大学でナノバイオメカニクス研究室による支援を認める。 NAKがマリー·キュリーIIFフェローシップによる支援を認めるものです。
資料
| Name | Company | Catalog Number | Comments |
| Cell culture incubator | Fisher Scientific Pte Ltd | Model: 371, S/No 318854-6055 | |
| Confocal microscope | Nikon A1R | Inverted confocal laser scanning microscope equipped with incubator chamber | |
| Dulbecco's Modified Eagle's Medium (DMEM) | Life Technologies | 11965-092 | |
| Fetal Bovine Serum (FBS) | Life Technologies | 10082-147 | |
| Fluorescent CellTracker dye CMTMR | Life Technologies | C2927 | |
| Glass-bottom dish | IWAKI Cell Biology | 3931-035 | 35 mm diameter dish with 12 mm diameter glass-bottom well |
| Hemocytometer | iN CYTO | DHC-N01 (Neubauer Improved) | |
| Microprocessor pH meter | Hanna Instruments | pH 211 | |
| Nutragen Collagen | Advanced BioMatrix | #5010-D | Acid-solubilized bovine collagen type I (stock pH ~ 2) |
| Objective lens | Nikon | CFI Super Plan Fluor ELWD ADM 20XC, W.D. 8.2-6.9mm, NA 0.45. | |
| Penicillin-Streptomycin | Life Technologies | 15140-122 | |
| pH meter | Sartorius | S/No 29153352 | Basic pH Meter PB-11 |
| Trypsin-EDTA | Life Technologies | 15400-054 |
参考文献
- Horwitz, R., Webb, D. Cell migration. Curr Biol. 13 (19), R756-R759 (2003).
- Liang, C. C., Park, A. Y., Guan, J. L. In vitro scratch assay: a convenient and inexpensive method for analysis of cell migration in vitro. Nat. Protoc. 2 (2), 329-333 (2007).
- Provenzano, P. P., Eliceiri, K. W., Inman, D. R., Keely, P. J. Engineering three-dimensional collagen matrices to provide contact guidance during 3D cell migration. Curr. Prot. Cell Biol. 10, 10-17 (2010).
- Friedl, P., Sahai, E., Weiss, S., Yamada, K. M. New dimensions in cell migration. Nat. Rev. Mol. Cell Biol. 13 (11), 743-747 (2012).
- Grinnell, F., Petroll, W. M. Cell motility and mechanics in three-dimensional collagen matrices. Annu. Rev. Cell. Dev. Biol. 26, 335-361 (2010).
- Wirtz, D., Konstantopoulos, K., Searson, P. C. The physics of cancer: the role of physical interactions and mechanical forces in metastasis. Nat. Rev. Cancer. 11 (7), 512-522 (2011).
- Sun, W., Kurniawan, N. A., Kumar, A. P., Rajagopalan, R., Lim, C. T. Effects of migrating cell-induced matrix reorganization on 3D cancer cell migration. Cell. Mol. Bioeng. 7 (2), 205-217 (2014).
- Achilli, M., Mantovani, D. Tailoring mechanical properties of collagen-based scaffolds for vascular tissue engineering: the effects of pH, temperature and ionic strength on gelation. Polymers. 2 (4), 664-680 (2010).
- Kurniawan, N. A., Wong, L. H., Rajagopalan, R. Early stiffening and softening of collagen: interplay of deformation mechanisms in biopolymer networks. Biomacromolecules. 13 (3), 691-698 (2012).
- Sun, W., Lim, C. T., Kurniawan, N. A. Mechanistic adaptability of cancer cells strongly affects anti-migratory drug efficacy. J. R. Soc. Interface. 11 (99), 20140638 (2014).
- Guzman, A., Ziperstein, M. J., Kaufman, L. J. The effect of fibrillar matrix architecture on tumor cell invasion of physically challenging environments. Biomaterials. 35 (25), 6954-6963 (2014).
- Wolf, K., et al. Physical limits of cell migration: control by ECM space and nuclear deformation and tuning by proteolysis and traction force. J. Cell Biol. 201 (7), 1069-1084 (2013).
- Friedl, P., Gilmour, D. Collective cell migration in morphogenesis, regeneration and cancer. Nat. Rev. Mol. Cell Biol. 10 (7), 445-457 (2009).
- Vedula, S. R. K., Ravasio, A., Lim, C. T., Ladoux, B. Collective cell migration: a mechanistic perspective. Physiology. 28 (6), 370-379 (2013).
- Petrie, R. J., Doyle, A. D., Yamada, K. M. Random versus directionally persistent cell migration. Nat. Rev. Mol. Cell Biol. 10 (8), 538-549 (2009).
- Miron-Mendoza, M., Seemann, J., Grinnell, F. The differential regulation of cell motile activity through matrix stiffness and porosity in three dimensional collagen matrices. Biomaterials. 31 (25), 6425-6435 (2010).
- Mouw, J. K., et al. Tissue mechanics modulate microRNA-dependent PTEN expression to regulate malignant progression. Nat. Med. 20 (4), 360-367 (2014).
- Wolf, K., et al. Collagen-based cell migration models in vitro and in vivo. Semin. Cell Dev. Biol. 20 (8), 931-941 (2009).
- Wong, L. H., Kurniawan, N. A., Too, H. -. P., Rajagopalan, R. Spatially resolved microrheology of heterogeneous biopolymer hydrogels using covalently bound microspheres. Biomech. Model. Mechanobiol. 13 (4), 839-849 (2014).
- Gupton, S. L., Waterman-Storer, C. M. Spatiotemporal feedback between actomyosin and focal-adhesion systems optimizes rapid cell migration. Cell. 125 (7), 1361-1374 (2006).
- Provenzano, P. P., Inman, D. R., Eliceiri, K. W., Trier, S. M., Keely, P. J. Contact guidance mediated three-dimensional cell migration is regulated by Rho/ROCK-dependent matrix reorganization. Biophys. J. 95 (11), 5374-5384 (2008).
- Wolf, K., et al. Multi-step pericellular proteolysis controls the transition from individual to collective cancer cell invasion. Nat. Cell Biol. 9 (8), 893-904 (2007).
- Jansen, K. A., Bacabac, R. G., Piechocka, I. K., Koenderink, G. H. Cells actively stiffen fibrin networks by generating contractile stress. Biophys. J. 105 (10), 2240-2251 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved