Method Article
ゲノム編集したヒト多能性幹細胞株の樹立:分離へのターゲティングから
要約
Genome editing of human pluripotent stem cells (hPSCs) can be done quickly and efficiently. Presented here is a robust experimental procedure to genetically engineer hPSCs as exemplified by editing the AAVS1 safe harbor locus to express EGFP and introduce antibiotic resistance.
要約
ヒト多能性幹細胞(hPSCs)のゲノム編集は人間開発を理解し、疾患の病態生理学を研究する、そこから遺伝的に制御され、臨床的に関連するプラットフォームを提供します。ゲノム編集のための部位特異的ヌクレアーゼ(のSSN)を採用することにより、それ以外の場合は同質遺伝子の設定で特定の遺伝子変異を保有する新しいHPSCラインの迅速な誘導が可能となります。ジンクフィンガーヌクレアーゼ(ZFNは)、転写活性化因子のようなエフェクターヌクレアーゼ(TALENs)とクラスタ化された定期的interspaced短い回文繰り返し(CRISPR)/ Cas9は、最も一般的に使用されるのSSNです。これらのヌクレアーゼのすべては、それによってゲノム遺伝子座で正確な遺伝子編集を促進する、指定されたサイトで、二本鎖DNA切断を導入することによって機能します。 SSN-瞑想ゲノム編集は、挿入/欠失変異を導入したり代替するか、細胞の内因性DNA修復機構、非相同末端結合(NHEJ)と相同性を向い修理(HDR)のうちの2つを利用します二重鎖切断部位での相同修理テンプレートを使用してゲノムえー。 hPSCsのエレクトロポレーションは、蛍光レポーターおよび抗生物質耐性カセットとして導入遺伝子を組み込むのSSNや修理テンプレートをトランスフェクションの効率的な手段です。エレクトロポレーション後に、抗生物質耐性のために選択することによって、修復構築物を組み込んだだけhPSCsを単離することが可能です。機械的にHPSCコロニーを分離し、ジェノタイピングを通じて標的部位での適切な統合を確認すると、正確に標的と遺伝的に均一な細胞株を単離することができます。このプロトコルの有効性は、ヒト多能性幹細胞におけるAAVS1セーフハーバー座に構築EGFPおよびピューロマイシン耐性を組み込むためにすべての3つのSSNのプラットフォームを利用して、ここで示されています。
概要
ゲノム編集技術が急速に分子および細胞生物学の1のための標準的なツールに進化しています。 hPSCsは、遺伝的に無傷の初代ヒト細胞の自己再生ソースを表し、ヒト多能性幹細胞(hPSCs)の遺伝子操作は、特に重要です。 hPSCsは、疾患のモデリングのための、または移植療法2,3のソースのような種々の細胞型に分化させることができます。ここでの実証は、AAVS1遺伝子座にレポーター構築物の標的組み込みのための内因性DNA修復機構に関連して、部位特異的ヌクレアーゼ(SSNの)の3つの異なるタイプを使用するプロトコルです。 hPSCsへのSSNのトランスフェクションの後、我々は、レポーターを保有する同質遺伝子の細胞集団を分離する方法を示します。
特に多能性細胞のゲノムを幹、ゲノムを操作する能力は、使用してのSSNは、ジンクフィンガーヌクレアーゼ(ZFNを)および転写activatのユーティリティなどの新しい現象ではありません遺伝子編集またはのようなエフェクターヌクレアーゼ(TALENs)が数年前に4-10実証されました。しかし、S。ピオゲネスCRISPR / Cas9技術11-13の出現により、遺伝子編集は14広くアクセスできるようになりました。すべてのSSNは、非相同末端結合(NHEJ)または相同性向かう修理(HDR)15のいずれかを使用して、内因性の細胞メカニズムによって修復された特定の標的部位1,4,5,11で二本鎖DNA切断(DSB)を導入。 HDRは、SSNと修復鋳型の同時トランスフェクションを介して導入される新規な要素を可能にしながら、NHEJは、誤りがちであり、遺伝子機能の喪失をもたらすフレームシフト変異を導入することができます。遺伝子編集を促進するDNA修復の基本原理は、各SSNのために大部分が同じであると考えられている一方で、プラットフォーム間のいくつかの違いに注意することができます。のZFNのデノボ設計は柔軟性およびヌクレアーゼの最適化16、公にのが使用することができます個々のZFNを設計するために利用可能な組立ライブラリおよびスクリーニングツールは、時間がかかることがあります。 ZFN媒介性標的化のための所望の遺伝子座が決定されると、ZFN対は、オンラインツールZiFit 17を設計することができます。設計後、ZFNはモジュール式には、プラスミドクローニング18のいくつかのラウンドを通じて組み立てることができます。また、事前検証済みのZFN 19、市販の多くがあります。 TALEヌクレアーゼは、オンラインツールと公に利用可能なコンポーネント17,20を用いて設計することができます。例えば、TALENsは急速FLASHアセンブリ21またはPCRベースの階層的なゴールデンゲートアセンブリ 22 を使用してを通じて、5 TALEリピートのブロックから組み立てることができます。 SSNの設計およびCRISPR / Cas9を使用して、建設の速度やすさ、広くアクセス可能なツールを編集するゲノムをしました。ガイドRNAの多重化は単一の構築物14でいくつかの遺伝子座を標的とするようにするためのショートガイドRNAを媒介CRISPR / Cas9の標的化もできます。デザイン標的遺伝子座に近接し、遺伝子編集のためのCas9のprotospacer隣接モチーフ(S.用NGGトリヌクレオチドはCas9をpyrogenes PAM)の識別のみを必要とします。 px330プラスミド14にPAMの20塩基対の5 '末端に対応するオリゴヌクレオチドを挿入することによって、構築物は、一のクローニング工程で組み立てることができます。 Sに加えて、 N.からピオゲネス Cas9、Cas9 5'-NNNNGATT-3 '(PAM)を認識し、 髄膜炎菌 (NmCas9)はhPSCs 23で効率的な遺伝子編集を可能にすることが示されています。
SSN設計の容易さの違いに加えて、各プラットフォームに固有の特性を有します。例えば、ZFNはとTALENsはCas9は主に平滑末端のDSBを生成すると考えられている間に4ヌクレオチドの5 'オーバーハング24を発生させるのFokIヌクレアーゼドメインを利用します。 ZFNは、TALENs、およびCas9は、オンオフ率標的DNAに、それらのタンパク質の安定性に異なり、DNAスキャンのモードすべてがCの理論的には、編集結果1の小さな差が生じるウルド。さらなる研究は、完全にこれらの違いの影響を理解するために必要とされるが、ここではすべての3つのプラットフォーム間で非常に堅牢で、容易に遺伝的に改変されたhPSCsを生成するために使用できるプロトコルを記述する。
かかわらず、SSNの選択の、エレクトロポレーションは、hPSCs 25にSSNのと相同性の修理テンプレートをトランスフェクトする堅牢な手順です。抗生物質耐性のための選択後にコロニーの生存数は、遺伝子座特異的なパラメータと編集方針(トランスジェニック挿入と選択のモードの例えば、大きさ)に依存します。ここで説明するプロトコルは、通常、約150〜400単一細胞由来のコロニーを生じます。
このプロトコルを使用してAAVS1遺伝子座での遺伝子編集は、以前のSSN 4,5の有効性を実証するために使用されています。 AAV-CAGGS-EGFP修理テンプレートは、遺伝子トラップstrを使用しています遺伝子座特異的にピューロマイシン耐性を付与するategy。簡単に言えば、修理テンプレートは、プロモーターのピューロマイシン耐性カセットの上流のスプライスアクセプター部位を含みます。 AAVS1遺伝子座におけるPPP1R12C遺伝子の最初のイントロン中に正しい組込みに際して、耐性カセットを編集する遺伝子のプロモーターから発現されます。この特定のAAVS1アッセイの堅牢性は、私たちは各SSNプラットフォームの効率を比較することができます。
SSNのを用いた遺伝子編集は任意の遺伝子を破壊および/または理論的に変更する能力与えられた強力です。 hPSCsは、その後、このようなニューロン26,27を肝細胞、心筋細胞28のようなヒトの細胞型の多数に分化させることができるようにhPSCsにこの戦略を適用すると、汎用性を提供します。加えて、患者由来の誘導多能性幹細胞の使用は、患者固有の遺伝的背景29における修復または既知の疾患を引き起こす変異の導入を可能にします、病気のメカニズムおよび患者自身のセル 30 を用いた試験治療を調査するためのプラットフォームを提供します。要約すると、hPSCsにおける遺伝子編集は人間開発や病気31の基本的な生物学を研究するための効率的で汎用性の高い手法です。
プロトコル
この原稿に記載された手順を見直し、カリフォルニア大学バークレー校の幹細胞研究監視委員会によって承認されました。
1.編集するための幹細胞を準備します
- 成長し、マイトマイシンC不活性化マウス胚性線維芽細胞の2.4×10 6細胞 /プレートを含む6ウェルプレート上での培養ヒト多能性幹細胞(hPSCs)(MEF)フィーダーは、ゼラチン32上に成長させました。ウェルあたりヒト胚性幹細胞培地(hESC媒体)3mlにhPSCsを維持し、37で成長 3%O 2/5%CO 2で℃のインキュベーター。
注意:このプロトコルの成功のためには、低酸素培養器でhPSCsを維持する必要はありません。しかし、それは時間と細胞数がそれに応じて調整する必要がありますので、hPSCsは、高O 2の環境でより速く増殖することに留意することが重要です。それはまた、低酸素に維持hPSCs自発分化33の下側速度を有することに留意すべきです。- TO 500ミリリットルのhESCメディアは380ミリリットルのDMEM / F12、75ミリリットルウシ胎児血清(FBS)を組み合わせて行い、25ミリリットルノックアウト血清代替(KSR)。グルタミン(1mMの最終濃度)を、5 mlの100×非必須アミノ酸、100単位/ mlのペニシリン - ストレプトマイシン(P / S)、塩基性線維芽細胞増殖因子(bFGF)(4 ngの/ mlの最終濃度)、および2-加えますメルカプトエタノール(5.5μMの最終濃度)。
- ガラスピペット、真空を使用してメディアの全容量(3 ml)を除去することにより、メディアを変更します。 3ミリリットル暖かいhESC媒体が血清学的ピペットを使用して交換してください。 hPSCsが約50%コンフルエント(1日目)になるまで繰り返したメディアは毎日変更されます。
- 1日(1日目)を標的とする前に、古い培地を除去し、10μMのY-27632を補足した温かみのhESCのメディアを追加し、hESCのメディアを変更します。
- また、1日目に、DR4マウス(2.4×10 6細胞 /プレート)34から薬剤耐性MEFフィーダー細胞の2個の6ウェルまたは10センチメートルプレートに1を準備します。
注意:一般的には、6ウェルプレートは、広告されています彼らは多くのメディアに対応するように、10cmプレートの上にvantageous。 6ウェルプレートは、異なるウェルからの異なるクローンが独立していることを確認してください。しかし、10cmのプレートは、このプロセスのために利用可能な顕微鏡に応じて、簡単にピッキングを可能にします。
2.編集多能性幹細胞
- 1.5mlチューブにCRISPR / Cas9 px330エンコーディングプラスミド( 図1)のZFN 1と2、TALEN 1および2、または15μgの5μgの各ピペッティングし、トランスフェクション溶液を調製します。このチューブに修理プラスミドのピペット30μgのにも。最後に、チューブにピペット1×リン酸緩衝生理食塩水(PBS)を300μlに音量を表示します。
注:300μlの下のトランスフェクション溶液の全容積を維持するために、300 ngの/μLの最小濃度で(任意のキットが適しています)ミディプレップとしてプラスミドを準備します。これは、プラスミドを線形化するために、フェノール/クロロホルム抽出を行う必要がない場合、またはendotoを使用することXIN無料プラスミドプレップキット。 - ガラスピペット、真空を使用してhPSCsからメディアを取り出します。細胞を洗浄するために、各ウェルにピペット2ミリリットル暖かい1×PBS。
- ガラスピペット、真空を使用してすぐに、PBSを削除します。各ウェル中の細胞に直接0.5 mlの0.25%トリプシン-EDTA溶液を加えます。インキュベーターに入れ、約10分間(37℃/ 5%CO 2/3%O 2)。またはフィーダー層まで、プレートを持ち上げを開始します。
- トリプシン反応を停止し、各ウェルに2ミリリットル暖かいesWashメディア(470ミリリットルDMEM / F12、25ミリリットルのFBS、100単位/ mlのP / S)を追加します。
- フィーダー細胞がシートとして外れていることを確認してください。全てのウェルを組み合わせ、十分に単50 mlのコニカルチューブにそれぞれの内容をピペット。 10ミリリットルの血清学的ピペットを用いて粉砕物の細胞。これは、フィーダー層を破壊する必要はありません。 hPSCsは穏やかな粉砕により、フィーダー層の外れます。細胞懸濁液の約15ミリリットルがあるはずです。
- voluを持って来るためにチューブにesWashメディアの25ミリリットルを追加します。私は最大40 mlの全。大きなフィーダーチャンクが1〜2分間、チューブの底に沈降することを許可します。新しい50mlコニカルチューブに血清用ピペットと預金を使用してチューブから上清(〜38ミリリットル)を取り外します。
- 190×gで5分間スピンダウン。ガラスピペットと真空を用いてチューブから上清を取り除きます。細胞ペレットを乱さないようにしてください。 500μlの1×PBSで細胞を再懸濁します。以前調製したプラスミドのトランスフェクション溶液を用いて結合します。この段階で細胞をカウントします。エレクトロポレーションにつき5から10000000細胞を使用してください。
- 、4ミリメートルのエレクトロポレーションキュベットに全体800μlの懸濁物をピペットで3〜5分間氷上に置きます。 250 V、500μF、∞抵抗、および4ミリメートルキュベットサイズ:次のパラメータを使用してエレクトロポレーションシステムの指数関数プログラムを使用して電気穿孔細胞。エレクトロポレーション後、3分間バック氷上のキュベットを配置します。
注:エレクトロポレーションシステムにエレクトロポレーションの時定数を観察します。時定数細胞数及びDNAの純度に応じて変化します。記載された条件および遺伝子パルサーIIを使用した場合のトランスフェクションの成功は、通常、10〜14ミリ秒の時間定数を有します。時定数は、これらの値から変化した場合より低いエレクトロポレーション効率が発生する可能性があります。 - 暖かいhESCのメディアは、10μMのY-27632を補足した18ミリリットル中にエレクトロポレーションした細胞を再懸濁します。プレート3 DR4フィーダー細胞との6ウェルプレートの各ウェルに、この単一細胞懸濁液1ml。インキュベーター(37℃/ 5%CO 2/3%O 2)に戻ります。
陽性コロニーの3選択
- 2日目、ガラスピペットと真空を使用してすべてのメディアを削除します。 10μMのY-27632を補足した温かみのhESC媒体3mlのと交換してください。 3日目、ガラスピペットと真空を使用してすべてのメディアを削除します。 Y-27632を含まない温かいhESC媒体3mlのと交換してください。 4日目、ガラスピペットと真空を使用してすべてのメディアを削除します。 3mlでのSelectIための抗生物質を補充した温かみのhESCのメディアを交換してくださいに。インキュベーター(37℃/ 5%CO 2/3%O 2)に戻ります。
注意:使用する抗生物質の種類は、カセットが修理テンプレートに含ま抵抗に依存します。 WIBR#3 hPSCs(NIHレジストリ0079)、0.5 / mlのピューロマイシン、70μgの/ mlのG418(ジェネティシン)、および35 / mlのハイグロマイシンを操作する場合は正常に使用されてきました。選択のための濃度は、約1週間以内に野生型細胞を死滅させるのに必要な抗生物質の最小濃度を確立することによって経験的に決定されるべきです。 - 日5-12、変化媒体毎日、抗生物質を補充した温かみのhESC媒体で古いメディアを毎回交換します。
注:細胞死を大量に期待しています。個々のコロニーを、8~10日の周りに明らかになるであろう。抗生物質を含まない正規のhESC媒体は、連続選択の12-14日後に使用することができます。細胞密度が高い場合、または細胞死が遅い場合には、培地の酸性化は避けるべきであり、それはメディアvoluを増加させる必要があるかもしれません私(4〜5ミリリットルまで)選択の最初の数日間。
4.ピッキング選択したコロニー(日12-14)
- 日12〜14のコロニーを観察します。解剖顕微鏡で選び、それらが分化し始めている細胞が含まれていないことを確実にするために準備ができているコロニーを観察します。おおよそのサイズは、800-1,200μMでなければなりません。コロニーは12日前にこのサイズに達すると、それは彼らが次に選んだすることをお勧めします。
注:各ターゲット実験では、目的の遺伝子型が分離されていることを確認するために必要な数のコロニーを選択します。例として、AAV-CAGGS-EGFP修理テンプレートとAAVS1編集は一貫して強固な統合が示されており、唯一の12〜24コロニーが約5〜10ヘテロ接合の標的クローンを得るために取りに行く必要がありました。他の標的の実験を採取するために多くのコロニー( 表1)を必要とする場合があります。正しいターゲッティング事象の頻度は、SSの効率などの要因に依存しますDSB、インサートのサイズ、および選択戦略を導入するN。正しく適切に標的化クローンを得るために必要なコロニーの数を減少させる、標的部位に組み込まれたときにここに提示AAVS1遺伝子座の遺伝子トラップ法の場合には、選択マーカーは、発現されます。 - ピッキングの前に、MEFを12ウェルプレートを準備する1日(2.4×10 6細胞 /プレート、1つのウェルは、各コロニーのために使用されます選びました)。
- ピッキングの日に、ガラスピペットと真空を用いて12ウェルプレートにMEFからすべてのメディアを削除し、1ミリリットルのhESCのメディアと交換してください。またピッキングの日に、ピックアップしようとしている6ウェルプレート上のhESCのメディアを変更します。
- フィーダー層からの個々のコロニーを機械的に解離用ガラスピペットを引き出します。イン・フードブンゼンバーナーの上にピペットを下げ、ガラスが可鍛性になるまで展開点で軟化。すぐに炎から削除して、角度のポイントを作成離れてピペットを引き出します。ブレイク狭いチャネルを残して、曲がった軸から約2cmを指します。 1-2秒間炎に暴露することによって、チャネルの端部を磨きます。
注:鈍い先端は、潜在的に、その後の採集物を汚染する、メディアに各コロニーからの細胞のより多くの量を除去することができるよう必要に応じてコロニーをP20ピペットチップを選ぶが、これは、文化のクローン性を減少させることができます。 - P1000のフィルターピペットチップを取り、狭い端部に吸引電球を取り付けることによって、デバイスを選んで組み立てます。ワイド端に引っ張らガラスピペットを挿入します。
- 組織培養フードに取り付けられた解剖顕微鏡のステージに選ばれるべきhPSCsの6ウェルプレートを置きます。ピッキング装置の電球を圧縮し、ゆっくりと10〜20の等しいサイズのピースに個々のコロニーを切除し、カット、メディアに作品をリリースしないように注意して。電球を解放することにより、ピペットにコロニーの切除したHPSC片を取ります。転送中にできるだけ少ないメディアを取るようにしてください。
- TRANSF12ウェルプレートMEFの1ウェルに、個々のコロニー、今壊れて植民地を解放し、ウェルに直接再び電球を圧縮えー。単一細胞由来のクローンの固有の識別を可能にするために、各ウェルにラベルを付けます。ガラスピペットを毎回変え、必要な数のコロニーを繰り返します。
注:ピッキングコロニーを学ぶのに時間がかかる場合があります。これは、対象となるコロニーを分離しようとする前にいくつかのコントロール細胞の実験の練習することをお勧めします。 - 穏やかに、各ウェルの中央に細胞の蓄積を回避するために、第一プレートを揺動、インキュベーター(37℃/ 3%O 2/5%CO 2)で12ウェルプレートを返します。
- 翌日とそれ以降の毎日は、ガラスピペット、真空を使用してメディアのフルボリュームを削除して、細胞が約50%コンフルエントになるまで温かいhESC媒体1.5mlのと交換してください(これは通常10〜12日かかります)。
- 10〜12日後、各ウェルから1〜2個のコロニーを選択し、再新しい12ウェルプレートに移すMEFpeatingは、レプリカプレートを生成するために、4.1から4.8を繰り返します。
- 元のMEFプレートの各ウェルに残っているコロニーから(下記参照)のDNAを抽出します。
- ガラスピペットと真空を使用して、すべてのウェルから培地を除去。ピペット1mlの細胞を洗浄し、各ウェルにPBSを1倍。ガラスピペット、真空を使用して、PBSを削除します。ピペット細胞溶解緩衝液250μlの(H 2 O中の最終濃度:10mMのトリスHCl、5mMのEDTA、0.2%SDS、200mMのNaCl、0.08 mg / mlのプロテイナーゼK)の各ウェルに。インキュベーターに入れ(37℃/ 3%O 2/5%CO 2)O / N。
- 次の日、ピペットそれぞれの内容だけでなく、個々に1.5ミリリットルチューブ。ピペット各チューブにイソプロピルアルコール250μlのDNAを沈殿させました。精力的にチューブを振ります。白色の沈殿物が表示されるはずです。
- テーブルトップ遠心機で13,000 rpmで3分間、各チューブをスピンダウン。スピンダウンした後、液体廃棄物の中にデカントすることにより、上清を捨てます。小さなDNAペレットの底に付着したままにしておく必要がありチューブ。 DNAペレットを洗浄するために、各チューブに250μlの70%エタノールをピペット。勢いよく振ります。テーブルトップ遠心機で13,000 rpmで3分間、各チューブをスピンダウン。
- スピンダウンした後、液体廃棄物の中にデカントすることにより、上清を捨てます。小さなDNAペレットをチューブの底に付着したままにしてください。チューブ内に残された液体が存在しないので、上清の残りをピペット。 5〜10分間、ベンチトップ上で乾燥オープンチューブを残します。乾燥後、250μlのTE緩衝液中にDNAを再懸濁します。 DNAを溶解させるために6時間37℃で置きます。
- あらかじめ最適化されたPCRまたはサザンブロット戦略を使用して、各サンプルを遺伝子型。
注意:AAVS1は、AAV-CAGGS-EGFP修理テンプレートを使用して標的とするため、サザンブロット戦略がら 、2009年4総合的なサザンブロット法プロトコルは、南部で見つけることができHockemeyerで見つけることができます2006年35のマルチプレックスPCRジェノタイピングのために。同じ実験を、プライマー及び条件がすることができます表2 36で見つかりました。 - 適切に標的にされていない井戸を捨て、日々適切に標的細胞上のhESCのメディアを変更し続けています。彼らは約50%コンフルエントである場合、ダウン細胞凍結プロトコルを凍結については、以下を参照してください。
- ある日、凍結前に、古い培地を除去し、10μMのY-27632を補足した温かみのhESCのメディアを追加し、hESCのメディアを変更します。
- 凍結の日にある2つの15 mlのコニカルチューブにダウン凍結されている12ウェルプレートのウェル当たり、溶液Aと溶液Bの0.5 mlずつを準備します。氷の上でソリューションを配置します。溶液A:50%のhESCのメディアと、50%のFBS;溶液B:80%FBS、20%ジメチルスルホキシド(DMSO)。
- 凍結前に、単一の細胞にHPSCコロニーを解離します。それに応じてボリュームをスケーリング、これを行うための手順2.2から2.8に従ってください。溶液Aを0.5mlで完全に再懸濁細胞ペレット
- 細胞懸濁液を均質にするために細胞懸濁液、上下ピペットに0.5ミリリットル溶液Bを追加します。タークE 2ミリリットルクライオチューブに細胞懸濁液(〜1ml)および預金の合計容量。しっかりとキャップをねじ込みます。
- -80℃の冷凍庫O / Nで置きクライオチューブ。凍結後の日は、-80℃の冷凍庫から凍結細胞を除去し、すぐに長期保存のために液体窒素タンクに置きます。
結果
ここでは、遺伝子工学的にHPSCラインを作成するには、3つの異なるSSNのプラットフォームと互換性のあるプロトコルを示しています。我々は、EGFPレポーターおよびピューロマイシン耐性カセット4を導入し 、修復テンプレートを使用してTALENs 5、以前に発表されたZFN 4を使用して AAVS1遺伝子座でWIBRの#3ヒト胚性幹細胞を標的としCRISPR / Cas9s 37。
我々は、未分化hPSCsの維持・拡大を可能にする細胞培養ワークフロー( 図2)のためのMEF上で私たちのhPSCsを培養し、それはまた、効果的かつスケーラブルなコストです。細胞が必要以上に成長している場合は、トランスフェクトされた多能性細胞の数及びその結果として得られる正しく標的HPSCのコロニーの数を減少させる、増加した分化の危険性があります。
我々はelectropSSNのプラットフォームあたり5.0×10 6個の細胞をoratedとDR4 MEFを、単一の6ウェルプレートに、各ターゲットから細胞を播種しました。選択後、各プラットフォームは、EGFP陽性コロニー( 図3)と、非標的ホモターゲットとヘテロで対象となるクローン(;表3、図4A、B)の組み合わせとなりました。ここで紹介する条件の下で、我々はAAVS1 TALENsは、ほとんどのEGFP陽性クローンが得られたことがわかります。この実験に用いた補修テンプレートは、EGFPレポーターおよびピューロマイシン耐性カセットの上流のスプライス受容部位で構成されています。この「遺伝子トラップ」戦略( 図1)を使用して 、構築物は、ピューロマイシン耐性カセットの発現を駆動するために、内因性プロモーターを用いて、AAVS1遺伝子座の最初のイントロンに挿入する必要があります。での発現を防止する必要があり、修理テンプレート内のピューロマイシン耐性遺伝子の発現を駆動するプロモーターの欠如ランダム統合のイベント。
したがって、我々はすべてのピューロマイシン耐性クローンがAAVS1サイトで標的にすることを期待しています。それはではなく、ほとんどのPCR戦略( 図1; 図4C)により、クローンのサブセットが内部プローブを用いたサザンブロットによって検出されているAAVS1遺伝子座における異常の統合を運ぶことに留意すべきである4。これらの統合イベントは、最も可能性の高いドナープラスミド4の倍数の統合につながる異種ターゲティング事象の結果です。私たちは、それぞれのSSNの実験から24個のコロニーを採取し、すべてのプラットフォームが非常に高いターゲティング効率を有しており、最小限の違いを示したことがわかりました。 PCRにより尋問のようTALENプラットフォームは、ほとんどのホモターゲットのクローン( 表3)を有しながら、CRISPR / Cas9は最も正確に標的のクローンが得られました。
/files/ftp_upload/53583/53583fig1highres.jpg "幅=" 700 "/>
遺伝子の 図1 の回路図、AAV-CAGGS-EGFP修理テンプレートを使用してAAVS1遺伝子座を編集しました。Hockemeyer らから変更された。2009年、この図の拡大版をご覧になるにはこちらをクリックしてください。
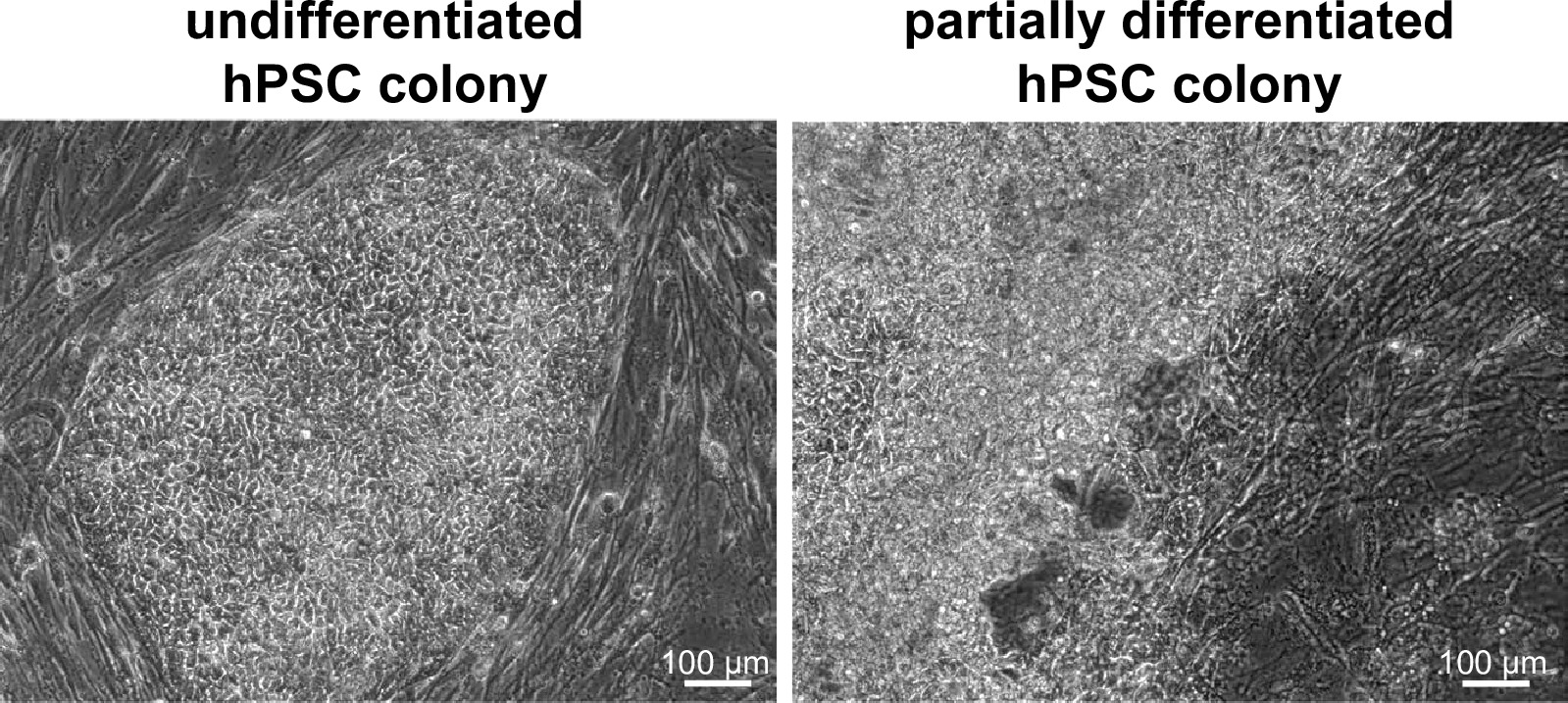
図WIBR#3セルの2コロニー。ターゲットの前にWIBR#3ヒト胚性幹細胞のコロニーの代表的な明視野像。分化の欠如と非理想的なもの(右)とは対照的に、理想的なコロニー(左)におけるフィーダー層から明確に分離に注意してください。 この図の拡大版をご覧になるにはこちらをクリックしてください。
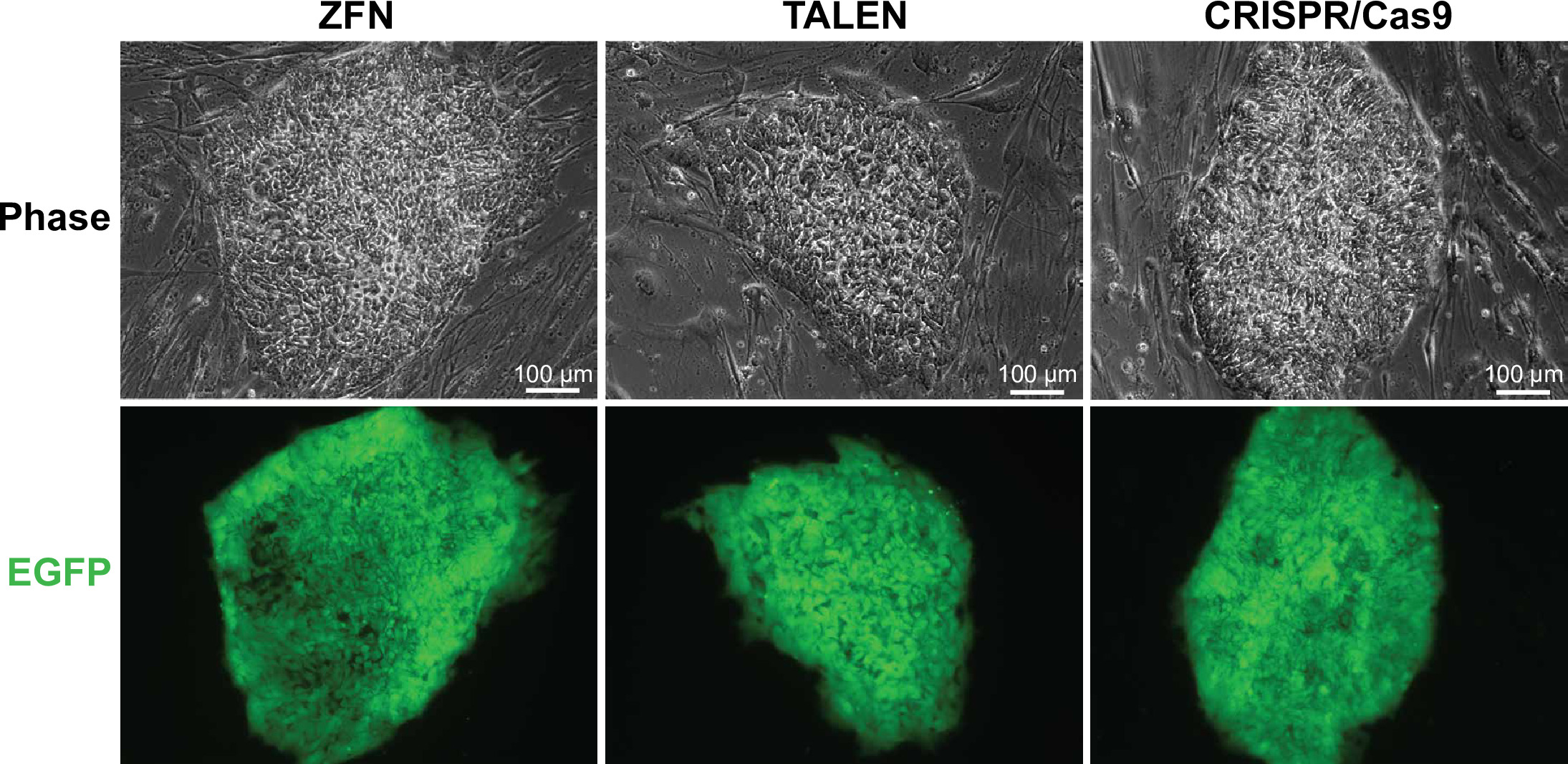
図3. EGFP陽性WIBR#3セル。WIBRの#AAVS1遺伝子座におけるEGFP発現修理テンプレートの標的と3細胞の代表的な画像。 ZFNで編集代表コロニーの画像、TALENsおよびCRISPR / Cas9が示されている。 この図の拡大版をご覧になるにはこちらをクリックしてください。
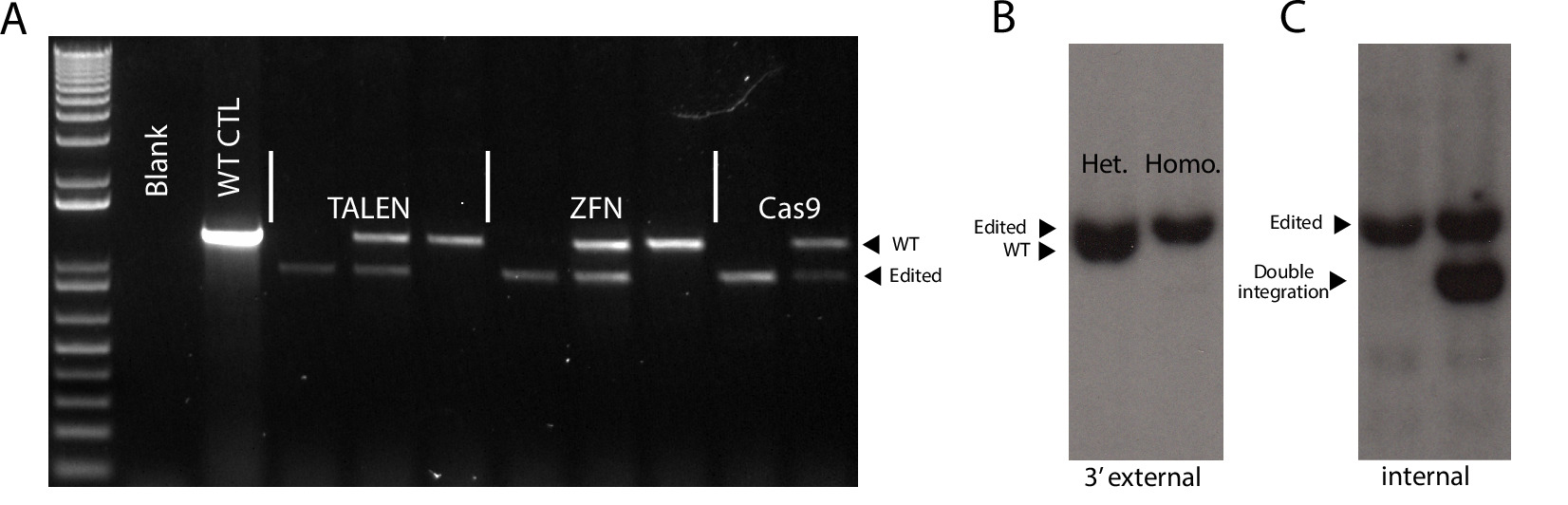
図4.ジェノタイピング戦略正しいターゲティングを確認する。(A)代表のPCR遺伝子型判定結果3 SSNのプラットフォーム間で、非標的ヘテロ接合およびホモ接合の標的クローンを示します。 WT CTLは野生型コントロールを=。 (B)Repreヘテロ接合の標的クローンおよび3 '外部プローブを用いて検出ホモ接合対象となるクローンを示すsentativeサザンブロットの結果。フラグメントサイズ:WT-6.5キロバイト、編集・6.9キロバイト(C)適切に編集したクローンと非ランダム二重統合とヘテロ接合クローンを示す代表的なサザンブロットの結果。フラグメントサイズ:適切に編集した-6.9キロバイト、異常な追加の統合-5キロバイトこの図の拡大版をご覧になるにはこちらをクリックしてください。
| 研究 | 遺伝子ターゲット | プラットフォーム | 修理テンプレートの種類 | 取り上げたクローンの# | ターゲティング効率 |
| セクストンら 、2014 | TPP1 | ZFN | GFP-ピューロ | 報告されていません | 報告されていません |
| セクストンら 、2014 | TERT | ZFN | ハイグロマイシン | 報告されていません | 報告されていません |
| Hockemeyer ら 、2009 | POU5F1 | ZFN | GFP-ピューロ | 31 | 39.0パーセント |
| Hockemeyer ら 、2009 | PITX3 | ZFN | GFP-ピューロ | 74 | 14.9% |
| Hockemeyer ら 、2011 | POU5F1 | TALEN | GFP-ピューロ | 68 | 91.0パーセント |
| Hockemeyer ら 、2011 | PITX3 | TALEN | GFP-ピューロ | 96 | 13.0% |
| マークルら 、2015 | VASA | Cas9 | レポーター、ジェネティシン | 139 | 94.0パーセント |
| マークルら</ em>の、2015 | CRH | Cas9 | レポーター、ジェネティシン | 30 | 93.0パーセント |
| マークルら 、2015 | HCRT | Cas9 | レポーター、ジェネティシン | 154 | 92.0パーセント |
| マークルら 、2015 | HMX2 | Cas9 | レポーター、ジェネティシン | 11 | 45.0パーセント |
| フォースターら 、2014 | LRG5-Nterm | ZFN | GFP-ピューロ | 報告されていません | 30.0% |
| フォースターら 、2014 | LRG5-CTERM | ZFN | GFP-ピューロ | 報告されていません | 14.0% |
| Soldner ら 、2011 | SNCA | ZFN | ピューロ | 96 | 1.0% |
カレとこの方法を用いて標的他の遺伝子の表1の説明、以前に発表された研究から取られた効率をターゲットsponding。4,5,38-41
| 軌跡 | シーケンス | ノート |
| AAVS1-Fプライマー | CTCTAACGCTGCCGTCTCTC | PCR条件:T mを= 57℃、35サイクル |
| AAVS1-WT-Rプライマー | GCTTCTCCTCTTGGGAAGTG | WTバンド:1273塩基対 |
| AAVS1-ターゲット-Rプライマー | CGTCACCGCATGTTAGAAGA | ターゲットバンド:992塩基対 |
| T2-Cas9ガイド | GGGCCACTAGGGACAGGAT | マリら 、2013年から |
| AAVS1-ZFN右 | TAGGGACAGGAT | Hockemeyer ら 、2009年から |
| AAVS1-ZFN左 | TGGGGTGTCACC | Hockemeyer ら 、2009年から |
| AAVS1-TALEN右 | TCCTAACCACTGTCTTT | Hockemeyer ら 、2011年から |
| AAVS1-TALEN左 | CCCCTCCACCCCACAGT | Hockemeyer ら 、2011年から |
プライマーとSSN標的化配列の表2のリスト。
| 標的構 築物 | EGFP +コロニー数 | ターゲット取り上げたクローン(PCRを検証) | (サザンブロットによる)前報告正しいターゲティング効率 |
| ZFN | 150 | 86.9パーセント(73.9パーセントのヘタ/ 13.0%ホモ) | 56%(50%HET / 6%のホモ) |
| TALEN | 412 | 91.3パーセント(47.8パーセントのヘタ/ 39.1パーセントホモ) | 47%(37.5%のヘタ/ 9.3%ホモ) |
| CRISPR-Cas9 | 235 | 95.7パーセント(69.5パーセントHET / 26.3%ホモ) | 報告されていません |
表3.比較EGFP陽性の数とはTALEN、ZFNを対象とし、CRISPR / Cas9ヒト幹細胞コロニー。PCRは、以前の実験4,5に適切な単一の統合検証サザンブロットと比較されるこの実験のAAVS1遺伝子座での統合を検証しました。
ディスカッション
遺伝子編集されたヒト多能性幹細胞の均質な集団を単離するため、ここで提示された方法は、標的遺伝子座でのみ異なる同質遺伝子HPSC株を生成するための強力なアプローチです。これらの細胞は、ヒト細胞の分化および発生のメカニズムを探索するためだけでなく、制御の遺伝的環境の中で単一遺伝子疾患の病態生理を理解するための理想的なシステムです。ここで実証されるように、それはAAVS1遺伝子座での標的組込みを達成するために、3つの独立したSSNの設計戦略(ZFNは、TALENs、およびCRISPR / Cas9)を使用することが可能です。これらの方法のそれぞれが独自の長所と短所があります。電位のZFNの利点とは、ある程度、TALENsは個々ヌクレアーゼ42のDNA結合ドメインを改善するために、反復エンジニアリングを可能にするそれらの設計の柔軟性です。このヌクレアーゼの最適化は、CRIで達成可能であるものを超えてのZFNとTALENsの特異性を増加させることができますSPR / Cas9システム。このような選択性は、標的特異性の高い程度を必要とする臨床応用のために重要であり得ます。 CRISPR / Cas9システムの主な利点は、その使いやすさです。 TALENとZFNコンストラクションキットは、パブリック( すなわち、Addgene〜21)が利用できるようにしてきたがpx330プラスミドを使用する場合にのみ必要なカスタマイズが20塩基対のオリゴヌクレオチド(あるとして、CRISPR / Cas9ベースのSSNは、構築する非常に容易ですデザイン14)。このシンプルさは、彼らの研究におけるゲノム編集を含めるように探している研究室のために有利であることがわかります。
遺伝子編集しHPSCラインを作成するためのヌクレオ43、などの代替トランスフェクション技術があります。しかし、エレクトロポレーションは、一貫して費用対効果4,5,25であることが示されています。ヌクレオはSSN効率ANの増加、直接核にCas9ガイドRNAのリボ核タンパク質複合体をトランスフェクトするために使用することができDの忠実度44。 MEFの上に成長hPSCsは、過剰な分化せずに多能性状態でhPSCsを維持するために、堅牢かつ安価な方法です。また、遺伝的に同一のコロニーの容易な単離を可能にします。あるいは、しかしながら、これらの培養条件は、フィーダベースの培養よりも高価になることができ、MEFのない培養hPSCsすることが可能です。さらに、全体のプロセスは非常にまれな編集イベントの単離または並列編集実験で多数の異なる細胞株の生成を可能にする、拡張可能です。
ここで説明するプロトコルは強固です。しかし、正しく編集したクローンを得ることができると効率に影響を与えるいくつかの重要なステップがあります。この方法のための最も重要なコンポーネントは、高品質のMEFと薬剤耐性のDR4のMEFをしています。単一hPSCsの生存が希薄で、低品質のMEFは未分化HPSCラインの分離を妨げます。第二に、Y-27632の使用でもあります異常な核型45を有する細胞に対して選択的な圧力を生成せずに、単一の細胞の生存を可能にするキー。第三に、十分な間隔コロニーを選ぶことは由来細胞株の遺伝的均質性を保証します。最後に、開口部は、複数のコロニーを新しいウェルに成長することを確認して、多くの小片にコロニーを破壊するのに十分に小さくなるようにガラスピペットを準備することが重要です。これは、遺伝子型決定のために単離したコロニーの原版を残し、文化の中で持っているレプリカプレートでよく間隔のサブクローンのピッキングが可能になります。このプロトコルで挑戦的な部分は、ガラスピペットの引き上げマニュアルです。これは、事前に実施されなければなりません。これは、ガラスピペットを必要としないクローンピッキングの複数の技術が存在することに留意すべきです。実験者は、彼らのために最も適したものを見つけるために奨励されています。
単純な修正によって克服することができるこのプロトコルには制限があります。 Oに意図された実験NLY編集された細胞を富化する別の方法を使用する必要があり、修理またはその修理テンプレート選択カセットを含まないものなしに関心の座を崩壊させます。陽性クローンを見つけるために選ばれなければならないコロニーの数は、選択の非存在下では大幅に増加することに注意してください。効率を改善する1つの戦略は、蛍光タンパク質を発現する非組み込みプラスミドを同時トランスフェクトすることです。細胞は2日間回復させた後、標的細胞は、蛍光活性化セルソーティングを使用して正の蛍光でソートし、29を再播種することができます。このプロセスは、エレクトロポレーションによりプラスミドでトランスフェクトされた細胞のために豊かにし、それによって細胞内での同時編集イベントの確率が高くなります。
ここに記載された技術は、同時に複数の遺伝子座14,46,47を標的とするために 、複数のガイドRNAを使用するように拡張することができます。多くのプロトコルは、hPSCsを区別するために設立されました異なる細胞型に、関心対象30の細胞型における種々の遺伝子操作を可能にします。全体的に、我々は関係なく、SSNの選択のhPSCsで効率的なゲノム編集の可能性を実証しました。我々は、この技術は、任意のゲノム遺伝子座での遺伝子編集された同質遺伝子HPSCラインを作成するように構成することができることを提案します。
開示事項
著者は全く競合する金融利益を宣言しません。
謝辞
この作品は、ヘレンBateupに脳研究財団のシード・グラント(BRFSG-2014から02)によってサポートされていました。ディルクHockemeyerは、エリソン医学財団のエイジングの新学者で、グレン財団だけでなく、ザ・Shurlとケイクルチ財団によってサポートされています。 DHはまた、NIHの助成金1R01CA196884-01によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| DMEM/F12 | Life Technologies | 11320082 | |
| Fetal Bovine Serum (HI) | Life Technologies | 10082-147 | |
| Knockout Serum | Life Technologies | 10828-028 | |
| Fibroblast Growth Factor - basic | Life Technologies | PHG0261 | |
| Pen/Strep | Life Technologies | 15140-122 | |
| Glutamine | Life Technologies | 25030-081 | |
| MEM NEAA | Life Technologies | 11140-050 | |
| 2-mercaptoethanol | Life Technologies | 21985-023 | |
| Y-27632 | Calbiochem | 688000 | |
| 6-well plates | Corning | 3506 | |
| 12-well plates | Corning | 3512 | |
| 4 mm Electroporation cuvettes | Bio-rad | 165-2081 | |
| X-cel gene pulser II | Bio-rad | 165-2661 | |
| 0.25% Trypsin-EDTA | Life Technologies | 25200-056 | |
| 10× Phosphate buffered saline (PBS) pH7.4 | Life Technologies | 70011-044 | |
| Puromycin | Life Technologies | A11138-02 | |
| Pasteur pipettes, plugged | VWR | 14672-412 | |
| Tris-HCl | Sigma-Aldrich | S5941 | |
| NaCl | Sigma-Aldrich | S9888 | |
| SDS | Sigma-Aldrich | L3771 | |
| EDTA | Sigma-Aldrich | E5134 | |
| Proteinase-K | Life Technologies | AM2544 | |
| Ethanol | VWR | TX89125-172SFU | |
| Isopropyl Alcohol | VWR | MK303216 | |
| TE Buffer | Life Technologies | 12090-015 | |
| Dimethyl Sulfoxide (DMSO) | Sigma-Aldrich | D8418 | |
| 1.8 ml Cryotubes | ThermoScientific | 377267 |
参考文献
- Carroll, D. Genome engineering with targetable nucleases. Annu Rev Biochem. 83, 409-439 (2014).
- Lancaster, M. A., Knoblich, J. A. Organogenesis in a dish: Modeling development and disease using organoid technologies. Science. 345 (6194), 1247125 (2014).
- Matsa, E., Burridge, P. W., Wu, J. C. Human stem cells for modeling heart disease and for drug discovery. Sci Transl Med. 6 (239), 239ps6 (2014).
- Hockemeyer, D., et al. Efficient targeting of expressed and silent genes in human ESCs and iPSCs using zinc-finger nucleases. Nat Biotechnol. 27 (9), 851-857 (2009).
- Hockemeyer, D., et al. Genetic engineering of human pluripotent cells using TALE nucleases. Nat Biotechnol. 29 (8), 731-734 (2011).
- Bozas, A., Beumer, K. J., Trautman, J. K., Carroll, D. Genetic analysis of zinc-finger nuclease-induced gene targeting in drosophila. Genetics. 182 (3), 641-651 (2009).
- Bibikova, M., Golic, M., Golic, K. G., Carroll, D. Targeted chromosomal cleavage and mutagenesis in drosophila using zinc-finger nucleases. Genetics. 161 (3), 1169-1175 (2002).
- Miller, J. C., et al. A TALE nuclease architecture for efficient genome editing. Nat Biotechnol. 29 (2), 143-148 (2011).
- DeKelver, R. C., et al. Functional genomics, proteomics, and regulatory DNA analysis in isogenic settings using zinc finger nuclease-driven transgenesis into a safe harbor locus in the human genome. Genome Res. 20 (8), 1133-1142 (2010).
- Urnov, F. D., et al. Highly efficient endogenous human gene correction using designed zinc-finger nucleases. Nature. 435 (7042), 646-651 (2005).
- Jinek, M., Chylinski, K., Fonfara, I., Hauer, M., Doudna, J. A., Charpentier, E. A programmable dual-RNA-guided DNA endonuclease in adaptive bacterial immunity. Science. 337 (6096), 816-821 (2012).
- Jinek, M., East, A., Cheng, A., Lin, S., Ma, E., Doudna, J. RNA-programmed genome editing in human cells. Elife. 2, e00471 (2013).
- Doudna, J. A., Charpentier, E. Genome editing. the new frontier of genome engineering with CRISPR-Cas9. Science. 346 (6213), 1258096 (2014).
- Cong, L., et al. Multiplex genome engineering using CRISPR/cas systems. Science. 339 (6121), 819-823 (2013).
- Jasin, M. Genetic manipulation of genomes with rare-cutting endonucleases. Trends Genet. 12 (6), 224-228 (1996).
- Urnov, F. D., Rebar, E. J., Holmes, M. C., Zhang, H. S., Gregory, P. D. Genome editing with engineered zinc finger nucleases. Nat Rev Genet. 11 (9), 636-646 (2010).
- Sander, J. D., Maeder, M. L., Reyon, D., Voytas, D. F., Joung, J. K., Dobbs, D. ZiFiT (zinc finger targeter): An updated zinc finger engineering tool. Nucleic Acids Res. 38, W462-W468 (2010).
- Carroll, D., Morton, J. J., Beumer, K. J., Segal, D. J. Design, construction and in vitro testing of zinc finger nucleases. Nat Protoc. 1 (3), 1329-1341 (2006).
- Chen, F., et al. High-frequency genome editing using ssDNA oligonucleotides with zinc-finger nucleases. Nat Methods. 8 (9), 753-755 (2011).
- Cermak, T., et al. Efficient design and assembly of custom TALEN and other TAL effector-based constructs for DNA targeting. Nucleic Acids Res. 39 (12), e82 (2011).
- Reyon, D., Tsai, S. Q., Khayter, C., Foden, J. A., Sander, J. D., Joung, J. K. FLASH assembly of TALENs for high-throughput genome editing. Nat Biotechnol. 30 (5), 460-465 (2012).
- Sanjana, N. E., Cong, L., Zhou, Y., Cunniff, M. M., Feng, G., Zhang, F. A transcription activator-like effector toolbox for genome engineering. Nat Protoc. 7 (1), 171-192 (2012).
- Hou, Z., et al. Efficient genome engineering in human pluripotent stem cells using Cas9 from neisseria meningitidis. Proc Natl Acad Sci U S A. 110 (39), 15644-15649 (2013).
- Podhajska, A. J., Szybalski, W. Conversion of the FokI endonuclease to a universal restriction enzyme: Cleavage of phage M13mp7 DNA at predetermined sites. Gene. 40 (2-3), 175-182 (1985).
- Costa, M., et al. A method for genetic modification of human embryonic stem cells using electroporation. Nat Protoc. 2 (4), 792-796 (2007).
- Chambers, S. M., Fasano, C. A., Papapetrou, E. P., Tomishima, M., Sadelain, M., Studer, L. Highly efficient neural conversion of human ES and iPS cells by dual inhibition of SMAD signaling. Nat Biotechnol. 27 (3), 275-280 (2009).
- Cai, J., et al. Directed differentiation of human embryonic stem cells into functional hepatic cells. Hepatology. 45 (5), 1229-1239 (2007).
- Kehat, I., et al. Human embryonic stem cells can differentiate into myocytes with structural and functional properties of cardiomyocytes. J Clin Invest. 108 (3), 407-414 (2001).
- Soldner, F., et al. Parkinson's disease patient-derived induced pluripotent stem cells free of viral reprogramming factors. Cell. 136 (5), 964-977 (2009).
- Wen, Z., et al. Synaptic dysregulation in a human iPS cell model of mental disorders. Nature. 515 (7527), 414-418 (2014).
- Chiba, K., Hockemeyer, D. Genome editing in human pluripotent stem cells using site-specific nucleases. Methods Mol Biol. 1239, 267-280 (2015).
- Jozefczuk, J., Drews, K., Adjaye, J. Preparation of mouse embryonic fibroblast cells suitable for culturing human embryonic and induced pluripotent stem cells. J Vis Exp. , (2012).
- Ezashi, T., Das, P., Roberts, R. M. Low O2 tensions and the prevention of differentiation of hES cells. Proc Natl Acad Sci U S A. 102 (13), 4783-4788 (2005).
- Tucker, K. L., Wang, Y., Dausman, J., Jaenisch, R. A transgenic mouse strain expressing four drug-selectable marker genes. Nucleic Acids Res. 25 (18), 3745-3746 (1997).
- Southern, E. Southern blotting. Nat Protoc. 1 (2), 518-525 (2006).
- Henegariu, O., Heerema, N. A., Dlouhy, S. R., Vance, G. H., Vogt, P. H. Multiplex PCR: Critical parameters and step-by-step protocol. BioTechniques. 23 (3), 504-511 (1997).
- Mali, P., et al. RNA-guided human genome engineering via Cas9. Science. 339 (6121), 823-826 (2013).
- Sexton, A. N., et al. Genetic and molecular identification of three human TPP1 functions in telomerase action: Recruitment, activation, and homeostasis set point regulation. Genes Dev. 28 (17), 1885-1899 (2014).
- Merkle, F. T., et al. Efficient CRISPR-Cas9-mediated generation of knockin human pluripotent stem cells lacking undesired mutations at the targeted locus. Cell Rep. 11 (6), 875-883 (2015).
- Soldner, F., et al. Generation of isogenic pluripotent stem cells differing exclusively at two early onset parkinson point mutations. Cell. 146 (2), 318-331 (2011).
- Forster, R., et al. Human intestinal tissue with adult stem cell properties derived from pluripotent stem cells. Stem Cell Reports. 2 (6), 838-852 (2014).
- Miller, J. C., et al. Improved specificity of TALE-based genome editing using an expanded RVD repertoire. Nat Methods. , (2015).
- Hohenstein, K. A., Pyle, A. D., Chern, J. Y., Lock, L. F., Donovan, P. J. Nucleofection mediates high-efficiency stable gene knockdown and transgene expression in human embryonic stem cells. Stem Cells. 26 (6), 1436-1443 (2008).
- Lin, S., Staahl, B. T., Alla, R. K., Doudna, J. A. Enhanced homology-directed human genome engineering by controlled timing of CRISPR/Cas9 delivery. Elife. 3, e04766 (2014).
- Watanabe, K., et al. A ROCK inhibitor permits survival of dissociated human embryonic stem cells. Nat Biotechnol. 25 (6), 681-686 (2007).
- Sakuma, T., Nishikawa, A., Kume, S., Chayama, K., Yamamoto, T. Multiplex genome engineering in human cells using all-in-one CRISPR/Cas9 vector system. Sci Rep. 4, 5400 (2014).
- Ousterout, D. G., Kabadi, A. M., Thakore, P. I., Majoros, W. H., Reddy, T. E., Gersbach, C. A. Multiplex CRISPR/Cas9-based genome editing for correction of dystrophin mutations that cause duchenne muscular dystrophy. Nat Commun. 6, 6244 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved