Method Article
液体クロマトグラフィー/質量分析法による有毒フグの改善されたポリメラーゼ連鎖反応 - 制限断片長多型ジェノタイピング
要約
液体クロマトグラフィー/質量分析によってフグ種を遺伝子型決定するための改良されたポリメラーゼ連鎖反応 - 制限断片長多型法が記載されています。逆相シリカモノリスカラム消化アンプリコンを分離するために使用されます。この方法は、ベース組成物を同定するのに有用であるオリゴヌクレオチドのモノアイソトピック質量を、解明することができます。
要約
ポリメラーゼ連鎖反応(PCR)-restriction断片長多型(RFLP)液体クロマトグラフィー/エレクトロスプレーイオン化質量分析(LC / ESI-MS)により毒性フグ種の遺伝子型を決定するための方法の改良が記載されています。 DNA抽出は、シリカ膜ベースのDNA抽出キットを用いて行われます。界面活性剤を含まないPCR緩衝液を用いてPCR増幅した後、制限酵素反応液を精製せずに溶液に添加されます。逆相シリカモノリスカラム及びフーリエ変換は、修正キングドントラップ分析器を有する高分解能質量分析計は、それぞれ、分離および検出のために使用される変換します。 400mMの1,1,1,3,3,3-ヘキサフルオロ-2-プロパノール、15mMのトリエチルアミン(pHは7.9)及びメタノールからなる移動相は、0.4 ml /分の流速で送達されます。 LC / ESI-MS分析のためのサイクル時間は、カラムの平衡化を含む、8分です。同位体分布モデルOを有するデコンボリューションソフトウェアオリゴヌクレオチドfの質量スペクトルから、対応するモノアイソトピック質量を計算するために使用されます。オリゴヌクレオチド(範囲26から79ヌクレオチド)の分析のために、質量精度は0.62±0.74 ppm以下(N = 280)であり、優れた精度と精度は、ロック質量標準を使用せずに180時間持続しました。
概要
質量分析(MS)は、イオン1,2に使用される核酸は、マトリックス支援レーザー脱離/イオン化(MALDI)およびエレクトロスプレーイオン化(ESI)の迅速かつ信頼性の高い同定のための受け入れられた技術です。 MALDI技術は、典型的には、飛行時間型(TOF)分析器と組み合わされます。しかし、MALDI-TOF MSのアプリケーションは短いオリゴヌクレオチドに限定されている(〜25ヌクレオチド(nt))その後の断片化、付加体形成と低イオン化効率1,2による。対照的に、ESIは、より長いオリゴヌクレオチド(> 100塩基)に適用可能であるが、複数の多くの電荷状態イオン荷電([M-NH] N-)生体高分子から同時に生成され、従って、分析物の質量は、上部を超えること分光器のm / z範囲の限界。これは、複雑なスペクトルの解釈を必要とする、 すなわち 、対応するマスに電荷状態系列の形質転換デコンボリューションを経由。
小分子の質量測定、モノアイソトピック質量のピーク、 すなわちの場合、各要素の唯一の最も一般的な同位体を含む分子の質量が最も豊富であり、3自然観察しました。分子量が増加するにつれて、より高いのm / z及びモノアイソトピックピークの同位体分布のシフトは、ベースラインノイズ3-5によって不明瞭になります。モノアイソトピックピークは10 kDaの3より大きい大衆のためにはもはや検出すると、平均分子量が測定ではなく、モノアイソトピック質量5のために使用されていません。このような場合、個々のピークのそれぞれ1ダによって分離された同位体分布のみを観察することができる場合TOF、フーリエキングドントラップ分析器6を修飾変換、またはフーリエ変換イオンサイクロトロン共鳴のような高分解能質量分析計分析装置は、分析のために使用されます。しかし、最も豊富なピークのありますometimes平均分子量5と一致していません。これらの問題は、正確な分析物を決定するための困難につながる可能性があります。
安定同位体の天然存在度の変動と平均分子量を決定する際の難しさを考えると、モノアイソトピック質量の測定は、生体分子3,4の質量特性評価に最適です。実際には、モノアイソトピックピークが観察できるかどうか、モノアイソトピック質量は、モデル検体4,7-10から計算理論的に観察された同位体分布パターンを比較することによって推定することができます。フィッティングアルゴリズム8は現在、独自のソフトウェアに組み込まれています。
ESI-MSの文脈において、DNA二重鎖、精製および脱塩の解離によるイオン抑制及び付加物形成2,10-14にイオン化の失敗を回避するために、直接測定するために必要とされます。これらの手順はtroublありますesomeは、しかしながら、完全に自動化された分析システムは、病原体15~20を検出するためのポリメラーゼ連鎖反応(PCR)、試料調製及びESI-MS含む商業的に開発されてきました。別のアプローチは、分離用液体クロマトグラフィー(LC)を導入されています。 LCが共存する物質からの検体のオンライン分離を提供し、21,22をイオン化する前に面倒な試料調製を必要としません。しかし、MS適合性移動相を用いた核酸の分離は、そのようなヌクレオチドのポリアニオン性質により、薬物、ペプチドおよびタンパク質などのほとんどの他の化合物に比べてより困難です。 LC / ESI-MSの最も成功した例は、イオン対逆相LCの使用を含みます。 1,1,1,3,3,3-ヘキサフルオロ-2-プロパノール(HFIP) -トリエチルアミン(TEA) -メタノールの移動相は、最初Apffel らによって提案された。短いオリゴヌクレオチド23を分離して検出します。 differentiatioのためのLC / ESI-MSジェノタイピングの応用病原体種、一塩基多型および短いタンデムリピートのNは、より長いオリゴヌクレオチド1,21,24-33を分離することができるキャピラリーポリマーモノリスカラムを使用して報告されています。
日本とアメリカでは、深刻な食中毒は誤認やフグの不適切な準備が原因で発生しているが、これはフグの分布と準備が厳密に食品の安全性の法律34,35によって制御されているにもかかわらずです。また、フグエキスを使用して、意図的な殺人は日本36で発生しました。したがって、フグ種の分化は、公衆衛生とフォレンジック調査の観点の両方から必要とされます。 トラフグはフグの他の種類よりもはるかに高価であるためさらに、分化能力はまた、食品詐欺の調査との関連で必要とされます。
ここで、Mを決定するための詳細な方法逆相シリカモノリスカラムおよび高分解能質量分析計を用いてLC / ESI-MSによるPCR産物のonoisotopic質量が記載されています。具体的には、アプローチは、MSを使用して、動物の種分化の最初の例であるPCR制限酵素断片長多型(RFLP)法37の使用に基づく毒性フグ種の分化を可能にするために開発されました。
プロトコル
1. DNA抽出
注:このようなゲル電気泳動およびLC / ESI-MSなどのDNA抽出とPCR後の検査のための別の部屋を使用してください。緩衝液1(含む塩酸グアニジン)、およびDNA抽出キットプロトコルに従って洗浄バッファー2(塩酸グアニジンを含まない)を洗浄するためにエタノールを加えます。魚試料は、日本の魚の卸売業者や小売市場から得ました。
- 0.5ミリリットルまたは1.5ミリリットルマイクロチューブに魚の組織の30から50ミリグラムを置きます。 (フィンと皮膚を除く)マイクロスパチュラを使用して組織をつぶします。
注:フィンの場合には、浸漬0.5 mlチューブを使用しています。 - 溶解バッファーと簡単にプロテイナーゼKと渦の40μlの180μlのを追加します。 2時間または一晩のためにブロックヒータで56℃でインキュベートします。簡単にインキュベーションの間15分毎にボルテックスして混合します。
注:完全な溶解は必要ありません。 - 13,000×gで5分間遠心分離します。遠心分離後、小AMO場合油のUNTは肝臓などの脂肪サンプルの場合の表面上に存在する、それを捨てます。新しい1.5 mlのマイクロ遠心チューブに上清を移します。
- グアニジン塩酸塩を含むカオトロピック緩衝液200μlを加え、ボルテックスで混和します。再びボルテックスすることにより、エタノール(99.5%)とミックスの200μlのを追加します。
- 2 mlのコレクションチューブに入れスピンカラムに混合物を転送します。 13,000×gで1分間遠心操作します。フロースルーを捨てます。
注意:グアニジンは、漂白剤との反応性が高い化合物を形成することができるので、無駄に漂白剤を追加しないでください。 - 13,000×gで1分間洗浄バッファー1(含む塩酸グアニジン)と遠心機の500μLを加えます。フロースルーとコレクションチューブを捨てます。
- キットで提供される新しい2 mlのコレクションチューブにスピンカラムを配置します。 2万×gで3分間洗浄バッファー2と遠心機の500μLを加えます。フロースルーとコレクションチューブを捨てます。新しいにスピンカラムを移します1.5ミリリットルマイクロチューブ。
- ピペットでスピンカラム・メンブレンの中央に溶出緩衝液200μl。 1分後、6,000×gで1分間遠心操作します。
- ピペット50使い捨てUVキュベットに溶出液μlの220から300 nmでの溶出液のUVスペクトルおよび分光光度計を用いて260 nmの吸光度を測定します。
- 260 nmにピークを有するDNAのUVスペクトルを確認します。簡単な方程式「DNA濃度(ngの/μL)は260 nmの×50の吸光度を=」を使用して、おおよそのDNA濃度を計算します。
注:フグのフィンから抽出された典型的なDNA濃度は63±30 ngの/μL(N = 20)です。 - DNA濃度を10ng /μl以上である場合は、5 ngの/μLまでのTris-EDTA(TE)緩衝液(pH8)でDNAサンプルを希釈します。
- -20℃でサンプルを保存したり、PCR増幅(セクション2)に進みます。
2. PCR
- 各EXTRのために25μlのPCRを設定しますDNA試料、陽性対照の汚染を回避するために、クリーンベンチの表1および2に従って氷上および陰性対照(代わりにDNA試料の超純水)(TE緩衝液pH8.0中の標的配列を有するオリゴヌクレオチドを合成した0.2μM)の作用。
注:簡単な操作のために、テンプレートDNAなしで全ての試薬を含むカクテルの十分な量を作り、それを分配します。 PCR産物の3 'オーバーハングの付加を回避するために、DNAポリメラーゼを有する校正活性を使用します。 - 表3に示したサイクルプログラムを使用してサーマルサイクラーでPCR増幅を行います。
3.酵素消化
- PCR溶液に100mMのトリス-HCl(pH7.5)、100mMのMgCl 2を、10mMのジチオスレイトール、および500mMのNaClを含む溶液2μlを添加します。
- 各制限酵素(DRA IおよびMSP I)の1μl加え、穏やかに混合します。目に37℃で30分間インキュベートermalサイクラー。
- MSP I.によって生成された粘着末端を埋め、残りのDNAポリメラーゼを活性化するために72℃で5分間インキュベート
- オプション:15(w / vの%)のクロスリンク比3.33の(%C、ビスアクリルアミドの割合)(重量%)と、ポリアクリルアミドゲル濃度(%のT)を用いてポリアクリルアミドゲル電気泳動によって反応液の各々2.5μlの分析38。蛍光DNA染色液を用いてゲルを染色し、琥珀色の画面を使用して、青色光トランスイルミに二本鎖DNAを観察します。
- 分析カラムを損傷することになる、目詰まりを防止するために0.2ミクロン遠心濾過装置を用いて反応溶液を濾過します。
- 分析まで-20℃でテーパー状ポリプロピレンバイアルとストアにろ液を転送します。
4. LC / ESI-MS分析
- 1 LボトルにTEAのHFIPの67.2グラム( 約 42ミリリットル)および1.52グラム( 約 2.0ミリリットル)を計量します。超純水(LC / MS GRAの955ミリリットルを追加します。ド)および試薬が完全に溶解するまでマグネチックスターラーを用いて攪拌します。溶液のアリコートを取り、pHをチェックし(7.85と7.95の間を、理想的でpH = 7.9)pHメーターを使用して。
注意:金属イオンによる汚染を避けるために、ボトルにアリコートを返さないでください。 pHが7.9でない場合は、pHを調整し、再度pH値を測定するためにHFIPまたはTEAのいずれかを少量加えます。 - 蒸発を防ぐために(家庭用)直接冷却ポータブル冷蔵庫を使用して、ボトルの冷却を液体クロマトグラフの線にボトルを接続します。 B.行目にメタノールで満たされた別の瓶を接続
- 液体クロマトグラフにガードカラムと分析カラム(2.0×50ミリメートル、= 30nmのメソ細孔サイズ)を接続します。カラムを平衡化するために、初期移動相(95%Aおよび5%B)を流れ始めます。
- 較正39として0.5 mg / mlとナトリウムトリフルオロ酢酸(pHは3.5)を準備します。
- トリフルオロ交流を50mg( 約 34μl)を計量ビーカー内のid。
- 超純水30mlを添加し、pHメーターを用いて10mMの水酸化ナトリウムでpH3.5に滴定します。
- 超純水で50mlに埋めます。アセトニトリル(LC / MSグレード)の50ミリリットルを加え、混ぜます。
- ワーキング溶液の一部を取り、室温で保管してください。 4°Cで溶液の残りの部分を格納します。
- 次のようにC-トラップの窒素圧力を減らします。
注:最近のフーリエ変換は、無傷のタンパク質分析に適している質量分析計を、変換圧力を制御するための制御ソフトウェアを有します。- 質量分析計の左側上部カバーを取り外します。 C-トラップ弁は、左前の角にあります。
- 制御ソフトウェアの機器ステータスウィンドウに、高真空圧力値を確認してください。
注:値は、一般的に、3×10 -8バーです。 - ロックを解除し、高真空プレスを減らすために非常にゆっくりとノブを反時計回りに回すためにノブを引いて1/3(典型的には、1×10 -8バール)にURE。示された圧力が遅れるようにして徐々に変更する必要があります。低圧に関する警告メッセージは無視してください。ロックするには、ノブを押してください。
- 安全のためにトップカバーを復元します。
- トリフルオロ酢酸ナトリウム39を使用して、質量分析計を調整します。
注:毎日の質量較正は、このプロトコルで置き換えることができる、しかし、製造元が推奨する推奨キャリブレーションは、このキャリブレーションの前に完了している必要があります。- 次のようにスキャンパラメータおよびチューニングソフトウェアの機器コントロールボックス内の加熱されたESI源のパラメータを設定します。スキャンタイプフルMSを。スキャン範囲m / zの 500-4,000;断片化(インソース衝突誘起解離(CID)電圧60 eVで、極性が負、シースガス流量5;補助ガス流量0、スイープガス、自動利得制御(AGC)は、1×10 6、最大注入時間50ミリ秒を標的率0を流れ、電圧3 kVのスプレー;キャピラリー温度320°C; S-レンズ高周波(RF)レベル100;ヒーター温度30℃です。
- マイナスイオンの入力リストは、m / zの 792.85908、1064.80870、1336.75832、1608.70794、1880.65755、2152.60717、2424.55679、2696.50640、2968.45602、3376.38045として監視します。
- 質量分析計(位置B)のインターフェースに近い加熱ESIプローブを移動します。プローブとチューブとシリンジを接続します。
- 埋め込まれたシリンジポンプを用いて10μL/分の流速で常に較正を注入。カスタマイズされたキャリブレーションテーブル内の唯一の6低質量イオン( のm / z 792.85908から2152.60717)を確認してください。
- 信号の強度が安定した後、キャリブレーションを続行します。キャリブレーション後、わずか6より高い質量のイオン( のm / z 1880.65755から3376.38045)をチェックし、キャリブレーションを続行します。失敗を避けるために、一度に全てのイオンを使用して校正することは避けてください。
- 次のようにスキャンパラメータおよびチューニングソフトウェアの機器コントロールボックス内の加熱されたESI源のパラメータを設定します。スキャンタイプフルMSを。スキャン範囲m / zの 500-4,000;断片化(インソース衝突誘起解離(CID)電圧60 eVで、極性が負、シースガス流量5;補助ガス流量0、スイープガス、自動利得制御(AGC)は、1×10 6、最大注入時間50ミリ秒を標的率0を流れ、電圧3 kVのスプレー;キャピラリー温度320°C; S-レンズ高周波(RF)レベル100;ヒーター温度30℃です。
- appropriaにプローブを移動します0.4ミリリットル/分(位置D)との流量のためのテ位置が不活性金属管を液体クロマトグラフに接続します。
- 次のようにチューニングソフトウェアの機器コントロールボックス内の加熱されたESI源のパラメータを設定します。シースガス流量50を、補助ガス流量15。ガス流量1のスイープ。電圧を2.5 kVのスプレー。キャピラリー温度350°C; S-レンズRFレベル100;ヒーター温度350℃です。
- 楽器の設定画面で、質量分析計の設定取得パラメータを次のようにメソッド時間8分。バルブ、質量分析計に3.5-6分、0から3.5と無駄に6-8分をそらします。ランタイム3.5〜6分。負極性;ソースCID電圧15.0 eVの中で、マイクロスキャン3。 14万( のm / z 200で)公称分解能; AGCの目標1×10 6;最大イオン時間50ミリ秒。スキャン1の数。スキャン範囲m / zの 700-3,500;スペクトルデータ型プロファイル。
- 液体クロマトグラフのパラメータを設定し、次のように:カラムオーブン温度20°C; samplEトレイ温度10℃。勾配プログラム0-0.5分5%B、0.5分で5〜30%のB、1-3.5分30〜40%のB、3.5-5分40〜98%のB、5~6分98%のB、6- 6.05分98-5%のB、6.05から8分で5%B。レート0.4ミリリットル/分を流します。
- パージは、バイアルの底部をタップすることで泡。サンプルラック上のサンプルバイアルを設定し、分析を開始するオートサンプラーを用いて、サンプルの1.0μLを注入します。
- ブラウザソフトウェアで、生データファイルを開き、すべての買収が正常に正および負の対照を含め、完了していることを確認します。
注:フグのDNAが正常に増幅し、消化された場合、通常、2個または3個のピークが4〜5.5分の保持時間で総イオン電流クロマトグラムにおいて観察されるであろう。
5.デコンボリューションと解釈
- デコンボリューションソフトウェアを起動します。実験型」自動抽出物(同位体解決)」を選択します。
- 次のパラメータを用いる方法を編集します。出力質量Mを、 S / NのthresholD 3、相対的な量のしきい値0;負電荷はい。 m / z範囲700-3,500;はい、現在のクロマトグラム(XIC)イオン抽出計算し、電荷キャリアのH +。検出された電荷3の最小数。同位体比のヌクレオチド;適合率80%。剰余しきい値0%。はい重複を考慮して、範囲3-50を充電します。最小強度1。予想される強度誤差3。
- 新しいメソッドとして保存し、それを選択します。
- 生データファイルが存在するディレクトリを選択します。分析すべき生データファイルを選択し、ボタン「キューに追加」をクリックします。
- デコンボリューションを開始するには[実行キュー]タブで[実行]ボタンをクリックします。
- キューが完了した後、行を選択し、デコンボリューションの結果を得るために上部のキャプションに「オープン結果」をクリックします。
- 表4を使用して、4から5.5分の保持時間にピーク由来モノアイソトピック質量の結果を解釈します。
結果
三商業的に入手可能なカラムは、100〜400μL/ minの流速で長いオリゴヌクレオチドの分離を評価しました。ワイドポアオクタデシル炭素鎖(C 18) -結合微粒子シリカカラム(a)は、市販のポリ(スチレン-ジビニルベンゼン)(PS-DVB)モノリスカラム(b)及びC 18 -結合シリカモノリスカラム( C)( 図1)と比較しました。 DNA二重鎖の三つの対(26、37および53塩基対)は、すべての列を使用して分離し、C 18 -結合シリカモノリスカラムは、その後の研究に使用しました。
T.の筋肉から抽出したDNA試料の分析のための代表的な結果rubripesは、 図2に示されている。同位体分離されたピークを、 図2aに示すように、正常モノアイソトピック質量を計算するために必要とされます。理論とMEASTのuredモノアイソトピック質量rubripesは、 図2bに示されています。エンドヌクレアーゼで消化し、精製することなく、単にPCR増幅の後に行われるので、MSP Iエンドヌクレアーゼによって生成された3 '付着末端は、残りのDNAポリメラーゼを用いて充填されます。合成DNAテンプレート由来のアンプリコンの分析によると、質量精度は-2.48から2.40 ppmの(平均0.62±0.74 ppmであり、n = 280)の範囲でした。したがって、3 ppmでの質量公差は、種( 表4)を区別するために使用しました。テーブルを使用して、市場や店から得たフグサンプルのすべてが適切に特定の種37として分化させました。
T.の合成DNAテンプレートから得られた試料の定期的な分析によりますメクラアブ属 、検体のすべてのモノアイソトピック質量は、正常でルのために±3ppmの内で決定しました。任意の質量キャリブレーションなしAST 180時間( 図3)。これは、質量校正が週単位で必要とされるであろうことを意味しています。
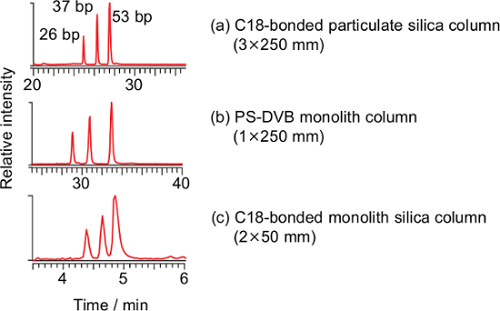
図1:Tの合成されたDNAテンプレートから派生したPCR-RFLP産物の総イオン電流クロマトグラム C 18 -結合微粒子シリカカラム(3 x 250 mmであり、粒径3μm)を、ポリ(スチレン-ジビニルベンゼン)(PS-DVB)モノリスカラム(B、1.0×250mm)で、およびC 18を 使用して pardalis -結合モノリスシリカカラム(C、本発明の方法)(a) のための条件 :流速0.2ミリリットル/分;メタノールの比が、0-2分5%、2-5分5%-25%、5〜20分間、25%-40%、20〜35分で40%-98%、35〜50分で98%、50〜 51分98%-5%、51-65分5%。条件(B):流速0.1 ml /分。メタノールの比が、0-2分5%、2-5分5%-25%、5〜35分間、25%-40%、35〜40分で40%-98%、40〜55分で98%、55- 56分98%-5%、56から70分5%。両方の列のための温度は20℃であった。 この図の拡大版をご覧になるにはこちらをクリックしてください。
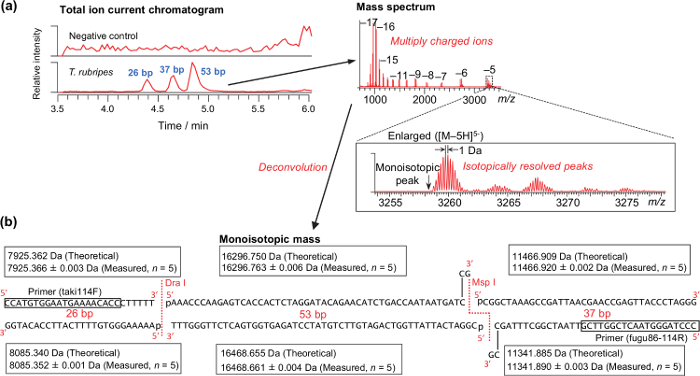
図2:T の生の筋肉を分析するための代表的な結果 rubripes。(a)は、典型的な総イオン電流クロマトグラムとマススペクトル。 (b)は Tの合成されたDNAテンプレートから派生したPCR-RFLP製品の理論と測定されたモノアイソトピック質量rubripes。 大きなを表示するには、こちらをクリックしてください。この図のRバージョン。
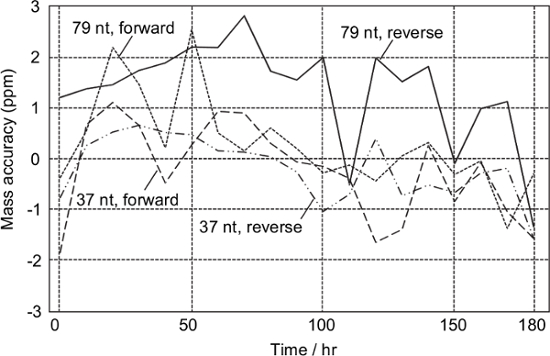
図 3:Tの合成DNAから誘導された安定性試験消化されたアンプリコンメクラアブ属は、すべて10時間を分析しました。質量較正は、テスト中に実行されませんでした。 この図の拡大版をご覧になるにはこちらをクリックしてください。
| 名 | シーケンス |
| lago86F | 5'-CCATGTGGAATGAAAACACC-3' |
| taki114F | 5'-AAAAACAAGAGCCACAGCTCTAA-3' |
| fugu86-114R | 5'-CCCTAGGGTAACTCGGTTCG-3' |
表1:PCRプライマー。
| PCR試薬 | ボリューム使用 | 最終濃度 |
| 超純水 | 15.75μlの | |
| 界面活性剤を含まない5Xバッファ | 5.0μlの | 1倍 |
| dNTPミックス(各10mM) | 0.5μlの | 0.2 mMの |
| プライマーミックス(TE緩衝液pH8.0中、10μMlago86F、taki114Fとfugu86-114Rのそれぞれを) | 1.0μlの | 0.4μMの各 |
| DNAポリメラーゼ(2.5単位/μl)を | 0.25μlの | 0.1単位/μlの |
| TE緩衝液pH8.0中の鋳型DNA(5.0から10 ngの/、抽出されたサンプルのためμlで陽性対照として合成DNAのための0.2μM) | 2.5μlの | 0.5から1.0のnグラム/抽出されたDNAおよび合成DNAのための20 nMのためのμlで |
| 合計:25μlの |
表2:25μlの規模PCRのコンポーネント。
| サイクル | 調子 | 関数 |
| 1 | 95℃で2分 | 最初の変性 |
| 2 | 95℃で30秒 | 変性 |
| 3 | 56℃で30秒 | アニーリング |
| 4 | 72℃で30秒 | 伸長 |
| 5 | 2-4(合計30サイクル)を繰り返します | |
| 6 | 72℃で7分 | 最後の伸長 |
| 7 | サンプルの除去まで12℃で保持します |
表3:PCRプログラム。
| 4.0〜5.5分の保持時間でピーク数 | 範囲内の第3のピークのモノアイソトピック質量(ダ) | 範囲内の第二のピークのモノアイソトピック質量(ダ) | フグ種 | ||
| 小さいストランド | より大きなストランド | 小さいストランド | より大きなストランド | ||
| 2 | → | → | 15890.707から15890.803 | 16177.531から16177.629 | L. inermis |
| 15921.702から15921.797 | 16146.537から16146.634 | L. gloveri | |||
| 15930.713から15930.809 | 16137.525から16137.622 | L. lunaris | |||
| 15937.697から15937.792 | 16131.537から16131.634 | L. wheeleri | |||
| 24173.034から24173.179 | 24566.905から24567.053 | T.のメクラアブ属 | |||
| 3 | 16295.706から16295.804 | 16467.610から16467.709 | 11341.851から11341.919 | 11466.875から11466.944 | T. pardalis T. snyderi T. ocellatus T. xanthopterus T. stictonotus |
| 11654.908から11654.978 | 11770.920から11770.991 | T. niphobles | |||
| 16296.701から16296.799 | 16468.605から16468.704 | → | → | T. rubripes | |
| 16311.701から16311.799 | 16452.610から16452.709 | → | → | T. poecilonotus T. exascurus | |
| 16320.713から16320.811 | 16443.599から16443.697 | → | → | T. porphyreus T. obscurus | |
| 16599.751から16599.851 | 16780.667から16780.767 | → | → | T. vermicularisが | |
表4:LC / ESI-MS由来モノアイソトピック質量からフグの分化。

表5:増幅されたDNA配列 (a)の配列が同一です。表4に記載されているように、他のフグ種のこと。 この表の拡大版をご覧になるにはこちらをクリックしてください。
ディスカッション
DNAを抽出するために、血液及び組織からDNAを抽出するための市販のキットには、軽微な修飾(プロテイナーゼKの量と溶解液の遠心分離)を用いて、製造業者のプロトコルに従って使用されています。しかし、任意の抽出キットであれば、細胞DNAをPCRのために適切な回収および純度で抽出できるように使用することができます。この方法は、筋肉、フィン、肝臓、卵巣、および皮膚37を使用してテストされています。フィンが原因、凍結、乾燥沸騰させ、焼きフグサンプルが正常に37試験したプロテイナーゼK新鮮な、と迅速な溶解を可能にする大きな表面積、特に適しています。
PCRプライマーセット( 表1)フグのために設計され、フグのミトコンドリア16SリボソームRNA遺伝子を増幅します。増幅する標的DNAは、114から115塩基対(属トラフグ )および86塩基対(属サバフグ属 )( 表5)です。メソッドのdevelopmeを容易にするために、NT、標的配列を有する合成されたオリゴヌクレオチドの代わりに本物のフグ標本を収集する基準のために使用しました。プライマーセットは、目的に応じて設定され、別のプライマーで置き換えることができ、しかし、酵素消化後の最終的なDNAの長さは、成功したデコンボリューションのために必要な質量スペクトルの品質を維持する観点から、NT 100未満であるべきです。加えて、標的オリゴヌクレオチドは、より長い、約75塩基以下である場合にCトラップ中の窒素圧力を減少させるべきであると質量スペクトルの品質が成功デコンボリューションのためには不十分です。そのような制限は、プロトコルセクションに記載したように他の方法でシャーシ内Cトラップするためのバルブを調整する必要があり、機器のソフトウェアにおける最近の進展を介して制御することができます。試料調製用としては、界面活性剤を含まない試薬の使用は、適切なピーク形状、十分なピーク強度と安定した保持時間31の点で、その後のLCのために重要です。
PS-DVBキャピラリモノリスカラム21,24-33、C 18逆相微粒子シリカカラム23,40,41および疎水性相互作用クロマトグラフィー(HILIC)カラム42は、LCのために使用されています/オリゴヌクレオチドのESI-MS分析。これらの中でも、キャピラリモノリスカラムは、分離能の点で他のものよりも優れているが、過去の研究で使用されるキャピラリーモノリスカラムは、社内製機器を必要とする低流量(2μL/分)で操作しましたマイクロLCに専念。簡単な操作を容易にするために、商業的に入手可能なカラムは、より高い流量(100-400μL/分)での長いオリゴヌクレオチドを分離するために評価しました。 DNA二重鎖(26、37および53塩基対)の3組は、上記カラムを用いて分離し、但し、C 18 -結合シリカ微粒子カラムおよびPS-DVBモノリスカラムのサイクル時間は、それぞれ、65及び70分でした、Cのそれに対し、 18 -結合シリカモノリスカラムは、8分( 図1)でした。考慮に迅速な分析を取って、C 18 -結合シリカモノリスカラムは、限られた分離能力にもかかわらず、我々の目的のために選択しました。しかしながら、改善された分離が必要とされる場合、残りの二つの列を使用してもよいです。理論的には、モノリスカラムの場合、そこには間隙容量はなく、したがって、効率的な分離27を有効に 、一貫性のある経路長を維持しながら、移動相は固相の孔を通って流れるように強制されることになります。このようなプロセスは、大きな分子の遅い拡散として、そのようなオリゴヌクレオチドとして具体的に生体高分子の分析で明らかにされるであろう。モノリスカラムの実用的な利点の一つは、背圧は粒子状シリカカラム27のそれよりも低いことです。高流速(400μL/分)と低いカラム温度(20°C)の最大背圧にもかかわらずシステムは、12.5 MPaで37でした。これは、長いオリゴヌクレオチドの迅速な分析のためのC 18 -結合シリカモノリスカラムの利点の最初の実証です。高い流速のため、界面でマイクロLCと正確な位置合わせのための専用装置が必要とされません。代わりに、加熱されたESIプローブは、DNA二本鎖を解離し、後述するように、DNAのイオン化を支援するために必要とされます。
イオン対クロマトグラフィーは、一般的に、オリゴヌクレオチドのMS適合性の分離のために使用されます。しかし、イオン対試薬は、一般に、ESIプロセスと干渉し、ESI-MSの感度を低下させます。従って、HFIPは、しばしば、オリゴヌクレオチドの感度を向上させるために、移動相のために使用されます。しかし、HFIP(沸点59°C)をメタノール(沸点65℃)の前に界面で急速に蒸発し、従って、溶媒のこの損失は、pHを増加させ、イオン対試薬の解離( すなわち 、TEA)を促進しますオリゴヌクレオチドから。本発明の方法は、350℃の高温の窒素ガスで溶出液をnebulizes加熱ESIプローブを採用しているので、この効果は、上で強調することができます。なぜなら微量金属イオン33との付加物形成の減少のジェノタイピング解析のために、代わりにHFIP-TEA緩衝液の、エルブとOberacherはcyclohexyldimethylammonium酢酸(pHは8.4 CycHDMAA)をお勧めします。著者らは、CycHDMAA自体が金属付加物の形成を抑制することが推定されます。文献にもかかわらず、かなりの付加物の形成は、本発明の方法において観察されませんでした。また、HFIP-TEA-メタノール系の注目すべき利点は、HFIP-TEA-メタノール系で得られたピーク面積は、Tの86塩基対のアンプリコンを分析する際CycHDMAA -アセトニトリル系から得られたものよりも17倍であったということですpoecilonotus(データは示さず)。 HFIP-TEA-メタノール系の一つの欠点は、しかし、CycHDMAA - アセトニトリル系を基準にしてコストの増加です。
モノアイソトピック質量の計算は、多価イオンの同位体ピークの分離が必要です。したがって、解像度は今回の解析のために重要です。必要な分解能は、検体の塩基対の長さに依存するが、数万の分解能を有する従来のTOF分析器は、短いオリゴヌクレオチドの分析のために制限され得ます。
表4に示す理論的モノアイソトピック質量は、分析物の対応する塩基組成から計算しました。あるいは、マディマンらは、正確な質量43の塩基組成を計算するソフトウェアアプリケーションを開発しました。同様のプログラムは、自動化されたESI-MSシステム16-18に統合されました。測定されたモノアイソトピック質量は常にユニークな塩基組成のOに対応していないため、これらのアルゴリズムの使用は、本方法の堅牢性を向上させることができます必然的な測定誤差に起因する3 ppm以下の質量公差翼。残念ながら、我々は本研究のためにこれらのソフトウェア製品を入手することができませんでした。
本発明の方法は、塩基組成の分析に基づいており、具体的にフグ用に設計された手順を必要としないので、本モノアイソトピック質量ベースの種決意はフグの分化のためだけでなく、他のDNA多型の検出のためだけでなく、適切であり得ます。 DNA多型の検出のためのように、専用のESI-MSシステムは、病原体15,17,18,20,30の検出などの診断用途に適していることができる、完全に自動化され、操作が容易です。逆に、本発明の方法は、したがって、研究用途に適した一般的な研究機器および装置で可能であると。 ESI-MSは、既に一塩基多型13,28,32、短いタンデム反復26ヒトDNA多型に適用されています、およびミトコンドリアDNAは16,19を analsis。マイクロRNAはまた、キャピラリーLC / ESI-MS 44を介して分析しました。これらの公開された出願はまた、本発明の方法によって実現することができます。また、この方法は、オリゴヌクレオチドおよびそのような抗生物質及び低温分離によりリボソームRNA 45との相互作用のような低分子量化合物との間の相互作用をモニターするために適切であり得ます。このような場合には、オリゴヌクレオチドおよび低分子量化合物を同時に検出する必要があり、LC / ESI-MSを使用することの利点です。
インスツルメンテーションは、サンガー配列決定およびリアルタイムPCRのような従来の技術のように平行分析を行うのに適していないという制限があります。さらに、本方法は、ベース組成物と同じ分子内の任意の塩基置換を区別することができない特定します。しかしながら、ここで説明MSベースのDNA分析は、まだ項のメリットを有していてもよいです例えば、ゲル電気泳動およびリアルタイムPCRのようなDNA結合色素ベースの技術と比較して精度のS。
開示事項
The authors have nothing to disclose.
謝辞
This work was supported by a Grant-in-Aid for Scientific Research by the Japanese Society for the Promotion of Science (15K08060).
資料
| Name | Company | Catalog Number | Comments |
| DNeasy Blood & Tissue Kit (50) | Qiagen | 69504 | DNA extraction kit |
| Proteinase K | Qiagen | 19131 | |
| Ethanol (99.5+ vol%) | Wako | 054-07225 | For dilution of wash buffers |
| TE buffer (pH 8.0) | Wako | 310-90023 | For dilution of DNA sample |
| PCR primer | Fasmac | NA | Purified with reverse-phase cartridge column by the supplier |
| Template DNA | Eurofins Genomics | NA | Purified by HPLC by the supplier |
| Ultrapurified water | NA | NA | Generated with a Milli-Q Direct water purification system (Merck Millipore), used for sample preparation |
| Detergent Free 5x Phusion HF Buffer | Thermo Fisher | F-520L | Use instead of the provided buffer of DNA polymerase |
| Pfu-X DNA polymerase | Jena Bioscience | PCR-207S | |
| dNTP mix (20 mM each) | Jena Bioscience | NA | Supplied with the DNA polymerase |
| 10× Universal buffer M | Takara Bio | NA | Containing 100 mM Tris-HCl (pH 7.5), 100 mM MgCl2, 10 mM dithiothreitol and 500 mM NaCl |
| Dra I | Thermo Fisher | FD0224 | Restriction enzyme |
| Msp I | Thermo Fisher | FD0544 | Restriction enzyme |
| 1,1,1,3,3,3-hexafluoro-2-propanol (HFIP) | Fluka | 42060-50ML | Eluent additive for LC-MS grade. |
| Triethylamine (TEA) | Fluka | 65897-50ML | Eluent additive for LC-MS grade. |
| Trifluoroacetic acid | Sigma-Aldrich | T6508 | For preparation of calibrant. |
| Sodium hydroxide (1.0 M) | Fluka | 72082 | Dilute to 10 mM with ultrapurified water and use for titration of trifluoroacetic acid. |
| Acetonitrile | Fluka | 34967 | For preparation of calibrant, LC/MS grade. |
| Methanol | Kanto | 25185-76 | For mobile phase, LC/MS grade. |
| Water | Thermo Fisher | W6-1 | For mobile phase, LC/MS grade. |
| Microcentrifuge tube (0.5 ml) | Eppendorf | 0030123301 | PCR clean grade |
| Microcentrifuge tube (1.5 ml) | Eppendorf | 0030123328 | PCR clean grade |
| UVette | Eppendorf | 0030106300 | Disposable UV cuvette. |
| Gel Green | Biotium | 41004 | Fluorescent DNA stain |
| Cosmospin filter G (0.2 μm) | Nakalai Tesque | 06549-44 | Made of hydrophilic polytetrafluoroethylene (PTFE) membrane. Any centrifugal filter unit (pore size, 0.2–0.5 μm) made of hydrophilic PTFE or another low-binding membrane is applicable. |
| 300 μl PP screw vial | American Chromatography Supplies | V0309P-1232 | |
| Preassembled screw cap and septa | Finneran | 5395-09R | |
| Monobis C18 analytical column (2.0×50 mm, mesopore size = 30 nm) | Kyoto Monotech | 2050H30ODS | Outside Japan, available for purchase from GL Sciences via its distributors. (http://www.glsciences.com/distributors/) |
| Monobis C18 guard column | Kyoto Monotech | GCSET-ODS3210 | Holder included. |
| Cadenza CW-C18 (3.0×250 mm) | Imtakt | CW036 | C18-bonded particulate silica column |
| ProSwift RP-4H (1.0×250 mm) | Thermo Fisher | 066640 | Poly(styrene-divinylbenzene) monolith column |
| Themo Mixer C | Eppendorf | 5382 000.023 | For digestion of fish tissues. |
| Spectrophotometer | Shimadzu | UV-3150 | Quantification of DNA concentration. |
| Thermal cycler | Bio-Rad | T100 | |
| Portable refrigerator | Twinbird | D-CUBE | For aqueous mobile phase to avoid evaporation of HFIP. This can be replaced by an ice box. |
| Ultimate 3000 liquid chromatograph | Thermo Fisher | NA | |
| Q Exactive mass spectrometer | Thermo Fisher | NA | Fourier transform mass spectrometer equipped with the modified Kingdon trap analyzer. |
| Protein deconvolution 3.0 | Thermo Fisher | NA | Use version 3.0 or higher having an isotopic patter model of nucleotide. |
参考文献
- Oberacher, H. On the use of different mass spectrometric techniques for characterization of sequence variability in genomic DNA. Anal. Bioanal. Chem. 391, 135-149 (2008).
- Banoub, J. H., Miller-Banoub, J., Jahouh, F., Joly, N., Martin, P., Banoub, J. H., Limbach, P. A. Chapter 1, Overview of recent developments in the mass spectrometry of nucleic acid and constituents. Mass spectrometry of nucleosides and nucleic acids. 1, 1-90 (2010).
- Yergey, J., Heller, D., Hansen, G., Cotter, R. J., Fenselau, C. Isotopic distributions in mass spectra of large molecules. Anal. Chem. 55, 353-356 (1983).
- Zubarev, R. A., Demirev, P. A. Isotope depletion of large biomolecules: Implications for molecular mass measurements. J. Am. Soc. Mass Spectrom. 9, 149-156 (1998).
- Null, A. P., Muddiman, D. C. Determination of a correction to improve mass measurement accuracy of isotopically unresolved polymerase chain reaction amplicons by electrospray ionization Fourier transform ion cyclotron resonance mass spectrometry. Rapid Commun. Mass Spectrom. 17, 1714-1722 (2003).
- Hu, Q., Noll, R. J., Li, H., Makarov, A., Hardman, M., Cooks, R. G. The Orbitrap: a new mass spectrometer. J Mass Spectrom. 40, 430-443 (2005).
- Senko, M. W., Beu, S. C., McLaffertycor, F. W. Determination of monoisotopic masses and ion populations for large biomolecules from resolved isotopic distributions. J. Am. Soc. Mass Spectrom. 6, 229-233 (1995).
- Horn, D. M., Zubarev, R. A., McLafferty, F. W. Automated reduction and interpretation of high resolution electrospray mass spectra of large molecules. J. Am. Soc. Mass Spectrom. 11, 320-332 (2000).
- Frahm, J. L., Mason, C. J., Muddiman, D. C. Utility of accurate monoisotopic mass measurements to confidently identify lambda exonuclease generated single-stranded amplicons containing 7-deaza analogs by electrospray ionization FT-ICR mass spectrometry. Int. J. Mass Spectrom. 234, 79-87 (2004).
- Frahm, J. L., Muddiman, D. C. Nucleic Acid analysis by fourier transform ion cyclotron resonance mass spectrometry at the beginning of the twenty-first century. Curr. Pharm. Des. 11, 2593-2613 (2005).
- Null, A. P., George, L. T., Muddiman, D. C. Evaluation of sample preparation techniques for mass measurements of PCR products using ESI-FT-ICR mass spectrometry. J. Am. Soc. Mass Spectrom. 13, 338-344 (2002).
- Null, A. P., Benson, L. M., Muddiman, D. C. Enzymatic strategies for the characterization of nucleic acids by electrospray ionization mass spectrometry. Rapid Commun. Mass Spectrom. 17, 2699-2706 (2003).
- Manduzio, H., Ezan, E., Fenaille, F. Evaluation of the LTQ-Orbitrap mass spectrometer for the analysis of polymerase chain reaction products. Rapid Commun. Mass Spectrom. 24, 3501-3509 (2010).
- Manduzio, H., Martelet, A., Ezan, E., Fenaille, F. Comparison of approaches for purifying and desalting polymerase chain reaction products prior to electrospray ionization mass spectrometry. Anal. Biochem. 398, 272-274 (2010).
- Hofstadler, S. A., et al. TIGER: the universal biosensor. Int. J. Mass Spectrom. 242, 23-41 (2005).
- Eduardoff, M., et al. Mass spectrometric base composition profiling: Implications for forensic mtDNA databasing. Forensic Sci. Int. Genet. 7, 587-592 (2013).
- Tang, Y. W., et al. Clinical accuracy of a PLEX-ID flu device for simultaneous detection and identification of influenza viruses A and B. J. Clin. Microbiol. 51, 40-45 (2013).
- Legoff, J., et al. Broad-range PCR-electrospray ionization mass spectrometry for detection and typing of adenovirus and other opportunistic viruses in stem cell transplant patients. J. Clin. Microbiol. 51, 4186-4192 (2013).
- Kiesler, K. M., Coble, M. D., Hall, T. A., Vallone, P. M. Comparison of base composition analysis and Sanger sequencing of mitochondrial DNA for four U.S. population groups. Forensic Sci. Int. Genet. 8, 226-232 (2014).
- Bacconi, A., et al. Improved sensitivity for molecular detection of bacterial and Candida infections in blood. J. Clin. Microbiol. 52, 3164-3174 (2014).
- Huber, C. G., Oberacher, H. Analysis of nucleic acids by on-line liquid chromatography-mass spectrometry. Mass Spectrom. Rev. 20, 310-343 (2001).
- Pourshahian, S., McCarthy, S. M., Bonilla, J. V., Srivatsa, G. S. Chapter 4, Analysis of oligonucleotides by liquid chromatography and mass spectrometry. Handbook of Analysis of Oligonucleotides and Related Products. , 137-166 (2011).
- Apffel, A., Chakel, J. A., Fischer, S., Lichtenwalter, K., Hancock, W. S. Analysis of Oligonucleotides by HPLC-Electrospray Ionization Mass Spectrometry. Anal. Chem. 69, 1320-1325 (1997).
- Premstaller, A., Oberacher, H., Huber, C. G. High-performance liquid chromatography-electrospray ionization mass spectrometry of single- and double-stranded nucleic acids using monolithic capillary columns. Anal. Chem. 72, 4386-4393 (2000).
- Oberacher, H., Oefner, P. J., Parson, W., Huber, C. G. On-Line Liquid Chromatography Mass Spectrometry: A Useful Tool for the Detection of DNA Sequence Variation. Angew. Chem. Int. Ed. Engl. 40, 3828-3830 (2001).
- Oberacher, H., Parson, W., Muhlmann, R., Huber, C. G. Analysis of polymerase chain reaction products by on-line liquid chromatography-mass spectrometry for genotyping of polymorphic short tandem repeat loci. Anal. Chem. 73, 5109-5115 (2001).
- Oberacher, H., Huber, C. G. Capillary monoliths for the analysis of nucleic acids by high-performance liquid chromatography-electrospray ionization mass spectrometry. Trends Anal. Chem. 21, 166-174 (2002).
- Oberacher, H., et al. Re-sequencing of multiple single nucleotide polymorphisms by liquid chromatography-electrospray ionization mass spectrometry. Nucleic Acids Res. 30, e67 (2002).
- Oberacher, H., Parson, W., Holzl, G., Oefner, P. J., Huber, C. G. Optimized suppression of adducts in polymerase chain reaction products for semi-quantitative SNP genotyping by liquid chromatography-mass spectrometry. J. Am. Soc. Mass Spectrom. 15, 1897-1906 (2004).
- Mayr, B. M., et al. Identification of bacteria by polymerase chain reaction followed by liquid chromatography-mass spectrometry. Anal. Chem. 77, 4563-4570 (2005).
- Oberacher, H., Niederstatter, H., Casetta, B., Parson, W. Some guidelines for the analysis of genomic DNA by PCR-LC-ESI-MS. J. Am. Soc. Mass Spectrom. 17, 124-129 (2006).
- Beer, B., et al. CYP2D6 genotyping by liquid chromatography-electrospray ionization mass spectrometry. Anal. Bioanal. Chem. 400, 2361-2370 (2011).
- Erb, R., Oberacher, H. Comparison of mobile-phase systems commonly applied in liquid chromatography-mass spectrometry of nucleic acids. Electrophoresis. 35, 1226-1235 (2014).
- Cohen, N. J., et al. Public health response to puffer fish (Tetrodotoxin) poisoning from mislabeled product. J. Food Prot. 72, 810-817 (2009).
- Cole, J. B., et al. Tetrodotoxin poisoning outbreak from imported dried puffer fish--Minneapolis, Minnesota 2014. MMWR Morb. Mortal. Wkly. Rep. 63, 1222-1225 (2015).
- Ohno, Y., et al. The influence of tetrodotoxin on the toxic effects of aconitine in vivo. Tohoku J. Exp. Med. 167, 155-158 (1992).
- Miyaguchi, H., Yamamuro, T., Ohta, H., Nakahara, H., Suzuki, S. Genotyping of Toxic Pufferfish Based on Specific PCR-RFLP Products As Determined by Liquid Chromatography/Quadrupole-Orbitrap Hybrid Mass Spectrometry. J. Agric. Food Chem. 63, 9363-9371 (2015).
- Sambrook, J. F., Russell, D. W. . Neutral polyacrylamide gel electrophoresis. in: Molecular Cloning: A Laboratory Manual. 1, 40-46 (2001).
- Moini, M., Jones, B. L., Rogers, R. M., Jiang, L. Sodium trifluoroacetate as a tune/calibration compound for positive- and negative-ion electrospray ionization mass spectrometry in the mass range of 100-4000 Da. J. Am. Soc. Mass Spectrom. 9, 977-980 (1998).
- Fountain, K. J., Gilar, M., Gebler, J. C. Analysis of native and chemically modified oligonucleotides by tandem ion-pair reversed-phase high-performance liquid chromatography/electrospray ionization mass spectrometry. Rapid Commun. Mass Spectrom. 17, 646-653 (2003).
- Chen, B., Bartlett, M. G. Evaluation of mobile phase composition for enhancing sensitivity of targeted quantification of oligonucleotides using ultra-high performance liquid chromatography and mass spectrometry: application to phosphorothioate deoxyribonucleic acid. J. Chromatogr. A. 1288, 73-81 (2013).
- Gong, L., McCullagh, J. S. Analysis of oligonucleotides by hydrophilic interaction liquid chromatography coupled to negative ion electrospray ionization mass spectrometry. J. Chromatogr. A. 1218, 5480-5486 (2011).
- Muddiman, D. C., Anderson, G. A., Hofstadler, S. A., Smith, R. D. Length and base composition of PCR-amplified nucleic acids using mass measurements from electrospray ionization mass spectrometry. Anal. Chem. 69, 1543-1549 (1997).
- Kullolli, M., Knorf, E., Arampatzidou, M., Tewari, M., Pitteri, S. J. Intact MicroRNA analysis using high resolution mass spectrometry. J. Am. Soc. Mass Spectrom. 25, 80-87 (2013).
- Cummins, L. L., Chen, S., Blyn, L. B., Sannes-Lowery, K. A., Drader, J. J., Griffey, R. H., Hofstadler, S. A. Multitarget affinity/specificity screening of natural products: finding and characterizing high-affinity ligands from complex mixtures by using high-performance mass spectrometry. J. Nat. Prod. 66, 1186-1190 (2003).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved