このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ケモスタットを用いた微生物の適応研究所進化のための手順
要約
ここでは、ケモスタット培養を使用した条件下での微生物の適応実験室進化を取得するためのプロトコルを提示します。また、進化した株のゲノム解析について説明します。
要約
Natural evolution involves genetic diversity such as environmental change and a selection between small populations. Adaptive laboratory evolution (ALE) refers to the experimental situation in which evolution is observed using living organisms under controlled conditions and stressors; organisms are thereby artificially forced to make evolutionary changes. Microorganisms are subject to a variety of stressors in the environment and are capable of regulating certain stress-inducible proteins to increase their chances of survival. Naturally occurring spontaneous mutations bring about changes in a microorganism's genome that affect its chances of survival. Long-term exposure to chemostat culture provokes an accumulation of spontaneous mutations and renders the most adaptable strain dominant. Compared to the colony transfer and serial transfer methods, chemostat culture entails the highest number of cell divisions and, therefore, the highest number of diverse populations. Although chemostat culture for ALE requires more complicated culture devices, it is less labor intensive once the operation begins. Comparative genomic and transcriptome analyses of the adapted strain provide evolutionary clues as to how the stressors contribute to mutations that overcome the stress. The goal of the current paper is to bring about accelerated evolution of microorganisms under controlled laboratory conditions.
概要
微生物が生存し、多様な環境に適応することができます。重度のストレスの下では、適応はランダムなゲノム変異とそれに続く正の選択1-3により有益な表現型の買収を経て発生する可能性があります。したがって、微生物細胞は、「適応進化」と呼ばれる最適な成長のために代謝または調節ネットワークを変更することにより、適合させることができます。このようなスーパーバグや堅牢な微生物株の発生の発生などの最近の重要な微生物の傾向は、非常に密接にストレスの多い条件下での適応進化に関連しています。定義された実験室条件の下で、我々は分子進化のメカニズムを研究、さらには様々なアプリケーションのための微生物の進化の方向を制御することができます。多細胞生物とは異なり、単細胞生物は、次のような理由から、適応実験室進化(ALE)に適しています:彼らは多くの人口を維持するため、すぐに再生成し、それはホーンを作成し、維持するのは簡単ですogeneous環境。 DNA配列決定技術と高スループット技術の最近の進歩と組み合わせることで、ALEは、全身規制の変更につながるゲノム変化を直接観察することができます。変異動態と人口の多様性も観測可能です。遺伝子工学戦略はALE株4,5の分析から決定することができます。
ケモスタット培養は定常状態の細胞を取得し、発酵工程6の生産性を増加させるために使用される方法です。新鮮な培地を加え、培養液は、工程(後者は、媒体とバイオマスを含む)中に回収されます。長期ケモスタット培養は、しかし、文化の定常状態の生産性を変化させ、培養物( 図1a)の間に自然突然変異と選択の蓄積をもたらします。様々な選択圧(ストレス)下で、突然変異の蓄積が促進されます。長期的にストレスの緩やかな増加ケモスタットは、コロニーの固体培地からの転送と、液体媒体からシリアル転送(繰り返しなど 、温度、pH、浸透圧、栄養飢餓、酸化、有毒な最終製品として、与えられたストレス因子に対して機能変異の連続選択を提供しますバッチ培養)は、研究者が進化した微生物( 図1Bおよび1C)を取得することを可能にします。ケモスタット培養は、複雑な方法が必要ですが、多様性のプール(複製と集団サイズの数)は、コロニー転送とシリアル転送技術によって得られるものよりも高くなっています。個々の細胞への安定的なストレス暴露とケモスタット培養物(定常状態)の間、細胞状態の変化を減少させたバッチ培養ベースの技術と比較して、ALEの他の利点です。高コハク酸条件にさらされた大腸菌のストレス誘発ALEは、この記事で紹介されています。
ジル/ ftp_upload / 54446 / 54446fig1.jpg "/>
図1:適応実験室進化の方法 (A)ケモスタット;。 (B)シリアル転送。 (C)コロニー転送。トップ数字はALEのための方法の概念を示し、下部の図は、ALEの間に成長した細胞の数を示している。 この図の拡大版をご覧になるにはこちらをクリックしてください。
プロトコル
1.機器の準備
- 入口ポートと出口ポートを含むケモスタットジャー(150〜250 ml)または三角フラスコ(250ml)に取得します。シリコンチューブ10〜100ミリリットル/ hrの流量を可能にするとポートを接続します。任意に、エアベント、吹出口、および温度制御された水の入口および出口ポートを使用します。
- 攪拌および温度制御を提供するケモスタットジャーに適した装置を得る(または回転振とう培養器を使用します)。
- 新鮮な培地を提供し、文化を収集するために、2つの蠕動ポンプを取得します。
- 媒体排出口と吸気口を含む貯蔵瓶(10〜20 L)を得ます。
- 希釈率(; L / S 13チューブすなわち 、ID 0.8ミリメートル、流量範囲0.06〜36ミリリットル/分)に適したシリコンチューブを入手します。
2.ミディアム調製と滅菌
- 初期ミディアム
- 0.3グラムのグルコース0.08 gでNH 4を溶解Cl、0.05グラムのNaCl、0.75グラムのNa 2 HPO 4・2H 2 O、および0.3グラムのKH 2 PO 4 90 mlの蒸留水ケモスタットジャー中(DW)。
- クランプを使用してチューブと一緒にケモスタットジャーを封印。通気口を密閉しないでください。
- 121℃で15分間オートクレーブ中でケモスタットの瓶を滅菌します。滅菌後、室温でケモスタットjarファイルを格納します。
- 0.02 gでのMgSO 4・7H 2 O 0.01グラムのCaCl 2、及び10mlのDW(溶液A)中の0.1mgのチアミンを溶解します。
- シリンジおよび予め滅菌シリンジフィルター(0.45μmの細孔のフィルター)を使用してフィルタソリューションA。
- Aは、ケモスタットジャーにろ液溶液を加えます。
- ストレスミディアム
- 30gのグルコース8 gでNH 4 Clで5 gでのNaCl 75 G 2のNa HPO 4・を溶解2H 2 O、30gのKH 2 PO 4、300gをコハク酸二ナトリウム六水和物( 硫酸ナトリウム・コハク・6H <サブ> 2 O;貯留瓶で9.9 L DWで、この実験で使用したストレッサー)。
- クランプを使用してチューブと一緒に貯留瓶を密封します。通気口を密閉しないでください。
- 121℃で15分間オートクレーブ内の貯留瓶を滅菌します。滅菌後、室温でjarファイルを格納します。
- 2 gでのMgSO 4・7H 2 O、1gのCaCl 2を、100mlのDW(溶液A)中の10 mgのチアミンを溶解します。
- シリンジおよび予め滅菌シリンジフィルター(0.45μmの孔径フィルタ)のフィルタ溶液A。
- Aが貯留瓶に濾液溶液を加えます。
- 無菌的に貯留瓶に滅菌シリコンチューブを接続し、蠕動ポンプを取り付けます。
- 高ストレスミディアム
- セクション2.2のようにメディアを準備したが、ストレッサーの高い濃度で( すなわち 、コハク酸適応3-5 G / L以上)。
注:このプロトコルは、ストレス番目への適応のためのものですATは、媒体を介して配信することができます。例えば、温度、攪拌、または照明などの物理的ストレス因子の場合には、栽培はそれに応じて設計する必要があります。
- セクション2.2のようにメディアを準備したが、ストレッサーの高い濃度で( すなわち 、コハク酸適応3-5 G / L以上)。
3.初期栽培
- 野生型E.の単一コロニーを接種します初期の培地4mlを含有する15mLの試験管中で大腸菌 。
- 37℃で12時間および220 rpmで振盪インキュベーター中で試験管をインキュベートします。
- 無菌的に転送ケモスタット瓶に前培養液の1ミリリットル。
- 6時間37℃で、通気(空気50ml /分)、撹拌(200rpm)しを提供する、ケモスタットの瓶をインキュベートします。
4.ストレス適応
- 無菌的ケモスタットジャーにポンプからシリコンチューブの端を接続します。
- 出口ポンプ(10ミリリットル/時以上)を起動し、文化を集めます。
注意:文化は、通常、4-8時間の初期培養後、対数期にあるべきです。 - Chの出口管から培養物の光学密度(600 nm)をECK。
- (0.1時間-1の希釈率に対応した10ミリリットル/時)入口ポンプを起動します。
- 24時間毎にチューブコンセントから600nmでの培養物の光学密度を確認してください。
- 96時間(9.6倍の売上高)以上のためのケモスタットを操作します。光学濃度が安定している場合、高負荷培地を含有するリザーバーを交換します。光学濃度が0.2未満である場合には、6時間供給入口ポンプを停止させます。入口ポンプを再起動し、光学濃度が0.2以上であることを確認してください。
- 徐々に高いストレッサー濃度を含有するリザーバーに変更することにより、ストレッサーの濃度を高めます。
- それはマイルストーンに到達するたびに適応文化のサンプルを取る( 例えば 、100グラム/ Lのコハク酸ストレスに適応株)さらなるゲノム解析のために、と店。
- サンプル保存のために、滅菌した80%グリセロールsolutiで培養試料(0.5ミリリットル)を混ぜます(0.5ミリリットル)に、-80℃で保管してください。
注:微生物がALE処理中にストレッサーを分解する能力を取得した場合、発酵ジャーの中ストレッサー濃度は、新鮮な貯水池のものと同じではありません。
ストレスに適応した株の5シングルコロニーの分離
- 同じストレッサーを含む寒天平板培地(1.6%寒天)を準備し、媒体の同一濃度で。
- ケモスタットからの出口文化(0.1 ml)をプレートし、16時間37℃でインキュベートします。
- 滅菌爪楊枝を用いてプレートから単一コロニーを選択し、同じストレッサーを含む15ミリリットル試験管内およびケモスタットと同じ中濃度でそれらを接種し、6時間インキュベートします。
- 転写培地50 mlを含む250 mlの三角フラスコに培養液1 mlです。収穫培養液0.5ミリリットル毎に1時間、および600nmでのODを測定します。適応straiの成長率を比較ストレッサー与えられた野生型株のものとn個。
結果
高コハク酸のストレス適応のため、野生型E.大腸菌 W3110株は、D = 0.1時間-1 270日間( 図2)でケモスタットで培養しました。
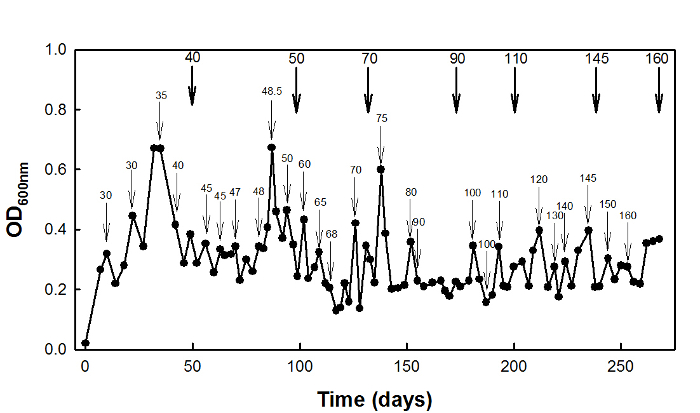
図2:Eの高コハク酸ストレス適応ケモスタット培養を使用して大腸菌 W3110。細い矢印は、ストレッサーの濃度が増加し?...
ディスカッション
微生物は、それらの急速な成長率と遺伝的多様性のほとんどすべての環境に適応することが可能です。適応研究室の進化は、与えられた条件の下で有益な自然突然変異を保有する生物個体を選択する方法を提供している、設計条件の下で進化した微生物を可能にします。
ケモスタット法は、以下の理由により転送技術より人工的に駆動される進化を達成するために、よ?...
開示事項
The authors have nothing to disclose.
謝辞
This study was financially supported by the Korean Ministry of Science, ICT and Future Planning (Intelligent Synthetic Biology Center program 2012M3A6A8054887). P. Kim was supported by a fellowship from the Catholic University of Korea (2015).
資料
| Name | Company | Catalog Number | Comments |
| Mini-chemostat fermentor | Biotron Inc. | - | manufactured by special order |
| silicon tubing | Cole-Parmer | Masterflex L/S 13 | tubing size can be varied depending on the dilution rate and the size of fermentor jar. |
| reservoir jar | Bellco | Media storage bottle | 20 L |
| chemicals | Sigma-Aldrich | - | reagent grade |
| glucose | Sigma-Aldrich | G5767 | ACS reagent |
| NH4Cl | Sigma-Aldrich | A9434 | for molecular biology, suitable for cell culture, ≥99.5% |
| NaCl | Sigma-Aldrich | 746398 | ACS reagent, ≥99% |
| Na2HPO4·2H2O | Sigma-Aldrich | 4272 | 98.5-101% |
| KH2PO4 | Sigma-Aldrich | 795488 | ACS reagent, ≥99% |
| MgSO4·7H2O | Sigma-Aldrich | 230391 | ACS reagent, ≥98% |
| CaCl2 | Sigma-Aldrich | 793639 | ACS reagent, ≥96% |
| thiamine·HCl | Sigma-Aldrich | T4625 | reagent grade, ≥99% |
| Na2·succinate·6H2O | Sigma-Aldrich | S2378 | ReagentPlus, ≥99% |
参考文献
- Rando, O. J., Verstrepen, K. J. Timescales of genetic and epigenetic inheritance. Cell. 128, 655-668 (2007).
- Kim, H. J., et al. Short-term differential adaptation to anaerobic stress via genomic mutations by Escherichia coli strains K-12 and B lacking alcohol dehydrogenase. Front Microbiol. 5, 476 (2014).
- Mendizabal, I., Keller, T. E., Zeng, J., Yi, S. V. Epigenetics and evolution. Integr Comp Biol. 54, 31-42 (2014).
- Lee, J. Y., Seo, J., Kim, E. S., Lee, H. S., Kim, P. Adaptive evolution of Corynebacterium glutamicum resistant to oxidative stress and its global gene expression profiling. Biotechnol Lett. 35, 709-717 (2013).
- Lee, J. Y., et al. Artificial oxidative stress-tolerant Corynebacterium glutamicum. AMB Express. 4, 15 (2014).
- Narang, A. The steady states of microbial growth on mixtures of substitutable substrates in a chemostat. J Theor Biol. 190, 241-261 (1998).
- Kwon, Y. D., Kim, S., Lee, S. Y., Kim, P. Long-term continuous adaptation of Escherichia coli to high succinate stress and transcriptome analysis of the tolerant strain. J Biosci Bioeng. 111, 26-30 (2011).
- Barrick, J. E., Lenski, R. E. Genome dynamics during experimental evolution. Nat Rev Genet. 14, 827-839 (2013).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25, 2078-2079 (2009).
- McKenna, A., et al. The Genome Analysis Toolkit: a MapReduce framework for analyzing next-generation DNA sequencing data. Genome Res. 20, 1297-1303 (2010).
- Deatherage, D. E., Barrick, J. E. Identification of mutations in laboratory-evolved microbes from next-generation sequencing data using breseq. Methods Mol Biol. 1151, 165-188 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved