このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
マイクロスケールの熱泳動を使用してATPのアプタマーの結合部位のマッピング
要約
MicroScale Thermophoresis (MST) is a sensitive technology to characterize aptamer-target interactions. This manuscript describes an MST protocol to characterize aptamer-small molecule interactions.
要約
Characterization of molecular interactions in terms of basic binding parameters such as binding affinity, stoichiometry, and thermodynamics is an essential step in basic and applied science. MicroScale Thermophoresis (MST) is a sensitive biophysical method to obtain this important information. Relying on a physical effect called thermophoresis, which describes the movement of molecules through temperature gradients, this technology allows for the fast and precise determination of binding parameters in solution and allows the free choice of buffer conditions (from buffer to lysates/sera). MST uses the fact that an unbound molecule displays a different thermophoretic movement than a molecule that is in complex with a binding partner. The thermophoretic movement is altered in the moment of molecular interaction due to changes in size, charge, and hydration shell. By comparing the movement profiles of different molecular ratios of the two binding partners, quantitative information such as binding affinity (pM to mM) can be determined. Even challenging interactions between molecules of small sizes, such as aptamers and small compounds, can be studied by MST. Using the well-studied model interaction between the DH25.42 DNA aptamer and ATP, this manuscript provides a protocol to characterize aptamer-small molecule interactions. This study demonstrates that MST is highly sensitive and permits the mapping of the binding site of the 7.9 kDa DNA aptamer to the adenine of ATP.
概要
分子間の相互作用は自然の基礎です。したがって、基礎および応用研究の多くの分野の科学者は、異なる種類の分子相互作用の基本的な原理を理解してみてください。マイクロ熱泳動(MST)は、バッファを自由に選択して、高速、正確な、コスト効率の高い、溶液中の分子間相互作用の品質管理特性評価を行うために、科学者を可能にします。 MSTを使用して、1,000以上の出版物は、ライブラリースクリーニング、結合事象の検証、競合アッセイ、および複数の結合パートナー1-8を用いた実験を含めた分析の種類を記述する、単独で2016年から、すでにあります。一般的に、MSTは、このような分子間相互作用の任意の種類の結合親和性(mmまでPM)、化学量論、および熱力学、のような古典的な結合パラメータ、の研究を可能にします。 MSTの大きな利点は、相互作用パートナーの大きさの独立した結合事象を研究する能力です。でもCHALlengingの小核酸アプタマーとの相互作用(15-30塩基)等の小分子、薬物、抗生物質、または代謝産物などの標的を定量することができます。
アプタマー-ターゲット相互作用を特徴づけるために、現在の最先端の技術は、実験室強く、非常に複雑されているか、またはアプタマー-小分子相互作用9,10を定量化することができません。表面プラズモン共鳴(SPR)はゲル状、19をプロービングにライン、アッセイ11,12と、本当にそのような等温滴定熱量測定(ITC)13-15、定組成溶離16、平衡ろ過17,18としてラベルフリー熱量測定アプローチを、ベースシフトアッセイ、stopped-流FL uorescence分光法20,21、蛍光異方性(FA)22,23、単一分子FL uorescenceイメージング24,25、およびバイオ層干渉(BLI)は26も不正確またはアプタマー、小分子と非互換ですか、相互作用。その他principaこれらのメソッドのリットルの問題は、低感度、高サンプル消費、固定化、表面の質量輸送の制限、および/またはバッファの制限です。これらの技術の唯一のいくつかは、凝集、吸着効果のための統合制御を提供します。
MSTは、アプタマーおよび小分子27-29、ならびにそのようなタンパク質30-33のような他の標的との間の相互作用を研究するために、この制限を克服するために、科学者のための強力なツールを表します。技術は、温度勾配を介して分子の動きに依存しています。呼ばれるこの有向運動、「熱泳動は、「サイズ、電荷、および分子34,35の水和殻に依存します。分子へのリガンドの結合を直接変更熱泳動移動度が得られ、これらのパラメータのうちの少なくとも1つを変化させます。小さなサイズを有するリガンドは、結合した状態に結合していないからサイズ変更の面で大きな影響がないかもしれませんが、彼らがdrを持つことができます水和殻および/または電荷にamatic効果。結合パートナーとの相互作用後の分子の熱泳動運動の変化は、基本的な結合パラメータ2,7,34,36,37の定量化を可能にします。
図1Aに示されているように、MSTデバイスは、蛍光検出の場合と同じ光学系を用いて、ガラスキャピラリー内のサンプル上に集束赤外線レーザーで構成されています。レーザは温度勾配(2-6℃のΔT)を確立しながら、トリプトファン6または蛍光標識された相互作用パートナー3,8のの固有のFL uorescenceを介してタンパク質の熱泳動的動きを監視することができます。空間、ΔTで生じる温度差はソーレーによって定量することができる高温の領域での枯渇または分子の蓄積につながるCOEF Fiのcient(S T)。
G "/>
C ホット加熱された領域内の濃度を表し、及びcの寒さは、初期の寒冷地での濃度です。
図1B、それぞれの時間スケールによって分離することができる別の相からなるMSTの移動プロファイル(時間トレース)における典型的なMSTの実験結果に示すように初期蛍光を正確出発蛍光を定義し、光退色またはphotoenhancementをチェックするために温度勾配が存在しない場合に最初の5秒で測定されます。温度ジャンプ(T-ジャンプ)において蛍光変化熱泳動移動前の段階を示しています。この蛍光の初期の減少は、量子収率uorophore FLの熱的変化に依存しています。定常状態分布に達するまで、熱泳動相が原因分子の熱泳動的動きのために、蛍光が減少(または増加)に従います。レーザがオフにされた後、図1(b)に示すように、逆TJUMPと蛍光増分子の同時逆拡散を観察することができます。基本的な結合パラメータにアクセスするために、相互作用パートナーの異なるモル比を分析し、比較します。光学可視分子が一定に保たれると非標識リガンドの増加量が供給され、一方、典型的には、16の異なる比率は、一つMST実験で研究されています。 2つの結合パートナーとの相互作用は、熱泳動の変化を誘導し、したがって、正規化されたFL uorescenceで、次のように計算されるF ノルム 、:
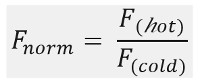
冷たい ホット &F Fは、MSTトレースのデFiのNEDの時点でFL uorescence強度を平均化表します。結合親和性(K dまたはEC 50値)CURVによって計算することができます。電子フィッティング( 図1C)。
全体的に、MSTは、いかなる種類の分子相互作用を研究するための強力なツールです。および25-ntの短い一本鎖DNAアプタマーDH25.42(7.9キロダルトン);この原稿は、小分子アデノシン三リン酸(0.5 kDaのATP)との間で挑戦的な相互作用を特徴づけるためのプロトコルを提供しています。原稿の間に、ATP分子上のアプタマーの結合部位は、ATPのアデニングループにダウンマッピングされます。
プロトコル
アプタマー作業ストックの調製
- 製造元の指示に従って、水にオリゴヌクレオチド(5-Cy5でCCTG GGGGAGTATTGCGGAGGAAGG-3、参照18からの配列)を溶解、100μMの最終濃度に達します。
- 結合緩衝液で200 nmまでのオリゴヌクレオチドストックを希釈することにより、アプタマー作業溶液を準備します(20 mMトリス、pHが7.6; 300mMのNaCl; 5mMのMgCl 2、0.01%のTween20)。
- 90℃で2分間混合物をインキュベート、サンプルはすぐに氷上で冷却し、室温でサンプルを使ってみましょう。
リガンド希釈シリーズの調製
- 各リガンド(アデノシン三リン酸(ATP)、アデノシン二リン酸(ADP)、アデノシン一リン酸(AMP)、アデニン、グアノシン三リン酸(GTP)、シチジン三リン酸(CTP)、デオキシアデノシン三リン酸(dATPを)、及びS-アデノシルメチオニン(SAM)のために; 10 mMストックずつ)、16ステップシリアルdilutiを準備200μlのマイクロ反応チューブ内に。
注:14,000×gで5分間のリガンド株式の遠心分離は、凝集物を除去するのに役立つことがあります。低容量は、低結合反応チューブは、チューブ壁に分子の吸着を避けることをお勧めします。 - 推定親和性より少なくとも50倍の最大濃度で開始し、各希釈段階で50%リガンド濃度を低下させます。
注:制御ソフトウェアに実装濃度ファインダーツールは、結合データをシミュレートし、希釈系列のための右の濃度範囲を見つけるのに役立ちます。 - マイクロ反応管に16から2をアプタマー結合緩衝液10μlを加え、チューブ1のリガンドストック(10 mM)の20μlのを入力します。
- 転送10管2に管1μlのピペッティングにより、数回上下適切に混ぜます。次のチューブに10μLを移し、残りのチューブのために、この希釈を繰り返します。
- 最後のチューブから10μlの過剰を捨てます。任意のbは避けてくださいufferの希釈効果。チューブ1およびチューブ2月16日のバッファは同一でなければなりません。
最終反応ミックスの調製
- ピペッティングの誤差を最小化するために個々の結合20μlの容量との反応(アプタマー使用液+それぞれのリガンド希釈液10μlを10μl)を準備します。わずか4μLの容量は、キャピラリーLL FiにSUFのfi cientです。
- 各リガンド希釈液10μlに200 nmのアプタマーワーキング溶液10μlを加え、ピペッティングにより、数回上下適切に混ぜます。
- 試料中に毛細血管を浸漬することにより、標準的なキャピラリーにサンプルLL室温とFiので5分間サンプルをインキュベートします。より長いインキュベーション時間は、いくつかの相互作用を必要とすることができます。しかし、5分が最も適しています。 NOT光学測定が行われる中央部、上、側面のみに毛細血管をタッチします。
- 目の上に毛細血管を配置電子キャピラリートレイとMSTデバイスを起動します。
4. MSTデバイスの起動
注:生成されたデータを解釈するためのソフトウェアデバイスに2つのプリインストールされたソフトウェア・パッケージを提供し、 '' " '分析制御実験条件の技術的なセットアップ用のソフトウェア'"。
- MSTデバイスに毛細管トレイを配置する前に、制御ソフトウェアを起動し、ドロップダウンメニュー ' "'温度制御で「手動温度制御を可能にする ''を選択することによって、全体的な所望の温度を調整します。このように、25℃に温度を調整します。
注:MST機器は、温度制御された22〜45°Cからすることができます。 - 温度が期待されるレベルに到達した後、MSTデバイスに毛細管トレイを配置するのを待ちます。
- Cy5の色素のための " ''赤にLEDチャネルを設定し、FL uorescencを得るためにLEDの電力を調整します標準センサーとMSTデバイスで300〜1000蛍光単位の電子信号。 25%のパワーLEDをこの研究で使用されています。
注:6000 18000蛍光単位は、高感度センサーとMSTのために推奨されています。
5.キャピラリースキャン
- 「コントロール」のソフトウェア上のキャピラリの位置を選択し、「開始キャップスキャン」をクリックすることで、試料の異なる品質面をチェックするために、毛細管スキャンを行います MSTの測定を開始する前に。
- ソフトウェアで(U字型またはピークを平坦化)蛍光増強/消光とこだわりの効果のためのキャピラリースキャンを点検します。
注:その他の蛍光の検出および取り扱いに関する詳細やこだわりの影響が議論に記載されています。
6. MST測定
注:MSTの測定を開始する前に、こだわりの影響を除外することを確認し、充実/クエンチング効果、またはピペッティングエラー、毛細管スキャンがフロリダ州uorescence信号がSUFのfi cientであることを示していることを確認してください。詳細については、説明を参照してください。
- ''コントロール」ソフトウェアで、それぞれのキャピラリ位置に希釈系列からのリガンドの濃度を割り当てアプタマーとリガンド(1:1)を混合する希釈工程を考えてみましょう。
- キャピラリ#1のリガンドの最高濃度(5 mm)を入力し、正しい希釈タイプを選択します(ここでは、1:1)、最大濃度をクリックすると、自動的に毛細血管の#2位の残りの濃度を割り当てるには、ドラッグ機能を使用16。最低濃度は152.6 nmです。
- 制御ソフトウェアの各セクションで(ここでは、100 nM)を蛍光増アプタマーの濃度を入力します。
- 、5秒間のFL uorescenceを検出し、デフォルト設定を使用して30秒間MSTを記録し、レーザーtの不活性化後にさらに5秒間蛍光を記録します O分子の逆拡散を監視します。
- 制御ソフトウェアの各セクションの20%のレーザー出力を調整します。
注:最高の信号対雑音比を受信し、非特異的効果を回避するために、20〜40%のレーザー出力が推奨されます。特定の場合において、より高いレーザー出力を結合していない及び結合した分子の良好な分離を得るために必要とされ得ます。 - コピー先のフォルダを選択した後、実験を保存して、 ''スタートMST測定」ボタンを押すことで、MSTの計測を開始します。
注:.ntpファイルは、保存先のフォルダに生成されます。この設定を使用して、一回の測定は、10〜15分間持続します。 - EC 50値のより正確な決意のために少なくとも2回の実験手順を繰り返します。
注:技術的再現性を試験するために、同一の毛細管が複数回(技術的反復)を走査することができます。
7. MSTデータ分析
NT ">注:解析ソフトウェアは測定中のFL yの上のデータの分析を可能にする分析ソフトウェアは、リガンド濃度37対MST時間トレースと正規化されたフロリダ州uorescence(F ノルム )の変化をプロットします。。- MST解析ソフトウェア(MO.Affinity分析)を起動し、保存先のフォルダから.ntpファイルをロードします。データ選択メニューでの解析タイプとして「MST」を選択します。
注:リガンド依存性蛍光効果の場合は、初期蛍光分析のために選択することができます。 - ドラッグ・アンド・ドロップするか、それぞれの実験下の「+」ボタンを押して、新たな分析にそれぞれの技術的あるいは生物学ラン(複数可)を追加します。
- 実験では、MSTのトレース、キャピラリースキャン、キャピラリー形状、初期蛍光、および漂白速度の特性に関する情報を得るために、それぞれの実験以下の情報ボタンを押してください。
注:これらの生データ缶アルそう分析の後の手順で検査されます。 - 視覚バンプやスパイクなどの目に見える、凝集および沈殿効果のためのMSTトレースを検査します。
注:検出および凝集効果の取り扱いの詳細については、議論をお読みください。 - 視覚的に平坦化またはU字型のピークとして表示毛細管スキャンと吸着効果のためのキャピラリー形状オーバーレイを、点検してください。視覚毛細管スキャンおよび蛍光効果の初期蛍光を検査します。視覚効果を光退色するための漂白率を検査します。
- 用量 - 応答モードに切り替えて、それぞれのボタンを押して、「エキスパート」モードに解析の設定を変更します。 MSTの評価戦略として「T-ジャンプ」を選択します。
- 曲線Fiのための設定については、「ヒル」モデルを選択します。結合パラメータを自動的に計算されます。 「比較結果」メニュー内の正規化のそれぞれのタイプを選択してデータを正規化します。エクスポート.XLSまたはPDFファイルなどのデータのいずれか。
注:バインディンググラフ下の表には、計算された結合パラメータをまとめたもの。
結果
この研究では、MSTは、ATPのDH25.42 DNAアプタマー18の結合部位を特徴付けるために適用されました。ランダム38-40一つ以上の蛍光体で標識タンパク質と小分子をATPの相互作用を特徴付けるまたはATP模倣他の研究とは対照的に、本研究では、5 '末端上の1つのCy5分子と7.9 kDaの一本鎖DNAアプタマーのラベル付きバージョンが含まれています。異なるATP誘導体...
ディスカッション
品質コントロール:
非特異的表面への試料物質の付着/吸着、ならびに凝集効果、親和性データの質に劇的な影響を与えます。しかし、わずか数最先端の技術は、これらの影響を監視し、回避するために、正確かつ迅速なオプションを提供しています。 MSTは、技術的なセットアップの段階的な最適化を可能にする、検出し、これらの問題を克服するのに役立つ統合された品...
開示事項
CEおよびTSは、生物物理学的分析サービスを提供し2bind社の従業員です。このビデオの記事のための出版手数料は2bind社によって支払われています。
謝辞
著者は何の確認応答がありません。
資料
| Name | Company | Catalog Number | Comments |
| Aptamer binding buffer | 20 mM Tris pH 7.6; 300 mM NaCl; 5 mM MgCl2; 0.01% Tween-20 | ||
| Fluorescently labeled ATP aptamer | IDT, Leuven, Belgium | sequence: DH25.42 50-Cy5-CCTGGGGGAGT- ATTGCGGAGGAAGG-3 | |
| ATP | Sigma Aldrich, Germany | A2383 | 10 mM stock solutions stored at - 20 °C |
| ADP | Sigma Aldrich, Germany | A2754 | 10 mM stock solutions stored at - 20 °C |
| AMP | Sigma Aldrich, Germany | A2252 | 10 mM stock solutions stored at - 20 °C |
| Adenine | Sigma Aldrich, Germany | A8626 | 10 mM stock solutions stored at - 20 °C |
| SAM | Sigma Aldrich, Germany | A7007 | 10 mM stock solutions stored at - 20 °C |
| dATP | Sigma Aldrich, Germany | 11934511001 | 10 mM stock solutions stored at - 20 °C |
| CTP | Sigma Aldrich, Germany | C1506 | 10 mM stock solutions stored at - 20 °C |
| GTP | Sigma Aldrich, Germany | G8877 | 10 mM stock solutions stored at - 20 °C |
| Monolith NT.115 | NanoTemper Technologies, Munich, Germany | MO-G008 | Blue/Red Channel MST device with standard detector, Monolith NT115 pico is MST device with high sensitivity detector |
| Monolith NT.115 capillaries Standard | NanoTemper Technologies, Munich, Germany | MO-K002 | |
| Eppendorf PCR tubes | Eppendorf, Germany | 30124537 | |
| Monolith control software. 2.1.33, pre-installed on the device | NanoTemper Technologies, Munich, Germany | ||
| MO.affinity analysis v2.1.1 | NanoTemper Technologies, Munich, Germany | ||
| Kaleidagraph 4.5.2 | Synergy Software |
参考文献
- Linke, P., et al. An Automated Microscale Thermophoresis Screening Approach for Fragment-Based Lead Discovery. J Biomol Screen. 21 (4), 414-421 (2015).
- Jerabek-Willemsen, M., et al. MicroScale Thermophoresis: Interaction analysis and beyond. Journal of Molecular Structure. 1077, 101-113 (2014).
- Zillner, K., et al. Microscale thermophoresis as a sensitive method to quantify protein: nucleic acid interactions in solution. Methods Mol Biol. 815, 241-252 (2012).
- Zhang, W., Duhr, S., Baaske, P., Laue, E. Microscale thermophoresis for the assessment of nuclear protein-binding affinities. Methods Mol Biol. 1094, 269-276 (2014).
- Wienken, C. J., Baaske, P., Rothbauer, U., Braun, D., Duhr, S. Protein-binding assays in biological liquids using microscale thermophoresis. Nat Commun. 1, 100 (2010).
- Seidel, S. A., et al. Label-free microscale thermophoresis discriminates sites and affinity of protein-ligand binding. Angew Chem Int Ed Engl. 51 (42), 10656-10659 (2012).
- Seidel, S. A., et al. Microscale thermophoresis quantifies biomolecular interactions under previously challenging conditions. Methods. 59 (3), 301-315 (2013).
- Schubert, T., et al. Df31 protein and snoRNAs maintain accessible higher-order structures of chromatin. Mol Cell. 48 (3), 434-444 (2012).
- McKeague, M., Derosa, M. C. Challenges and opportunities for small molecule aptamer development. J Nucleic Acids. 2012, 748913 (2012).
- Ruscito, A., DeRosa, M. C. Small-Molecule Binding Aptamers: Selection Strategies, Characterization, and Applications. Front Chem. 4, 14 (2016).
- Chang, A. L., McKeague, M., Liang, J. C., Smolke, C. D. Kinetic and equilibrium binding characterization of aptamers to small molecules using a label-free, sensitive, and scalable platform. Anal Chem. 86 (7), 3273-3278 (2014).
- Chang, A. L., McKeague, M., Smolke, C. D. Facile characterization of aptamer kinetic and equilibrium binding properties using surface plasmon resonance. Methods Enzymol. 549, 451-466 (2014).
- Jing, M., Bowser, M. T. Methods for measuring aptamer-protein equilibria: a review. Anal Chim Acta. 686 (1-2), 9-18 (2011).
- Sokoloski, J. E., Dombrowski, S. E., Bevilacqua, P. C. Thermodynamics of ligand binding to a heterogeneous RNA population in the malachite green aptamer. Biochemistry. 51 (1), 565-572 (2012).
- Burnouf, D., et al. kinITC: a new method for obtaining joint thermodynamic and kinetic data by isothermal titration calorimetry. J Am Chem Soc. 134 (1), 559-565 (2012).
- Mannironi, C., Scerch, C., Fruscoloni, P., Tocchini-Valentini, G. P. Molecular recognition of amino acids by RNA aptamers: the evolution into an L-tyrosine binder of a dopamine-binding RNA motif. RNA. 6 (4), 520-527 (2000).
- Jenison, R. D., Gill, S. C., Pardi, A., Polisky, B. High-resolution molecular discrimination by RNA. Science. 263 (5152), 1425-1429 (1994).
- Huizenga, D. E., Szostak, J. W. A DNA aptamer that binds adenosine and ATP. Biochemistry. 34 (2), 656-665 (1995).
- Lee, E. R., Baker, J. L., Weinberg, Z., Sudarsan, N., Breaker, R. R. An allosteric self-splicing ribozyme triggered by a bacterial second messenger. Science. 329 (5993), 845-848 (2010).
- Wickiser, J. K., Cheah, M. T., Breaker, R. R., Crothers, D. M. The kinetics of ligand binding by an adenine-sensing riboswitch. Biochemistry. 44 (40), 13404-13414 (2005).
- Jucker, F. M., Phillips, R. M., McCallum, S. A., Pardi, A. Role of a heterogeneous free state in the formation of a specific RNA-theophylline complex. Biochemistry. 42 (9), 2560-2567 (2003).
- Zhao, Q., Lv, Q., Wang, H. Aptamer fluorescence anisotropy sensors for adenosine triphosphate by comprehensive screening tetramethylrhodamine labeled nucleotides. Biosens Bioelectron. 70, 188-193 (2015).
- Zhang, D., et al. A sensitive fluorescence anisotropy method for detection of lead (II) ion by a G-quadruplex-inducible DNA aptamer. Anal Chim Acta. 812, 161-167 (2014).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule imaging of an in vitro-evolved RNA aptamer reveals homogeneous ligand binding kinetics. J Am Chem Soc. 131 (29), 9866-9867 (2009).
- Elenko, M. P., Szostak, J. W., van Oijen, A. M. Single-molecule binding experiments on long time scales. Rev Sci Instrum. 81 (8), 083705 (2010).
- Zichel, R., Chearwae, W., Pandey, G. S., Golding, B., Sauna, Z. E. Aptamers as a sensitive tool to detect subtle modifications in therapeutic proteins. PLoS One. 7 (2), 31948 (2012).
- Baaske, P., Wienken, C. J., Reineck, P., Duhr, S., Braun, D. Optical thermophoresis for quantifying the buffer dependence of aptamer binding. Angew Chem Int Ed Engl. 49 (12), 2238-2241 (2010).
- Entzian, C., Schubert, T. Studying small molecule-aptamer interactions using MicroScale Thermophoresis (MST). Methods. 97, 27-34 (2016).
- Valenzano, S., et al. Screening and Identification of DNA Aptamers to Tyramine Using in Vitro Selection and High-Throughput Sequencing. ACS Comb Sci. 18 (6), 302-313 (2016).
- Jauset Rubio, M., et al. beta-Conglutin dual aptamers binding distinct aptatopes. Anal Bioanal Chem. 408 (3), 875-884 (2016).
- Breitsprecher, D., et al. Aptamer Binding Studies Using MicroScale Thermophoresis. Methods Mol Biol. 1380, 99-111 (2016).
- Stoltenburg, R., Schubert, T., Strehlitz, B. In vitro Selection and Interaction Studies of a DNA Aptamer Targeting Protein A. PLoS One. 10 (7), 0134403 (2015).
- Kinghorn, A. B., et al. Aptamer Affinity Maturation by Resampling and Microarray Selection. Anal Chem. 88 (14), 6981-6985 (2016).
- Duhr, S., Braun, D. Why molecules move along a temperature gradient. Proc Natl Acad Sci U S A. 103 (52), 19678-19682 (2006).
- Braun, D., Libchaber, A. Trapping of DNA by thermophoretic depletion and convection. Phys Rev Lett. 89 (18), 188103 (2002).
- Duhr, S., Arduini, S., Braun, D. Thermophoresis of DNA determined by microfluidic fluorescence. Eur Phys J E Soft Matter. 15 (3), 277-286 (2004).
- Jerabek-Willemsen, M., Wienken, C. J., Braun, D., Baaske, P., Duhr, S. Molecular interaction studies using microscale thermophoresis. Assay Drug Dev Technol. 9 (4), 342-353 (2011).
- He, K., Dragnea, V., Bauer, C. E. Adenylate Charge Regulates Sensor Kinase CheS3 To Control Cyst Formation in Rhodospirillum centenum. MBio. 6 (3), 00546 (2015).
- Brvar, M., et al. Structure-based discovery of substituted 4,5'-bithiazoles as novel DNA gyrase inhibitors. J Med Chem. 55 (14), 6413-6426 (2012).
- Pogorelcnik, B., et al. 4,6-Substituted-1,3,5-triazin-2(1H)-ones as monocyclic catalytic inhibitors of human DNA topoisomerase IIalpha targeting the ATP binding site. Bioorg Med Chem. 23 (15), 4218-4229 (2015).
- Jhaveri, S., Rajendran, M., Ellington, A. D. In vitro selection of signaling aptamers. Nat Biotechnol. 18 (12), 1293-1297 (2000).
- Khavrutskii, L., et al. Protein purification-free method of binding affinity determination by microscale thermophoresis. J Vis Exp. (78), (2013).
- Ramakrishnan, M., et al. Probing cocaine-antibody interactions in buffer and human serum. PLoS One. 7 (7), 40518 (2012).
- Chen, M., et al. Antiviral activity and interaction mechanisms study of novel glucopyranoside derivatives. Bioorg Med Chem Lett. 25 (18), 3840-3844 (2015).
- Wan, C., et al. Insights into the molecular recognition of the granuphilin C2A domain with PI(4,5)P2. Chem Phys Lipids. 186 (4,5), 61-67 (2015).
- Harazi, A., et al. The Interaction of UDP-N-Acetylglucosamine 2-Epimerase/N-Acetylmannosamine Kinase (GNE) and Alpha-Actinin 2 Is Altered in GNE Myopathy M743T Mutant. Mol Neurobiol. , (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved