Method Article
リボソーム DNA に基づいて RNA サンプルの DNA 汚染の評価
要約
ここでは、RNA サンプルでゲノム DNA (gDNA) 汚染をトレースするためのプロトコルを提案する.提案手法では、リボソーム DNA (rDNA) 遺伝子の内部転写スペーサー領域 (ITS) に特異的なプライマーを利用しています。メソッドは、ほとんどの真核生物と原核生物の DNA 汚染の信頼性と機密性の高い検出に適しています。
要約
遺伝子発現変動とトラン スクリプトの存在量の定量化のため広く使用されて 1 つの方法は、逆のトランスクリプション量的なリアルタイム PCR (RT qPCR) です。正確な敏感な信頼性が高く、再現性のある結果を提供します。いくつかの要因は、RT qPCR の特異性、感度に影響を与えます。RNA のサンプルを汚染残留ゲノム DNA (gDNA) は、それらの 1 つです。遺伝子発現の分析で gDNA 汚染による非特異的増幅はトラン スクリプト レベルの豊富なを過大評価して Rt-qpcr 結果に影響を与えることができます。QRT PCR によって gDNA を検出する一般的に、プライマーを用いた遺伝子間領域または興味の遺伝子のイントロンに焼鈍のペアします。残念ながら、イントロン/エクソンのアノテーションがまだ知られていないすべての遺伝子のため脊椎動物、細菌、原生生物、菌類、植物、および無脊椎動物の後生動物種から。
ここで提案するプロトコル リボソーム DNA (rDNA) を使用して、サンプルの rna gDNA 汚染の検出のためのプライマーをベースします。RDNA のユニークな特徴に基づく: その発現の自然、非常に節約されたシーケンス、およびゲノムの高周波。また事例研究としてプライマーの一意のセット リボソーム DNA (rDNA) イネ科の保存領域に基づいて設計されました。これらのプライマー対の普遍性は、メルトのカーブ解析と agarose ゲル電気泳動によってテストされました。方法で、私たちが rDNA のプライマーは、イネ科 gDNA 汚染の試金のため適用することができます、方法、それを他の原核生物と真核生物の種に簡単に使用できます。
概要
転写制御遺伝子セットを面白いか、シグナル伝達ネットワークの探索は、生物学的イベント1に関与する複雑な分子機構を理解に不可欠です。現在、qPCR 分析は最も広く遺伝子発現研究 (ゲノム) DNA または RNA (トランスクリプトーム) のいずれかを対象とするそれぞれメチロームとトランスクリプトーム解析を可能にするためのアプローチを使用します。QPCR 続く逆のトランスクリプション (RT) は、トランスクリプトーム解析2の生物学的研究のさまざまな領域における遺伝子発現レベルを測定するために使用されています。他の伝統的な北の交配などの方法、組織特異的検出原交配、リボヌクレアーゼの保護試金 (RPA), と半 RT-PCR 法、正確性、利便性、スピード、そして広いダイナミック レンジを経由と比較してください。qPCR ベースのアッセイは、非常に顕著な3,4です。メッセンジャー RNA (mRNA)、RNA の開始材料の量と質などの信頼性の定量化のために考慮する必要があるいくつかの重要な要因があります。さらに、非特異的増幅、RT-qPCR の効率性と PCR 効率を5,6を考慮されなければなりません。
GDNA の存在は、固有の問題 RNA の抽出中にため、一部では、DNA および RNA の7のような理化学的性質。GDNA と相補的 DNA (cDNA) mRNA サンプル由来のシーケンス id のため Rt-qpcr 結果の精度に影響を与えるが非特異的増幅は発生できます。残り gDNA の豊富の過大評価につながる遺伝子発現解析8で mRNA のターゲットします。
基本的には、非特異的増幅はほとんどプライマー二量体形成または gDNA、適切なコントロールのサンプルを使用して、どちらの形で得られる非特異的背景増幅から発生します。そのようなサンプルは、テンプレート コントロール (NTC) して、逆転写酵素コントロール (NRT)、それぞれ。研究されているサンプルで gDNA 汚染のレベルが異なる gDNA に対する感度は、遺伝子解析によって大きく異なりますので、成田コントロールがサンプル/アッセイのペアごとに必要です。これは実質的にコストと Rt-qpcr プロファイリング研究労働増加しますが、これらのコントロールが必要な7,9です。
GDNA 汚染を取扱うことの代替方法がありますプライマー組の遺伝子間領域または利息10の遺伝子と大きなイントロンの側面またはエクソン エキソン接合、すなわちにまたがるプライマーの使用のイントロンに焼鈍アニーリングのサイトが存在しない成熟した mRNA シーケンス1,4。ただし、多く脊椎動物、細菌、原生生物、菌類、植物、および無脊椎動物の後生動物種からすべての遺伝子のイントロン/エクソンのアノテーションはまだ知られています。さらに、多くの真核生物的偽遺伝子に由来する複製イベントがあります。さらに、イントロンのプライマー デザイン gDNA の非増幅とは限りません。クロマチンとしてDNaseをゲノム領域のアクセシビリティ異なります、ターゲットの異なる染色体10異なるプライマー対を設計する勧めします。
真核生物のゲノムは、リボソーム形成に必要なリボソームのサブユニット rDNA 遺伝子のコピーを千まで取囲むことができます。これらの rDNA 遺伝子頻繁、単一またはタンデム繰り返し配列11で構成されます。大サブユニット (LSU) と小サブユニット (SSU) を含むコードする Rrna (図 1) は、RNA ポリメラーゼによる転写が私 (RNA ポル私)。結果前 Rrna は、さらに its 1 領と ITS2 2 つの内部転写スペーサー領域を排除することによって処理されます。最終製品として 3 成熟 Rrna、17-18 s rRNA (SSU)、5.8 s、および 25-28 s rRNA (LSU) は、生成された12。rDNA 遺伝子は非常に節約されたシーケンスと多重遺伝子族の代表です。彼らはゲノムの高頻度で発生、潜在的に 1 つ以上の染色体13の存在です。転写スペーサーの劣化や、rRNA の処理は、核小体の高速プロセスです。園児の度が高いため遺伝子コピー数と検出可能な未処理 RNA premolecules の比率は低コピー イントロン配列や, 前駆体と比較して低いです。これらの機能を行う rDNA 遺伝子のほとんどの真核生物と原核生物3gDNA 汚染の信頼性の高い、高感度の検出に適しています。
ここで RNA サンプルで gDNA 汚染を検出するための新規手順を説明します。保存 rdna に基づく普遍的なプライマーのセットは、いくつかのイネ科植物種間で gDNA 試金のため提示されます。DNA をテンプレートとして使用融解曲線分析による提案のプライマーの普遍性と特異性を調べた。我々 のプロトコルのみイネ科のために適当ではないも簡単に他の真核生物と原核生物の種に適応することができます。
プロトコル
注: 任意の組織を使用ことができます。
1. 核酸抽出
- 組織サンプルの 100 mg を 2.0 mL チューブに入れて、2 つの 5 mm ステンレス ビーズを追加、30 25 30 Hz で組織をホモジナイズしてください s (均質化期間および組織の種類によって周波数) の RNA と DNA の両方。
- 製造元の指示に従って総 RNA を分離します。
- 製造元の指示に従って全 DNA を分離します。
- 260 と 280 nm で吸光度を測定することによって、純度と RNA サンプルの量を制御します。
- 260 と 280 nm で吸光度を測定することによって、純度と DNA のサンプルの量を制御します。
: 注意核酸は 260 の波長の光を吸収する nm (A260)、波長 280 の光の吸光度 (A280) は、タンパク質の量を定量化する使用ことができます、サンプルに存在するフェノール類。したがって、A260/A280 nm の比率は、DNA のサンプルから抽出した RNA の純度を評価する使用できます。≥1.8 の範囲の A260/280 値と > 2.0 はそれぞれ DNA および RNA のための「純粋な」であると考えられています。低い A260/280 値は、タンパク質や有機化学物質による汚染を示します可能性があります。 - DNA の品質をテストするには、0.7% の agarose のゲル電気泳動を実行します。ゲルを準備し、1 x TRIS ホウ酸 EDTA バッファーで実行 (TBE: 89 mM トリス、89 mM ホウ酸、2 ミリメートルの EDTA) 100 V で 30 分高品質 gDNA のように、シャープの高分子量の分画法バンド低分子量 (低分子) 分子の範囲でないの塗抹標本。
- チェックは、製造元の指示に従って数量、純度、整合性、グアニジン チオシアン酸 (GTC) agarose ゲル電気泳動やキャピラリー電気泳動チップによる変性条件下に RNA を分離しました。
- 60 ° C に寒天を冷却した後 5 mM GTC を 1 x TBE の標準的な 1% の agarose のゲルに追加して GTC ゲルを準備します。
注: GTC は有毒である、だからヒューム フードの調剤し、適切な個人用保護具を着用します。 - 読み込みバッファーを変化させる RNA の準備: 95% ホルムアミド、10 mM EDTA pH 8.0 0.1% ブロモフェノール ブルー 0.1% キシレン cyanole、臭化エチジウム 10 μ L。
注: ホルムアミド、エチジウム ブロマイドは有毒でありドラフトで分配する必要があります。 - 読み込みバッファー、熱 70 ° C で 5 分間混合物を変化させる RNA の総 RNA の負荷 1 5 μ g、ゲルにロードする前に氷の上に置き、100 V 45 分負荷 DNA または RNA 分子量マーカーの GTC ゲルと一緒に標準として RNA を分離RNA のサンプル。
- エチジウム ブロマイドでゲルを染色し、紫外光下で画像キャプチャ システムを用いてバンドを視覚化します。真核生物、実行条件の変化でそのまま総 RNA が少なくとも 2 つシャープされ、オフ rRNA のバンド (28 秒・ 18 秒) 2:1 の強度比と表示されます。
- 60 ° C に寒天を冷却した後 5 mM GTC を 1 x TBE の標準的な 1% の agarose のゲルに追加して GTC ゲルを準備します。
- DNase 処理による gDNA の痕跡を削除 (DNase私 RNase フリー)。10 μ L の総ボリュームの RNase フリー チューブに追加: 0.1 - 1 μ g の総 RNA、 DNase MgCl2、私は、1 μ L の 10 倍反応バッファーの 1 つのユニット。37 ° C で 30 分間混合物を孵化させなさい50 ミリメートルの EDTA の 1 μ L を追加し、10 分の 65 ° C で培養反応を停止させます。
- 製造元のプロトコルに従って、DNase 無料RNase A を使用して gDNA 抽出物から RNA の痕跡を削除します。RNase の 5 μ L を追加全 DNA を 10 mg/mL,-80 ° C で 1 h. ストア RNA と DNA の抽出のための 37 ° C で、
2. プライマー デザイン gDNA アッセイの rDNA 領域から
メモ: フルレングス rdna には (its 1 領と ITS2) endonucleolytic 亀裂のシリーズで成熟した rRNA 分子が削除されており (図 1) が低下し、2 つの領域が含まれています。
- 興味の種 (http://www.ncbi.nlm.nih.gov) NCBI から rDNA 塩基配列を取得します。検索データベースの最適なキーワードは、「内部転写スペーサー」
- 領域を節約された内部転写スペーサー領域 (ITSs)、SSU、および LSU を見つけるため BLASTn 検索にターゲットのヌクレオチド シーケンスを入力します。
- どちらかが ITSs シーケンスを側面プライマーを選択または成熟した rRNA には存在しない、ITSs シーケンスを増幅します。
- Its 1 領または ITS2 シーケンスを並ぶプライマーの設計: ClustalW による様々 な種から保存された領域を配置します。AlleleID ソフトウェアとイチイ/クロス-種分析後並ぶ領域の特異的なプライマーを設計します。2 つのプライマー組増幅 SSU-5.8 s と 5.8 s-LSU amplicons に設計できますベース its 1 領と ITS2 の並ぶ地域それぞれ。私アンプリコン長さが少なくとも 300 の増加これらの産物は、その地域にまたがるが、ので bp gDNA から amplicons。この増加は、感度を下げます。
- Its 1 領を並べる: 選択 SSU と 5.8 s rRNA シーケンス。イネ科用の選択したプライマー: SSU、SF: CGTAACAAGGTTTCCGTAGGTG、r: GGTTCACGGGATTCTGCAAT。このプライマー (SF: 順方向と r: 逆) SSU の部分的な地域、its 1 領のフルレングスと 5.8 s の部分的領域増幅 rDNA。
- ITS2 の側面: 5.8 s、LSU のシーケンスを選択します。イネ科用の選択したプライマー: f: ATTGCAGAATCCCGTGAACC LSU 一致シーケンスを LR: TGCTTAAAYTCAGCGGGTAGYC。このプライマーは、5.8 s の部分的な地域、フルレングス、ITS2 および LSU (図 1) の部分的な地域を増幅します。
注: プロモーター領域、SSU の非常に節約された地域 ITSs に基づくプライマー デザインの場合 5.8 s、および LSU が識別されます。前方および逆のプライマーの 5.8 s rRNA の開花植物14保存モチーフに基づいて設計されていた。前方および逆プライマーをもとにデザインした SSU と LSU はイネ科、地域をそれぞれ保存されています。表 1にそれぞれの種に対して SSU、LSU のプライマーの発散であります。
- ITSs シーケンスを増幅するプライマー: このプロトコルの設計 its 1 領プライマーはAeluropusに基づくそのシーケンス (NCBI taxid 数: 110873)。プライマーを使用して: 前方: GGTATGGCGTCAAGGAACACT、逆: ATAGCATCGCTGCAAGAGGT。プライマーのペア、インシリコによる産物によると 60 からサイズの範囲を 200 bp。これはまた、qPCR 分析の推奨サイズです。
- Its 1 領または ITS2 シーケンスを並ぶプライマーの設計: ClustalW による様々 な種から保存された領域を配置します。AlleleID ソフトウェアとイチイ/クロス-種分析後並ぶ領域の特異的なプライマーを設計します。2 つのプライマー組増幅 SSU-5.8 s と 5.8 s-LSU amplicons に設計できますベース its 1 領と ITS2 の並ぶ地域それぞれ。私アンプリコン長さが少なくとも 300 の増加これらの産物は、その地域にまたがるが、ので bp gDNA から amplicons。この増加は、感度を下げます。
- これらの推奨事項を考慮しながらプライマーを選ぶ: GC の内容: 40-60%、プライマー長さ: 18-23 基本、PCR 製品長さ: 60 160 bp (特別にそのプライマー)、融点 (Tm): 60 ° C、最終の両方のプライマーの Tm が 5 ° C 以上とプリムに違いはありませんers は彼ら自身を補完するものではありませんまたはプライマーのパートナーします。
- プライマーの特異性とコピーの数を確認します。プライマー ブラスト プログラム (https://www.ncbi.nlm.nih.gov/tools/primer-blast/) によって選択したプライマー シーケンスのインシリコ解析を実行します。
- プライマー ブラスト提出ページを開きます。フォームのプライマーのパラメーター] セクションでは、両方のプライマー シーケンスを入力します。パラメーター セクションをチェックするプライマーのペアの特異性、生物名 (または生物グループ名) を入力し、ゲノム データベースを選択します。これらの設定は、製品長さ、染色体上の位置を含むターゲット シーケンスとプライマー パラメーターについては特異性を与えるし、数をコピーします。
3. rDNA に基づくプライマー DNA テンプレートの検証 qPCR ステップを実行します。
注: qPCR gDNA をテンプレートとして使用を実行することによって設計されたプライマーの機能を検証しなければなりません。いくつか並列反応を行い、ピペッティングのミスの削減、マスター ミックスの準備をお勧めします。マスター ミックスの反応混合物プラス ~ 10% の合計数に相当する数量を準備します。
- PCR 反応管内の DNA のテンプレートを除くすべての反応コンポーネントを混合することによってマスター ミックスを準備します。マスター ミックスを準備する 1 つの反作用から必要に応じて高級: 5 μ L SYBR グリーン (サイバー) ミックス (2 x)、0.3 μ L のプライマー (0.3 μ M 各の前方および逆プライマー)、マスターし、RNase フリー水 10 μ L に最終的な音量を調整します。分析テンプレート gDNA の 1 μ L で約 ≤200 ng を使用します。
注: 解凍、アセンブル、すべての試薬、コンポーネント、および氷の上の反応混合物を保ちます。 - 光の 96 ウェル プレートにマスター ミックスの約数。各ウェルに gDNA の 1 μ L をピペットし、光板フィルム シールでカバーします。スピン、サーマルサイクラーの場所します。
- 次の条件下でリアルタイム サーマルサイクラーの qPCR アッセイを実行: 95 ° C で 10 分間続いて 95 ° C の 40 のサイクル ステップ 15 s と 60 ° C で 1 分 60 ° C の熱処理/拡張子でデータ集録を実行するため。
- 増幅手術後対象 55 ° C ~ 95 ° C から連続蛍光測定による融解曲線解析をすべて PCR の反作用通常、1 つのデータ ポイント各サイクルで収集温度の段階的増加サイクルあたり 0.5 の ° c。
注: 各プライマーのペアのマスター ミックスの少なくとも 2 つの非テンプレート コントロール (NTC) が含まれます。少なくとも 3 つの複製ですべてのアッセイを実行します。 - 融解曲線分析によるプライマーの特異性を確認します。単一しきい値サイクルと減算カーブ フィット法曲線を分析します。
注: 個々 の鋭いピーク時の外観は均一個々 の増幅を示します。プライマー二量体の製品は、低温での個々 のピークとして表示できます。 - Agarose のゲルの電気泳動によって各私アンプリコンのサイズを検証します。
- TBE バッファー 100 mL で 3 g アガロースを混合することによって 3% の agarose のゲルの準備 (TBE: 89 mM トリス、89 mM ホウ酸、2 ミリメートルの EDTA)。
- DNA と読み込みバッファー x 6 の 1-2 μ L と 5-10 μ L の PCR の製品を混ぜます。3% の agarose のゲルの DNA の梯子の横の PCR の製品をロードします。100 V 45 分で TRIS ホウ酸 EDTA バッファー x 1 の電気泳動の分離を実行します。
- エチジウム ブロマイドや他のデュアルインターカレーション剤がゲルを染色し、紫外光下で画像キャプチャ システムを用いてバンドを可視化します。
注: DNA デュアルインターカレーション エージェント (例えば、臭化エチジウム)、発癌性と取り扱い、個別に調剤。ユニークな鋭いバンド (サイズに関しておよびプライマー二量体や人工背景増幅なし) の外観は、私アンプリコンの特異性を確認します。
4. gDNA RNA テンプレートと汚染試金プロシージャ
注: DNase、浄化された RNA による治療後、サンプルは、rDNA 特定のプライマーによってテストされます。ITSs のイントロンのような機能の処理のため増幅に使用するこれらの地域増幅した信号を検出できますない無料の DNA RNA サンプルで。これに基づいて、qPCR で増幅信号が検出された場合は agarose のゲルのバンド観察 (インシリコ解析による推定)、予想サイズこれ gDNA 汚染原因である必要があります。このセクションで実行される手順は、セクション 3 と似ていますが、すべてのサンプルの cDNA は gDNA 代わりにテンプレートとして使用されます。
- RNA テンプレート PCR 反応管内を除くすべての反応コンポーネントを混合することによってマスター ミックスを準備します。1 つの反作用のためのミックス組み合わせをマスター: 5 μ L サイバー マスター ミックス (2 x)、0.3 μ L のプライマー (0.3 μ M 各の前方および逆プライマー ミックス)、および RNase フリー水 10 μ L に最終的な音量を調整します。分析のためには、1 μ L のボリュームの約 500 ng テンプレート RNA を使用します。
- 光の 96 ウェル プレートにマスター ミックスの約数。ピペットの各ウェルに 1 μ L の RNA と光板フィルム シールでそれをカバーします。遠心分離機、サーマルサイクラーの場所。
注:、少なくとも 2 つの NTC コントロールとそれぞれの試金のための 2 つの肯定的な gDNA コントロールが含まれます。3 技術的なレプリケーションのすべてのアッセイを実行します。 - 次の条件下でリアルタイム サーマルサイクラーの qPCR アッセイを実行: 95 ° C で 10 分間続いて 95 ° C の 40 のサイクル ステップ 15 s と 60 ° C で 1 分 60 ° C の熱処理/拡張子でデータ集録を実行するため。
- 増幅手術後対象 55 ° C ~ 95 ° C から連続蛍光測定による融解曲線解析をすべて PCR の反作用通常、1 つのデータ ポイント各サイクルで収集温度の段階的増加サイクルあたり 0.5 の ° c。
- 電気泳動ゲルの 3% の agarose で実行して、すべての PCR の製品を確認します。
注: 任意のバンドや NTC 反応のピークの出現はおそらくバンドまたは RNA サンプルのピークの存在は gDNA 汚染の結果、低温融解曲線では通常見られるプライマー二量体形成に関連します。RDNA のプライマー、まずすべての RNA のサンプルをテストするのにはお勧め、下流用cDNA 合成、遺伝子発現解析などの汚染された dna の使用されます。
5. cDNA の統合のため RT-PCR のステップと qPCR 解析
- DNaseを解凍-RNA および cDNA 合成試薬を室温で扱われます。解凍後、試薬をスピンダウンします。RNA の 1 μ g とオリゴ (dT) 18 の 1 μ L を追加ヌクレアーゼ フリー チューブに入門。RNase フリー水 12 μ L の容量を調整する、優しく、ミックスし、氷の上保存します。
- ダウン 5 分スピン 65 ° C で反応の孵化によってテンプレート RNA の二次構造を溶かし、氷にバイアルをクールします。
- 次のように反応のマスター ミックス (各反作用のため最終巻 20 μ L) を準備: 逆転写酵素 (200 U/μ L) の 1 μ L、4 μ L 反応バッファー (5 x) のRNase阻害剤 (20 U/μ L)、1 μ L、dNTP ミックス (10 mM) の 2 μ L。穏やかに混合し、氷上バイアルをクールします。RNA を含む準備のチューブに 19 μ L を追加します。
- 42 ° C で 60 分の反作用を孵化させなさい、逆転写酵素を終了する 5 分間の 70 の ° C で、孵化させなさい。氷の上 RT 反応を設置し、(3 と 4 のセクションで説明) とルーチン qPCR プロシージャによって遺伝子発現解析に進みます。
結果
葉の組織の RNA サンプルで gDNA 汚染の有無を検証する rDNA のプライマーの使用を提案します。QPCR 分析と gDNA 汚染測定のフローチャートは図 2に示します。提案するプロトコルの 2 つの相補的な戦略は、rDNA に基づくプライマー デザインのため使用された: 1) 特異的プライマーは、ITSs のシーケンスから選ばれた、2) 普遍的なプライマーは、ITSs 並ぶ領域から選ばれました。概念実証のAeluropus littoralisのために特定のプライマーおよびプロトコルで指定されている、イネ科の種に基づく普遍的なプライマーを設計しました。5.8 s 前方および逆のプライマーは、開花植物、コケ植物、藻類や菌類の14のいくつかの注文間の類似性を示す保存された 14 の塩基対 (bp) をモチーフに選ばれました。設計されたプライマーの機能は、表 2に与えられています。普遍性、SSU の 5.8 s、および LSU プライマーは、BLASTn、によってチェックされ、プライマー相同性結果モチーフのロゴとして図 3に表示されます。それぞれの種の発散のプライマーと同様、相同性解析に含まれている種のリストは、表 1.プライマーの特異性は、プライマー爆発でチェックだった。全ゲノム シーケンスが使用可能なある種の rDNA 遺伝子の染色体の位置が推定されました。例えば、イネ、シロイヌナズナrDNA の遺伝子はトウモロコシ2 つの異なる染色体の 3 つの異なった染色体にあります。
融解曲線解析テンプレートとして DNA を用いた its 1 領と ITS2 山腹 amplicons の rDNA のプライマーの qPCR 検証行った。記載されている図 4と図 5、 を含むイネ科種ないプライマー二量体形成と単一の鋭いピークの観察実験的プライマーの特異性が確認されました。コムギ、オオムギ、イネ、アルファルファ、双子葉類、キュウリやタバコ、シロクローバ alexandrinum、ソラマメ、 シロイヌナズナ。電気泳動のサイズ分離による増幅物の更なるテストを示したユニークなバンド。予想通り、バンドはサイズ (図 6Aおよび6B) に様々 な種のサンプルから派生します。興味深いことに、イネ科 3種だけ有用である他のイネ科の種が、また、シロイヌナズナなど他の植物種およびエンドファイト菌viz に特化された普遍的なプライマーを使用します。Piriformospora インド。
設計されたプライマー (its 1 領) の妥当性は、 A. littoralis gDNA をテンプレートとして使用して、qPCR で確認されました。ないプライマー二量体形成と単一のピークが観察されました。A. littoralisのみならずタバコとシロクローバ alexandrinumを除く他のすべての種のために (プライマー) としてA. littoralis its 1 領プライマーが単一の鋭いバンドを生成する意外にも、生成される 2 つのバンド (図 6)。すべての RNA のサンプルでそのまたはその側面のプライマーによって gDNA 汚染分析を行った。GDNA 汚染分析では、増幅のプレートの概略、結果の解釈は、図 7で示されます。
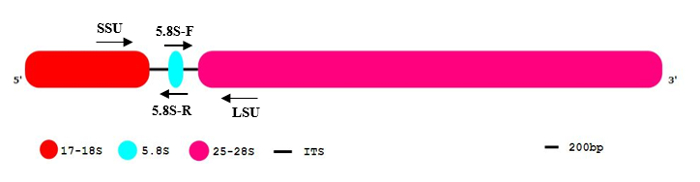
図 1: 真核生物の rDNA シーケンス組織の一般的なパターン。
真核生物の rDNA セグメントを含む 17-18 歳 (赤) (青)、5.8 s、25-28 s rRNA (ピンク)。内部転写スペーサー (ITS) は黒い線として示されます。5´and 3´ は、DNA 分子の方向を示します。この図の拡大版を表示するのにはここをクリックしてください。
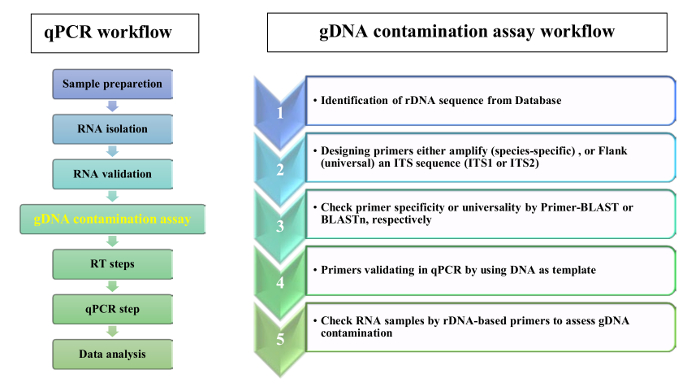
図 2: RT qPCR と gDNA 汚染の試金のためのワークフロー 。この図の拡大版を表示するのにはここをクリックしてください。
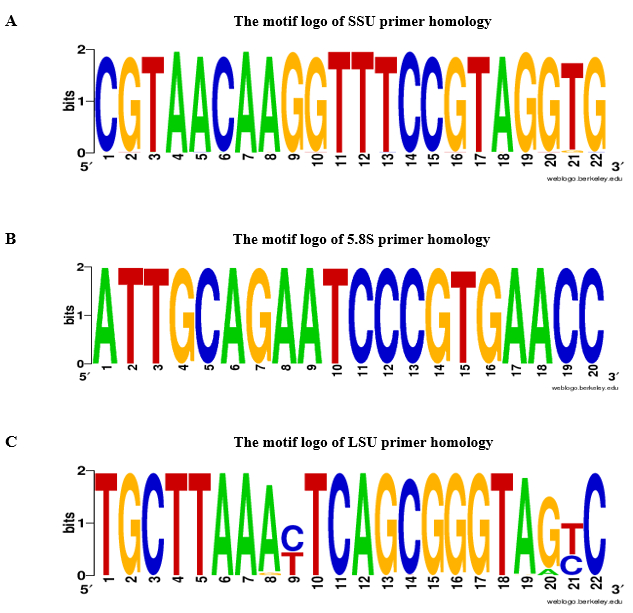
図 3: A. SSU、5.8 s b. と c. LSU プライマー相同のモチーフのロゴ。5.8 s、SSU の LSU プライマーをモチーフにしたロゴが 2,000 の緑の植物の記録に基づく BLASTn によって建設された (NCBI taxid 数: 33090) カットオフ値 e ≤10-10と。A-アデニン T チミン、G はグアニン、C シトシン。この図の拡大版を表示するのにはここをクリックしてください。
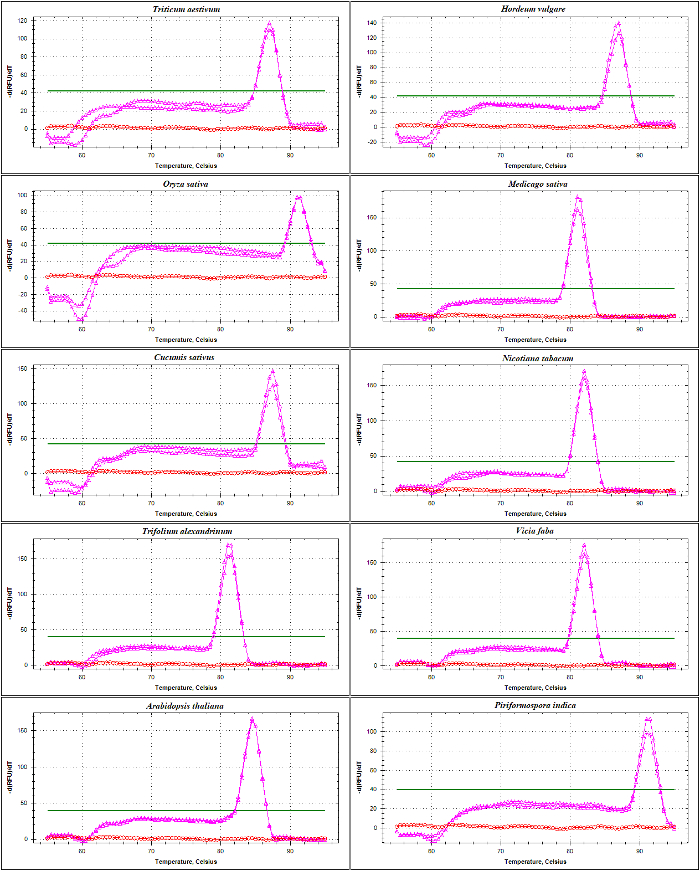
図 4: 種で its 1 領並ぶ私アンプリコンの融解曲線解析。
この増幅増幅 SSU と 5.8 s-R プライマー、17-18 歳未満エンコード地域からシーケンス、its 1 領全体の流れ、5.8 s の部分シーケンスの一部が含まれています。Amplicons 生成 (ピンク)、コムギ、オオムギ、イネ、ウマゴヤシ分子、キュウリ、タバコ、から (赤) NTC の融解曲線が示されています。シロクローバ alexandrinum、ソラマメ、シロイヌナズナ、およびPiriformospora インディカ。フラット太字の行は、ベースラインのしきい値を示します。この図の拡大版を表示するのにはここをクリックしてください。
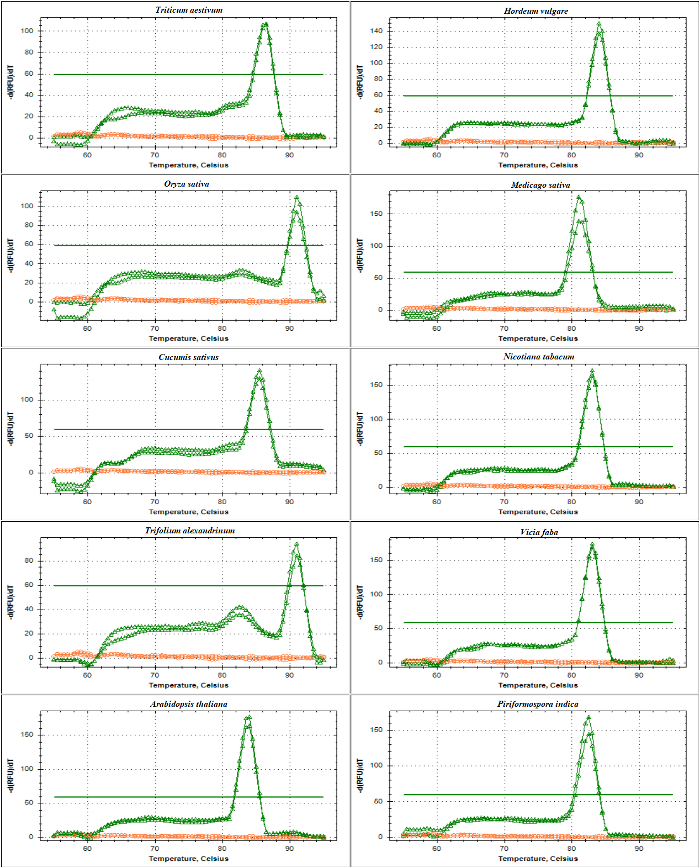
図 5: 種 ITS2 並ぶ私アンプリコンの融解曲線解析。
この私アンプリコン、5.8 s の使用によって生成されます-F、LSU のプライマー。説明私アンプリコンには 5.8 S 部のシーケンス、ITS2、全体の流れ、25、28 秒の部分シーケンスが含まれています。Amplicons (緑)、コムギ、オオムギ、イネ、ウマゴヤシ分子、キュウリ、タバコ、から生成された NTC (赤) の融解曲線が示されています。シロクローバ alexandrinum、ソラマメ、シロイヌナズナ Piriformospora インディカ。この図の拡大版を表示するのにはここをクリックしてください。
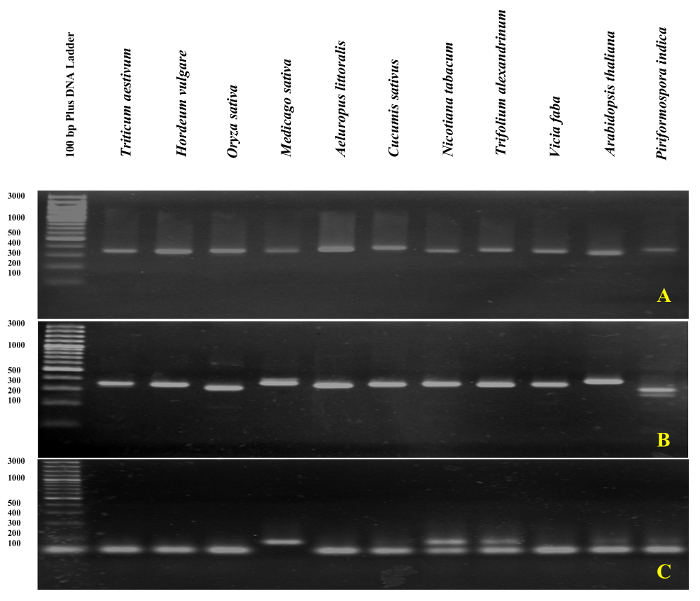
図 6: Agarose のゲルの rDNA に基づく PCR の製品の分析。
(A)ITS2 側面 its 1 領側面の増幅(B)、および its 1 領(C)が 3% の agarose のゲルで実行されました。この図の拡大版を表示するのにはここをクリックしてください。

図 7: ITSs のイントロンのような機能は、gDNA 汚染を検出できるプライマーを設計に見なすことができます。
ピークまたは qPCR 分析で予想されるサイズのバンド gDNA RNA サンプルの汚染を示します。Unk: 不明なサンプル、pos: 肯定的な制御、NTC: 非テンプレート コントロール。この図の拡大版を表示するのにはここをクリックしてください。
| プライマー | 属 | Taxid ID | 種 | 発散プライマー | ||
| SSU | シロイヌナズナ | 3701 | タチスズシロソウ、シロイヌナズナ、 ・ リラータ | - | ||
| ソラマメ | 3904 | ビロサ、アメリカーナ、unijuga、amoenane、発生学、craccamal、擬似 orobus、新称、スギ、ramuliflora 、ソラマメ | ||||
| シロクローバ | 3898 | alexandrinum、montanum、resupinatum 、シロクローバ | - | |||
| タバコ | 4085 | タバコ、栽培、otophora、picilla、bigelovii、palmeri、tomentosiformis、ユスラウメ、digluta、kawakamii、スパティフィラム、nesophila、solanifolia のシダ、実生、arentsii、その分布、wigandioides、undulata、モザイク、noctiflora、petunioides、ハブ、miersii、pauciflora、減衰、尖圭コンジローム、群落、アラタ、ヨーロッパアカマツ、ルスティカとsuaveolens | - | |||
| キュウリ | 3655 | anguria、メロン、ダイコン | CGTAACAAGGTTTCCGTAGGKG | |||
| Aeluropus | 110873 | - | 発見ないプライマー | |||
| ウマゴヤシ | 3877 | イネ、lupulina、pamphylica、ルナタ、口ばしります、皺状舌と分子 | - | |||
| イネ | 4597 | サティバ、glumipatula、・ ルフィポゴン barthii glaberrima 点状、longistaminata、meridionalis、nivara、meridionalis 、 longistaminata | - | |||
| コムギ | 4564 | ほとんど、urartuとハプロイド | - | |||
| オオムギ | 4512 | オカダンゴムシにおける、marinum、brevisubulatum 、 bogdanii | - | |||
| LSU | シロイヌナズナ | 3701 | ・ ペトラエア、シロイヌナズナ、 ・ リラータ | TGCTTAAACTCAGCGGGTAATC | ||
| ソラマメ | 3904 | sylvatica、tetrasperma、サティバ、産、剛毛、における裂、報、orobus、orobus、bithynicaとソラマメ | TGCTTAAATTCAGCGGGTAGCC | |||
| シロクローバ | 3898 | 見せかけ、クロサンショウウオ、resupinatum、ナッツ、subterraneum、strictum、ochroleucon、glomeratum、squamosum、ornithopodioides 、シロクローバ | TGCTTAAATTCAGCGGGTAGCC | |||
| タバコ | 4085 | タバコ、栽培、otophora、picilla、bigelovii、palmeri、tomentosiformis、ユスラウメ、digluta、kawakamii、スパティフィラム、nesophila、solanifolia のシダ、実生、arentsii、その分布、wigandioides、undulata、モザイク、noctiflora、petunioides、ハブ、miersii、pauciflora、減衰、尖圭コンジローム、群落、アラタ、ヨーロッパアカマツとsuaveolens | TGCTTAAACTCAGCGGGTAGTC | |||
| キュウリ | 3655 | メロ、ritchieiおよび品種 | TGCTTAAACTCAGCGGGTAGTC | |||
| Aeluropus | 110873 | lagopoide、pungens 、 littoralis | TGCTTAAATTCAGCGGGTAATC | |||
| ウマゴヤシ | 3877 | ruthenica、サティバ、lupulina、アラビカ、やわらかく、ミニマ | TGCTTAAATTCAGCGGGTAGCC | |||
| pamphylica、ルナタ、口ばしります、皺状舌 | TGCTTAAACTCAGCGGGTAGTC | |||||
| イネ | 4597 | サティバ、glumipatula、・ ルフィポゴン、barthiial、glaberrima、australiensis、アスパラガス、australiensis、ridleyi、malampuzhaensis、アルタ、nivara、・ ルフィポゴン、meridionalis 、 longistaminata | TGCTTAAACTCAGCGGGTAGTC | |||
| コムギ | 4564 | において、spelta、turgidum、dicoccoides、petropavlovskyi、urartu 、ハプロイド | TGCTTAAACTCAGCGGGTAGTC | |||
| オオムギ | 4512 | オカダンゴムシにおける、性、secalinum、brevisubulatum 、 bogdanii | TGCTTAAACTCAGCGGGTAGTC | |||
| 退化のプライマーはヌクレオチドの専門語の IUPAC システムとして定義されています。 | ||||||
表 1: rDNA のプライマーを選ぶ対象種のリスト。
SSU 結合部位にわたって高い配列相同性を示した LSU 結合部位と比較しては、属を与えられました。
| 私アンプリコンの長さ | 増幅領域 | シーケンス | プライマー名 | 私アンプリコン | |
| 332-405 bp | SSU、its 1 領の全体シーケンスと 5.8 s の部分シーケンスの部分シーケンス | CGTAACAAGGTTTCCGTAGGTG | SSU | Its 1 領側面 | |
| GGTTCACGGGATTCTGCAAT | 5.8 S-R | ||||
| 318-361 bp | 5.8 s、全体の流れ ITS2 および LSU の部分シーケンスの部分シーケンス | ATTGCAGAATCCCGTGAACC | 5.8 S ・ F | ITS2 側面 | |
| TGCTTAAAYTCAGCGGGTAGYC | LSU | ||||
| 100-200 bp | ITS 1 領 | GGTATGGCGTCAAGGAACACT | ITS 1 領 F | ITS 1 領 | |
| ATAGCATCGCTGCAAGAGGT | ITS 1 領-R | ||||
表 2: プライマー シーケンスします。
ディスカッション
定量的 PCR による遺伝子発現解析は、近年広く適用されています。この急速なコスト効果の高いと自動化された方法の主な利点は、その精密で正確な結果です。ただし、これらの利点から最適利点を得る qPCR 実験に使用するパラメーターのセットアップの明確な理解が必要です。QPCR 遺伝子発現解析で信頼性の高い結果が表示されます、RNA サンプル3,15プライマー二量体または gDNA 汚染に起因する非特異的増幅は避けるべきです。GDNA 汚染8RNA 転写レベルが過大評価することが期待されます。ここでは、rDNA 遺伝子のユニークな特徴は、RNA サンプルで gDNA 汚染分析のためと考えられました。
このプロトコルで使われる rDNA の基礎的性質:リボソーム遺伝子から成っている 2 つの ITSs、its 1 領と ITS2、および 3 つの rRNA エンコーディング遺伝子、17-18 歳、すなわち、5.8 s、25-28 サブユニット12。2 つの ITS 領域はリボソームのサブユニットのコーディング シーケンスの一部ではないです。RRNA を成熟への前駆物質を処理する少なくとも 3 つの酵素活性によって削除される: ヘリカーゼ、エンドヌクレアーゼ、エキソヌクレアーゼ活性。リボソーム RNA をコードするトラン スクリプトとして転写されると ITSs を含む主な製品は、確かにあります。処理は非常に高速と qPCR 法の検出限界値以下、ITS を含む検出可能な前駆体分子の量は、核小体で行われる。したがって、its 1 領または ITS2 は、プライマーの側面により増幅されるとき、増幅検出できますなし RNA サンプルで gDNA 汚染が存在しない限り。真核生物のゲノムにおける rDNA 遺伝子の数は、千コピーの染色体11のシングルまたはタンデム配列で整理されると推定されました。このプロトコルでは各反応/試金で使用される gDNA 汚染を検出する成田ではなく、別の方法を提案する.
の利点と既存のメソッドに関して制限:成田は通常準備された RNA のサンプルがきれいか gDNA によって汚染されたかどうかをテストする使用されます。RNA の異なるサンプル間 gDNA 汚染は均一に分布しない gDNA を反応感度は遺伝子解析によって著しい影響を受けるので、成田コントロールが各サンプル/アッセイ ペア7,15必要です。これは実質的にコストを追加され、労働の多くを処理するときのサンプル同時に3,9。文献に記載されている他の代替方法には、イントロン、gDNA の検出のための特異的プライマーあるいはイントロンの側面またはエクソン エキソンジャンクションにまたがる設計プライマーの使用が含まれます。これらのメソッドの制限はイントロン配列情報、イントロン/エクソン構造の不完全な注釈および遺伝子のイントロンの不在または金利1,4,10 的偽遺伝子の使用不能から生じます.進化のため、rDNA 遺伝子は発現と非常に節約された遺伝子ファミリーとして存在します。高度のゲノムの豊富なと13の異なる染色体上に存在です。他のコーディングや nonconding 遺伝子と比較して、rDNA 遺伝子表示 gDNA 汚染の検出に最適です。比較トランスクリプトーム解析による rRNA キャリブレータ qPCR データの正規化は cDNA の違いなど、いくつかの問題のため推奨されません (ランダム hexamer プライミングとプライミング ポリア) の準備、rRNA と mRNA の豊富に大きな違い、と誤解を招く可能性があります生成する異なる器官結果10,16。しかし、我々 だけ言及している問題、gDNA 汚染測定のための利点です。たとえば、高いターゲット サイト豊富なゲノムと異なる染色体上の局在について rDNA のプライマーは大幅 gDNA 既存手法と比較して検出感度を向上します。
RDNA に基づく他の生物への versality: rDNA 遺伝子はほとんどの有機体の識別された遺伝子家族。RDNA に基づく手法は、他の真核生物と原核生物 (プロトコル 2 - 5) に簡単に適応させること gDNA 汚染の試金のための単純な非常に敏感な経済システムを表します。事例研究として我々 はここでいくつかの Poceae の種 (図 4および図 5) でこのメソッドの有用性を実証しました。使用されるプライマーは表示への移転その他 Poceae 種の種間の rDNA サブユニットの非常に節約された構造のための率が高いです。ゲノム配列に十分な情報がプライマー デザインのため利用できない場合、この問題がより重要になります。したがって、その側面プライマーの 1 つの種のために設計は、近縁種で使用できます。また、5.8 s ・ F ・ R プライマーは、高い類似性は、ほとんどの顕花植物14保存モチーフに基づいて選ばれました。高スループット シーケンス技術は完全に知られているゲノムの数を増やすがほとんど生物のエキソン ・ イントロン アノテーションが完了していないと、それが頻繁エクソン エクソン国境にまたがるにプライマーを設計することが可能。提案手法を説明どのように rDNA のプライマー分析/プライマーの組み合わせごとに高価な成田コントロールを排除することを目標に原核生物と真核生物の qPCR 分析 gDNA 汚染の試金のため適用することができます。
開示事項
著者は競合する金融興味を持ってないです。
謝辞
この研究は、遺伝子と農業バイオ テクノロジー研究所の Tabarestan (GABIT)、サリ農業科学および天然資源大学 (SANRU) によって支えられました。非生物的ストレス ゲノミクス ジュニア研究グループ資金が供給された IZN (ドイツ、ハレ (ザーレ) 作物植物研究の学際的な中心。原稿の批判的読解ありがとうロンダ マイヤー。
資料
| Name | Company | Catalog Number | Comments |
| Maxima SYBR Green / ROX qPCR Master Mix (2X) | Thermo Scientific | K0221 | |
| TissueLyser II | QIAGEN | 85300 | |
| RevertAid H Minus First Strand cDNA Synthesis Kit | Thermo Scientific | K1631 | |
| GeneRuler 100 bp Plus DNA Ladder | Thermo Scientific | SM0321 | |
| 96 well WHT/CLR | Bio-Rad | HSP9601 | |
| Microseal B film | Bio-Rad | MJ-0558 | |
| Low tube strip CLR | Bio-Rad | TLS0801 | |
| Flat cap strips | Bio-Rad | TCS0803 | |
| NanoDrop 2000 | Peqlab | ND-2000 | |
| RNaseZAP | Ambion | 9780 | |
| Centrifuge | Eppendorf | 5810 R | |
| Agilent RNA 6000 Nano Kit | Agilent Technologies | 5067-1511 | |
| 2100 Electrophoresis Bioanalyzer | Agilent Technologies | G2939AA | |
| RNase A, DNase and Protease-free | Thermo Scientific | EN0531 | |
| DNase I, RNase-free | Thermo Scientific | EN0523 | |
| TRIZOL Reagent | Ambion | 15596026 | |
| CFX96 Touch Real-Time PCR Detection System | BIO RAD | 1855195 | |
| PCR tube, 0.2 mL, RNase-free | Stratagene | Z376426 | |
| Guanidine thiocyanate for molecular biology | Sigma-Aldrich | G9277 | |
| Agarose - Nucleic Acid Electrophoresis | Sigma-Aldrich | A9414 | |
| Boric Acid for molecular biology | AppliChem | A2940 | |
| bromophenol blue | AppliChem | A2331 | |
| ethidium bromide | AppliChem | A1151 | |
| Gel documentation system | BIO RAD | Gel Doc 2000 |
参考文献
- Bustin, S. A., Nolan, T. Pitfalls of quantitative real-time reverse-transcription polymerase chain reaction. J Biomol Tech. 15 (3), 155-166 (2004).
- Gutierrez, L., Mauriat, M., Pelloux, J., Bellini, C., Van Wuytswinkel, O. Towards a systematic validation of references in real-time RT-PCR. Plant Cell. 20 (7), 1734-1735 (2008).
- Hashemi, S. H., Nematzadeh, G., Ahmadian, G., Yamchi, A., Kuhlmann, M. Identification and validation of Aeluropus littoralis reference genes for Quantitative Real-Time PCR Normalization. J Biol Res (Thessalon). 23 (1), 18(2016).
- Bustin, S. A. Absolute quantification of mRNA using real-time reverse transcription polymerase chain reaction assays. J. Mol. Endocrinol. 25 (2), 169-193 (2000).
- Andersen, C. L., Jensen, J. L., Orntoft, T. F. Normalization of real-time quantitative reverse transcription-PCR data: a model-based variance estimation approach to identify genes suited for normalization, applied to bladder and colon cancer data sets. Cancer Res. 64 (15), 5245-5250 (2004).
- Bustin, S. A., et al. The MIQE guidelines: minimum information for publication of quantitative real-time PCR experiments. Clin Chem. 55 (4), 611-622 (2009).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40 (7), e51(2012).
- Galiveti, C. R., Rozhdestvensky, T. S., Brosius, J., Lehrach, H., Konthur, Z. Application of housekeeping npcRNAs for quantitative expression analysis of human transcriptome by real-time PCR. RNA. 16 (2), 450-461 (2010).
- Laurell, H., et al. Correction of RT-qPCR data for genomic DNA-derived signals with ValidPrime. Nucleic Acids Res. 40, e51(2012).
- Caldana, C., Scheible, W. R., Mueller-Roeber, B., Ruzicic, S. A quantitative RT-PCR platform for high-throughput expression profiling of 2500 rice transcription factors. Plant Methods. 3 (1), 7(2007).
- Lawrence, R. J., Pikaard, C. S. Perspectives Chromatin Turn Ons and Turn Offs of Ribosomal RNA Genes. Cell Cycle. 3 (7), 880(2004).
- Boisvert, F. M., van Koningsbruggen, S., Navascues, J., Lamond, A. I. The multifunctional nucleolus. Nat Rev Mol Cell Biol. 8 (7), 574-585 (2007).
- Alvarez, I., Wendel, J. F. Ribosomal ITS sequences and plant phylogenetic inference. Mol Phylogenet Evol. 29 (3), 417-434 (2003).
- Jobes, D. V., Thien, L. B. A conserved motif in the 5.8 S ribosomal RNA (rRNA) gene is a useful diagnostic marker for plant internal transcribed spacer (ITS) sequences. Plant Mol Biol Report. 15 (4), 326-334 (1997).
- Padhi, B. K., Singh, M., Huang, N., Pelletier, G. A PCR-based approach to assess genomic DNA contamination in RNA: Application to rat RNA samples. Anal Biochem. 494, 49-51 (2016).
- Dheda, K., et al. Validation of housekeeping genes for normalizing RNA expression in real-time PCR. Biotechniques. 37 (1), 112-114 (2004).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved