このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
原子間力顕微鏡を用いた単一タンパク質分子の力分光法
要約
詳細な手順と機械的性質および原子間力顕微鏡を用いた単一タンパク質分子のアンフォールディング経路を機械的に測定するための戦略について述べる。また選択と良い単一蛋白質分子の録音の正当化のための参照として代表的な結果を述べる.
要約
ネイティブ 3 D 構造にそのアミノ酸配列からのタンパク質の折り畳み過程の定量生物学の重要な問題では。原子間力顕微鏡 (AFM) ストレッチと特定のアンフォールディングとリフォールディング特性の直接証拠を与える単一タンパク質分子の弛緩を有効にしてこの問題に対処することができます。原子間力顕微鏡による単一分子力分光 (原子間力顕微鏡 SMF) は、高エネルギーは従来のばら積み (生化学) 測定不可能なタンパク質立体を一貫して測定するための手段を提供します。AFM SMF の原則を示すため、公開された多数の論文じゃない簡単に網羅的なプロトコルの不足のための SMF 実験を実施できます。本研究では、AFM の原理を簡単に説明して SMF の実験からのよい結果を達成するためにガイドラインとしてプロトコル、手順、およびデータ分析の詳細を広く。単一蛋白質機械的展開測定の代表的な SMF 結果を示すし、一般的、いくつかの戦略をトラブルシューティング問題が発生いたします。
概要
原子間力顕微鏡による単一分子力分光 (SMF) の進歩は、機械操作と単一タンパク質分子の正確な評価に有効にしています。この特性は、タンパク質力学1,2,3タンパク質-リガンド相互作用4、蛋白質蛋白質の相互作用5、フォールディングについて知見を生産している、タンパク質ベース設計材料6,7,8。SMF は特にタンパク質を調べる場合に役立ちます AFM によるストレッチとして展開、継続的に増加輪郭線長を生じ、その剛性によると徐々 に拡張するタンパク質分子内における化学的および物理的債をできます。このタンパク質分子の overstretching 破裂イベントの結果力拡張曲線の急激な遷移を生成 (したり強制的にピーク)。力ピーク値は、力学の展開プロセス中にタンパク質の展開力と構造変化に関する直接的な情報を提供します。AFM を用いた最初の研究の一つは、タイチン1を測定し、タンパク質のアンフォールディングとリフォールディング集中化学薬品、極端な温度のような不自然な変性剤を使用せず生理条件下での新しい側面を発見します。
ここで原子間力顕微鏡だけを考えても SMF 実験をさまざまな楽器に行った.AFM は、4 つの主要な要素で構成されます: プローブ、検出器、試料ホルダー、圧電スキャナー。プローブは、片持ちの揺れる端に鋭い先端です。キャリブレーション後フックの法則を使用して力を正確に判断するカンチレバーの背面から反射されるレーザー光を用いた測定接続されている分子のストレッチ中に、片持ち梁の曲げ。ダイオード センターからのレーザービームの変位に比例した電圧を作り出す象限フォト ダイオード検出器に反射レーザー ビーム プロジェクト。流体の蛋白質のサンプルの基板は、サブナノ精度で制御することができます 3 D 圧電ステージ上にマウントされます。フォト ダイオード検出器からの電圧を読み取り、コンピューター制御の電圧供給を通じて 3 D ステージを制御コンピューター。これらの圧電アクチュエータの段階通常容量が装備されているまたは歪みゲージ センサー位置正確に測定圧電変位してフィードバック制御システムを通じて正しい履歴。圧電コント ローラーからセンサー信号出力は、工場出荷時校正は、ピエゾの電圧定数を使用して距離に変換されます。引っ張る実験例力拡張曲線は図 2に示します。
AFM SMF 実験の 2 つのタイプがある: 一定速度、一定力測定を引っ張るします。Oberhauserらのコンスタントフォース SMF 測定値の説明します。9、ここで我々 は一定速度の測定に焦点を当てる一方。典型的な原子間力顕微鏡等速引張実験は、カンチレバー先端に対して基板をゆっくり移動するピエゾに電圧を提供することによって行われます。典型的な実験は最初表面に対して押すとヒントです。引きの測定は、接触の引き出すため先端から基板を移動によって開始されます。場合蛋白質の最初の接触は先端、それが取得され、変位に対する力の展開のトレースで測定されます。先端と接触し、基板を戻され、タンパク質の折り畳みを決定力変位からことができます、リラックスしたトレースを測定します。
プロトコル
1. 蛋白質の準備
- DNA のクローニング。
- 興味、例えば NI10C10、DNA シーケンスの DNA シーケンスの合成または経由でPCR 標準分子生物学技術11を使用してホストの有機体から分離します。合成中にまたは (Addgene #74888)12プラスミド pEMI91 のモジュールに対応する PCR プライマーの 5' 末端にサイトを配置することによって、制限のサイトと興味の遺伝子を逃げ。
- 別にプラスミド pEMI9112と制限のサイトのペアを持つ興味の DNA シーケンスの両方は、タンデム I91 を繰り返します (図 1参照) で両側の興味のシーケンスになりますのでダイジェストします。制限のサイトの標準的なプロトコルに従います。
- ゲル電気泳動を使用して消化、製品を浄化し、T4 DNA リガーゼ、次の標準的なプロトコルを使用している製品を縛る。結紮、プラスミッドの浄化のためのエシェリヒア属大腸菌のセルにプラスミッドを変換し、標準的なプロトコルを使用してプラスミッドの浄化します。T7 プライマーまたは内部 pEMI91 プライマーを使用して、シーケンスが正常に変換を確認するシーケンス。
- 変換です。
- C41 などのタンパク質発現細胞を削除 (DE3) pLysS 細胞、-80 ° C のフリーザー アイスで完全に雪解けから。
- セルに DNA のプラスミッドの 1 μ L を追加し、簡単にかき混ぜるそれは気泡を導入し、細胞を温めると、上下にピペッティングは勧められません。
- セルと 30 分間氷の上プラスミドを含む培養管を孵化させなさい。
- 熱衝撃 45 42 ° C の水浴中で細胞 s。
- 2 分間氷にチューブを返します。
- セル内流体培養基 LB の 950 μ L を置き、37 ° C で 1 時間 250 rpm で振る
- 100 μ g/mL アンピシリン含有 LB プレートに変換の約 200 μ L を配置します。PEMI91 以外のプラスミドを使用している場合は、そのプラスミッドのため適切な抗生物質を使用します。37 ° C でインキュベーターで一晩プレートを配置します。次の日、インキュベーターから削除板パラフィルム、ラップし、ストア冷凍 1 ヶ月。
- 蛋白質の表現。
- プレートに単一の細菌のコロニーを滅菌ピペット チップを触れると、LB の上下にピペッティングで一晩成長 37 ° C で 50 mL のチューブに 100 μ g/mL アンピシリンと LB ブイヨン培地 15 mL を接種します。
- 100 μ g/mL アンピシリンと LB ブイヨン培地 1 L に 15 mL 文化を転送し、37 ° C で 4-12 h に振る (まで外径600 > 0.8)。
- プラスミッドの準備の文化の 10 mL を収集します。
- 0.2-1 mM イソプロピル-β-D-thiogalactopyranoside (IPTG) を追加し、一晩式室温に温度を下げます。
- 次の日 4 本のチューブに分割し、40 分間 4,000 × gで遠心分離によって細胞を収穫し、数時間-80 ° C でペレットを凍結します。
- タンパク質12の挿入されたカセットに対応するプライマー配列の細胞から抽出したプラスミドを送信し、挿入 DNA の忠実度を確認します。
- 蛋白質の浄化。
- 室温で 30 分間凍結細胞の 1 つの管を解凍します。
- 解凍細胞換散バッファー (38 mL の水、グリセリン、50 mM トリス塩酸 pH 7.6, 150 mM の NaCl、1 mM の CaCl2、10 μ g/mL DNase、1 mM PMSF、1 mM TCEP、500 μ G/ml リゾチーム 2 mL) を中断し、1 時間氷の上を振る。
- いくつかの時間-80 ° C で分離細胞を凍結し、再室温で解凍。
- 4 ° C で 30 分間 13,100 × gでダウン ライセート スピンします。
- 特定のタグ (例えば、連鎖球菌タグまたはタグ) の重力流列を上清を実行します。
- 連鎖球菌性タグまたはタグと使用前に 4 ° C でストアの適切なバッファーを用いた遠心フィルターを使用してバッファー交換を実行します。
2. サンプル準備のためをスライドします。
- ピラニア溶液を用いたガラス スライドを準備します。
- 場所 10-30 個の 40 mL ガラス製ビーカーにガラス カバー スリップ (半径 7.5 mm) のラウンドします。
注:フードでは、この手順と次の手順を実行します。このステップの間に危険な化学物質に対処するための標準的な手順に従います。 - (18.4 M) の濃硫酸 30 mL を追加します。
- 30% 過酸化水素の 10 mL を追加します。
- 95 ° C に 10-30 分のための混合物を熱します。
- (再使用または破棄) を別の廃棄物容器にデカント ピラニア ソリューションが慎重に。
- ピラニア ソリューションを削除する脱イオン水でスライドをすすいでください。
- 40 mL のアセトンにスライドを中断します。
- 廃棄物容器にアセトンを捨て、再 40 mL のエタノールにスライドを停止します。
- 慎重に、時に 1 つのスライドを抽出し、アルゴンまたは浄化された空気と乾燥きれいな鉗子を使用します。
- 場所きれいな、乾燥したスライド金蒸着まで真空下でまたは長期保管のためのアルゴン中。
- 場所 10-30 個の 40 mL ガラス製ビーカーにガラス カバー スリップ (半径 7.5 mm) のラウンドします。
- (省略可能) ゴールド コーティング スライドを準備します。
注:この手順には、主要な大学で多くのクリーン ルーム設備で利用できる真空 (電子ビーム) 蒸発器の使用が必要です。- 5 つの顕微鏡スライド (25 mm × 75 mm の長方形) を glasscutter を使用して半分にカットします。
- それぞれの 10 の中央に両面粘着テープを適用半分顕微鏡スライド。
- 付箋の粘着面をカットし、両面テープに適用されるように、メモの粘着面です。付箋は、一般的に以下の接着剤はまだしっかりと将来の破損を防ぐためにスライドに添付します。その後、それらの半分の顕微鏡スライド ガラス スライド ホルダーとして処理できます。
- ホルダーの粘着面の各コーナーに (セクション 2.1.10) から 4 つのきれいなガラス スライドを慎重に押します。
- 電子ビーム蒸発器にガラス スライドを移動します。70 を適用する蒸発器の仕様に従うクロムと 300 nm の表面に金の nm。
- アルゴン中ゴールド コーティング スライドを保存します。
3. サンプル準備
-
スライドを準備します。
- きれいな鉄のディスク (直径 15 mm) を選択し、粘着タブを添付します。
- 粘着タブのカバー作品の品をはずします。
- (セクション 2.1.10) からきれいなガラスまたは (セクション 2.2.7) から金の一部を選択、しっかりと鉄のディスクの粘着面に配置このサンプル スライドになります。
- スライド上にタンパク質を沈殿物します。
- ポリプロテインを dialyze 実験 (25 mM トリス-HCl、150 mM の NaCl と pH 7.6) のバッファーにバッファー交換カラム 0.5 mL 遠心フィルターなどを用いるします。13,000 rpm で 10 分間の列でタンパク質をスピン列を逆にし、2 分間 1,000 rpm で回転して新しいバッファーにタンパク質を溶出します。
- 280 で吸光度を測定することによりおおよそのタンパク質濃度を決定する nm、分光光度計を使用しています。
- 10-100 μ g/ml 100 μ L の最終巻にタンパク質を希釈します。
- スライドの中央にタンパク質溶液の 60 μ L を適用します。これは実験と制御されていないサンプルの動きの間に接着剤の膨張を引き起こす可能性がある任意の液体ガラスと鉄スライド間のギャップに下入力をさせないこの段階でつけてください。
注:ゴールドのスライド、疎水性の球状液滴を形成、スライド ガラスは親水性、タンパク質溶液が広がる可能性があります。 - 10-60 分間室温で放置サンプルを聞かせてください。この間、原子間力顕微鏡のセットアップを開始する次の手順に進みます。
4. 原子間力顕微鏡 (AFM) セットアップ
注:以下は、原子間力顕微鏡をセットアップするための一般的な説明およびある特定の細部が使用特定の機器によって異なる場合があります。器械使用は部分的に家造られた、ショル13で詳細に記述されています。
- 原子間力顕微鏡セルにカンチレバーをマウントします。
- アプリケーションの適切なプロパティを持つ片持梁を選択します。使用は 4-10 pN/nm 低展開力のバネ定数とカンチレバー (10 〜 50 pN)、バネ定数と使用のカンチレバー 15 100 pN/nm 高い展開力の。
- 慎重に端から片持ち梁をピックアップし、セルを保持するプローブに配置。
- 場所でしっかりと保持細胞は、カンチレバーを保持続行する前に確認してください。
- レーザーの配置のための AFM ヘッドに保持セルを配置します。
- AFM ヘッドにレーザーを合わせます。
- 倒立顕微鏡上に頭を置き、頭にレーザーを駆動するための AFM ヘッドにバッテリー パックを接続します。
- 顕微鏡検出器にカメラを接続して、テレビまたはモニターの上にレーザー光を視覚化できます。
- カンチレバーの先端にあるので、レーザーを位置します。
- サンプル ヘッドをマウントします。
- 各セルを保持するプローブのポートに 10 μ L のバッファーをフラッシュします。
- 培養されているサンプルのスライドと培養液からの流体の 40 μ L をデカントします。スライド上のバッファーの 40 μ L を追加します。上記の圧電磁石にサンプル スライドを配置します。
- 原子間力顕微鏡ステージが高い位置にあることを確認します。AFM カンチレバーがサンプル液滴になるので、ステージ上に AFM ヘッドを配置します。
- センター フォト ダイオード上に反映されたレーザー ビーム。
- 紙の小片をカットし、AFM フォト ダイオードの前に置きます。
- ノブを使用して用紙にレーザー スポットになる集中的かつ明るい原子間力顕微鏡レーザーの位置を変更します。
- レーザーがゼロ (A + B ・ C ・ D) を 2 つの領域をヒットすべての象限 (A + B + C + D) で全信号を最大にするフォト ダイオードと上の 2 つの間の差信号を発生上下ように AFM ヘッド ミラーを調整します。
5. 原子間力顕微鏡校正
- パワー スペクトルを測定します。
- AFM に接続されているフィルター、帯域幅をフルに AFM ヘッド信号のフィルター設定を有効にします。
- 自体 AFM に関する信号にノイズが追加されますこの圧電が消灯していることを確認します。
- AFM ソフトウェア14を使用して、パワー スペクトルの計算ごとの 1,024 データ ポイントからの 512 の計算の平均値を測定します。
- カンチレバーの振動の主要なモードに対応する最初のピークの間でパワー スペクトル密度を統合するのに AFM ソフトウェア15を使用します。
- フォト ダイオードの感度を計算します。
- 自体 AFM、圧電コント ローラーをオンし、500 Hz (低域通過フィルター) にフィルター設定を変更します。
- AFM ソフトウェアでゼロの周り変動すべき差信号を監視します。迅速 micropositioners を使用して数百マイクロメートル下 AFM ヘッドを移動します。頭を下へ移動を繰り返し、一貫性のあるジャンプの差信号を監視します。表面は非常に近くのとき信号がジャンプを開始高さで増加しています。
- すぐに偏向信号は表面から頭がわずかに動いて、表面との接触時に飽和します。
- Afm によるソフトウェアの入力コントロールを使用して、100 5,000 nm の上下を移動することによって、表面を見つけるにピエゾ電圧を調整します。それでも表面が到達しない場合は、手動で頭とサンプル間の距離を減らすために続けます。
- AFM ソフトウェアでスキャン サイズ約 500 の引き抜き実験を行う nm 先端は、約 100 の連絡先、ピエゾ旅行の nm。
- AFM の先端が基板表面と接触して残る圧電変位曲線とフォト ダイオード信号の線形領域の勾配を測定します。
- ばね定数と斜面と統合されたパワー スペクトルを用いた感受性を計算 (
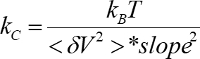 )。
)。
6. データ集録
- 準備。
- AFM に接続されているフィルター、フィルター設定をサンプリング周波数は、少なくとも 2 倍の帯域 (ナイキスト基準)、低域カットオフの上限を与えるに設定します。
- 測定。
- スキャン サイズを展開ポリタンパク質 (アミノ酸 × 0.365 数) に加えて基板に対して押すことを許可する約 40% の総理論的なサイズに設定します。たとえば、9 x I91 構築を引いて約 300 の理論展開長さを必要がのでスキャン サイズ、約 420 nm nm。
- AFM ソフトウェア表面かスキャン サイズの 80% にあるカンチレバーを位置 (340 についてこの場合、サーフェスから nm)。
- AFM ソフトウェアで 300 nm/秒に最初のスキャン速度を設定します。
注:この速度は、低速または高速の用途に変更できます。 - 引き抜き実験を実行し、圧電、フォト ダイオードの信号の位置を測定します。原子間力顕微鏡のソフトウェアを使用すると、スキャンを開始します。
- AFM ソフトウェアで約 10,000 の録音まで測定を実行し続けます。
注:通常、肯定的な制御分子のピックアップ率は 0.516、だから 10,000 は、分析するのに十分なデータを収集できるようにする必要の周りです。
7. データ解析
- データを正規化します。
- 各記録のための拡張機能を使用して、拡張子を計算 = 変位 - F/kc (kc は 5.2.7 から)。
- 力ピークが存在しない力拡張トレースの前後いずれかの平均を取ることによってベースライン力レベルを決定し、カンチレバーが表面に対して押していません。全体の力拡張曲線は、この平均値で、シフトことができます。
- ゼロの y 軸に沿って拡張機能が揃うようにデータを変換します。これは分子を拡張トレースの先頭をゼロに設定する必要がありますのでです。
- 興味の蛋白質を識別します。
- 単一分子の指紋を提供する少なくとも 4 つの I91 イベントが付いている録音を識別します。これらの録音は、真の「単一分子測定」をキャプチャします。
- さらなる分析のための印の単一分子イベント イベントを保存します。
- 輪郭の長さの増分を決定します。
- 録音ごとに展開イベントにワームのようなチェーン モデルに合うよう
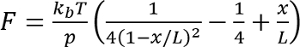 、
、 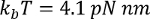 、室温でpは永続化長さ (通常 0.4 を 1 nm)、 xはナノメートルの拡張子、 Lは輪郭線長ナノメートル。
、室温でpは永続化長さ (通常 0.4 を 1 nm)、 xはナノメートルの拡張子、 Lは輪郭線長ナノメートル。
注:ワームのようなチェーンのフォームは正確に補間されたソリューション、Bouchiatらのより厳密な数値解法を発見17. 蘇らに代替のフィッティング モデルを見つけることができます18。 - L は、2 つの連続した展開イベントの決定し、この違いは鋳造の値の差を計算「輪郭の長さの増分」.
- 録音ごとに展開イベントにワームのようなチェーン モデルに合うよう
- 破断力を決定します。
- 展開曲線に最大の低下の前に最高点を取ることによって特定の展開イベントの破断力を計算します。
結果
この議定書から代表的な結果は、図 2のとおりです。両方のパネルは、タンパク質から代表的な力-伸長曲線を示します。上部下部 I91 蛋白質、蛋白質の金利、NI10C 分子の側面を示しています I91 エンベロープタンパク質の結果を示しています。これらの録音は I91 の特徴的な力を表示 (200 pN) と長さの増減値を輪郭 (28 nm) の配置および AFM ?...
ディスカッション
プロトコルの重要なステップは、単一分子イベント「指紋」をポジティブ コントロールとして機能する 1.1.2、の手順に従って、エンベロープタンパク質の使用です。一般に、ある必要があります開くポリタンパク質蛋白質のイベント (I91、つまり約 28 約 200 pN と輪郭線長さ増分の展開力 nm) 興味の蛋白質が折り畳まれたされていることを明確に締結します。たとえば、興味の蛋白質は、どち?...
開示事項
著者が明らかに何もありません。
謝辞
この作品は、全米科学財団助成金 MCB 1244297 と PEM に MCB 1517245 によって支持されました。
資料
| Name | Company | Catalog Number | Comments |
| AFM Specimen Discs, 15mm diameter | Ted Pella, Inc. | 16218 | Serve as base for glass substrate |
| Round Glass Coverslips, 15mm diamiter No.1 Thick | Ted Pella, Inc. | 26024 | serve as glass substrate and base for gold coating |
| Adhesive Tabs | Ted Pella, Inc. | 16079 | Paste on AFM Specimen Discs to provide a sticky face for attaching glass coverslips |
| STD Multimode head assembly | Bruker Nano Inc. | 1B75C | AFM head |
| Glass probe holder | Bruker Nano Inc. | MTFML-V2 | Glass probe holder for scanning in fluid with the MultiMode AFM. |
| Microlever AFM probes | Bruker Nano Inc. | MLCT | Silicon Nitride cantilevers with Silicon Nitride tips, ideal for contact imaging modes |
| AFM probes with Au coated tips | Bruker Nano Inc. | OBL-10 | Cantilevers for pulling on proteins with low unfolding force |
| Multifunction Data Acquisition (DAQ) Card,16-Bit, 1 MS/s (Multichannel), 1.25 MS/s (1-Channel), 32 Analog Inputs | National Instruments | PCI-6259 | Data Acquisition for signals from AFM head and Piezo Actuators |
| LISA Linear Piezo Stage Actuators | Physik Instrumente LP | P-753.11C | Piezo Actuator to control the position of substrate and perform pulling measurements |
| XY Piezo Stage | Physik Instrumente LP | P-541.2CD | Piezo Actuator to control the position of substrate and scan on substrate surface |
参考文献
- Rief, M., Gautel, M., Oesterhelt, F., Fernandez, J. M., Gaub, H. E. Reversible Unfolding of Individual Titin Immunoglobulin Domains by AFM. Science. 276 (5315), 1109-1112 (1997).
- Fisher, T. E., Oberhauser, A. F., Carrion-Vazquez, M., Marszalek, P. E., Fernandez, J. M. The study of protein mechanics with the atomic force microscope. Trends in Biochemical Sciences. 24 (10), 379-384 (1999).
- Ng, S., Rounsevell, R., Steward, A., Randles, L., Clarke, J. Single molecule studies of protein folding by atomic force microscopy(AFM). Abstracts of Papers of the American Chemical Society. 227, U545-U545 (2004).
- Rico, F., Chu, C., Moy, V. T., Braga, P. C., Ricci, D. . Methods in Molecular Biology. 736, 331-353 (2011).
- Muller, D. J., Dufrene, Y. F. Atomic force microscopy as a multifunctional molecular toolbox in nanobiotechnology. Nature Nanotechnology. 3 (5), 261-269 (2008).
- Lv, S., et al. Designed biomaterials to mimic the mechanical properties of muscles. Nature. 465 (7294), 69-73 (2010).
- Kim, M., et al. Nanomechanics of Streptavidin Hubs for Molecular Materials. Advanced Materials. 23 (47), 5684-5688 (2011).
- Gonzalez, M. A., et al. Self-Adhesive Hydrogels from Intrinsically Unstructured Proteins. Advanced Materials. , (2017).
- Oberhauser, A. F., Hansma, P. K., Carrion-Vazquez, M., Fernandez, J. M. Stepwise unfolding of titin under force-clamp atomic force microscopy. Proceedings of the National Academy of Sciences. 98 (2), 468-472 (2001).
- Li, Q., Scholl, Z. N., Marszalek, P. E. Capturing the Mechanical Unfolding Pathway of a Large Protein with Coiled-Coil Probes. Angewandte Chemie International Edition. 53 (49), 13429-13433 (2014).
- Davis, L. . Basic methods in molecular biology. , (2012).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. A Modular, Non-Degenerate Polyprotein Scaffold for Atomic Force Spectroscopy. Biomacromolecules. , (2016).
- Scholl, Z. N. . The (Un) Folding of Multidomain Proteins Through the Lens of Single-molecule Force-spectroscopy and Computer Simulation. , (2016).
- Pawlak, K., Strzelecki, J. Nanopuller-open data acquisition platform for AFM force spectroscopy experiments. Ultramicroscopy. 164, 17-23 (2016).
- . Nanopuller Available from: https://sourceforge.net/projects/nanopuller/ (2018)
- Scholl, Z. N., Marszalek, P. E. Improving single molecule force spectroscopy through automated real-time data collection and quantification of experimental conditions. Ultramicroscopy. 136, 7-14 (2014).
- Bouchiat, C., et al. Estimating the persistence length of a worm-like chain molecule from force-extension measurements. Biophysical journal. 76 (1), 409-413 (1999).
- Su, T., Purohit, P. K. Mechanics of forced unfolding of proteins. Acta. 5 (6), 1855-1863 (2009).
- Steward, A., Toca-Herrera, J. L., Clarke, J. Versatile cloning system for construction of multimeric proteins for use in atomic force microscopy. Protein science. 11 (9), 2179-2183 (2002).
- Scholl, Z. N., Josephs, E. A., Marszalek, P. E. Modular, Nondegenerate Polyprotein Scaffolds for Atomic Force Spectroscopy. Biomacromolecules. 17 (7), 2502-2505 (2016).
- Hoffmann, T., et al. Rapid and Robust Polyprotein Production Facilitates Single-Molecule Mechanical Characterization of β-Barrel Assembly Machinery Polypeptide Transport Associated Domains. ACS. 9 (9), 8811-8821 (2015).
- Dudko, O. K., Hummer, G., Szabo, A. Theory, analysis, and interpretation of single-molecule force spectroscopy experiments. Proceedings of the National Academy of Sciences of the United States of America. 105 (41), 15755-15760 (2008).
- Popa, I., Berkovich, R., Alegre-Cebollada, J., Rivas-Pardo, J. A., Fernandez, J. M. Halotag Tethers to Study Titin Folding at the Single Molecule Level. Biophysical journal. 106 (2), 391a (2014).
- Yu, H., Siewny, M. G., Edwards, D. T., Sanders, A. W., Perkins, T. T. Hidden dynamics in the unfolding of individual bacteriorhodopsin proteins. Science. 355 (6328), 945-950 (2017).
- Rico, F., Gonzalez, L., Casuso, I., Puig-Vidal, M., Scheuring, S. High-speed force spectroscopy unfolds titin at the velocity of molecular dynamics simulations. Science. 342 (6159), 741-743 (2013).
- He, Y., Lu, M., Cao, J., Lu, H. P. Manipulating protein conformations by single-molecule AFM-FRET nanoscopy. ACS nano. 6 (2), 1221-1229 (2012).
- Fotiadis, D., Scheuring, S., Müller, S. A., Engel, A., Müller, D. J. Imaging and manipulation of biological structures with the AFM. Micron. 33 (4), 385-397 (2002).
- Edwards, D. T., Faulk, J. K., LeBlanc, M. A., Perkins, T. T. Force Spectroscopy with 9-μs Resolution and Sub-pN Stability by Tailoring AFM Cantilever Geometry. Biophysical journal. 113 (12), 2595-2600 (2017).
- Dudko, O. K., Mathe, J., Szabo, A., Meller, A., Hummer, G. Extracting kinetics from single-molecule force spectroscopy: Nanopore unzipping of DNA hairpins. Biophysical. 92 (12), 4188-4195 (2007).
- Scholl, Z. N., Li, Q., Yang, W., Marszalek, P. E. Single-molecule Force Spectroscopy Reveals the Calcium Dependence of the Alternative Conformations in the Native State of a βγ-Crystallin Protein. Journal of Biological Chemistry. 291 (35), 18263-18275 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved