このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
コメットアッセイを用いた DNA 損傷の in Vitro評価
要約
コメット ・ アッセイは、単一および二重鎖 DNA を含む DNA 損傷の中断を検出する効率的な方法です。化学療法の治療効果を評価するためがん細胞における DNA 損傷を測定するアルカリ性と中性の彗星の試金を記述します。
要約
DNA 損傷は、その寿命の間にセルごとに一般的な現象であるし、ゲノム DNA の化学構造の変化として定義されています。ラジオや化学療法などのがんの治療法は、癌の進行を抑える細胞周期停止やアポトーシスにつながる追加の DNA 損傷の膨大な量を紹介します。実験的ガン治療中に DNA 損傷の定量評価は、遺伝毒性エージェントの有効性を正当化するための重要なステップです。本研究では単一を定量化できますコメット試験として知られている単一細胞電気泳動アッセイに着目し、体外で二重鎖 DNA の区切り。コメット ・ アッセイは、効率的かつ容易に行える、DNA 損傷定量化法、低時間/予算要求、高い再現性。ここでは、我々 は olaparib/テモゾロミド併用療法 U251 神経膠腫細胞の遺伝毒性影響を評価することで臨床研究のコメット ・ アッセイの有用性を強調表示します。
概要
最初、1984 年は、中性条件1下核から断片を DNA の移行を示すことによっての Ostling と・ ジョハンソンによって開発コメット ・ アッセイが使用されました。テクニックは、シンら、アルカリ条件は、特異性と再現性アッセイ2に大幅に増加を示すによって後で開発されました。以来、中立コメットアッセイをほとんど使用して対し、アルカリ コメットアッセイは少量のシングルを含む、DNA 損傷に対してより敏感、二重鎖 DNA 切断、アルカリ不安定なサイト DNA DNA 二本鎖 DNA 切断を検出するか、Dna-タンパク質の架橋と不完全切除に関連付けられている DNA 一本鎖切断修復サイト3,4。両方試金断片化された DNA の可視化および DNA 損傷を定量的に評価する簡単な方法を提供できます。コメット ・ アッセイはin vitroとin vivo遺伝毒性学的研究法が機密とみなされ、初期医薬品候補の選択、環境モニタリング、人間バイオモニタ リングなど、様々 な研究分野に適用基礎 DNA 損傷研究し、5を修復します。
アッセイの原理は、電場下で断片化された DNA 核体 (「彗星頭」とも呼ばれる) のうちの移行、agarose のゲル (「彗星テール」とも呼ばれる) の DNA 汚れを形成します。ヌクレオチドは染色、この単一細胞の電気泳動によって形成された「彗星」を分析することによって DNA 損傷の程度を定量化することができます。テール モーメントの計算は、さまざまな実験的グループの間で DNA 損傷を比較するさらに助けることができます。DNA 損傷の検出の伝統的な方法と比較して、コメット ・ アッセイは直接、敏感な安価な比較的単純です。
放射線治療と化学療法は、癌治療のための一般的な戦略一本鎖の生成、二重鎖 DNA 染色体6に改します。DNA 修復阻害剤の最近の進歩の併用療法によりより効果的な遺伝毒性効果としたがって、貧血、感染症、骨髄抑制7,などの全身性の副作用が減る可能性があります。8します。 本研究で示したポリ (adp-リボース) ポリメラーゼ (PARP 阻害剤, olaparib (Ola)9の調査。PARP は豊富な核蛋白質である、10ポリ (adp-リボース) ポリマーを形成することにより DNA 塩基除去修復を担当。テモゾロミド (TMZ) は、経口投与可能なアルキル化剤で神経膠腫患者の治療に広く使用されています。DNA 損傷を定量化するコメット ・ アッセイを用いたテモゾロミドと深く olaparib を組み合わせて olaparib/テモゾロミド併用療法は神経膠腫の治療に効果的な戦略を提案する神経膠腫細胞における DNA 損傷を高めることを紹介します。テモゾロマイドだけで11と比較。
プロトコル
1。 準備試薬
- 1x PBS
- 900 mL dH 2 O 100 mL 10 x PBS を希釈し、pH メーターを使用して 7.4 に pH を調整します。室温で保管します 。
- 換散ソリューション (LS)
- 2.5 M の NaCl、100 ミリメートル二ナトリウム EDTA、10 mM Tris ベース、および 200 mM NaOH 900 mL dH 2 O の準備; よく完全に溶解する混合物を許可するように約 20 分かかります。PH メーターを使用して 10 の pH を調整します。追加 1% ナトリウム ラウリル油脂肪酸サルコシンと 1% トリトン X-100 1,000 mL に最終的な音量を調整して。使用する前に、少なくとも 30 分のための 4 ° C に涼しい 。
- アルカリ電気泳動ソリューション (AES)、pH > 13
- 準備 200 mM NaOH と 1 mM ナトリウム EDTA 800 mL dH 2 O. 調整 pH と pH であることを確認してください > 13。1,000 mL に最終的な音量を調整します。使用する前に新鮮なを確認し、使用する前に、少なくとも 30 分のための 4 ° C に冷却します 。
- 中立電気泳動 s sollution (ファミコン)
- 氷酢酸と 100 mM 1,000 mL dH 2 O. 調整トリス酢酸ナトリウム ベースと 300 mM 9.0 pH を混合して準備 1,000 mL 中立電気泳動バッファー。使用する前に、少なくとも 30 分のための 4 ° C に涼しい 。
- DNA の沈殿物の解決 (DPS)
- 10 mL 7.5 M アンモニウム酢酸ストックの準備。DNA の沈殿物溶液の 50 mL 43.3 mL 95% エタノール 6.7 mL 7.5 M 酢酸アンモニウムを混在させます。室温で保管します 。
- 染色ソリューション
- 追加 1 μ L 10,000 緑色蛍光核酸染色 (例えば、SYBR グリーン) 30 mL トリス EDTA バッファー (10 mM トリス-HCl、1 mM ナトリウム EDTA、pH 7.4) とストア x 4 ° c. の保護から光です 。
- 1% 低融点アガロース
- 1% 低溶解点 agarose (100 mL dH 2 O で 1 g) 電子レンジで溶かします。旋回 agarose agarose が完全に溶融していることを確認するすべての 15-20 秒。使用する前に、少なくとも 20 分の 37 ° C の水浴の agarose を配置します 。
- 中古暖かいピペット チップ
- アガロースをピペッティングする前に 3 mm、37 ° C で暖かい P200 ピペット チップの狭い両端を切っています 。
2。彗星のスライドを準備
- コーティング スライド
- 2 〜 3 分または agarose が完全に溶融するまで電子レンジで溶かす 1% アガロース (100 mL dH 2 O に 1 g)。Agarose にガラス顕微鏡スライドを浸し、糸くずのワイプを使用してスライドの 1 つの側面を拭く 。
- は、空気乾燥または乾燥時間 50 ° C で熱を平らな面にスライドを置く; 乾燥後透明アガロース フィルムを形成する必要があります。使用する前に 37 ° C でコーティング スライドを配置します 。
- 単一細胞懸濁液の準備
- 文化と治療神経膠腫細胞
- 10 DMEM ハム F-12 培に U251 MG 細胞の培養 37 ・ # で 10 μ G/ml ストレプトマイシン、100 U/mL ペニシリン %fbs176;5% CO 2 C.
- は、3 分の 1 mL のトリプシンを用いた細胞を消化し、FBS と DMEM ハム F-12 培地を使用してトリプシンを中和します。収集 15 mL チューブの 4 分の 300 x g でスピン、培地を吸引、2 x 10 の 5 セル/mL の 1x PBS で細胞を中断します
。 注: セルのサンプルは、すぐに測定を開始する前に準備されるべき、すべてのサンプルは光から DNA の損傷を防ぐために暗いまたは淡色表示環境で処理する必要があります 。
- 併用細胞懸濁液 1% 溶融低融点アガロース (37 ° C) での比率で 1:10 (v/v)、上下、ピペットで穏やかに混合し、すぐにスライドに 30 μ L をピペットします。側を使用、薄層の形成を確保する agarose/細胞の混合物を広めるためピペット チップ 。
- 10 分 30 分ゲル化時間時間を増やす高湿度環境でのサンプルの付着を向上させるため、暗闇の中で 4 ° C でフラット スライドを配置します 。
- 4 ° C LS 一晩に 1 時間の暗闇の中でスライドを没頭します 。
- 文化と治療神経膠腫細胞
3。単一の細胞電気泳動
- アルカリ (ステップ 3.2) に進むまたはアッセイ ニュートラル (ステップ 3.3) 彗星
- アルカリ コメットアッセイの
- そっと LS からスライドを削除、余分なバッファーをドレインし、優しく AES で 4 で 1 時間浸漬° C DNA の巻き戻しを許可します。暗闇の中のスライドを保持します 。
- 電気泳動のスライド トレイに追加済みチルド AES (電気泳動ユニットのサイズによって異なります) スライド上 0.5 cm を超えないように、内スライドとキャップ カバーを置きます。電源電圧を 1 V/cm (電極間の長さ) に設定し、4時 30 分実行 ° C
- ドレイン余分な電気泳動ソリューションのスライドから。DH 2 O 室温で 5 分間の 2 回のスライドをゆっくり浸す 。
- は軽く 70% エタノール室温で 5 分間のスライドを浸します。手順 4 に進みます 。
- 中性コメットアッセイの
- そっと LS からスライドを削除、余分なバッファーをドレインし、4時 30 分の NES 優しく浸す ° C. 保つ暗闇の中スライド 。 電気泳動スライド トレイに
- 追加中古冷蔵ニュートラル電気泳動バッファーは 0.5 cm スライド (電気泳動ユニットのサイズによって異なります) を超えていない、内側スライドとキャップ カバーを置きます。電源電圧を 1 V/cm (電極間の長さ) に設定し、4時 45 分に実行 ° C
- はスライドから余分なバッファーをドレイン。DPS で室温で 30 分間のスライドを軽く浸す 。
- は軽く 70% エタノール室温で 30 分間のスライドを浸します。手順 4 に進みます 。
4。彗星のスライドを染色
- 暗闇の中で 10-15 分の 37 ° C でスライドを乾燥します 。
- 場所 50-100 μ L 緑蛍光核酸染色液にそれぞれ乾燥アガロースと暗闇の中で室温で 15 分間染色します 。
- DH 2 O のスライドを簡単にリンスし、暗闇の中で 37 ° C で完全に乾燥します。画像の収集及び分析に進みます 。
5。画像取得・解析
注: 可視化と DNA 切断の定量化は落射蛍光顕微鏡と彗星の分析ソフトウェアに基づいています (材料の表 を参照してください) 12.
- スライド ホルダー付け顕微鏡でスライドを配置。Agarose のゲルは対物レンズに直面していることを確認します。ランダムに 10 倍の対物レンズを用いた蛍光顕微鏡ステンド グラス彗星スライドから画像をキャプチャします。エッジと気泡の周りの領域を避ける 。
- 各彗星の尾は水平方向に分散することを確認します。彗星のヘッドが左と右から尾に属します 。
- は、暗い背景と明るい DNA 染色バイナリ TIF 形式で各画像を保存します。ソフトウェアを使用してイメージを読み込む、" 分析対象のファイルを選択して " ボタンは、ツールバーの左側にあります。イメージ ビュー ウィンドウ ( 図 1) を表示する必要があります 。
- は、画面上測定フレームを描画し、セルの彗星に従ってそのサイズを調整します。クリックして、" 調整 " 頭、彗星と、画像によると尾のしきい値を設定するボタンをクリック、" 測定を始める " ボタン ( 図 1).
- フレームを使用してセルを選択し、マウスでクリックして、測定をアクティブにする、、" 彗星を試金 " ボタン; 強度画像に現れる、" プロファイル " 選択した測定パラメーターを持つウィンドウ。クリックして結果を保存することができます、" の結果を格納 " ボタン ( 図 1).
注: ソフトウェアは、彗星の尾、尾の DNA、テール モーメント (TM) とオリーブ テール モーメント (OTM) の割合の長さを含むパラメーターを計算します。尾の瞬間、フォローとして数式によって計算される:

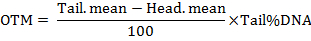
- 、少なくとも 50 セルを分析治療あたり 。
結果
議定書は、彗星の試金実行とデータ分析 (図 1) ステップバイ ステップ ワークフローを説明します。ドキソルビシン処理 U251 細胞 (1 μ M、20 h) の彗星の尾が長く高い DNA 強度、化学療法 (図 2) により断片化された DNA のかなりの蓄積を示唆があったことを示したアルカリ性と中性の彗星の試金からの結果。
ディスカッション
コメット ・ アッセイは、細胞レベルでの単一および二重鎖 DNA 切断を測定するための効率的なツールです。アッセイは、研究については遺伝毒性とバイオモニタ リング13基本病変、DNA クロスリンク、医薬品開発およびアルカリ敏感なサイトに至る「黄金の標準」として広く適用されています。本研究では、我々 はそれぞれアルカリ性と中性の彗星の試金のための 2 つの異?...
開示事項
著者が明らかに何もありません。
謝辞
この研究は、NIH、NCI、CCR の学内研究プログラムによって支えられました。すべての著者は、CCR、NCI、NIH から学内の研究助成金を受け取った。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| 10x PBS(Ca++, Mg++ free) | TEKnova | P0196 | |
| NaCl | Sigma | S5886 | |
| EDTA | TEKnova | E0308 | |
| Trizma base | Sigma | T1503 | |
| NaOH | Sigma | 72068 | |
| Sodium lauryl sarcosinate | Sigma | L7414 | |
| Triton X-100 | Sigma | 93443 | |
| Sodium acetate | Sigma | 32318 | |
| Glacial acetic acid | Sigma | 695092 | |
| Ammonium acetate | Sigma | A1542 | |
| SYBR Green | Invitrogen | S33102 | |
| Low melting point agarose | Invitrogen | 16520 | |
| Agarose | Invitrogen | 16500 | |
| 95% ethanol | WARNER-GRAHAM | #64-17-5 | |
| Trypsin | GIBICO | 25300-054 | |
| Name | Company | Catalog Number | Comments |
| Consumables | |||
| Glass tissue slides | ELECTRON MICROSCOPY SCIENCES | 63422-11 | |
| Kimwipes | KIMberly-Clark | ||
| 1.5 mL Microcentrifuge Tubes | DENVILLE | ||
| Pipette Tips | SHARP | ||
| Name | Company | Catalog Number | Comments |
| Equipments | |||
| Microwave | Avanti | ||
| Waterbath | PRECISION | ||
| Horizontal electrophoresis chamber | TREVIGEN | Cometassay ES II | |
| Power supply | Bio-Rad | ||
| Incubator | Quincy Lab | Model 12-140E | |
| Fluorescent microscope | Zeiss | LSM700 | |
| Micropipettor | Eppendorf |
参考文献
- Ostling, O., Johanson, K. J. Microelectrophoretic study of radiation-induced DNA damages in individual mammalian cells. Biochem Biophys Res Commun. 123 (1), 291-298 (1984).
- Singh, N. P., McCoy, M. T., Tice, R. R., Schneider, E. L. A simple technique for quantitation of low levels of DNA damage in individual cells. Exp Cell Res. 175 (1), 184-191 (1988).
- Tice, R. R., et al. Single cell gel/comet assay: guidelines for in vitro and in vivo genetic toxicology testing. Environ Mol Mutagen. 35 (3), 206-221 (2000).
- Shah, A. J., Lakkad, B. C., Rao, M. V. Genotoxicity in lead treated human lymphocytes evaluated by micronucleus and comet assays. Indian J Exp Biol. 54 (8), 502-508 (2016).
- Azqueta, A., Collins, A. R. The essential comet assay: a comprehensive guide to measuring DNA damage and repair. Arch Toxicol. 87 (6), 949-968 (2013).
- Goldstein, M., Kastan, M. B. The DNA damage response: implications for tumor responses to radiation and chemotherapy. Annu Rev Med. 66, 129-143 (2015).
- Gavande, N. S., et al. DNA repair targeted therapy: The past or future of cancer treatment?. Pharmacol Ther. 160, 65-83 (2016).
- Torgovnick, A., Schumacher, B. DNA repair mechanisms in cancer development and therapy. Front Genet. 6, 157 (2015).
- Weston, V. J., et al. The PARP inhibitor olaparib induces significant killing of ATM-deficient lymphoid tumor cells in vitro and in vivo. Blood. 116 (22), 4578-4587 (2010).
- Brown, J. S., O'Carrigan, B., Jackson, S. P., Yap, T. A. Targeting DNA Repair in Cancer: Beyond PARP Inhibitors. Cancer Discov. 7 (1), 20-37 (2017).
- Lu, Y., et al. Chemosensitivity of IDH1-Mutated Gliomas Due to an Impairment in PARP1-Mediated DNA Repair. Cancer Res. 77 (7), 1709-1718 (2017).
- Konca, K., et al. A cross-platform public domain PC image-analysis program for the comet assay. Mutat Res. 534 (1-2), 15-20 (2003).
- Valverde, M., Rojas, E. Environmental and occupational biomonitoring using the Comet assay. Mutat Res. 681 (1), 93-109 (2009).
- Collins, A. R. The comet assay for DNA damage and repair: principles, applications, and limitations. Mol Biotechnol. 26 (3), 249-261 (2004).
- Karbaschi, M., Cooke, M. S. Novel method for the high-throughput processing of slides for the comet assay. Sci Rep. 4, 7200 (2014).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved