このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ライブの一次繊毛で高速超解像顕微鏡の応用
要約
最近我々 は様々 なタンパク質の一次線毛による生細胞内透過輸送ルートの三次元 (3 D) 空間の場所を割り当てられます。ここで実験のセットアップ、生体試料のプロセスおよびアプローチを新しくイメージング 3 D 超解像蛍光のデータ解析の適用と課題については、一次繊毛を住んでいます。
要約
一次繊毛は、多くの真核細胞の表面に微小管の突出を細胞運動、シグナル伝達タンパク質のユニークな補体の関数を批判的に含みます。繊毛は自分のタンパク質を合成することができないので、ほぼ 200 のユニークな毛様体タンパク質は細胞質と一次繊毛の間取り引きされる必要があります。しかし、それはまだ既存の現在の制限のためライブの一次繊毛のこれらの蛋白質の輸送経路の三次元 (3 D) 場所をマップする技術的な課題のテクニック。挑戦を征服するには、最近開発して、輸送経路のための 3 D の空間的位置を決定するため、シングル ポイント エッジ励起サブ回折 (速度) 顕微鏡と呼ばれる高速仮想 3 D 超解像顕微鏡を採用ゾル性細胞質の両方の生きているセルの一次繊毛膜タンパク質。この記事で、顕微鏡、毛様体タンパク質の蛍光蛋白質標識、ライブの繊毛に個々 の蛋白質の実時間単一分子追跡および達成を発現する細胞の準備の詳細設定がデモンストレーションします。3 D 空間の確率密度に毛様体タンパク質の輸送ルートのマップします。
概要
以来、1873 年にエルンスト ・ アッベが述べたように、従来の光学顕微鏡の分解能は、客観的1,2から光の回折による限られた約 200 nm と考えられています。現在、超解像光学顕微鏡技術は、この制限を破るし、サブ回折 (< 200 nm) 解像度とダイナミックな画像を取得できます。技術は一般に 2 つの広範なカテゴリに分類: サンプル3で fluorophores の非線形光学応答のため、サブの回折照明ボリュームを生成、排出枯渇 (STED) 顕微鏡ベース アプローチを刺激センサーの光学顕微鏡観察 (パーム) と確率論的光再建顕微鏡 (嵐)-ベースの fluorophores の重心をローカライズし、この重心を再構成する数学的な関数を利用した超解像技術超解像画像4,5を形成。現在、比較的単純な光のセットアップのためパームと嵐が広く採用のみ生物学的製剤の長いビデオの各フレームでの同時の小さなサブセットをアクティブにします。これによりより正確な局在化のため 2次元ガウシアンフィッティング蛍光スポットの点拡がり関数 (PSF) と呼ばれる、ビデオの各フレームで蛍光標識したタンパク質の。蛍光標識した分子の 2次元位置は、生物学的準備1,2の超解像画像を生成する単一のイメージング平面上、重ね合わせることが。これらの分子の局在化しながら超解像顕微鏡方法確かに革命をどのように生体試料のイメージングを行った、まだ克服すべき課題があります。たとえば、嵐とパーム生体試料の固定後最高の分解能を達成できしたがって電子顕微鏡のような制限である蛍光標識したタンパク質の静的な表現を提示します。さらに、タンパク質のダイナミクスを捉えることである非常に長いフレーム レートで、生きた細胞の蛍光標識蛋白質ごとの高空間分解能を達成するためにサンプルをイメージする必要があります。したがって、これらの主要な技術的なハードルを克服するために必要です。
生細胞における高速移動蛋白質または Rna の検出に適しています高い時空間分解能を得るためには、私たち研究室 (図 1)6,7,の超解像顕微鏡を開発しました。8. 顕微鏡のいくつかの主要な技術の進歩は正常に小分子の核細胞質輸送を追跡する私たちを有効にしていた、ネイティブ核を介してウイルス mRNA、タンパク質孔複合体 (Npc)6,7,8. Npc など一次繊毛の生細胞におけるサブミクロン回転対称形構造の高分子の高速移動を追跡する簡単に、顕微鏡の次の機能が使用されます: (1) 傾斜または垂直照明 PSF により焦点面 (図 1); 小さな回折限界ボリューム内単一分子の励起(2) 傾斜の PSF は大きくアウト フォーカス蛍光を回避でき、信号対雑音比を向上させる。100-500 kW の光学濃度 (3)/cm2照明 PSF の高速検出速度 (> 500 Hz) で単一 fluorophores が付いてから収集する光子の数千をことができます。(4) 高速検出速度も大幅分子拡散は 1 つの主要な要因の生細胞における蛍光分子の移動の空間軌道を決定する単一分子空間定位エラー (< 10 nm) が減少します。分子の移動の単一分子の局在化の欠陥を引き起こします。(5) 老舗 2D 3d 変換アルゴリズムは、NPC または一次繊毛中分子の輸送ルートの 3 D 空間確率密度マップを提供するために私たちを有効にします。それはデカルトと円筒の調整システムの間の変換処理を使用して、3 D 空間の確率密度 3 D ではなくマップ単一分子追跡 (図 2) を生成することは注目に値する。以前は、電子顕微鏡データでは NPC9,10と一次繊毛11両方回転対称形の構造があることを明らかにしました。原則として、ランダムに拡散分子 NPC または一次繊毛を移動する必要があります回転対称形の分布。図 2に示すように、ランダムにシリンダーの中の分子の拡散の数が多いがおよそ均一の結果さらに NPC の回転対称形分布と断面生成空間2 つの隣接リング (図 2E) 間の非常に小さい各サブ領域内で配布します。この均一な分布を導く円筒における θ 次元空間分布は一定であります。その後、3 D 座標 (R, X, θ) は 2 D 座標 (R, X, 定数) であると簡略化できます。実際には、デカルトと円筒形のシステムの間の変換プロセスは (X, Y) は 2 D から 2 D (R, X, 定数) です。一定の θ図 2電子の空間密度pを指し、 Aの方程式を使用して計算されます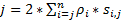 。
。
最終的には、単一分子追跡研究生物学の広範なアプリケーションには、したがって、特定の生物学的ニッチ12,13,14を入力するテクニックの茄多が開発することは当然です。顕微鏡の場合と同様であります。以前は、3 D 変換アルゴリズムと相まって、この手法が通過する Npc、diffraction サイズと回転対称の生物学的構造6による分子の 3 D 輸送ルートを解決する開発されました。本稿で一次繊毛がエクセレント モデル細胞小器官と同様に表示されます。一次繊毛は、プロジェクトのほとんどの哺乳類細胞15,16,17の表面からアンテナのように、円筒の器官 (~ 125 nm radius) です。外部信号を受信し、成長と代謝15,16に通常関連付けられている細胞内の応答を送信に責任があります。したがって、構造タンパク質のフラックス、膜貫通受容体と細胞内メッセンジャーの伝送のリサイクル一次繊毛の重要な責任です。一次繊毛、細胞体の間の連接は転移の地帯または11,18,19を発生する必要がありますこのすべてのタンパク質輸送、TZ と呼ばれる重要な選択性バリア 20。TZ のゲーティング機能に加えて少なくとも 2 つの輸送プロセス, 鞭毛内輸送と受動拡散この地域16,21,蛋白質の動きを担当すると考えられています。22. ヒトの健康の観点から主な毛損失と下流シグナルの後の規制緩和は多くの癌の特性。さらに、バルデ-ビードル症候群や多嚢胞性腎疾患など、多くの遺伝病は、欠陥のある蛋白質輸送23に関連付けられます。サブの回折限界サイズと TZ のたんぱく質の選択的輸送の複雑なプロセスの両方一次繊毛この技術のための主なターゲットを作る。この方法で毛様体膜貫通タンパク質、ソマトスタチン受容体 3 (SSTR3) の追跡を紹介24、Alexa Fluor 647 と IFT、IFT2025、溶かされた GFP の分子とラベルのコンポーネントで外部ラベルします。
プロトコル
1. NIH 3T3 細胞に備えて在庫から顕微鏡
- 1.5 週間実験前に 37 ° C で融解し、細胞を 3 ml のダルベッコ変更されたワシの媒体 (DMEM) 110 mg/ml の 25 cm2細胞培養用フラスコに転送することによって凍結ストックから NIH 3T3 細胞の新鮮な文化を回復します。ピルビン酸ナトリウム、2 mM グルタミン、10% 牛胎児血清 1% ペニシリン/ストレプトマイシン
- 37 ° c 5% CO2インキュベーターでセルを孵化させなさい。
- 細胞周期の均質性を確保するための実験の日の前に、少なくとも 3 回のすべての 2 日間、約 80% の confluency にセルを分割します。37 ° C で 2 分の 0.25% トリプシンで細胞を trypsinize、トリプシンを吸引、培地 2 mL に置き換えます。ピペット繰り返しするに細胞塊を破る目的セル数、メディアの容量をもたらす媒体は、3 mL をバックアップします。
注: NIH 3T3 以前遺伝子組み換えされた NPHP-4、鈍痛、TZ26mCherry に C 末端融合蛋白質を表現します。mCherry は蛍光色素で、定量的 TZ 選択性バリアをローカライズし一次繊毛を方向づける 561 nm 照明と興奮することができます。 - 実験、プレートの前に 2 日間 35 mm ガラス下部にセル 1.1 のステップとして同じ媒体の 1.5 mL で 60-70% の confluency に皿し、インキュベーターにセルを返します。
- 1 日、実験の前に、目的のプラスミドを細胞化学的に transfect します。ミックス 500-1000 目的プラスミドの ng は (下のメモを参照) 30 分吸引メディア、35 から抗生物質がない低下した血清中メディアの 0.25 mL のトランスフェクション試薬と 1:2.5 比 mm ガラス底皿と 0.25 mL プラスミッド交換/トランスフェクション試薬ミックス プラス抗生物質なしの減らされた血清のメディアの余分な 1.25 mL。低下した血清中のメディアは、成功したトランスフェクションを促進する一次繊毛の成長を誘導すると同様、実験を実行するセルを十分な長さで生きている維持の目的を果たします。セルを次の日に実験のためのインキュベーターに戻ります。
注: IFT20 の単一分子追跡を実行すると、gfp の C 末端に融合した遺伝子組み換え IFT20 を含むプラスミドは使用です25。単一分子を実行するとき、SSTR3 の追跡受容体ペプチド (AP) ドメインにその N 末端に融合した遺伝子組み換え SSTR3 を含んでいるプラスミッド、gfp の C 末端は使用される22。SSTR3 構築に加えて共同表されるビオチン リガーゼ ・ ビラを含んでいるプラスミッドとトランスフェクション メディアで 10 μ M のビオチンと補われなければなりません。・ ビラは、ビオチンを小胞体のレベルで新たに合成された AP SSTR3 GFP 分子の AP ドメインにアタッチします。3 つのストレプトアビジン、上 4 つのビオチン結合部位を共役 Alexa647 平均では、可能性があります、補われる蛍光ラベルにセル22,27 の外部表面で AP SSTR3 GFP の分子イメージングの前にメディアに.GFP および AlexaFluor647 はこのメソッドで使用されます。ただし、安定性に優れた写真と量子収率に同様に持っている場合は、他の蛍光プローブを使用できます。 - Alexa647 共役外部ラベル SSTR3 の構造、ガラス底からメディア実験の前に 1 h を皿、リン酸緩衝生理食塩水 (PBS) 1 mL で 5 回セルを洗浄し、1 μ M を添加した低下した血清中メディアの 1 mL を追加削除を使用してかどうかストレプトアビジン。
- 実験する前に 15 分以上ガラス下皿からメディアを削除 5 回 1 mL の PBS と transfected とラベル付きセルを洗浄して.
- ガラス下皿に 1 mL の画像バッファー (20 mM HEPES、110 mM KOAc, 5 mM 2 mM MgOAc、NaOAc 1 mM グリコールエーテルジアミン四酢酸、pH 7.3) を配置します。
注: 画像バッファー内セル、3 h よりは、もはや実行可能です。したがって、実験のみ 2 h は、皿ごとに実行されます。
2. 顕微鏡
注: 速度顕微鏡のセットアップを含む 1.4 NA 100 × 油浸のアポクロマート対物レンズ、35 mW 633 nm He-ne レーザー、50 mW 固体 488 nm および 561 nm レーザ、オンチップ乗算利得を搭載した倒立蛍光顕微鏡電荷結合素子カメラと顕微鏡ソフトウェア パッケージ データ取得と処理 (図 1)。個々 のチャンネル用に、GFP、mCherry、および Alexa647 は 488 で興奮している nm の, 561 nm、または 633 nm のレーザー、それぞれ。単一分子追跡一点照明が個々 の蛍光標識した分子を追跡する使用されます。落射蛍光イメージング、凹面レンズ、照明の均一なフィールドにビームを展開するレーザー照明パスに配置されます。蛍光排出量を同じ目的によって収集された、ダイクロイック フィルター (405、488 561/635) と発光フィルター (405/488/561/635)、によってフィルターされ、単一分子追跡のための 500 Hz、2 Hz で動作上の CCD カメラで撮像落射蛍光イメージング。
- 顕微鏡のステージにガラスの底板を貼る、正しく目的の構造を表現しているセルを探します。適切な細胞が検出されたら、レーザーの単一ポイント照明に対応する画像平面上の場所とプライマリの繊毛の基部に NPHP4 mCherry スポットに合わせます。
- NPHP4 mCherry と IFT20 GFP またはデジタル顕微鏡のソフトウェア パッケージを使用している場合、「フォーカス コントロール」ウィンドウの「カメラ」タブで「スナップ」機能を使用して AP SSTR3 GFP の落射蛍光イメージをキャプチャ (材料の表を参照してください).
注: これらのイメージは、その後単一分子の場所への参照として機能します。 - 参照画像を取得すると、ローカル ラベルの単一分子の濃度を低減します。写真-ブリーチ 20 の 1 mW のレーザー照射で TZ s または蛍光強度は蛍光バック グラウンドの近くまで。
注: とき正確な濃度を制御できる 0.1 1 nM 単一分子をラベルを使用します。 - 単一分子追跡の準備をするには、~0.15 mW の GFP の付いた単一分子または分子の Alexa647 ラベルの付いた ~0.5 mW レーザー照明電力を削減します。
- レーザー パワーと撮像パラメーターを設定すると、すぐに最大利得と発達 2 ms フレーム レート、単一分子イメージングのため適切な照明レーザーを行うし、非 photobleached、運ばれる時、単一分子をラベルを記録「フォーカス コントロール」ウィンドウの「カメラ」タブで「ストリーム」ボタンをクリックして TZ の photobleached 地方。
注: ビデオの 2 分以上は無視できるレベルに毛様体のドリフトの影響を最小限に抑えるために取り込む必要があります。 - 単一分子ビデオをキャプチャした後 (aoi) の各分子の励起包括的な領域で PSF の重心を正確にローカライズ ジェレス研究室によって垣間見ることなどの 2次元ガウシアンフィッティング アルゴリズムを使用して動画を処理します。
- 精度ですべての単一分子の場所を選択して < 10 nm および単一分子場所 2D ガウス機能装備の分布に基づく繊毛のセンターを修正します。
注: 2 D を 3 D 変換アルゴリズムに使用して IFT20 GFP と AP SSTR3 ルートの 3 D 輸送ルート明確に表示されます毛様体鞭毛や繊毛膜、それぞれ。
3. 2D 3 D への変換から
- 繊毛の分子 (信号対雑音比 > 11) を通過する数千ローカライズが収集されると、X の次元として、繊毛の長い軸を選択します。場所の Y 次元ヒストグラムを確認し、10 nm 刻みでビンの合計を取得します。
注: 2 D 3 D への変換からが手または任意のソフトウェアやプログラミング言語によって評価されます。著者は、Matlab と Python 2.7 変換を実装しました。
結果
このセクションは Alexa647 に 〜 15 nm の外部リンカーによって接続されている SSTR3 の輸送ルートを検討する主な繊毛の TZ で顕微鏡を実行から得られたデータを示します (図 3A)。デュアル 3 D 変換アルゴリズム検証の目的を提供しています。Alexa647 だけで一次繊毛としたがって、3 D の輸送ルートの外部表面のラベルする必要があります...
ディスカッション
このプロトコルでは、顕微鏡の一次繊毛、効率的なタンパク質輸送に大きく依存して細胞シグナリング オルガネラへの応用について説明します。顕微鏡に、TZ の中心一点照明を通過するときは、蛍光標識した分子 (< 10 nm) 場所高解像度を提供できます。以前それは NPC6,7,8を通じて人身売買蛋白質を調査に適用されています...
開示事項
著者は利益相反を宣言しません。
謝辞
いくつかのプラスミドの提供ありがとう博士クリステン四重奏団、他 (アナーバー、ミシガン州大学)、博士グレゴリー Pazour (マサチューセッツ大学医学部)。プロジェクトは、国立衛生研究所 (NIH の GM097037、GM116204、大学院に GM122552) からの助成金によって支えられました。
資料
| Name | Company | Catalog Number | Comments |
| 25 cm2 tissue culture dish | Corning | VV-01936-00 | |
| Penicillin/streptomycin | ThermoFisher | 15140122 | |
| Fetal bovine serum | ThermoFisher | 10438018 | |
| DMEM | ThermoFisher | 10566-016 | |
| OPTIMEM | ThermoFisher | 31985062 | |
| Trypsin | ThermoFisher | 25300054 | |
| Phosphate buffered saline | Sigma-Aldrich | P3813-1PAK | |
| Transit LT1 | Mirus | MIR 2300 | |
| 35 mm glass bottom dish | MatTek | P35GCOL-0-14-C | |
| AlexaFluor 647-conjugated streptavidin | ThermoFisher | S21374 | |
| Biotin | Sigma-Aldrich | B4501-100MG | |
| 633 nm He-Ne laser | Melles Griot | 25-LHP-928-249 | |
| 561 nm solid state laser | Coherent | OBIS 561-50 LS | |
| 488 nm solid state laser | Coherent | 1185053 | |
| Inverted fluorescence microscope | Olympus | IX81 | |
| 1.4-NA 100× oil-immersion apochromatic objective | Olympus | UPLSAPO 100× | |
| On-chip multiplication gain charge-coupled-device camera | Roper Scientific | Cascade 128+ | |
| Dichroic filter | Semrock | Di01- R405/488/561/635-25x36 | |
| Emission filter | Semrock | NF01-405/488/561/635-25X5.0 | |
| Slidebook 6.0 | Intelligent Imaging Innovations | digital microscopy software |
参考文献
- Huang, B., Bates, M., Zhuang, X. Super-resolution fluorescence microscopy. Annu Rev Biochem. 78, 993-1016 (2009).
- Leung, B. O., Chou, K. C. Review of super-resolution fluorescence microscopy for biology. Appl Spectrosc. 65, 967-980 (2011).
- Willig, K. I., Rizzoli, S. O., Westphal, V., Jahn, R., Hell, S. W. STED microscopy reveals that synaptotagmin remains clustered after synaptic vesicle exocytosis. Nature. 440, 935-939 (2006).
- Betzig, E., et al. Imaging intracellular fluorescent proteins at nanometer resolution. Science. 313, 1642-1645 (2006).
- Rust, M. J., Bates, M., Zhuang, X. Sub-diffraction-limit imaging by stochastic optical reconstruction microscopy (STORM). Nat Meth. 3, 793-796 (2006).
- Ma, J., Yang, W. Three-dimensional distribution of transient interactions in the nuclear pore complex obtained from single-molecule snapshots. Proc Natl Acad Sci USA. 107, 7305-7310 (2010).
- Ma, J., Goryaynov, A., Sarma, A., Yang, W. Self-regulated viscous channel in the nuclear pore complex. Proc Natl Acad Sci USA. 109, 7326-7331 (2012).
- Ma, J., et al. High-resolution three-dimensional mapping of mRNA export through the nuclear pore. Nat Comm. 4, (2013).
- Akey, C. W., Radermacher, M. Architecture of the Xenopus nuclear pore complex revealed by three-dimensional cryo-electron microscopy. J Cell Biol. 122, 1-19 (1993).
- Akey, C. W. Interactions and structure of the nuclear pore complex revealed by cryo-electron microscopy. J Cell Biol. 109, 955-970 (1989).
- Czarnecki, P. G., Shah, J. V. The ciliary transition zone: from morphology and molecules to medicine. Trends Cell Biol. 22, 201-210 (2012).
- Elf, J., Li, G. -. W., Xie, X. S. Probing transcription factor dynamics at the single-molecule level in a living cell. Science. 316, 1191-1194 (2007).
- Anzalone, A., Annibale, P., Gratton, E. 3D orbital tracking in a modified two-photon microscope: an application to the tracking of intracellular vesicles. J Vis Exp. , (2014).
- Ritter, J. G., Veith, R., Veenendaal, A., Siebrasse, J. P., Kubitscheck, U. Light sheet microscopy for single molecule tracking in living tissue. PloS one. 5, 11639 (2010).
- Marshall, W. F., Nonaka, S. Cilia: tuning in to the cell's antenna. Curr Biol. 16, 604-614 (2006).
- Scholey, J. M., Anderson, K. V. Intraflagellar transport and cilium-based signaling. Cell. 125, 439-442 (2006).
- Yang, T. T., et al. Superresolution pattern recognition reveals the architectural map of the ciliary transition zone. Sci Rep. 5, 14096 (2015).
- Craige, B., et al. CEP290 tethers flagellar transition zone microtubules to the membrane and regulates flagellar protein content. J Cell Biol. 190, 927-940 (2010).
- Kee, H. L., et al. A size-exclusion permeability barrier and nucleoporins characterize a ciliary pore complex that regulates transport into cilia. Nat Cell Biol. 14, 431-437 (2012).
- Najafi, M., Maza, N. A., Calvert, P. D. Steric volume exclusion sets soluble protein concentrations in photoreceptor sensory cilia. Proc Natl Acad Sci USA. 109, 203-208 (2012).
- Nachury, M. V., Seeley, E. S., Jin, H. Trafficking to the ciliary membrane: how to get across the periciliary diffusion barrier. Annu Rev Cell Dev Biol. 26, 59-87 (2010).
- Ye, F., et al. Single molecule imaging reveals a major role for diffusion in the exploration of ciliary space by signaling receptors. Elife. 2, 00654 (2013).
- Ross, A. J., et al. Disruption of Bardet-Biedl syndrome ciliary proteins perturbs planar cell polarity in vertebrates. Nat Genetics. 37, 1135-1140 (2005).
- Handel, M., et al. Selective targeting of somatostatin receptor 3 to neuronal cilia. Neuroscience. 89, 909-926 (1999).
- Follit, J. A., Tuft, R. A., Fogarty, K. E., Pazour, G. J. The intraflagellar transport protein IFT20 is associated with the Golgi complex and is required for cilia assembly. Mol Biol Cell. 17, 3781-3792 (2006).
- Awata, J., et al. NPHP4 controls ciliary trafficking of membrane proteins and large soluble proteins at the transition zone. J Cell Sci. 127, 4714-4727 (2014).
- Howarth, M., Ting, A. Y. Imaging proteins in live mammalian cells with biotin ligase and monovalent streptavidin. Nat Protoc. 3, 534-545 (2008).
- Huang, B., Wang, W., Bates, M., Zhuang, X. Three-dimensional super-resolution imaging by stochastic optical reconstruction microscopy. Science. 319, 810-813 (2008).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved