Method Article
血液ベース ROS1 の検出のためのテストし、デジタル ポリメラーゼ連鎖反応を使用してリボ核酸を循環から融合の成績証明書を RET
要約
リボ核酸 (cRNA) 血液からを循環の検出は、臨床診断で満たされていない必要性です。ここで我々 は非小細胞肺癌患者特異的かつ高感度デジタル ポリメラーゼ連鎖反応を使用してから cRNA を特徴付ける方法を説明します。テストは、72 時間以内に融合の亜種を検出する設計要件を満たします。
要約
単離と腫瘍由来循環リボ核酸 (cRNA) 血液ベース液生検のための解析手法を開発しました。血液から回復 cRNA の堅牢な検出は、臨床診断の重要なニーズにソリューションを表します。テストは cRNA を安定させる防腐剤を含む採血管に全血のコレクションに始まります。このテスト システムでは、無料の携帯、exosomal、および血小板関連 RNA をプラズマから分離されます。CRNA は逆相補的 DNA (cDNA) を転写、デジタルのポリメラーゼの連鎖反応 (dPCR) を使用して増幅されます。サンプル制御遺伝子と同様、両方のターゲット マーカーが評価されます。テストの検証には、検出、精度、および堅牢性の研究分析サンプルの制限が含まれています。再現性をもってこれらの研究の結果開発した方法は、ROS1 の融合に複数のバリアントを検出 (C Ros イヌ 1; 8 亜種) と RET (トランスフェクション イヌ; 中に再配置 8 亜種)。サンプル処理ワークフローは、テストの結果は、サンプル受領後 72 時間以内に一貫して生成できるように最適化されています。
概要
上は非小細胞肺癌 (NSCLC) の 25% に患者には、十分な組織診断の時にテストできるがありません。組織が利用可能な場合でもそれが十分の量や品質を実行するは推奨されません分子テスト1,2。において分子プロファイリングの生検から十分な組織、患者の結果、数週間以上待ってまたは分子の結果3,4せずに治療を開始する必要があります。ただし、有益な分子診断は、非小細胞肺癌と診断された患者の複数ターゲットを絞った治療法の選択肢の出現を与えられた利用できるが重要です。液体の生検から循環細胞遊離 DNA (cfDNA) のテストは、テスト4,5,6伝統的な組織の課題への解決策です。CfDNA と似たような dPCR ベースのワークフローを使用して、迅速な結果の生成のための非小細胞肺癌における実用的な変異の電流試験オプションを含める変異 ΔE746 A750、L858R、egfr 遺伝子の耐性変異 T790M は感作性上皮成長因子受容体 (EGFR)、KRAS 癌 (KRA) は、バリアントと B Raf イヌ (BRAF) バリアント V600E。として広く採用されるに至らなかったフィールドによって、循環腫瘍由来メッセンジャー RNA (mRNA) 液体の生検から分離されたことと、重要な臨床情報7,8,9もできます。以前開発して棘皮動物微小管関連タンパク質のような 4 未分化リンパ腫受容体チロシンキナーゼ (EML4 ALK) 融合の亜種の血漿10から多重検出の方法で報告します。本研究で我々 は高次多重 RNA ターゲット、ROS1 と RET、各アッセイ内 8 つの融合のバリエーションをカバーするこれらのメソッドを拡張しました。目的は、以前非小細胞肺癌と診断された患者の血漿からこれらの融合の亜種を検出するための迅速な敏感な具体的で、再現性のある手法を開発することでした。
テスト プロセスは、医師のオフィスの血のコレクションの管の11を安定させる RNA を使用して開始されます。これらの管には、セル RNase 阻害剤、防腐剤が含まれています。サンプルは、一元的なカレッジのアメリカの病理学者キャップ認定/臨床実験室の改善改正 (CLIA) に一晩出荷優先順位-有能な人材による処理のための認定研究所 (臨床検査)。臨床検査によって受信されると、処理の各ステップは承認された標準操作手順書 (SOP) の下で行われています。循環、RNA の分離に使用される、プラズマを回復する全血を遠心血液中またはエクソソームと血小板の7,8,9などの部分をカプセル化する内無料。これらのコンパートメントからの RNA を隔離するには、いくつかの抽出方法の比較に基づいて RNA 回復のシステムを選択しました。隔離された RNA は集中と逆 cDNA に転写します。ROS1 と RET ターゲット トラン スクリプト変換10を最大化する cDNA の合成方法の最適化中にいくつかの逆転写酵素や遺伝子特定のプライマーを行った。これは低豊富な腫瘍由来の融合の亜種などの成績証明書を循環にとって重要です。最後に、我々 は RET や ROS1 融合変種の制御遺伝子、β-グルクロニダーゼ (GUSB) 多重検出を実現する dPCR のプライマーとプローブの濃度を最適化されています。私たちは、このレポートに記載されている分析検証研究を実行する前に最終的なロック プロトコルに最適化スタディのそれぞれから最高の条件を組み合わせます。このプロトコルとこれらの結果は、循環で稀な融合のルーチンの検出のための迅速・高感度なワークフローの基礎を提供します。
プロトコル
明記しない限り、以下の試薬メーカーの指示に従います。PCR の試金は、ROS1 と RET 融合を検出する市販の製品です。
1. RNA の逆のトランスクリプション (RT) のための準備と作業-dPCR: 研究室のベスト プラクティス
- RNA での作業時は、RNase フリーの環境を作成します。
- 市販スプレー汚染 RNases を不活化するように設計を使用します。
- 使用は、RNase フリー試薬、ヒント、およびチューブを認定しました。RNases の導入またはサンプルのクロスコンタミネーションを防ぐために障壁ピペッターに調整するためのヒントを使用します。
- 常にあなたのサンプルに服から落ちてから粉じんを防ぐために白衣を着用します。研究室コート特定の RNA の処理で使用するを指定します。
- 皮膚の RNases からのサンプルの汚染を防ぐために手袋を着用します。手袋を頻繁に変更します。
注:研究所表面は RNase で汚染されて以来、彼らは環境にさらされていると仮定します。肌、髪、ドアノブ、冷凍庫ハンドル、ペン/マーカー等にお問い合わせください手袋は、もはや RNase フリーになると見なされます。 - 使用する RNase 不活化スプレー前にピペッターに調整、benchtops、遠心分離機、および他の作業面を消毒します。
- 可能であれば、RNA を使用のみのための機器のセットを維持します。
- 粉じん試料に落下または作業エリアの汚染を防ぐために RNA のサンプルを使用する場合は、ラボではエリア内の空気の流れの中断を最小限に抑えます。
- ストアは、-80 ° C に RNA を精製しました。
- これにより低下することができます複数の RNA のサンプル、凍結解凍の繰り返しは避けてください。
2. ポジティブ コントロールの分析、RNA 素材の生成
- 利息10の融合のバリエーションに対して公開された mRNA シーケンスを用いた合成 DNA を設計します。
- 特定の融合のバリアントのための融合サイトと並ぶ PCR 増幅をカバーするためのそれぞれの側に十分な長さを含む融合の mrna を選択します。
- 濃縮した血小板血漿を使用してキャプチャした RNA を循環のサイズを模倣する 50-250 nt 間の塩基配列を選択します。
- T7 プロモーターを追加 (5'-CAGAGATGCATAATACGACTCACTATAGGGAGA-3') 5' ターゲット シーケンスの末尾に。
- フラグメントの二重鎖デオキシリボ核酸 (DNA) 合成シーケンスを注文します。
- 10 ng/μ L の最終的な集中するトリス-EDTA (TE) バッファーの合成 DNA を再構築します。
- 60 ng 合成 DNA を RNA生体外のトランスクリプションを使用してに変換します。
- フェノール/グアニジン系試薬12を使用して RNA 転写産物を浄化します。
- DNase が含まれて私は、RNase フリーの残留テンプレート DNA を削除します。
- 精製の in vitro RNA 特定の染料と標準市販蛍光光度計を使用して RNA 濃度を測定します。選択した基準の許容範囲内で RNA を確認します。希釈が必要な可能性があります。
- RNA のゲルの染色と高範囲 50-250 nt サイズ範囲を含む RNA はしご混合 2% アガロースゲルを用いたゲル電気泳動の成功に転写を確認します。
- それぞれの in vitro RNA ゲルにロード 500 ng。
- 5 V/cm でゲルを実行します。
- 照明を使用して単一のバンドを視覚化し、結果を文書化します。
- (2.1.2 の手順で設計に基づく) 融合バリエーションごとに予想される転写サイズを確認します。
- 各の in vitro RNA by RT dPCR 一致したバリエーション固有で PCR の試金を使用しての検出を確認 (5-8 このプロトコルの手順を参照)。
- オプション: 各融合亜種と制御遺伝子 GUSB 含む体外RNA の等モル混合物を準備します。
- 2.9 ステップが実行された場合: 各バリエーション固有で PCR の試金 (このプロトコルの手順 5-8 を参照してください) を使用して dPCR によって制御混合物に含まれる融合の亜種の検出を確認します。
- 0.25 から 2.5 fg10まで濃度をテストすることによって分析ポジティブ コントロールに必要な入力濃度を決定します。必要なコピー数の出力に基づいて濃度を選択します。
- 次の確認、肯定的な制御 (手順 4.4) と-80 ° C でストアで使用するため分析 RNA の 10 μ L の単回使用因数を準備します。
3. ドナーの標本
- 携帯無料 RNA 防腐剤を含む 10 mL 採血管 (BCT) の 10 mL ひと全血検体を収集します。
注:研究使用を了承するものとすべての人間のドナーとドナー固有識別情報を収集またはテスト中に使用しなければなりません。 - BCT の製造元によって指定された時間枠内で全血サンプルを処理します。
- プールの正常ひと血漿は、分析のポジティブ コントロール内で使用する商業ソースから購入できます。肯定的な制御 (手順 4.4) で使用する単回使用、プールされた正常ひと血漿、-80 ° C でストアの 1 mL 因数を準備します。
4. プラズマからの RNA を循環の回復
注: この手順中にすぐに動作するように重要です。
- 20 分間 200 x g で全血チューブを遠心します。
- 血清ピペットを使用して遠心採血管から血漿の 4 mL までを収集します。邪魔やバフィー コート層を吸引しないように注意します。
- エクソソーム、血小板、プラズマから RNA を細胞に取り込むことができます市販のキットを使用して循環の RNA を分離します。各バッチと一緒に肯定的な制御サンプルからの RNA を隔離します。
-
臨床サンプルの各バッチに対して肯定的な制御を次のとおり準備します。
- プール 1 mL 通常血中因数 (ステップ 3.3) を解凍します。
- 10 μ L 分析 RNA 因数 (手順 2.12) を解凍します。
- ライセートの血漿にエタノールを追加したら、正常ひと血漿サンプルに 10 μ L 分析 RNA を追加することによって肯定的な制御を準備します。
- 100 μ L ヌクレアーゼ フリー水サンプルを溶出します。すぐに RNA をクリーンアップ続行と濃度。
- サンプルをぬれた氷の上格納され、覆われて、1 時間があります。
- RNase フリー水の 9 μ L の溶出し、列ベースのメソッドを使用して RNA を集中します。
- 手順 5 に進んでくださいまたは 1 時間濡れた氷のサンプルを保ちます。
5 cDNA に RNA の転写を逆します。
- 集中循環 RNA サンプルを cDNA ランダムプライマー (コンポーネントの表 1を参照) を含む市販の逆転写反応キットを使用してに変換します。
注:遺伝子特異的プライマーは省略可能であり、テスト バリエーションを設計できます。プライマーは、ターゲット RNA シーケンスに基づいて設計されています。ステップ 2.1 から融合バリアント配列を使用します。- コントロールのない逆転写酵素のサンプル コントロールのない RNA のサンプルなど (表 1参照)。
- 市販回転カラム濃縮 DNA を使用して逆転写反応から cDNA を分離します。
注:この手順は、無料 deoxynucleotide 三リン酸塩 (dNTPs)、プライマー、酵素の除去を促進します。 - PCR の反作用ですぐに cDNA を使用または-80 ° C で保存します。
6. デジタル PCR
注: この PCR は液滴特定デジタル PCR (を参照してくださいテーブルの材料)。
-
PCR ミックス注意。
- 使い捨て可能な実験室のコートとニトリル手袋を着用します。
- PCR の使用専用試薬の準備領域で試薬を混ぜます。試薬のみの準備エリアに cDNA を処理しません。
- 光からそれらを保護するために働いている間プローブをカバーします。過剰な光することができます写真のプローブに接続されている蛍光色素を漂白剤します。
- カバー ミックスの輸送し、光から保護、cDNA の前に別の中古増幅領域に追加することです。
- 追加テストする cDNA PCR クリーン フードに PCR ミックス中古増幅エリアに位置します。
- 最終反応体積表 2に従って 20 μ L の PCR ミックスを準備します。
- PCR ミックス + PCR プレートに cDNA を配布します。
注:板レイアウトの使用ガイドをお勧めします。 - 取り外し可能なプレート シーラーを使用してプレートをカバーします。
- 井戸の下部にサンプルを収集するために簡単にプレートを遠心します。
- 10 の低設定にプレート シェーカーでミックス s。
- 井戸の底でサンプルを収集するために簡単にプレートを遠心します。
- プレート シーラーを削除します。手動または自動の液滴生成システムどちらかと PCR cDNA ミックスの液滴生成を実行します。
- 手動液滴生成の液滴生成のカートリッジのサンプル井戸に 20 μ L の PCR ミックスを転送します。液滴生成油の 70 μ L を追加します。ゴムのガスケットと転送カートリッジ液滴生成を開始する手動液滴形成配列装置でカバーします。以下の液滴生成液滴をメーカーの推奨のヒントを使用して新鮮な PCR プレートに転送します。アスピレーション及び 6 に 5 滴をゆっくり、ディスペンス s 液滴カートリッジまたは板の先端の開口部に触れることがなく、それぞれ。
- 自動化された液滴生成のためホイルシールと液滴形成配列装置へ転送プレートをシールします。すべてのヒントは、カートリッジを確保して液滴生成を開始する前に、場所のプレート。
- 以下の液滴生成と新鮮な PCR プレートへの液滴の転送シール箔板シーラーと熱サイクル板と表 3の設定を使用します。
- サーマルサイクラー、実行が完了した後、液滴のリーダーを使用してプレートを読みます。リーダー ソフトウェア コントロール、サンプルなどの場所を識別するためのプレート レイアウトを作成して読み取りを開始するソフトウェアにロードします。
7. データ分析、レビュー、および結果の生成
- 市販のソフトウェアを使用して、結果を読むプレートを分析します。
- 二次元 (2 D) 振幅プロットを表示するには、[分析] メニューに移動します。
- 液滴のデータを調べることによって、データの全体的な品質を評価します。
- [イベント] メニューを使用して、承認済みイベントの総数数字データを評価します。ウェルあたり未満 10,000 のイベントが存在する場合追加問題のデータを慎重に評価します。
- 異常な蛍光振幅についてデータを確認します。振幅が大きく違いとレプリケート サンプルの濃度の違いは、貧しい処理またはサンプルの混合を示します。
- 質の悪い水滴や問題のサンプルを示す 45 度軸のスプレー パターンを持つ液滴クラスターのノートを作る。
- 最初肯定的な制御、No 逆転写酵素 (いいえ RT)、およびなし RNA 制御 (NRC) のデータを調べます。すべてのコントロールのサンプルを選択し、2 D プロットによってクラスターの品質を調べます。適切な閾値の液滴クラスター間の明確な分離が明らかになる必要があります。
- アッセイ バリエーションごとにコントロールの井戸に基づくしきい値を設定します。
- 場合制御遺伝子の人口 (5'-hexachloro-フルオレセイン-CE ホスホロアミダイト プローブのラベル)、y 軸、およびバリアントの遺伝子人口から二重否定液滴人口を分離する十字ツールを使用して 2D プロットに設定された閾値を提示 (ラベルの付いたフルオレセイン amidite OR 6 carboxyfluorescein プローブ)、x 軸。
- 合計は、1 つのサンプルの複製もからコピーします。
- 検出されたバリアントのコピーの数とテスト結果を表現します。
注:通常の正または負のサンプルを呼び出すため分析のカットオフ値を決定を実行するには、健康なドナー サンプル確定プロセスを (少なくとも 10 の個々 のサンプル) を設定しての突然変異のための任意の検出可能なバック グラウンド信号上のカットオフを確立します。関心します。また、正または負の結果を呼び出すために必要な制御遺伝子コピー数を確立します。この制御遺伝子のカットオフは、内部の品質管理 (QC) が処理される各 RNA サンプルの質と量を評価するためとして機能します。
8. (オプション) のセルラインを使用して RT dPCR 反応条件の検証
- 融合の亜種の検出を確認するには、ROS1 または RET 融合を表現する市販のセルラインを使用して興味の mRNA。次の手順します。
- 冷凍状態から直接換散のためベース ソリューションでフラッシュ凍結細胞をホモジナイズしてください。均質化の前にも簡単な融解 RNA の劣化と損失を引き起こすことができます。
- RNA 用に設計されたシリカ膜のスピンの列を使用して RNA を分離します。
- RNA 特定試薬と基準、蛍光光度計を用いた RNA 試料の濃度を測定します。
- プラズマまたは別の商業源からの野生型 RNA の背景に隔離された RNA を希釈します。
- CDNA、デジタル PCR とデータ分析、レビュー、RNA の転写の逆から手順を実施し、結果の生成目的のバリアントの検出を確認するこのプロトコルに記載されています。
結果
このプロトコルでは、非小細胞肺癌患者 (図 1A)、プラズマ内でドライバー変異の測定で使用するための RNA 融合亜種の検出のために開発された試験システムについて説明します。融合 mRNA 非小細胞肺癌の人口における最も一般的な RET と ROS1 再編成の式からの製品には、13,14,15,16,17が識別されます。多重 PCR の試金は単一の反作用の内で非小細胞肺癌のターゲットごとに 8 つの最も一般的なトラン スクリプトのバリエーションを検出する、設計されていた。ROS1軌跡で最も一般的な転座CD74、 SDC4、 SLC34A2、 EZRやTPM3遺伝子 (図 1B) の 5' 部分とのアソシエーションを生成します。RET遺伝子座で最も一般的な転座は、アッセイが六つのエキソン接合をカバー KIF5B並置に します。覆われている追加のRETパートナーには、これらのCCDC6とTRIM33 (図 1C) が含まれます。合計では、法は、約 88% をカバー ROS1 の非小細胞肺癌患者人口17で発生すること知られている RET の変異の 99%。
分析コンポーネントの特異性は最初 ROS1 または RET 融合成績多重アッセイの mRNA シーケンスを含む 8 つの個々の in vitro Rna を使用して評価されます。各 RNA 種は、各多重バージョンを構成する個々 のバリアント アッセイに対してテストされました。これらのアッセイの交差反応性はなく、デモは 100% 設計内解析的特異性多重アッセイ (データは示されていない)。テスト プロトコルの検出下限値を決定するには、アッセイに含まれる融合バリアントを表現する細胞株由来の総 RNA は 5%、1%、0.2%、0.04% の濃度で通常の RNA の背景に混合しました。多重 RET と ROS1 バリアント PCR の試金 0.2% 融合バリアント (図 2A B) 同様に少しを検出しました。さらに、5% の準備オフのターゲット細胞株 (EML4 ALK 融合転写産物発現) 派生 RNA 検出されなかった多重 ROS1 と RET の分析でさらに特異性 (図 2A B) を示します。
両方 ROS1 RT dPCR プロセスの精密な品質検査を行ったし、等モルの in vitro Rna から成る中国と分析制御材料 (高、中、低) の 3 つの濃度で逆のトランスクリプションおよび 3 dPCR 経由で処理されました。同じ日 (日中)、2 つの演算子 (間演算子) と連続 3 日間 (間日) 内で別の機会に。精度のテストからの結果は、関心の融合転写制御遺伝子、GUSB 内部の品質管理指標 (図 2-D) として含まれている両方の正確な検出を示した。
GUSB 内部統制に加えて臨床サンプルの各バッチはバッチ コントロールのセットが実行されました。肯定的な制御には、GUSB のため、各 RT-dPCR でテスト融合バリエーションを表す RNA として分析の in vitro RNA の解析体外の混合物から開発されました。この RNA は正常ひと血漿ライセート中に RNA の抽出にスパイクだったし、プロトコルを通して臨床サンプルと一緒に処理されました。制御なしの逆転写酵素 (RT なし) は、RNA 抽出ワークフローにおける汚染物質の有無を確認し、RNA のプライマーの特異性を示す陰性対照だった。いいえ RT 制御は、肯定的な制御と同じ素材を使用して生成されましたが、cDNA 合成反応内酵素は含まれません。ないの RNA 制御 (NRC) は、逆転写反応コンポーネントの転写物の汚染の有無を確認する否定的なコントロールです。このコントロールが導入された cDNA 合成段階でワークフローと RNA テンプレートではなく反応で水が追加されました。なし RT と NRC のコントロールは正確な結果が配信される場合、両方のチャネルに負である必要があります。表 1は、各コントロールの逆転写反応コンポーネントを示します。ROS1 のこれらのコントロールのそれぞれの 2 D プロットの例が表示されます (図 3 A ~ C) と RET (図 3 E G) マルチプレックスアッセイ。融合亜種フルオレセイン amidite (FAM) プローブを用いて検出された GUSB、制御遺伝子の 5'-hexachloro-フルオレセイン-CE ホスホロアミダイト (HEX) プローブを用いた検出された間、y 軸沿いに表され、x 軸上です。これらのバッチ コントロールは、アッセイの堅牢性を決定するため 21 日間にわたって評価されました。核融合肯定的な液滴と GUSB 制御遺伝子液滴 ROS1 と RET の認められた (図 3D, H) 研究のコース上で実行すべての 21 の実行.すべての否定的なコントロール (いいえ RT および NRC) は、全体の 21 日 (データは示されていない) の間で否定的な結果をもたらした。
トラブルシューティングを行う能力は、検査設定で実行する任意のテスト プロトコルの重要なコンポーネントです。ここでは、我々 は RT dPCR プロトコルを使用してサブ最適な結果の実世界の例を提供します。最初のない逆転写酵素コントロール (図 4A) の重要性を示す例 2 D プロットです。この例では、酵素の不足により cDNA 変換はありませんでしたにもかかわらず肯定的な突然変異体の液滴が存在しました。この結果はターゲットからゲノム DNA を増幅 dPCR プライマーによる可能性が高かった。この例では、イントロンにまたがるアッセイのデザインは、ゲノム DNA の増幅を防ぐが。また、RNase フリーの DNase 酵素は汚染の DNA を除去するために使用できますが、これは推奨されませんまれなターゲットの検出のため、いくつかの RNA 分解酵素と潜伏中に発生します。次の使用例 2 D プロットは両方のチャネル (図 4B) に肯定的な液滴と NRC だったこれには、RT dPCR セットアップでいくつかの時点で汚染が示されます。この場合は、テストで使用される可能性のある汚染された試薬を破棄、徹底的にすべての機器を消毒して新鮮な反応コンポーネントを再テストをお勧めします。3 番目の例 2 D プロット (図 4C) 45 ° 線に沿って液滴のスプレーとして提示します。これは多くの場合せん断と液滴の合体が原因です。液滴は損傷しやすく、慎重な液滴の熱サイクルにかける前の処理が不可欠です。自動化された液滴生成の使用をお勧めしますが利用可能。手動で転送すると水滴が生成された場合する推奨ワイドボア ヒントを選択し、慎重な分注技術を採用します。溶滴移行遅い吸引と各撮影場所 5-6 秒以上と調剤を必要と、それはピペット先端開口は液滴カートリッジを触れたり、よくない不可欠です。調剤、液体レベルでピペット チップを保つし、液滴ディスペンサー (ビューのデモのビデオ) は、それをゆっくりと上げます。最終的な 2 D プロット例を示します (図 4) の正と負の液滴集団間の分離の欠如。これは、いくつかの原因があります。換散バッファーの高い劣化 DNA の過剰使用される洗剤など強い PCR 阻害剤には、分離の損失が生じます。この場合、cDNA を合成して dPCR (ようこのプロトコルの手順 5 で記述されている) のクリーンアップ手順を追加することを検討してください。最後に、サブ最適な増幅条件のために分離の欠如することができ、PCR のステップの最適化も考慮する必要があります。
図 5を表す 984 現実世界患者内のデータは、ターンアラウンド時間のサンプルし、このテストのワークフローの迅速な性質を示します。結果は、48 時間以内 (79% 例) 領収書のサンプルと 95% の場合、72 時間以内として、早ければ治療医師に報告されました。結論としての使用の急速なテスト システムを提供できる RNA 採血管、血と RT dPCR 内部適切な最適化されたプロトコルおよびバッチ コントロールに従って実行から最適化された RNA 抽出手順を循環を安定化融合 RNA 亜種非小細胞肺癌に関連する高精度で検出。
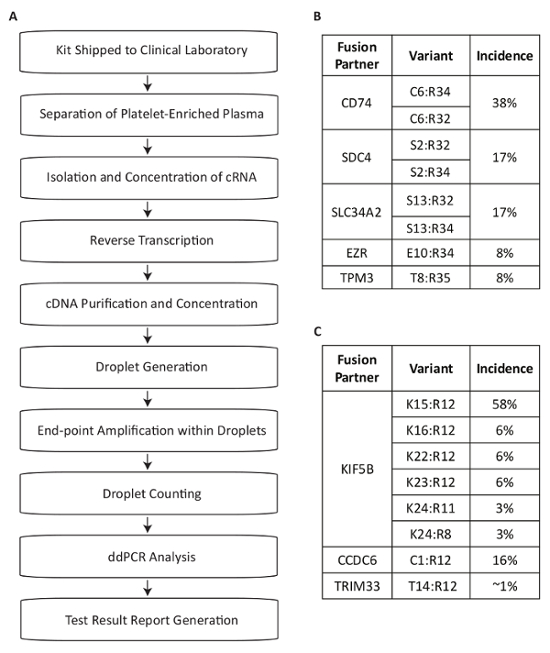
図 1: 非小細胞肺癌で最も普及している RET と ROS1 亜種にアッセイ固有を使用して融合バリアント検出のための血液サンプル処理手順の概要です。(A) 全血が描画され、臨床検査室に標本コレクション キット内で、BCT を出荷時にサンプル テストを開始します。RNA の循環を回復濃縮した血小板血漿内の複数のソースから逆遺伝子特定プライミングと転写の dPCR で使用するため精製で。サンプルは、液滴生成 (乳剤) から成っている市販のシステムを使用して処理される増幅、および液滴を数えます。市販のソフトウェアを使用してデータを分析します。テストの結果は文書化し、テストを要求する医師に報告します。プロセスは、結果リリースに領収書のサンプルから 72 時間の期間内に動作するように設計されています。ROS1 (B) と (C) RET の 8 つのバリエーションは、多重アッセイ内で覆われています。許可を得て Biodesix ウェブサイトから適応。この図の拡大版を表示するのにはここをクリックしてください。
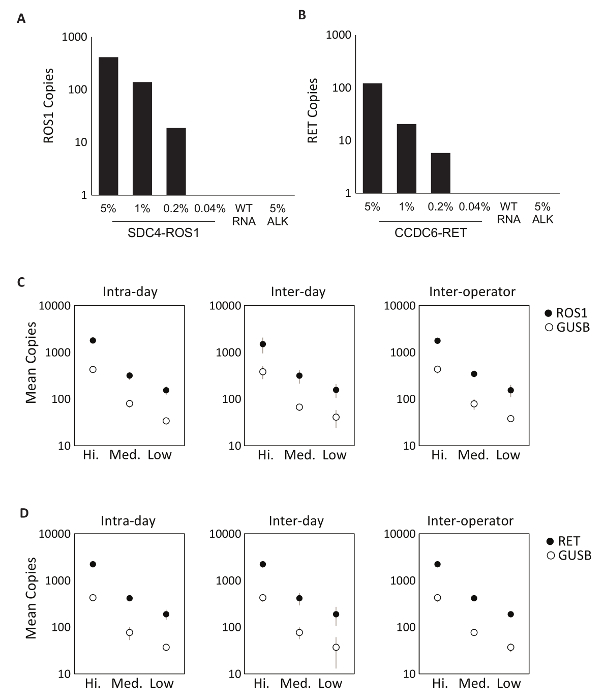
図 2: 分析検証します。表現する細胞 (A) (B) CCDC6 RET 融合と SDC4 ROS1 融合総人間野生型 RNA (WT RNA) のバック グラウンドで希釈されました。各融合バリアント検出限界は 0.2% 周波数各バリアントの試金のための定義済みの条件を使用してに設立されました。このしきい値を超えるすべてのサンプルには、制御遺伝子の少なくとも 21 のコピーも含まれています。野生型 RNA のバック グラウンドで 5% EML4 ALK (アルク) 標準は、否定的な結果によって確認されたアッセイの特異性を示すテストされました。分析の多重 RNA 基準高、中で測定し、ROS1 (C) と (D) 中国と精度の低濃度が同じ日 (日中) に 3 失点、3 日間連続 (1 日間) と 3 つの動作を評価し2 つの独立した演算子 (間演算子)。コピー数と標準偏差の意味が表示されます。許可を得て Biodesix ウェブサイトから適応。この図の拡大版を表示するのにはここをクリックしてください。
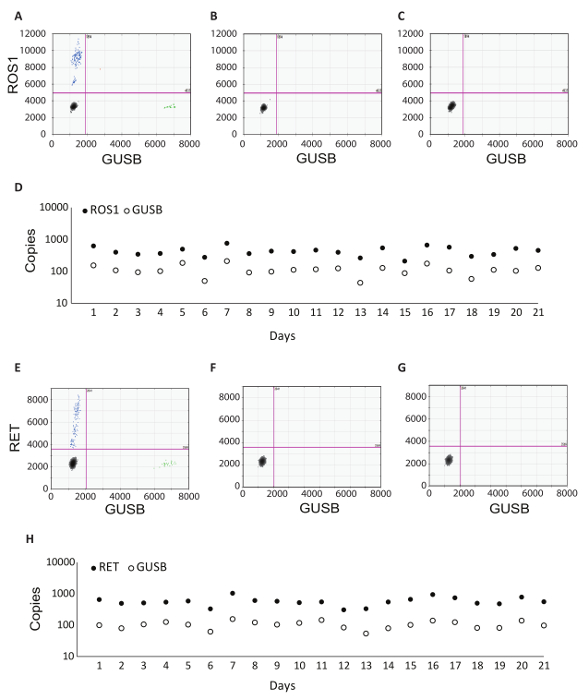
図 3: バッチ コントロールの例と堅牢性データを処理します。ROS1 多重アッセイ dPCR の 2次元プロットの結果 (A) 肯定的な制御、(B) なしの逆転写酵素の制御、および (C) ない RNA テンプレート コントロール。(D) コントロール (土日祝日を除く) 21 の連続した日に実行されました。陽性コントロール 141 ± 439 ROS1 のための標準偏差 ± コピー数を意味します。ない逆転写酵素とはテンプレート コントロールもそれぞれの日に実行された、これらがすべて負 (データは示されていない)。RET 多重アッセイ dPCR の 2次元プロットの結果 (E) 肯定的な制御、(F) (G) と逆転写酵素操縦不能ない RNA テンプレート コントロール。(H) コントロール (土日祝日を除く) 21 の連続した日に実行されました。RET 陽性コントロール 182 +/-586 の標準偏差 ± コピーを意味します。ない逆転写酵素とそれぞれの日に実行されたも、すべて陰性だったないのテンプレート コントロールは、表示されません。許可を得て Biodesix ウェブサイトから適応。この図の拡大版を表示するのにはここをクリックしてください。
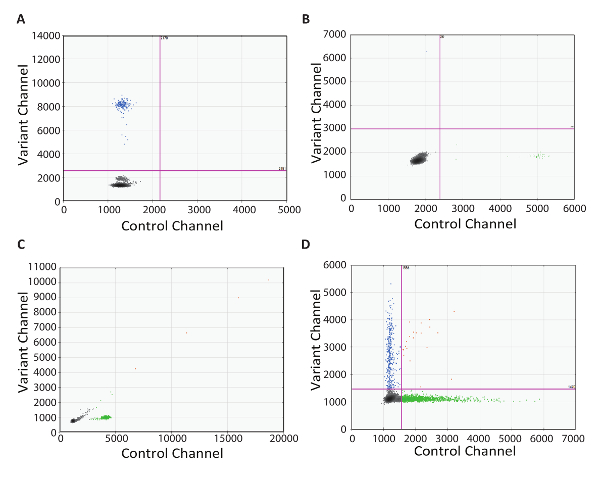
図 4: RT dPCR をトラブルシューティングします。2D プロット表す最適 dPCR 得られた結果 (A) ないの逆写コントロールでは、ないの RNA コントロールでは、(C) (B) 汚染汚染がある場合せん断とし (D の結合) 不十分な PCR の状態または PCR 阻害を最適化します。この図の拡大版を表示するのにはここをクリックしてください。
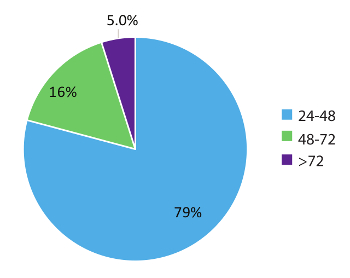
図 5: 所要時間 (TAT).TAT (単位は時間) でコンパイルされたテスト RNA バリアントを要求する (n = 984)。週末、祝日、およびサンプルの開催データを除外 > 実験室テスト依頼フォーム臨床情報が不完全なため 24 h。この図の拡大版を表示するのにはここをクリックしてください。
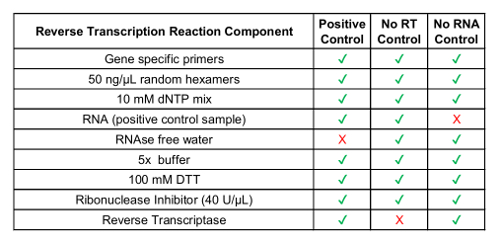
表 1: プロセス コントロールの逆のトランスクリプション試薬の準備。
| コンポーネント | ボリューム |
| dPCR スーパーミックス プローブ x 2 (ない-2'-デオキシウリジン 5'-三リン酸塩) | 10 Μ L |
| 20 x バリエーション ターゲットのプライマー ・ プローブ セットします。 (250 nmol/L FAM 450 nmol/L プライマー プローブ) | 1 Μ L |
| 制御対象のプライマー ・ プローブ セット × 20 (450 nmol/L プライマー、250 nmol/L ヘックス プローブ) | 1 Μ L |
| ヌクレアーゼ フリー水 | 1 Μ L |
| cDNA | 7 Μ L |
表 2: dPCR マスター ミックスの準備。
| ステップをサイクリング | 温度 | 時間 | # サイクル | ランプに入る率 |
| 酵素活性化 | 95 ° C | 10 分 | 1 | 〜 2 oC/s |
| 変性 | 94 ° C | 30 s | 40 | |
| 焼鈍/拡張機能 | 55 ° C | 1 分 | ||
| 酵素を不活性 | 98 ° C | 10 分 | 1 | |
| (省略可能) 保持します。 | 4 ° C | 無限 | 1 | 〜 1 oC/s |
表 3: 熱サイクリング条件。
ディスカッション
RET と ROS1 再編成は、非小細胞肺癌人口18内ドライバー変異の ~ 3% を一緒に作る。まれですが、これらの遺伝的変異の検出が不可欠です。これらの変化と非小細胞肺癌患者に起因する新規蛋白質13異常キナーゼ活性を阻害する標的治療から寄与するかもしれない。いくつかそのような療法は、他は臨床試験19の RET に効くことが示されている間既に ROS1 陽性非小細胞肺癌で使用するために FDA によって承認されています。
デジタル PCR の技術は、液体生検アプリケーション20に最適な感度を提供します。この技術の使用のための重要な採用があった循環の非小細胞肺癌4,6,21,22,23症例における腫瘍の突然変異の測定のための DNA の携帯無料.CfDNA、に加えて循環腫瘍 RNA (図 1A)10から非小細胞肺癌患者における最も一般的な融合亜種の堅牢な測定のために設計されたプロトコルを開発しました。
私たちの確立されたプロトコルは、0.2% (図 2) までの検査の分析限界のことができます。RT dPCR は非常に特定および敏感な法は PCR 法で検出するための多重は選ばれ知られている融合のバリアント型のパネルに制限されます。したがって、非小細胞肺癌患者の人口内の適切なカバレッジを確保する多重分析に含まれるへの融合を慎重に選択する必要があります。正常に RET と ROS1 同時に ROS1 または RET 遺伝子の再編成から 8 融合変形結果を検出し、それぞれ 99% および RET と ROS1 の肯定的な人口の 88% をカバーのための試金を考案したが (図 1B C)17。
前述のように本研究では最終テストのワークフローにはバッチ結果の一貫性を確保するためのコントロールが含まれます。これらのコントロールには、肯定的な分析の標準として一緒に汚染またはバッチ (図 3) 内で発生する PCR 阻害がないことを確認 2 つの否定的なコントロールの両方が含まれます。アッセイの堅牢性を確保するため、(図 3D, H) 21 日間にわたってバッチ コントロールを使用して、検討がなされました。これらのデータは、このプロトコルの中で確立された、RNA プロセスの一貫性を示しています。
よい実験室の練習および適切な RNA 処理は、頑健で正確な結果を保証する部品です。実験スペースや専用機器は RNA、各使用後に機器の洗浄、RNase フリー試薬および消耗品を使用、汚染 RNases を減らすを助けるすべての作業スペースに不活化、RNase のスプレーを適用することを使用します。良心的な処理専用の白衣を含む技術者によって RNA サンプルの RNA 抽出手順をすばやく作業手袋変更を頻繁に、氷の上保管サンプル サンプルの整合性を維持する非常に重要です。RNA の逆は cDNA に転写されており、サンプルされて劣化を招きにくくより安定したフォームになります。RNA の整合性をサポートする方法のほか PCR のコンポーネントおよびサンプルは、偽肯定的な結果につながる可能性が交差汚染を防ぐために隔離エリアに維持しなければなりません。必要があります株式 PCR 試薬および PCR マスター ミックスの準備は、PCR テンプレートとは別に保管され、試薬、RNA および cDNA サンプルを含むすべての中古増幅された材料から増幅されたテンプレート (ポスト PCR) を分離するため細心の注意する必要があります。最後に、適切な生成および増幅前に乳化 PCR ミックスの取扱い、液滴の整合性および最適な dPCR 状態を維持するため中央にあります。一貫性のある、正確な結果を取得するためこのプロトコルの実行中に以下のような注意が重要です。すべてのデータは、すべて QC 基準が満たされていること確かに結果のリリース前に訓練を受けた専門家調査されるべき。最適ではない結果 (図 4)、場合バッチは技術スタッフと実験室ディレクターによって見直される必要があります、再処理が必要な場合があります。
領収書のサンプルと本研究に使用したテスト内のサンプル結果の 95% から 24 時間後に早くも RT dPCR 結果を作り出すことができる (n = 984) 領収書 (図 5) の時間から 72 時間以内に順序付けの医師に報告されました。このターンアラウンド タイム提供医師は多くにより、適切な治療の開始時期に分子の情報を必要とします。これらの結果が従来の組織生検を使用して得られるよりも早く通常できます。非小細胞肺癌や他の癌バイオ マーカー追加と同様の循環 RNA に基づくアプローチを使用して開発できるし、同じ高速時間-する-の結果から恩恵を受けるでしょう。たとえば、RT dPCR を使用してプログラムされた死配位子 1 (PD L1) mRNA の転写産物の測定免疫療法オプションについて医師に通知できます。また、液体生検の有用性の関心の高まりと治療効果のモニタリングに dPCR があります。ゲノム特定のバリアントのテストを用いた腫瘍の復活の以前の適応により、患者が介護24をイメージング等の標準によって症候性前に治療法を調整する医師。本研究で報告されているものなどのプロトコルは、非侵襲性、感度、迅速なターンアラウンド時間、費用対効果などの監視に最適です。記載法は、最小限の誤検出率と、急速な治療の決定を容易にし、組織ベースのテスト4で経験したいくつかの制限を回避するサンプルの領収書から 72 時間以内の結果を提供します。
我々 のプロトコルとデータ低豊富な RNA の亜種として臨床検査血液ベースの突然変異の可能性を識別するための堅牢なテスト システムを示しています。実用的なドライバーを持っていないそれらの患者のための急速なターゲットを絞った液体生検によって識別される突然変異アプローチのようにこの 1 つより広範なゲノムやプロテオーム組織や血液からテストの追加もより広範な臨床情報を提供可能性があります。治療計画をサポートします。
開示事項
陛下、L.J.、比較、ギャップの従業員し、株式保有ギャップ、L.J.、Biodesix、株式会社陛下が Biodesix、循環非小細胞の遺伝的変異を検出するための診断テスト システムの特許申請で共同発明者肺癌。
謝辞
私たちは私たちの協力に感謝、スティーヴン ・ ジョーンズ、Nia キャリングトン、博士ディアナ Maar、博士はサマンサ クーパーのアッセイ デザイン デジタル生物学センター (バイオ ・ ラッド株式会社 CA) からサポート。Nezar Rghei、博士 Moemen アブダラ (Norgen Biotek、カナダ) RNA の抽出のプロトコルを最適化する際の重要なアドバイスシャノン ・ キャンベル、スコット ・ サーストン、ジェフ Fensterer、シャノン マーテローについて Joellyn Enos テスト要件と商業を監視します。
資料
| Name | Company | Catalog Number | Comments |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1604071 |
| Ultrapure Distilled Water (DNAse, RNAse Free) (500 mL) | Life Technologies | 10977-015 | 1809353 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1604071 |
| Nuclease-free water (molecular grade) | Ambion | AM9938 | 1606077 |
| Phosphate Buffered Saline 1X, Sterile | Amresco | K812-500mL | 1446C189 |
| Phosphate Buffered Saline 1X, Sterile – 500 mL | Invitrogen | 10010023 | 1916C092 |
| RNase Zap (Life Tech) (250 mL) | Ambion | AM9780 | 353952 |
| Beta-Mercaptoethanol (BME) (250 mL) | CalbioChem | 6050 | W105B |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56054611 |
| OmniPur Ethyl Alcohol | CalbioChem | 4455-4L | 56238638 |
| Isopropyl Alcohol | VWR | 0918-4L | 2116C416 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 403648 |
| TranscriptAid T7 High Yield Transcription Kit | Thermo Scientific | K0441 | 288461 |
| DNase I | Thermo | K0441 | 371299 |
| QIAzol Lysis Reagent | Qiagen | 79306 | 54809699 |
| 20x TE buffer pH 8.0 | Alfa Aesar | J62388 | R13C548 |
| UltraPure Agarose | Invitrogen | 16500-100 | 552730 |
| 10x TBE buffer | Invitrogen | AM9863 | 353065 |
| Cell-Free RNA BCT | Streck | 218976 | 60110327 |
| Cell-Free RNA BCT | Streck | 218976 | 61900327 |
| Cell-Free RNA BCT | Streck | 218976 | 61480327 |
| Cell-Free RNA BCT | Streck | 218976 | 62320327 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 585849 |
| Plasma/Serum Circulating and Exosomal RNA Purification Kit (Slurry Format) 50 preps | Norgen | 42800 | 588308 |
| Lysis Buffer | Norgen | 21205 | A5F61E |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 585848 |
| RNA Cleanup and Concentration Micro-Elute Kit (Norgen) 50 preps | Norgen | 61000 | 588309 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC186976 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188077 |
| DNA Clean and ConcentratorTM- 5 200 preps (samples) | Zymo | D4014 | ZRC188413 |
| Collection Tubes 500 pack | Zymo | C1001-500 | N/A |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 391657 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 392504 |
| SuperScript IV First Strand Synthesis System 200 rxn (samples) | Life Technologies | 18091200 | 448001 |
| SuperScript IV Reverse Transcriptase | Life Technologies | 18090200 | 451702 |
| Qubit HS RNA Assay Kit (500) | Life Technologies | Q32854 | 1745264 |
| Qubit assay tubes (500) | Life Technologies | Q32856 | 13416Q311 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64031651 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64063941 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065740 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64065741 |
| ddPCR Supermix for Probes (no dUTP) | Bio-Rad | 1863023 | 64079083 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64025320 |
| ddPCR Buffer Control for Probes | Bio-Rad | 1863052 | 64052358 |
| gBlock KIF5B-RET K15:R12 | IDT | 151004172 | 4-Oct-16 |
| gBlock KIF5B-RET K16:R12 | IDT | 151004173 | 4-Oct-16 |
| gBlock KIF5B-RET K22:R12 | IDT | 151004174 | 4-Oct-16 |
| gBlock KIF5B-RET K23:R12 | IDT | 151004175 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R11 | IDT | 151004176 | 4-Oct-16 |
| gBlock KIF5B-RET K24:R8 | IDT | 151004177 | 4-Oct-16 |
| gBlock CCDC6-RET C1:R12 | IDT | 151004178 | 4-Oct-16 |
| gBlock TRIM33-RET T14:R12 | IDT | 151004179 | 4-Oct-16 |
| RET exon 8 RT Gene Specific Primer | IDT | 150554385 | 28-Sep-16 |
| 5’-CTCCACTCACACCTG-3’ | IDT | 150554385 | 28-Sep-16 |
| RET exon 11 RT Gene Specific Primer | IDT | 150554384 | 28-Sep-16 |
| 5’-GCAAACTTGTGGTAGCAG-3’ | IDT | 150554384 | 28-Sep-16 |
| RET exon 12 RT Gene Specific Primer | IDT | 150554383 | 28-Sep-16 |
| 5’-CTGCCTTTCAGATGGAAG-3’ | IDT | 150554383 | 28-Sep-16 |
| gBlock CD74-ROS1 C6:R34 | IDT | 152324366 | 15-Nov-16 |
| gBlock CD74-ROS1 C6:R32 | IDT | 152324367 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R32 | IDT | 152324368 | 15-Nov-16 |
| gBlock SDC4-ROS1 S2:R34 | IDT | 152324369 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R32 | IDT | 152324370 | 15-Nov-16 |
| gBlock S13del2046-ROS1 S13del2046:R34 | IDT | 152324371 | 15-Nov-16 |
| gBlock EZR-ROS1 E10:R34 | IDT | 152324372 | 15-Nov-16 |
| gBlock TPM3-ROS1 T8:R35 | IDT | 152324373 | 15-Nov-16 |
| ROS1 exon 34 RT Gene Specific Primer | IDT | 152704983 | 21-Nov-16 |
| 5’-CCTTCCTTGGCACTTT-3’ | IDT | 152704983 | 21-Nov-16 |
| ROS1 exon 35 RT Gene Specific Primer | IDT | 152704985 | 21-Nov-16 |
| 5’-CTCTTGGGTTGGAAGAGTATG-3’ | IDT | 152704985 | 21-Nov-16 |
| ALK Gene Specific Primer | IDT | 140035422 | 26-Aug-16 |
| 5’-CAGTAGTTGGGGTTGTAGTCG-3’ | IDT | 140035422 | 26-Aug-16 |
| EML4-ALK Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| SLC34A2-ROS1 Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| CCDC6-RET Cell line pellet | Horizon Discovery | N/A | 11-Jun-15 |
| Human Brain Total RNA | Ambion | AM7962 | 1703548 |
| PrimePCR ddPCR Expert Design Assay: K15:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K16:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K22:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K23:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R11 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: K24:R8 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C1:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: T14:R12 | Bio-Rad | N/A | 17-Aug-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: C6:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S2:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R32 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: S13del2046:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: E10:R34 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: T8:R35 | Bio-Rad /Biodesix | N/A | 6-Dec-16 |
| (version 2) | Bio-Rad | 12003909 | 213939881 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 13-Dec-16 |
| PrimePCR ddPCR Expert Design Assay: ROS1 Multiplex (version 3.2) | Bio-Rad | N/A | 20170112v3.2 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851151 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 207383915 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 195995635 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 212851152 |
| PrimePCR ddPCR Gene Expression Probe Assay: GUSB, Human | Bio-Rad | 10031257 | 213949301 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 20160914 |
| PrimePCR ddPCR Expert Design Assay: EML4-ALK | Bio-Rad | 12003909 | 211383227 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 1065C220 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052953 |
| Droplet Generation Oil for Probes | Bio-Rad | 186-3005 | 64052358 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 1065C320 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64052952 |
| Automated Droplet Generation Oil for Probes (20x96) | Bio-Rad | 186-4110 | 64064127 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000065883 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084276 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000079928 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084395 |
| DG8 Cartridges for QX100/QX200 Droplet Generator | Bio-Rad | 186-4008 | C000084634 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20160627 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161107 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161206 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20161216 |
| Droplet Generator DG8 Gasket | Bio-Rad | 186-3009 | 20170125 |
| Pipet Tips for Automated Droplet Generator | Bio-Rad | 1864120 | PR125340 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206894 |
| DG32 Cartridge for Automated Droplet Generator (10-96 well plates) | Bio-Rad | 186-4108 | 206893 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 1409850 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 100402 |
| Pierceable Foil Heat Seal | Bio-Rad | 1814040 | 145851 |
| Microseal 'B' seals | Bio-Rad | MSB1001 | BR00428490 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64039089 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049253 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64049255 |
| ddPCR Droplet Reader Oil | Bio-Rad | 186-3004 | 64081870 |
| DNA Lo Bind Tube 0.5 mL | Eppendorf | 22431005 | E1629620 |
| DNA Lo Bind Tube 1.5 mL | Eppendorf | 22431021 | F16698K |
| DNA Lo Bind Tube 2 mL | Eppendorf | 22431048 | E160610I |
| 50 mL Conicals, Polypropylene (25) | Thermo | 339652 | G5ZF5W8118 |
| TempAssure PCR 8-Strips, Optical Caps, Natural, polypropylene (120) | USA Scientific | 1402-4700 | 16202 |
| For Rainin LTS Pipettors 0.5-20 µL tips | Pipette.com | LF-20 | 40155-642C4-642C |
| For Rainin LTS Pipettors 5-200 µL tips | Pipette.com | LF-250 | 40154-642C4-642B |
| Tips LTS 200 ul Filter 960/10 RT-L200F (10 boxes) | Rainin | 17002927 | 1635 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F1175551-1108 |
| Pipet Tips, 10 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1181-3710 | F118054L-1720 |
| Pipet Tips, 100 ul TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1180-1740 | 0014961Q-2501 |
| Pipet Tips, 200 ul TipOne RPT ultra low retention filter tip refill cassette, sterile | USA Scientific | 1180-8710 | E116684P-1540 |
| Pipet Tips, 1000 ul XL TipOne RPT ultra low retention filter tip refill cassette, sterile (10x96) | USA Scientific | 1182-1730 | F118815P |
| 5 mL Standard Racked Gilson-fit Reference Tips | Scientific Specialties | 4411-00 | 14312 |
| Combitips advanced, 0.1 mL Biopur | Eppendorf | 003 008 9618 | F165414H |
| Combitips advanced, 0.2 mL Biopur | Eppendorf | 0030 089.626 | F166689J |
| Combitips advanced, 5 mL Biopur | Eppendorf | 0030.089 669 | F166054J |
| Combitips advanced, 50 mL Biopur | Eppendorf | 003.008.9693 | F166055I |
| Reagent Reservoir | VWR | 89094-680 | 141500 |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165029I |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | F165028G |
| Twin tec PCR Plate 96, semi-skirted, Clear | Eppendorf | 951020303 | E163697P |
| Twin tec PCR Plate 96, semi-skirted, Green | Eppendorf | 951020346 | F166183K |
| Equipment Type | Equipment ID | ||
| Analytical Balance | EQP0125 | ||
| Cryogenic Freezer 1, -80oC | EQP0095 | ||
| Refrigerator 6.1 cu ft GP06W1AREF | EQP0139 | ||
| -20oC Freezer | EQP0140 | ||
| Beckman Coulter Microfuge 22R | EQP0025 | ||
| Beckman Coulter Microfuge 22R | EQP0124 | ||
| Thermo Scientific Hereaus Megafuge 8 | EQP0104 | ||
| Mini Centrifuge | EQP0131 | ||
| Mini Centrifuge | EQP0136 | ||
| Mini Centrifuge | EQP0134 | ||
| Mini Centrifuge | EQP0235 | ||
| Mini Centrifuge | EQP0216 | ||
| Thermo Scientific HeraTherm Incubator | EQP0105 | ||
| Pipette 0.1 - 2.5 μL | EQP0182 | ||
| Pipette 0.1 - 2.5 μL | EQP0072 | ||
| Pipette 0.1 - 2.5 μL | EQP0070 | ||
| Pipette 0.5-10 μL | EQP0218 | ||
| Pipette 0.5-10 μL | EQP0075 | ||
| Pipette 0.5-10 μL | EQP0169 | ||
| Pipette 0.5-10 μL | EQP0074 | ||
| Pipette 0.5-10 μL | EQP0147 | ||
| Pipette 2 - 20 μL | EQP0128 | ||
| Pipette 2 - 20 μL | EQP0160 | ||
| Pipette 2 - 20 μL | EQP0018 | ||
| Pipette 2 - 20 μL | EQP0146 | ||
| Pipette 10 - 100 μL | EQP0079 | ||
| Pipette 10 - 100 μL | EQP0181 | ||
| Pipette 10 - 100 μL | EQP0085 | ||
| Pipette 10 - 100 μL | EQP0077 | ||
| Pipette 20 - 200 μL | EQP0088 | ||
| Pipette 20 - 200 μL | EQP0087 | ||
| Pipette 20 - 200 μL | EQP0231 | ||
| Pipette 100 - 1000 μL | EQP0050 | ||
| Pipette 100 - 1000 μL | EQP0158 | ||
| Pipette 100 - 1000 μL | EQP0217 | ||
| Pipette 100 - 1000 μL | EQP0082 | ||
| Pipette 100 - 1000 μL | EQP0183 | ||
| Pipette 100 - 1000 μL | EQP0083 | ||
| Pipette 5 mL | EQP0153 | ||
| Timer | S/N 140623950 | ||
| Hamilton SafeAire VAV Fume Hood | EQP0206 | ||
| Biosafety Cabinet | EQP0205 | ||
| Biosafety Cabinet | EQP0204 | ||
| Qubit 3.0 | EQP0102 | ||
| Benchmark Digital Heat Block | EQP0108 | ||
| Benchmark Digital Heat Block | EQP0231 | ||
| Polaroid Z2300 Instant Print Digital Gel Camera with WiFi and 16GB SDHC memory card | EQP0111 | ||
| Electrophoresis Power Unit | EQP0113 | ||
| Electrophoresis Small Gel Box | EQP0116 | ||
| Maestro Transilluminator | EQP0118 | ||
| Microwave | EQP0215 | ||
| Multichannel 8-well Pipette 2 - 20 μL | EQP0207 | ||
| Multichannel 8-well Pipette 10 - 100 μL | EQP0090 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0094 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0161 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0162 | ||
| Rainin Multichannel 8-well Pipette 50 μL | EQP0163 | ||
| Vortex Genie 2 | EQP0052 | ||
| Vortex Genie 2 | EQP0007 | ||
| Vortex Genie 2 | EQP0132 | ||
| Vortex Genie 2 | EQP0137 | ||
| Vortex Genie 2 | EQP0135 | ||
| Air Clean PCR Workstation | EQP0203 | ||
| Air Clean PCR Workstation | EQP0096 | ||
| Air Clean PCR Workstation | EQP0148 | ||
| Air Clean PCR Workstation | EQP0097 | ||
| QX200 Droplet Generator | EQP0202 | ||
| QX200 Droplet Generator | EQP0121 | ||
| Automated Droplet Generator | EQP0179 | ||
| PX1 PCR Plate Sealer | EQP0123 | ||
| PX1 PCR Plate Sealer | EQP0186 | ||
| C1000 Touch Cycler w/96W FS RM | EQP0120 | ||
| S1000 Cycler w/96W FS RM | EQP0174 | ||
| S1000 Cycler w/96W FS RM | EQP0173 | ||
| T100 Thermal Cycler | EQP0180 | ||
| T100 Thermal Cycler | EQP0175 | ||
| QX200 Droplet Reader | EQP0194 | ||
| QX200 Droplet Reader | EQP0122 |
参考文献
- Ignatiadis, M., Lee, M., Jeffrey, S. S. Circulating Tumor Cells and Circulating Tumor DNA: Challenges and Opportunities on the Path to Clinical Utility. Clin Cancer Res. 21 (21), 4786-4800 (2015).
- Alix-Panabieres, C., Pantel, K. Clinical Applications of Circulating Tumor Cells and Circulating Tumor DNA as Liquid Biopsy. Cancer Discov. , (2016).
- Paxton, A. Is Molecular AP testing in sync with guidelines. CAP Today. , (2014).
- Sacher, A. G., et al. Prospective Validation of Rapid Plasma Genotyping for the Detection of EGFR and KRAS Mutations in Advanced Lung Cancer. JAMA Oncol. , (2016).
- Sozzi, G., et al. Quantification of free circulating DNA as a diagnostic marker in lung cancer. J Clin Oncol. 21 (21), 3902-3908 (2003).
- Oxnard, G. R., et al. Noninvasive detection of response and resistance in EGFR-mutant lung cancer using quantitative next-generation genotyping of cell-free plasma DNA. Clin Cancer Res. 20 (6), 1698-1705 (2014).
- Best, M. G., et al. RNA-Seq of Tumor-Educated Platelets Enables Blood-Based Pan-Cancer, Multiclass, and Molecular Pathway Cancer Diagnostics. Cancer Cell. 28 (5), 666-676 (2015).
- Rodriguez, M., et al. Different exosome cargo from plasma/bronchoalveolar lavage in non-small-cell lung cancer. Genes Chromosomes Cancer. 53 (9), 713-724 (2014).
- Kalluri, R. The biology and function of exosomes in cancer. J Clin Invest. 126 (4), 1208-1215 (2016).
- Mellert, H., et al. Development and Clinical Utility of a Blood-Based Test Service for the Rapid Identification of Actionable Mutations in Non-Small Cell Lung Carcinoma. J Mol Diagn. 19 (3), 404-416 (2017).
- Qin, J., Williams, T. L., Fernando, M. R. A novel blood collection device stabilizes cell-free RNA in blood during sample shipping and storage. BMC Res Notes. 6, 380 (2013).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. Biotechniques. 15 (3), 532-537 (1993).
- Kohno, T., et al. Beyond ALK-RET, ROS1 and other oncogene fusions in lung cancer. Transl Lung Cancer Res. 4 (2), 156-164 (2015).
- Takeuchi, K., et al. ROS1 and ALK fusions in lung cancer. Nat Med. 18 (3), 378-381 (2012).
- Rimkunas, V. M., et al. Analysis of receptor tyrosine kinase ROS1-positive tumors in non-small cell lung cancer: identification of a FIG-ROS1 fusion. Clin Cancer Res. 18 (16), 4449-4457 (2012).
- Tsuta, K., et al. RET-rearranged non-small-cell lung carcinoma: a clinicopathological and molecular analysis. Br J Cancer. 110 (6), 1571-1578 (2014).
- Forbes, S. A., et al. COSMIC: somatic cancer genetics at high-resolution. Nucleic Acids Res. 45 (1), 777-783 (2017).
- Salgia, R. Diagnostic challenges in non-small-cell lung cancer: an integrated medicine approach. Future Oncol. 11 (3), 489-500 (2015).
- Cagle, P. T., Raparia, K., Portier, B. P. Emerging Biomarkers in Personalized Therapy of Lung Cancer. Adv Exp Med Biol. 890, 25-36 (2016).
- Vogelstein, B., Kinzler, K. W. Digital PCR. Proc Natl Acad Sci U S A. 96 (16), 9236-9241 (1999).
- Oxnard, G. R., et al. Association Between Plasma Genotyping and Outcomes of Treatment With Osimertinib (AZD9291) in Advanced Non-Small-Cell Lung Cancer. J Clin Oncol. 34 (28), 3375-3382 (2016).
- Reckamp, K. L., et al. A Highly Sensitive and Quantitative Test Platform for Detection of NSCLC EGFR Mutations in Urine and Plasma. J Thorac Oncol. 11 (10), 1690-1700 (2016).
- Yanagita, M., et al. A prospective evaluation of circulating tumor cells and cell-free DNA in EGFR mutant non-small cell lung cancer patients treated with erlotinib on a phase II trial. Clin Cancer Res. , (2016).
- Abbosh, C., et al. Phylogenetic ctDNA analysis depicts early-stage lung cancer evolution. Nature. 545 (7655), 446-451 (2017).
- Chomczynski, P. A reagent for the single-step simultaneous isolation of RNA, DNA and proteins from cell and tissue samples. BioTechniques. 15 (3), 532-534 (1993).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved