このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
同様に効果的な二値分類性能を持つ複数のバイオ マーカーのサブセットを選択
要約
既存のアルゴリズムは、バイオ マーカー検出データセットの 1 つのソリューションを生成します。このプロトコルは、複数の同様に有効な解の存在を示し、提案の挑戦のためのデータセットの調査生物医学の研究のためのユーザーフレンドリーなソフトウェアを示します。コンピューター科学者は、彼らのバイオ マーカーのこの機能が検出アルゴリズムを入力もできます。
要約
バイオ マーカー検出 '高速 'オミックス研究者より重要な医学問題の 1 つは、ほぼすべての既存のバイオ マーカー検出アルゴリズムは、与えられたデータセットの最適化されたパフォーマンスの測定と 1 つのバイオ マーカーのサブセットを生成.しかし、最近の研究では、同様に効果的なまたは同一の分類性能を持つ複数のバイオ マーカーのサブセットの存在を実証しました。このプロトコルは、二値分類のパフォーマンス、ユーザー定義カットオフよりバイオ マーカーのサブセットを検出するためのシンプルで簡単な方法論を提示します。プロトコルは、データの準備と読み込み、ベースライン情報の要約、パラメータ チューニング、バイオ マーカーのスクリーニング、結果可視化解釈、バイオ マーカー遺伝子アノテーションとで結果と可視化の輸出で構成されています出版物の品質。スクリーニング戦略提案のバイオ マーカーは、直感的なバイオ マーカー検出アルゴリズムを開発するための一般的な規則を示します。ユーザーフレンドリーなグラフィカル ユーザー インターフェイス (GUI) は、プログラミング言語の Python では、生物医学の研究の結果に直接アクセスすることができますを使用して開発されました。ソース コードとマニュアルの kSolutionVis は、http://www.healthinformaticslab.org/supp/resources.php からダウンロードできます。
概要
二値分類、最も一般的の 1 つを調査し、最も正確な差別のパワー1,のサンプルの 2 つのグループの訓練を受けて分類モデルの構築に挑戦的なデータマイニングで生物医学分野における問題を使用2,3,4,5,6,7します。 ただし、バイオメディカル分野で生成された大きなデータは、本来「大 p 小さな n」パラダイム、機能サンプル6,8,9の数より大きい数を持つ。したがって、生物学者は、過問題8,9を避ける分類アルゴリズムを活用する前にフィーチャーの寸法を減らさなければなりません。診断バイオ マーカーは、健康的な制御サンプル10,11から特定の病気の患者を分離検出されたフィーチャのサブセットとして定義されます。患者は通常、肯定的なサンプルとして定義されて、健常者が負サンプル12として定義されます。
最近の研究では、生体データセット5ため同一または同様に効果的な分類の公演で 1 つ以上のソリューションが存在することを示唆しています。ほぼすべての機能選択アルゴリズムは、確定的なアルゴリズムは、同じデータセットを 1 つだけ解決です。遺伝的アルゴリズム同時に同様の性能を持つ複数のソリューションを生成可能性がありますが、彼らはまだ与えられたデータセット13,14の出力として最高のフィットネス関数で 1 つのソリューションを選択ましょう。
機能選択アルゴリズムは、フィルターまたはラッパー12として大体グループ化できます。フィルター アルゴリズム選択機能仮定に基づくバイナリ クラス ラベルを重要な個別連想ランキング トップ -k機能がお互い15,16,17 の独立しました。.この前提が成立しないほとんどすべての現実世界のデータセット、ヒューリスティック フィルタ ルール アルゴリズムを実行する、多くの場合、例えば、mRMR (冗長性を最小と最大の関連性)、Wilcoxon テストによる機能フィルター (WRank)アルゴリズム、および ROC (受信者操作特性) プロット (ROCRank) をフィルタ リング アルゴリズムを用いた。それははるかに小さい問題、それぞれのだけ 2 つの変数が含まれている最大依存関係機能選択アルゴリズムと比較する一連の組合せの推定問題を近似 mRMR、効率的なフィルター アルゴリズムは、したがってより堅牢な18,19ペア共同確率を使用します。しかし、それでは、関連性を高めることができますし、このように個別に役に立たないが、組み合わせたときにのみ有用ないくつかの機能の組み合わせをミスの機能間の相互作用を計測、mRMR 可能性がありますいくつかの機能の有用性を過小評価。WRank アルゴリズムは、どのように識別機能のサンプルは、2 つのクラス間と外れ値20,21の堅牢性のために知られている非パラメトリック スコアを計算します。さらに、ROCRank アルゴリズムは, 二値分類性能22,23どのように重要な ROC の下で領域曲線 (AUC) の特定の機能を評価します。
その一方で、ラッパーは繰り返しヒューリスティック ルールによって生成された特定の機能サブセットの定義済みの分類子のパフォーマンスを評価し、最高のパフォーマンス測定24の機能のサブセットを作成します。ラッパーは、一般的に分類性能のフィルターよりも優れているが、遅い25を実行します。正則ランダム フォレスト (RRF)26,27アルゴリズムが機能重要性スコアは、ジニ指数によって評価される各ランダム フォレスト] ノードにトレーニング データのサブセットの機能を評価することによって、貪欲な規則を使用するたとえば、.新機能の選択は、そのインフォメーション ・ ゲインが選ばれた機能の改善しない場合罰せられることでしょう。さらに、マイクロ アレイ (PAM)28,29アルゴリズムもラッパーのアルゴリズムの予測分析クラスのラベルのそれぞれの重心を計算し、全体に向けた遺伝子重心を圧縮する機能を選択しますクラスの重心。PAM は遠く隔った機能の堅牢です。
トップの分類性能を持つ複数のソリューション、特定のデータセットの必要があります。まず、決定的アルゴリズムの最適化の目標は、必ずしも生物学的サンプルの理想的ではない、数式、例えば、最小誤り率30、によって定義されます。第二に、データセットと同様の効果的なまたは同一公演を複数、大幅に異なるソリューションがあります。ほとんどすべての既存機能選択アルゴリズムは、ランダムに出力31としてこれらのソリューションのいずれかを選択します。
本研究は、任意の指定されたバイナリ分類データセットと同様のパフォーマンスと複数の機能選択解情報分析プロトコルをご紹介いたします。情報技術やコンピューターがコーディングに精通しているない最も生物学者を考慮した、ユーザーフレンドリーなグラフィカル ユーザー インターフェイス (GUI) は生体二値分類データセットの迅速分析を容易にするために開発されました。分析のプロトコルは、データの読み込みと集計、パラメーター調整、パイプラインの実行と結果の解釈で構成されています。単純なクリックで研究者、バイオ マーカーのサブセットと文書品質可視化のプロットを生成することができます。プロトコルは、2 つの二値分類データセット急性リンパ芽球性白血病 (すべて) のすなわちALL1 と ALL212のトランスクリプトームを使用してテストされています。ALL1 と ALL2 のデータセットは、広範な研究所ゲノム解析センター、http://www.broadinstitute.org/cgi-bin/cancer/datasets.cgi でご利用いただけますからダウンロードされました。ALL1 には 12,625 機能を持つ 128 のサンプルが含まれています。これらのサンプルの 95 は B 細胞すべてと 33 T 細胞は、すべて。ALL2 には 12,625 機能同様の 100 サンプルが含まれます。これらのサンプルの再発を受けた 65 患者としなかった患者 35 例があります。ALL1 はだった 4 つのフィルターと 4 つのラッパーが 96.7% と 10012を達成する 8 特徴選択アルゴリズムの 6 の最小精度の簡単なバイナリ分類データセットです。ALL2 は 83.7% 精度12よりも優れて実現する上記の 8 の機能選択アルゴリズムのより困難なデータセットです。この最高の精度は相関機能選択 (CFS)、ラッパー アルゴリズムによって検出された 56 機能で実現されました。
プロトコル
注: 次のプロトコルは、情報の分析手順の詳細との主要なモジュールの擬似コードをについて説明します。自動解析システムは、Python バージョン 3.6.0 と Python モジュール パンダ、abc、numpy、scipy、sklearn、sys、PyQt5、sys、mRMR、数学および matplotlib を使用して開発されました。本研究で使用される材料は、材料のテーブルに表示されます。
1 データ行列を準備し、クラスのラベル
- 図 1 aに示されているように、タブ区切りまたはコンマ区切りの行列ファイルとしてデータ マトリックス ファイルを準備します。
メモ: 各行は、機能のすべての値、最初のアイテムは機能名。機能はマイクロ アレイを用いたトランスクリプトーム データセットの probeset id または、methylomic データセットのメチル化値のシステイン残基のような別の値 ID があります。各列は、サンプル名をされている最初の項目と特定のサンプルの特徴量を与えます。行は、タブ (図 1 b) またはカンマ (図 1) で列に区切られます。行列をタブ区切りファイルは、ファイル拡張子 .tsv によって認識され、マトリックスのコンマで区切られたファイルに拡張子 .csv。このファイルは、Microsoft Excel などのソフトウェアから .tsv または .csv 形式いずれかとして行列を保存生成ことがあります。データ行列を生成するコンピューター化によっても。 - タブ区切りまたはカンマ区切り行列ファイルとして (図 1)、データ行列ファイルに似ていますクラスファイル ラベルを準備します。
注: 最初の列は、サンプル名を与えるし、各サンプルのクラスのラベルは列のクラスをタイトルで与えられます。最大限の互換性は、追加の列を追加する可能性があります、コーディング プロセスでと見なされます。クラスのラベル ファイルは、.tsv または .csv ファイルとしてフォーマット可能性があります。列クラス内の名前可能性があります条項は、およびサンプルの 2 つ以上のクラスがあります。ユーザーは、次の分析のためのクラスの任意の 2 つを選択可能性があります。
2. データ行列をロードし、クラスのラベル
- データ マトリックスおよびクラスのラベルをソフトウェアにロードします。負荷データ マトリックスユーザー指定のデータ行列ファイルを選択してボタンをクリックします。負荷クラス ラベルクラスのラベルの対応するファイルを選択する] ボタンをクリックします。
注: 両方のファイルが読み込まれた後、kSolutionVis は 2 つのファイル間の互換性のルーチン画面を行います。 - 機能とデータ マトリックス ファイルからのサンプルを要約します。データ マトリックス ファイルのサイズを見積もります。
- サンプルおよびクラスのラベル ファイルからクラスをまとめます。クラスのラベル ファイルのサイズを見積もります。
- 各サンプル データ マトリックスからクラスのラベルがあるかどうかをテストします。クラスのラベルとサンプル数をまとめたものです。
3. 要約、データセットのベースライン統計を表示
- 任意の指定されたキーワードを入力することがなく集計の方法をクリックし、ソフトウェア 20 インデックス機能と対応する機能名が表示されます。
注: ユーザーは、そのベースライン統計とすべての入力サンプルの間で対応する値の分布を参照してくださいに検索したい機能名を指定する必要があります。 - キーワード、例えば「1000_at」、テキスト ボックスの集計の対象とする特定機能を検索する機能を提供します。要約この特定機能のベースライン統計を取得するボタンをクリックします。
注: キーワードがターゲットの機能名、ユーザーの検索プロセスを促進することで任意の場所表示されます。 - 与えられたキーワードと 1 つ以上の機能を見つけるに集計」ボタンをクリックし、1 つの特定の機能をまとめたは、上記の手順を続行するユニークな機能 ID を指定します。
4. クラスのラベルと上位ランクのフィーチャの数を決定します。
- (中央)図 2に示すように、クラス正と負クラス、ドロップ ダウン ボックスで正 ("P (33)") と負 ("N (95)") クラスの名前を選択します。
注: バランスの取れたバイナリ分類データセット、すなわち、正と負のサンプル数の違いは最小限を選択する勧めします。かっこでも指定は、2 つのドロップ ダウン ボックスで各クラスのラベルの名前の後のサンプル数です。 - 上位機能 ( pTopXパラメーター) の数として 10 を選択ドロップダウン ボックスTop_X (?)機能サブセットの包括的な画面。
注: ソフトウェアは自動的にP- 正と負のクラスを比較する各機能の t 検定によって計算された値によってすべての機能をランク付けします。小さいP- 機能値のサンプルの 2 つのクラス間より識別力があります。包括的な上映モジュールは負荷が高いです。PTopXパラメーターは既定では 10 です。ユーザーが満足を見つけるまでに 50 から 10 の範囲内のこのパラメーターを変更できます良い分類性能を持つサブセットの機能します。
5. 別の公演のためシステムのパラメーターを調整します。
- 選択した分類子極端な学習マシン (ニレ) のドロップダウン ボックスAcc/bAcc (?)のパフォーマンスの測定精度 (Acc) (pMeasurement) を選択します。このパラメーターの別のオプションは、測定精度のバランス (bAcc)。
注: FN、TN、TP と FP それぞれ真陽性、偽陰性、真陰性、偽陽性の数であります。測定Accをいいます (TP+TN)/(TP+FN+TN+FP)、バランスの取れたデータセット6に最適します。Accを最適化された分類子負サンプル数は、肯定的なものよりもはるかに大きい場合負のクラスにすべてのサンプルを割り当てる傾向しますが、あります。BAccが (Sn + Sp) として定義されている/2、Sn = TP/(TP+FN)、Sp = TN/(TN+FP) 正しく予測された料金は正と負のサンプルのそれぞれ。したがって、bAcc は 2 つのクラスで予測性能を正規化し、以上 2 つのアンバランス クラス バランスのとれた予測パフォーマンスにつながる可能性があります。Accは、 pMeasurementの既定の選択です。ソフトウェア分類性能を計算するのにデフォルトでエルム分類子を使用します。また、ユーザーは、SVM (サポートベクター マシン)、KNN (k 最近傍)、意思決定ツリー、または Naïve Bayes から分類子を選択可能性があります。 - 入力ボックスで指定されたパフォーマンス測定のためのカットオフ値 0.70 ( pCutoffパラメーター) を選択pCutoff:。
注: 両方Accと駆け込んでの 0 と 1 の間の範囲し、ユーザー指定値 pCutoff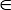 [0, 1] 一致するソリューションを表示するカットオフとして。ソフトウェアは包括的な機能サブセットのスクリーニングを行い、 pCutoffの適切な選択より直感的かつ明示的な 3 D の可視化を行います。PCutoff は0.70 の既定値。
[0, 1] 一致するソリューションを表示するカットオフとして。ソフトウェアは包括的な機能サブセットのスクリーニングを行い、 pCutoffの適切な選択より直感的かつ明示的な 3 D の可視化を行います。PCutoff は0.70 の既定値。
6. パイプラインを実行し、インタラクティブな可視化の結果
- パイプラインを実行し図 2 (下) に示すように、可視化のプロットを生成する分析ボタンをクリックします。
注意: 左側のテーブルは、すべての機能サブセットおよび彼らのpMeasurement 5を前述のようにニレ、分類子の 10 倍のクロス検証戦略による計算を使用します。2 つの 3 D 散布と 2 ライン プロット現在のパラメーター設定と機能サブセットのスクリーニング プロシージャが生成されます。 - PMeasurementカットオフの既定値 (パラメーター piCutoff、入力ボックスの値)、および 10 として最高の機能サブセット ( piFSNumパラメーター) の数の既定値として 0.70 を選択します。
注: パラメーター pTopX、 pMeasurement、およびpCutoffを使用して、パイプラインが実行されます。検出された機能のサブセットがさらにあります上映カットオフpiCutoffを使用すると、しかしpiCutoffすることはできませんpCutoffより小さい。したがって、 piCutoffは、 pCutoffとして初期化され、パフォーマンス測定 ≥ piCutoffと機能のサブセットのみが視覚化されます。PiCutoffのデフォルト値はpCutoffです。時々 kSolutionVis 検出の多くのソリューションと最高のpiFSNumのみ (デフォルト: 10) 特徴集合が視覚化されます。ソフトウェアによって検出機能サブセットの数がpiFSNumより小さい場合は、すべての機能のサブセットが視覚化されます。 - 収集し、図 3に示すように、ソフトウェアによって検出機能を解釈します。
注: 左側のボックスでテーブル検出機能サブセットおよび彼らのパフォーマンスの測定値を示しています。最初の 3 列の名前は、"F1"、"F2"、"F3"です。各機能のサブセットで 3 つの機能が 1 つの行にそのランキング順に与えられている (F1 < F2 < F3)。最後の列は、各機能サブセットのパフォーマンス測定 (AccまたはbAcc) を与えるし、その列名 (AccまたはbAcc) pMeasurementの値です。
7. 3 D 散布プロット視覚化を解釈し、3 D の散布を使用して同様に効果的な二値分類性能と機能のサブセットを解釈
- 図 3 (ミドル ボックス) に示すように、ソフトウェアによって検出 (AccまたはbAcc) 最高の分類性能とトップ 10 の機能サブセットの 3 D 散布を生成する分析ボタンをクリックします。彼らのランクの昇順機能サブセットの 3 つの機能を並べ替え、F1、F2、F3 軸、すなわちF1 と 3 つの機能のランクを使用して < F2 < F3。
注: ドットの色は、対応する機能サブセットのバイナリ分類性能を表します。データセットは、同様に効果的なパフォーマンス測定の複数の機能のサブセットがあります。したがって、インタラクティブおよび簡体字の散布が必要です。 - 0.70 入力ボックスに値を変更pCutoff: 図 3 (右のボックス) に見られるように分析とパフォーマンス測定 ≥ piCutoff機能サブセットの 3 D 散布を生成する] をクリックします。3 D チューニングを手動で 3 D 散布の視野角を調整する新しいウィンドウを開くボタンをクリックします。
注: 上記と同じ方法では、各機能のサブセットをドットで表されます。3 D の散布は、デフォルトの角度で生成されました。3 D 可視化やチューニングによって、別のウィンドウを容易にするためには3 D チューニングボタンをクリックして表示されます。 - 検出された機能のサブセットの冗長性を減らすために削減をクリックします。
注: ソフトウェアはさらに機能のサブセットの冗長性を最小限に抑える機能三つ子を選択して場合は、mRMR 特徴選択アルゴリズムを使用してこの関数も提供.縮小ボタンをクリックすると、kSolutionVis 機能三つ子でこれらの冗長機能を削除し、テーブルを再生成し、上記 2 つの散布します。機能三つ子の削除された機能は表にキーワードで置き換えられます。F1、F2、F3 軸なしの値はpiFSNum (F1、F2、F3 の正常値の範囲は [1, top_x]) の値として表示されます。したがって、「外れ値」3 D ドット プロットする[なし]値が含まれる点があります。手動で調整可能な 3 D プロットは、補足資料で「3 d ドット プロットの手動チューニング」でもあります。
8. 遺伝子注釈および人間の病気との関連付けを検索します。
注: 手順 8 に 10 は DNA および蛋白質のシーケンス レベルから遺伝子に注釈を付ける方法を示しています。まず、上記の手順からバイオ マーカー ID ごとの遺伝子の記号がデビッド32、データベースから取得される、DNA および蛋白質のレベルからこの遺伝子の記号をそれぞれ分析する、2 つの代表的な web サーバーが使用されます。サーバー GeneCard はある特定の遺伝子記号の包括的な機能アノテーションを提供し、人間データベース (OMIM) でオンライン メンデル遺伝病遺伝子連合の最も包括的なキュレーションを提供します。蛋白質の最も包括的なデータベースである UniProtKB サーバーとサーバー グループに基づく予測システム (GPS) はシグナリングのリン酸化の非常に大きなリストのキナーゼを予測します。
- コピーして web ブラウザーにデビッド データベースの web リンクを貼り付け、このデータベースの web ページを開きます。図 4 aに見られる遺伝子 ID への変換リンクをクリックし、Id 38319_at/38147_at/33238_at (図 4 b) データセット ALL1 の最初マーカー サブセットの機能を入力します。遺伝子リストのリンクをクリックし、図 4 bに示すように、リストの提出をクリックします。興味の注釈を取得し、遺伝子リストの表示(図 4) をクリックします。遺伝子記号 (図 4).のリストを得る
注: ここで取得した遺伝子記号は、次の手順でさらに機能上の注釈の使用されます。 - コピーして web ブラウザーに遺伝子カード データベースの web リンクを貼り付け、このデータベースの web ページを開きます。データベース クエリの入力ボックスで遺伝子の名前 CD3D を検索し、表 1と図 5 aに示すように、この遺伝子カード33,34, 遺伝子の注釈を見つけます。
注: 遺伝子カードは、命名法、ゲノミクスやプロテオミクス、細胞内の局在と関与する経路、他の機能モジュールを提供する包括的な遺伝子のナレッジ ベースです。PDB/PDB_REDO35Entrez の遺伝子36、OMIM37UniProtKB38のような様々 な他生物医学データベースへの外部リンクも提供しています。機能名は、標準的な遺伝子記号ではない場合、は、39を変換する ENSEMBL データベースを使用します。CD3D は、遺伝子 T 細胞受容体 T3 デルタ チェーンの名前です。 - コピーして web ブラウザーに OMIM データベースの web リンクを貼り付け、このデータベースの web ページを開きます。遺伝子の名前 CD3D を検索し、表 1と図 5 bに示すように、データベース OMIM37, この遺伝子の注釈を見つけます。
注: OMIM は継承可能な疾患遺伝子の接続の最も包括的かつ権威の源の 1 つとして今すぐ機能します。OMIM は40疾患に関連する遺伝子の突然変異をカタログにビクター ・ マキューズィック博士によって開始されました。OMIM 今 15,000 ひと遺伝子と 12 月現在 8,500 以上の表現型を 1st 2017 をカバーします。
9. 符号化された蛋白質と翻訳後修飾に注釈を付ける
- コピーして web ブラウザーにデータベース UniProtKB の web リンクを貼り付け、このデータベースの web ページを開きます。UniProtKB のクエリの入力] ボックスで遺伝子の名前 CD3D を検索し、表 1と図 5に示すように、データベースの38、この遺伝子の注釈を見つけます。
注: UniProtKB は、名称と機能情報の両方を含む蛋白質のアノテーションの豊富なソースを収集します。このデータベースには、PDB/PDB_REDO35、OMIM37, Pfam41など他の広く使用されているデータベースへの外部リンクも提供します。 - コピーして web ブラウザーに GPS の web サーバーの web リンクを貼り付け、この web サーバーの web ページを開きます。UniProtKB データベース38からバイオ マーカー遺伝子 CD3D によって符号化される蛋白質シーケンスを取得し、表 1と図 5に示すように、オンライン ツール、GPS を使用して蛋白質の翻訳後修飾 (PTM) 残基を予測します。
注: 生体システムは動的であり、複雑な既存のデータベースが既知の情報だけを収集します。したがって、オフライン プログラムと同様に、生体の予測オンライン ツール仮定されたメカニズムを補完するために有用な証拠があります。GPS は、開発および改善のために 12 年7,42をされているし、蛋白質の特定のペプチッド シーケンス43,44PTM 残留を予測するために使用可能性があります。ツールは、タンパク質の細胞内局在45と転写因子結合モチーフ46他の中の予測など、様々 なトピックで研究も承ります。
10. タンパク質間相互作用とその豊かな機能モジュールに注釈を付ける
- コピーし web ブラウザーに web サーバー文字列の web リンクを貼り付けて、この web サーバーの web ページを開きます。CD3D と P53 遺伝子のリストを検索し、データベース文字列47を使用して彼らの管弦楽に編曲された特性を見つけます。同じプロシージャは、別の web サーバー、デビッド32でも行うことが。
注: 個々 の遺伝子の前述の注釈、ほか遺伝子のグループのプロパティを調査する使用可能な多くの大規模な情報ツールがあります。最近の研究は、個別に悪いマーカー遺伝子が多く改良された遺伝子設定5を構成可能性があります示した。したがって、それはより複雑なバイオ マーカーの画面に計算コストの価値があります。データベース文字列可能性がありますまたは予測知られている相互作用の接続を視覚化し、デビッド サーバーは、照会された遺伝子47、32で重要な表現型関連付け機能モジュールを検出ことがあります。その他の各種の大規模な情報分析ツールも承ります。
11. 輸出の生成されたバイオ マーカーのサブセットと可視化のプロット
- さらなる分析のため .tsv または .csv テキスト ファイルとして検出されたバイオ マーカーのサブセットをエクスポートします。すべての検出されたバイオ マーカーのサブセットのテーブルの下でテーブルをエクスポート] ボタンをクリックし、として保存するテキスト形式を選択します。
- 可視化プロットを画像ファイルとしてエクスポートします。各プロットで保存ボタンをクリックし、として保存する画像形式を選択します。
注: ソフトウェアは、ピクセル形式 .png とベクトル形式の紋章をサポートします。ピクセルの画像は、ベクトル画像がジャーナル出版の目的のために必要な任意の解像度に変換される、コンピューター画面での表示に適しています。
結果
このワークフロー (図 6) の目標は、二値分類データセットの同じような効率を持つ複数のバイオ マーカーのサブセットを検出することです。全体のプロセスは、2 つの例のデータセット ALL1 と ALL2 最近公開されたバイオ マーカー検出から抽出した12,48の研究によって例証されます。ユーザーは、補足?...
ディスカッション
本研究は、二値分類のユーザー指定したデータセットに容易に続くマルチ ソリューション バイオ マーカー検出と評価プロトコルを提示します。ソフトウェアは、使いやすさと柔軟性のあるインポート/エクスポート インターフェイス ソフトウェアの GUI を使用して簡単にそのデータセットを調査する医学研究者を許可する、さまざまなファイル形式の重点を置きます。本研究では、以前多?...
開示事項
我々 は本レポートに関する利害の対立があります。
謝辞
この作品は、中国の科学 (XDB13040400) 院と吉林大学からスタートアップ助成金の戦略的な重点研究課題によって支えられました。匿名のレビューや生体テスト ユーザーは、使いやすさと kSolutionVis の機能の改善に関する建設的なコメントの認められました。
資料
| Name | Company | Catalog Number | Comments |
| Hardware | |||
| laptop | Lenovo | X1 carbon | Any computer works. Recommended minimum configuration: 1GB extra hard disk space, 1 GB memory, 2.0MHz CPU |
| Name | Company | Catalog Number | Comments |
| Software | |||
| Python 3.0 | WingWare | Wing Personal | Any python programming and running environments support Python version 3.0 or above |
参考文献
- Heckerman, D., et al. Genetic variants associated with physical performance and anthropometry in old age: a genome-wide association study in the ilSIRENTE cohort. Scientific Reports. 7, 15879 (2017).
- Li, Z., et al. Genome-wide association analysis identifies 30 new susceptibility loci for schizophrenia. Nature Genetics. 49, 1576-1583 (2017).
- Winkler, T. W., et al. Quality control and conduct of genome-wide association meta-analyses. Nature Protocols. 9, 1192-1212 (2014).
- Harrison, R. N. S., et al. Development of multivariable models to predict change in Body Mass Index within a clinical trial population of psychotic individuals. Scientific Reports. 7, 14738 (2017).
- Liu, J., et al. Multiple similarly-well solutions exist for biomedical feature selection and classification problems. Scientific Reports. 7, 12830 (2017).
- Ye, Y., Zhang, R., Zheng, W., Liu, S., Zhou, F. RIFS: a randomly restarted incremental feature selection algorithm. Scientific Reports. 7, 13013 (2017).
- Zhou, F. F., Xue, Y., Chen, G. L., Yao, X. GPS: a novel group-based phosphorylation predicting and scoring method. Biochemical and Biophysical Research Communications. 325, 1443-1448 (2004).
- Sanchez, B. N., Wu, M., Song, P. X., Wang, W. Study design in high-dimensional classification analysis. Biostatistics. 17, 722-736 (2016).
- Shujie, M. A., Carroll, R. J., Liang, H., Xu, S. Estimation and Inference in Generalized Additive Coefficient Models for Nonlinear Interactions with High-Dimensional Covariates. Annals of Statistics. 43, 2102-2131 (2015).
- Li, J. H., et al. MiR-205 as a promising biomarker in the diagnosis and prognosis of lung cancer. Oncotarget. 8, 91938-91949 (2017).
- Lyskjaer, I., Rasmussen, M. H., Andersen, C. L. Putting a brake on stress signaling: miR-625-3p as a biomarker for choice of therapy in colorectal cancer. Epigenomics. 8, 1449-1452 (2016).
- Ge, R., et al. McTwo: a two-step feature selection algorithm based on maximal information coefficient. BMC Bioinformatics. 17, 142 (2016).
- Tumuluru, J. S., McCulloch, R. Application of Hybrid Genetic Algorithm Routine in Optimizing Food and Bioengineering Processes. Foods. 5, (2016).
- Gen, M., Cheng, R., Lin, L. . Network models and optimization: Multiobjective genetic algorithm approach. , (2008).
- Radovic, M., Ghalwash, M., Filipovic, N., Obradovic, Z. Minimum redundancy maximum relevance feature selection approach for temporal gene expression data. BMC Bioinformatics. 18, 9 (2017).
- Ciuculete, D. M., et al. A methylome-wide mQTL analysis reveals associations of methylation sites with GAD1 and HDAC3 SNPs and a general psychiatric risk score. Translational Psychiatry. 7, e1002 (2017).
- Lin, H., et al. Methylome-wide Association Study of Atrial Fibrillation in Framingham Heart Study. Scientific Reports. 7, 40377 (2017).
- Wang, S., Li, J., Yuan, F., Huang, T., Cai, Y. D. Computational method for distinguishing lysine acetylation, sumoylation, and ubiquitination using the random forest algorithm with a feature selection procedure. combinatorial chemistry & high throughput screening. , (2017).
- Zhang, Q., et al. Predicting Citrullination Sites in Protein Sequences Using mRMR Method and Random Forest Algorithm. combinatorial chemistry & high throughput screening. 20, 164-173 (2017).
- Cuena-Lombrana, A., Fois, M., Fenu, G., Cogoni, D., Bacchetta, G. The impact of climatic variations on the reproductive success of Gentiana lutea L. in a Mediterranean mountain area. International journal of biometeorology. , (2018).
- Coghe, G., et al. Fatigue, as measured using the Modified Fatigue Impact Scale, is a predictor of processing speed improvement induced by exercise in patients with multiple sclerosis: data from a randomized controlled trial. Journal of Neurology. , (2018).
- Hong, H., et al. Applying genetic algorithms to set the optimal combination of forest fire related variables and model forest fire susceptibility based on data mining models. The case of Dayu County, China. Science of the Total Environment. 630, 1044-1056 (2018).
- Borges, D. L., et al. Photoanthropometric face iridial proportions for age estimation: An investigation using features selected via a joint mutual information criterion. Forensic Science International. 284, 9-14 (2018).
- Kohavi, R., John, G. H. Wrappers for feature subset selection. Artificial intelligence. 97, 273-324 (1997).
- Yu, L., Liu, H. Efficient feature selection via analysis of relevance and redundancy. Journal of machine learning research. 5, 1205-1224 (2004).
- Wexler, R. B., Martirez, J. M. P., Rappe, A. M. Chemical Pressure-Driven Enhancement of the Hydrogen Evolving Activity of Ni2P from Nonmetal Surface Doping Interpreted via Machine Learning. Journal of American Chemical Society. , (2018).
- Wijaya, S. H., Batubara, I., Nishioka, T., Altaf-Ul-Amin, M., Kanaya, S. Metabolomic Studies of Indonesian Jamu Medicines: Prediction of Jamu Efficacy and Identification of Important Metabolites. Molecular Informatics. 36, (2017).
- Shangkuan, W. C., et al. Risk analysis of colorectal cancer incidence by gene expression analysis. PeerJ. 5, e3003 (2017).
- Chu, C. M., et al. Gene expression profiling of colorectal tumors and normal mucosa by microarrays meta-analysis using prediction analysis of microarray, artificial neural network, classification, and regression trees. Disease Markers. , 634123 (2014).
- Fleuret, F. Fast binary feature selection with conditional mutual information. Journal of Machine Learning Research. 5, 1531-1555 (2004).
- Pacheco, J., Alfaro, E., Casado, S., Gámez, M., García, N. A GRASP method for building classification trees. Expert Systems with Applications. 39, 3241-3248 (2012).
- Jiao, X., et al. DAVID-WS: a stateful web service to facilitate gene/protein list analysis. Bioinformatics. 28, 1805-1806 (2012).
- Rappaport, N., et al. Rational confederation of genes and diseases: NGS interpretation via GeneCards, MalaCards and VarElect. Biomedical Engineering OnLine. 16, 72 (2017).
- Rebhan, M., Chalifa-Caspi, V., Prilusky, J., Lancet, D. GeneCards: integrating information about genes, proteins and diseases. Trends in Genet. 13, 163 (1997).
- Joosten, R. P., Long, F., Murshudov, G. N., Perrakis, A. The PDB_REDO server for macromolecular structure model optimization. IUCrJ. 1, 213-220 (2014).
- Maglott, D., Ostell, J., Pruitt, K. D., Tatusova, T. Entrez Gene: gene-centered information at NCBI. Nucleic Acids Research. 39, D52-D57 (2011).
- Amberger, J. S., Bocchini, C. A., Schiettecatte, F., Scott, A. F., Hamosh, A. OMIM.org: Online Mendelian Inheritance in Man (OMIM(R)), an online catalog of human genes and genetic disorders. Nucleic Acids Research. 43, D789-D798 (2015).
- Boutet, E., et al. the Manually Annotated Section of the UniProt KnowledgeBase: How to Use the Entry View. Methods in Molecular Biology. 1374, 23-54 (2016).
- Zerbino, D. R., et al. Ensembl 2018. Nucleic Acids Res. , (2017).
- McKusick, V. A., Amberger, J. S. The morbid anatomy of the human genome: chromosomal location of mutations causing disease. Journal of Medical Genetics. 30, 1-26 (1993).
- Finn, R. D., et al. The Pfam protein families database: towards a more sustainable future. Nucleic Acids Research. 44, D279-D285 (2016).
- Xue, Y., et al. GPS: a comprehensive www server for phosphorylation sites prediction. Nucleic Acids Research. 33, W184-W187 (2005).
- Deng, W., et al. GPS-PAIL: prediction of lysine acetyltransferase-specific modification sites from protein sequences. Scientific Reports. 6, 39787 (2016).
- Zhao, Q., et al. GPS-SUMO: a tool for the prediction of sumoylation sites and SUMO-interaction motifs. Nucleic Acids Research. 42, W325-W330 (2014).
- Wan, S., Duan, Y., Zou, Q. HPSLPred: An Ensemble Multi-Label Classifier for Human Protein Subcellular Location Prediction with Imbalanced Source. Proteomics. 17, (2017).
- Zhang, H., Zhu, L., Huang, D. S. WSMD: weakly-supervised motif discovery in transcription factor ChIP-seq data. Scientific Reports. 7, 3217 (2017).
- Szklarczyk, D., et al. STRING v10: protein-protein interaction networks, integrated over the tree of life. Nucleic Acids Research. 43, D447-D452 (2015).
- Chiaretti, S., et al. Gene expression profile of adult T-cell acute lymphocytic leukemia identifies distinct subsets of patients with different response to therapy and survival. Blood. 103, 2771-2778 (2004).
- Rowley, J. D., et al. Mapping chromosome band 11q23 in human acute leukemia with biotinylated probes: identification of 11q23 translocation breakpoints with a yeast artificial chromosome. Proceedings of the National Academy of Sciences of the United States of America. 87, 9358-9362 (1990).
- Rabbitts, T. H., et al. The chromosomal location of T-cell receptor genes and a T cell rearranging gene: possible correlation with specific translocations in human T cell leukaemia. Embo Journal. 4, 1461-1465 (1985).
- Yin, L., et al. SH2D1A mutation analysis for diagnosis of XLP in typical and atypical patients. Human Genetics. 105, 501-505 (1999).
- Brandau, O., et al. Epstein-Barr virus-negative boys with non-Hodgkin lymphoma are mutated in the SH2D1A gene, as are patients with X-linked lymphoproliferative disease (XLP). Human Molecular Genetics. 8, 2407-2413 (1999).
- Burnett, R. C., Thirman, M. J., Rowley, J. D., Diaz, M. O. Molecular analysis of the T-cell acute lymphoblastic leukemia-associated t(1;7)(p34;q34) that fuses LCK and TCRB. Blood. 84, 1232-1236 (1994).
- Taylor, G. M., et al. Genetic susceptibility to childhood common acute lymphoblastic leukaemia is associated with polymorphic peptide-binding pocket profiles in HLA-DPB1*0201. Human Molecular Genetics. 11, 1585-1597 (2002).
- Wadia, P. P., et al. Antibodies specifically target AML antigen NuSAP1 after allogeneic bone marrow transplantation. Blood. 115, 2077-2087 (2010).
- Wilson, D. M., et al. 3rd et al. Hex1: a new human Rad2 nuclease family member with homology to yeast exonuclease 1. Nucleic Acids Research. 26, 3762-3768 (1998).
- O'Sullivan, R. J., et al. Rapid induction of alternative lengthening of telomeres by depletion of the histone chaperone ASF1. Nature Structural & Molecular Biology. 21, 167-174 (2014).
- Lee-Sherick, A. B., et al. Aberrant Mer receptor tyrosine kinase expression contributes to leukemogenesis in acute myeloid leukemia. Oncogene. 32, 5359-5368 (2013).
- Guyon, I., Elisseeff, A. An introduction to variable and feature selection. Journal of machine learning research. 3, 1157-1182 (2003).
- John, G. H., Kohavi, R., Pfleger, K. . Machine learning: proceedings of the eleventh international conference. , 121-129 (1994).
- Jain, A., Zongker, D. Feature selection: Evaluation, application, and small sample performance. IEEE transactions on pattern analysis and machine intelligence. 19, 153-158 (1997).
- Taylor, S. L., Kim, K. A jackknife and voting classifier approach to feature selection and classification. Cancer Informatics. 10, 133-147 (2011).
- Andresen, K., et al. Novel target genes and a valid biomarker panel identified for cholangiocarcinoma. Epigenetics. 7, 1249-1257 (2012).
- Guo, P., et al. Gene expression profile based classification models of psoriasis. Genomics. 103, 48-55 (2014).
- Xie, J., Wang, C. Using support vector machines with a novel hybrid feature selection method for diagnosis of erythemato-squamous diseases. Expert Systems with Applications. 38, 5809-5815 (2011).
- Zou, Q., Zeng, J., Cao, L., Ji, R. A novel features ranking metric with application to scalable visual and bioinformatics data classification. Neurocomputing. 173, 346-354 (2016).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved