このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
イン・シトゥバイオプロセスにおける単細胞形態のリアルタイム測定のための顕微鏡検査
要約
細胞懸濁液中の単一細胞の大きさを直接監視するために、その上の顕微鏡装置の光光学を開発した。リアルタイム測定は、光光学滅菌可能プローブを自動画像解析に結合して行います。形態学的変化は、成長状態および栽培条件に依存して現れる。
要約
微生物バイオプロセスにおけるその過程でのモニタリングは、主に培地の化学的および物理的性質(例えば、pH値および溶存酸素濃度)に制限される。それにもかかわらず、細胞の形態は、成長状態、製品蓄積および細胞ストレスに依存して変化するので、最適な条件のための適切な指標となり得る。さらに、単一細胞サイズ分布は、栽培条件に関する情報だけでなく、集団の不均一性に関する情報も提供する。このような情報を得るために、生体反応器における細胞懸濁液中の単一細胞サイズ分布のモニタリングを直接可能にするために、その上の顕微鏡装置1における光光学を開発した。自動画像解析は、ニューラルネットワークモデルに基づいて顕微鏡に結合され、ユーザーに付け込まれた画像でトレーニングされます。顕微鏡の捕捉から得られるいくつかのパラメータは、代謝活性のような細胞の関連する特徴を処理するために相関している。これまで、その場で提示された顕微鏡プローブシリーズを適用して、糸状菌懸濁液中のペレットサイズを測定した。微細藻類培養における単細胞サイズを区別し、脂質蓄積に関連させるために用いられた。細胞粒子の形状は、酵母培養における出芽に関連していた。顕微鏡分析は、一般に、(i)画像取得、(ii)粒子同定、(iii)データ分析の3つのステップに分けることができます。すべてのステップは生物に適応する必要があり、したがって、信頼性の高い結果を達成するために特定の注意を引いた情報が必要です。細胞形態の変化を直接ラインまたはインライン(バイパス)で監視する機能により、プロセス開発や生産規模でのモニタリングと制御のためのリアルタイム値が可能になります。オフラインデータがリアルタイム データと相関している場合、セル サイズに対する未知の影響を持つ現在の退屈なオフライン測定は不要になります。
概要
細胞の形態学的特徴は、生理学的状態に関連することが多く、形態と機能との間の接続は、多くの用途に存在する。単一細胞の形態は、成長の状態、細胞の年齢、浸透性および他の潜在的な細胞ストレスまたは製品の蓄積の影響を受ける。細胞の形態学的変化は、多くの場合、培養物の成長活力の尺度である。細胞内産物合成は、藻類における脂質蓄積及び細菌における封入体形成、とりわけ、細胞サイズと同様に関連している。細胞凝集は、最近2を要約したように調査する価値がある別の要因であることができます。
集団不均一性は、個々の細胞の形態学的特徴に基づいて定量化することができる。研究は、培養中の不均一性が有意であり得るかもしれないことを示し、例えば、大規模な生産条件3の下では、全体の収量は、サブ集団4の低性能の影響を受ける可能性がある。
通常、細胞の形態学的特徴の評価は、手動サンプリングまたは光光学デバイスに結合されたバイパスフローチャンバによって行われる。これはいくつかの制限につながります:取得されたデータの限られた量は、統計的に信頼性の高い測定値をほとんど提供できません。サンプリングと結果のアクセシビリティの間の時間遅延は、プロセスのダイナミクスと比較して長すぎる可能性があります。そして最も重要なのは、サンプリング手順(サンプリングポートの位置、測定前のサンプルの前処理、サンプリングまたはバイパスチューブ内の好ましくない条件)は、サンプル手順自体がすでにセルに影響を与える可能性があるため、バイアス誤差を引き起こす可能性があります。形態。最後に、サンプリング中またはバイパスソリューションで汚染される危険性が常に高く、その場で滅菌できない場合があります。
in situ顕微鏡(ISM)の適用は、これらの問題のいくつかを回避することができます。細胞が自動的に検出された場合、その形態学的特徴の正しい同定を5.これまで、この方法の主な制限は、(i)画像の評価時間、situアプリケーションでは長すぎた、および(ii)特に高細胞密度で画像の解像度が低かった。ISMの最初の解決策は、機械的サンプリング、プローブの希釈、またはバイパスシステム6、7に制限されたが、さらなるアプローチは、細胞懸濁液を直接8の捕捉を可能にする。
ISMの最近の進歩は、単一細胞ベースで細胞のインラインまたはライン監視を可能にし、これはかなり高い細胞濃度で細胞懸濁液に直接リアルタイムで形態学的パラメータの分布を提供する。細胞の主要パラメータのオフライン分析を通じて、結合された自動細胞検出およびISMによって提供される情報との相関関係を同定することができる。次に、単一細胞形態で測定不能なパラメータを推定する新しいソフトセンサ設計が達成されます。
このレポートでは、ISM は光光学プローブを自動画像解析に結合することによって行われます。ISMは、高解像度CCDカメラで調整可能な測定ギャップで既知の焦点範囲内の画像のキャプチャを可能にするシングルロッドセンサプローブで構成されています[MM-Ho = CCD GT2750(2750x2200)およびMM 2.1 = CMOS G507c(2464x2056)]。フラッシュライト照明はトランスミッションによって行われます。従って、光はカメラ9の反対側から起点となり、その強度を調整することができる。細胞は液体の流れとこのギャップを連続的に通過する。したがって、代表的なサンプル母集団が得られる。プローブは、細胞懸濁液に到達するようにバイオリアクターに直接取り付けることができ、または滅菌可能なバイパスで使用することができます。センサシェルは殺菌前にシステムに接続され、光学部品はその後シェルに取り付けられる。
これまで、関連する工業用微生物、例えば、糸状菌(直径200μm以上)、異栄養性微細藻類クリプトヘコディニウムコニウム(平均細胞径20μm)、および酵母サッカロミセス・セレビシエ(平均細胞径5μm)を、この装置または類似の装置で調査した。
糸状菌は、特定の栽培条件下でペレットを形成する傾向がある。これらは数百μmまでの大きさです。真菌細胞のヒファは、流体相における流体力学的ストレスへの依存性において異なる長さを発達させる。これは、代謝および成長活性、基質の取り込みおよび製品放出に影響を及ぼす。ISMは、ペレットの大きさ分布とペレットの端における低バイオマス密度のゾーンの幅を同定するために適用された(独自の未公開データ)。
C.コニの大きさは、細胞が窒素制限の下で多価不飽和脂肪酸ドコサヘキサエン酸(DHA)を蓄積すると15~26μmの間で変化する。このバイオテクノロジーDHA製造プロセスは、細胞が分裂して小さくなる成長段階と、細胞が生成物を蓄積して大きくなる生産段階の2つの部分で構成されています。したがって、細胞サイズは、増殖またはDHA産生のいずれかが良好であったプロセス状態を決定するために使用された。最後に、細胞サイズとDHA含有量との相関関係が見つかった。この場合、ISMは、サンプリング、細胞破壊、および一般的なガスクロマトグラフィー分析10を必要とせずに、細胞内DHA蓄積をリアルタイムで監視することができる。
出芽酵母は、通常、3〜8μmの間のサイズです。一度に成熟状態にある細胞の割合は、出芽指数(BI)で説明されるように、増殖活力11、12、及び組換えタンパク質分泌との関係に関する情報を提供し、13であることが証明されている。ISMの助けを借りて、出芽および非出芽酵母細胞(芽の有無にかかわらず細胞)を区別した14.ストレス条件はまた、酵母集団内の細胞サイズのより広範な変動をもたらし得るが、最近スケールダウン栽培で示されているように、大規模な栄養限定飼料栽培の条件を模倣した3。
したがって、ISMは、最適な培養条件の同定、またはプロセス制御の目的のために、バイオプロセスのすべての段階において、単一細胞レベルで増殖活力および製品形成を監視する可能性を有する。ここで説明する方法は、単一細胞を用いた微生物用途に焦点を当てていますが、ヒトおよび動物細胞、細胞凝集体および糸状生物のペレットのようなより大きな粒子にも適用可能です。
プロトコル
注:パラメータをそれぞれの微生物および培養条件に適合させるためには、以下のステップが必要です。プローブ設定の調整は、経験豊富なユーザーのために約20分間続きます。ツールと手順の詳細な説明は、SOPAT GmbH の対応するプローブマニュアルに記載されています。一般に、次のプロトコルで示されるツールが必要です: (i) プローブ調整および画像取得用のプローブコントローラ。(ii) 取得した画像に対する注釈のためのフィジー(ImageJ)(iii) 人工ニューラルネットワーク(ANN)トレーニングおよびワークフロー作成のためのSOPATサポート(iv) ワークフローで既に取得した画像を使用したデータバッチ処理のためのBatcher。(v) バッチ処理された画像の結果の視覚化と評価のための結果アナライザー。(vi) 自動リアルタイム測定と結果可視化のためのモニター 。
1. ハードウェアパラメータの設定
- 実験または遠心分離機中に達成される可能性のある最も高い細胞濃度の培養を準備し、この濃度を達成するためにペレットを再中断します。この場合、乾燥バイオマス濃度の65gL-1がS.セレビシエ栽培に選択された。
- 予想される範囲が完全に覆われるように、最高濃度から最低濃度までの範囲の異なる希釈を準備します。少なくとも4種類の濃度をお勧めします。
- 従来の顕微鏡法で微生物の細胞サイズ範囲を同定する。各セルの予想最大直径(dmax)を定義します。この値は、S. セレビシエの場合は 8 μm に設定されます。
- セルの予想dmaxの5xと10xの2つの測定ギャップを選択します。
- 最大ストロボスコープ強度を選択します。最も高いセル濃度のギャップに対してストロボスコープの強度を選択し、光強度が最も低い画像 (最も暗い画像) でセルが引き続き表示されるようにします。
- ストロボスコープの最小強度を選択し、両方のギャップにストロボスコープの強度を選択して、光強度が最も高い画像(最も明るい画像)でセルが引き続き表示されるようにします。最も低いセル濃度を使用します。
- ストロボスコープの強度とテストが必要な濃度範囲の両方について、各測定ギャップに対して最も鮮明な画像を生成する 1 つのフォーカス位置を選択します (フォーカスの詳細については、ステップ 2 を参照してください)。後で画像データにコメントが付けられるように、セルに適切に焦点を当てます(ステップ4を参照)。
- ギャップ幅とストロボスコープ強度の両方で、以前に調製した細胞濃度の希釈シリーズ(ステップ2を参照)を測定します。
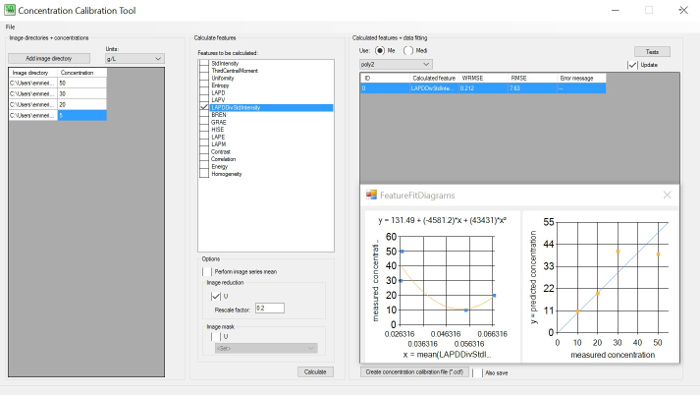
図1:濃度キャリブレーションツール左 GUI: 既知の濃度でイメージ ディレクトリ (最小 3) を設定します。中央 GUI: イメージ ディレクトリで計算する機能を選択します。右の GUI: 加重二乗平均平方誤差 (WRMSE) を選択して最小値を識別します。WRMSEと任意の画像特徴と細胞濃度との間の最良の相関。この図の大きなバージョンを表示するには、ここをクリックしてください。
- 希釈系列実験を評価します。
- 濃度キャリブレーション(CoCa)ツール(図1参照)を使用して、抽出された画像特徴(明るさまたはシャープネス)と、ユーザーが提供する以前に測定された濃度(例えば、ドライバイオマスまたは細胞数)との間の最適な相関関係を特定します。詳細については、ソフトウェアマニュアルの指示に従ってください。
- 画像フィーチャから抽出された情報と、オフライン測定と比較して、さまざまな濃度で抽出された情報間の最適な相関関係を特定します。図 1の凡例を参照してください。
- 濃度相関曲線を考慮した中で最も合理的な測定ギャップとストロボスコープ強度を選択し、最小の加重二乗平均平方誤差(WRMSE)を生じる特徴を持ちます。
注:測定ギャップは実験中に固定され、ストロボスコープの強度は細胞濃度に応じて調整することができます。
2.オフライン測定
- 厚さ計の助けを借りて、ステップ1に従って所望の測定ギャップを調整します。
- SOPAT ダッシュボードでグラフィカル ユーザー インターフェイスプローブ コントロールを開きます。
- ソフトウェアサブセクションのアンプに目的のプローブを接続し、接続を押します。
- 再生ボタンを押してストリーミングを開始します (ライブ ビュー)。
- エタノールを隙間に噴霧して測定ギャップをきれいにし、光学紙でほこりや汚れを丁寧に拭き取ります。センサーのガラスにCamControlのライブ ビューを使用してパーティクルが含めなくなっていることを確認します。
メモ:粒子とほこりの乱れ測定と自動セル識別。 - 測定ギャップにドライ光学用紙を入れ子にします。[プローブコントロール]タブを開き、ストロボスコープの強度を調整して用紙を視覚化します。用紙の単一の繊維がはっきりと見えるまで、結合ネジを回します。
- 培養スープでチューブを充填します。培養液に顕微鏡を浸し、隙間が細胞懸濁液で完全に覆されるようにします。タブプローブコントロールを開き、セクション1に従って目的のストロボスコープ強度を調整します。フォーカス結合ねじを微調整してセルに焦点を当てます。実験中にフォーカスを変更してはいけません
注:5〜6 mLの培養液を50mL円錐円錐円管に添加し、測定ギャップを十分に浮かべる。 - トリガーごとの GUI フレームでトリガーするメニューで、ユーザー インターフェイスのタイム ポイント [-]あたりのフレーム数を定義します。トリガーごとにフレーム数を 200 フレームに設定します。
注: フレーム数は、統計的に信頼できる結果に必要な最小値に減らすことができます。これは、代表的な形態細胞サイズ分布を得るために必要なサンプルサイズに依存する(ステップ5も参照)。 - GUI フレームレートでトリガするメニューでフレームレート[Hz]を定義します。前のフレームから移動するパーティクルが次のフレームに表示されないことを保証するフレーム レートを選択します。
注:これは200フレームのテストトリガで証明することができます。繰り返しキャプチャされるパーティクルのイメージを検査します。この場合は、フレームレートを下げます。オフライン測定の場合は、1 Hz をお勧めします。 - 取得したイメージを保存するディレクトリを[一般]メニューで設定します。
- [イメージ トリガー取得の開始] ボタンをアクティブにして、イメージの取得を実行します。培養懸濁液でチューブをゆっくりと上下に動かして、測定ギャップを通る流れを誘導します。
- 各測定後にステップ2.5を繰り返します。
- 取得した画像を確認します。セルは注釈に十分な鋭さでなければなりません。繰り返しキャプチャされるパーティクルのイメージを検査します。この場合は、フレームレートを下げます。
- 次の経路を選択して設定を保存します。
3. 粒子識別
- 人工ニューラルネットワーク(ANN)(トレーニングセット)のトレーニング用に粒子に注釈を付けます。
- 取得したイメージをアノテーション ツール "フィジー, ImageJ"に読み込むには、ファイルをメイン "ImageJ" ウィンドウにドラッグ アンド ドロップします (図 2の GUI "Fijiツールを参照)。
- ROI マネージャを開くには、[分析] |ツール |ROI マネージャ:
- 選択ツールを選択します。ワンド(トレース)ツール、フリーハンド、楕円形、または楕円形の選択をお勧めします。
- 前に説明した選択ツールでアテンションされるパーティクルの周囲に円を描画し、ブラシ ツールで調整します。
- [t]を押して、ROI マネージャに注釈を追加します。
- 約 15 個の画像に対象のすべてのオブジェクト (識別対象のセル) をマークします。
注:すべての必要な情報、すなわち、異なる形状、サイズ、細胞の濃度、明るさなどをカバーするために、最初から5つの画像、その間から5つの画像、および実験の終わりから5つの画像を使用します。 - セルの形状 (例えば、セル周期の異なるフェーズ) のために、セルを異なるサブクラスに分類する必要があるかどうか、またはすべてのセルが同じクラスであるかどうかを決定します。
- 選択した各パーティクルの名前を適宜変更します。クラスごとに 1 つの名前または省略形を設定し、クラスのパーティクルごとにカウンタ(cell_1、cell_2など) を設定します。クラスごとに少なくとも 50 個のパーティクルに注釈を付ける。
- 気泡や、未溶解のメディアコンポーネントなどの他の粒子など、プロセスに関連しないため、検出してはならないオブジェクトに注釈を付けないでください。
注: これらのイベントは ANN のトレーニング手順に含まれず、バックグラウンドと見なされます。 - 焦点が合っていないセルに注釈を付けないでください。
- 画像に可能な限り一貫性のある注釈を付けます。疑問がある場合は、[無視]というラベルを適用できます。ANN はラベル付けされた構造のみを認識するため、使用を乱用しないことを強くお勧めします。

図 2: フィジー ツールのユーザー インターフェイス警告されたイメージを使用してトレーニング セットが作成されます。2 つのクラスで構成される手動注釈が示され、注釈付きパーティクルのリストが ROI マネージャに表示されます。クラスごとに異なる名前と色を設定できます。この図の大きなバージョンを表示するには、ここをクリックしてください。
- 通知されたオブジェクトと画像を ZIP 形式で保存し、プラットフォームへのアップロードまたは電子メールで ZIP ファイルを送信して、トレーニング ネットワークにファイルを送信します。
注: 通常、画像上の分類されたオブジェクトの適切な予測を識別するには、多くの反復トレーニングラウンドが必要です。各トレーニング ラウンドは、プログラムによって返されるワークフローにつながります。 - トレーニング済みのオブジェクト認識アルゴリズムでワークフロー (*.wf) を使用して、ダッシュボードで開始できるデータ バッチ進行状況プログラムBatcherでテスト イメージを分析します。
- 誤検知イベントとネガティブイベントの定量化を通じて、テスト画像のオブジェクト検出を確認します。
- 誤検知事象の検出を定量化する:粒子が誤って細胞として検出され、正しく分類されていない細胞、および輪郭が十分に識別されていない細胞。
- 偽陰性イベント (そのように認識されないセル) を定量化します。
- ダッシュボードでプログラムを起動して、ツールの結果アナライザーで結果を視覚化します。
- 目的の結果ファイルを[ファイル]を使用して読み込む |ファイルまたはファイルを読み込む |フォルダのインポート:
- グラフで結果を視覚化する |グラフGUI でグラフを作成します。
- 分布チャート、感度プロット、時間の経過に従う特性、測量ポイント上の特性、およびフィーチャーとフィーチャーのいずれかのオプションを選択します。
メモ:結果アナライザの使用に関するマニュアルは、システムに付属しており、サポートからも入手できます。 - 結果が受け入れられる場合は、実験のすべての取得画像に対してBatcherでワークフローを実行します。同時に、プローブコントローラ(*.pcfg)から保存された設定をワークフロー(*.wf)と組み合わせることで、監視プログラムを作成できます。
注: このワークフローは、このカルチャ メディアの今後の実験を監視するためにも使用できます。 - 結果が受け入れられない場合は、トレーニング セットの注釈を確認するか、別の反復トレーニング ラウンドを続行します (手順 4.2 を参照)。
4. サンプルサイズ定量
- 検出されたパーティクル間で許容される標準偏差(σ)を設定します。
注: 標準偏差は、セル サイズの均質性に平行に変化します。最大標準偏差は、サイズの不均一度が最も高いサンプルを示します。 - 期待される測定値の分散(e)に対して、信頼区間の振幅または所望の精度を設定します。
- 認められた誤差(α)を5%(z1-α/2 = 1.96)から10%(z1-α/2 = 1.64)の間に設定します。
- 方程式 1 から各クラスから識別されるセルの数を計算します。
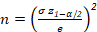 [方程式 1]
[方程式 1]
注: セルの数に基づいて、取得する必要があるイメージの数をデータ ポイントごとに定義できます。 - 実験のランダムタイムポイントに対して感度解析を実行し、n個の粒子の分析が平均フェレット直径とDv90の変動性が5%未満であることを確認します。結果アナライザで自動的に計算できます。
5.オンライン(バイパス)またはインライン測定
- まず、ハードウェアとソフトウェアの設定を生物とプロセス(濃度または媒体)の関数として設定するために、まずオフライン測定手順を実行します(ステップ2を参照)。
- 前のセクションから保存した設定をアップロードするには、[読み込み] ボタンを選択し、次のパスを選択します。
- プローブをフローセルまたはバイオリアクターに接続します。
メモ:その場合、ピンチフランジで測定を行うことができます。 - 殺菌を行う。
メモ:器械の調査ぬれた材料だけが蒸気殺菌によって殺菌可能である。プローブの濡れた長さは 6 ~ 222 mm です (図 3)。
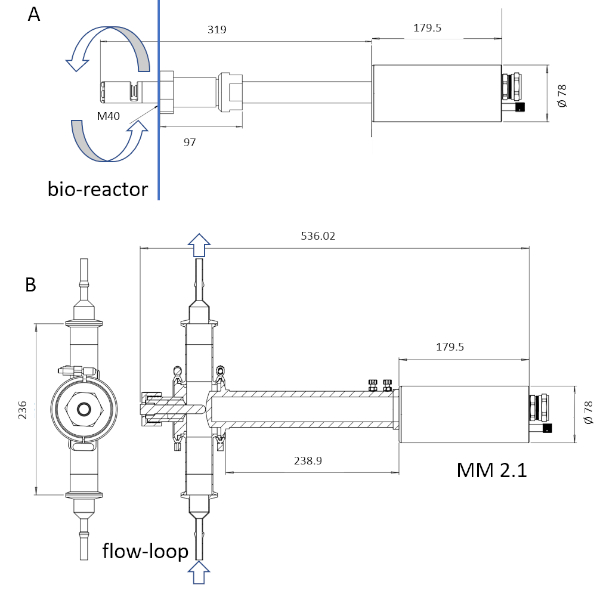
図 3: ISM デバイスのスケッチプローブMM-Ho(A)はバイオリアクターに直接取り付け可能であるのに対し、プローブMM 2.1(B)はバイパスとして使用できます。培養ブロス循環は、各画像に矢印でマークされています。変換係数は、MM-Ho の場合は 0.166 μm pix-1、MM 2.1の場合は 0.087 μm pix-1です。この図の大きなバージョンを表示するには、ここをクリックしてください。
- [トリガ間隔 [s]」フィールドで、GUIトリガで画像取得率を定義します。
注:プロセスダイナミクスに応じて、画像取得率を調整することができます。例えば、3時間のラグフェーズが予想される場合、新陳代謝シフトや製品の蓄積を監視する場合よりも、取得率を低くすることができます。通常、これは分の範囲ではるかに短い取得時間を必要とします。例えば、バッチ酵母栽培中に5〜10分の間の画像取得シーケンスは、プロセスダイナミクスを捕捉するのに十分な情報を提供する。 - 手順 2.10 の説明に示すように、フレーム レートを定義します。
注:機械的な攪拌はギャップを通して流量を増加させることができるので、ラインおよびライン測定では、フレームレートを増加させることができます。 - [選択したプローブのストリーミングを開始する] ボタンをアクティブにしてイメージの取得を開始します。
- [選択したプローブのストリーミングを停止する] ボタンを使用して実験が終了したら、取得を停止します。
注:1回目の実行では、培養の接種直前に取得を開始し、ステップ4に進みます。次の実行では、ダッシュボードで[監視] プログラムを開き、作成したワークフローを選択します (手順 4 を参照)。[再生] ボタンを押して、カルチャの接種直前に監視を開始します。
結果
ISMによる酵母培養における細胞サイズ検出と、出芽細胞と非出芽細胞を区別する自動画像検出が成功した。ストロボスコープの強度と測定ギャップの選択の両方に許容範囲があり、粒子同定は影響を受けません。例えば、S.セレビシエ細胞は、4gL-1の乾燥バイオマス濃度で11%の変動範囲内で種々のストロボスコープ強度で測定した。対応する画像は鋭い?...
ディスカッション
同一または非常に類似したデバイスをここで提示するISMは、真菌、微細藻類、酵母細胞の形態学的ダイナミクスを測定するために使用され、増殖活性の決定を可能にし、藻類の場合には細胞内産物蓄積を可能にした。センサーは可動部品を持っておらず、標準的なポートを通して、または滅菌可能なバイパスで、任意の標準的な攪拌タンクバイオリアクターに直接適用可能です。酵母は藻類?...
開示事項
著者は申告するものが何もない。
謝辞
著者らは、ZIM-Koopの枠組みにおけるドイツ連邦経済エネルギー省の支援に感謝している。ZF 4184201CR5.
資料
| Name | Company | Catalog Number | Comments |
| Sensor MM 2.1 - MFC | SOPAT GmbH, Germany | n.a. | Inline Monocular Microscopic probe Version 2.1 with a Mirco Flow Cell |
| Sofware version v1R.003.0092 | SOPAT GmbH, Germany | n.a. | |
| Thickness gauge | n.n. | It can be any supplier, DIN 2275:2014-03 | |
| Ethanol 70% | n.n. | It can be any supplier | |
| SOPAT manual Version 2.0.5 | SOPAT GmbH, Germany | ||
| Optical lense paper | VWR | 470150-460 | |
| Fiji, ImageJ | open source | ||
| 50 mL conical centrifuge tubes | It can be any supplier |
参考文献
- Maaß, S., Rojahn, J., Hänsch, R., Kraume, M. Automated drop detection using image analysis for online particle size monitoring in multiphase systems. Computers & Chemical Engineering. 45, 27-37 (2012).
- Lemoine, A., Delvigne, F., Bockisch, A., Neubauer, P., Junne, S. Tools for the determination of population heterogeneity caused by inhomogeneous cultivation conditions. Journal of biotechnology. 251, 84-93 (2017).
- Marbà-Ardébol, A. M., Bockisch, A., Neubauer, P., Junne, S. Sterol synthesis and cell size distribution under oscillatory growth conditions in Saccharomyces cerevisiae scale-down cultivations. Yeast. 35 (2), 213-223 (2017).
- Xiao, Y., Bowen, C. H., Liu, D., Zhang, F. Exploiting nongenetic cell-to-cell variation for enhanced biosynthesis. Nature chemical biology. 12 (5), 339-344 (2016).
- Beutel, S., Henkel, S. In situ sensor techniques in modern bioprocess monitoring. Applied microbiology and biotechnology. 91 (6), 1493 (2011).
- Belini, V. L., Wiedemann, P., Suhr, H. In situ microscopy: A perspective for industrial bioethanol production monitoring. Journal of microbiological methods. 93 (3), 224-232 (2013).
- Havlik, I., et al. Monitoring of microalgal cultivations with on-line, flow-through microscopy. Algal Research. 2 (3), 253-257 (2013).
- Suhr, H., Herkommer, A. M. In situ microscopy using adjustment-free optics. Journal of biomedical optics. 20 (11), 116007 (2015).
- Panckow, R. P., Reinecke, L., Cuellar, M. C., Maaß, S. Photo-Optical In-Situ Measurement of Drop Size Distributions: Applications in Research and Industry. Oil Gas Sci. Technol. - Rev. IFP Energies. 72 (3), 14 (2017).
- Marbà-Ardébol, A. -. M., Emmerich, J., Neubauer, P., Junne, S. Single-cell-based monitoring of fatty acid accumulation in Crypthecodinium cohnii with three-dimensional holographic and in situ microscopy. Process Biochemistry. 52, 223-232 (2017).
- Porro, D., Vai, M., Vanoni, M., Alberghina, L., Hatzis, C. Analysis and modeling of growing budding yeast populations at the single cell level. Cytometry. Part A : the journal of the International Society for Analytical Cytology. 75 (2), 114-120 (2009).
- Brauer, M. J., et al. Coordination of growth rate, cell cycle, stress response, and metabolic activity in yeast. Molecular biology of the cell. 19 (1), 352-367 (2008).
- Puxbaum, V., Gasser, B., Mattanovich, D. The bud tip is the cellular hot spot of protein secretion in yeasts. Applied microbiology and biotechnology. 100 (18), 8159-8168 (2016).
- Marbà-Ardébol, A. M., Emmerich, J., Neubauer, P., Junne, S. Vol. P2. Prozessmesstechnik. , 222-225 (2017).
- Marbà-Ardébol, A. -. M., Emmerich, J., Muthig, M., Neubauer, P., Junne, S. Real-time monitoring of the budding index in Saccharomyces cerevisiae batch cultivations with in situ microscopy. Microbial cell factories. 17 (1), 73 (2018).
- Marquard, D., Schneider-Barthold, C., Düsterloh, S., Scheper, T., Lindner, P. Online monitoring of cell concentration in high cell density Escherichia coli cultivations using in situ Microscopy. Journal of biotechnology. 259, 83-85 (2017).
- Marquard, D., et al. In situ microscopy for online monitoring of cell concentration in Pichia pastoris cultivations. Journal of biotechnology. 234, 90-98 (2016).
- Camisard, V., Brienne, J., Baussart, H., Hammann, J., Suhr, H. Inline characterization of cell concentration and cell volume in agitated bioreactors using in situ microscopy: application to volume variation induced by osmotic stress. Biotechnology and bioengineering. 78 (1), 73-80 (2002).
- Böhm, A., Ücker, A., Jäger, T., Ronneberger, O., Falk, T. ISOODL: Instance segmentation of overlapping biological objects using deep learning. , 1225-1229 (2018).
- Davey, H. M. Life, Death, and In-Between: Meanings and Methods in Microbiology. Applied and environmental microbiology. 77 (16), 5571-5576 (2011).
- Lodolo, E. J., Kock, J. L., Axcell, B. C., Brooks, M. The yeast Saccharomyces cerevisiae-the main character in beer brewing. FEMS yeast research. 8 (7), 1018-1036 (2008).
- Albertin, W., et al. Population size drives industrial Saccharomyces cerevisiae. alcoholic fermentation and is under genetic control. Applied and environmental microbiology. 77 (8), 2772-2784 (2011).
- Gomes, J., Chopda, V. R., Rathore, A. S. Integrating systems analysis and control for implementing process analytical technology in bioprocess development. Journal of Chemical Technology and Biotechnology. 90 (4), 583-589 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved