Method Article
新しいウイルスのゲノムを発見する感染葉からの RNA の解析とウイルス粒子粗抽出 DNA の結合解析
要約
二本鎖 DNA のゲノムを持つ植物ウイルスを識別するために新しいアプローチをご紹介します。我々 は感染葉から DNA や RNA を抽出し、次世代シーケンシングを行う標準的な方法を使用します。Bioinformatic ツールは、コンティグにシーケンスを組み立てる、ウイルスのゲノムを表すコンティグを識別し、分類グループにゲノムを割り当てます。
要約
このメタゲノム アプローチは、環状 DNA ゲノムを持つ植物ウイルスとそのトラン スクリプトを識別するために使用されます。機械的に別のホストに再接種することはできませんまたはそのホストで低価で発生する植物 DNA ウイルス頻繁伝染性材料の大きい価を達成するために伝達が困難です。感染した葉は、最適な pH とイオン組成ほとんど被膜パラ レトロ ウイルスを浄化するためお勧めします穏やかなバッファーの地です。尿素は、ウイルス粒子をトラップ封入体を分割する、細胞成分を溶解するために使用されます。さらに差動遠心分離は植物の汚染物質からのウイルス粒子の分離を提供します。プロテイナーゼ K 処理、カプシドを削除します。その後、ウイルス DNA は集中、次世代シーケンサー (NGS) の使用します。NGS データは生成された dataset にウイルスの配列のサブセットを識別する NCBI BLASTn を投稿されたコンティグをアセンブルに使用されます。並列パイプラインの RNA は標準の列に基づく RNA 抽出法を用いて感染葉から分離されます。その後、リボソーム枯渇は mRNA およびウイルスの転写産物のサブセットを豊かにする行われます。RNA シーケンス (seq RNA) から派生した組み立てシーケンスは、このデータセットにウイルスの配列のサブセットを識別する NCBI BLASTn を堤出されました。我々 の研究では、2 つのデータセットの 2 つの関連のフルレングス badnavirus ゲノムを識別されます。植物ウイルスのゲノム配列を再構成する小さな rna の総人口を抽出する別の一般的なアプローチのこのメソッドお勧めします。この後者のメタゲノム パイプライン回復ウイルス関連シーケンス レトロ-議事録の作成要素は、植物ゲノムに挿入されます。これは、さらに積極的に感染性病原体を識別する生化学的または分子アッセイと結合されます。この研究に記載されているアプローチは、可能性が高いアクティブなウイルスに感染しているウイルスを複製する代表的なシーケンスを回復します。
概要
新たな病害はドライブ正しい因果エージェントを特定する新しいツールを開発する研究者です。新規または定期的なウイルス病の最初の報告は、モザイク、リーフ、静脈をクリア、小人症、萎れ、病変、壊死、奇形など一般的に発生する症状や他の症状に基づいています。病気の因果のエージェントが他の汚染病原体と区別するため、新しいウイルスを報告のための標準は適切なホストに伝達し、元ホスト種の健康な植物に接種によって病気を再現します。この方法では制限が多く属植物ウイルスが昆虫または適切なホストしたり、元の宿主への伝送のための他のベクトルに依存しています。この場合、適切なベクターを検索することができます長期化するベクトルの所コロニーを確立する難しさがあります、さらに努力が実験的伝送のプロトコルを考案する必要。成功伝送研究のための条件を達成できない場合、作業を下回る標準新しいウイルス病を報告するため。非常に低価で彼らの自然なホストで発生するウイルス、研究者が研究を遂行するための十分な感染株を維持するために伝播のための代替ホストを指定する必要があります。少数の植物だけに感染するウイルス種のこの株式文化1を成長のための障害することができます。
近年、科学者は、高スループットにより、NGS とメタゲノム アプローチ知られている病気に関係のない場合がありますが、分類学上の種と属に割り当てることが環境に存在するウイルスの配列を明らかにするに多く採用します。2,3,4. 検出および異なる環境で遺伝物質の分類にそのようなアプローチ ウイルス多様性自然の中や特定の生態系にその存在を記述する方法を提供しますが必ずしもを定義するフレームワークを確認していません。明らかな病気の発病。
Badnavirus属は、pararetroviruses のCaulimoviridae家族に属しています。これらのウイルスは、約 7 に 9 kb の円形の二重鎖 DNA のゲノムを持つ図形の被膜です。すべての pararetroviruses は、RNA 中間を介して複製されます。Pararetroviruses はエピソームとして存在し、植物の染色体 DNA5,6の独立した複製。これらのウイルスの集団遺伝的複雑なウイルス集団の野外研究を示します。さらに、植物ゲノムの範囲にわたって高スループット シーケンスによって得られる情報には、非嫡出統合イベントによって植物のゲノムに挿入される badnavirus ゲノム フラグメントの多数の例が発見されています。これらの内因性の badnavirus シーケンスが必ずしも感染症7,8,9,10,11に関連付けられてあります。その後、病の因果剤として新しい badnaviruses を識別するために NGS の使用 episomal ゲノムの個体の多様性だけでなく、内因性シーケンス12,13の発生複雑です。
新規 pararetrovirus ゲノムの検出のない 1 つの最適なパイプライン、病の因果剤としてこれらのウイルスを識別するために 2 つの一般的なアプローチがあります。1 つの方法感染葉から小さな RNA シーケンスを豊かにし、ウイルス genome(s)14,15,16,17を再構成するこれらの系列を組み立てることです。別のアプローチは、ローリング サークル増幅 (RCA) 円形の DNA ウイルスのゲノムの18を増幅します。RCA の成功は、葉と選択した組織にウイルス力価の年齢によって異なります。RCA の製品は制限の消化力を受ける、直接19,20,21を塩基配列のプラスミドにクローニングします。
カンナ黄色モザイク ウイルス(CaYMV) は、badnavirus をゲノムの 565 の bp フラグメントだけがずっと以前に感染したカンナ22から分離したカンナ、黄斑疾患の病因として記載されています。ゲットウにともなって(開花ジンジャーの CaYMV 識別の現代的な研究CaYMV-Ap)23。本研究の目的は、感染したカンナ ユリから完全な badnavirus のゲノム配列を回復するだった。我々 は工場の汚染物質からウイルスを浄化し、その後この準備からウイルス DNA を隔離するためのプロトコルを記述し、NGS 用 DNA ライブラリを準備します。このアプローチは、中間分子増幅の手順の必要性を排除できます。我々 はまた、RNA シーケンス NGS の感染植物から mRNA を分離各核酸準備を使用して実施された RNA シーケンスが含まれています。組み立てられたコンティグ核酸 (BLASTn) バイオ テクノロジーと情報 (NCBI) の基本的なローカル配列検索ツールのナショナル センターを使用して両方のデータセットのBadnavirus分類群に関連する発見されました。2 つの badnavirus 種24のゲノムを同定しました。
プロトコル
1 コビーらによって標準的な方法を使用して差動遠心分離による一般ウイルス浄化25
- まず、病気にかかった植物からの葉の 80-100 g を切るし、200 mL のバッファーを研削を使用して 4 ° C でローカルワ リング ミキサーで挽く (0.5 M NaH2PO4, 0.5 M Na2HPO4 (pH 7.2). 0.5% (w/v) Na2SO3)。実験室のコートおよびこの手順のすべてのステップのための手袋を着用します。
- その後、1.0 L ビーカーに磨砕液 (300 mL) を転送します。化学フード内ホモジネートする尿素の 18 g と 10% 非イオン性洗剤 (t10 月 C6H4-(OCH2CH2)9オハイオ) 25 mL を追加します。
注: この手順お勧め安全メガネおよび個人的な保護のための簡単な呼吸マスクを着用します。 - 簡単にフードのマグネチックスターラーで攪拌し、箔とビーカーをカバーします。寒い部屋に覆われて箔ビーカーを転送し、4 ° C で一晩マグネチックスターラーで攪拌
- 遠心分離機の回転子ボトル (250 mL 容器) と、固定角ロータの 4,000 x g で 4 ° C で 10 分間遠心するホモジネートを転送します。化学の発煙のフード、上澄みと寒冷紗の 4 層を介してフィルターを回復します。
- 38.5 mL ポリプロピレン製遠心管と 2.5 時間 4 ° C で 40,000 × g で遠心分離機の中で磨砕液を分割します。通常、管の下部に緑餌、チューブの長さに沿って白いペレットの存在を確認します。上清を離れて注ぎ、両方のペレットを保持氷の上にサンプルを配置します。
注: 緑のペレットには、葉緑体、澱粉および他の細胞器官が含まれています。 - 化学フードで働いて、ゴム警官を使って餌を区切ります。懸濁液は一晩ソリューションに完全に溶解するため材料を許可する 4 ° c を維持しながら 1-2 時間にわたって ddH2O の 1 mL にそれぞれのローターのボトルに白いペレットを再懸濁します。6,000 g x と残りの残骸を削除する 10 分のための 4 ° C での懸濁液を遠心します。
- 遠心分離機のウイルス粒子をペレットへの 4 ° C で 2 時間 136,000 x g で濃厚。バッファー (50 mM トリス-HCl、pH 7.5、5 mM MgCl2) 1 mL にペレットを再懸濁します。
注: 省略可能な手順は DNAse とウイルス粒子を扱う、私は非以上 DNA、つまり、葉緑体およびミトコンドリア DNA 汚染することを削除する 37 ° C で 10 分間 (10 μ G/ml)。DNAse を不活性化し、私は 1 mm EDTA を追加します。 - 15 分の 37 ° C で 2 μ g/μ L プロティナーゼ K の 40 μ L でウイルス粒子を混乱させます。
- 有機抽出によるウイルス粒子の DNA を回復する化学フード内部の作業します。急性の健康影響に対する保護のため抽出時に研究室コート、手袋、顔面シールドを着用します。サンプルにフェノール ・ クロロホルム ・ イソアミル アルコール (49:50:1) の 1 ボリュームを追加、16,000 x g に 5 分は上層の水相と新しい管への転送を削除するため、室温で 20 s. 遠心の手で振る。この抽出 2 回以上を繰り返します。制度的化学廃棄26ガラスの廃瓶に置くことで有機相を破棄します。
- エタノールの沈殿物を使用して DNA を集中します。0.3 M 酢酸ナトリウム (pH 5.2) の最終的な集中を使うと 95% のエタノールの 2.5 倍の量。-20 ° C、30-60 分および DNA26のペレットを 10 ~ 20 分 13,000 × g で遠心分離にサンプルを配置します。
- 研究室のベンチで働いて、1 mL の 0.1 mM TE バッファー (pH 8.0) で DNA ペレットを再懸濁します。ファイラー (通常使用されるポリメラーゼの連鎖反応 (PCR) のクリーンアップ) 商業ゲルろ過クロマトグラフィによる懸濁液塩と NGS を妨げる可能性がある低分子量物質を除去するために。
- エチジウム ブロマイド染色、製剤の品質を表示するのにを使用して 1% アガロースゲル電気泳動によってサンプルを分析します。Nanodrop 分光光度計を用いた DNA の品質を評価します。
注: 260 λ および 280 の λ 1.85 と 2.0 の間でサンプルの吸光度の比は、準備が不純物の「クリーン」があり、目的の品質のことに通常を示します。 - DNA の品質を分析 (5 pg は 10 を使用 ng) チップを用いたキャピラリー電気泳動装置です。
注: 品質の出力は、x 軸に沿ったサイズで配布の DNA のフラグメントを表すクリーンの峰を示しています。ピークの高さは、フラグメントの豊富さを示します。ギザギザのピークを示す部分的劣化フラグメントまたは化学汚染物質。丸い曲線を表す貧しい品質を示す DNA の塗抹
2. ライブラリの準備使用 DNA とエマルジョン系クローン増幅 (emPCR 増幅)
注: ライブラリは、通常お客様指向を貫く NGS 施設で用意しています。
- DNA をフラグメントに変換するネブライザーを使用して DNA (> 200 ng) のソリューションをせん断します。マニュアルの手順27によると商業的なアダプターを縛る。
- 製造元の指示28,29,30によると DNA のサンプルの emPCR 増幅を行います。3 回洗浄手順を繰り返し、各洗浄した後、minicentrifuge のビーズ用ペレット 10 s. 破棄上清各洗浄後。
メモ: 手順は、キットに付属商業洗浄バッファーで洗浄によってキャプチャ ビーズの作製と開始します。emPCR は、NGS のテンプレートの増幅に使用されます。 - 熱変性 DNA または RNA 95 ° C で 2 分をクリックし、使用する準備ができるまで 4 ° C。使用 2 億 30 μ L の最終巻で 500 万キャプチャ ビーズ DNA ・ RNA の分子は、DNA ・ RNA のサンプルとモックのサンプルと同様、核酸サンプルでは、次の手順を行うと並んでモック サンプルを準備します。
- ボルテックス 10 のエマルジョン オイルの管によって乳化を実行最大速度で s は、プラスチック製のプラットフォーム ホモジナイザーと互換性のあるチューブを攪拌にコンテンツ全体 (4 mL) を注ぐ。プラットフォーム上で 5 分間 2,000 rpm でエマルジョンをミックスする攪拌チューブを配置します。
- 8 ストリップ キャップ チューブや 96 ウェルのプレートには、乳剤の 100 μ 因数を調剤します。チューブをキャップ プレート シールや製造元の推奨プログラム28を使用して emPCR を実行します。
メモ: PCR の完了後は、乳剤がそのまま進み、井戸を確認します。乳剤が壊れている場合は、全体をしっかりを破棄します。 - 白衣を着用し、増幅された DNA ビーズ (ADB) を収集するために化学フードで働きます。真空吸引井戸からエマルジョン 50 mL のチューブでビーズを収集しています。イソプロパノールの 100 μ L で 2 回井戸をリンスし、同じ 50 mL のチューブにリンスを吸引します。
- 収集した渦エマルジョン、35 mL の最終巻をイソプロパノールで ADB を再懸濁します。930 x g で 5 分間削除で ADB の上澄みをペレットし、バッファーを強化 10 mL を加えます。渦 ADB とそれぞれ洗浄し、洗浄ステップを 2 回繰り返して 40 mL 最終巻遠心分離機にイソプロパノールと破棄上清を追加し、洗ってください。
- イソプロパノールの代わりにエタノールを使用して最終的な洗浄を行います。追加 35 mL 最終巻、渦、ペレットにバッファーを高める 930 x g で 5 分間でビーズ上澄みを除去が、バッファーを強化 2 mL を残します。
- 微量遠心チューブに懸濁液を転送し、簡潔に ADB をペレットに遠心分離機します。上清を廃棄後に、1 mL の強化バッファーで 2 回 ADB ペレットをすすいでください。遠心分離機、各洗浄後上澄みを廃棄します。
- DNA ライブラリ ビーズ濃縮の準備をするには、ビーズに 1 N NaOH の 1 mL を追加します。渦 ADB とし、室温で 2 分間インキュベートします。遠心し、上清を捨てます。この洗浄手順をもう一度繰り返します。
- アニーリング バッファー、渦、ADB の 1 mL を追加し、室温で 2 分間インキュベートします。簡単に遠心し、上清を捨てます。再度アニーリング バッファーの 100 μ L を使用してこの手順を繰り返します。
- Dna 配列のプライマーをアニール、Seq プライマー A の 15 μ L と Seq プライマー B キットに付属の 15 μ L を追加します。簡単にボルテックスによってミックスし、65 ° C 5 分 2 分の氷への転送でのヒート ブロックに遠心チューブを配置します。
- アニーリング バッファー 1.0 mL で 3 回洗います。5 s と破棄するたびに上清の渦。
- シーケンス処理の前に商業ビーズ カウンターを使用してビーズの数を測定します。少なくとも 500,000 の豊かなビーズがあるはずです。
注: ビーズ カウンターは、指定された微量遠心チューブにビーズを測定する特殊な装置です。
3. 一般的な mRNA 合成から始まる感染カンナは CaYMV を使用して報告された診断プライマーの RT-PCR 法によりテストする葉 dsDNA の分離と
- 以降の手順で研究室コートと個人保護用ラテックス手袋を着用します。研究室のベンチで働いて、葉から 12 個の試料を収集し、サンプルを液体窒素に突入します。均質化のビーズ工場を使用します。総植物 RNA の隔離のための標準列ベースの方法を提供する商業キットを使用します。20 の粉砕と手ふれするキットによって提供されるグアニジン イソチオ シアン酸の溶解バッファーを追加 s。
- エタノールを加え、キットの指示に従って、徹底的に混ぜます。各の磨砕液を膜に RNA を結合するスピン列に追加します。3 回洗浄し、回収管24に RNA を溶出します。
- 260 λ と λ 280。 RNA の整合性を検証 1% アガロースゲル電気泳動エチジウム ブロマイドで染色を使用して吸光度の比を測定する分光光度計を使用して RNA を定量化します。
注: 1.85 と 2.0 の間の吸光度比は、準備が必要な品質であることを示します。DNase の RNA を扱う私は 37 ° C で 10 分間 (10 μ G/ml)RNase フリー水31で RNA を集中するのに商業回転カラムを使用します。プール RNA サンプルを続行する前にします。 - リボソーム RNA の植物を削除するのに削除、rRNA のキットを使用します。微量遠心チューブと洗浄を 2 回に分注の磁気ビーズ RNase フリーと水します。渦管に再懸濁します、分注チューブ マグネット スタンドに、液体をオフに待ちます。上澄みを廃棄し、磁性体粒子の再懸濁のソリューションに置き換え。再懸濁します RNase 阻害剤の 1 μ L を追加し渦。
注: このようなキットは、oligo dT mRNA に交配させる磁気ビーズにバインドを使用します。メソッドは、成績証明書24を回復するのに標準マグネット ビーズ分離技術を使用します。 - Rna の 1.25 μ g に組み合わせる 500 ng RNase フリー水と反応バッファー キットによって提供されます。50 ° c. で 10 分の混合物を置く暑さから削除し、RNAse フリー水で洗浄の磁気ビーズを追加します。渦を簡単に、5 分間室温で設定します。
- マグネット スタンドに配置し、オフに液体を待ちます。新鮮な遠心チューブに上清を転送します。氷の上を設定します。
- エクソソームと 200 の濃縮ソリューション ベースのキャプチャ方法を使用して cDNA ライブラリを準備する RNA の ng。
注: 二重鎖 cDNA ライブラリ通常仕事の顧客志向を貫く NGS 施設で用意しています。 - 商業の RNA 断片化ソリューション (0.136 g ZnCl2と 100 ミリメートル トリス塩酸 pH 7.0) を使用して RNA をフラグメントします。18 μ L の RNA 溶液 2 μ L を加える (200 ng の合計)。スピンの遠心で簡単にチューブ、30 s、や氷への転送のための 70 の ° C でサンプルを配置します。2 μ L の 10 mM トリス塩酸 pH 7.5 の 0.5 M EDTA pH 8.0 および 28 μ L を使用して反応を停止します。
- 10 分の室温で混合することによって磁性ビーズにバインド RNA は、磁気コンセントレーターを使用ビーズを収集し、上清を捨てます。70% エタノール 200 μ L で 3 回ビーズを洗浄します。破棄それぞれ洗浄し、空気が乾燥し、室温 3 分の小球形にされたビーズ 10 mM トリス塩酸 pH 7.5 の 19 μ L で再懸濁します。
- 70 ° C 10 分の加熱により断片化された RNA にランダムなプライマーをアニールし、2 分準備最初ストランドと標準的な商業 cDNA 合成キットを使用して 2 番目の鎖 cDNA のため氷の上管。
- 磁気ビーズ コンセントレーターを使用して二重鎖 cDNA を浄化します。3 回 800 μ L の 70% エタノールで洗浄します。破棄それぞれ洗浄し、空気乾燥 3 分間室温でペレットは、10 mM トリス塩酸 pH 7.5 の 16 μ L で再懸濁します。磁気ビーズ コンセントレーターを使用すると、ソリューションにある二本鎖 cDNA からビーズを分離します。新しい 200 μ L の PCR チューブに分注して cDNA を削除します。
- Taq のポリメラーゼおよび商業図書館準備キットによって提供されるナノメーターの混合物を使用してフラグメント終わり修理を実施します。市販のキットでは、25 ° C で 10 分間商業リガーゼを使って二重鎖 cDNA の各端に追加する希釈アダプターを提供します。
4. ウイルス製剤と dsDNA mRNA から調製したライブラリから調製した DNA ライブラリの NGS
- 標準高スループット パイロシークエンス楽器を使用し、DNA 配列の直接読み出しを生成するすべての推奨メーカーのプロトコルに従います。蛍光に分類されたヌクレオチドを含む商業シーケンス試薬を使用します。
注記: 詳細は、計測器の製造元の指示を参照してください。 - < 700 跪くの平均の長さとコンティグの最初のセットを生成する読み取りを自動的にアセンブルするゲノムのアセンブリのソフトウェアを使用してポスト シーケンス解析を行う品質管理を行なう iPlant/サイヴァース ウェブサイト上の FastQC ソフトウェアを使用します。塩基配列データ32のチェックします。マッピングと私アンプリコン ソフトウェアを使用してより小さいシーケンス読み取り24から長いシーケンスを再構築していく Phred スコア ≥ 30 でシーケンスを選択します。
注: 詳細は製造元の指示を参照してください。 - NCBI BLASTn 解析メガブラスト デフォルト モジュールとして Viridplantae を使用してこれらの組み立てられたコンティグを提出 (TaxID: 33090) やウイルス (TaxID: 10239)33名制限の個体として。レポートに報告されたBadnavirusのゲノムに高い類似性を示すコンティグの亜種を収集します。
- 1 つまたは複数の候補フルレングス ウイルスのゲノムを表す結合された足場が正しく標準 badnavirus ゲノムと同じ組織があるフレームのシーケンスを生成することを確認します。これを行うには、ソフトウェアを描画プラスミドに候補全長ウイルスゲノムを入力します。最初 15 のヌクレオチドは、tRNA に会った(TGGTATCAGAGCGAG) を badnaviruses の間で保存性が高いので構成されていますを確認します。ゲノムの 3' 末端に近い電位起こる信号を検索します。小さな 2 つの Orf と 1 つの大規模な ORF ポリタンパク質をエンコードの存在を確認する完全なゲノムの注釈を付けます。ExPASy ポータルを使用して翻訳 badnavirus ORF1 と ORF2、ORF3 翻訳製品34を識別するツールです。
注: この科学的なソフトウェアは無料と、環状 DNA を生成、識別すべて開いたリーディング フレーム、およびシーケンスがフルの長さの円形のゲノム DNA を表すことを確認する即時出力を提供します。 - DNA および RNA の解析35,36から得られたウイルスのゲノムを比較するのに複数のシーケンスの比較ツール、筋肉、CLUSTALW、オープン ソースを使用します。
- 30 badnavirus 種の全ゲノム シーケンスを取得して .fasta 形式のドキュメントとしてエクスポート NCBI 塩基配列データベースを検索します。NGS によって得られたウイルスのゲノム シーケンスと共にシーケンスの進化の遺伝学的解析を行うソフトウェアにシーケンスをアップロードします。複数の配列アラインメントと筋37を使用して最大尤度ツリーを生成します。
5. 感染植物からのウイルスのゲノムの PCR 増幅によるDe Novoシーケンスの評価
- PCR プライマー38する無料オンライン Primer3 ツールに新たに同定されたフルレングス badnavirus ゲノム配列 (.fasta 形式) を入力します。ウイルス genome(s) の全体の長さに沿って 1,000-1,500 bp の千鳥生産プライマー セットを識別します。合成、PCR のプライマーを提供サービス施設にシーケンスを送信します。
注: 出力は、一般的の溶ける温度と導入されたシーケンスに沿って正確なプライマー場所許容プライマー対を識別します。 - 研究室のベンチで作業、実験用の上着と手袋を身に着けている、植物素材39から DNA を分離する標準的な常磁性セルロース粒子を含む自動化された方法を使用してウイルス感染および健康な制御の葉から DNA の 5 μ g を分離します。葉材 (20-40 mg) を微量遠心チューブに液体窒素で凍結し、ビーズのミルを使用してを挽きます。微量遠心チューブに換散バッファーのサンプルを組み合わせて、RNase A を各サンプルに追加します。渦サンプル 10-20 s と簡単にスピン固体粒子を削除するサンプルです。
注: 常磁性セルロース粒子高 DNA 結合の能力を持っている、純粋な DNA の高収率を隔離します。DNA の隔離のための標準的な商業シリカ カラム法効率的にさまざまな植物種から DNA を抽出しません。その結果、メソッドの数十存在する個々 の植物種の効率を改善する手順の変更です。以上 25 草本被子植物種40より高品質の DNA が得られますので、自動化された常磁性セルロース粒子法が選ばれました。 - 自動化された常磁性 DNA の隔離のための商業試薬カートリッジを使用します。ヌクレアーゼ フリー水の 300 μ L を各商業試薬カートリッジと転送植物ライセートを同じカートリッジに追加します。カートリッジ ラックには置、溶出チューブに近い井戸のプランジャーに、溶出チューブに溶出バッファーを置きます。核酸自動分離機にカートリッジをロードし、植物 DNA 分離プロトコル41,42を実行します。
- 重複する PCR の製品のセットを派生する PCR を行います。PCR の拡大のサイクルで 35 各前方および逆のプライマーの 5 μ M を使用します。次のサイクリング条件を使用: 95 ° C、60 s、7 ~ 10 分除去するためにパッケージ化されたゲルろ過カラムを使用塩の 45 s、および 72 ° C で最終的な拡張と 72 ° C 1-2 分で延長は 50 ° C で熱処理、低分子材料で変性 ステップ 1.231。
- 50 に縛ること PCR の製品の量を決定するベクトルに PCR の製品の 3:1 のモル比を計算する線形 pGEM プラスミド43の ng。コントロール挿入 DNA およびが効率的に動作するかどうかを決定するために使用します。一晩 4 ° C で T4 DNA リガーゼ (3 U/μ L) を使って結紮を行う変換が商業的 JM109 有能なエシェリヒア属大腸菌を準備し、セル。使用は、効率的な変換のための肯定的な制御としてノーカット プラスミド DNA の 100 pg を制御します。LB 寒天培地プレート上に変換されたセルの 100 μ L をプレート結紮プラスミド26を回復する抗生物質と青/白で。37 ° C で 16-24 h 用の版を孵化させなさい
メモ: pGEM ベクトル β-ガラクトシダーゼをコードする lacZ 遺伝子があります。変換された細菌が平板上に成長した 100 μ g/mL アンピシリン、0.5 mM IPTG、80 μ g/mL 5-bromo-4-chloro-3-indoyl-β-D-galactopyranosidase を含む (X-gal) が青く β-ガラクトシダーゼ活性のためなります。LacZ遺伝子を混乱させる方法で pGEM プラスミドが線形化されます。PCR の製品の挿入を含む植民地は lacZ の遺伝子を妨害し、X gal を代謝されません。これらの植民地は白されています。こうして植民地 (青と白)26の色によって、挿入なしでそれらから挿入して植民地を区別できます。 - 標準列に基づくプラスミド分離キット39を使用して 3 つのコロニーからの DNA を分離します。変換製品の 3 つのプラスミドをシーケンスします。NGS のプロデュース・ デ ・ ノボ組み立てウイルス ゲノムと各 DNA シーケンスを比較します。CLUSTALW を使用して、シーケンスを配置し、適切に順序付けられていることを確認します。
結果
この変更されたウイルスの精製法は、ウイルス Dna NGS とバイオインフォマティクスによる 2 つのウイルス種を識別するのに役立ちますの濃縮を提供されます。磨砕液は 2.5 時間 40,000 × g で遠心分離後、管の下部に緑のペレットと長さに沿って白いペレットがあった。緑餌は 1 つの遠心管に再停止され、白いペレットは 2 つの遠心管に再停止されます。PCR は標準、CaYMV PCR 診断のプライマーを使用して遂行し、可溶化された白いペレット, 緑のペレット (図 1A) ない物が検出されました。124-133 nm の長さ (図 1B) を測定被膜粒子観察し、粗野な準備のサンプル透過型電子顕微鏡により調べた。ほとんどの badnaviruses の予測されたモーダル長さ以内です。DNA は白と緑のペレットから抽出され、別々 に再停止されます。図 1C、緑から抽出した DNA の 5 μ L を読み込むと白いペレット サンプル (緑の割合のための DNA の 1.6 μ g)、白、分数の DNA の 3.1 μ g 0.8% の agarose ゲルの電気泳動と次の臭化エチジウム DNA を分析染色。緑の分数には、白い小数生産低分子量 DNA (図 1C) と同様、高分子量 DNA の 2 つのバンドに対し低分子量 DNA が含まれています。100 V で 40 分間図 1Cに示したゲルを実行し、車線 3 のスミアは明確なバンドを生成するゲルの電圧の下がるべきであることを示唆しています。これらのデータは、白いペレットがウイルス粒子の濃縮だったことを提案します。白のサンプルから抽出した DNA (0.6 μ g/mL) 濃度は低いが、NGS は、10 の最小値を必要とするための十分な続行する DNA の ng。断片化された Dna は、NGS のライブラリを準備する使用されました。
並行して、RNA が RNA シーケンス高スループットは感染したカンナ植物 (図 1D) から抽出されました。標準的なワークフローは、ライブラリの準備、NGS、コンティグを作成して (図 1E) のウイルスのゲノム配列を識別するため行った。出発物質として DNA と RNA を使用してからの出力結果を比較しました。
原油ウイルス準備から分離された DNA を用いた ngs 188,626 生 DNA 読み取りを取得しました。13,269 コンティグに組み立てられた読み取りと BLASTn は塩基配列の NCBI データセットを検索する使用された (Viridplantae TaxID を使用して: 33090 とウイルス TaxID: 制限の生物として 10239) (図 1E)。NCBI BLASTn 結果では、 de novo組み立てコンティグの 93% が細胞系列、22% は不明であったその 0.3% がウイルス コンティグ (図 2A) を明らかにしました。コンティグ細胞系列はミトコンドリアとして識別されたように、分類や葉緑体 DNA の大部分。ウイルス コンティグのデータセット内ウイルス コンティグの 32% (Badnavirus シーケンスではなかった)、 Caulimoviridaeのメンバーに関連していた、これらの 58% に関連していたBadnavirus.ウイルス コンティグの 29% が非常によく似 (e < 1 10-30x) CaYMV 分離 V17 ORF3 遺伝子 (EF189148.1) に、サトウキビ被膜ウイルス分離バタヴィア D、完全なゲノム (FJ439817.1)、バナナ連勝 CA ウイルスゲノム (を完了KJ013511)。この集団内で 2 つの完全な長さのゲノムに似ている長いコンティグがあった。
< 500 跪く Contig アセンブリの長さを読む平均高スループット RNA シーケンス生成 153,488 洗浄個別シーケンス リード 8,243 コンティグまで低下。これらが提出され NCBI BLASTn (Viridplantae TaxID を使用して: 33090 とウイルス TaxID: 制限の生物として 10239) 植物細胞系列、23% のカテゴリに、コンティグの 76% を配置出力は不明であったし、0.1% はウイルス コンティグ (として分類されました。図 2B). ウイルス コンティグの 0.1% の人口の人口を綿密に検討決定これらの 68% がCaulimoviridae (図 2B) に割り当てられました。この集団内で 3 つの大きなコンティグの類似性が高い識別された (10-30X e < 1) CaYMV 分離 V17 ORF3 遺伝子 (EF189148.1)、サトウキビ被膜ウイルス分離バタヴィア D、完全なゲノム (FJ439817.1) およびバナナ連勝CA のウイルス完全なゲノム (KJ013511)。私たち 3 コンティグを調べると、手動でフルレングスのウイルスゲノムを生成するこれらの 2 つに参加。
我々 は、2 つのフルレングスのウイルスのゲノムの存在を確認する相互足場として DNA および RNA シーケンスによって生成されるウイルスのゲノムの長さコンティグを比較しました。1 つのフルレングスのウイルス ゲノム 6,966 の bp だったカンナ関連黄斑ウイルス1 (CaYMAV-1) (図 3A) を仮称します。2 番目のゲノムは 7,385 bp とゲットウにともなって(CaYMV-Ap01) (図 3A) に感染する CaYMV のバリアント。
最後に、各ウイルスのクローン 〜 1,000 bp の断片に設計されたプライマーを用いて差分 9 品種を表す 227 カンナ植物の人口の両方のゲノムを検出します。多くの場合個々 の植物は両方のウイルスに感染しました。12 植物の CaYMAV 1 の CaYMV Ap01 RT-PCR 検出の例を提供しています。これらの 3 つが CaYMV Ap01 は陽性であり (図 3B) 両方のウイルス陽性であった 9。

図 1: ウイルス核酸製剤と NGS ワークフロー 。(A) アガロースゲル (1.0%) CaYMV ゲノムの 565 bp PCR のフラグメントの電気泳動。緑餌サンプル (3 車線) ではなく白いペレット (レーン 1、2) から調製したサンプルで 2 つの PCR 産物が検出されました。肯定的な制御 (+) は、感染した植物が標準的な常磁性セルロース粒子を含む自動化された方法を使用して分離された DNA 増幅 PCR の製品を表します。レーン L にはサンプル車線で線形 DNA バンドのサイズを測定するための標準として使用される DNA の梯子が含まれます。閲覧によって感染したカンナの葉の粗分別回収白いペレットの透過電子顕微鏡によるウイルス粒子の (B) の例です。(C) Agarose (0.8%) グリーン (1 車線) から回復 DNA の電気泳動のゲルし、ホワイト (2 車線) ペレット パネル A の PCR によってその陽性レーン 2 の横に赤と黄色のドットは、白の割合で発生する 2 つの高分子量 DNA バンドを識別します。(1%) の (D) には、列に基づく RNA 精製によって回復の総 RNA の電気泳動がアガロースゲルします。レーン L にはサンプル車線で線形バンドのサイズを測定するための標準として使用される DNA の梯子が含まれます。レーン 1-6 には、リボ枯渇と核酸分離、ライブラリの準備、シーケンス、contig アセンブリ、およびウイルスのゲノムの RNA シーケンス (E) スケマティック パイプ ラインのための 1 つのサンプルを集めた感染カンナ葉から分離された RNA が含まれています。発見。この図の拡大版を表示するのにはここをクリックしてください。
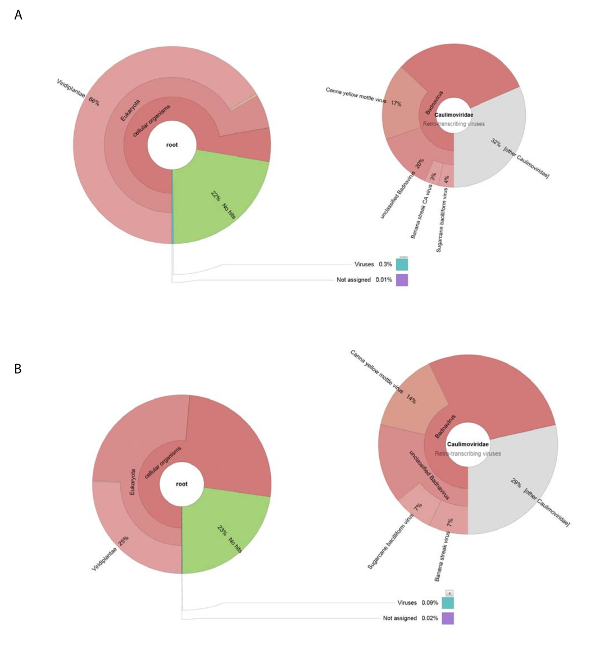
図 2: アイスランドクローナ グラフ コンティグの分類カテゴリを視覚化します。(A) 粗ウイルス準備から組み立てのコンティグの分類の生息左図のグラフ。右のグラフは、 Caulimoviridae家族、 Badnavirus属および 3 つの密接に関連種に関連付けられているウイルス コンティグの割合を示しています。(B) 左側のパネルは、その分類学的分布に基づく RNA シーケンスから派生したコンティグの豊かさを示しています。右側はCaulimoviridae家族、 Badnavirus属および 3 つの密接に関連種に関連付けられているウイルス コンティグの人口内のコンティグの豊かさを描いたグラフです。この図の拡大版を表示するのにはここをクリックしてください。
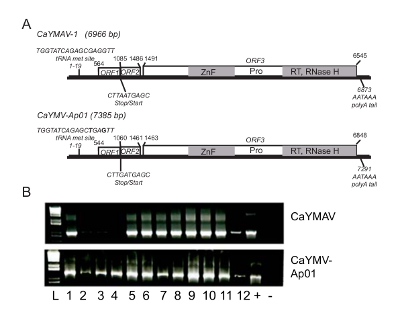
図 3.CaYMAV 1、CaYMV Ap01 ゲノムのキャラクタリゼーション。カンナ黄色斑紋ウイルス 1 に関連付ける(CaYMAV) とカンナ黄色斑紋ウイルス由来のゲノムに類似の図式化 (A)ゲットウにともなって (CaYMV Ap01).塩基配列の位置 1-10 はゲノムのスタートとして識別され、tRNAに会ったアンチコドン サイトほとんど badnavirus ゲノムの典型的なが含まれています。オープンリーディング フレーム (ORF) 1 と 2 の翻訳の停止と開始位置は、隣接しています。これらの蛋白質がある未知の関数。ORF3 は亜鉛指 (ZnF)、プロテアーゼ (プロ)、逆転写酵素 (RT) と RNAse H ドメインを含む蛋白です。3' 多信号シーケンスは、両方のウイルスのゲノムの保存されています。(B) RT - pcr はウイルスに感染した葉と CaYMAV と CaYMV Ap01 を検出するプライマーから単離した RNA を用いて行った。12 植物の同じ人口で 3 つに感染した CaYMV Ap01 のみ、残りの CaYMAV と CaYMV Ap01 感染していたに対し。肯定的な制御を示します (+) (-) がネガティブ コントロールを示し。この図は、Wijayasekaraらから再現/変更24権限を持つ。この図の拡大版を表示するのにはここをクリックしてください。
ディスカッション
近年、植物ウイルスなどウイルス様粒子 (VLP) またはウイルス特定 RNA または DNA2,3,44、豊かな自然環境生物多様性を勉強するさまざまな方法を採用されています。 45,46 。これらのメソッドは、NGS とバイオ情報解析が続いています。本研究の目的は、耕された植物の一般的な疾患の病原を見つけることだった。病気は、エンベロープを持たない被膜粒子、565 の bp のフラグメントだけがクローンとして作られた47をされている未知のウイルスの結果であると報じられました。この情報は、仮に属Badnavirus Caulimoviridae家族内にウイルスを割り当てる前の研究者のために十分だった。カンナ カンナ ユリ斑病がこの研究で説明するメタゲノム アプローチを使用して、単一の badnavirus の結果であったという仮説を立てた事前レポートしながら我々 は病気が 2 つ仮 badnavirus 種24によって引き起こされたことを決定しました。したがって、病気の原因物質を発見するメタゲノム アプローチを使用しての強さは我々 は今の状況を識別できる、1 つ以上の原因があるかもしれません。
我々 のアプローチは、DNA および RNA の配列データを組み合わせることは徹底され、2 つのアプローチを使用して結果が一貫性のある結果が得られた、2 つの関連のウイルスの存在を確認した例も示します。我々 は caulimoviruses の分離の変更手順を採用し、ウイルス核酸が濃縮されたウイルスのカプシド内で保護されていたことサンプルを制作します。サービス研究所 DNA シーケンシングを行う契約しました。De novoシーケンスの本質的な概念はその DNA ポリメラーゼに DNA 合成の連続サイクル中にヌクレオチド DNA のテンプレートの繊維にラベルの付いた蛍光灯が組み込まれています。コンティグ組み立て続く ngs コンティグ ウイルスとして識別されたいくつかのコンティグを生産バイオ情報ワークフローに提出されました。2 つのウイルスのゲノム10,24,48,49,50の更なる確証はリボ枯渇 RNA 製剤から得られた RNA シーケンス データのバイオインフォマティクス解析から得た。興味深い結果が DNA および RNA シーケンスによって、シーケンスの人口を回復を学ぶことだった 1 つは、非ウイルスやウイルスの核酸のような分布を提供しました。DNA および RNA シーケンスのシーケンスの < 0.5% はウイルスの起源だった。内ウイルスCaulimoviridae家族に属していたシーケンス 78 82% の人口。DNA および RNA シーケンスから組み立てられたウイルス コンティグを比較すると、両方のデータセットに 2 つの組み立てられたゲノムが発生したことを確認した.
新しいウイルスのゲノムを識別する唯一の DNA の配列を使用しての関心事は、オープンの環状 DNA である badnavirus ゲノムです。我々 はゲノムの不連続性を重複シーケンスがコンティグからゲノムのアセンブリのための障害を示すかもしれないと推測。結果を配列する DNA の最初の検査では、2 つの類似ウイルス ゲノムを明らかにしました。我々 はこれらのゲノムがどちらかは検討されていない種の遺伝的多様性を表す仮説や共同感染同じ表される 2 種植物24。したがって、NGS DNA および RNA シーケンスによって得られるデータセットの集合的なバイオ情報解析には、2 つの完全な長さのゲノムの存在の確認が有効になります。
メタゲノム研究、カリフラワー モザイク ウイルス(CaMV;、caulimovirus)3から DNA を回復する手順に基づく植物ホモジュネートから VLP と核酸を抽出する方法を開発したもう一つの報告があります。このアプローチには、RNA の小説と非栽培植物 DNA ウイルス シーケンスが識別されます。耕された植物の病気の原因物質を発見する本研究で使用される caulimovirus の隔離プロシージャから派生した手順は、派生して自然感染植物24から VLP を抽出するための手順とは異なりです。両方の変更方法の成功は、caulimovirus 分離フレームワーク プロシージャ一般的かもしれない植物ウイルスのメタゲノム研究のための貴重な出発点を示唆しています。
開示事項
著者が明らかに何もありません。
謝辞
科学の進歩と技術応用研究プログラム フェーズ II AR 132-053-2; オクラホマ センターによって資金が供給された研究・農業園芸作物のオクラホマ部研究助成プログラム。HongJin 黄博士と NSF (EOS 0132534)、(2P20RR016478-04、1P20RR16478 02 5P20RR15564 03) NIH からの補助金によって支えられた大須バイオインフォマティクスの中核施設に感謝します
資料
| Name | Company | Catalog Number | Comments |
| NaH2PO4 | Sigma-Aldrich St. Louis MO | S5976 | Grinding buffer for virus purification |
| Na2HPO4 | Sigma-Aldrich | S0751 | Grinding buffer for virus purification |
| Na2SO3 | Thermo-Fisher Waltham, MA | 28790 | Grinding buffer for virus purification |
| urea | Thermo-Fisher | PB169-212 | Homogenate extraction |
| Triton X-100 | Sigma-Aldrich | X-100 | Homogenate extraction |
| Cheesecloth | VWR Radnor, PA | 21910-107 | Filter homogenate |
| Tris | Thermo-Fisher | BP152-5 | Pellet resuspension& DNA resuspension buffers |
| MgCl2 | Spectrum, Gardena, CA | M1035 | Pellet resuspension buffer |
| EDTA | Spectrum | E1045 | Stops enzyme reactions |
| Proteinase K | Thermo-Fisher | 25530 | DNA resuspension buffer |
| phenol:chloroform:isoamylalcohol | Sigma-Aldrich | P2069 | Dissolve virion proteins |
| DNAse I | Promega | M6101 | Degrade cellular DNA from extracts |
| 95% ethanol | Sigma-Aldrich | 6B-100 | Virus DNA precipitation |
| Laboratory blender | VWR | 58984-030 | Grind leaf samples |
| Floor model ultracentrifuge &Ti70 rotor | Beckman Coulter, Irving TX | A94471 | Separation of cellular extracts |
| Floor model centrifuge and JA-14 rotor | Beckman Coulter | 369001 | Separation of cellular extracts |
| Magnetic stir plate | VWR | 75876-022 | Mixing urea into samples overnight |
| Rubber policeman | VWR | 470104-462 | Dissolve virus pellet |
| 2100 bioanalyzer Instrument | Agilent Genomics, Santa Clare, CA | G2939BA | Sensitive detection of DNA and RNA quality and quantity |
| 2100 Bioanalyzer RNA-Picochip | 5067-1513 | Microfluidics chip used to move, stain and measure RNA quality in a 2100 Bioanalyzer | |
| 2100 Bioanalyzer DNA-High Sensitive chip | 5067-4626 | Microfluidics chip used to move, stain and measure DNA quality in a 2100 Bioanalyzer | |
| Nanodrop spectrophotometer | Thermo-Fisher | ND-2000 | Analysis of DNA/RNA quality at intermediate steps of procedures |
| Plant total RNA isolation kit | Sigma-Aldrich | STRN50-1KT | Isolate RNA for RNA-seq |
| RNase-free water | VWR | 10128-514 | Resuspension of DNA and RNA for NGS |
| RNA concentrator spin column | Zymo Research, Irvine, CA | R1013 | Prepare RNA for RNA-seq |
| rRNA removal kit | Illumina, San Diego, CA | MRZPL116 | Prepare RNA for RNA-seq |
| DynaMag-2 Magnet | ThermoFisher | 12321D | Prepare RNA for RNA-seq |
| RNA enrichment system | Roche | 7277300001 | Prepare RNA for RNA-seq |
| Agarose | Thermo-Fisher | 16500100 | Gel analysis of DNA/RNA quality at intermediate steps of procedures |
| Ethidium bromide | Thermo-Fisher | 15585011 | Agarose gel staining |
| pGEM-T +JM109 competent cells | Promega, Madison, WI | A3610 | Clone genome fragments |
| pFU Taq polymerase | Promega | M7741 | PCR amplify virus genome |
| dNTPs | Promega | U1511 | PCR amplify virus genome |
| PCR oligonucleotides | IDT, Coralvill, IA | Custom order | PCR amplify virus genome |
| Miniprep DNA purification kit | Promega | A1330 | Plasmid DNA purification prior to sequencing |
| PCR clean-up kit | Promega | A9281 | Prepare PCR products for cloning |
| pDRAW32 software | ACAClone | Computer analysis of circular DNA and motifs | |
| MEGA6.0 software | MEGA | Molecular evolutionary genetics analysis | |
| Primer 3.0 | Simgene.com | ||
| Quant-iT™ RiboGreen™ RNA Assay Kit | Thermo-Fisher | R11490 | Fluorometric determination of RNA quantity |
| GS Junior™ pyrosequencing System | Roche | 5526337001 | Sequencing platform |
| GS Junior Titanium EmPCR Kit (Lib-A) | Roche | 5996520001 | Reagents for emulsion PCR |
| GS Jr EmPCR Bead Recovery Reagents | Roche | 5996490001 | Reagents for emulsion PCR |
| GS Junior EmPCR Reagents (Lib-A) | Roche | 5996538001 | Reagents for emulsion PCR |
| GS Jr EmPCR Oil & Breaking Kit | Roche | 5996511001 | Reagents for emulsion PCR |
| GS Jr Titanium Sequenicing kit* | Roche | 5996554001 | Includes sequencing reagents, enzymes, buffers, and packing beads |
| GS Jr. Titanium Picotiter Plate Kit | Roche | 5996619001 | Sequencing plate with associated reagents and gaskets |
| IKA Turrax mixer | 3646000 | Special mixer used with Turrax Tubes | |
| IKA Turrax Tube (specialized mixer) | 20003213 | Specialized mixing tubes with internal rotor for creating emulsions | |
| GS Nebulizers Kit | Roche | 5160570001 | Nucleic acid size fractionator for use during library preparations |
| GS Junior emPCR Bead Counter | Roche | 05 996 635 001 | Library bead counter |
| GS Junior Bead Deposition Device | Roche | 05 996 473 001 | Holder for Picotiter plate during centrifugation |
| Counterweight & Adaptor for the Bead Deposition Devices | Roche | 05 889 103 001 | Used to balance deposition device with picotiter plate centrifugation |
| GS Junior Software | Roche | 05 996 643 001 | Software suite for controlling the instrument, collecting and analyzing data |
| GS Junior Sequencer Control v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Run Processor v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS De Novo Assembler v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Reference Mapper v. 3.0 | Roche | (Included in item 05 996 643 001 above) | |
| GS Amplicon Variant Analyzer v. 3.0 | Roche | (Included in item 05 996 643 001 above) |
参考文献
- Dijkstra, J., Jager, C. P. . Practical Plant Virology : Protocols and Exercises. , (1998).
- Roossinck, M. J. Plant virus metagenomics: biodiversity and ecology. Annu Rev Genet. 46, 359-369 (2012).
- Melcher, U., et al. Evidence for novel viruses by analysis of nucleic acids in virus-like particle fractions from Ambrosia psilostachya. J Virol Methods. 152 (1-2), 49-55 (2008).
- Stobbe, A. H., Schneider, W. L., Hoyt, P. R., Melcher, U. Screening metagenomic data for viruses using the e-probe diagnostic nucleic Acid assay. Phytopathology. 104 (10), 1125-1129 (2014).
- Borah, B. K., et al. Bacilliform DNA-containing plant viruses in the tropics: commonalities within a genetically diverse group. Mol Plant Pathol. 14 (8), 759-771 (2013).
- Bousalem, M., Douzery, E. J., Seal, S. E. Taxonomy, molecular phylogeny and evolution of plant reverse transcribing viruses (family Caulimoviridae) inferred from full-length genome and reverse transcriptase sequences. Arch Virol. 153 (6), 1085-1102 (2008).
- Geering, A. D., et al. Banana contains a diverse array of endogenous badnaviruses. J Gen Virol. 86, 511-520 (2005).
- Kunii, M., et al. Reconstruction of putative DNA virus from endogenous rice tungro bacilliform virus-like sequences in the rice genome: implications for integration and evolution. BMC Genomics. 5, 80 (2004).
- Laney, A. G., Hassan, M., Tzanetakis, I. E. An integrated badnavirus is prevalent in Figure germplasm. Phytopathology. 102 (12), 1182-1189 (2012).
- Gambley, C. F., Geering, A. D., Steele, V., Thomas, J. E. Identification of viral and non-viral reverse transcribing elements in pineapple (Ananas comosus), including members of two new badnavirus species. Arch Virol. 153 (8), 1599-1604 (2008).
- Gayral, P., et al. A single Banana streak virus integration event in the banana genome as the origin of infectious endogenous pararetrovirus. J Virol. 82 (13), 6697-6710 (2008).
- Lyttle, D. J., Orlovich, D. A., Guy, P. L. Detection and analysis of endogenous badnaviruses in the New Zealand flora. AoB Plants. 2011, 008 (2011).
- Le Provost, G., Iskra-Caruana, M. L., Acina, I., Teycheney, P. Y. Improved detection of episomal Banana streak viruses by multiplex immunocapture PCR. J Virol Methods. 137 (1), 7-13 (2006).
- Singh, K., Talla, A., Qiu, W. Small RNA profiling of virus-infected grapevines: evidences for virus infection-associated and variety-specific miRNAs. Funct Integr Genomics. 12 (4), 659-669 (2012).
- Alfson, K. J., Beadles, M. W., Griffiths, A. A new approach to determining whole viral genomic sequences including termini using a single deep sequencing run. J Virol Methods. 208, 1-5 (2014).
- Kreuze, J. F., et al. Complete viral genome sequence and discovery of novel viruses by deep sequencing of small RNAs: a generic method for diagnosis, discovery and sequencing of viruses. Virology. 388 (1), 1-7 (2009).
- Zheng, Y., et al. VirusDetect: An automated pipeline for efficient virus discovery using deep sequencing of small RNAs. Virology. 500, 130-138 (2017).
- James, A. P., Geijskes, R. J., Dale, J. L., Harding, R. M. Molecular characterisation of six badnavirus species associated with leaf streak disease of banana in East Africa. Annals of Applied Biology. 158 (3), 346-353 (2011).
- Baranwal, V. K., Sharma, S. K., Khurana, D., Verma, R. Sequence analysis of shorter than genome length episomal Banana streak OL virus like sequences isolated from banana in India. Virus Genes. 48 (1), 120-127 (2014).
- Sukal, A., Kidanemariam, D., Dale, J., James, A., Harding, R. Characterization of badnaviruses infecting Dioscorea spp. in the Pacific reveals two putative novel species and the first report of dioscorea bacilliform RT virus 2. Virus Res. 238, 29-34 (2017).
- BÖmer, M., Turaki, A. A., Silva, G., Kumar, P. L., Seal, S. E. A sequence-independent strategy for amplification and characterisation of episomal badnavirus sequences reveals three previously uncharacterised yam badnaviruses. Viruses. 8 (7), (2016).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. Canna yellow mottle virus detected in Canna in Florida. Plant Health Progress. , 2-4 (2004).
- Zhang, J., et al. Characterization of Canna yellow mottle virus in a new host, Alpinia purpurata, in Hawaii. Phytopathology. 107 (6), 791-799 (2017).
- Wijayasekara, D., et al. Molecular characterization of two badnavirus genomes associated with Canna yellow mottle disease. Virus Res. 243, 19-24 (2018).
- Covey, S. N., Noad, R. J., al-Kaff, N. S., Turner, D. S. Caulimovirus isolation and DNA extraction. Methods Mol Biol. 81, 53-63 (1998).
- Sambrook, J., Fritsch, E. F., Maniatis, T. . Molecular cloning: A laboratory manual. 2nd edn. , (1989).
- Radford, A. D., et al. Application of next-generation sequencing technologies in virology. J Gen Virol. 93, 1853-1868 (2012).
- Kanagal-Shamanna, R. Emulsion PCR: Techniques and Applications. Methods Mol Biol. 1392, 33-42 (2016).
- Getts, D. R., et al. Targeted blockade in lethal West Nile virus encephalitis indicates a crucial role for very late antigen (VLA)-4-dependent recruitment of nitric oxide-producing macrophages. J Neuroinflammation. 9, 246 (2012).
- van Dijk, E. L., Jaszczyszyn, Y., Thermes, C. Library preparation methods for next-generation sequencing: tone down the bias. Exp Cell Res. 322 (1), 12-20 (2014).
- . Gel filtration principles and methods. GE Healthcare. , (2010).
- Goff, S., et al. The iPlant Collaborative: Cyberinfrastructure for Plant Biology. Frontiers in Plant Science. 2, (2011).
- Lin, Z., et al. Next-generation sequencing and bioinformatic approaches to detect and analyze influenza virus in ferrets. J Infect Dev Ctries. 8 (4), 498-509 (2014).
- Artimo, P., et al. ExPASy: SIB bioinformatics resource portal. Nucleic Acids Res. 40, 597-603 (2012).
- Edgar, R. C. MUSCLE: a multiple sequence alignment method with reduced time and space complexity. BMC Bioinformatics. 5, 113 (2004).
- Hung, J. H., Weng, Z. Sequence Alignment and Homology Search with BLAST and ClustalW. Cold Spring Harb Protoc. 2016 (11), (2016).
- Sohpal, V. K., Dey, A., Singh, A. MEGA biocentric software for sequence and phylogenetic analysis: a review. Int J Bioinform Res Appl. 6 (3), 230-240 (2010).
- Untergasser, A., et al. Primer3--new capabilities and interfaces. Nucleic Acids Res. 40 (15), 115 (2012).
- Dhaliwa, A. DNA extraction and purification. Mater Methods. 3, 191 (2013).
- Moeller, J. R., Moehn, N. R., Waller, D. M., Givnish, T. J. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Moeller, J. R., et al. Paramagnetic cellulose DNA isolation improves DNA yield and quality among diverse plant taxa. Appl. Plant Sci. 2 (10), (2014).
- Grooms, K. . Review: Improved DNA Yield and Quality from Diverse Plant Taxa. , (2015).
- Nishimori, A., et al. In vitro and in vivo antivirus activity of an anti-programmed death-ligand 1 (PD-L1) rat-bovine chimeric antibody against bovine leukemia virus infection. PLoS One. 12 (4), 0174916 (2017).
- Rojas, M. R., Gilbertson, R. L., Roossinck, M. J. . Plant Virus Evolution. 1, 27-51 (2008).
- Roossinck, M. J. The big unknown: plant virus biodiversity. Curr Opin Virol. 1 (1), 63-67 (2011).
- Roossinck, M. J., Martin, D. P., Roumagnac, P. Plant Virus Metagenomics: Advances in Virus Discovery. Phytopathology. 105 (6), 716-727 (2015).
- Momol, M. T., Lockhart, B. E. L., Dankers, H., Adkins, S. . Plant Health Progress. , (2004).
- Eni, A., Hughes, J. D., Asiedu, R., Rey, M. Sequence diversity among badnavirus isolates infecting yam (Dioscorea spp.). Archives of Virology. 153 (12), 2263-2272 (2008).
- Harper, G., et al. The diversity of Banana streak virus isolates in Uganda. Arch Virol. 150 (12), 2407-2420 (2005).
- Muller, E., Sackey, S. Molecular variability analysis of five new complete cacao swollen shoot virus genomic sequences. Arch Virol. 150 (1), 53-66 (2005).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved