Method Article
ストレス曝露に伴うエピジェネティックな変化を識別するためにラット メチル Seq プラットフォーム
要約
ここでは、プロトコルとメチル Seq エピゲノムのプラットフォームでは、慢性的なストレス暴露に伴うエピジェネティックな変化を識別するためにラットのモデルを使用しての実装について述べる。結果は、ラット メチル Seq プラットフォームのラットにおけるストレス暴露から生じる可能性のメチル化の相違点を検出できることを示します。
要約
幅広い動物のゲノムが利用可能になると、これらの動物モデルにおけるエピジェネティックな変化を捉えることができますツールのニーズがあります。ラットは、エピジェネティックなツールが洞察力の力学的情報を提供する多くの薬理学的・行動研究を補完することが 1 つの特定のモデル動物であります。このため、ラット、ラット遺伝子 DNA のメチル化レベルを評価するため SureSelect ターゲットをキャプチャ システム (メチル Seq と呼ばれる) を適応しました。ラットの設計では、プロモーター、CpG の島、島の海岸、すべて RefSeq 遺伝子から GC の豊富な地域を対象としました。
ラット実験プラットフォームを実装するには、男性スプレイグ ラットは 3 週間、その後採血したゲノム DNA の抽出の後の慢性的な可変ストレスにさらされました。メチル Seq ライブラリは、剪断、アダプター結紮、ターゲット濃縮、重亜硫酸塩の変換、多重化によりラットの DNA のサンプルから構築されました。次世代シーケンシング プラットフォームのライブラリ結果し、応力負荷及び無負荷ラットの DNA の間 Dmr を識別するためにシーケンスされた読み取りを行った。プラットフォームの堅牢性を確認するための重亜硫酸塩ピロシーケンスによる最有力候補 Dmr が独立して検証。
結果は、ラット メチル Seq プラットフォームがストレスにさらされることによってメチル化変化をキャプチャすることができます便利なエピジェネティックなツールであることを示します。
概要
高スループット シーケンスの進歩は、豊富なモデルと非モデル生物のゲノムにつながっています。このようなシーケンスのアベイラビリティは、遺伝学、比較ゲノミクス、トランスクリプトミクス研究を促進しています。たとえば、利用可能なゲノム シーケンスが豊かにヒストンの修正1、または重亜硫酸塩の配列との関連に基づく DNA チップ Seq 実験からシーケンサーのデータを整列させるため非常に有用なによる DNA メチル化を測定します。非メチル化シトシン2の重亜硫酸塩の変換から形成されるウラシルを検出します。しかし、遺伝子の機能に影響を与えることができます特異の規制シーケンスの注釈付きデータの不足のためのデザインに使用可能なゲノム シーケンス データを組み込むエピゲノムのプラットフォームの実装で遅延があった。
特に、DNA メチル化は methylomic プラットフォームを構築するため利用可能なゲノムのデータを活用して DNA に最も広く研究のエピジェネティックな修飾の 1 つであります。1 つのような例は精神医学4,5腫瘍から様々 な分野で用いられている人間のメチローム3に、アレイ ・ ベースのプラットフォームです。残念なことに、ヒト以外の動物のモデルの類似のプラットフォームが不足している、事実上ない広く使われているプラットフォームを活用しているゲノムのシーケンスの最初の設計があります。
ヒト以外の動物モデルの methylomic 風景を評価する一般的な方法は、縮約表現の重亜硫酸塩の配列 (RRBS)6です。このアプローチは、包括的な methylomic 風景を提供しながらコストと2 ゲノムの大規模な遺伝子貧しい地域で限られた機能情報のため下位の読み取りの深さカバレッジを提供する重亜硫酸塩全ゲノム シーケンスのコストを克服します。.RRBS は制限のダイジェストと一般的遺伝子プロモーター付近、遺伝子調節7に役割を果たすと考え CpG の島など高い GC 豊富なシーケンスの豊かにするゲノム DNA のサイズ選択に含まれます。RRBS 法は重要な研究の数で使用されています、制限酵素への依存はない注目すべき課題と制限なし。例えば、RRBS の GC 豊富なシーケンスの濃縮は電気泳動によって制限酵素とその後のサイズ選択によって認識特定シーケンスの存在に完全に依存。これはサイズの選択中にこれらの制限のサイトが含まれていない任意のゲノム領域を除外することを意味します。また、異種間の比較は、同じ制限のサイトが異なる種の間で同じ遺伝子座で存在しない限りに挑戦しています。
RRBS の限界を克服する方法の 1 つは、プラットフォームの設計、公開されたゲノム シーケンスの活用強化方法を使用することです。アレイ ・ ベースの人間プラットフォームは、対立遺伝子特定 (重亜硫酸塩の変換後 TG 対 CG) ターゲット焼鈍およびプライマー拡張機能の特定の Cpg に対するプライマー プローブを使用します。そのデザインだけでなく利用できる人間のゲノム シーケンス、エンコードと ENSEMBL8など、お問い合わせの複数行から取得した実験的検証規制地域を反映します。その広い使用にもかかわらず人間の methylomic の調査で、モデル動物に類似したプラットフォームが存在しません。さらに、アレイ ・ ベースの形式は、プローブ配置可能な表面積の大きな制約を配置します。過去数年間、キャプチャ プローブ設計と次世代シーケンシングの高スループット機能によって与えられるターゲット特異性を結合する努力をしました。このような努力は、メチル化9,10脳固有または糖質コルチコイド誘発性の違いを識別するために使用されたマウスのゲノム (マウス メチル Seq) のシーケンス ベースのターゲット濃縮システムになりました。他モデルと非モデル動物の類似のプラットフォームは、これらの動物のエピゲノム研究を容易にするために必要です。
ここでは、ラットの methylomic 分析を実施するこの新しいプラットフォームの実装を示す.ラットは薬理学、代謝、神経内分泌学、および動作に重要な動物モデルを務めています。たとえば、薬物毒性、肥満、ストレス応答、または薬物中毒を生じさせる基になるメカニズムを理解がますます必要があります。これらの条件に関連付けられている methylomic の変更をキャプチャできる高速プラットフォームのメカニズムの私達の理解を増加させます。ラットのゲノムはまだ規制地域の注釈を欠いている、以来、私たちは非冗長のプロモーター、CpG の島、島の海岸11を組み込むし、以前ラット メチル Seq プラットフォーム12に GC 豊富なシーケンスを識別しました。
特異的メチル化する慢性変動応力 (CVS)13のラットモデルを用いて、ラットのゲノムの SureSelect ターゲット濃縮 (総称、メチル Seq) プラットフォームの成功の設計と実装を評価するには強勢と強調した動物との間の領域。当社のプラットフォームの設計、プロトコル、および実装は、生物ゲノム シーケンスですでに入手可能ですが、不十分な注釈付きのままの包括的かつ公平なエピジェネティックな調査を実施する必要がある研究者の役に立つかもしれません。
プロトコル
やガイドラインの遵守すべて関連する規制・制度、機関動物ケアとジョンズ ・ ホプキンス医学利用委員会などを含むすべての実験を行った。
1. 動物
- 4 週齢で男性思春期ラットを取得します。温度でポリカーボネート ラット ケージに動物の家-12 h、12 h 暗いサイクル 0600 h で光で発症した部屋の湿度制御を提供する水に自由にアクセスできる動物と。
- 輸送に伴うストレスを減らすために 1 週の順応するラットを許可します。ペア住宅分離ストレスを排除し、5 週齢、慢性的な変数を開始 (N = 16) 動物は、3 週間 (CVS) 療法を強調します。
2. 慢性変動応力
- 予測不可能なルーチンを維持する不規則な時に一度朝 (9-11) と午後 (1-15) に、CVS の養生法を管理します。一晩穏やかなストレッサーを組み込みます。「CVS 療法が含まれています: 1) 3 h 拘束シリンダー;2) 10 分泳ぐ;3) 3 h ケージ傾斜 4) 1 h 遅い振動プラットフォーム;・ 5) 1 h 4 ° C の冷たい部屋で。
注: 一晩ストレス社会叢生 (ケージごと 5)、社会的孤立、濡らした寝具、食事制限、ライトにあります。ストレス療法の典型的な週単位のスケジュールは、表 1に提供されます。
3. 内分泌を試金します。
-
コルチコステロン (CORT) 尾血 (~ 50 mL) サンプリングを使用してのレベル収集、実験を通して週 2 回同時に (9) CVS 養生法を前に毎週の CVS の途中で一度ベースライン ホルモン レベル (0 日目) の確立を決定 (日4,11 と 18 日)、CVS のすべての 7 日後 (日 7、14)、CVS (21 日) の結論。毎日ストレス療法前に血液サンプルを収集します。
- RIA とゲノム DNA の抽出の安楽死 (25 日) の間に 1 つの最終的なトランクの血液サンプルを収集します。
- (600 x g、4 ° C, 10 分) 血液細胞から血漿を分離するすべての血液サンプルを遠心します。ピペット プラズマ (清) と試料-80 ° C で保存
- 解凍し、ラジオイムノアッセイ (RIA) が CORT のレベルを決定するため、プラズマを使用します。3 週間血中 CORT ストレス療法の堅牢性を確認するストレス動物の上昇しているを確認します。
4. 動作
- CVS の養生法の後 (23-24 日)、高架上の不安様行動のそれぞれの動物を評価プラス (EPM)14の迷路。
- 300 s と時間、センターでスコア EPM 装置上の動物ビデオ カメラのレコードを使用して、腕を閉じるし、オープン腕を。
5. ラット メチル Seq のデザイン
- UCSC のゲノムのブラウザーを使用して、取得 CpG の島と島の海岸 (± 1 kb の CpG の島の側面)、各 RefSeq 遺伝子とから利用可能な他のシーケンスのプロモーター (± 各 TSS の 1 kb) の非冗長のゲノム座標 (ラットは rn4 アセンブリの 2004 年 11 月)関連する文献。
注意: ラット メチル Seq の以前のアレイ ・ ベースのメチル化プラットフォームから追加の GC 豊富なシーケンスは12を追加されました。5 kbps を超える地域、スキップされた 1 kbps に続いて、500 bps の交互になる地域採取。230 万 Cpg; 111 Mbps から成っている最終的なラット メチル Seq デザイン594 bps の地域の平均サイズ。228,800 ユニークな軌跡を対象します。 - 市販ターゲット キャプチャ デザイン ソフトウェアに適切なプローブ デザインのゲノム座標のコンパイルされたリストを入力します。
6. Genomic DNA からラット メチル Seq ライブラリーの構築
注: バッチの効果を除去するために複数のサンプルを同時に処理し、それに応じてスケール マスター ミックス アップ。市販 DNA 抽出キットを使用して DNA を抽出します。高品質 DNA 収量カラムベースまたは沈殿法 (260/280 比 1.8 〜)。フェノール ベースのメソッドの使用は推奨されません。溶出または低 TE バッファー (10 mM テ、0.1 ミリメートルの EDTA、pH 8.0) で DNA を再懸濁します。
- サンプル準備
注: すべてのステップは DNA 結合の磁気ビーズを用いて、確認ビーズの少なくとも 30 分室温に慣れるし、使用前によく混合します。- DNA をせん断
- 各サンプルの初期の二重鎖 DNA の濃度を決定するため、蛍光光度計を使用します。希釈 > gDNA 低 TE バッファー (10 mM テ、0.1 ミリメートルの EDTA、pH 8.0) 低 DNA 結合マイクロ遠心チューブ用の 50 μ L に 1 μ g。
- 等温超音波発生装置を使用してサンプルをせん断 (10% のデューティ サイクル、5 強度、バースト、60 s、周波数掃引、4 ° C の 6 サイクルあたり 200 サイクル)。
- DNA のサイズと量を測定する電気泳動に基づくシステムを使用して DNA の品質を評価します。
注: 推奨 DNA 量は 1 μ g または 3 μ g です。最低の入力量をする必要がありますが開始材料限られている場合、> 500 ng、低額に悪影響が生成ライブラリの質と量。
- DNA 修復を終了します。
- ラット メチル Seq キットを使用して、氷の上終わり修理マスター ミックスを調製します。各サンプルにミックスの 52 μ L を追加し、サーマルサイクラー温水ふた (20 ° C 30 分、4 ° C ホールド) なしで孵化させなさい。
修理-終わりマスター ミックス (サンプルごと)。
水の 35.2 μ L
最後修理バッファーの 10 μ L (10 倍)
DNTP ミックスの 1.6 μ L
T4 DNA ポリメラーゼの 1 μ L
Klenow DNA ポリメラーゼの 2 μ L
T4 ポリヌクレオチド キナーゼの 2.2 μ L - DNA 結合磁性体ビーズ 180 μ L と 1 サンプルあたり作りたての 70% のエタノールの 400 μ L を使用してサンプルを浄化します。各サンプルにビーズの 180 μ L を追加し、室温で 5 分間インキュベートします。ビーズをペレット、上清を除去、70% エタノール 200 μ L でペレットを再懸濁します。エタノールを削除し、一度洗浄を繰り返します。
- 磁気プレートを使用してビーズをペレットし、できるだけ多くのエタノールを削除します。37 ° C で乾燥 heatblock ビーズ ペレットが完全になるまで 3-5 分間乾燥させます。ヌクレアーゼ フリー水 44 μ で再懸濁します、上澄みの約 42 μ L を収集します。
停止ポイント: DNA 修復が終了した後のサンプルがありますに密封され、-20 ° C で保存
- ラット メチル Seq キットを使用して、氷の上終わり修理マスター ミックスを調製します。各サンプルにミックスの 52 μ L を追加し、サーマルサイクラー温水ふた (20 ° C 30 分、4 ° C ホールド) なしで孵化させなさい。
- アデニル酸 3' を終了します。
- 氷上アデニル マスター ミックスを調製します。各サンプルに 9 μ L ミックスを追加し、サーマルサイクラー温水ふた (37 ° C 30 分、4 ° C ホールド) なしで孵化させなさい。
アデニル マスター ミックス (サンプルごと):
Klenow バッファーの 5 μ L
DATP の 1 μ L
Klenow DNA ポリメラーゼの 3 μ L - DNA 結合磁性体ビーズ 90 μ L と 1 サンプルあたり作りたての 70% のエタノールの 400 μ L を使用してサンプルを浄化します。各サンプルにビーズの 90 μ L を追加し、室温で 5 分間インキュベートします。ビーズをペレット、上清を除去、70% エタノール 200 μ L でペレットを再懸濁します。エタノールを削除し、一度洗浄を繰り返します。
- 磁気プレートを使用してビーズをペレットし、できるだけ多くのエタノールを削除します。37 ° C で乾燥 heatblock ビーズ ペレットが完全になるまで 3-5 分間乾燥させます。ヌクレアーゼ フリー水の 35 μ L で再懸濁します清約 33.5 μ L を収集しています。
- 氷上アデニル マスター ミックスを調製します。各サンプルに 9 μ L ミックスを追加し、サーマルサイクラー温水ふた (37 ° C 30 分、4 ° C ホールド) なしで孵化させなさい。
- メチル化アダプターを縛る。
- 氷上結紮マスター ミックスを調製し、ミックスの 16.5 μ L を各サンプルに追加します。サーマルサイクラー (20 ° C、4 ° C ホールドの 15 分間) 加熱蓋なしで孵化させなさい。
結紮マスター ミックス (サンプルごと):
水の 2.5 μ L
2.5 μ L メチル Seq のメチル化アダプター
T4 DNA リガーゼ バッファーの 10 μ L (5 x)
T4 DNA リガーゼの 1.5 μ L - DNA 結合磁性体ビーズ 90 μ L と 1 サンプルあたり作りたての 70% のエタノールの 400 μ L を使用してサンプルを浄化します。各サンプルにビーズの 90 μ L を追加し、室温で 5 分間インキュベートします。ビーズをペレット、培養上清を削除および 70% のエタノールの 200 μ L でペレットを再懸濁します。エタノールを削除し、一度洗浄を繰り返します。
- 磁気プレートを使用してビーズをペレットし、できるだけ多くのエタノールを削除します。37 ° C で乾燥 heatblock ビーズ ペレットが完全になるまで 3-5 分間乾燥させます。ヌクレアーゼ フリー水の 22 μ L で再懸濁します、上澄みの約 22 μ L を収集します。バイオアナライザーを使用して品質を評価します。
注: DNA の総容量は 500 未満 ng, せん断, およびプロセス追加 DNA 以降の手順に進む前に。30 bps 以上で平均 DNA サイズが増えない場合は、試薬の新しい、T4 DNA ポリメラーゼ、Klenow、または T4 リガーゼが古い可能性がありますを確認します。
停止ポイント: メチル化アダプターを縛ること後のサンプルがありますに密封され、-20 ° C で保存
- 氷上結紮マスター ミックスを調製し、ミックスの 16.5 μ L を各サンプルに追加します。サーマルサイクラー (20 ° C、4 ° C ホールドの 15 分間) 加熱蓋なしで孵化させなさい。
- DNA をせん断
- 交配
- 低 DNA 結合マイクロ遠心チューブ用にサンプルを転送し、未満 3.4 μ L. 西川 3.4 μ L をサンプルにサンプル ボリュームを抑える加熱真空濃縮を使用します。
注: 集中サンプルがすべて液体の前に真空濃縮から削除されることを確認する約 〜 3 μ L のサンプルを蒸発させます。 - 常温で氷の上のメチル Seq ブロック ミックス交配バッファーを準備します。各サンプルにメチル Seq ブロック ミックスの 5.6 μ L を追加し、サーマルサイクラー (95 ° C 65 ° C ホールド 2 分の 65 ° C 5 分) で孵化させなさい。
交配バッファー (サンプルごと):
6.63 メチル Seq Hyb 1 μ L
メチル Seq Hyb 2 0.27 μ
メチル Seq Hyb 3 の 2.65 μ L
メチル Seq Hyb 4 3.45 μ
メチル Seq ブロック ミックス (サンプルごと):
メチル Seq インデックス ブロック 1 の 2.5 μ L
メチル Seq ブロック 2 の 2.5 μ L
メチル Seq ブロック 3 の 0.6 μ L - RNase ブロック ミックスとキャプチャ ライブラリ交配の組合せを準備します。各サンプルにキャプチャ ライブラリ交配の組合せの 20 μ L を追加し、65 ° C 少なくとも 16 時間インキュベートします。
RNase ブロック ミックス (サンプルごと):
RNase ブロックの 0.5 μ L
水の 1.5 μ L
(サンプルごと) ライブラリ交配の組合せをキャプチャします。
交配バッファーの 13 μ L
RNase ブロック ミックスの 2 μ L
ラット メチル Seq の 5 μ L をキャプチャ ライブラリ
注: は、非特異的結合を防ぐために交配の組合せを追加する場合、 65 ° C で反応を続ける。 - 新しい 8 よくストリップ管にサンプルあたりストレプトアビジン磁気ビーズの分注 50 μ L。200 μ L メチル Seq 結合バッファーのビード洗浄しなさい。磁気プレートを使用して、ビーズをペレットし、3 洗浄の合計のための各洗浄の上清を除去します。最終的な洗浄の後に、メチル Seq 結合バッファーを 200 μ l 添加のストレプトアビジン ビーズを再懸濁します。
- 洗浄ストレプトアビジン磁気ビーズを 200 μ l 添加するサンプルを追加し、回転ミキサーを使用して 30 分間室温でインキュベートします。96 ウェル プレート サンプルと事前 65 ° C に温めてサーマルサイクラーの場所ごとの帳票の井戸にメチル Seq 洗浄バッファー 2 の因数の 200 μ L を混合しながら
- インキュベーション後、磁気板を用いたストレプトアビジン磁気ビーズをペレットし、ビーズを 200 μ L で再懸濁しますメチル Seq 洗浄バッファー 1。室温で 15 分間インキュベートします。ペレット、上澄みを廃棄し、磁気プレートを使用します。
- ビード メチル Seq 洗浄バッファー 2 で 3 回洗浄しなさい: 洗浄バッファー 2 (手順 6.2.5 で予め加温) 200 μ L でビーズ ペレットを再懸濁します、サーマルサイクラー (65 ° C, 10 分) のビーズをインキュベートし、ビーズをペレットします。磁気プレートを使用して各洗浄後上澄みを廃棄します。
注: は、非特異的結合を防ぐために洗浄バッファー 2 を追加する際に交配反応 65 ° c を維持します。 - 洗浄のビーズにメチル Seq 溶出バッファーの 20 μ L を追加し、20 分使用ペレット ビーズと新しいストリップ チューブに上清転送磁気プレートは室温で孵化させなさい。ビーズを破棄します。
注: 抱卵中の重亜硫酸塩の変換試薬を準備します。
- 低 DNA 結合マイクロ遠心チューブ用にサンプルを転送し、未満 3.4 μ L. 西川 3.4 μ L をサンプルにサンプル ボリュームを抑える加熱真空濃縮を使用します。
- 重亜硫酸塩の変換
注: 適切な試薬と市販の重亜硫酸塩の変換キットからの指示を使用して溶出 ssDNA の重亜硫酸塩の変換を実行します。- 前の手順から、上澄みに 130 μ L 準備重亜硫酸塩の変換試薬を追加します。2 つの井戸に均等に 150 μ L 反応のそれぞれに分割します。サーマルサイクラー (2.5 h、4 ° C ホールドの 64 ° C) で孵化させなさい。
注: 150 μ L 反応は、均質な温度を確保するため 2 つの井戸に均等に分かれています。2.5 h をインキュベートした後すぐに次のステップに進みます。 - バッファーのバインドの 600 μ L を追加することによって列をスピン洗浄バッファーの 100 μ L で 1 回洗浄しサンプルをバインドします。遠心分離機 (15,000 × g、1 分) の列のすべての重亜硫酸塩の変換手順の間、流れを破棄します。
- Desulphonation バッファーの 200 μ L を列に追加することによって Desulphonate のサンプル。15-20 分繰り返し遠心室温でインキュベートし、流れを破棄します。
- 200 μ L の洗浄バッファーで 2 回列を洗います。10 μ L の溶出バッファーを列、室温で 3 分間インキュベートし、遠心分離 (15,000 × g、1 分) に追加することによって各サンプルを溶出します。20 μ L の合計のため溶出のステップを繰り返します。
- PCR 反応氷の上のマスター ミックス 1 を準備します。ミックスの 82 μ L を各サンプルに追加します。次のプログラムでサーマルサイクラーで孵化させなさい。
PCR 反応マスター ミックス 1 (サンプルごと):
水の 30 μ L
メチル Seq PCR マスター ミックスの 50 μ L
メチル Seq PCR1 プライマー F の 1 μ L
メチル Seq PCR1 プライマー R の 1 μ L
サーマルサイクラー プログラム:
ステージ 1、1 サイクル: 95 ° C 2 分
ステージ 2、8 サイクル: 95 ° C 30 s, 60 ° C 30 s, 72 ° C 30 s
ステージ 3、1 サイクル: 72 ° C 7 分
ステージ 4、1 サイクル: 4 ° C ホールド - DNA 結合磁性体ビーズ 180 μ L と 1 サンプルあたり作りたての 70% のエタノールの 400 μ L を使用してサンプルを浄化します。各サンプルにビーズの 180 μ L を追加し、室温で 5 分間インキュベートします。ビーズをペレット、上清を除去、70% エタノール 200 μ L でペレットを再懸濁します。エタノールを削除し、一度洗浄を繰り返します。
- 磁気プレートを使用してビーズをペレットし、できるだけ多くのエタノールを削除します。37 ° C で乾燥 heatblock ビーズ ペレットが完全になるまで 3-5 分間乾燥させます。ヌクレアーゼ フリー水の 21 μ L で再懸濁します、上澄みの約 19.5 μ L を収集します。
- 前の手順から、上澄みに 130 μ L 準備重亜硫酸塩の変換試薬を追加します。2 つの井戸に均等に 150 μ L 反応のそれぞれに分割します。サーマルサイクラー (2.5 h、4 ° C ホールドの 64 ° C) で孵化させなさい。
- インデックスの作成
- 反応 PCR マスター ミックス 2 氷の上を準備します。25.5 μ L を追加する各サンプルのマスター ミックス 2。個々 のサンプルにプライマーをインデックス商業 5 μ l 添加し熱 cycler で孵化させなさい。
PCR 反応マスター ミックス 2 (サンプルごと):
25 μ L メチル Seq PCR マスター ミックス
0.5 μ L メチル Seq 共通インデックス プライマー
サーマルサイクラー プログラム:
ステージ 1、1 サイクル: 95 ° C 2 分
ステージ 2、6 サイクル: 95 ° C 30 s, 60 ° C 30 s, 72 ° C 30 s
ステージ 3、1 サイクル: 72 ° C 7 分
ステージ 4、1 サイクル: 4 ° C ホールド
注: 追加サイクル (2-3) 開始の DNA 濃度が推奨値未満の場合は必要があります。 - DNA 結合磁性体ビーズ 90 μ L と 1 サンプルあたり作りたての 70% のエタノールの 400 μ L を使用してサンプルを浄化します。各サンプルにビーズの 90 μ L を追加し、室温で 5 分間インキュベートします。ビーズをペレット、上清を除去、70% エタノール 200 μ L でペレットを再懸濁します。エタノールを削除し、一度洗浄を繰り返します。
- 磁気プレートを使用してビーズをペレットし、できるだけ多くのエタノールを削除します。37 ° C で乾燥 heatblock ビーズ ペレットが完全になるまで 3-5 分間乾燥させます。ヌクレアーゼ フリー水の 24 μ L で再懸濁します、上澄みの約 24 μ L を収集します。
- 濃度と bp のサイズ、バイオアナライザーに高感度 DNA 検出試薬を使用して評価します。
メモ: バイオアナライザー DNA ライブラリの存在を検出する場合は、追加の DNA の準備の手順を繰り返します。
停止点: 浄化後インデックス付きサンプル密封可能性があり、-20 ° C で保存 - 使用する適切な次世代シーケンシング プラットフォームのサンプルのプール。
- ライブラリのサイズと指定されたボリュームの数量に基づいて DNA モル濃度を決定するバイオアナライザー濃度データを用いて低 TE バッファー (6.1.1.1) と希釈し、15 の最終的な集中にすべてのサンプルを組み合わせた午後。
注: ライブラリを定量化のより敏感な方法は、結紮アダプターを対象とするプライマーを使用して量的なリアルタイム PCR です。 - 次世代シーケンサーのレーンあたり 4 サンプルの十分な車線数にプールされたサンプルを実行します。
注: たとえば場合 16 ライブラリのサンプルは一意にインデックスを作成し、結合、実行ライブラリ以上 4 車線、車線あたりのサンプル数 4 に相当。
- ライブラリのサイズと指定されたボリュームの数量に基づいて DNA モル濃度を決定するバイオアナライザー濃度データを用いて低 TE バッファー (6.1.1.1) と希釈し、15 の最終的な集中にすべてのサンプルを組み合わせた午後。
- 反応 PCR マスター ミックス 2 氷の上を準備します。25.5 μ L を追加する各サンプルのマスター ミックス 2。個々 のサンプルにプライマーをインデックス商業 5 μ l 添加し熱 cycler で孵化させなさい。
7. 次世代シーケンサーのシーケンス
- 次世代シークエンシング マシンでシーケンスが続くメチル Seq ライブラリのクラスタ リングの制度的シーケンス コアにサンプルを送信します。
8. Dmr を識別する分析
- ビスマルク15、亜硫酸水素変換、プラス鎖のゲノムを生入力読み取りを合わせて、内部シーケンス アライナ16,17、ボウタイ 2.0 を起動するを実装します。次の配置、品質管理を行い、各 CpG メチル化推定値を代入する、Bismark_methylation_extractor を使用します。
- Bioconductor で BS Seq パッケージ18 Dmr のリストを生成します。フィルター、Dmr が 3 連続した Cpg と P 値よりも大きいことに基づいて < 0.05。
注: ゲノム座標、最寄りの RefSeq 遺伝子までの距離、それぞれの DMR、平均 %cpg メチル化値 P 値 (例えば、強勢と強調)、2 つの比較のグループ DMR を内の Cpg の数が含まれています DMR リストを生成し、FDR (虚偽の発見率) の値。すなわちDMR リストを使用します。、検証パイロシークエンス プライマーを設計するためのゲノム座標。
9. 重亜硫酸塩ピロシーケンスによる検証
-
プライマー デザイン
- 重亜硫酸塩 PCR のためのプライマーおよびパイロシークエンス設計します。Nested pcr 法は、DMR の 150-400 bps を増幅するように (外と入れ子になった)、PCR のプライマーの 2 つのセットをデザインします。
注: 一般に、設計されたプライマーは少なくとも 24 拠点のアニール温度シーケンスの複雑さの損失からアカウントに少なくとも 4-5 連続しない G の (C の逆プライマー) と長い。ビオチン標識し、HPLC 精製入り込まれたプライマーの 1 つになります。ただし、標準的なプライマーは agarose のゲルの反応を解決することによって PCR のステップを最適化する最初を注文する必要があります。- デザイン、パイロシークエンス プライマーをアッセイは、相補的なビオチンを対象はちょうど 1-2 塩基上流に試金する Cpg の鎖します。各パイロシークエンス プライマー 30 bps 下流を分析することが確実に、必要に応じて複数パイロシークエンス プライマーを設計します。
- Rt1-m4 は、次を使用します。
外 – F TGTAYGATTTTGGTTATYGTAAAT rRT1M4
外-R AACTTACAAATTTCACCAACTCA rRT1M4
rRT1M4 ネスト-F GTGGGTTAYGTGGATAATATATAG
rRT1M4 ネスト-R AATCACTTACCATTCTCTCTCTAACTA
rRT1M4 Pyro1 TAYGTGGATAATATATAGAT
rRT1M4 Pyro2 GATAGTTATTTGGYGAGTTAG
rRT1M4 Pyro3 GAGTATTTGGAGGAGTTGAT
rRT1M4 Pyro4 GGATTTTAATATTTGGT
- 重亜硫酸塩 PCR のためのプライマーおよびパイロシークエンス設計します。Nested pcr 法は、DMR の 150-400 bps を増幅するように (外と入れ子になった)、PCR のプライマーの 2 つのセットをデザインします。
-
ラット血液 gDNA の重亜硫酸塩の変換のため、市販のキットを使用します。
注: 重亜硫酸塩の変換の手順は、次の変更と市販キットから適応されている: ステップ 1 で、50-100 ng の血 gDNA を追加し、20 μ L を水で希釈します。ステップ 9 でサンプルごとの 20 μ L を溶出します。- 重亜硫酸塩の変換試薬製造元のプロトコルに従ってを準備し、希釈された gDNA を組み合わせます。サーマルサイクラー (2.5 h、4 ° C ホールドの 64 ° C) で孵化させなさい。
- スピン列および遠心分離機 (15,000 × g, 1 分) で変換された gDNA をバッファーのバインドを追加します。洗浄列は、一度 Desulphonation バッファーを列に追加し、室温で 15 分間インキュベートします。遠心分離機 (15,000 × g、1 分)。
- 洗浄バッファーと遠心分離機 (15,000 × g、1 分) 列を洗います。遠心分離 (15,000 × g、2 分) と洗浄手順を繰り返します。20 μ l 添加溶出バッファーと遠心分離機 (15,000 × g、1 分) が溶出します。
-
PCR の拡大
- 外の PCR マスター ミックスを準備します。3.5 μ L の重亜硫酸塩の変換された gDNA をマスター ミックスの 21.5 μ L を追加し、サーマルサイクラー プログラムを実行します。
外 PCR マスター ミックス:
水の 16.25 μ L
ポリメラーゼ バッファーの 2.5 μ L [10 x]
DNTP [10 mM] の 0.5 μ L
前方のプライマーの 1 μ L [0.1 μ M]
逆のプライマーの 1 μ L [0.1 μ M]
Taq DNA ポリメラーゼの 0.25 μ L [5000 U/mL]。
サーマルサイクラー プログラム:
ステージ 1、1 サイクル: 94 ° C 4 分
ステージ 2、47 サイクル: 94 ° C 1 分、53 ° C 30 s 72 ° C 1 分
ステージ 3、1 サイクル: 72 ° C 8 分、4 ° C ホールド - ネステッド PCR マスター ミックスを準備します。外の PCR から 2 μ L のサンプルをマスター ミックスの 23 μ l 添加し PCR サーマルサイクラーの外部プログラムを繰り返します。(1 x TAE バッファー、1% の agarose のゲル) のゲル電気泳動による PCR の製品の品質を評価します。
ネステッド PCR マスター ミックス:
水の 17.75 μ L
ポリメラーゼ バッファーの 2.5 μ L [10 x]
DNTP [10 mM] の 0.5 μ L
前方のプライマーの 1 μ L [0.1 μ M]
逆のプライマーの 1 μ L [0.1 μ M]
Taq DNA ポリメラーゼの 0.25 μ L [5000 U/mL]
注: ネストされた PCR のため順方向または逆プライマーでなければならないビオチン化
- 外の PCR マスター ミックスを準備します。3.5 μ L の重亜硫酸塩の変換された gDNA をマスター ミックスの 21.5 μ L を追加し、サーマルサイクラー プログラムを実行します。
-
パイロシークエンス
- バッファーのバインドの 38 μ L、水の 35 μ L および 2 μ L のサンプルあたりストレプトアビジン コーティング セファローズ 4 b ビーズを含むマスター ミックスを作る。96 ウェル プレートでのマスターの組合せの 75 μ L とネストされた PCR の製品の 5 μ L を追加します。15-60 分間プレート シェーカーを振る。
- 揺れながらパイロシークエンス アッセイ プレートのウェルにプライマー (0.5 μ M で希釈アニーリング バッファー) の 12 μ L を追加します。
- 後揺れ、結合反応洗浄バッファーを使用して洗浄手順を実行します。水で満たされた谷で真空ツールを配置し、板からサンプルを収集します。70% エタノール、水酸化ナトリウム (0.2 M)、トリス酢酸緩衝液 (10 mM、pH 7.4) を含む半分満たされた谷で真空ツールが水没します。ビーズを転送する HS アッセイ プレートの真空と場所真空ツールから外します。
- ヒート ブロックにプレートを置き、2 分 5 分間冷却し、パイロ プログラムを開始許可プレートの 80 ° C で孵化させなさい。
結果
ラット メチル Seq プラットフォームの成功の実装は、いくつかの条件によって異なります。図 1研究の全体的なワークフローと進む前に必要な特定の品質管理 (QC) 手順を強調表示します。考慮すべき最初の要因の 1 つは、メチロームの間で発生するエピジェネティックな変化の大きさを決定する動物モデルおよびストレス療法の堅牢性です。私たち慢性変動応力 (CVS) 療法を生成するための十分な厳しさのする必要が以来、私たち動物の仕事はコルチコステロン (CORT) 露出は DNA メチル化19,20の変化につながることができます私たちの前の観察に基づいて、ラット血中 CORT レベルを強調しました。典型的な毎週 CVS 療法は表 1に示すように、成っていた日常ストレッサーの午前、午後と一晩慣れを防ぐためには常に変更されて、ストレス反応を減少します。3 週間の養生法の中で重点を置かれた動物展示血中 CORT のレベルが大幅上昇 [日 4-21 を制御: 32.7 3.7 ng/mL、ストレス: 103.0 11.9 ng/mL (平均 SEM)、P = 10-4、図 2 ax 2.2] 強勢、上動物を制御します。一貫して、これらの動物はまた EPM と短い時間で (図 2 b) 開いて腕の中で閉じられた腕の中で大幅により多くの時間によって示されるように、高架上の大きい不安様行動プラス迷路 (EPM) を示した。これらの結果は、重要な内分泌およびこれらの変更された特定の DNA のメチル化の署名に関連付けられているかどうかを調査するために私たちをリードして、行動の変化につながった CVS 露出を示しています。
我々 は成功したメチル Seq 図書館建設に不可欠ないくつかのチェックポイントを強調します。超音波処理、複数の洗浄・精製、ターゲット濃縮する必要は十分な量の DNA から始まる、重亜硫酸塩の変換の手順が順次完成したライブラリの DNA の量を減らします。ただし、PCR 増幅のいくつかのステップでは、DNA テンプレートの損失を軽減するため、過剰な PCR のサイクル数は高い重複読み取りを導入できます。現在のラット メチル Seq 研究ラットごと gDNA を血液の 2 g 使用しています。我々 は DNA 量として最低 500 とメチル Seq ライブラリを作ることができる注意してください ng。小さい材料を開始できます (蛍光活性化細胞ソーティング) FACS により分離された DNA からライブラリを生成またはニードル パンチ、後続のシーケンスのためのライブラリの不足量を生産のリスクの増加はあるものの。QC は、DNA 分子の重量、数量、およびモル濃度を提供のバイオアナライザーのサンプルの 1 L の電気泳動によって実行されます。バイオアナライザーの使用を必要とする 3 つの重要なステップは、: 1) (~ 170 bp、赤、図 3) DNA の十分な剪断を確認する超音波処理ステップを次せん断の DNA (~ 200 bp、青、図 3) pcr 法による、その後増幅を確保するための平均サイズの変化によって示される 2) 次のアダプターの ligation のステップ・ 3) 数量とシーケンス処理用ライブラリのサイズを確保する最後のライブラリの精製ステップを次します。
R パッケージ BSSeq と Bioconductor の BSmooth シーケンス データ18重亜硫酸塩の分析に使用されました。彼らは、ツール、品質制御を実行するシーケンス リードを整列させるための方法と領域 (Dmr) をメチル化特異的に識別します。BSmooth ソフトウェアは、ゲノム配列の重亜硫酸塩変換する raw 入力読み取りの配向による CpG レベル測定の概要を取得する内部シーケンス アライナとボウタイ 2.016,17を呼び出します。整列の読み取りは、体系的な配列および下流解析が偏る可能性がありますベースの呼び出しエラーを特定しようとすると、厳密な品質管理手順を過されています。プロットのシリーズは、視覚的にフィルタ リングするこのプロセスを支援するために生成されます。シーケンスのメトリックがドキュメント関連情報他 (表 2) の中の CpG カバレッジあたりと一直線に並べられた読み取り、% ターゲットの数なども生成されます。一度データがフィルタ リング、スムージング/正規化アルゴリズムを実行する、すべての CpG がすべて QC に基づいて推定されるメチル化値を割り当てられている各サンプルから読み取り、Cpg メチル化のより正確な通話を確保するための近隣からの見積もりシーケンスのカバー率が低い場合でも、ステータス。この値は、各 CpG サイトのメチル化の確率の平滑化された推定値を提供します。2 つの治療群と以上に最も大きく異なるランキング ゲノム領域間の各サンプルの平滑化メチル化の見積もりの平均値を比較すると、Dmr の一覧が生成されます (表 3)。
応力負荷及び無負荷グループ間 DMR とラット主要組織適合性遺伝子Rt1 m4のプロモーターに位置していたトップは、アクセントのない動物 (図 4 a) よりもすべての Cpg のメチル化レベルを展示動物を強調しました。DMR と強調し、アクセントのない動物 (メチル Seq でシーケンス 8 やないシーケンス 8) の全体のコホートにおける血液 DNA メチル化レベルに対して設計されたプライマー メチル Seq プラットフォームとデータ分析の成功の実装を確認するには重亜硫酸塩パイロシークエンスによって評価しました。結果は試金される 12 の Cpg から 10 間で DNA のメチル化の大幅な増加を示す (5.1-10.4% メチル化、P 変化 < 0.037、図 4 b)。KEGG パスウェイ解析は、すべてのストレスに関連付けられている経路を識別するために重要な名目上の Dmr で実行されました。一貫して、DMR 関連付けられている経路には、糖尿病、心血管疾患、がん (表 4) などの慢性的なストレス暴露と関連する疾患が関与しています。21,22,23 10 CpG のメチル化レベルはストレスへのエピジェネティックなデータと露出度と関連を示すためには、それぞれの動物の平均 3 週 CORT レベルに比較しました。結果、ささやかな、内分泌およびメチル化のデータ相関 (R2= 0.54、P = 0.001、図 5)。
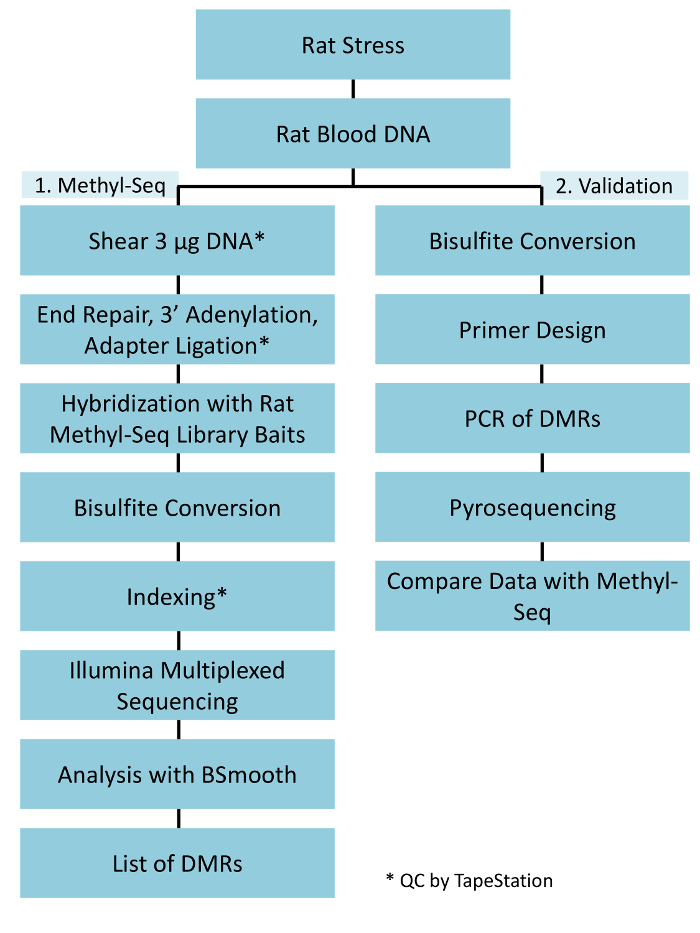
図 1: ラット メチル Seq プラットフォームの全体ワークフロー 。1 g の血液から抽出したゲノム DNA の強調し、対照群は最初配列決定、分析、およびターゲット識別のためメチル Seq ライブラリを構築するために処理します。もう一つ 100 ng の DNA の特定のエピジェネティック ターゲットの独立した検証重亜硫酸塩パイロシークエンスによって使用されます。この図の拡大版を表示するのにはここをクリックしてください。
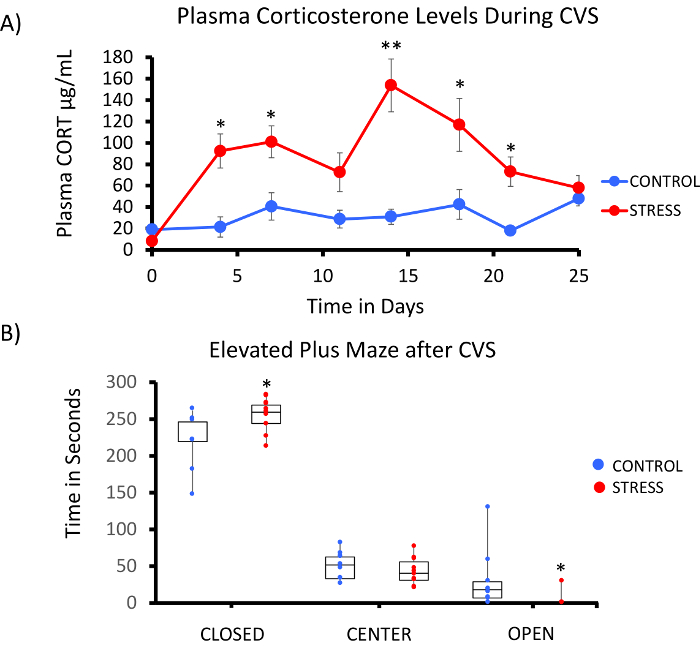
図 2: 慢性変動応力 (CVS) への暴露がラットの内分泌と行動の変化につながります。(A) コルチコステロン (コート) の複数サンプリング デモンストレーションの堅牢性 3 週 CVS 療法。朝は毎日ストレス療法前に採血。(B) 強調した動物は高架式の開いた腕かの時間プラス迷路 (EPM) なり、閉じられた腕の中でより多くの時間を過ごした。それぞれの動物のデータ ポイントとひげが表示されます。統計的有意性のスチューデントの T 検定を行った。* P < 0.05 * * P < 0.01 と * * * P < 0.001。この図の拡大版を表示するのにはここをクリックしてください。
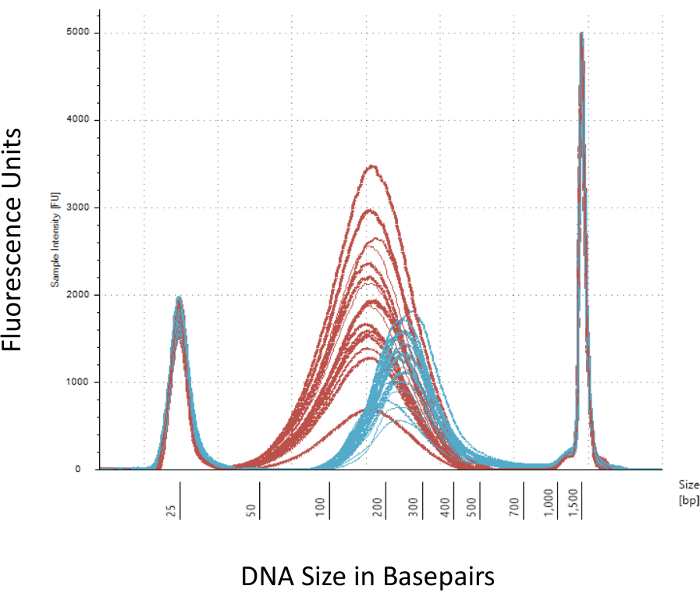
図 3: せん断とアダプター結紮ラット DNA をバイオアナライザーの定量します。赤と青の曲線は、量と次の等温超音波発生装置のアダプターの ligation それぞれせん断ゲノム DNA (赤) のサイズを表示します。各行には 1 つのサンプルと、赤と青の曲線 (終了-修理、3'-アデニル、およびサンプルのクリーンアップ) いくつかのステップの間に DNA の両方の損失を反映し、アダプターの結紮による bp サイズの増加します。鋭いピーク 25 bp、1500 bp が読み込みバッファーに追加されている標準のマーカー。この図の拡大版を表示するのにはここをクリックしてください。
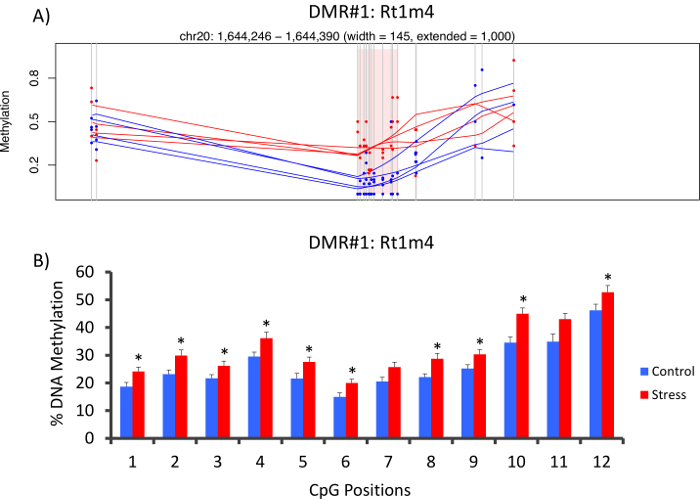
図 4: CVS によるエピジェネティックな変化がラット メチル シーケンスで検出されたラット メチル Seq データ (A) 解析が強調 (赤) と制御 (ブルー) ラット特異的メチル化領域 (DMR) としてRt1m4遺伝子のプロモーターを関与しています。Rt1m4 DMR (ピンクの灰色の領域) のグラフィカルな出力は、各グループ (赤または青のライン)、およびそれぞれの動物 (赤または青ドット) の % メチル化レベルで各 CpG (灰色の垂直線)、4 つのサンプルを表示します。(B) DMR 内 12 Cpg は重亜硫酸塩ピロシーケンスによる検証されました。SEM は、意味としては、バー グラフを表し、統計的有意性のスチューデントの T 検定を行った。* P < 0.05。この図の拡大版を表示するのにはここをクリックしてください。
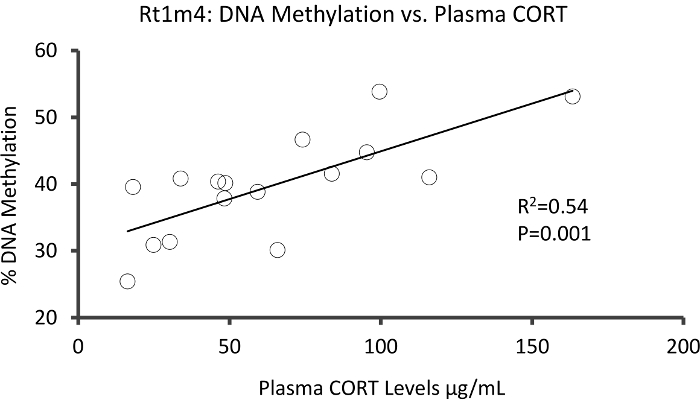
図 5: 線形回帰分析を示し, %dna のささやかな相関Rt1m4の 10 分の CpG のメチル化と 3 週プラズマ コート レベルの両方のストレスを意味動物 (N = 16) を制御します。重点を置かれた動物からのデータは、赤い円によって表されます。この図の拡大版を表示するのにはここをクリックしてください。
| 1 週間 | 日 1 | 日 2 | 日 3 | 日 4 | 日 5 | 日 6 | 日 7 |
| 午前 | 拘束 | 泳ぐ | 冷蔵室 | 泳ぐ | 拘束 | シェーカー | 泳ぐ |
| 午後 | シェーカー | ケージの傾き | 拘束 | シェーカー | 冷蔵室 | 拘束 | 冷蔵室 |
| 一晩 | 食べ物を制限します。 | 寝具を濡らす | 分離 | 点灯 | 混雑 | 点灯 | 寝具を濡らす |
表 1: 慢性変動ストレス療法 (CVS) の典型的な週間スケジュール。
| シーケンスの指標 | ストレス1 | コントロール1 |
| (n = 4) | (n = 4) | |
| ペア (PER) 最後の読み取り | 89,290,397 | 80,165,674 |
| 一意に割り当てたペア終了読み取り (UMPER) | 39,200,255 | 35,013,406 |
| 配置率/マッピング効率 (UMPER/) | 44% | 44% |
| 重複読み取り (UMPER %) | 73% | 65% |
| 重複 UMPER | 10,481,031 | 12,306,018 |
| 読みの深さカバレッジの平均 (x) (同) | 6 x | 6 x |
| Cpg (N) | 12,056,878 | 12,056,878 |
| Cpg のによって (x) | 2 x | 2 x |
| 少なくとも 10 の読み取り (N) と Cpg | 481,383 | 595,850 |
| 同 (X) とは、少なくとも 10 の読み取り Cpg の | 19 | 19 |
| ターゲット Cpg (プローブ ターゲット領域の完全な重複) に | 1,923,872 | 2,007,638 |
| ターゲット Cpg の (x) 同上 | 7 x | 8 倍速 |
| 少なくとも 10 の読み取り (N) とターゲット Cpg に | 428,249 | 531,419 |
| ターゲットによって (x) とは、少なくとも 10 の読み取り Cpg の上 | 18 倍 | 18 倍 |
| ターゲット (プローブ ターゲット領域の 1 つ以上の塩基対重複 PER) (UMPER) | 8,277,715 | 9,369,523 |
| (重複除外 UMPER)」の目標の % | 78% | 77% |
| ターゲット (総拠点マップ) Mb | 125 mb | 128 mb |
| ターゲットの平均読み取りの深さカバレッジ (x) (同) | 9 x | 10 倍 |
| 1各グループの被験者の平均値に基づくシーケンス メトリック |
表 2: シーケンス ラット メチル Seq プラットフォームから得られる指標です。
| chr | スタート | 終わり | 遺伝子 | 距離 | areaStat | meanDiff | ストレス | コントロール | 方向 |
| chr20 | 1,644,246 | 1,644,390 | RT1 M4 | in_gene | 93.03 | 0.22 | 0.33 | 0.11 | ゲイン |
| chr5 | 160,361,352 | 160,361,564 | LOC690911 | in_gene | -70.75 | -0.19 | 0.72 | 0.91 | 損失 |
| chr3 | 61,138,281 | 61,138,330 | RGD1564319 | 265569 | 61.79 | 0.21 | 0.94 | 0.72 | ゲイン |
| chr2 | 143,064,811 | 143,065,010 | Ufm1 | 8569 | -59.48 | -0.11 | 0.13 | 0.24 | 損失 |
| chr7 | 30,764,111 | 30,764,284 | Ntn4 | in_gene | 57.04 | 0.21 | 0.94 | 0.73 | ゲイン |
| chr17 | 12,469,112 | 12,469,218 | Idnk | 41996 | -50.91 | -0.13 | 0.74 | 0.88 | 損失 |
| chr7 | 47,101,725 | 47,101,930 | Pawr | in_gene | -50.54 | -0.12 | 0.64 | 0.76 | 損失 |
| chr5 | 76,111,248 | 76,111,822 | Txndc8 | 151703 | -50.38 | -0.11 | 0.85 | 0.96 | 損失 |
| chr11 | 80,640,132 | 80,640,356 | Dgkg | in_gene | -50.07 | -0.16 | 0.73 | 0.89 | 損失 |
| chr8 | 71,759,248 | 71,759,411 | Mir190 | 210226 | -47.84 | -0.17 | 0.58 | 0.75 | 損失 |
テーブル 3: トップ 10 特異的メチル化領域。各 DMR の出力テーブルは左から右の列に表示されます: 染色体位置 (chr) 座標 (開始/終了)、遺伝子名、トランスクリプションからの距離は、サイトを開始、差分領域統計間強調し, 対照群 (areaStat)、意味する。メチル化 (meanDiff)、平均のメチル化レベルの各 DMR を強調し、管理グループ (ストレス ・ コントロール)、およびメチル化の方向をコントロールから変更します。
| KEGG パスウェイ用語 | 遺伝子数 | % | P 値 | Benjamini |
| 糖尿病 | ||||
| II 型糖尿病 | 12 | 0.1 | 3.6 x 10-4 | 9.8 x 10-3 |
| 心血管疾患 | ||||
| 血管平滑筋収縮 | 18 | 0.1 | 1.6 x 10-3 | 3.6 x 10-2 |
| 不整脈原性右室心筋症 (ARVC) | 13 | 0.1 | 4.0 x 10-3 | 7.1 の x 10-2 |
| 拡張型心筋症 | 14 | 0.1 | 7.6 x 10-3 | 1.2 倍 10-1 |
| 神経機能 | ||||
| 長期増強 | 11 | 0.1 | 1.5 倍の 10-2 | 1.4 倍の 10-1 |
| シグナル伝達 | ||||
| MAPK シグナル伝達経路 | 35 | 0.2 | 10-4 x 2.4 | 10-3 x 9.9 |
| カルシウム シグナル伝達経路 | 22 | 0.1 | 1.2 10-2倍 | 1.4 倍の 10-1 |
| ケモカイン シグナル伝達経路 | 21 | 0.1 | 1.2 10-2倍 | 1.3 10-1 |
| がん | ||||
| 癌の経路 | 42 | 0.3 | 10-5 x 4.1 | 3.4 x 10-3 |
| 神経膠腫 | 15 | 0.1 | 10-5 x 4.4 | 2.4 x 10-3 |
| 非小細胞肺癌 | 10 | 0.1 | 7.9 x 10-3 | 10-1 x 1.1 |
| 大腸がん | 13 | 0.1 | 8.4 x 10-3 | 10-1 x 1.1 |
| 慢性骨髄性白血病 | 12 | 0.1 | 1.2 10-2倍 | 1.3 10-1 |
Dmr ラット メチル シーケンスから特定の表 4: KEGG パスウェイ解析
ディスカッション
本研究では、設計し、ラットのゲノムのメチル Seq プラットフォームを実装します。ストレスのモデルラットにその有用性を発揮し、実験的・解析的パイプラインが比較の 2 つのグループ間の特異的メチル化領域を提供できる私たちを発揮します。
プラットフォームの実装を成功させるためには、いくつかの重要なステップは、観察する必要があります。最初、初期 DNA の質と量は最終的なメチル Seq ライブラリの量と質に大きな影響を与える。私たちの DNA の測定が現在の二本鎖 DNA の量を反映するために、蛍光光度計、分光光度計よりもむしろを使いました。バイオアナライザーは分子サイズとアダプター結紮後せん断に続く DNA の量を測定していました。「シフト」これらのステップ間の分子のサイズの検証は、以降の手順でアダプターを介した PCR を受ける各 DNA 断片の両端にアダプターの存在を確認することが重要です。DNA アダプター結紮ステップの終わりに残りの数量は、少なくとも 100 からも重要であるライブラリ製品の ng が十分な量がターゲットの濃縮と重亜硫酸塩の変換の手順後に利用できるようにするこの手順で必要です。最終的な高感度測定は、ライブラリを正しく次世代シーケンサーの後続のクラスタ リングのため薄くことができるように構築されたメチル Seq ライブラリで実行されました。最後に、重亜硫酸塩パイロシークエンスは解析パイプラインの精度を評価するために非常に量的な独立したメソッドとして採用されました。元のサンプルおよび付加的な動物を使用して、レプリケーションを使用して最終的な検証は、DNA メチル化の実験が生物学的に重要な変更を検出できるようにする重要なステップです。
我々 もが発生した場合、プロトコルからの逸脱または問題が発生したかどうかいくつかのガイドラインが含まれます。まず、最後修理、アダプター結紮または磁気ビーズ精製工程中にあまりにも多くの DNA を失うことが可能です。また、量の DNA を開始小さなことができる (< 200 ng) 限られた組織/DNA 可用性または蛍光活性化細胞選別などさまざまな濃縮方法の実施のため。DNA の過度の損失を補償することができるまたは低 DNA ライブラリ構築プロトコル全体量を開始 2 つのライブラリの増幅段階でサイクル数を増やすことがあります。ただし、いいえ追加より 2-3 サイクルが推奨、過剰なテンプレートの増幅が配列されている重複読み取りの数の増加につながる可能性があります。これらの重複は、パーセントのメチル化計算でバイアスを防ぐために、アライメント」ステップ時に除外されます。第二に、以上 30 bps によって DNA の平均サイズが向上しない場合、は、試薬の新しい、T4 DNA ポリメラーゼ、Klenow、または T4 リガーゼが古い可能性がありますを確認します。市販の交換用試薬を使用することができます。
また、予測の Dmr がパイロシークエンス、DNA メチル化の違いが存在しないか、分析で予測されたものよりも大幅に少ないが、検証が可能です。候補地域の貧しい人々 の検証もときパイロシークエンス結果がメチル化を確認しないなど、多くのゲノム解析の一般的な問題であるか、効果量は分析によって予測よりもはるかに小さい。BSmooth は、複数の Cpg のウィンドウを横切る 1 つ分析パッケージ「平滑化」のメチル化レベルです。現在の実験では、BSmooth には DMR のメチル化レベルの重亜硫酸塩ピロシーケンスによる検証が関与しています。しかし、おそらくこと BSmooth によって予測されるメチル化レベルとピロシーケンスによるこれらの不一致があります。不一致はすべて持ち、連続の Cpg DNA のメチル化の 50% 以上によって異なる場合がありますまたは Cpg メチル化値を持つために除外されたを含む内の Cpg のメチル化平均値を推定するスムージング機能から生じるサブしきい値には、深さがお読みください。MethylKit24などの R パッケージは、Cpg または単一 Cpg のメチル化レベルが相関関係が強くピロシーケンスによる検証の小さい窓を識別するために使用できます。異なるパッケージを実装する、予測地域や差分のメチル化 Cpg をピロシーケンスによる試験データの信頼性が保証されます。また、元メチル Seq ライブラリを resequenced し、読み取りの深さを増加する読み取りファイルに追加できます。メチル化レベルの定量は読み取りの数によって半定量的決定 [(# of CpGs)/(# TpGs + Cpg)]、与えられた CpG がその % メチル化値の精度を向上させるための読み取りの深さを増やします。本研究では我々 だけ Cpg メチル化値が少なくとも 10 の読み取りによって定められた、各 CpG の 19 x の全体的な読み取り範囲を達成すると見なされます。
ラット メチル Seq プラットフォームは、制限なしです。全ゲノムの重亜硫酸塩の配列よりも費用対効果は、他の方法よりもかなり高価です。それにもかかわらず、費用のほとんどは、キャプチャ システムで、シーケンサーの車線を購入するためだった。クロス組織比較少ない大きい (25-70%) の違い12 DNA メチル化のために必要とする、必要に応じての読み取りの深さに応じてレーンあたりより多くのサンプルを多重化し、大容量プラットフォームを使用してコストを削減することができます。また、サンプル準備は他の方法よりも時間がかかるです。一方、次世代シーケンスを組み込む他のプルダウンのアプローチと同様に、作業負荷に追加された重亜硫酸塩の変換および精製手順を追加します。全体的にみて、メチル Seq プラットフォームは、全ゲノム シーケンスにコスト効果の高い代替手段で、マイクロ アレイ ベースのプラットフォームによって試金されるそれらよりもかなり多くである以上 230 万 Cpg で塩基対の解像度を提供します。までに、市販の人間そしてマウス メチル Seq プラットフォームは、サル脳25,26アルコール依存的変化、マウス脳9、および血液脳神経発達学的遺伝子を文書化に使用されています。グルココルチコイド10のターゲット。更に、制限酵素認識シーケンスに関係なく特定の地域をターゲットにする能力はそれクロス種の比較のための理想的なプラットフォームを作る。本研究では、ゲノム methylomic ツールの恩恵なしを多くの薬理、代謝、および行動実験を行い、ラットに対するメチル Seq プラットフォームを設計しました。我々 のデータは、それがストレスのモデルラットに Dmr を検出する使用ことができますと全体的な血中 CORT など他の生理学的パラメーターの相関を見る。
メチル Seq プラットフォームは、規制地域を文書化する十分な実験的証拠がない可能性がありますシーケンスのゲノムを持つ動物のエピジェネティックな実験に最適です。このような領域が利用できる場合、追加の領域をカスタム設計および現在のバージョンに接続されている可能性があります。さらに、プラットフォームは、豊かなターゲットに制限酵素認識によって拘束されていないので、比較ゲノミクスに最適です。たとえば、特定の制限のサイトを抱いてそれかどうかに関係なく興味の任意の遺伝子のプロモーター領域をキャプチャできます。同様に、マウスまたは関心のゲノム内に保存されます人間で識別されたそれらのような規制領域をキャプチャできます。
開示事項
原稿は、アジレント ・ テクノロジーからコンテストの賞金の一部です。
謝辞
この研究は、NIH グラント MH101392 (RSL) と次の賞を受賞し基礎からのサポートによって賄われていた: NARSAD 若手研究者賞、マーガレット アン価格調査基金、チャールズ t. バウアー財団は、パン屋でジェームズ ・華気分障害学者基金財団法人、およびプロジェクトの一致財団 (RSL)。
資料
| Name | Company | Catalog Number | Comments |
| Radioimmuno assay (RIA) | MP Biomedicals | 7120126 | Corticosterone, 125I labeled |
| Master Pure DNA Purification Kit | Epicentre/Illumina | MC85200 | |
| Thermal-LOK 2-Position Dry Heat Bath | USA Scientific | 2510-1102 | Used with 1.5 mL tubes |
| Vortex Genie 2 | Fisher | 12-812 | Vortex Mixer |
| Ethyl alcohol, Pure | Sigma-Aldrich | E7023 | 100% Ethanol, molecular grade |
| Centrifuge 5424 R | Eppendorf | - | Must be capable of 20,000 x g |
| Qubit 2.0 | ThermoFisher Scientific | Q32866 | Fluorometer |
| Qubit dsDNA BR Assay Kit | ThermoFisher Scientific | Q32850 | |
| Qubit dsDNA HS Assay Kit | ThermoFisher Scientific | Q32851 | High sensitivity DNA detection reagents |
| Qubit Assay Tubes | ThermoFisher Scientific | Q32856 | |
| SureSelectXT Rat Methyl-Seq Reagent Kit | Agilent Technologies | G9651A | Reagents for preparing the Methyl-Seq library |
| SureSelect Rat Methyl-Seq Capture Library | Agilent Technologies | 931143 | RNA baits for enrichment of rat targets |
| IDTE, pH 8.0 | IDT DNA | 11-05-01-09 | 10 mM TE, 0.1 mM EDTA |
| DNA LoBind Tube 1.5 mL | Eppendorf | 22431021 | |
| Covaris E-series or S-series | Covaris | - | Isothermal sonicator |
| microTUBE AFA Fiber Pre-Slit Snap-Cap 6x16mm (25) | Covaris | 520045 | |
| Water, Ultra Pure (Molecular Biology Grade) | Quality Biological | 351-029-721 | |
| Veriti 96 Well-Thermal Cycler | Applied Biosystems | 4375786 | |
| AMPure XP Beads | Beckman Coulter | A63880 | DNA-Binding magnetic beads |
| 96S Super Magnet | ALPAQUA | A001322 | Magnetic plate for purification steps |
| 2200 TapeStation | Agilent Technologies | G2965AA | Electrophresis-based bioanalyzer |
| D1000 ScreenTape | Agilent Technologies | 5067-5582 | |
| D1000 ScreenTape High Sensitivity | Agilent Technologies | 5067-5584 | |
| D1000 Reagents | Agilent Technologies | 5067-5583 | |
| D1000 Reagents High Sensitivity | Agilent Technologies | 5067-5585 | |
| DNA110 SpeedVac | ThermoFisher Scientific | - | Vacuum Concentrator |
| Dynabeads MyOne Streptavidin T1 magnetic beads | Invitrogen | 65601 | Streptavidin magnetic beads |
| Labquake Tube Rotator | ThermoFisher Scientific | 415110Q | Nutator Mixer is also acceptable |
| EZ DNA Methylation-Gold Kit | Zymo Research | D5006 | Bisulfite conversion kit. Contains Binding, Wash, Desulphonation, and Elution buffers |
| Illumina Hi-Seq 2500 | Illumina | - | Next-generation sequencing machine |
| PCR and Pyrosequencing Primers | IDT DNA | Variable | |
| Taq DNA Polymerase with ThermoPol Buffer - 2,000 units | New England BioLabs | M0267L | |
| Deoxynucleotide (dNTP) Solution Set | New England BioLabs | N0446S | |
| Pyromark MD96 | QIAGEN | - | Pyrosequencing machine |
| Ethyl Alcohol 200 Proof | Pharmco-Aaper | 111000200 | 70% Ethanol solution |
| Sodium Hydroxide Pellets | Sigma-Aldrich | 221465 | 0.2 M NaOH denature buffer solution |
| Tris (Base) from J.T. Baker | Fisher Scientific | 02-004-508 | 10 mM Tris Acetate Buffer wash buffer solution |
| PyroMark Gold Q96 Reagents (50x96) | QIAGEN | 972807 | Reagents required for pyrosequencing |
| PyroMark Annealing Buffer | QIAGEN | 979009 | |
| PyroMark Binding Buffer (200 mL) | QIAGEN | 979006 | |
| Streptavidin Sepharose High Performance Beads | GE Healthcare | 17-5113-01 | Streptavidin-coated sepharose beads |
| PyroMark Q96 HS Plate | QIAGEN | 979101 | Pyrosequencing assay plate |
| Eppendorf Thermomixer R | Fisher Scientific | 05-400-205 | Plate mixer. 96-well block sold separately (cat. No 05-400-207) |
| SureDesign Website | Agilent Technologies | - | Target capture design software (https://earray.chem.agilent.com/suredesign/) |
| UCSC Genome Browser | University of California Santa Cruz | - | rat Nov 2004 rn4 assembly |
| Agilent Methyl-Seq Protocol | Agilent Technologies | - | https://www.agilent.com/cs/library/usermanuals/public/G7530-90002.pdf |
参考文献
- Barski, A., et al. High-resolution profiling of histone methylations in the human genome. Cell. 129 (4), 823-837 (2007).
- Meissner, A., et al. Genome-scale DNA methylation maps of pluripotent and differentiated cells. Nature. 454 (7205), 766-770 (2008).
- Bibikova, M., et al. High density DNA methylation array with single CpG site resolution. Genomics. 98 (4), 288-295 (2011).
- Naumov, V. A., et al. Genome-scale analysis of DNA methylation in colorectal cancer using Infinium HumanMethylation450 BeadChips. Epigenetics. 8 (9), 921-934 (2013).
- Wockner, L. F., et al. Genome-wide DNA methylation analysis of human brain tissue from schizophrenia patients. Translational Psychiatry. 4, e339 (2014).
- Meissner, A., et al. Reduced representation bisulfite sequencing for comparative high-resolution DNA methylation analysis. Nucleic Acids Research. 33 (18), 5868-5877 (2005).
- Smith, Z. D., Gu, H., Bock, C., Gnirke, A., Meissner, A. High-throughput bisulfite sequencing in mammalian genomes. Methods. 48 (3), 226-232 (2009).
- Slieker, R. C., et al. Identification and systematic annotation of tissue-specific differentially methylated regions using the Illumina 450k array. Epigenetics Chromatin. 6 (1), 26 (2013).
- Hing, B., et al. Adaptation of the targeted capture Methyl-Seq platform for the mouse genome identifies novel tissue-specific DNA methylation patterns of genes involved in neurodevelopment. Epigenetics. 10 (7), 581-596 (2015).
- Seifuddin, F., et al. Genome-wide Methyl-Seq analysis of blood-brain targets of glucocorticoid exposure. Epigenetics. 12 (8), 637-652 (2017).
- Irizarry, R. A., et al. The human colon cancer methylome shows similar hypo- and hypermethylation at conserved tissue-specific CpG island shores. Nature Genetics. 41 (2), 178-186 (2009).
- Lee, R. S., et al. Adaptation of the CHARM DNA methylation platform for the rat genome reveals novel brain region-specific differences. Epigenetics. 6 (11), 1378-1390 (2011).
- Jankord, R., et al. Stress vulnerability during adolescent development in rats. Endocrinology. 152 (2), 629-638 (2011).
- Pellow, S., Chopin, P., File, S. E., Briley, M. Validation of open:closed arm entries in an elevated plus-maze as a measure of anxiety in the rat. Journal of Neuroscience Methods. 14 (3), 149-167 (1985).
- Krueger, F., Andrews, S. R. Bismark: a flexible aligner and methylation caller for Bisulfite-Seq applications. Bioinformatics. 27 (11), 1571-1572 (2011).
- Langmead, B., Salzberg, S. L. Fast gapped-read alignment with Bowtie 2. Nature Methods. 9 (4), 357-359 (2012).
- Langmead, B., Trapnell, C., Pop, M., Salzberg, S. L. Ultrafast and memory-efficient alignment of short DNA sequences to the human genome. Genome Biology. 10 (3), R25 (2009).
- Hansen, K. D., Langmead, B., Irizarry, R. A. BSmooth: from whole genome bisulfite sequencing reads to differentially methylated regions. Genome Biology. 13 (10), R83 (2012).
- Lee, R. S., et al. Chronic corticosterone exposure increases expression and decreases deoxyribonucleic acid methylation of Fkbp5 in mice. Endocrinology. 151 (9), 4332-4343 (2010).
- Lee, R. S., et al. A measure of glucocorticoid load provided by DNA methylation of Fkbp5 in mice. Psychopharmacology. , (2011).
- Bose, M., Olivan, B., Laferrere, B. Stress and obesity: the role of the hypothalamic-pituitary-adrenal axis in metabolic disease. Current Opinion in Endocrinology, Diabetes, and Obesity. 16 (5), 340-346 (2009).
- Brydon, L., Magid, K., Steptoe, A. Platelets, coronary heart disease, and stress. Brain, Behavior, and Immunity. 20 (2), 113-119 (2006).
- McKlveen, J. M., et al. Chronic Stress Increases Prefrontal Inhibition: A Mechanism for Stress-Induced Prefrontal Dysfunction. Biological Psychiatry. 80 (10), 754-764 (2016).
- Akalin, A., et al. methylKit: a comprehensive R package for the analysis of genome-wide DNA methylation profiles. Genome Biology. 13 (10), R87 (2012).
- Cervera-Juanes, R., Wilhelm, L. J., Park, B., Grant, K. A., Ferguson, B. Alcohol-dose-dependent DNA methylation and expression in the nucleus accumbens identifies coordinated regulation of synaptic genes. Translational Psychiatry. 7 (1), e994 (2017).
- Cervera-Juanes, R., Wilhelm, L. J., Park, B., Grant, K. A., Ferguson, B. Genome-wide analysis of the nucleus accumbens identifies DNA methylation signals differentiating low/binge from heavy alcohol drinking. Alcohol. 60, 103-113 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved