Method Article
CRISPR/Cas9リボヌクレオタンパク質媒介精密遺伝子編集チューブエレクトロポレーション
要約
ここに提示される、チューブエレクトロポレーションを用いて哺乳動物細胞における効率的なCRISPR/Cas9リボヌクレオタンパク質媒介遺伝子編集のためのプロトコルである。
要約
CRISPR関連タンパク質9(Cas9)に代表される遺伝子編集ヌクレアーゼは、生物医学研究の主流ツールになりつつあります。トランスフェクションによる標的細胞へのCRISPR/Cas9要素の正常な送達は、効率的な遺伝子編集の前提条件です。このプロトコルは、異なるタイプの哺乳動物細胞に対する一本鎖オリゴデオキシヌクレオチド(ssODN)ドナーテンプレートと共に、CRISPR/Cas9リボヌクレオプロテイン(RNP)のチューブエレクトロポレーション(TE)機械媒介送達が堅牢につながることを示しています。正確な遺伝子編集イベント。まず、TEを適用して、ウサギ線維芽細胞細胞におけるインターロイキン2受容体サブユニットγ(IL2RG)遺伝子およびセピアプテリン還元酵素(SPR)遺伝子における疾患を引き起こす変異を誘導するためにCRISPR/Cas9 RNPおよびsSODNを誘導した。3.57%-20%の正確な突然変異率は、細菌TAクローニングシーケンシングによって決定されたとおりに達成された。同じ戦略は、表皮成長因子受容体(EGFR)、ミオシン結合タンパク質C、心臓(Mybpc3)、ヘモグロビンサブユニットベータ(HBB)を含むいくつかの臨床的に関連する遺伝子上のヒトiPSCで使用された。一貫して、高精度の突然変異率を達成した(11.65%-37.92%)深いシーケンス(DeepSeq)によって決定されます。本研究は、CRISPR/Cas9 RNPのチューブエレクトロポレーションが哺乳動物細胞における遺伝子編集のための効率的なトランスフェクションプロトコルを表していることを示している。
概要
CRISPR/Cas9は遺伝子編集のために最も一般的に使用されるプログラム可能なヌクレアーゼである。これは、ゲノム内の標的配列と隣接するプロトスペーサー隣接モチーフ(PAM)配列の単一ガイドRNA(sgRNA)媒介認識を介して動作します。Cas9ヌクレアーゼは、PAM配列1の上流に3つのヌクレオチドに位置する二本鎖DNA破断(DSB)を生成する。DSB は、エラーが発生しやすい非相同エンド 結合 (NHEJ) または相同指向修復 (HDR) 経路を介して修復されます。HDR経路を介して正確な遺伝子編集を達成するために、ドナーテンプレートは、多くの場合、プラスミドDNA(pDNA)または一本鎖オリゴデオキシヌクレオチド(ssODN)の形式で提供される。
CRISPR/Cas9およびsgRNAは、Cas9タンパク質およびgRNA2、3のリボヌクレオタンパク質(RNP)複合体の3つのフォーマットで細胞に送達することができる。Cas9 mRNA および sgRNA4,5;または必要なプロモーターを含むプラスミドDNA(pDNA)、駆動sgRNA、およびCas9コード領域6、7、8を含む。多くのグループは、CRISPR/Cas9がRNPとして送達されるとき、遺伝子編集効率が多くの場合、核酸9と比較してRNPのはるかに小さいサイズに起因するpDNAまたはmRNAフォーマットで達成されるものよりも優れていることを実証しています。さらに、新規チューブエレクトロポレーション(TE)機械が複数の細胞タイプ9における遺伝子編集用途において特に有効であることが以前に示されている。
本研究で提示されるのは、いくつかの臨床的に関連する遺伝子座で異なる種の哺乳動物細胞にCRISPR/Cas9 RNPを送達するためのTEを利用するステップバイステッププロトコルである。この新しいTEトランスフェクション技術と高いHDRレート現象は、生物医学研究における幅広い応用を見出すかもしれない。
プロトコル
すべての動物の維持、ケア、および使用手順は、ミシガン大学の機関動物ケアおよび使用委員会(IACUC)によって審査され、承認されました。
1. 細胞の調製
-
アメリカン・タイプ・カルチャー・コレクション(ATCC)からヒトiPSC(ACS-1030)を取得する。サプライヤーの指示に従い、細胞培養インキュベーター(37°Cで5%CO2)でフィーダーフリー細胞培養培地(材料表参照)を用いた人工細胞外マトリックス上の培養iPScsを行う。
- トランスフェクションの2時間前に、iPSCを10μM Rho関連、タンパク質キナーゼ(ROCK)阻害剤Y27632を含むコイルコイルで処理する(解離されたヒトhiPSCのアポトーシスを低減し、hiPSCの生存率とクローニング効率を向上させる使用)多能性に影響を与える)。
- トランスフェクションの場合は、iPSCを細胞剥離液(材料表参照)で37°Cの単一細胞に5分間分離し、細胞数をカウントします。
-
ウサギの耳皮膚組織生検の一次培養を用いてウサギ線維芽細胞培養を確立し、前述の10.
- 0.5 cm x 0.5 cmの耳の皮の生検はウサギの耳の先端から得られる。耳のティッシュから髪を剃ります。
- 5%ペニシリン連鎖マイシンでダルベッコのリン酸緩衝生理食べ物(DPBS)で2xをすすいで。耳組織を新しい6cm組織培養皿に移し、ティッシュを小片(約1.0mm x 1.0mm)に切ります。組織が乾燥するのを防ぐために、胎児の牛血清を数滴追加します。
- 細断された組織を10cmの組織培養皿に広げ、次に培養培地の10mLを加える。ウサギの線維芽細胞は、10%の胎児ウシ血清でダルベッコの修飾イーグルの培地(DMEM)で培養される。組織培養皿を細胞培養器(37°Cで5%CO2)に入れる。
- めっき後3~5日後に、トリプシン-EDTAを使用して細胞数を2分間37°Cで消化します。
2. gRNAとドナーオリゴスの設計と合成
- 各遺伝子について、オンラインツールを使用して標的遺伝子の配列に基づいて設計ガイドRNAを導く(例えば、)。
- 目的のDNA配列に貼り付け。
- ゲノムとプロトスペーサー隣接モチーフ(PAM)を選択します。入力DNA配列の可能なガイドシーケンスは、出力ページに表示されます。より高い予測効率と低いオフターゲット電位を持つgRNAを選択することをお勧めします。
- gRNAを転写するための商用ベンダーによるDNAの合成メーカーの指示に従ってgRNA合成キットを用いてgRNAのインビトロ転写を行う。
- gRNA合成キットに含まれるRNA精製マイクロカラムを用いてgRNAを精製する。濃度を測定し、次に-80 °CにgRNAを保存します。
- 各変異部位の ssODN ドナー テンプレートを設計します。SSODN は、IDT などの商用ベンダーによって合成できます。一般に、各ssODNは長さが120〜160ヌクレオチド(nt)であり、左相同アームでは60〜80nt、右相同性アームでは60〜80ntからなる。編集されたDNAの再切断を防ぐために、PAMでのサイレント変異を可能な限りssODNに導入する必要があります。CRISPR切断部位は、可能な限り意図されたゲノム変化に近い位置に配置する必要があります。
3. Cas9 RNPおよびsSODNのチューブエレクトロポレーション
- セクション1で説明するようにセルを調調べる。
- エレクトロポレーションバッファーの20 μLで2-3 x 105セルを再中断します。ピペットを上下に注意深く、シングルセルサスペンションを作り出します。
- Cas9 RNPトランスフェクションの場合、室温(RT)で0.67 μgのGRNAを10〜15分間、Cas9-NLSタンパク質のプレミックス2 μgを使用します。次に、形成されたRNP複合体を2 μgのssODNと細胞と穏やかに混合する。
- チューブエレクトロポレーションキットによって提供されるユニバーサルフィットピペットチップを使用して、細胞混合物を20 μLエレクトロポレーションチューブに移します。より良いエレクトロポレーションを達成するために、転送中に気泡の形成を避けるようにしてください。
- エレクトロポレーションチューブをエレクトロポレータのスロットに入れ、「Go」を押して仕上げます。各セルタイプのメーカーの推奨パラメータに従ってください。例えば、ヒトiPSCおよびウサギ線維芽細胞の場合、電圧セットは420V、パルス時間は30msです。
- エレクトロポレーション後、細胞培養部に記載の予温められたY-27632含有培養培地の1mLにヒトiPS細胞を移移す。ウサギの線維芽細胞の場合は、10%の胎児ウシ血清でDMEMに移します。
- 再懸濁した細胞を12ウェル細胞培養プレートの1ウェルにプレートする。
- 毎日培養媒体を変更します。Y-27632は、ヒトiPSC培養培地24時間ポストエレクトロポレーションから除去される。
4. 遺伝子編集事象の解析
- エレクトロポレーション後72hの細胞を収穫する。ウサギ線維芽細胞またはヒトiPSC用細胞剥離溶液に対するトリプシン-EDTAを用いて培養プレートから細胞を消化する。遠心分離機の後、350 mLのリシスバッファー(1MトリスHCl、5M NaCl、0.5 M EDTA、pH 8.0、10%SDS、20 μLの20μLを添加し、1mLのリシスバッファー当たり20μLを添加し、次いで55°Cでインキュベートする)
- 標準的な手順を使用してフェノールクロロホルムでゲノムDNAを抽出します。
- 高忠実度のDNAポリメラーゼを用いて標的領域を含む100~200bp DNA断片を増幅し、ゲル抽出キットを使用するか、PCR SVミニキットを使用して直接PCR製品からゲルからDNA断片を精製します。
- 細菌コロニーシーケンシングによる遺伝子編集効率を決定するには、精製されたPCR産物をTOPO TAクローニングキットを用いてpCR4-TOPOベクターにライガゼします。細菌クローンをランダムにピックアップし、TOPO TAクローニングキットが提供するユニバーサルシーケンシングプライマーを使用してインサートを配列します。
- 深いシーケンシングによって遺伝子編集効率を決定するには、DNAシーケンシングコア内のCRISPRアンプリコンシーケンシングのステップ4.3から精製されたPCR製品(〜100〜200 bp)を送ります。
結果
ウサギ線維芽細胞に対するCas9 RNPおよびsSODNのTE
哺乳動物細胞へのCas9 RNPのTE媒介送達の全体的なプロセスを図1に示す。まず、IL2RG遺伝子ではC231YおよびQ235X変異が生成され、R150G変異はウサギ線維芽細胞のSPR遺伝子で産生された。IL2RGおよびSPR遺伝子における機能喪失変異は、それぞれ一次免疫不全11および運動および認知障害12を引き起こすことが知られている。
特定のsgRNA設計を図2Aに示す。対象領域を増幅するために使用されるプライマーは、表 3に示されています。ssODN のシーケンスを表1に示します。遺伝子編集率は、細菌TAクローニング(図2B)によって決定した。IL2RG C231遺伝子座では、配列決定された28個のクローンのうち、1つ(3.57%)正確なC231Y突然変異を運んだ, 4 (14.28%)挿入または欠失(インデル)突然変異を運び、残りの23(82%)野生型でした。IL2RG Q235遺伝子座では、配列された27個のクローンのうち、2つ(7.41%)正確なQ235X突然変異を運んだ、3つの運ばれたインデル突然変異(11.11%)残りは野生型でした。SPG R150遺伝子座では、配列された20個のクローンのうち、5個(25%)が並配列された正確なR150G突然変異を運んだ, 10 (50%)インデル突然変異を運び、残りは野生型であった。
人間のiPSCに対するCas9 RNPとsSODNのTE
TE は、Cas9 RNP および sSODN をヒト iPSC に提供し、EGFR、Mybpc3、および HBB 遺伝子の臨床的に関連する遺伝子座を標的にするために使用されました。EGFR T790近位領域における点変異は、EGFR13の活性化変異を有する非小細胞肺癌(NSCLC)患者におけるEGFRチロシンキナーゼ阻害剤に対する耐性を付与する。Mybpc3におけるエキソン16におけるフレームシフト変異は、肥大型心筋症14に関与している。HBB遺伝子におけるE6V点変異は鎌状赤血球病15につながる。
特定のsgRNA設計を図3Aに示す。対象領域を増幅するために使用されるプライマーは、表 3に示されています。ssODN のシーケンスを表1に示します。遺伝子編集率はDeepSeq(図3B)によって決定された。EGFR遺伝子座では、15.68%の対減が正確な点突然変異(6,315読み取り)、22.75%がインデル突然変異(9,162回)、残りの61.57%が野生型(24,797読み取り)であった。Mybpc3遺伝子座では、37.92%が正確な4bp TGAA欠失(11,654読み取り)、2.24%がインデル変異(410回読み取り)、残りの59.84%が野生型(18,692読み取り)であった。HBB遺伝子座では、11.65%が正確なE6V突然変異(6,565読み取り)、23.35%がインデル突然変異(13,163読み取り)、残りの65%が野生型(36,644読み取り)であった。

図1:Cas9 RNPのチューブエレクトロポレーションのフローチャート。

図 2:ウサギ線維芽細胞の遺伝子編集。(A) ターゲットシーケンスの図。ボックスは、ターゲット遺伝子座を示します。下線付きの文字は gRNA シーケンスに対応します。赤い色の文字は PAM シーケンスを示します。(B) 遺伝子編集イベントのTAクローニング結果。ボックスは、正確に変異した遺伝子座を示します。示すインデル配列は、1つの対立遺伝子タイプのみを代表する。その他のインデルシーケンスは表示されません。この図のより大きなバージョンを表示するには、ここをクリックしてください。
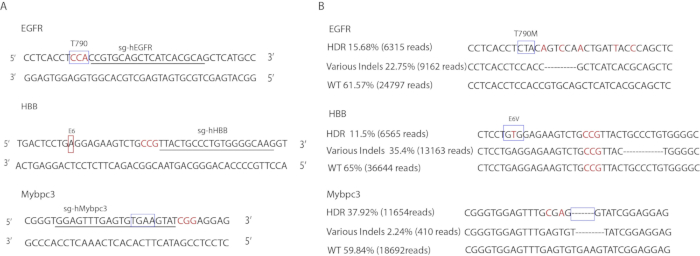
図 3: ヒトiPSCの遺伝子編集(A) ターゲットシーケンスの図。ボックスは、ターゲット遺伝子座を示します。下線付きの文字は gRNA シーケンスに対応します。赤い色の文字は PAM シーケンスを示します。(B) 遺伝子編集事象のディープセック結果ボックスは、正確に変異した遺伝子座を示します。赤い色の文字は、ドナーテンプレートで導入されたサイレント変異を示します。示すインデル配列は、1つの対立遺伝子タイプのみを代表する。その他のインデルシーケンスは表示されません。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| 軌跡 |
オリゴ配列 | |||||||
| (標的変異) | ||||||||
| ウサギ IL2RG (C231Y) | アグググガットグマクガクガアクタクタカグトッチャグトッチャグトググツッタッタッタットガットガットガットガットガットガットガットガットガットガットガットガットガットガットガットガットガット ガッガグガッカッカアアクトカアクトカアググターアアググトグスグック | |||||||
| ウサギ IL2RG (Q235X) | アグググガットグマクガクガアクタクタカグトッチャグトグググググトッタッタッタットGTGTGGGTタグタグタグタグ ガッガグガッカッカアアクトカアクトカアググターアアググトグスグック | |||||||
| ウサギ SPR | gacctccatctctctctctgcgcgcgcgcgcgcctgcgcctgcctgcctgcctggcctgcttcttctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctctgtcttctctctctctctctctctctctctctctctctctctctctctctctctctctctctggggggggctctctggctgctgctgctgctggggggg cgctgtac | |||||||
| (R150G) | ||||||||
| 人間のEGFR | ACGTGGGGCCAGCGGACCCGCGCGCGCGCGCGCGCGCGCGCCTCTCTCTACCTACACACACTAACTGATTACCC アグットカッCTTTCGGCGCGCCTCTCTCTCTGGCTGGGGGGGGGGGGGGGGAAアガアガアガアガアカアガットッチャッカッカグタック | |||||||
| (T790に近い点変異) | ||||||||
| ヒューマンマイブプ3 | GCCCCCTGTGTTCATGCGCGCGCGGAGGAGGAGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGGTTTGCGCGGG GGAGGGGGCGCAGTCAATGGTTTAGAAGAGGGGGGGGGGGGGGGGGGGGGGGGGGGGCAT | |||||||
| (4-bp削除) | ||||||||
| ヒューマン HBB | TCTGACACAACTGTGTTCTTCTTCTTCTTCACACACACACACACACACACACACACACACAGCGCGCCTCTGGGGガガアグトグツグタクタクタクグCC CTGTGGGGCAAGGTGAAGGGGAGGGGGGGGGGGGGGGGCGCAG | |||||||
| (E6V) | ||||||||
表 1: ssODN のシーケンス。
| ステップ | 問題 | 考えられる理由 | ソリューション |
| 2.1年 | 低いインデル率 | 貧弱なガイドRNA設計、ガイドRNAストック>6ヶ月、低ガイドRNA濃度 | ガイドRNAを再設計し、新しいガイドRNAを生成/注文します。 |
| 2.3年 | 低いPGE効率 | 貧しいドナーDNA設計、低効率ガイドRNA、ドナーDNAの誤量または質の悪いDNA | 相同性アームの長さを増やし、PAM変異を導入し、ドナーDNAにサイレント変異を導入し、より効率的なガイドRNAを使用し、ガイドRNAに対するCas9タンパク質の比率を最適化する。 |
| 3.4年 | トランスフェクションに失敗しました | 細胞バッファー混合物をエレクトロポレーションチューブに移す際に形成された気泡、電圧/持続時間の設定が正しくない | 気泡の形成を避け、電圧/持続時間の設定を調整してください。 |
| 3.6年 | エレクトロポレーション後の低細胞生存率 | 単一ヒトipscの低い生存率 | エレクトロポレーション後にROCK阻害剤を添加し、細胞数を増やす。 |
| 4.1年 | 失敗した PCR | 高い GC コンテンツ、または繰り返しシーケンス | PCR条件を最適化し、PCRシステムにDMSOを追加します。 |
表 2: 頻繁な問題のトラブルシューティング ガイド。
| プライマー名 | シーケンス | メモ |
| Rb-IL2RG-F | カガカグマクガッググツック | ウサギIL2RG DNA断片を増幅する場合 |
| Rb-IL2RG-R | TGCCAGAGAカカカアガガックガック | |
| Rb-SPR-F | GTACTTGGAGGGAGAGG | ウサギSPR DNA断片を増幅する場合 |
| RB-SPR-R | CTCAGCCCTGACACTGGGGG | |
| H-EGFR-F | ツガッグッカグググスガアック | ヒトEGFR DNA断片を増幅する場合 |
| H-EGFR-R | アカグトガッカグタクトグッグ | |
| H-Mybpc3-F | ATGCCCCGTGCTTTGAAC | ヒトMybpc3 DNA断片を増幅するための |
| H-Mybpc3-R | TCAGGGGAGCCAACCCTCAT | |
| H-HBB-F | タアックトガタッカッカック | ヒトHBB DNA断片を増幅する |
| H-HBB-R | キャットトットクトガカアクト |
表 3: 手順 4.3 で使用されるプライマー。
ディスカッション
このチューブエレクトロポレーション法は、CRISPR/Cas9 RNPおよびssODNをウサギおよびヒト細胞に送り出すのに有効であり、堅牢な精密遺伝子編集(PGE)を導いた。TE と他の従来のエレクトロポレーション デバイスとの主な違いは、2 つの電極がチューブの上部と下部にあり、サンプルが完全に装填され、エレクトロポレーション時に密封されるチューブの使用です (図1)。対照的に、従来のキュベットでは、電極は側面にあり、サンプルはエレクトロポレーション中に完全に密閉されていません。この新しい設計は気泡の発生を減らし、その結果、電気電圧の配分を改善し、その結果、細胞死の減少および高いトランスフェクション効率9につながる気泡のサイズを圧縮する。本研究では、高いPGE率(15%-37%)ヒトiPSCにおけるEGFR、Mybpc3およびHBB遺伝子を標的に達成された。これらの結果は、ヒト幹細胞9で高いPGE率が達成された以前の報告と一致している。
疾患を引き起こす変異は、ウサギ細胞のIL2RGおよびSPR遺伝子を標的とした。近年、IL2RGノックアウトウサギは、ヒトX結合重篤な複合免疫不全(SCID-X1)16、17のモデルとして製造されている。本研究は、患者IL2RG変異(例えば、C231YおよびQ235X)がウサギ細胞で効率的に生成できることを示し、患者変異を担うSCID-X1ウサギモデルを作成する可能性を実証する。また、SPR R150G変異がウサギ細胞で効率的に作成できることも実証された。この突然変異は、小児12の運動および認知障害を引き起こす。これらのIL2RGおよびSPR変異ウサギモデルは、一度生成されると、翻訳研究のための貴重な前臨床モデルとして役立つ可能性がある。これらの単原性疾患に対する遺伝子編集ベースの治療薬を確立するためにも使用できます。
CRISPR/Cas9媒介遺伝子編集アプリケーションの懸念事項の1つは、オフターゲット編集イベントです。インデル率は、前述の9を用いた方法を用いて、本研究で用いられるsgRNAの予測トップオフターゲット部位(表S1)で分析した。合計で、7つの潜在的なトップオフターゲット遺伝子座は、表S2に記載されているプライマーを使用して、sg-rb-IL2RG-01、sg-rb-SPRの5つ、sg-hEGFRの場合は7つ、sg-hBbpc3の場合は5つ、sg-hHBB用は7つ、表S2に記載されているプライマーを用いて分析した。T7E1アッセイ(図S1)では、これらのsgRNAを用いたCRISPR/Cas9媒介遺伝子編集のオフターゲットリスクを最小限に抑えるオフターゲットインデルは明らかにされませんでした。また、チューブエレクトロポレーション方法自体がオフターゲット編集を引き起こしたり増加させたりしないことを示します。それにもかかわらず、望ましくないオフターゲット編集を減らすか、または排除するための努力が必要です。全ゲノムシーケンシングは、臨床応用に使用されることを意図した細胞に対してそのような事象を除外する必要があるかもしれない。
技術レベルでは、CRISPR/Cas9 RNPチューブエレクトロポレーションによる効率的な正確なゲノム編集を達成するための重要な要素と考えられています。まず、予測された低いターゲット電位を持つ効率的なsgRNAを選択することをお勧めします。PEGアプリケーションに使用する前に、選択したsgRNAのインデル効率を検証することが重要です。ソフトウェアが検証ステップで良好な sgRNA が失敗すると予測することはまれではありません。
第二に、高いPGEを達成するために、可能な限りssODNドナーにPAM変異を誘導することが推奨される。その根拠は、そうすることで、ドナーテンプレートの統合が妨げられた後にCRISPR/Cas9を再切断するということです。場合によっては、PGE 自体が PAM 変異を導入します。他のケースでは、PAM配列にサイレント変異を導入することができる。PAM突然変異が不可能な場合は、sgRNA配列に対応するドナーにいくつかの無声変異を含めようとすることをお勧めします。
第三に、特にTEに関連し、細胞およびRNP混合物をエレクトロポレーションチューブに移す際に気泡の形成を避ける必要がある。TEチューブの設計は既に気泡の形成を最小限に抑えますが、慎重な取り扱いはさらに減少し、気泡の形成を完全に回避する可能性があります。CRISPR/Cas9リボヌクレオプロテインのチューブエレクトロポレーションの適用で発生する可能性のある頻繁な問題に関するトラブルシューティングガイドを、正確な遺伝子編集を媒介する問題を表2に示す。
結論として、チューブエレクトロポレーションは、高いPGEレートを達成するために、哺乳動物細胞にCRISPR/Cas9 RNPおよびsSODNを送達するための有効な手段であることをここで実証されています。この新しいTEトランスフェクション技術とその堅牢な精密遺伝子編集速度は、遺伝子編集アプリケーションの開発を容易にする可能性があります。
開示事項
J.C.は、チューブエレクトロポレーターのメーカーであるセレトリックスLLCで働いています。L. M. J. , J. S. , D. Y. Y.
謝辞
この研究は、国立衛生研究所(R21OD023194からJX)によってサポートされました。本研究は、ミシガン大学医療センターの翻訳科学・治療研究センター(CAMTraST)が支援するコアサービスを活用しました。
資料
| Name | Company | Catalog Number | Comments |
| Accutase | STEMCELL Technologies | 792 | Cell detachment solution for human iPSCs, first used in Step 1.1.2. |
| Cas9 Nuclease 3NLS | IDT | 1074182 | Cas9 protein, first used in Step 3.3. |
| DMEM | Thermo Fisher | 11965092 | For cell culture, first used in Step 1.2.3. |
| DPBS | Thermo Fisher | 1708075 | For preparing cell culture, first used in Step 1.2.2. |
| EDTA | Lonza | 51201 | For making lysis buffer, first used in Step 4.1. |
| Electroporation buffer | Celetrix | 13–0104 | The electroporation buffer, first used in Step 3.2. |
| Electroporation tubes | Celetrix | 20 μL: 12–0107; 120 μL: 12–0104 | The electroporation tube, first used in Step 3.4. |
| Electroporator | Celetrix | CTX-1500A LE | The tube electroporation machine, first used in Step 3.5 |
| Fetal bovine serum | Sigma Aldrich | 12003C | For cell culture, first used in Step 1.2.2. |
| Forma CO2 Incubators | Thermo Fisher | Model 370 | For cell culture, first used in Step 1.1. |
| Gel Extraction Kit | Qiagen | 28115 | For gel purification, first used in Step 4.3. |
| Human induced pluripotent stem cells | American Type Culture Collection | ACS-1030 | Human iPSCs, first used in Step 1.1. |
| Matrigel | Corning | 354277 | Artificial extracellular matrix; for precoating cell culture plate, first used in Step 1.1. |
| mTeSR 1 medium | STEMCELL Technologies | 85850 | Feeder-free cell culture medium for human iPSCs, first used in Step 1.1. |
| PCR SV mini | GeneAll | 103-102 | For PCR product purification, first used in Step 4.3. |
| Penicillin-Streptomycin | Thermo Fisher | 15140163 | For preparing cell culture, first used in Step 1.2.2. |
| Phenol-chloroform | Thermo Fisher | 15593031 | For DNA extraction, first used in Step 4.2. |
| Precision gRNA Synthesis Kit | Invitrogen | A29377 | For the generation of full length gRNA (guide RNA), first used in Step 2.4. |
| Proteinase K Solution | Thermo Fisher | AM2548 | For DNA extraction, first used in Step 4.1. |
| Q5 high-fidelity DNA polymerase | NEB | M0491 | For PCR amplification, first used in Step 4.3. |
| Sodium dodecyl sulfate | Sigma Aldrich | L3771 | For making lysis buffer, first used in Step 4.1. |
| TA Cloning Kit | Thermo Fisher | K457502 | For TA clone sequencing, first used in Step 4.4. |
| Tissue Culture Dish (10 cm) | FALCON | 353003 | For cell culture, first used in Step 1.2.3. |
| Tissue Culture Dish (12 well) | FALCON | 353043 | For cell culture, first used in Step 3.7. |
| Tissue Culture Dish (6 cm) | FALCON | 353004 | For cell culture, first used in Step 1.2.2. |
| Tris HCl | Thermo Fisher | BP1757-500 | For making lysis buffer, first used in Step 4.1. |
| Trypsin-EDTA | Thermo Fisher | 25200056 | For cell digestion, first used in Step 1.2. 4. |
| Universal Fit Pipette Tips | Celetrix | 14-0101 | For electroporation, first used in Step 3.4. |
| Y27632 | LC Labs | Y-5301 | The apoptosis inhibotor, first used in Step 1.1.1. |
参考文献
- Cong, L., et al. Multiplex genome engineering using CRISPR/Cas systems. Science. 339 (6121), 819-823 (2013).
- Mout, R., et al. Direct Cytosolic Delivery of CRISPR/Cas9-Ribonucleoprotein for Efficient Gene Editing. ACS Nano. 11 (3), 2452-2458 (2017).
- Zuris, J. A., et al. Cationic lipid-mediated delivery of proteins enables efficient protein-based genome editing in vitro and in vivo. Nature Biotechnology. 33 (1), 73-80 (2015).
- Miller, J. B., et al. Non-Viral CRISPR/Cas Gene Editing In Vitro and In Vivo Enabled by Synthetic Nanoparticle Co-Delivery of Cas9 mRNA and sgRNA. Angew Chem Int Ed Engl. 56 (4), 1059-1063 (2017).
- Finn, J. D., et al. A Single Administration of CRISPR/Cas9 Lipid Nanoparticles Achieves Robust and Persistent In Vivo Genome Editing. Cell Reports. 22 (9), 2227-2235 (2018).
- Liang, C., et al. Tumor cell-targeted delivery of CRISPR/Cas9 by aptamer-functionalized lipopolymer for therapeutic genome editing of VEGFA in osteosarcoma. Biomaterials. 147, 68-85 (2017).
- Luo, Y. L., et al. Macrophage-Specific in Vivo Gene Editing Using Cationic Lipid-Assisted Polymeric Nanoparticles. ACS Nano. 12 (2), 994-1005 (2018).
- Wang, H. X., et al. Nonviral gene editing via CRISPR/Cas9 delivery by membrane-disruptive and endosomolytic helical polypeptide. Proceedings of the National Academy of Sciences of the United States of America. 115 (19), 4903-4908 (2018).
- Xu, X., et al. Efficient homology-directed gene editing by CRISPR/Cas9 in human stem and primary cells using tube electroporation. Scientific Reports. 8 (1), 11649 (2018).
- Du, F., et al. Beneficial effect of young oocytes for rabbit somatic cell nuclear transfer. Cloning Stem Cells. 11 (1), 131-140 (2009).
- Allenspach, E., Rawlings, D. J., Scharenberg, A. M., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Friedman, J., et al., Adam, M. P., et al. . GeneReviews(R). , (1993).
- Hidaka, N., et al. Most T790M mutations are present on the same EGFR allele as activating mutations in patients with non-small cell lung cancer. Lung Cancer. 108, 75-82 (2017).
- Ma, H., et al. Correction of a pathogenic gene mutation in human embryos. Nature. 548 (7668), 413-419 (2017).
- Vakulskas, C. A., et al. A high-fidelity Cas9 mutant delivered as a ribonucleoprotein complex enables efficient gene editing in human hematopoietic stem and progenitor cells. Nature Medicine. 24 (8), 1216-1224 (2018).
- Song, J., et al. Bacterial and Pneumocystis Infections in the Lungs of Gene-Knockout Rabbits with Severe Combined Immunodeficiency. Frontiers in Immunology. 9, 429 (2018).
- Song, J., et al. Production of immunodeficient rabbits by multiplex embryo transfer and multiplex gene targeting. Scientific Reports. 7 (1), 12202 (2017).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved