Method Article
特定および効率的なタンパク質枯渇を達成するための分解性の調整
要約
ここで、β-est AIDシステムを用いて酵母サッカロマイセスセレビシエに関心のあるタンパク質を効果的かつ具体的に枯渇させるプロトコルを提示する。
要約
植物性オーキシン結合受容体TIR1は、アウキシンの存在下で特定の補助誘導性デグロン(AID)モチーフを含むタンパク質を認識し、分解を標的とする。このシステムは、AIDモチーフでタグ付けされた標的タンパク質がauxin添加時に分解されるような多くの非植物真核生物で利用される。TIR1 式のレベルは重要です。過剰な発現は、アウキシンが存在しない場合でもAIDタグ付きタンパク質の分解を引き起こしますが、発現が低いと、枯渇が遅くなります。β-エストラジオール誘導性AIDシステムを作成し、β-エストラジオール誘導性プロモーターの制御下でTIR1を発現した。TIR1のレベルは、auxin添加の前にβ-エストラジオールでインキュベーションの時間を変更することによっても起立可能である。このプロトコルは、AIDシステムを使用して標的タンパク質を急速に枯渇させる方法について説明する。適切なβエストラジオールインキュベーション時間は、標的タンパク質の豊富さによって異なります。したがって、効率的な枯渇は、また、オーキシン独立性の枯渇を最小限に抑える最適なタイミングに依存します。
概要
温度感受性変異体などの条件付き突然変異は、必須タンパク質の研究のための強力なツールであり、寛容な条件下で細胞の成長を可能にするが、非寛容な条件下で機能の喪失を引き起こす。しかし、細胞代謝は、欠陥を誘発するために必要な成長条件の変化によって深刻に動揺し、また、オフターゲット効果を作成することができます。目的とするタンパク質が条件的に隔離されるいくつかの方法が開発され、その発現は、小分子を添加することによって2、3を制御する。 このプロトコルは、オーキシンおよび補助誘導性デグロン(AID)システムを使用して、標的タンパク質を効率的に枯渇させます。
AIDシステムは、アウキシン(このプロトコルではインドール-3-酢酸(IAA)が使用される植物に起源を有し、SCF U3ユビキチンリエーゼ複合体4のメンバーであるTIR1とのAux/IAAタンパク質の相互作用を刺激する。SCF複合体相互作用は、Aux/IAAファミリータンパク質のポリユビキチン化を引き起こし、プロテアソーム5,6による分解をもたらす。
このシステムは、酵母細胞内でオリザサティバ(osTIR)からTIR1タンパク質を発現させることにより、酵母サッカロマイセスセレビシエ7、8での使用に適応し、内因性酵母と相互作用することができる。SCF コンプレックス。目的のタンパク質は、Aux/IAAタンパク質IAA17をモチーフにタグ付けされ、分解を標的としました。IAA17の機能的切り捨ては、後にAID*8、9、10など、アラビドプシス・タリアナIAA17の43アミノ酸オーキシン感受性モチーフを含み、エピトープタグと共に開発された。検出。
このシステムは、最初に発芽酵母7、8における使用に適応し、酵母GALプロモーターからosTIR1タンパク質を発現した。発現は、ガラクトースを唯一の炭素源として成長培地にシフトする必要があり、残念ながら、細胞代謝11に広範囲の変化を伴う希釈シフトをもたらす。一方、TIR1の構成的発現は、発現レベルが高い場合はアウクシン/IAA12が存在しない場合に標的タンパク質の分解を引き起こす可能性がある一方、TIR1発現が低いと非効率な枯渇を引き起こすことが報告されている。β-est AIDという改良されたAIDシステムは、細胞代謝への影響を最小限に抑えながら、標的タンパク質に合わせて調節可能な誘導性プロモーターの制御下にある、β-est AIDという改良されたシステムが開発されました。これを達成するために、VP16ウイルス転写活性化剤がエストロゲン受容体と4本のZn指DNA結合ドメイン(DBD)に融合する人工転写因子(ATF)が構築された。βエストラジオール(エストロゲン)が存在する場合、ATFは核に入り、そのプロモーター(Z4EVpr)13,12に結合することによってosTIR転写を誘導することができる。
osTIR発現は、通常、β-エストラジオール12を添加した後、約20分で検出可能である。しかしながら、タグ付けされたタンパク質をオーキシンで効率的に枯渇させるosTIR発現の最適な持続時間は、auxin添加前の枯渇を回避しつつ、各標的タンパク質について経験的に決定する必要がある。このインキュベーション前のおおよその時間は、サッカロマイセスゲノムデータベース(SGDhttps://www.yeastgenome.org/)の豊富な値から推定することができます。図1に見られるように、豊富なタンパク質であるDcp1(2880~4189分子/細胞)は、β-エストラジオールによる40分の潜伏を必要とし、オーキシン非依存性の枯渇は認められない。はるかに少ない豊富なタンパク質、Prp2(172〜211分子/細胞)は、インキュベーション前のわずか20分後に強く枯渇する。この最初の推定時間の前または後に10〜20分(20分は推奨される最小時間である)の2つの追加の事前インキュベーション時間をテストすることをお勧めします。最適なインキュベーション時間は、ターゲットタンパク質がアウクシンを添加する前に枯渇していない時間であり、一度オーキシンを添加すると、枯渇が許容されるか、タンパク質レベルが可能な限り最小限に近づく。したがって、図1bから、プリインキュベーションの30分を有するPrp22の場合、レベルはauxin添加後10分あまり低下していない。これは、インキュベーション前の40分とIAAとの15分を比較すると、追加の枯渇がほとんどない場合、10分以上のオーキシンでインキュベーションを行ったり、30分以上のプレインキュベートを行ったりしても、特に非auxinの証拠があるので、利点はありません。40分で依存的な枯渇。プレインキュベーションの40分のDcp1(タンパク質レベルがauxin加算前に約100%である最後の点)の場合、15〜20分のオーキシンによる枯渇が許容される。細胞代謝14に対する二次的な影響を減らすために、枯渇時間をできるだけ短くしておくことをお勧めします。
本稿では、osTIR発現に対するβ-エストラジオールインキュベーションのタイミングを最適化し、オーキシンを添加する前にIAA添加時に急速な標的タンパク質枯渇を達成することにより、β-est AIDシステムを使用する方法を示す。
プロトコル
注:グラフィカルな概要については、図 2を参照してください。
1. ひずみ準備
-
ura3-strainを用いて、β-est AIDシステム(すなわち、β-エストラジオール応答転写因子(ATF)およびosTIRをコードする遺伝子およびAID*タグを標的タンパク質にタグ付けする(手順の概要については図3および表1を参照)。
- pZTRK(G418耐性マーカー)またはpZTRL(LEU2マーカー)プラスミド(酵母遺伝資源センターから入手可能)をura3-酵母株に変換するか、またはプラズミドをテンプレートとして使用してゲノム用PCR産物を製造する統合。
- PCRは、プラスミドpZTRKまたはpZTRLのいずれかから高忠実度ポリメラーゼを使用してATF(プラスミドマップ上でZ4EVATFとマークされた)およびosTIRを増幅する。ゲノム領域に相同性を持つ50~60個のベース3'エクステンションを持つプライマーを使用して、相同組換え16による直接統合を行う。2 つの成分を個別または一緒にゲノム統合する場合は、プライマーと条件の表 1を参照してください。
注:株pZ4EV-NTR1はゲノムに既に統合された成分を有する(酵母遺伝資源センターから入手可能)。 - ロングチン手順17を使用して標的タンパク質が AID* タグ付けされていることを確認します (図 3bおよび表 1を参照)。
- β エストラジオールおよび IAA が存在しないひずみの成長分析を実行して、AID* タグが成長に影響を与えるかどうかを判断し、ステップ 1.5 で使用する成長率を予測します。
2. 枯渇に対する一般的な手順
- すべてのサンプルを収集するために必要なカルチャの量を計算します。例えば、0.8のOD600での培養の10 mLは、単一のサンプルに対するタンパク質、RNAおよびDNA抽出に十分であり、6つのサンプルでは、少なくとも60mLの培養が必要である。
- 一晩培養から、OD600 0.1~0.2で十分な新しい培養を設定し、30°Cで成長させる。YPDA などの豊富な媒体を推奨しますが、他の成長条件を使用できます。
酵母エキス 10グラム ペプトン 20グラム グルコース 20グラム アデニン硫酸 40 mg H2O へ 1 L
注:オートクレーブまたはフィルター殺菌;フィルター殺菌は、サンプル採取に用いられるメタノール中のオートクレーブ沈殿によって生成されるペプチド/糖複合体として好ましい。 - サンプルを受け取る準備をします。
- メタノールの意図したサンプル量の30~50%をチューブに入れます。例えば、10mLのサンプルを採取する場合は、5mLのメタノールを15mLのファルコンチューブに入れ、チューブをしっかりと閉じます。閉じたら、チューブにラベルを付け、ドライアイスまたは-80 °Cで冷やします。
注意:ヒュームフードにメタノールを分配します。 - サンプルの長期保存用に1.5mLチューブにラベルを付け、氷の中に入れ、冷却します。
- 氷上で十分なH2 O(サンプル当たり少なくとも1mL)を冷却します。
- メタノールの意図したサンプル量の30~50%をチューブに入れます。例えば、10mLのサンプルを採取する場合は、5mLのメタノールを15mLのファルコンチューブに入れ、チューブをしっかりと閉じます。閉じたら、チューブにラベルを付け、ドライアイスまたは-80 °Cで冷やします。
- 文化の成長を予測する。サンプルを収集するためのターゲットODは約0.7〜0.8ですが、インキュベーション前工程(osTIRを誘導するβエストラジオールによるインキュベーション)は、サンプルが得られるまでに培養がほぼ適切なODに達するように、早めに開始する必要があります。収集。
注:この開始ODを推定できるように、実験で使用する条件で成長曲線を実行することをお勧めします。 - プリインキュベーションの開始の目標ODに達したら、サンプル(通常は10mL)を、冷たいメタノールを含む予め準備されたチューブに取り込む。短時間反転して混ぜてドライアイスに戻します。
注:試料は約5分後に水氷に移動することができます。 - すぐにβ-エストラジオール、1 μL/mLの培養(最終濃度10μM)を加える。サンプルの収集とβ-エストラジオールの添加までの時間を短縮するために、ピペットでβ-エストラジオールを事前に測定しておきます。激しく旋回して急速に混ぜます。
- 以前のように培養を増殖させ続ける(ステップ2.2)、最適な時間のためにβエストラジオールを用いてインキュベート(これは「インキュベーション前工程」)(最適なインキュベーション時間の決定のために図1を参照)。
- IAA(オーキシン)を追加する準備をします。ステップ2.10に必要なIAAの体積を取り上げます(すなわち、培養mL当たりIAAの0.5 μL)。これにより、ステップ 2.20 が高速になります。
- ステップ 2.5 としてサンプルを収集します。
- 直ちにIAA 0.5 μL/mLの培養量を、ステップ2.8で調製した750 μMの最終濃度に加えます。激しく旋回して急速に混ぜます。
- 実験計画に従って、ステップ 2.5 としてサンプルを収集します。単一のサンプルか、タンパク質が確実に枯渇することが予想される時に、または枯渇の時間の経過に複数のサンプル。例えば、5分間隔はタイミングに便利で、タンパク質レベルの範囲を提供します。図 1に示すように、最適化戦略は、適切な時間を示します。
- サンプルを処理します。
- まだ行われていない場合は、氷の上にサンプルを置く。サンプルが凍結していないことを確認します。彼らが持っている場合は、手で穏やかに暖かく、温度が局所的に上昇しないように常に反転します。
注:これは、サンプルの温度を評価することができ、それは常に寒さを感じる必要がありますので、手で行うのが最善です。氷の上に置けこれは一時停止ポイントではありません - すべてのサンプルが流体になったら、次のステップに進みます。 - すべてのサンプルが収集され、凍結されなくなったら、3,500 x gで2分間スピンします(可能な場合は4°C)。
- メタノール/ミディアムミックスを注ぎ、氷の上に戻します。すべての液体が取り除かれていない場合は心配しないでください。
- セルペレットを氷冷H2 Oの1mL(ステップ2.3.3から)で再ステーペンし、氷上の標識付き1.5mLチューブ(ステップ2.3.2で調製)に移します。
- 15,000 x gで短時間(例えば、合計10回)でスピンし、細胞を再ペレットし、氷の上に戻し、液体を取り除きます。
- 吸引によってH2 Oを取り除く。細胞ペレットは-20 °C、または-80 °Cで長期保存できます。
- まだ行われていない場合は、氷の上にサンプルを置く。サンプルが凍結していないことを確認します。彼らが持っている場合は、手で穏やかに暖かく、温度が局所的に上昇しないように常に反転します。
- ウェスタンブロット分析18によってタンパク質が枯渇したレベルを確認してください。
注:十分なタンパク質19および/または核酸は、ほとんどの目的のために単一の細胞ペレットから抽出することができるが、希少なRNA種はより多くのサンプル量を必要とする場合がある。
結果
枯渇の代表的な例を図 1に示します。この図で提示された3つの実験は、タンパク質Prp2、Prp22およびDcp1の枯渇に対する最適化実験であった。低い存在量、スプライセオソームPrp2およびPrp22タンパク質は、β-エストラジオールを用いた40分前インキュベーション後に20%未満に枯渇し、続いて15分のオーキシンが続いた。より長いインキュベーション時間は、より速い枯渇につながるが、また、auxin添加の前に望ましくないタンパク質の枯渇を示す。これに対し、より豊富なDcp1は同じ処理で約30%まで枯渇したが、潜伏前の60分は、同じオーキシン処理で13%に枯渇し、アウキシンが添加される前に枯渇した。β-エストラジオールによるプリインキュベーションの50分とオーキシンによる15分は、より短い時点で同様の結果を達成した可能性があり、より最適であったであろう。
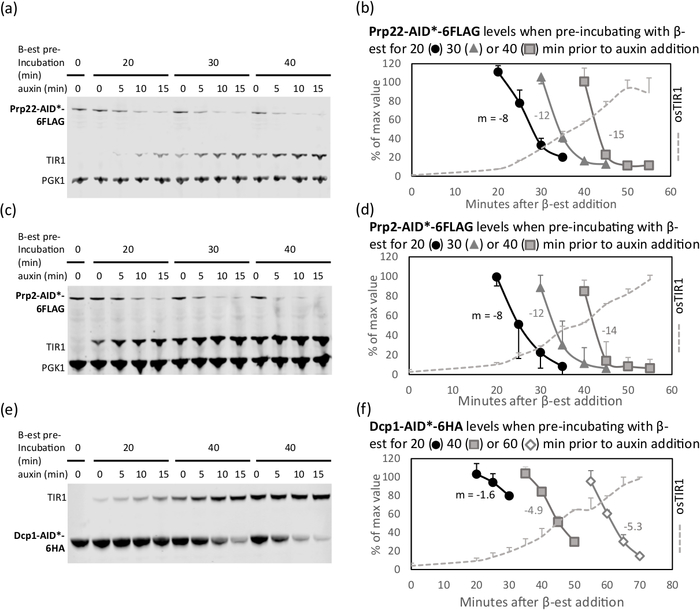
図1:βエストラジオールプリインキュベーションの持続時間を調節することにより枯渇率を調整することができる。標的タンパク質のウェスタンブロット18:(aおよびb) Prp22-AID*-6FLAG, (cおよびd) Prp2-AID*-6FLAG, および ( eおよびf)Dcp1-AID*-6HA, β-エストラジオール(β-est)で事前インキュベートされた培養物から 20, 30, 40, またはオーキシン加算12の前に60分.各レーンに同量のタンパク質を積み込んだ。Pgk1 は、Pgk1 と Dcp1 が共に移行するパネルeを除き、視覚的な読み込み制御として検出されます。パネルa、cおよびeにおけるタンパク質バンドの定量は、それぞれパネルb、d、およびfに示されている。 枯渇率の尺度として、各曲線の線形断面(初期値の100%から30%)に対して傾き(m)を計算した。最適なインキュベーション前の時間は、タンパク質レベルが依然として非誘導レベル(100%)に近い時間です。そして、その後の枯渇率は速いです。Dcp1(f)の場合、タンパク質がオーキシンの不在で分解し始めたため、潜伏前の60分が長すぎますが、この時間コースではタンパク質が著しく枯渇しないため、20分は短すぎます。40分前インキュベーション後、タンパク質が約70%枯渇し、20分でさらに枯渇するが、二次的な影響を及ぼす可能性もあるため、オーキシンを使用して15分を使用することができます。誤差バーは、2つの生物学的反復の標準偏差を表します。各実験について、1つの代表的なブロットが示されている。この図は、以前のパブリケーション9から派生しています。この図のより大きなバージョンを表示するには、ここをクリックしてください。
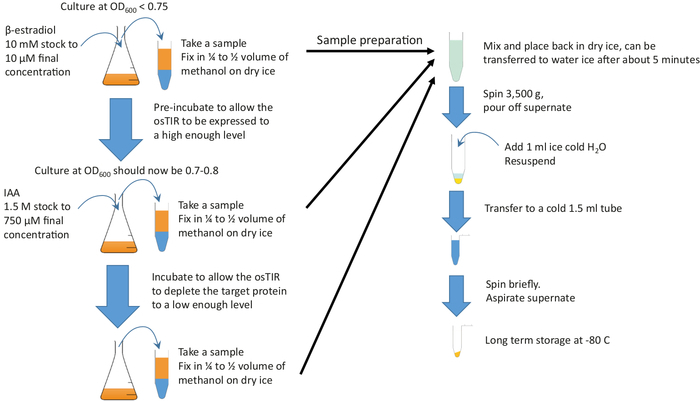
図 2: グラフィカルな概要。プレインキュベーションを開始するために、豊富な培地および必要な温度で成長する十分な培養にβ-エストラジオールを添加する。枯渇を開始するためにIAA(オーキシン)を追加する前に、必要なインキュベーション前の時間の成長を続けます.インキュベーション前および枯渇時間は枯渇するタンパク質に依存しますが、プリインキュベーションは20~60分の範囲であることが多く、枯渇時間は通常10~20分の順で、10mLサンプルはプレインキュベーションおよびデュリンの開始時と終了時に採取する必要があります。g 枯渇。これらのサンプルは、ペレットおよび貯蔵する前に冷たいメタノールで急速に固定される。この図のより大きなバージョンを表示するには、ここをクリックしてください。
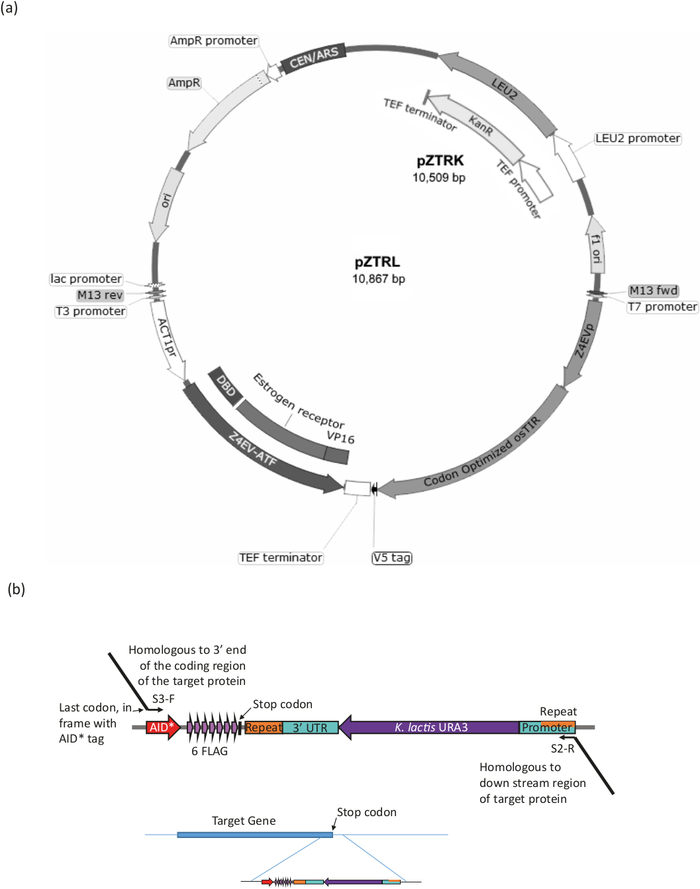
図3:Bエスト系のひずみ生成。(a) AID*システムを用いて酵母株を生成するには、pZTRL(LEU2)またはpZTRK(カナマイシン(G418)耐性)のいずれか、プラスミドを株に導入するか、または、代わりに、ATFおよびosTIRをゲノムに挿入することができる。3'エンドプライマーからPCRによって生成されたフラグメントの相同的な組み換え(図3bおよび表1を参照)。(b) 標的タンパク質のC末端タグ付けは、プラスミドpURA3-AID*-6FLAGの適切な領域のPCR増幅によって達成される(pURA3_AID*-6HAはタグのみで異なり、まったく同じ方法で処理することができる)、LongtineプライマーS3-FおよびS2-Rは、標的タンパク質の3'末端に相同的な3'拡張を伴う。フォワードプライマーエクステンションには、AID* タグの開始を持つフレーム内の最後のアミノ酸コドンを含める必要があり、ストップコドンを含めてはなりません。リバース プライマー拡張は、コーディング領域の下流の領域にする必要があります。いったんゲノムに挿入されると、URA3マーカーを失った細胞(マーカーの両端で見つかった同一領域間の相同組換えによって)は、URA3細胞を対敗する5-FOAによる増殖によって選択することができる。この図のより大きなバージョンを表示するには、ここをクリックしてください。
| a. プライマーシーケンス | |||||||
| ターゲット | 場所 | プライマー | 名前 | シーケンス | Tm (°C) | ||
| pZTRL | 516の | F | pZTRL_F | <-相同の領域->GCGACAGCCACCGACTTCG | 61.23 | ||
| 7897の | R | pZTR_R | CGCCGCCTCTCTTTAGA<-相同学の領域(RC)-> | 61.30 | |||
| pZTRK | 9154年 | F | pZTRK_F | <-相同学の領域->ACGTTGAGCCATTAATATTTTGCッタック | 59.40台 | ||
| 5897の | R | pZTR_R | CGCCGCCTCTCTTTAGA<-相同学の領域(RC)-> | 61.30 | |||
| pURA3-AID*-6フラグまたはpURA3_AID*-6HA | F | S3-F | <-相同の領域->CGTACGCTGCTCGAC | 59.21台 | |||
| R | S2-R | アトッガトットガグクCG<-相同性の領域(RC)-> | 52.76の | ||||
| pZRTL/Kはβ-est AIDシステムを増幅する | |||||||
| pURA3-AID*-6FLAG/6HAを増幅して標的タンパク質にタグ付けするAID*およびエピトープタグ(ロンチン手順) | |||||||
| <-相同の領域-> | システムが挿入される側面領域に同種の領域。この領域が長いほど、変更が成功する可能性が高くなります。50 ~ 100 のベースをお勧めします。 | ||||||
| <-相同学の領域 (RC)-> | システムが挿入される側面領域に同種の領域は、逆補数を使用することを忘れないでください。上記のように、この領域が長いほど良いです。 | ||||||
| Tm (°C) | 50 mM NaCl を使用した %GC メソッドを使用した Tm | ||||||
| b. PCRミックス | |||||||
| コンポーネント | 体積(μL) | ||||||
| テンプレート | <10 | ||||||
| NEB プシオン HF バッファー (5x)* | 100人 | ||||||
| フォワードプライマー 100 μM | 2.5年 | ||||||
| リバースプライマー 100 μM | 2.5年 | ||||||
| dNTP 各 10 mM | 10歳 | ||||||
| H2O | 500まで | ||||||
| *NEBプシオンGCバッファ(5x)も使用できますが、好ましくはありません | |||||||
| この混合物を作り、それぞれ50μlミックスの10チューブに分割し、表1 cとしてPCRを実行します。 | |||||||
| PCRがアガロースゲルで動かされたことを確認してください | |||||||
| すべての成功した反応を1つのチューブに結合し、エタノール沈殿物 | |||||||
| PCRによって生成されたすべての材料で酵母を変換します | |||||||
| c. PCR条件 | |||||||
| ステップ | 気温 (°C) | 時間 | |||||
| 初期の非核化 | 98歳 | 30 s | |||||
| 25-35 サイクル | 変性 | 98歳 | 10 s | ||||
| アニール | 45~60歳 | 20 s | |||||
| 拡張子 | 72歳 | 30 s/kb | |||||
| 最終拡張 | 72歳 | 10分 | |||||
| 保持 | 8 | ||||||
| ロンチンプライマーセット(S3-FおよびS2-R)の場合は45°C、pZTRL/Kプライマー用は60°C | |||||||
| ロンティン PCR は 3 分間、pZTRL/K では 3 分間延長します。 | |||||||
表1:プライマー配列、PCRミックスおよびPCR条件。
ディスカッション
十分に最適化されたプロトコルは、標的タンパク質の迅速かつ効率的な枯渇を生成することができます。β-エストラジオールを使用して近似プリキュベーション時間を決定することは重要であり、これは枯渇の再現性を増加させるが、インキュベーション前の小さな変動は許容できる。一方、タンパク質レベルが急速に低下するにつれて、補助添加後のタイミングに注意が必要です。
このアプローチの利点は、β-エストラジオールおよびIAAインキュベーション時間との事前インキュベーション時間の組み合わせによって調整された枯渇を達成できることである。例えば、必要に応じて、標的タンパク質は、インキュベーション前の時間を短縮することによってよりゆっくりと枯渇させることができる。
β-est AIDシステムはOsTIRが構成的に発現されるシステムに対して一定の利点を提供する。例えば、標的タンパク質が生存に不可欠である場合、osTIRの調節発現は、標的タンパク質の早期枯渇を回避することができる。さらに、osTIRの発現は、標的タンパク質の豊富さと分解に対する感受性に合わせて調整することができ、枯渇は速いか遅いかのどちらかでありうる。2つの小分子エフェクター、β-エストラジオールおよびオーキシンは、ここで使用される条件下で酵母代謝を乱さない、ラパマイシンとは異なり、アンカーアウェイシステム1で使用される。
いくつかのタンパク質をタグ付けすると、その機能が破壊されることに留意すべきです, これは、任意の標的枯渇システムの問題である.この場合、C 端末タグが機能しない場合は、N 端末タグが機能する可能性があります。また、すべてのタンパク質が効率的に枯渇するわけではありません。例えば、標的タンパク質上のAIDタグは、osTIRタンパク質にアクセスできない場合があります。したがって、AIDタグの後、各標的タンパク質は、成長に対するタグの任意の効果をテストし、β-エストラジオール前インキュベーションおよびオーキシン処理のタイミングが最適化される前に、枯渇が有効であるかどうかを決定する必要があります。
このAID*システムは非常に簡単で、タンパク質、DNAまたはRNA分析または顕微鏡のようなさらなる成長を伴わない後続の実験手順と互換性がある。さらに、システムは、新生RNA20を浄化するためにチオラベリングと組み合わせるとうまく動作します。
このシステムは、酵母細胞の代謝に影響を与えることなく、タンパク質を枯渇させる迅速で、特異的で再現可能な手段を提供する。
開示事項
著者は何も開示していない。
謝辞
このプログラムを始めてくださったジェーン・リード、開発のためのバーバラ・テルルー、"ウラ・ルーパー"の構成物のためのヴァヒド・アスランザデ、多くの有益な議論のためのスサナ・デ・ルーカスのおかげで。この研究は、メキシコのコンセホ・ナシオナル・デ・シエンシア・イ・テクノロジア(CONACYT)とエジンバラ大学生物科学部からのGIMOへの奨学金、IEM[105256]へのウェルカム博士号の学生シップ、ウェルカム・ファンド[104648]からJDベッグスへの奨学金によって支援されました。.細胞生物学のためのウェルカムセンターでの仕事はウェルカムコア資金[092076]によってサポートされています。
資料
| Name | Company | Catalog Number | Comments |
| Adenine sulphate | Formedium | DOC0230 | |
| Agar | Formedium | AGA03 | |
| Β-estradiol | Sigma Aldrich | E2758-1G | 10 mM solution in ethanol. Store at -20 °C |
| DMSO | Alfa Aesar | 42780 | DMSO should be solid at 4 °C |
| Glucose | Fisher Scientific | G/0500/60 | |
| IAA 1H-Indole-3-acetic acid | Across Orgainics | 122150100 | Auxin analogue. 1.5 M in DMSO. The solution will be a russet colour and darken as time goes on; a deep red solution should be discarded and a new one made. Store at -20 °C. |
| Methanol | Fisher Scientific | M/4000/PC17 | CAUTION Toxic and flammable |
| Phusion High-Fidelity DNA Polymerase | NEB | M0530 | |
| Peptone | Formedium | PEP03 | |
| SCSM single drop-out –ura | Formedium | DSCS101 | |
| Yeast Extract | Formedium | YEA03 | |
| Yeast nitrogen base without amino acids with amonium sulphate | Formedium | CYN0410 |
参考文献
- Haruki, H., Nishikawa, J., Laemmli, U. K. The Anchor-Away Technique: Rapid, Conditional Establishment of Yeast Mutant Phenotypes. Molecular Cell. 31, 925-932 (2008).
- Bellí, G., Garí, E., Piedrafita, L., Aldea, M., Herrero, E. An activator/repressor dual system allows tight tetracycline-regulated gene expression in budding yeast. Nucleic Acids Research. 26, 942-947 (1998).
- Alexander, R. D., et al. RiboSys, a high-resolution, quantitative approach to measure the in vivo kinetics of pre-mRNA splicing and 3′-end processing in Saccharomyces cerevisiae. RNA. 16, 2570-2580 (2010).
- Deshaies, R. J., Joazeiro, C. A. P. RING Domain E3 Ubiquitin Ligases. Annual Review of Biochemistry. 78, 399-434 (2009).
- Tan, X., et al. Mechanism of auxin perception by the TIR1 ubiquitin ligase. Nature. 446, 640-645 (2007).
- Teale, W. D., Paponov, I. A., Palme, K. Auxin in action: signalling, transport and the control of plant growth and development. Nature Reviews Molecular Cell Biology. 7, 847-859 (2006).
- Nishimura, K., Fukagawa, T., Takisawa, H., Kakimoto, T., Kanemaki, M. An auxin-based degron system for the rapid depletion of proteins in nonplant cells. Nature Methods. 6, 917-922 (2009).
- Morawska, M., Ulrich, H. D. An expanded tool kit for the auxin-inducible degron system in budding yeast. Yeast. 30, 341-351 (2013).
- Kubota, T., Nishimura, K., Kanemaki, M. T., Donaldson, A. D. The Elg1 Replication Factor C-like Complex Functions in PCNA Unloading during DNA Replication. Molecular Cell. 50, 273-280 (2013).
- Brosh, R., et al. A dual molecular analogue tuner for dissecting protein function in mammalian cells. Nature Communications. 7, 11742 (2016).
- DeRisi, J. L., Iyer, V. R., Brown, P. O. Exploring the metabolic and genetic control of gene expression on a genomic scale. Science. 278, 680-686 (1997).
- Mendoza-Ochoa, G. I., et al. A fast and tuneable auxin-inducible degron for depletion of target proteins in budding. Yeast. , (2018).
- McIsaac, R. S., et al. Synthetic gene expression perturbation systems with rapid, tunable, single-gene specificity in yeast. Nucleic Acids Res. 41, e57 (2013).
- Prusty, R., Grisafi, P., Fink, G. R. The plant hormone indoleacetic acid induces invasive growth in Saccharomyces cerevisiae. PNAS. 101, 4153-4157 (2004).
- Geitz, D., St Jean, A., Woods, R. A., Schiest, R. H. Improved method for high efficiency transformation of intact yeast cells. Nucleic Acids Research. 20, 1425 (1992).
- Widlund, P. O., Davis, T. N. A high-efficiency method to replace essential genes with mutant alleles in yeast. Yeast. 22, 769-774 (2005).
- Longtine, M. S., et al. Additional modules for versatile and economical PCR-based gene deletion and modification in Saccharomyces cerevisiae. Yeast. 14, 953-961 (1998).
- Eaton, S. L., et al. A Guide to Modern Quantitative Fluorescent Western Blotting with Troubleshooting Strategies. Journal of Visualized Experiments. , e52099 (2014).
- Volland, C., Urban-Grimal, D., Géraud, G., Haguenauer-Tsapis, R. Endocytosis and degradation of the yeast uracil permease under adverse conditions. Journal of Biological Chemistry. 269, 9833-9841 (1994).
- Barrass, J. D., et al. Transcriptome-wide RNA processing kinetics revealed using extremely short 4tU labeling. Genome Biology. 16, 282 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved