Method Article
ゼブラフィッシュにおけるin situハイブリダイゼーション染色のジェノタイピングと定量化
要約
遺伝子編集技術により、研究者はゼブラフィッシュ変異体を生成して遺伝子機能を比較的容易に調べることができます。ここでは、ゼブラフィッシュにおけるin situハイブリダイゼーションシグナルの並列的な胚ジェノタイピングおよび定量化を行うガイドを提供する。この公平なアプローチは、in situハイブリダイゼーションに基づくフェノタイプ解析においてより高い精度を提供します。
要約
in situ ハイブリダイゼーション (ISH) は、研究者が mRNA 分布を研究することを可能にする重要な技術であり、何十年もの間、発生生物学において重要な技術となっています。従来、ほとんどの遺伝子発現研究は、イシュシグナルの視覚的評価に依存していましたが、特にサンプルアイデンティティが先験的に知られている場合には、偏りが起こりやすい方法です。このバイアスを回避し、ISH信号をより正確に定量する方法について以前に報告しました。ここでは、この方法を適用して、ISH染色された胚に関心のある遺伝子の発現レベルを定量化し、その発現レベルを対応する遺伝子型と相関させる簡単なガイドを提示する。この方法は、混合遺伝子型のサンプル中の空間的に制限された遺伝子発現シグナルを定量化するのに特に有用であり、従来の視覚的スコアリング方法に代わる公平で正確な代替手段を提供する。
概要
ゲノム編集技術(ZFN、TALEN、そして最近ではCRISPR/Cas9)の導入により、これらのシステムを利用して生体内の特定の遺伝子の機能を研究する世界中の研究室が大幅に増加しました。ゼブラフィッシュは特に遺伝子操作に適しており、最近では過去1,2で多くの変異体が生成されている。発生生物学者にとって、胚発生における遺伝子変異のフェノタイプな結果を評価する最も一般的な方法の1つは、その中でハイブリダイゼーション(ISH)である。ホモ接合体変異体を野生型または異種兄弟から分離する明らかな形態学的欠陥がない場合、異なる遺伝子型を正確に正確に識別できることが不可欠である。
従来のISHは、シグナル強度の質的分析に依存して、目的の遺伝子と選択されたマーカー遺伝子との間の調節相互作用に関する結論を導き出します。有用ですが、これらの分析は技術的な変動に苦しんでおり、研究者の期待に偏っている可能性があります。従って、イシュ染色胚をイメージングした後、対応する遺伝子型の予備知識なしに遺伝子発現を定量する方法が開発された。その後、遺伝子型と遺伝子発現3を定量的に相関させる効率的なDNA抽出とジェノタイピングが行われました。イシュ後の胚のジェノタイピングは4,5以前に使用されているが、画像ベースのISHパターンの定量は、いくつかの研究6、7とは別に広く用いられていない。最も一般的な選択肢は、ISH染色された細胞8、9、10の視覚的スコアリングまたはカウントに依存し、再現性と研究者バイアスが低い傾向があります。この方法は、runx1またはgata2bのような空間的に制限された発現パターンを有する遺伝子の変化を研究するのに特に有用であり、両方とも血原性内皮11,12と呼ばれる大動脈床細胞の制限されたサブセットで発現する。
ここでは、フィジー13を用いた画像解析による定量化の実用ガイドと、DNA抽出とジェノタイピングプロトコルを提供することを目指す。これは、以前に公開された方法3を視覚的に示すことを目的としたものです。我々の方法は、ISHによって検出された遺伝子発現の変動と、特定の遺伝子型への遺伝子発現レベルの偏った割り当てを正確に表すことを可能にする。
プロトコル
動物を対象とする手続きは、1986年の動物(科学的手続き)法によって規制されており、内務省および地元の動物福祉倫理審査機関によって承認されています。
1. 画像ISH染色胚
- グリセロール溶液(1x PBSバッファーで50%~80%)を調製し、混合して溶液を均質化する(例えば、少なくとも5分間ローラーに放置する)。この溶液は室温で数ヶ月間保つことができる。
- その後、ハイブリダイゼーション14、15、16、17、3mLパスツールピペットでグリセロール溶液に胚を移し、少なくとも5分間沈着させる。24hpf(受精後数時間)より古い胚を撮像する場合、それらは18に記載されているように漂白され得る。
- イメージング後に、ISH染色された胚を転写するのに十分なPCRチューブを準備し、標識します。
- 3 mL パスツールピペットを使用したガラスのうつ病スライドでウェルの底に 100% グリセロールを加えます。
- 3 mL パスツールピペットを使用して、デジタル カメラと底面と上面の照明を備えたステレオ顕微鏡の下で、必要に応じて単一の ISH 染色された胚をスライドと方向に移します。
注:イメージング用の胚を配置するには、ゲルローディングピペットチップを使用しますが、他のツール(鉗子、解剖針など)も同様に適切です。 - 第1の胚を用いて、所望の倍率で照明および露光時間を調整する。同じ実験ですべての胚に対してこれらの条件を使用します(すなわち、ヘテロ接合変異体インクロスから40個の胚をイメージングする場合は、照明、暴露時間および倍率がすべて同じであることを確認してください)。
- 必要な数の胚を画像化します。各画像に一意の番号を付けます。イメージング後、同じ番号でラベル付けされたPCRチューブ/プレートに胚を移します。
注: 画像は TIF ファイルとして保存する必要がありますが、他の形式でも十分です。- 必要に応じて、PCRチューブ/プレート中の過剰なグリセロールを除去します。
注:この時点で、胚は室温で数週間PCRチューブに保存することができます。
- 必要に応じて、PCRチューブ/プレート中の過剰なグリセロールを除去します。
2. DNAを抽出し、ISH染色された胚を遺伝子型にする
注:ここでは、95%-100%3のDNA抽出効率を持つHoTSHOT法19に基づくゲノムDNAを分離するために、信頼性の高い安価な方法を使用する。
- イメージングが完了したら、各チューブに40〜75μLのアルカリ性リシスバッファー(例えばHoTSHOT)を加えます。
- 95°Cで約30分間インキュベートし、チューブを4°Cまで冷却してから、等量の中和バッファーを加えます。4 °Cでの夜間インキュベーションは、PCR効率を向上させる可能性があります。
注:この時点で、ゲノムDNAはジェノタイピングに使用したり、必要になるまで-20°Cで保存することができます。 - 適切な方法を有するジェノタイプサンプル(例えば、HRMA、RFLP)3、20、21は目的の突然変異に必要である。
- 各サンプルに対応する遺伝子型に注意してください(例えば、スプレッドシートソフトウェアを使用)。
3. ISH染色胚の画素強度を定量化する(フィジーソフトウェアを用いた画像解析)
- in situ ハイブリダイゼーション(ISH)染色信号強度を定量化するには、3に説明されているようにすべての画像を 8 ビットグレースケールに変換する。画像がとして保存された場合TIF ファイルでは、バッチ変換にフィジー マクロを使用します 3。または、他の形式(例えば、.)で画像を変換します。にします) にします。適切なソフトウェアを使用した TIF を、フィジーを使用して 8 ビットのグレースケールに変換します。便宜上、以下は、以前に公開された手順 3 を変更して、いくつかの変更を行った手順です。
- フィジーで画像を開き、編集 >反転して画像を反転します。次に、イメージの種類を 8 ビットに変更します (イメージ > 種類 > 8 ビット)。
- ポリゴン選択ツールを使用して、信号を含む領域の周囲の画像に、関心のある領域(ROI)を手動で描画します。
- ROI マネージャーを開くには、t を押します。ROI マネージャーの[測定] コマンドを使用して、ROI の強度を測定します。[結果] ウィンドウの平均値をスプレッドシート ソフトウェアにコピーします。
- 同じROIを移動し、元の領域と同じサイズと形状を確実に、染色を含まないゼブラフィッシュの領域に移動する。手順 3.4 を繰り返して、背景を測定します。
- ISHシグナルの平均画素強度を求めるには、各胚の染色領域の平均強度値から背景領域の平均強度値を差し引く。
- 各強度値を遺伝子型に割り当てます(ステップ 2.3 から)。
4. 適切な統計検定で結果を分析
- 1つのQ-Qプロット上のすべての値をプロットして、正規分布からの偏差を特定します。
注:通常の分布は、コルモゴロフ-スミルノフ検定またはシャピロ・ウィルク検定でも検証できます。しかし、大きなサンプルサイズの場合、これらのテストでは誤検出のリスクが高くなります。 - 正規分布からの偏差が強い場合は、(ln または sqrt 関数を使用して) すべての値を変換して、処理を進める前に、それらが正規分布であることを確認します。
- 95%の信頼度を持つ2尾分散分析を用いて、各遺伝子型(wt vs. ヘテロ接合体対突然変異体)に割り当てられた値(必要に応じて変換された)の値の差を分析し、レヴェンの検定とウェルチ補正との分散の等値を考慮する。各遺伝子型ペアのペアワイズ比較では、Tukey(等分散)またはゲームハウエル(不等分散)のポストホック検定を使用します。
- 変換にもかかわらず値が正規分布していない場合は、ノンパラメトリック検定(Kruskall-Wallis)を使用して、ランク付けされた値と、ペアワイズのボンフェローニ補正によるポストホックダンの多重比較検定の差を分析します。比較。
- 結果を最適に表現するために、(ステップ 3.6 から) 変換されていない値をドットプロットとしてプロットします。
結果
ここでは、他の場所に公開されている画像定量と胚ジェノタイピングのためのパイプラインの実用的なアプリケーションについて説明します3.このメソッドのワークフローを図 1に示します。この方法を使用する方法を説明するために、ishは、runx1W84X/+22インクロスから33 hpf胚のdnmt3bb.1に対して実行されました(図2)。130個の胚を、プロトコルで詳述されているのと同じ照明条件を使用して画像化し、固有の番号でラベルを付けた。イメージング後、各胚をジェノタイピング用PCRチューブに移した。この時点で、各画像にピクセル強度値を帰属させるために画像分析を行った。その後、その遺伝子型を対応する画像に割り当て、統計的分析のためにその遺伝子型に従ってグループ化されたピクセル強度値を示します。以前の観測値 5 と一致して、runx1W84X/W84X変異体 (図 2A,B)3でdnmt3bb.1式の減少が検出されました。興味深いことに、runx1W84x/+ヘテロ接合系胚は、野生型の兄弟と比較してdnmt3bb.1発現(図2A,B)に有意な差を示さなかったが、Runx1の1コピーは適切なレベルでdnmt3bb.1発現を維持するのに十分であることを示唆した。
多くのゼブラフィッシュ変異体は、モルフォリーノオリゴヌクレオチド(MO)のような他の機能技術を使用して検出できる胚性表現型を示すことができず、それ以外の場合があります。この不一致は、オフターゲット効果、母体タンパク質補償、過型アレーレ23、または遺伝的補償24、25、26、27の最近発見された現象を含む多くの原因に起因することができる。この例では、lmo4aMOを用いて以前に公表されたデータから、lmo4aの形態素28でrunx1が減少することを示唆したので、runx1発現がlmo4a uob100突然変異体において減少したか失われたかを尋ねた。ここで、解析では野生型とlmo4auob100ホモ接合体変異体3との間のrunx1発現に有意な差は見なかった(図3A、B)。さらに単一胚qPCRによる分析は、lmo4auob100変異体におけるrunx1発現の小さいが有意な減少があったことを示した(図3C)。したがって、画像定量化では、発現レベルのわずかな違いを検出できない可能性があります。あるいは、検出した遺伝子型の違いが無く、qPCR実験では、lmo4aとrunx1の両方が発現するテレンシェファロンのような他の組織におけるrunx1発現の変化を検出しています。研究者は常にqPCRのような独立した方法で結果を検証する必要がありますが、例えばフローサイトメトリーによって関心のある組織のために理想的に富む。
ISH のバックグラウンドが高い場合(図3D)、この領域のピクセル強度値が非常に高いため、シグナル値からの減算は負の数を生み出し、そのような場合はそれらの胚は分析から除外されます。私たちの経験では、これはrunx1-プローブ胚3の約0.4%で起こったが、実験、プローブまたは試薬のバッチによって異なる可能性がある。それは方法の限界かもしれませんが、高いバックグラウンドの低頻度は全体的な結果に影響を与える可能性は非常に低いです。
背景補正のための異なる領域を選択する効果をテストするために、我々はまず、バックグラウンド補正のための異なる領域を使用して、28 hpf胚におけるrunx1 ISHシグナルのピクセル強度を測定した(図4)。4つの異なる領域が選択された:トランク領域(R1、およびR2)に2つ、黄身領域に1つ(染色されていないが、バックグラウンド染色を蓄積する可能性が高い)とROIに対する小さな領域の前(R4、図4B)。これらの領域におけるピクセル強度の測定は、ROIといずれかのバックグラウンド領域との間の強度の相対的に安定した差を示した(図4C)。しかし、R3 は常に非常に高い値を示しました (ROI の値を上回る)。反転と8ビットへの変換後、黄身領域は非常に明るく見えるので、背景補正として使用するには適していません。R2はROIに近いが、いくつかのISH信号を含んでいた、と補正のためにそれを使用すると、R1(さらに後方に位置し、ISH信号から離れて)またはR4のいずれかと比較すると、平均ピクセル強度を減少させた。したがって、R1 または R4 はバックグラウンド修正に使用できる適切な領域です (R4 の領域が R1 の領域よりも小さい場合でも)。次に、Runx1式を比較する際に R1 または R4 を使用した結果の影響を比較したいと考えました。このために、我々は、交差したdll4+/-ヘテロ接合体29を、ランダムに選択された野生型とdll4-/-胚でrunx1発現を分析した(図4E)。背景補正にR1またはR4を使用して個々の値に影響を与えたが、同じ遺伝子型内の平均画素強度は有意に異なっていなかった(図4E)。さらに、runx1式を比較すると、R1 または R4 領域を背景補正として使用する遺伝子型間で、平均強度値が類似しています (それぞれ、R1=16.3 と μR4=18.2)。まとめると、背景領域の選択は重要ですが、主な基準は黄身領域(バックグラウンド染色の蓄積を起こしやすい)を含まず、背景の画素強度値を歪める可能性のある(特定の)染色を含むべきではない、と結論付けました。
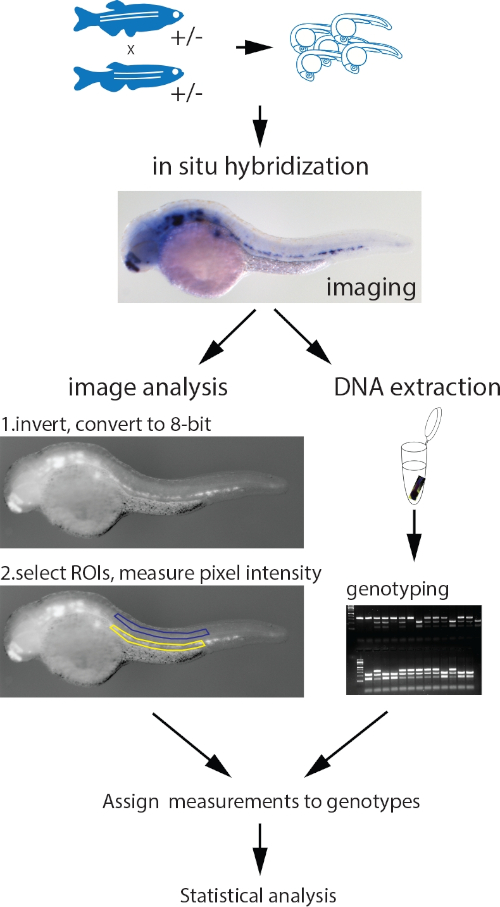
図1:パラレル画像定量とジェノタイピングプロトコルのワークフロー変異対立遺伝子の魚のヘテロ接合から採取された胚は、標準的なISHプロトコルを用いて測定された遺伝子についてプローブされる。イメージング後、0.2 mL PCRチューブでリシスバッファーを直接胚に加え、95°Cで30分インキュベーションを行い、HotSHOTプロトコルを使用してゲノムDNAを抽出します。このDNAは、PCR、PCRおよび制限断片長多型(RFLP)、KASPアッセイ、またはその他の適切な方法による胚の遺伝子型化に使用されます。並行して、各胚の画像は反転され、8ビットグレースケールに変換されます。ISH信号(黄色)と背景(青)を含む同一の形状とサイズのROIは、手動で選択され、測定されます。対応する遺伝子型に割り当てられた測定値は、統計的に分析されます。ドブジキら3より大きいバージョンの図を見るには、ここをクリックしてください。
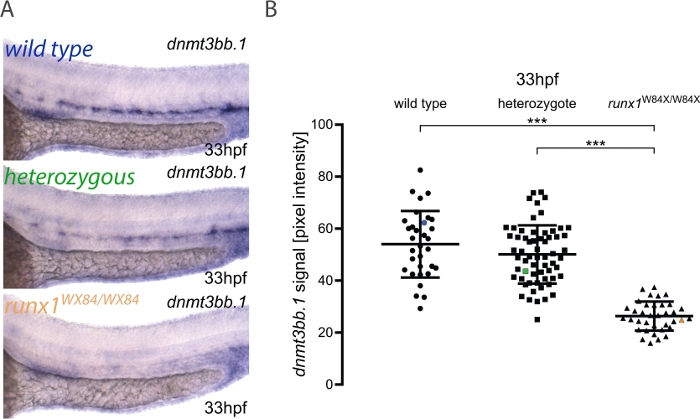
図2:runx1変異体における画像定量は、ISHによるdnmt3bb.1発現の減少レベルを明らかにする。(A)33hpf野生型(青)、runx1 +/W84X(緑)およびrunx1W84X/W84X(オレンジ)胚におけるISHの例画像は、背部大アルタにおけるdnmt3bb.1発現を示す。(B) runx1W84X/W84X胚(n=36)のdnmt3bb.1 mRNAのピクセル強度値は、野生型(n=32)およびヘテロ接合体(n=62)と比較して有意に減少する(ANOVA、p<0.001)。 変動係数は、野生型、ヘテロ接ウト基、変異基の場合、それぞれ24%、22%、および21%です。青、緑、オレンジ色のデータポイントは、パネルAの画像の例に対応しています。バーは平均 ± s.d. ***p<0.001 (ゲームハウエルポストホックテスト)を表します。ドブジキら3より大きいバージョンの図を見るには、ここをクリックしてください。
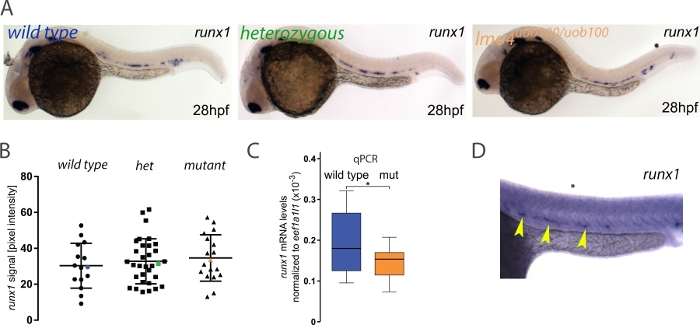
図3: lmo4auob100変異体におけるISHによるrunx1発現レベルの測定(A)28hpf野生型(青)、ヘテロ接合(緑)およびlmo4auob100/uob100(オレンジ)胚におけるrunx1のISHの代表的な画像は、背側大小小田における発現を示す。(B) runx1 mRNAシグナルの定量 ISHによって検出された、28 hpf野生型(n=15)から、ヘテロ接合体lmo4a+/-(het)(n=34)およびlmo4auob100/uob100突然変異体(n=18)の1つのクラッチからの胚は、異なる遺伝子型(ANOVA、>p.0.p.0.)間でrunx1ピクセル強度に有意差を示さない。青、緑、オレンジ色のデータポイントは、パネルAの画像の例に対応しています。バーは、単一の野生型(青;n=12)およびlmo4auob100/uob100(ミュート、オレンジ;n=12)胚で正規化されたrunx1 mRNAレベル(2-ΔCt)を表示するボックスプロットを表し、qRT-PCRによって測定され、突然変異体のrunx1のレベルが減少した。*p < 0.05 (tテスト)(D)高い背景を示す28 hpf胚(runx1に染色された黄色の矢印)に対するISH実験の例。ドブジキら3より大きいバージョンの図を見るには、ここをクリックしてください。
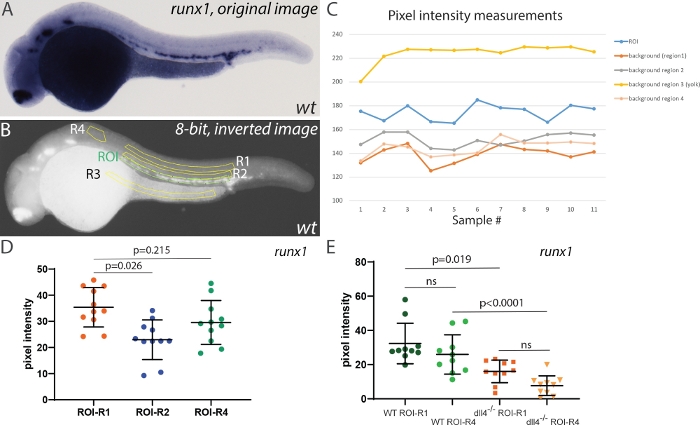
図4:測定結果に対する背景強度補正の効果(A)28hpfで野生型胚におけるrunx1 ISH染色の代表的な画像。(B) 反転して 8 ビットに変換した後の同じイメージ。関心領域(ROI)が緑色でハイライトされ、背景補正に使用される4つの異なる領域(R1-R4)が黄色で強調表示されます。(C) パネル B に示す全ての領域における生の画素強度測定は、R3(黄身)の強度がROIの実際のISH信号(n=11)よりも一貫して高いことに注意してください。(D) R1、R2、R4 バックグラウンド領域を使用する ROI のRunx1式レベル。ROI、R1、R2、R3~28500 ピクセルのエリア。R4~8500 ピクセル。バックグラウンド補正(ROI-R3)が一貫して負の値を生成したため、R3 の背景はこの比較では使用されません。(E) バックグラウンド補正に R1 または R4 を使用する野生型およびdll4-/-変異体のRunx1式レベル (サンプルごとに n=10)。パネルDおよびEにおける統計解析は、ピクセル強度値が正規分布していないと仮定して、非パラメトリックなKruskal-Wallis検定を用いて行った。この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
遺伝子発現を定量化するためにこの方法を使用する場合、いくつかの要因を考慮する必要があります。測定間の変動を低減するために、イメージング条件は実験全体(照明、露光時間、胚位置など)を維持する必要があります。重要な点は、サンプル間の染色の違いがマスクされる可能性があるため、サンプルの過剰染色を避けることです。例えば、ゼノプス・レービス胚30におけるEto2の不在時のVegfA発現の減少は、24時間にわたる染色を注意深くモニタリングすることによってのみ検出することができた。したがって、その発現を最もよく表す各遺伝子の適切な染色レベルを、飽和状態に達することなく経験的に決定することが良い習慣です。オーバーステインは、変換された 8 ビットグレースケール画像の背景ピクセル強度を人工的に増加させ、定量結果を歪めます。極端な場合、胚組織のバックグラウンドレベルは、選択されたROIのISHシグナルよりも高く、これらのサンプルは分析から除外されるべきである。同様の現象が見られたのは、未染色の黄身の背景補正の適合性をテストした場合です(図4)。反転して8ビットに変換すると、黄身領域の暗いピクセルは胚のISHシグナルよりも明るくなり、背景補正された値を否定的にレンダリングします。したがって、背景補正のために黄身の使用を避けてください。胚の色素領域(例えば、26/28 hpf以降の目または胴体の背側部分)のバックグラウンドシグナルを測定することは、定量結果を均等に歪め、また避けるべきである。イシュ18の前または後にゼブラフィッシュ胚を漂白するためのプロトコルがあり、イメージングが推奨される前に24 hpfより古い胚を漂白する。
この方法は、定義された領域のピクセル強度を、同等の非染色領域の背景ピクセル強度に対して測定する必要があるため、ユビキタスまたはほぼユビキタスに発現した遺伝子の定量には適切ではない。その代わり、背景ピクセルの強度を測定する領域を容易に同定できる空間的に制限された分布を持つ遺伝子の発現を測定する場合に適しています。今回の追加分析では、バックグラウンド補正に小さい面積(3~4倍小さい)を使用すると、ROIと同等の面積を使用する場合と同様の結果が得られます。これは、バックグラウンド補正のために胚の明らかに染色されていない領域を使用できる限り、より広い空間ドメインで発現される遺伝子に対する方法の適用性を拡張する(したがって、強度測定のためにより大きなROIを必要とする)。
最後に、実験者のバイアスを最小限に抑えるために、ジェノタイピングを並列または画像定量後に実行することを提案する。2人目の実験者に匿名化されたサンプルの定量を繰り返し、最初の測定値と比較するように依頼すると、実験者のバイアスを減らすのにも役立ちます。定量化する画像がジェノタイピングを必要としない治療(例えば、野生型対化学阻害剤または野生型対MOノックダウン)の比較からである場合、測定を行う実験者は、その同一性に目がくらむべきである。サンプル。
開示事項
著者らは、彼らが競合する財政的利益を持っていないと宣言します。
謝辞
オックスフォードとバーミンガムのバイオメディカルサービスのスタッフに、素晴らしいゼブラフィッシュの夫に感謝したいと思います。T.D.はウェルカムトラスト染色体および発生生物学博士課程奨学金(#WT102345/Z/13/Z)によって資金提供されました。R.M.とM.K.は英国心臓財団(BHF IBSRフェローシップFS/13/50/30436)から資金提供を受けており、彼らの寛大な支援に感謝しています。R.M.は、オックスフォードのBHFリサーチ・エクセレンス・センター(RE/13/1/30181)からの支援を認めています。
資料
| Name | Company | Catalog Number | Comments |
| 0.2 mL PCR tubes (8-strips with lids) | StarLab | A1402-3700 | 96-well plates are equally appropriate for sample handling but beware of cross contamination between samples |
| 3 mL Pasteur pipettes | Alpha Laboratories | LW4114 | |
| Cavity slides | Brand | BR475535-50EA | |
| Digital Camera (Qimaging Micropublisher 5.0) | Qimaging | ||
| Eppendorf Microloader tips | Eppendorf | 10289651 | the tips are used to orient the embryos for imaging in glycerol |
| Excel | Microsoft | ||
| F3000 Fiber Optic Cold Light Source | Photonic | ||
| Fiji | |||
| Glycerol | Sigma | G5516-1L | |
| Graphpad Prism 8.01 | GraphPad Software, Inc. | we prefer to use Graphpad, but other statistics software packages are also suitable (e.g. SigmaPlot or SPSS) | |
| HotSHOT alkaline lysis buffer | 25 mM NaOH, 0.2 mM disodium EDTA, pH 12 | ||
| HotSHOT neutralization buffer | Tris HCl 40 mM, pH 5 | ||
| PBS (10X) pH 7.4 | Thermofisher | 70011044 | |
| Stereomicroscope with illumination stand (Nikon SMZ800N) | Nikon | ||
| Thermocycler | Thermofisher |
参考文献
- Varshney, G. K., et al. High-throughput gene targeting and phenotyping in zebrafish using CRISPR/Cas9. Genome Research. 25 (7), 1030-1042 (2015).
- Varshney, G. K., et al. CRISPRz: a database of zebrafish validated sgRNAs. Nucleic Acids Research. 44 (D1), D822-D826 (2016).
- Dobrzycki, T., Krecsmarik, M., Bonkhofer, F., Patient, R., Monteiro, R. An optimised pipeline for parallel image-based quantification of gene expression and genotyping after in situ hybridisation. Biology Open. 7 (4), bio031096 (2018).
- Bresciani, E., et al. CBFbeta and RUNX1 are required at 2 different steps during the development of hematopoietic stem cells in zebrafish. Blood. 124 (1), 70-78 (2014).
- Gore, A. V., et al. Epigenetic regulation of hematopoiesis by DNA methylation. Elife. 5, e11813 (2016).
- Fan, Y., et al. Tissue-Specific Gain of RTK Signalling Uncovers Selective Cell Vulnerability during Embryogenesis. PLoS Genetics. 11 (9), e1005533 (2015).
- Wen, B., et al. GATA5 SUMOylation is indispensable for zebrafish cardiac development. Biochimica et Biophysica Acta. 1861 (7), 1691-1701 (2017).
- Espin-Palazon, R., et al. Proinflammatory signaling regulates hematopoietic stem cell emergence. Cell. 159 (5), 1070-1085 (2014).
- Peterkin, T., Gibson, A., Patient, R. Redundancy and evolution of GATA factor requirements in development of the myocardium. Developmental Biology. 311 (2), 623-635 (2007).
- Genthe, J. R., Clements, W. K. R-spondin 1 is required for specification of hematopoietic stem cells through Wnt16 and Vegfa signaling pathways. Development. 144 (4), 590-600 (2017).
- Kalev-Zylinska, M. L., et al. Runx1 is required for zebrafish blood and vessel development and expression of a human RUNX1-CBF2T1 transgene advances a model for studies of leukemogenesis. Development. 129 (8), 2015-2030 (2002).
- Butko, E., et al. Gata2b is a restricted early regulator of hemogenic endothelium in the zebrafish embryo. Development. 142 (6), 1050-1061 (2015).
- Schindelin, J., et al. Fiji: an open-source platform for biological-image analysis. Nature Methods. 9 (7), 676-682 (2012).
- Lleras Forero, L., et al. Segmentation of the zebrafish axial skeleton relies on notochord sheath cells and not on the segmentation clock. Elife. 7, (2018).
- Jowett, T., Yan, Y. L. Double fluorescent in situ hybridization to zebrafish embryos. Trends in Genetics. 12 (10), 387-389 (1996).
- Thisse, C., Thisse, B. High-resolution in situ hybridization to whole-mount zebrafish embryos. Nature Protocols. 3 (1), 59-69 (2008).
- Narayanan, R., Oates, A. C. Detection of mRNA by Whole Mount in situ Hybridization and DNA Extraction for Genotyping of Zebrafish Embryos. Bio-protocol. , e3193 (2019).
- Monteiro, R., Pouget, C., Patient, R. The gata1/pu.1 lineage fate paradigm varies between blood populations and is modulated by tif1gamma. EMBO JOURNAL. 30 (6), 1093-1103 (2011).
- Truett, G. E., et al. Preparation of PCR-quality mouse genomic DNA with hot sodium hydroxide and tris (HotSHOT). Biotechniques. 29 (1), 52-54 (2000).
- Wilkinson, R. N., Elworthy, S., Ingham, P. W., van Eeden, F. J. A method for high-throughput PCR-based genotyping of larval zebrafish tail biopsies. Biotechniques. 55 (6), 314-316 (2013).
- Parant, J. M., George, S. A., Pryor, R., Wittwer, C. T., Yost, H. J. A rapid and efficient method of genotyping zebrafish mutants. Developmental Dynamics. 238 (12), 3168-3174 (2009).
- Jin, H., et al. Definitive hematopoietic stem/progenitor cells manifest distinct differentiation output in the zebrafish VDA and PBI. Developement. 136 (4), 647-654 (2009).
- Stainier, D. Y. R., et al. Guidelines for morpholino use in zebrafish. PLoS Genetics. 13 (10), e1007000 (2017).
- El-Brolosy, M. A., Stainier, D. Y. R. Genetic compensation: A phenomenon in search of mechanisms. PLoS Genetics. 13 (7), e1006780 (2017).
- Rossi, A., et al. Genetic compensation induced by deleterious mutations but not gene knockdowns. Nature. 524 (7564), 230-233 (2015).
- Ma, Z., et al. PTC-bearing mRNA elicits a genetic compensation response via Upf3a and COMPASS components. Nature. 568 (7751), 259-263 (2019).
- El-Brolosy, M. A., et al. Genetic compensation triggered by mutant mRNA degradation. Nature. 568 (7751), 193-197 (2019).
- Meier, N., et al. Novel binding partners of Ldb1 are required for haematopoietic development. Development. 133 (24), 4913-4923 (2006).
- Kettleborough, R. N., et al. A systematic genome-wide analysis of zebrafish protein-coding gene function. Nature. 496 (7446), 494-497 (2013).
- Leung, A., et al. Uncoupling VEGFA functions in arteriogenesis and hematopoietic stem cell specification. Developmental Cell. 24 (2), 144-158 (2013).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved