このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
ゲノムワイド往復半角化分析によるサッカロマイセス酵母の種間の熱耐性の遺伝学的マッピング
要約
シーケンシング(RH-seq)を介した相互異形性は、種間の形質の違いの遺伝的基礎をマッピングする強力な新しい方法である。ヘミジゴットのプールはトランスポゾン変異によって生成され、その適合性は、高いシーケンシングを使用して競争力のある成長を通じて追跡されます。得られたデータの分析は、形質の基礎となる遺伝子を特定します。
要約
現代遺伝学の中心的な目標は、野生の生物が表現型で異なる方法と理由を理解することです。現在までに、この分野は、種の個体間の交配から組換え子孫間のDNA配列変異体と表現型との関係をトレースするリンケージおよびアソシエーションマッピング法の強度に大きく進歩した。これらのアプローチは、強力ですが、生殖的に分離された種間の形質の違いに適していません。ここでは、互換性のない種に容易に適用できる自然形質変動のゲノム全体の解剖のための新しい方法について説明する。私たちの戦略、RH-seqは、相互ヘミジゴット試験のゲノム全体の実装です。我々はそれを利用して、その姉妹種S.パラドックスに対する酵母サッカロマイセスセレビシエの顕著な高温増殖の原因となる遺伝子を同定した。RH-seqはトランスポゾン変異を利用して相互ヘミジゴットのプールを作成し、ハイスループットシーケンシングを介して高温競争を通じて追跡されます。ここで説明した RH-seq ワークフローは、新芽酵母クレードの古代の複雑な形質を解剖する厳格で公平な方法を提供し、遺伝的マッピングのためのゲノムカバレッジを確保するためにリソース集約的な深いシーケンシングが必要であることを警告します。シーケンシングコストが低下するにつれて、このアプローチは真核生物全体での将来の使用に大きな期待を持っています。
概要
フィールドの夜明け以来、それは野生の個体間の変動の機械的基礎を理解するために遺伝学の主要な目標となっています。関心のある形質の根底にある遺伝子座をマッピングすると、新しい遺伝子は診断や薬剤の標的として即座に使用され、進化の原理を明らかにすることができます。この目的に向けた業界標準は、リンケージまたはアソシエーション1を介して集団全体の遺伝子型と表現型の関係をテストすることです。これらのアプローチは強力であり、彼らは1つの主要な制限を持っています - 彼らは相互不妊の個人間の交差から組換え子孫の大きなパネルに依存しています。そもそも子孫を形成することができない種の研究では役に立たない。したがって、この分野は、生殖的に単離された種2の間の形質の違いを公平に解剖する能力がほとんど持っていなかった。
本研究では、種間の形質変動の遺伝的基盤のゲノムスケール調査に関する新しい方法RH-seq3の技術的基盤を報告する。このアプローチは、相互ヘミジゴット試験4、5の非常に平行なバージョンであり、これは最初に2つの遺伝的に異なる背景の間の対立性の差異の表現効果を評価する方法として考案された特定の軌跡(図1A)。このスキームでは、2つの異なる個体が最初にハイブリッドを形成するために交配され、そのゲノムの半分はそれぞれの親から来ている。この背景には、複数の株が生成され、それぞれが各親の軌跡の対立遺伝子の中断または削除されたコピーを含む。これらの株は、それらは、関心のある軌跡を除いてゲノムのいたるところに浸留し、ハプロイドとみなされ、それぞれが1つの親の対立遺伝子しか欠け、残りの対立遺伝子が由来するので、多角質である。他の親。これらの相互ヘミジゴット株のフェノタイプを比較することにより、遺伝子座の変異体が相互の間の唯一の遺伝的差異であるため、操作された遺伝子座におけるDNA配列変異体が関心のある形質に寄与するかどうかを結論付けることができる。ヘミジゴット株。このようにして、種間の遺伝的差異を、よく制御された実験セットアップでそれらの間の見型的な違いに結び付けが可能である。現在までに、この試験の応用は候補遺伝子フレームワーク、すなわち候補遺伝子座の自然変動が形質に影響を与える可能性があるという仮説が既に手に入っている場合である。
以下では、酵母をモデルシステムとして用いて、ゲノムスケールの相互半角膜スクリーンのプロトコルをレイアウトする。我々の方法は、種間で生存可能な無菌F1ハイブリッドを生成し、トランスポゾン変異形成を受けることによって、ヘミジゴット変異体のゲノム補体を作成する。我々は、ヘミジゴットをプールし、シーケンシングベースのアッセイでそれらの表現型を測定し、所定の遺伝子の2つの親の対立遺伝子を有するプールのクローン間の頻度の違いをテストする。その結果、種間の変異体が関心のある形質に影響を与える遺伝子座のカタログが得られます。我々は、2つの新芽酵母種、サッカロマイセスセレビシエとS.パラドックスの間の熱耐性の違いの遺伝的基礎を解明するためにRH-seqワークフローを実装し、6年前に約500万年前に発散した。
プロトコル
1. 形質転換のためのピギーバク含有プラスミドの調製
- 単一のコロニーにストリークは、LB +カルベニシリン寒天プレート上にプラスミドpJR487を収容する大腸菌株を収容する。37°Cで1泊、または単一のコロニーが現れるまでインキュベートします。
注:プラスミドpJR487がクローン化された方法の説明は、前の作品3で見つけることができます。 - 2LガラスフラスコにpJR487を含有する大腸菌の単一コロニーを有するLB+カルベニシリンの1Lを100μg/mLで接種する。飽和するまで200 rpmで振りながら37°Cで一晩成長します(OD600 ≥ 1.0)。
- 製造元の公開プロトコルで指示されているように、大規模なプラスミド準備キットを使用して培養物からプラスミドDNAを精製します(詳細は材料の表を参照)。溶出バッファーの5 mLを37°Cに温めた5mLで10分間インキュベーションした後にDNAを溶出させる。
- 分光光度計を用いてプラスミドDNAの量と質を測定します(詳細は材料表を参照)。
- ステップ1.2~1.4を繰り返し、A260で少なくとも11mgのプラスミドDNAの合計が単離されるまで:A280比が少なくとも1.8個単離されるまでである。これは、効率に応じて、いくつかの準備を取る場合があります。
- すべてのプラスミドの準備物を1つのチューブに混ぜ、溶出バッファーまたは水で20 mLまでの総容積を持って来る。分光光度計で最終的な量と品質を再度測定します。プラスミドの濃度は、この最終20 mL体積において少なくとも538 ng/μLでなければなりません。濃度が538 ng/μLより高い場合は、溶出バッファーまたは水でプラスミドを538 ng/μLに希釈します。プラスミドは、使用するまで数週間まで4°Cで保存することができる。
2. 標的化されていないゲノム全体の相互ヘミジゴットのプールを作成する
- 形質転換のためのハイブリッド酵母細胞の調製
- -80 °Cの冷凍庫ストック株からYPD寒天プレート上の単一のコロニーにJR507をストリークアウト。26 °Cで2日間、またはコロニーが現れるまでインキュベートします。
注:JR507は、S.セレビシアDBVPG1373およびS.パラドキスZ1(四面体解剖顕微鏡を用いて)3のハプロイド胞子の単細胞交配を介して作られたハイブリッド株である。 - JR507の単一のコロニーで250 mLのガラスフラスコで液体YPDの100 mLを接種し、28°C、200 rpmで24時間、または静止相に達するまで振ります。
- 翌日、一晩培養の600nm(OD600)で光学密度を測定する。新鮮な液体YPDで一晩培養の一部を0.2のOD 600と500 mLのOD600に新しい1Lガラスフラスコに戻すことによって、新しい文化を作成します。
注: 一晩培養に OD600が 5.0 で、C が光学密度で V がボリュームである場合の逆希釈の計算例: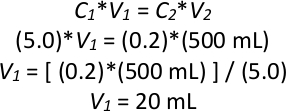
したがって、飽和一晩培養の20mLを液体YPDの480 mLに添加し、0.2のOD600で合計500mLの培養を行う。 - ステップ 2.1.3 をさらに 3 回繰り返し、4 つの 1 L ガラスフラスコで 0.2 の OD600で合計 4 つの 500 mL 培養を行い、4 つの新しい培養物すべてに同じ一晩培養を使用します。28°Cで6時間(2~3世代)振り、200rpmでインキュベートします。
- 500 mL 培養物のうち 2 つを組み合わせて、1 L 培養を作成します。残りの 2 つの 500 mL 培養を組み合わせて、別の 1 L 培養を作成します。この時点で、2 つの 1 L カルチャがあります。これらの 1 つの L 培養のそれぞれは、次の手順で pJR487 を使用して変換されます。
- -80 °Cの冷凍庫ストック株からYPD寒天プレート上の単一のコロニーにJR507をストリークアウト。26 °Cで2日間、またはコロニーが現れるまでインキュベートします。
- pJR487をハイブリッド酵母細胞に変換
- 1L培養物を20本のプラスチック円錐管に20個の50mLのアリコットに分割し、合計40本のチューブを使用します。20本のチューブを脇に置き、一度に20本のチューブで次の手順を実行します。
- 酵母細胞をペレットに1,000 x gで3分間20本のチューブのそれぞれを遠心分離する。上清を捨てます。
- 渦によって無菌H2 Oの25 mLで各ペレットを再懸濁する。1,000 x gで3分間遠心分離機。上清を捨てます。
- 渦によって1x TE、0.1 M LiOAcバッファーの5 mLで各ペレットを再中断します。1,000 x gで3分間遠心分離機。上清を捨てます。
- 手順 2.2.4 を繰り返します。細胞が遠心分離している間、39.52%ポリエチレングリコール、0.12 M LiOAcおよび1.2xトリス-EDTAバッファー(12 mMトリス-HClおよび1.2 mM EDTA)の溶液の少なくとも120 mLを調製する。氷の上に保管してください。
- 形質転換のためにプラスミドDNAを調用するために、まず100°Cでサケ精子DNAの4 mLを5分間沸騰させ、すぐに氷の上で5分間冷却する。次いで、pJR487の20mL(セクション1で得られた)を538ng/μLの濃度で混合し、24mLの総体積に対して冷却されたサケ精子DNAの4mLと混合する。使用するまで氷の上に保管してください。
- 各細胞ペレットの上にサケ精子DNAと混合プラスミドDNAの600 μLを追加します。まだ再中断しないでください。
- ステップ2.2.5で作られたPEG-LiOAc-TE溶液の3 mLを各ペレットに追加します。上下にピペッティングし、渦を巻くことによってペレットを再中断します。
- 室温で10分間各チューブをインキュベートします。
- 39°Cに設定された水浴で26分間、各チューブにヒートショックを与えます。
注:数分ごとに、各チューブを反転して、細胞がチューブの底部に沈着するのを防ぎます。 - 1,000 x gで3分間各チューブを遠心分離します。上清を廃棄し、渦によってYPDの10 mLで各ペレットを再懸濁する。新しいガラスフラスコにすべての20チューブを組み合わせます。細胞の総体積は〜200 mLでなければなりません。
- 66.6 mLのセルを新しい1Lガラスフラスコに移し、液体YPDで500mLの体積まで持ち上げます。形質転換細胞の200 mL全体を使用するには、さらに2回繰り返します。新しい 500 mL 培養ごとに OD600を測定します (OD600 ~0.35-4 を期待します)。
- 3つのフラスコを28°Cで2時間振り、200rpmで回復(<1世代)。
- 3つのフラスコのそれぞれに300mg/mL G418の0.5 mLを加え、最終的な濃度300 μg/mL G418に加え、28°C、200rpmで振り返ります。
注: この手順の前に、変換されたハイブリッド セルは変換から回復しています。G418の添加に伴い、プラスミドpJR487の存在が選択される。形質転換中にプラスミドを取り上げなかった細胞は死に始める。 - 残りの 20 本の円錐形のチューブで、手順 2.2.2 ~ 2.2.14 を繰り返します。この時点で 6 つの 1 L ガラスフラスコがあり、それぞれに G418 が追加された 500 mL のセルがあります。
- 28°Cで細胞の6つのフラスコをすべてインキュベートし、200rpmで振り、約2日間、または各フラスコで約2.3のOD600に達するまで。6 つのフラスコをすべて組み合わせて、単一のカルチャを作成します。
注: このカルチャ内のすべてのセルは下流のステップでは使用されませんが、このような大容量を使用する目的は、できるだけ多くの固有の変換イベントを作成し、それらをすべてプールすることによって、単一の変換全体のバイアスを正規化することです。一緒に。 - 2.2.16 で作成されたカルチャを使用して、YPD + G418 (300 μg/mL) の 500 mL で 2 つの新しい 1 L フラスコを 0.2 の OD600に接種します。廃棄できる残りカルチャがあります。
- 28°Cで1Lフラスコを一晩でインキュベートし、200rpmで振り、それぞれが〜2.2(〜3.5世代)のOD600に達するまで。両方のカルチャを単一のカルチャに結合し、結合されたカルチャの OD600を再度測定します。
注:この時点で、培養物は、プラスミドpJR487を収容する細胞からほぼ完全に構成されるべきである。細胞の集団の一部では、PiggyBacトランスポゾンはプラスミドからプラスミドを発現するトランスポザーゼによってプラスミドからゲノムに転置されるであろう。しかし、トランスポザーゼの発現を継続すると、選択の過程で転置が起こし、遺伝子型と表現型の関係が不明瞭になります。次のいくつかのステップの目的は、プラスミドの存在に対してカウンターセレクションを実行し、トランスポザーゼの発現がこれ以上ないことを確実にすることである。得られたプールは、トランスポゾンをゲノムに統合する有無にかかわらず細胞の混合物であるが、その後のマッピングステップ中にトランスポゾンを含む細胞のみが検出される。トランスポザーゼが発現する変換の時間は、プラスミド符号化が失われる前に、変異後の所定のクローンが複数のトランスポジソン挿入を持つ可能性を支配し得る。一度に1つの遺伝子の分析で「二次的」変異として現れるこれらの頻度は、変異発生後に定義されたコロニー数を配列し、そのDNAを組み合わせ、独立した挿入数を配列することによって推定することができる。プール内の位置。 - 1,000 x gで3分間、この培養の遠心分離機25 mL。25 mL にあるセルの合計 OD600単位の数を計算します (下の計算例を参照)。上清を廃棄し、渦によって1.85 OD600/mLの細胞懸濁液を作成するのに十分なH2 Oで再懸濁する。
注: 結合培養の OD600が 2.2 であった場合の水中の細胞の再懸濁の計算例: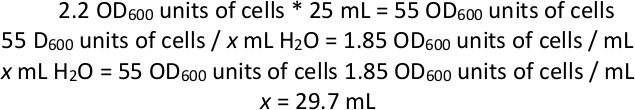
そこで、細胞培養の25mLを紡糸し、上清を廃棄した後、細胞と水の総体積を最大29.7mLまで持ち込むのに十分なH2Oを細胞に加える(細胞ペレットも体積を持つため、H2Oの29.7mL未満を添加する)。 - ガラスビーズを使用して、5-FOAを持つ12の大きな正方形の完全な人工寒天プレートのそれぞれに水中に再懸濁した細胞のプレート1 mL。各プレートを28°Cで1~2日間、またはプレート上に芝生が形成されるまでインキュベートします。
- 小さな無菌スクイージを使用して、6枚のプレートのそれぞれから細胞をこすり落とし、35 mLの無菌水を含むチューブに入れます。他の6枚のプレートで、細胞と水の合計2本のチューブを繰り返します。すべてのセルサスペンションを1つのチューブに組み合わせます。水をブランクとして使用して、この懸濁液のOD600を測定します。OD600/mLの細胞濃度を水で44.4 OD600単位/mLにします。私たちの経験では、転置効率(URA-であるKAN+細胞の割合)は平均50%です。
- 保存するセルの-80 °C冷凍庫ストックの数を決定します。各アリコートは、将来的に単一の実験に使用することができます。
注: プールの生成にかかる時間を考えると、偶発的な誤用や反復実験の実行時に複数のバイアルを保管してください。20~30株は妥当な数です。 - 各冷凍庫のストックは10%DMSOの1 mLの細胞の40 OD600単位を含んでいる。DMSOの100 μLに900 μLの細胞を加えます。作成されたフリーザーストックの合計数に対して繰り返します。将来の使用のために-80 °Cでそれぞれを保管してください。
3. プール形式での相互ヘミジゴットの選択
- -80°C冷凍庫から、室温でセクション2からプールされた相互ヘミジゴットの単一のアリコートを解凍する。
注:アリコートが解凍したら室温で長時間座らせないようにし、すぐに使用してください。 - 1 mL アリコート全体を使用して、250 mL のガラスフラスコに 150 mL の液体 YPD を接種します。この培養物のOD600を測定し、次いで28°Cでインキュベートし、200rpmで振り、〜7時間、または培養が2〜3個の集団倍増を経るまで。この時点で、培養は選択中の培養物を接種するために使用される準備ができています。
注: 計算例: 元のフラスコの OD600が 0.25 を測定する場合は、少なくとも 1.0 の OD600に達するまでカルチャをインキュベートします。「タイムゼロ」(T-0)でサンプルポイントが望まれる場合、選択前にヘミジゴテ集団を調べるための方法として、ペレット当たりの培養量を1,000xgで3分間遠心分離し、上清を廃棄し、 -80 °C. - 成長したヘミジゴットプールを使用して、高温(39°C)と許容温度(28°C)の両方で、適切なレプリケートスキームで選択するための培養物を接種します。少なくとも、各温度で3つの生物学的複製選択培養物を、合計6つの選択培養物に設定する。
- 液体YPDが付いている2 Lガラスフラスコの合計500 mLの各選択培養を作成し、0.02のOD600に接種する。各選択培養物を28°Cまたは39°Cのいずれかで100rpmで振り、6~7個の集団倍増が起こった(~1.28-2.56のOD600に相当)。すべての選択カルチャの最終的な OD600を可能な限り密接に一致するようにしてください。
注:28 °Cの選択培養物は、39°Cの選択培養物よりも速く成長します。その結果、39°Cの選択培養物は、インキュベーターでより長い期間を過ごします。インキュベーターで費やされた合計時間数に関係なく、準備が整った各フラスコで次の手順を進めます。私たちの経験では、28°Cまたは39°Cの培養物は、それぞれ〜12または〜18時間かかり、約2.0のODに達しました。長い選択は、小さなフィットネス効果を増幅する利点を持つことができますが、また、任意の1つの遺伝子/対立遺伝子のトランスポゾン変異体間のフィットネスの最終的な分布にノイズを導入するde novoの背景変異が発生することを可能にします。そのため、RH-seq実験では選択時間を制限することが重要です。
- 液体YPDが付いている2 Lガラスフラスコの合計500 mLの各選択培養を作成し、0.02のOD600に接種する。各選択培養物を28°Cまたは39°Cのいずれかで100rpmで振り、6~7個の集団倍増が起こった(~1.28-2.56のOD600に相当)。すべての選択カルチャの最終的な OD600を可能な限り密接に一致するようにしてください。
- 各選択培養から細胞ペレットを収穫する。ライブラリの準備とシーケンシングのための技術的な複製として、各選択培養物からこのボリュームの少なくとも4つのペレットから、1,000 x gで7 OD600単位の細胞と遠心分離機を得るために必要な体積を計算します(セクション4および5を参照)。、以下)。上清を捨て、-80°Cで保管してください。
注: 選択フラスコの最終 OD600が 2.0 の場合の例: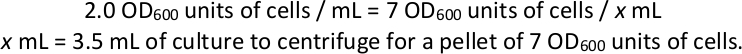
4. Tn-seqライブラリ構築とイルミナシーケンシングにより、トランスポゾン変異型ヘミジゴットの豊富さを判定
- シークエンスされるセクション3から各細胞ペレットを氷上で融解する。
- メーカーの指示に従って酵母gDNA精製キットを使用して、各細胞ペレットから総ゲノムDNA(gDNA)を分離します。65°Cに温めた溶出バッファーの50μLでDNAを再中断します。
- フッ素計を用いて各ペレットからのgDNA量を定量します。次の手順を使用して Tn-seq の次世代シーケンシング (NGS) ライブラリーを作成するために各セル ペレットに必要な gDNA の最小総量は 1 μg です。
注: ライブラリの作成には 1 μg 未満の gDNA を使用できますが、ライブラリの最終的な量と品質は損なわれます。 - Tn-seq ライブラリ7を作成するための確立されたプロトコルに従ってください。このプロトコルに固有の次の関連情報に注意してください。
- gDNAせん断、修理およびアダプタライゲーションを終了した後、PCRを介してトランスポゾンを含むgDNAを増幅します。この PCR では、PiggyBac トランスポゾンアダプターと NGS アダプターに固有の次のフォワード プライマーとリバース プライマーをそれぞれ使用します。
フォワード (N – ランダムヌクレオチド)
5' ATGATACCGACCACCGAGATCTACCTCTTCTACACGACG
CTTCCGATCTNNNNNNNナグナガットカアットカアガガットGCGTCAATCTCAATCTCAATCTCAATAGCGTCAAT3'
リバース(Nsのストレッチは、多重化に使用される一意の6bpインデックスを表します。インデックスの詳細については、以下を参照してください)
5' CAAGCAGAAGAGGGGAGAGAGAGGAGGAGATNNNNNNGTGACTGGAGTCAG
ACGTGTGTGCTCTTCCGCT 3' - サイズ選択ビーズを含む付属のクリーンアップ手順を使用して、最終的なライブラリ内のクローンフラグメントの比率を最小限に抑えます。
注: 選択培養に関するこれまでの最小反復要件に従った結果、シーケンス用に 24 個の gDNA サンプルが存在します。シーケンスの現在のコストを考えると、各サンプルが単独で実行されることはほとんどありません。同じレーン上のサンプルを組み合わせるには、複数のリバースプライマーを作成し、それぞれに固有の 6 ベース ペア インデックスを作成します。異なるインデックスを持つサンプルは、同じシーケンス レーンに結合し、その後計算で分離できます。
- gDNAせん断、修理およびアダプタライゲーションを終了した後、PCRを介してトランスポゾンを含むgDNAを増幅します。この PCR では、PiggyBac トランスポゾンアダプターと NGS アダプターに固有の次のフォワード プライマーとリバース プライマーをそれぞれ使用します。
- 8 レーンにわたる NGS テクノロジを使用して、各ライブラリからシーケンス シングルエンド 150 bp 読み取りします。
注:必要なシーケンシング読み取りの量は、前のステップで調製したライブラリの品質に大きく依存します(すなわち、相互ヘミジゴット由来のDNAを表すトランスポゾンDNAを実際に含むライブラリ内のDNAの割合)。これに寄与する主な要因は2つあります。第 1 に、統合トランスポゾンのないセルはプール作成中に対して逆選択されないため、各培養はトランスポゾンの有無にかかわらずセルの組み合わせになります。第二に、トランスポゾン含有逆半血のゲノム内であっても、ゲノムのほとんどは配列を含むトランスポゾンではなく、このgDNAはやむを得ずライブラリー製剤の一部となる。トランスポゾン含有DNAの最終的なPCR増幅の目的は、これら2つのバックグラウンドgDNA源に対するトランスポゾン含有DNAの比率を増加させる。この増幅がより効率的であるほど、読み取りの割合が高いほど、ダウンストリーム解析に使用できるようになります。ライブラリの品質が低いほど、読み取りの割合が増えるほどトランスポゾン DNA は含まれないため、より多くのシーケンシングを行う必要があります。上記の制約を考えると、8レーンのシーケンシングは、相互ヘミジゴットの豊富さを合理的な程度に追跡することができる。シーケンシングを増やすと、より深い分析が可能になります。
5. トランスポゾン挿入とRH-seq解析の位置のマッピング
注: 次のデータ分析は、カスタム Python スクリプト (https://github.com/weiss19/rh-seq でオンラインで見つかった) で行われましたが、他のスクリプト言語を使用してやり取りできます。以下に、プロセスの主要な手順の概要を示します。個々のレプリケート読み取りファイルを組み合わせることに注意しない限り、次の手順を実行します。
- アダプターのシーケンスを読み取りから除外し、インデックスに従って各反復の読み取り値を分離します。
- トランスポゾンゲノム接点を含む読み取りを検索します。これを実現するには、トランスポゾン CAGACTATCTTTTTagGGTTAA の最後の 20 個の基本ペアを各読み取り内で検索します。このシーケンスを含まないすべての読み取りは破棄します。
注:私たちの経験では、トランスポゾンの終わりにマッピングする読み取りの割合は83-95%です。 - トランスポゾンの3'終端の下流のシーケンスのみを含むトランスポゾン含有読み取りをトリミングします。この配列を酵母ゲノムにマッピングすることにより、読み取りごとにトランスポジット挿入のゲノムコンテキストを決定します(下図5.4)。
- BLAT または同等のマッピング ツールを使用して、トランスポゾンの下流のシーケンスを S. cerevisiae DBVPG1373 x S.パラドカスZ1 ハイブリッド ゲノムにマッピングします (スクリプト名: map_and_pool_BLAT.py)。
- トランスポゾンの3'終端の下流で使用可能なシーケンスの50未満の基本ペアがある読み取りを破棄します。短いシーケンスは、一意にマップするのが困難です。
- BLAT を使用する場合は、ID = 95、タイル サイズ = 12 というパラメーターを使用します。
- S. cerevisiae S288c および S. パラドックス CBS432 の参照ゲノムの最新バージョンを連結してマッピングに使用する基本的なハイブリッドゲノムを作成します。
注:ハイブリッドゲノム全体の個々の遺伝子のゲノム境界を説明する基本的な注釈ファイルは、上記のGithubリポジトリで見つけることができます(ファイル名:YS2+CBS432+plasmid_clean)。ハイブリッドゲノム内の単一の場所にマップする読み取りのみを使用します(つまり、S. cerevisiae または S. パラドックスのいずれかに固有です)。ゲノム全体の挿入イベントの均一な頻度が期待されます。ゲノム間の挿入位置の分布は、他の場所で報告されています 3.
- 一意のトランスポゾン挿入位置にマッピングされる読み取りの合計数を集計し、1 つのトランスポジット挿入変異クローンのセルから発生したと推測します。1 つのライブラリーからのすべてのこのような値の合計は、そのライブラリーのマップされた読み取りの合計数と呼ばれます。
- 互いに 3 つの基本ペア内に複数の挿入マッピングがある場合は、これらすべてを 1 つの挿入ポイントに結合し、すべての読み取り値を最も高い読み取り数を持つ単一の場所に割り当てります。この値ninsertは、gDNAが配列された細胞ペレット内の挿入クローンの豊富さを表す。この時点で、nインサートのリストがあり、それぞれが一意のマッピングトランスポジット挿入の豊富さ、シーケンスされたセルペレットごとに1つのリストがあります。
注:PiggyBacトランスポゾンは、ゲノム内のTTAA配列、4塩基配列に挿入します。したがって、互いの 3 つの基本ペア内の挿入マッピングは、同じ TTAA サイトから発生している必要があると推測します。 - シーケンスされた各ライブラリからの合計読み取りの数は若干異なるので、比較する場合は、すべてのファイル間で n挿入値を正規化します。この操作は、個々のライブラリ (nペレット)からマップされた読み取りの合計数を表し、すべてのライブラリ 、ペレット>のすべての nペレットの平均を取ります。個々のライブラリのデータ内の各nインサートにペレット>/nペレットの比率を掛け、インサートを計算し、所定のトランスポゾン挿入クローンの正規化された豊富さを計算します。
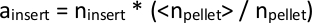
または、DESeq28 (スクリプト名: total_reads_and_normalize.py) などの利用可能なツールを使用して、ライブラリサイズを推定することもできます。 - すべてのライブラリにマップされたすべての挿入のセットを集計します。一部のライブラリで見つかったが、他のライブラリでは見つからない挿入の場合は、下流の計算にinsert = 1 を設定します。
- 読み取りをフィルタリングして、アニテーション ファイルに従って遺伝子内に含まれる挿入を見つけます (スクリプト名: remove_NC_and_plasmid_inserts.py)。
- 一意の挿入ごとに、各選択の技術的な反復 (各カルチャは 28 °C または 39 °C)、インサート>テクニカル(スクリプト名: combine_tech_reps_V2.py) を計算します。
- 一意の挿入ごとに、各温度ですべての insert>技術的な平均を取ることによって、各温度の生物学的反復全体の平均量を計算します。同時に、挿入ごとに変動係数を計算し、CV挿入、 インサート>テクニカル(スクリプト名: combine_bio_reps.py)全体を計算します。
注:この時点で、各温度、28°Cおよび39°Cについて、固有のトランスポジット挿入、それらの平均豊富さと各生物学的反復間の変動係数のリストがあります。私たちの実験のためのこれらのデータは、他の場所で報告されています 3. - 28 °C または 39 °C、挿入>合計> 1.1、および CV挿入、合計≤ 1.5 (スクリプト名: filter_insert.py) を持つもののすべての挿入のリストをフィルター処理します。
- 一意の挿入ごとに、ログ2(<挿入>合計,28 °C / インサーt>合計,39 °C)を計算します。この値は、特定のトランスポゾン挿入変異クローンの「熱耐性」を表します(スクリプト名: fitness_ratios.py)。
- 遺伝子と対立遺伝子(S.セレビシエまたはS.パラドックス)によってユニークな挿入物をすべてソートし、各対立遺伝子の挿入数を集計する。各対立遺伝子に少なくとも5個の挿入を持つ遺伝子のみが分析できるように遺伝子をフィルタリングします(スクリプト名:organize_and_filter_genes.py)。
注:各対立遺伝子に対する複数のユニークな挿入は、その相互ヘミジゴットの熱耐性のより正確な尺度を可能にします。対立遺伝子あたりに必要な挿入数を減らすことは可能ですが、この測定の精度を損ない、より多くの遺伝子をテストできるようにすることで、複数の検査負担を増大させます。さらに、対立遺伝子あたりの挿入数が少なすぎる遺伝子をフィルタリングすると、非常に異なる表現型を付与する二次部位変異を抱える個々のヘミジゴットクローンの試験結果への影響を軽減するのに役立ちます。 - 上記のフィルタリング後にデータセットに残っている各遺伝子について、S.セレビシアの全挿入物の耐熱性(対数2比)を、マン・ホイットニーU検定を用いたS.パラドックス対立遺伝子のサーチ耐性(log2比)と比較する。あるいは、回帰モデルを実装し、DESeq28 (スクリプト名: mann_whitney_u.py) から調整することもできます。
- Benjamini-Hochberg法を用いた複数のテストのp値を補正する。
- 有意なp値を持つ遺伝子(例えば、≤ 0.01)は、2つの種間の熱耐性の違いに重要な遺伝子の候補である。
結果
我々はS.セレビシエとS.パラドックスを交配して無菌ハイブリッドを形成し、トランスポゾン変異形成を行った。各変異型クローンは、1つの遺伝子の1つの対立遺伝子が破壊されるヒプロイドハイブリッドであるヘミジゴットであった(図1A、図2)。我々は39°Cでの成長によって互いにヘミツゴテスを競い合い、対照として別?...
ディスカッション
以前の統計的遺伝的方法に比べてRH-seqの利点は、数倍です。リンケージおよびアソシエーション分析とは対照的に、RH-seqは単一遺伝子マッピング分解能を提供します。したがって、特定の種の個体間の形質変動の研究や、特異的な違いについても重要な有用性を持つ可能性が高い。また、ゲノム全体の相互異端異端分析における以前の試みは、遺伝子欠失変異体のコレクションを使用し、そ?...
開示事項
著者は何も開示していない。
謝辞
J.ループ、R.ハックリー、I.グリゴリエフ、A.アーキン、J.スカーカーが元の研究に貢献してくれたことに感謝します。B.ブラックマン、S.コラデッティ、A.フラムホルツ、V.グアッチ、D.コシュランド、C.ネルソン、A.サシクマールがディスカッションを行いました。また、PiggyBacプラスミドのJ.デュエバー(UCバークレー大学バイオエンジニアリング学科)に感謝します。この研究は、R01 GM120430-A1と、米国エネルギー共同ゲノム研究所(DOE科学ユーザー施設)のRBBへのコミュニティシーケンシングプロジェクト1460によってサポートされました。後者によって行われた作業は、契約No.の米国エネルギー省科学局によってサポートされました。DE-AC02-05CH11231。
資料
| Name | Company | Catalog Number | Comments |
| 1-2 plasmid Gigaprep kits | Zymo Research | D4204 | The number of kits required depends on how efficient your preps are in each kit. This kit comes with 5 individual plasmid prep columns. Run 1 L of saturated E. coli culture through each prep column, as using more than 1 L per column can cause clogging of the prep filter, leading to low yield and poor quality DNA. |
| 10X Tris-EDTA (TE) buffer (100 mM Tris-HCl and 10 mM EDTA) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 1M LiOAc | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| 300 mg/mL Geneticin (G418) | Gibco | 11811023 | |
| 52% polyethylene glycol (PEG) 3350 | Sigma | 1546547 | Dissolve in water and filter sterilize through a 0.22 μm filter before use. 1X trafo mix: 228 uL 52% PEG, 36 uL 1M LiOAc, 36 uL 10X TE buffer |
| Autoclaved LB liquid broth | BD Difco | 244620 | Make LB liquid broth using your powder from any brand, and milliQ water. Autoclave it before use. |
| Carbenicillin stock in water (100 mg/mL) | Any | N/A | Filter sterilize through a 0.22 μm filter before use. |
| Complete synthetic agar plates (24.1cm x 24.1cm) with 5-fluoroorotic acid (5-FOA) [0.2% drop-out amino acid mix without uracil or yeast nitrogen base (YNB), 0.005% uracil , 2% D-glucose, 0.67% YNB without amino acids, 0.075% 5-FOA] | 5-FOA: Zymo Research, Drop-out mix: US Biological, Uracil: Sigma, D-glucose: Sigm), YNB: Difco | 5-FOA: F9001-5, Drop-out mix: D9535, Uracil: U0750, D-glucose: G8270, YNB: DF0919 | |
| DMSO | Any | N/A | |
| E. coli strain carrying pJR487 (CEN-/ARS+ piggyBac-containing plasmid) | N/A | N/A | Request from Brem lab. |
| Hybrid yeast strain JR507 (S. cerevisiae DBVPG1373 x S. paradoxus Z1, URA-/URA-) | N/A | N/A | Request from Brem lab. |
| Illumina Hiseq 2500 | used for SE-150 reads | ||
| Large shaking incubators with variable temperature settings | Any | N/A | |
| LB + carbenicillin agar plates (100 μg/mL) | Agar: BD Difco | Agar: 214010 | Make LB agar plates as normal and add carbenicillin to 100 μg/mL before drying. |
| Nanodrop spectrophotometer | Thermo Scientific | ND-2000 | |
| Qubit Fluorimeter | Thermo Scientific | Q33240 | |
| Salmon sperm DNA | Invitrogen | 15632011 | |
| Water bath at 39°C | Any | N/A | |
| Yeast fungal gDNA prep kit | Zymo Research | D6005 | |
| Yeast peptone dextrose (YPD) liquid media | BD Difco | Peptone: 211677, Yeast Extract: 212750 | Add filter-sterilized D-glucose to 2% after autoclaving. |
| YPD + G418 agar plates (300 μg/mL) | Agar: BD Difco | Agar: 214010 | Make YPD agar plates as normal and add G418 to 300 μg/mL before drying. |
| YPD agar plates | Agar: BD Difco | Agar: 214010 |
参考文献
- Flint, J., Mott, R. Finding the molecular basis of quantitative traits: successes and pitfalls. Nature Reviews Genetics. 2, 437-445 (2001).
- Allen Orr, H. The genetics of species differences. Trends in Ecology and Evolution. 16, 343-350 (2001).
- Weiss, C. V., et al. Genetic dissection of interspecific differences in yeast thermotolerance. Nature Genetics. 50, 1501-1504 (2018).
- Stern, D. L. Identification of loci that cause phenotypic variation in diverse species with the reciprocal hemizygosity test. Trends in Genetics. 30, 547-554 (2014).
- Steinmetz, L. M., et al. Dissecting the architecture of a quantitative trait locus in yeast. Nature. 416, 326-330 (2002).
- Scannell, D. R., et al. The Awesome Power of Yeast Evolutionary Genetics: New Genome Sequences and Strain Resources for the Saccharomyces sensu stricto Genus. G3 (Bethesda). 1, 11-25 (2011).
- Wetmore, K. M., et al. Rapid quantification of mutant fitness in diverse bacteria by sequencing randomly bar-coded transposons. MBio. 6, e00306-e00315 (2015).
- Love, M. I., Huber, W., Anders, S. Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biology. 15, 550 (2014).
- Wilkening, S., et al. An evaluation of high-throughput approaches to QTL mapping in Saccharomyces cerevisiae. Genetics. 196, 853-865 (2014).
- Kim, H. S., Huh, J., Riles, L., Reyes, A., Fay, J. C. A noncomplementation screen for quantitative trait alleles in saccharomyces cerevisiae. G3 (Bethesda). 2, 753-760 (2012).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved