このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
金属イオン認識の構造とメカニズムを決定するイオン移動質量分析技術と金属結合オリゴペプチドの酸化還元活性
要約
イオンモビリティ質量分析および分子モデリング技術は、設計された金属結合ペプチドおよび銅結合ペプチドメタノバクチンの選択的金属キレート性能を特徴付けることができる。金属キレートペプチドの新しいクラスを開発することは、金属イオンの不均衡に関連する疾患の治療につながるのに役立ちます。
要約
エレクトロスプレーイオン化(ESI)は、質量、全体的な電荷、金属結合相互作用、および立体形状を節約しながら、水相ペプチドまたはペプチド複合体をガス相に移すことができる。ESIとイオンモビリティ質量分析(IM-MS)の結合は、その状態測定、プロトネーション状態に関連するペプチドの質量対電荷(m/z)および衝突断面(CCS)の同時測定を可能にする器械的な技術を提供し、そして立体形状。ペプチド複合体の全体的な電荷は、1)ペプチドの酸性および塩基部位および2)金属イオンの酸化状態のプロトン化によって制御される。したがって、複合体の全体的な電荷状態は、ペプチド金属イオン結合親和性に影響を与える溶液のpHの関数である。ESI-IM-MS分析では、ペプチドおよび金属イオン溶液は水性のみの溶液から調製され、pHは希希水性酢酸または水酸化アンモニウムで調整されます。これにより、pH依存性および金属イオン選択性を特定のペプチドに対して決定することができる。さらに、ペプチド複合体のm/zおよびCCSは、B3LYP/LanL2DZ分子モデリングと共に、複合体の金属イオン協調および三次構造の結合部位を識別するために使用することができる。結果は、ESI-IM-MSが一連の代替メタノバクチンペプチドの選択的キレート性能を特徴付け、銅結合ペプチドメタノバクチンと比較する方法を示した。
概要
銅イオンと亜鉛イオンは、生物に不可欠であり、酸化保護、組織成長、呼吸、コレステロール、グルコース代謝、ゲノム読み取り1を含むプロセスに不可欠です。これらの機能を有効にするために、シスのチオレート、彼の2、3のイミダゾール、(よりまれに)メチオニンのチオエーテル、およびGluとAspのカルボキシレートは、選択的に金属を活性化部位に組み込む。メタロエンザイム。これらのコーディネーショングループの類似性は、HisおよびCysリガンドが正しい機能を確保するためにCu(I/II)またはZn(II)のいずれかを選択的に組み込む方法に関する興味深い疑問を提起する。
選択的結合は、多くの場合、Zn(II)またはCu(I/II)イオン濃度4を制御するペプチドの獲得および密売によって達成される。Cu(I/II)は反応性が高く、酵素に酸化的損傷や急激な結合を引き起こすため、その自由濃度は、細胞内の様々な場所に安全に輸送する銅シャペロンおよび銅調節タンパク質によって厳しく調節される。その恒常主義を制御する 5,6.銅代謝または恒常性の破壊は、メンケスおよびウィルソン病7ならびに癌7およびプリオン8およびアルツハイマー病9などの神経疾患に直接関与する。
ウィルソン病は、目、肝臓、脳のセクションの銅レベルの増加に関連しており、Cu(I/II)の酸化還元反応が活性酸素種を産生し、肝無胞および神経変性を引き起こす。既存のキレート療法は、小さなチオールアミノ酸ペニシラミンおよびトリエチレネテトラミンである。あるいは、メタノトロフィー系銅獲得ペプチドメタノバクチン(mb)10、11はCu(I)12に対する高い結合親和性のために治療可能性を示す。メチロシヌス・トリコスポリウムOB3bのメタノバクチン(mb-OB3b)がウィルソン病の動物モデルで研究された場合、銅は肝臓から効率的に除去され、胆汁13を通して排泄された。インビトロ実験では、mb-OB3bが肝臓サイトゾル13に含まれる銅メタロチオネインから銅をキレートできることを確認した。レーザーアブレーション誘導結合血漿質量分析イメージング技術は、ウィルソン病肝サンプル14、15、16における銅の空間分布を調査し、mb-OB3bであることを示した。わずか8日17の短い処理期間で銅を除去します。
mb-OB3bはまた、Ag(I)、Au(III)、Pb(II)、Mn(II)、Co(II)、Fe(II)、Ni(II)、およびZn(II)18、19を含む他の金属イオンと結合する。生理的Cu(I)結合部位に対する競争は、mb-OB3b複合体からCu(I)を置き換えることができるためAg(I)によって示され、Ag(I)およびNi(II)の両方もCu(I)19によって置き換えることができないMbへの不可逆的な結合を示す。近年、2His-2Cys結合モチーフを有する一連の代替メタノバクチン(amb)オリゴペプチドが20、21、及びそのZn(II)およびCu(I/II)結合特性を特徴とする。彼らの一次アミノ酸配列は類似しており、それらはすべて2His-2Cysモチーフ、Proおよびアセチル化N終産を含有する。2His-2Cysモチーフはmb-OB3bの2つのエネチオールオキサゾロン結合部位を置き換えるため、主にmb-OB3bとは異なります。
イオンモビリティ質量分析(ESI-IM-MS)と組み合わせたエレクトロスプレーイオン化は、質量電荷(m/z)と衝突を測定するため、ペプチドの金属結合特性を決定するための強力な計測器技術を提供します。断面(CCS)を使用しながら、溶液相から質量、電荷、立体形状を保存します。m/zおよびCCSは、ペプチオメトリー、プロトネーション状態、および立体構造形状に関連する。種に存在する各要素の同一性および数が明示的に同定されるため、ストイチオメトリーが決定される。ペプチド複合体の全体的な電荷は、酸性および塩基部位の原発状態および金属イオンの酸化状態に関する。CCSは、複合体の三次構造に関連する回転平均サイズを測定するので、ペプチド複合体の立体形状の情報を提供する。複合体の全体的な電荷状態はまた、pHの機能であり、カルボキシル、His、CysおよびTyrなどのデプロトン化された基本部位または酸性部位が金属イオンの潜在的な結合部位でもあるため、ペプチドの金属イオン結合親和性に影響を与える。分析のために、ペプチドおよび金属イオンは希希水性酢酸または水酸化アンモニウムによって調整されたpHと水溶液で調製される。これにより、pH依存性および金属イオン選択性がペプチドに対して決定され始める。さらに、ESI-IM-MSによって決定されたm/zおよびCCSをB3LYP/LanL2DZ分子モデリングと共に使用して、複合体の金属イオン協調および三次構造の種類を発見することができる。この記事に示す結果は、ESI-IM-MSが一連のアンブペプチドの選択的キレート性能を特徴付け、銅結合ペプチドmb-OB3bと比較する方法を明らかにする。
プロトコル
1. 試薬の調製
- 培養メチロシヌストリコスポリウムOB3bは、Cu(I)フリーmb-OB3b 18、22、23を分離し、使用するまで-80°Cで試料を凍結乾燥し、保存する。
- アンブペプチド(アンブ1、アンブ2、アンブ4、>70%純度)を合成し、サンプルを凍結乾燥させ、使用するまで-80°Cで保存します。
- 購入>98%純化マンガン(II)塩化物、コバルト(II)塩化物、塩化物(II)塩化物、銅(II)塩化物、銅(II)硝酸塩、銀(I)硝酸塩、亜鉛(II)塩化物、鉄(III)塩化物および鉛(II)。
- アンブ種およびHPLCグレード以上の水酸化アンモニウム、氷河酢酸、アセトニトリルの衝突断面を測定するためのキャリブラントとして使用されるポリDL-アラニンポリマーを購入します。
2. ストックソリューションの準備
-
ペプチドストックソリューション
- 正確に重量を量り、少なくとも3つの有意な数字を使用して、mb-OB3bまたは1.7 mLプラスチックバイアル中のmb-OB3bまたはambの質量の質量。
注:計量質量は、ペプチドの溶解度に応じて、1.00 mLの脱イオン化(DI)水が加えられた場合に、12.5 mMまたは1.25 mMのいずれかを得るべきである。 - ピペを使用して、計量されたペプチドサンプルに1.00 mLの脱イオン水(>17.8 MΩ cm)を加え、12.5 mMまたは1.25 mM溶液を得ます。キャップをしっかりと置き、少なくとも20回の反転と十分に混ぜます。
- マイクロピペットを使用して、ペプチドサンプルから個別にラベル付けされた1.5 mLバイアルに50.0 μLアリコートを分配し、使用するまで-80°Cに保存します。
- 正確に重量を量り、少なくとも3つの有意な数字を使用して、mb-OB3bまたは1.7 mLプラスチックバイアル中のmb-OB3bまたはambの質量の質量。
-
金属イオンストックソリューション
- 正確に重量を量り、少なくとも3つの有意な数字を使用して、1.7 mLバイアル中の金属塩化物または硝酸銀の10.0〜30.0mgの質量を用いて。
注:DI水の1.00 mLを添加すると、計量質量は125 mMを得るべきです。 - 1.7 mLバイアルの計量金属サンプルに1.00mLのDI水を加え、125mM溶液を得ます。キャップをしっかりと置き、少なくとも20回の反転と十分に混ぜます。
- 正確に重量を量り、少なくとも3つの有意な数字を使用して、1.7 mLバイアル中の金属塩化物または硝酸銀の10.0〜30.0mgの質量を用いて。
- 水酸化アンモニウムストック溶液:DI水で99.5%酢酸溶液の57 μLを1.00mLの最終体積に希釈することにより、1.0M酢酸溶液を調剤します。DI水で21%水酸化アンモニウム溶液の90μLを1.00mLの最終体積に希釈することにより、水酸化アンモニウム溶液を1.0Mmmmmmmに調作します。1.0M溶液の100 μLを取り、0.10 Mおよび0.010 M酢酸および水酸化アンモニウム溶液を調作することにより、各溶液の2つの連続した希釈を行います。
- ポリDL-アラニンストック溶液:1.0mgのPAを計量し、1.0mLのDI水に溶解して1,000ppmを与えることによってポリ-DL-アラニン(PA)を調製する。よく混ぜる。マイクロピペを使用して、50.0 μLアリコートを分配し、それぞれを1.7 mLバイアルに入れ、-80°Cに保存します。
3. エレクトロスプレーイオン移動質量分析
- 0.1Mの氷河酢酸、0.1Mの水酸化アンモニウム、そして最後にDI水の約500 μLでESI入口チューブと針毛細血管を徹底的に洗浄します。
- 1,000 ppm PAストック溶液の50.0 μLアリコートを解凍し、450 μLのDI水で希釈して100ppm PAを与えます。この溶液のピペット100.0 μLを1.00mLに希釈し、500μLのDI水と500μLのアセトニトリルを用いて10ppm PA溶液を与える。
- 議論セクションで説明するように、ネイティブESI-IM-MS条件を使用して、それぞれ10分間10ppm PA溶液の負および正イオンIM-MSスペクトルを収集します。
- 12.5 mMまたは1.25 mMアンブストック溶液の50.0 μLアリコートを解凍し、0.125 mMアンブの最終濃度を与えるためにDI水で連続的な希釈を行います。各希釈を十分に混ぜます。
- 125mM金属イオンストック溶液のピペ100.0 μLを、1.7mLバイアルに入れ、DI水で1.00mLに希釈し、12.5mMの金属イオンを与える。さらに2つの連続した希釈を繰り返し、最終的な0.125 mMの金属イオン濃度を与えます。各希釈を十分に混ぜます。
- 0.125 mMアンブのピペ200.0 μLを1.7mLバイアルに入れ、500μLのDI水で希釈し、溶液を十分に混合します。
- 1.0M酢酸溶液の50 μLを加えてサンプルのpHを3.0に調整します。
- pH調整サンプルに0.125 mM金属イオンの200.0 μLを追加します。DI水を加えて、サンプルの最終容積1.00mLを得て、十分に混合し、試料をRTで10分間平衡させます。
- 鈍い鼻注射器を使用して、サンプルの500 μLを取り、それぞれ5分間の負および正イオンES-IM-MSスペクトルを収集します。サンプルの残りの500 μLを使用して、校正されたマイクロpH電極を使用して最終pHを記録します。
- 手順 3.6 ~ 3.9 を繰り返し、手順 3.7 を変更して pH を 4.0、5.0、6.0、7.0、8.0、9.0、または 10.0 に調整しながら、0.010 M、0.10 M、または 1.0 M 酢酸または 1.0 M の酢酸またはアンモニウム水酸化物溶液の新しいボリュームを追加します。
- 10ppm PA溶液の負イオンおよび正イオンESI-IM-MSスペクトルをそれぞれ10分間収集します。
4. アンブサンプルの金属イオン滴定の調製
- 手順 3.1 ~ 3.5 に記載されている手順に従います。
- 0.125 mMアンブのピペ200.0 μLを1.7mLバイアルに入れ、500.0μLのDI水で希釈し、溶液を十分に混合します。
- 0.010 M水酸化アンモニウム溶液の80 μLを加えて、サンプルのpHをpH = 9.0に調整します。
- 0.125 mM金属イオン溶液の28 μLを追加し、金属イオンの0.14モル相当量を与え、DI水を加えてサンプルの最終容積を1.00 mLにし、完全に混合し、試料をRTで10分間平衡化させます。
- 鈍い鼻注射器を使用して、サンプルの500 μLを取り、それぞれ5分間の負および正イオンESI-IM-MSスペクトルを収集します。サンプルの残りの500 μLを使用して、校正されたマイクロpH電極を使用して最終pHを記録します。
- 手順 4.2 ~ 4.5 を繰り返し、ステップ 4.3 を変更して 0.125 mM 金属イオン溶液の適切な体積を追加し、0.28、0.42、0.56、0.70、0.84、0.98、1.12、1.26、または 1.40 モルルの同等量を与えます。
- 10ppm PA溶液の負イオンおよび正イオンIM-MSスペクトルをそれぞれ10分間収集する。
5. ESI-IM-MS pH滴定データの解析
- IM-MSスペクトルから、どの荷電種のアンブが存在するかを特定し、それらが理論的なm/z同位体パターンに一致させることによって存在する。
- MassLynxを開き、クロマトグラムをクリックしてクロマトグラムウィンドウを開きます。
- [ファイル]メニューに移動し、IM-MSデータ ファイルを検索して開きます。
- 右クリックしてクロマトグラムを横切ってドラッグして解放することにより、IM-MSスペクトルを抽出します。スペクトル ウィンドウが開き、IM-MS スペクトルが表示されます。
- スペクトルウィンドウで、[ツール]と[同位板]モデルをクリックします。同位体モデリングウィンドウで、アンブ種の分子式を入力し、[充電イオンを表示]ボックスをオンにして充電状態を入力します。[OK]をクリックします。
- IM-MSスペクトル内のすべての種を識別し、そのm/z同位方位化範囲を記録するために繰り返します。
- 各amb種について、偶然のm/z種を分離し、m/z同位元パターンを使用して到着時間分布(ATD)を抽出して識別します。
- MassLynxでドリフトスコープをクリックしてプログラムを開きます。DriftScope で[ファイル]をクリックし、[開く]をクリックして IM-MS データ ファイルを検索して開きます。
- マウスと左クリックを使用して、アンブ種の m/z 同位元パターンを拡大します。
- [選択] ツールとマウスの左ボタンを使用して、同位板パターンを選択します。[現在の選択を受け入れる]ボタンをクリックします。
- 偶然のm/z種を分離するには、選択ツールとマウスの左ボタンを使用して、amb種の同位元パターンに合わせたATD時間整列を選択します。[現在の選択を受け入れる]ボタンをクリックします。
- ATD をエクスポートするには、ファイルに移動します |MassLynx にエクスポートし、[ドリフト時間を保持]を選択し、適切なフォルダにファイルを保存します。
- ATDの高等部を決定し、種の集団の尺度としてATD曲線の下の領域を統合します。
- MassLynxのクロマトグラムウィンドウで、保存されたエクスポートされたファイルを開きます。[プロセス] をクリックします|メニューから統合します。[ApexTrack ピーク統合] ボックスをオンにして[OK]をクリックします。
- クロマトグラムウィンドウに示すように、高等 ATD (tA)と統合領域を記録します。保存されたすべての amb および PA IM-MS データ ファイルに対して、この手順を繰り返します。
- 各滴定点の正イオンまたはマイナスイオンのすべての抽出されたアンブ種に対して統合ATDを使用して、相対パーセンテージスケールに正規化します。
- 各pHでアンブ種とその統合ATDのアイデンティティをスプレッドシートに入力します。
- 各 pH について、統合 ATD の合計を使用して、個々の amb の ATD をパーセンテージ スケールに正規化します。
- 各アンブ種の強度対pHのパーセント強度をグラフにプロットし、各種の人口がpHの関数としてどのように変化するかを示します。
6. 衝突断面
- スプレッドシートを使用して、HEバッファガス28で測定されたPA負25、26および正27イオンのCCS(Ω)を、以下の式1を使用して修正されたCCS(Ωc)に変換します。ec = 電子電荷 (1.602×10-19 C);mN2 = N2ガスの質量 (Da);とmイオン= イオン質量。29歳
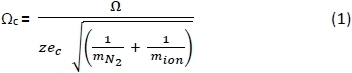
- PAキャリブラントおよびアンバ種の平均到着時間(tA)を、以下の式2を使用してドリフト時間(tD)に変換し、ここで:c =増強されたデューティサイクル遅延係数(1.41)、およびm/zはペプチドイオンの質量対電荷である。

- PA キャリブラントの tDと Ωcをプロットします。次に、次に示す式 3の最小二乗回帰適合値を使用して、A' と B の値を決定します。B は IM デバイスの非線形効果を補正します。

- これらのA値とB値と、AMBsのATDからの高調度tD値を使用して、方程式3を使用してΩcを決定し、方程式 1を使用して Ω を決定します。この方法は、約2%25、26、27の推定絶対誤差を持つペプチド種にCCSを提供する。
7. 計算方法
- ベッケ3パラメータハイブリッド機能30とダニングベーシスセット31と電子コア電位32、33、34で構成される理論のB3LYP/LanL2DZレベルを使用して、見つけます。観察されたm/z amb種35のすべての可能なタイプの調整のための幾何学に最適化された適合子。
注: 計算をビルドして送信する方法の詳細については、補足ファイルの GaussView の使用法を参照してください。 - 各適合子の予測自由エネルギーを比較し、シグマプログラム36からイオンスケールレナード・ジョーンズ(LJ)法を使用して理論CCSを計算する。
- 最も低い自由エネルギー適合体から、IM-MS測定CCSに同意するLJ CCSを展示するコンフォーマーを決定し、実験で観察された適合体の三次構造と調整の種類を特定します。
結果
amb1の金属結合
amb1(図1A)のIM-MS研究20は、銅イオンと亜鉛イオンの両方がpH依存様式でamb1に結合していることを示した(図2)。しかし、銅と亜鉛は、異なる協調部位における異なる反応機構を介してamb1に結合した。例えば、Amb1にCu(II)を加えると、ジスルフィドブリ?...
ディスカッション
重要なステップ:ESI-IM-MSを介した検査のためのソリューションフェーズの動作の保存
ネイティブESIインストゥルメンタル設定は、ペプチオメトリー、電荷状態、および立体構造を保存するために使用する必要があります。ネイティブ条件の場合、コーン電圧、温度、ガスフローなどのESIソースの条件を最適化する必要があります。また、ソース、トラップ、イオンモビリティ、...
開示事項
著者は何も開示していない。
謝辞
この資料は、1764436、NSF機器サポート(MRI-0821247)、ウェルチ財団(T-0014)、エネルギー省(TX-W-20090427-0004-50)およびL3コミュニケーションズの下で国立科学財団が支援する作業に基づいています。.私たちは、シグマプログラムを共有するためのカリフォルニア大学サンタバーバラのバウアーのグループに感謝し、ビデオで技術を実証するためのアヨバミ・イレサンミ。
資料
| Name | Company | Catalog Number | Comments |
| acetonitrile HPLC-grade | Fisher Scientific (www.Fishersci.com) | A998SK-4 | |
| ammonium hydroxide (trace metal grade) | Fisher Scientific (www.Fishersci.com) | A512-P500 | |
| cobalt(II) chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 255599-5G | |
| copper(II) chloride 99.999% | Sigma-Aldrich (www.sigmaaldrich.com) | 203149-10G | |
| copper(II) nitrate hydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 229636-5G | |
| designed amb1,2,3,4,5,6,7 peptides | Neo BioLab (neobiolab.com) | designed peptides were synthized by order | |
| designed amb5B,C,D,E,F peptides | PepmicCo (www.pepmic.com) | designed peptides were synthized by order | |
| Driftscope 2.1 software program | Waters (www.waters.com) | software analysis program | |
| Freeze-dried, purified, Cu(I)-free mb-OB3b | cultured and isolated in the lab of Dr. DongWon Choi (Biology Department, Texas A&M-Commerce) | ||
| glacial acetic acid (Optima grade) | Fisher Scientific (www.Fishersci.com) | A465-250 | |
| Iron(III) Chloride Anhydrous 98%+ | Alfa Aesar (www.alfa.com) | 12357-09 | |
| lead(II) nitrate ACS grade | Avantor (www.avantormaterials.com) | 128545-50G | |
| manganese(II) chloride tetrahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203734-5G | |
| MassLynx 4.1 | Waters (www.waters.com) | software analysis program | |
| nickel chloride hexahydrate 99.99% | Sigma-Aldrich (www.sigmaaldrich.com) | 203866-5G | |
| poly-DL-alanine | Sigma-Aldrich (www.sigmaaldrich.com) | P9003-25MG | |
| silver nitrate 99.9%+ | Alfa Aesar (www.alfa.com) | 11414-06 | |
| Waters Synapt G1 HDMS | Waters (www.waters.com) | quadrupole - ion mobility- time-of-flight mass spectrometer | |
| zinc chloride anhydrous | Alfa Aesar (www.alfa.com) | A16281 |
参考文献
- Dudev, T., Lim, C. Competition among Metal Ions for Protein Binding Sites: Determinants of Metal Ion Selectivity in Proteins. Chemical Reviews. 114 (1), 538-556 (2014).
- Sovago, I., Kallay, C., Varnagy, K. Peptides as complexing agents: Factors influencing the structure and thermodynamic stability of peptide complexes. Coordination Chemistry Reviews. 256 (19-20), 2225-2233 (2012).
- Sóvágó, I., Várnagy, K., Lihi, N., Grenács, &. #. 1. 9. 3. ;. Coordinating properties of peptides containing histidyl residues. Coordination Chemistry Reviews. 327, 43-54 (2016).
- Rubino, J. T., Franz, K. J. Coordination chemistry of copper proteins: How nature handles a toxic cargo for essential function. Journal of Inorganic Biochemistry. 107 (1), 129-143 (2012).
- Robinson, N. J., Winge, D. R. Copper Metallochaperones . Annual Review of Biochemistry. 79, 537-562 (2010).
- Scheiber, I. F., Mercer, J. F. B., Dringen, R. Metabolism and functions of copper in brain. Progress in Neurobiology. 116, 33-57 (2014).
- Tisato, F., Marzano, C., Porchia, M., Pellei, M., Santini, C. Copper in Diseases and Treatments, and Copper-Based Anticancer Strategies. Medicinal Research Reviews. 30 (4), 708-749 (2010).
- Millhauser, G. L. Copper and the prion protein: Methods, structures, function, and disease. Annual Review of Physical Chemistry. 58, 299-320 (2007).
- Arena, G., Pappalardo, G., Sovago, I., Rizzarelli, E. Copper(II) interaction with amyloid-beta: Affinity and speciation. Coordination Chemistry Reviews. 256 (1-2), 3-12 (2012).
- Kim, H. J., et al. Methanobactin, a copper-acquisition compound from methane-oxidizing bacteria. Science. 305 (5690), 1612-1615 (2004).
- Di Spirito, A. A., et al. Methanobactin and the link between copper and bacterial methane oxidation. Microbiology Molecular Biology Reviews. 80 (2), 387-409 (2016).
- Kenney, G. E., Rosenzweig, A. C. Chemistry and biology of the copper chelator methanobactin. ACS Chemical Biology. 7 (2), 260-268 (2012).
- Summer, K. H., et al. The biogenic methanobactin is an effective chelator for copper in a rat model for Wilson disease. Journal of Trace Elements in Medicine and Biology. 25 (1), 36-41 (2011).
- Hachmoeller, O., et al. Investigating the influence of standard staining procedures on the copper distribution and concentration in Wilson's disease liver samples by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 44, 71-75 (2017).
- Hachmoeller, O., et al. Spatial investigation of the elemental distribution in Wilson's disease liver after D-penicillamine treatment by LA-ICP-MS. Journal of Trace Elements in Medicine and Biology. 44, 26-31 (2017).
- Hachmoeller, O., et al. Element bioimaging of liver needle biopsy specimens from patients with Wilson's disease by laser ablation-inductively coupled plasma-mass spectrometry. Journal of Trace Elements in Medicine and Biology. 35, 97-102 (2016).
- Mueller, J. C., Lichtmannegger, J., Zischka, H., Sperling, M., Karst, U. High spatial resolution LA-ICP-MS demonstrates massive liver copper depletion in Wilson disease rats upon Methanobactin treatment. Journal of Trace Elements in Medicine and Biology. 49, 119-127 (2018).
- Choi, D. W., et al. Spectral and thermodynamic properties of Ag(I), Au(III), Cd(II), Co(II), Fe(III), Hg(II), Mn(II), Ni(II), Pb(II), U(IV), and Zn(II) binding by methanobactin from Methylosinus trichosporium OB3b. Journal of Inorganic Biochemistry. 100, 2150-2161 (2006).
- McCabe, J. W., Vangala, R., Angel, L. A. Binding Selectivity of Methanobactin from Methylosinus trichosporium OB3b for Copper(I), Silver(I), Zinc(II), Nickel(II), Cobalt(II), Manganese(II), Lead(II), and Iron(II). Journal of the American Society of Mass Spectrometry. 28, 2588-2601 (2017).
- Sesham, R., et al. The pH dependent Cu(II) and Zn(II) binding behavior of an analog methanobactin peptide. European Journal of Mass Spectrometry. 19 (6), 463-473 (2013).
- Wagoner, S. M., et al. The multiple conformational charge states of zinc(II) coordination by 2His-2Cys oligopeptide investigated by ion mobility - mass spectrometry, density functional theory and theoretical collision cross sections. Journal of Mass Spectrom. 51 (12), 1120-1129 (2016).
- Bandow, N. L., et al. Isolation of methanobactin from the spent media of methane-oxidizing bacteria. Methods in Enzymology. 495, 259-269 (2011).
- Choi, D. W., et al. Spectral and thermodynamic properties of methanobactin from γ-proteobacterial methane oxidizing bacteria: a case for copper competition on a molecular level. Journal of Inorganic Biochemistry. 104 (12), 1240-1247 (2010).
- Pringle, S. D., et al. An investigation of the mobility separation of some peptide and protein ions using a new hybrid quadrupole/travelling wave IMS/oa-ToF instrument. International Journal of Mass Spectrometry. 261 (1), 1-12 (2007).
- Forsythe, J. G., et al. Collision cross section calibrants for negative ion mode traveling wave ion mobility-mass spectrometry. Analyst. 14 (20), 6853-6861 (2015).
- Allen, S. J., Giles, K., Gilbert, T., Bush, M. F. Ion mobility mass spectrometry of peptide, protein, and protein complex ions using a radio-frequency confining drift cell. Analyst. 141 (3), 884-891 (2016).
- Bush, M. F., Campuzano, I. D. G., Robinson, C. V. Ion Mobility Mass Spectrometry of Peptide Ions: Effects of Drift Gas and Calibration Strategies. Analytical Chemistry. 84 (16), 7124-7130 (2012).
- Salbo, R., et al. Traveling-wave ion mobility mass spectrometry of protein complexes: accurate calibrated collision cross-sections of human insulin oligomers. Rapid Communications in Mass Spectrometry. 26 (10), 1181-1193 (2012).
- Smith, D. P., et al. Deciphering drift time measurements from travelling wave ion mobility spectrometry-mass spectrometry studies. European Journal of Mass Spectrometry. 15 (2), 113-130 (2009).
- Becke, A. D. Density-functional thermochemistry. III. The role of exact exchange. Journal of Chemical Physics. 98 (7), 5648-5652 (1993).
- Dunning, T. H., Hay, P. J. Gaussian basis sets for molecular calculations. Modern Theoretical Chemistry. 3, 1-27 (1977).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for potassium to gold including the outermost core orbitals. Journal of Chemical Physics. 82 (1), 299-310 (1985).
- Hay, P. J., Wadt, W. R. Ab initio effective core potentials for molecular calculations. Potentials for the transition metal atoms scandium to mercury. Journal of Chemical Physics. 82 (1), 270-283 (1985).
- Wadt, W. R., Hay, P. J. Ab initio effective core potentials for molecular calculations. Potentials for main group elements sodium to bismuth. Journal of Chemical Physics. 82 (1), 284-298 (1985).
- . Gaussian 09, Revision C.01. Gaussian, Inc. , (2012).
- Wyttenbach, T., von Helden, G., Batka, J. J., Carlat, D., Bowers, M. T. Effect of the long-range potential on ion mobility measurements. Journal of the American Society of Mass Spectrometry. 8 (3), 275-282 (1997).
- Choi, D., et al. Redox activity and multiple copper(I) coordination of 2His-2Cys oligopeptide. Journal of Mass Spectrometry. 50 (2), 316-325 (2015).
- Rigo, A., et al. Interaction of copper with cysteine: stability of cuprous complexes and catalytic role of cupric ions in anaerobic thiol oxidation. Journal of Inorganic Biochemistry. 98 (9), 1495-1501 (2004).
- Vytla, Y., Angel, L. A. Applying Ion Mobility-Mass Spectrometry Techniques for Explicitly Identifying the Products of Cu(II) Reactions of 2His-2Cys Motif Peptides. Analytical Chemistry. 88 (22), 10925-10932 (2016).
- Choi, D., Sesham, R., Kim, Y., Angel, L. A. Analysis of methanobactin from Methylosinus trichosporium OB3b via ion mobility mass spectrometry. European Journal of Mass Spectrometry. 18 (6), 509-520 (2012).
- Martell, A. E., Motekaitis, R. J. NIST Standard Reference Database 46. Institute of Standards and Technology. , (2001).
- Pesch, M. L., Christl, I., Hoffmann, M., Kraemer, S. M., Kretzschmar, R. Copper complexation of methanobactin isolated from Methylosinus trichosporium OB3b: pH-dependent speciation and modeling. Journal of Inorganic Biochemistry. 116, 55-62 (2012).
- Amin, E. A., Truhlar, D. G. Zn Coordination Chemistry: Development of Benchmark Suites for Geometries, Dipole Moments, and Bond Dissociation Energies and Their Use To Test and Validate Density Functionals and Molecular Orbital Theory. Journal of Chemical Theory and Computation. 4 (1), 75-85 (2008).
- Sorkin, A., Truhlar, D. G., Amin, E. A. Energies, Geometries, and Charge Distributions of Zn Molecules, Clusters, and Biocenters from Coupled Cluster, Density Functional, and Neglect of Diatomic Differential Overlap Models. Journal of Chemical Theory and Computation. 5 (5), 1254-1265 (2009).
- Lillo, V., Galan-Mascaros, J. R. Transition metal complexes with oligopeptides: single crystals and crystal structures. Dalton Transactions. 43 (26), 9821-9833 (2014).
- Choutko, A., van Gunsteren, W. F. Conformational Preferences of a beta-Octapeptide as Function of Solvent and Force-Field Parameters. Helvetica Chimica Acta. 96 (2), 189-200 (2013).
- Angel, L. A. Study of metal ion labeling of the conformational and charge states of lysozyme by ion mobility mass spectrometry. European Journal of Mass Spectrometry. 17 (3), 207-215 (2011).
- Kelso, C., Rojas, J. D., Furlan, R. L. A., Padilla, G., Beck, J. L. Characterisation of anthracyclines from a cosmomycin D-producing species of Streptomyces by collisionally-activated dissociation and ion mobility mass spectrometry. European Journal of Mass Spectrometry. 15 (2), 73-81 (2009).
- El Ghazouani, A., et al. Copper-binding properties and structures of methanobactins from Methylosinus trichosporium OB3b. Inorganic Chemistry. 50 (4), 1378-1391 (2011).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved