Method Article
定量染色体立体構造捕捉による胚体におけるエンハンサー・プロモーター接触の同定(4C)
要約
我々は、胚性幹細胞から生成された胚体における高スループットシーケンシングに続く定量染色体立体構造捕捉の適用を報告する。この技術は、胚性幹細胞分化中に所定の遺伝子の推定エンハンサーとプロモーター領域との接触を同定し、定量することを可能にする。
要約
哺乳類の開発中、細胞の運命は、遺伝子発現の特異性、タイミング、および空間パターンを定義する規制ネットワークの確立を通じて決定される。多能性幹細胞由来の胚体(EBs)は、主な3つの生殖層の分化を研究し、細胞運命指定時に調節回路を定義する一般的なモデルとなっている。組織特異的エンハンサーがプロモーターと相互作用することによってこれらのネットワークで重要な役割を果たしていることはよく知られていますが、関連する標的遺伝子に割り当てることはまだ困難です。これを可能にするために、開発中のエンハンサー・プロモーター接触とそのダイナミクスを研究するために定量的アプローチが必要です。ここでは、EB分化モデルにおける同精促進剤とのエンハンサーおよびその接触を定義する4C法を適合させました。この方法では、頻繁に切断制限酵素、超音波処理、およびネストライゲーション媒介PCRプロトコルを、市販のDNAライブラリ調製キットと互換性のある使用します。続いて、4Cライブラリはハイスループットシーケンシングを行い、バイオインフォーマット的に分析され、選択されたプロモーターとの接触を有するすべての配列の検出と定量が可能になる。結果のシーケンシングデータは、分化中のエンハンサー・プロモーター接触のダイナミクスに関する情報を得るためにも使用することができる。EB 分化モデルに関して説明されている手法は、簡単に実装できます。
概要
マウスでは、3.5日齢の胚の内細胞塊(ICM)は、胚多能性幹細胞を含む。ICMはさらに4.5日目にエピブラストに発展し、胚の主な3つの胚層である外胚、中胚、および内胚細胞を生成する。ICM内の多能性細胞は生体内に一過性しか存在しないが、マウス胚性幹細胞(mESC)1、2、3の樹立により1,2,3培養中に捕捉することができる。mESCは未分化状態のままで無限に増殖するが、内因的および外因性刺激の際には、多能性状態を抜け出し、3つの発達生殖層22、44の細胞を生成することもできる。興味深いことに、小さな液滴で懸濁液中で培養すると、mESCは3つの胚層5のすべてに分化する3次元凝集体(すなわち、EB)を形成する。EB形成アッセイは、初期の系統指定プロセスを研究するための重要なツールです。
系統指定の間、各生殖層の細胞は特定の遺伝子発現プログラム4を獲得する。遺伝子の正確な時空間的発現は、コアプロモーター、エンハンサー、サイレンサー、および絶縁,体6、7、8、97を含む多様6なシス調節要素によって調節される。8,9エンハンサーは、典型的には数百塩基対にまたがる調節DNAセグメント、組織特異的遺伝子発現8を調整する。エンハンサーは、局所クロマチン構造88,1010を調節する転写因子および補因子との結合によって活性化または沈黙される。推定エンハンサーを同定するために一般的に使用される技術は、ゲノム全体のクロマチン免疫沈降とそれに続くシーケンシング(ChIP-seq)、およびシーケンシング(ATAC-seq)技術を用いたトランスポザーゼ対応クロマチンのアッセイである。したがって、活性エンハンサーは、特定の活性ヒストンマークと増加局所DNAアクセシビリティ1111、12、13、1412,13,によって特徴付けられる。さらに、発達促進剤は、その同精促進剤88,99との物理的相互作用を必要とすると考えられている。実際に、エンハンサー・プロモーターの接触を妨害するエンハンサー変異体および欠失が発達奇形15を引き起こす可能性が示されている。そのため、発達遺伝子発現を制御する機能エンハンサーの同定に関する付加的な情報を提供する新しい技術が必要とされている。
染色体立体構造捕捉(3C)技術16の開発以来、染色体接触のマッピングは、調節要素間の物理的距離を評価するために集中的に使用されてきた。重要なことに、3C技術のハイスループット変異体が最近開発され、クロマチン断片17間の接触の固定、消化、結紮、および回復のための異なる戦略を提供する。中でも、Hi-Cの中ではゲノムワイド18の3Cライゲーション製品のシーケンシングを可能にする一般的な技術となっている。しかし、エンハンサー・プロモーター接触の分析に適した解像度に達するために必要な高いシーケンシングコストは、この技術を特定の遺伝子座の研究には現実的でありません。したがって、より高,い解像度19、20、21、2220,21で標的座を分析する代替方法が開発された。1922これらの方法の1つ、すなわち4Cは、1対全戦略として知られており、視点として選択されたサイトに接触するすべてのシーケンスを検出することができます。しかし、標準4C技術の欠点は、必要な逆PCRであり、異なるサイズの断片を増幅し、小さな製品を支持し、ハイスループットシーケンシング後のバイアス定量化である。近年、UMI-4Cは、この問題23を回避する定量的および標的染色体接触プロファイリング用に、一意の分子識別子(UMI)を用いた4C技術の新しい変形が開発されている。このアプローチでは、頻繁なカッター、超音波処理、およびネストライゲーション媒介PCRプロトコルを使用し、比較的均一な長さの分布を有するDNA断片の増幅を伴う。この均質性は、短い配列のためのPCRの好みの増幅プロセスにおけるバイアスを低減し、空間的に接続された分子/断片の効率的な回復および正確なカウントを可能にする。
ここでは、EB 分化中のリネージュ有益な転写因子のプロモーターとエンハンサー間のクロマチン接触を同定および定量するために UMI-4C 技術を適応させるプロトコルについて説明する。
プロトコル
1. マウス胚性幹細胞からの胚性の体生成
- MESC無血清培地を調製:DMEM/F12および神経基底培地を1:1の割合で混合した。培地は、MEM非必須アミノ酸溶液(1x)、ピルビン酸ナトリウム(1mM)、L-グルタミン(2mM)、ペニシリンストレプトマイシン(100 U/mL)、β-メルカプトエタノールを添加した。 (50 μM)、N2およびB27サプリメント(1x)、PD0325901(1 μM)、CHIR99021(3μM)、白血病阻害因子(LIF)(1,000 U/mL)。
- EB分化培地を調製する:DMEMは10%胎児ウシ血清(FBS)、MEM非必須アミノ酸(1x)、ピルビン酸ナトリウム(1mM)、L-グルタミン(2mM)、ペニシリンストレプトマイシン(100 U/mL)、β-メルカプトエタノール(50μM)を添加した。
- mESC培養液中に0.1%(w/v)ゼラチンをあらかじめコーティングした10cmプラスチック皿の培養メック。
- mESCが60%の合流度に達したら、培養培地を取り除き、2mLの殺菌されたPBSで1倍静かに洗います。
- PBSを完全に取り除き、2 mLの細胞剥離培地を加えます。培養皿を37°Cで5分間インキュベートします。
- 8 mLのEB分化培地をディッシュに加えて反応を不活性化する。
- mESCコロニーを上下15〜20回ピペット化して単細胞懸濁液を得た。
- 室温(RT)で300xgのg細胞を5分間遠心し、上清を慎重に取り除きます。
- カウント細胞(例えば、ヘモサイトメーターを用いて)。
- 細胞ペレットをEB分化培地で再懸濁し、濃度を2 x 104細胞/mLに調整します。
- 15 cm培養皿の蓋を反転させ、200 μLのマルチチャンネルピペットを使用して、20 μLの再懸濁液(約400細胞/ドロップ)を蓋に堆積させます。
- 蓋を慎重に底のチャンバーに反転させ、37°Cの吊り下げ液で3日間、CO2と湿度95%で3日間インキュベートします。
- 10 mLのPBSで蓋を軽く洗い、EB含有懸濁液を50mLのプラスチックチューブに移して、EBを回収します。
- チューブをRTに30分間置き、重力で底に沈みます。上清を慎重に取り除いてください。
- 新鮮なEB分化培地10mLでEBを穏やかに再中断し、10cmの細菌学的ペトリ皿に移します。
- 逆顕微鏡を使用して、3-6日後にEB形成を確認してください。生成される EJB は、丸く、同種のサイズにする必要があります。
- 5%CO2と95%の湿度で37°C2で培養をインキュベートします。EB は 3 つの胚芽層に対して引き続き区別され、さまざまなタイムポイントで解析できます。
2. エブの解離
- 2~3個の10cmの皿から50mLのプラスチックチューブにEBを集めろ。5分間RTで300 x gでエブを遠心分離し、慎重に上清を取り除きます。
- 10 mL の PBS で EJB を再中断します。遠心分離機のエブはRTで300×gで3分間、上清を取り除く。 g
- トリプシン-EDTAの2 mLを追加する(0.25%)ペレットにし、37 °Cで15分間チューブをインキュベートし、ピペットを3分間上下にして単一細胞懸濁液を得る。
- 8 mLのEB分化培地を加えてトリプシン反応を停止します。EBの解離を顕微鏡で確認し、細胞を数えます。
3. 固定
- 1 x 106細胞/mLで新鮮なEB培養培地中の細胞を再懸濁する。50 mL チューブの場合、最大 4.5 x 107 個のセルを 45 mL の培地に使用します。
- 37%の在庫(6ヶ月以上前ではない)からパラホルムアルデヒドを1%の最終濃度に加えます。
注意:パラホルムアルデヒドは有害な化学物質であるため、適切な安全衛生規則に従い、パラホルムアルデヒドを取り扱ってください。 - 回転の下でRTで10分間インキュベートします。
- 0.125 Mの最終濃度にグリシンを加えてホルムアルデヒドをクエンチする。
- 回転の下でRTで5分間インキュベートします。
- 固い細胞を氷に移し、これから4°Cで冷たく保ちます。
- 細胞を冷蔵遠心分離機でg5分間300xgでペレット化する。
- 上清を捨てて、冷たいPBS(5 x 106細胞の1mL)でペレットを再懸濁し、1.5 mLの安全ロックチューブに移します。
- 4°Cで5分間300xgの細胞をペレットにし、上清を捨て、ペレットを液体窒素中でスナップフリーズします。 g-80°Cで保存するか、以下のプロトコルに進みます。
4. 細胞の分解と制限酵素の消化
- 細胞ペレットを、調製したばかりの氷冷リシスバッファー(10 mM Tris-HCl pH = 8.0、10 mM NaCl、0.2%イゲパルCA630、および1xプロテアーゼ阻害剤)を2〜5 x 106細胞あたり0.25 mLに静かに再懸濁します。5 mL のリシスバッファーを準備するには、表 1を参照してください。
- 氷の上で15分間細胞をインキュベートします。
- 遠心分離機 1,000 x gで 5 分 4 °C.上清を捨てて、核を含むペレットを保管してください。
- ペレット化した核を500 μLの冷たいリシスバッファーで洗浄します。
- ペレットを0.5%SDSの50 μLの1.5mLチューブ内の1xバッファ2に静かに再懸濁し、62°Cの加熱ブロックで10分間チューブをインキュベートします。
- 加熱ブロックからチューブを取り出し、10%トリトンX-100の25 μLを含む170 μLの消化バッファーを加え、SDSを急冷します。過度の発泡を避け、ピペットでよく混ぜます。
- 37°Cで15分間インキュベートします。
- 25 μLの消化バッファーを加え、反転して混合し、未消化制御として8 μLを取ります。未消化の対照試料を-20°Cに保ちます。残りの核に100 U MboI制限酵素(25 U/μLストックの4μL)を加え、クロマチンを37°Cで2時間、回転下で消化します。MboIの100 Uの別のアリコートを追加し、さらに2時間インキュベートします。
- さらに100 UのMboIを加え、37°Cで一晩回転してインキュベートする。
- 翌日、さらに100 UのMboIを加え、37°Cで3時間インキュベートします。
- 8 μLを消化された対照サンプルとして取ります。80 μLのTEバッファー(10 mM Tris pH = 8,1 mM EDTA)および10 μLのプロテイナーゼK(10 mg/mL)を加えることで、ステップ4.8から脱架橋された制御サンプルおよび未消化制御サンプル。65°Cで1時間インキュベートする。
- 0.6%のゲルで20 μLのアリコートを実行し、消化効率を確認します。正常なダイジェストは、主に3.0-0.5 kbの範囲の断片を示しています。
5. 近接ライゲーションと架橋反転
- MboI消化サンプルを加熱ブロックで65°Cで20分間インキュベートし、MboIを不活性化し、RTに冷却します。
- RTで1,000 x gで5分間チューブを遠心し、上清を取り除き、ペレットを200μLの新鮮なリガーゼバッファーに溶解します。
- 各サンプルに1,000 μLのライゲーションマスターミックスを加えます。ライゲーションマスターミックスの1,000 μLを調製するには、表2を参照してください。
- 転向して混ぜ、遅い回転(9 rpm)でRTの夜にインキュベートする。
- RNAおよびタンパク質残基を除去するには、100 μLのプロテイナーゼK(10 mg/mL)と10μLのRNase A(10 mg/mL)を加えて下さいます。サンプルを55°Cで45~60分間インキュベートします。
- 65°Cでサンプルを4時間続けて続けます。
6. DNAのせん断とサイズの選択
- RTに冷却チューブ。
- 遠心分離機 1,000 x gで 4 °C で 5 分間
- サンプルを2mLチューブ内の3つの400 μLアリコートに分割し、2μLのグリコーゲン(20mg/m)、40μLの酢酸ナトリウム(3M、pH = 5.2)、各チューブに100%エタノールの2.5倍の体積(1mL)を加えます。反転して混ぜ、-80°Cで45〜60分間インキュベートします。
- 4°Cで16,000 x gの遠心分離機を25分間回転させた後、チューブを氷の上に置き、ピペットで慎重に上清を取り除きます。
- 70%エタノールの800 μLで再懸濁してDNAペレットを洗浄します。遠心分離機 16,000 x gで 4 °C で 5 分間
- 上清を取り除き、70%エタノールの800 μLでもう一度洗浄を行います。
- ペレットを1xトリスバッファー(10 mM Tris-HCl、pH =8)の130 μLに溶解し、37°Cで15分間インキュベートしてDNAを完全に溶解します。必要に応じて、ピペットを使用して沈殿物を再中断します。
- DNAの収量を測定します。2.5~5 μgのクロマチンは、1 x 106個の細胞に対して期待できます。3C製品の±200 ngを0.6%アガロースゲルで実行してライゲーションを確認します。成功したライゲーションは、主にDNA断片を示す > 3 kb.サンプルは-20°Cで保存してください。
- 1x Trisバッファボリューム(1μg)の100 μLで10ng/μLの超音波処理に適した0.65 mLチューブでサンプルを希釈します。ライブラリの準備に使用される標準的な量は3 μgですので、必要に応じて3つの別々のチューブで超音波処理を行います。
- 超音波器上の次のパラメータを使用して150-700 bp(平均= 400-500 bp)のサイズにDNAを剪断:サイクル:60 sオフの20 sの6-8。これにより、Illuminaシーケンサーを使用したハイスループットシーケンシングライブラリ調製に適したDNAが得られます。
- シアドDNAを通常の新しいセーフロックチューブに移します。同じサンプルから複数の超音波処理をプールします。
- RTでDNA精製ビーズのボトルを温めます。これからは、低いバインドのヒントを使用します。
- 1.8倍のビーズをDNAチューブに加え、穏やかに再中断します。
- RTで5分間インキュベートします。
- 磁気ラックでビーズを収集します。マグネティックラックにチューブを入したまま、作りたての80%エタノールを1mLでビーズ2倍洗います。
注:残留液滴を含むすべてのエタノールを除去します。 - RTでビーズを短時間(2〜3分)空気乾燥させます。
注:5分以上のビーズを乾燥させないでください。これによりDNA収率が低下します。 - 90 μL の 1x Tris バッファー (10 mM Tris-HCl、pH = 8) のビーズを再中断して、DNA を溶出します。
- DNAの収率を測定し、1.5%ゲルで5μLアリコートを分析します。予音前の収量に比べて非常に少ない損失があるはずです。
7. シーケンシングのためのライブラリの準備
- ライブラリ準備キットから15 μLのマスターミックスを追加します。せわされたDNAの末端を修復するには、10倍末修復反応バッファーの10 μLと5 μLのエンドリペア酵素ミックスを組み合わせます。
- RTで30分間インキュベートします。
- 1.1倍のDNA精製ビーズを加え、穏やかに再懸濁します。
- RTで5分間インキュベートします。
- 磁気ラックでビーズを収集します。マグネティックラックにチューブを入したまま、1 mLの新しく作りたての80%エタノールでビーズを2回洗います。エタノールを取り除く。
- RTでビーズを2〜3分間エアドライし、42 μLの1x Trisバッファ(10 mM Tris-HCl、pH =8)でビーズを再中断してDNAを溶出させます。
- 各サンプルに8μLのdA-tailingマスターミックスを加えます。dA-tailingマスターミックスを準備するには、5 μLの10倍のdA-テーリング反応バッファーと3 μLのクレノウフラグメントエキソマイナスを組み合わせます。
- 37°Cで30分間インキュベートします。
- 2μLの子牛の腸内アルカリホスファターゼ(CIP)を加えてDNAを脱リン酸化します。
- 37°Cで30分間インキュベートし、50°Cで60分インキュベートします。
- 1.1倍のDNA精製ビーズを加え、穏やかに再懸濁します。
- RTで5分間インキュベートします。
- 磁気ラックでビーズを収集します。マグネティックラックにチューブを入したまま、作りたての80%エタノールを1mLでビーズ2倍洗います。
- RTでビーズを短時間(2〜3分)空気乾燥します。35 μLの1x Trisバッファ(10 mM Tris-HCl、pH = 8)のビーズを再中断してDNAを溶出させます。
- アダプターライゲーション反応を行います。表 3に示すように、アダプター/リガーゼ濃度を低減して使用します。
- 20°Cで15分間インキュベートします。
- ウラシルDNAグリコシラーゼとDNAグリコシラーゼエンドヌクレアーゼVII(例えば、USER)酵素の混合物の3μLを加え、ピペット処理によって混合し、37°Cで15分間インキュベートする。
- 水で100μLにボリュームを上げ、96°Cで5分沸騰させ、サンプルを氷の上に置きます。
- 1.1倍のDNA精製ビーズを加え、穏やかに再懸濁します。
- RTで5分間インキュベートします。
- 磁気ラックでビーズを収集します。マグネティックラックにチューブを入したまま、作りたての80%エタノールを1mLでビーズ2倍洗います。
- RTでビーズを2〜3分間エアドライし、50 μLの1x Trisバッファ(10 mM Tris-HCl、pH = 8)でビーズを再中断してDNAを溶出させます。
8. 4Cクロマチン相互作用ライブラリの増幅と精製
- 10 μLのライブラリを使用して4Cライブラリを増幅し、最初のPCRを実行します。PCR のセットアップとプログラムは、表 4を参照してください。
- 入れ子になった PCR を実行します。ネストされた PCR のセットアップとプログラムは、表 5を参照してください。
- 各ライブラリーの PCR 製品をプールし、1.1x DNA 精製ビーズで精製します。
- DNAの収率を測定し、1.5%ゲルで5μLアリコートを分析します。
- ライブラリの濃度を調整し、ライブラリをシーケンスします。インデックスが作成されている場合、ライブラリはシーケンスの前にプールできます。
結果
吊り下げ低下におけるESC分化誘導の6日後、さらなる分析に用いた同種のEB集団を得た(図1)。我々は、UMI-4C法23を適応し、EBs24における系統特異的遺伝子のプロモーターにおける特異的クロマチン相互作用を24定量化した。異なるステップで代表的な品質管理ゲルを使用するプロトコルの概略図の概要を図 2Aに示します。第1の品質管理は、MboI制限酵素消化の効率を決定するために行った。効率的な消化は3kbp未満のフラグメントサイズを示した(図2B)。なお、mESCおよびEBクロマチン消化は難しく、未消化のクロマチンが残留することがあった。第2の品質管理は、ほとんどの断片が現在>3kbpであることを確認するために、結紮後に行った(図2B)。次いで、超音波処理後に得られたクロマチン断片をゲル電気泳動法により解析した。400~500bpのフラグメントサイズが予想された(図2B)。
脱リン酸化およびシングルエンドアダプターのライゲーションの後、目的の標的を増幅するために2ラウンドのPCRを行った。ネストされたアプローチは、各軌跡に対して2つのプライマーのセットを設計するために使用されました。これは特異性を改善するのに役立ちました。各標的は、PCR条件を最適化するために2つの異なるプライマーペアで別々に増幅された(すなわち、それぞれT遺伝子座のPou5f1遺伝子座およびプライマーペアのプライマーペアAとBとプライマーペアCとD)を、400bp前後のDNAスミアを生じさせた(図2C)。あるいは、標的AとCを同時に増幅するためにマルチプレックスPCRを行い(図2D)を同時に増幅し、精製後に同様のフラグメントサイズを生じた(図2D)。4Cライブラリ調製に使用されるプライマー (Pou5f1およびTの軌跡)は、表6に記載されています。
データ分析では、生シーケンシング読み取りは最初にrefence mm10マウスゲノムに対して整列し、すべて複製され、低品質(<20)読み取りが削除された。各餌について、読み取り断片数を算出した各制限フラグメントに関する情報を取得し、生の接触プロファイルを得た。次に、対象領域は、餌までの距離が2kbpおよび250kbpのすべての制限フラグメントとして定義された。各制限フラグメントのサイズは、対象領域内の生接触の総数の5%の閾値に達するまでプロファイルを平滑化するために、隣接する制限フラグメントを順次集約することによって増加した。複製が統合され、条件が比較されたことを確認するために、制限フラグメントレベルに傾斜とランダム・インターセプトの両方を含めた。図 3に示すように、条件ごとの平均プロファイルとそれらの間の折り目の変化をプロットしました。EB分化の間、増強剤と多能性遺伝子Pou5f1のプロモーターとの接触は減少し、一方で、チェンデドリン病系統有益転写因子Tのエンハンサープロモーター接触が増加する(図3)、これらの発達エンハンサーに関する機能的洞察を提供する。
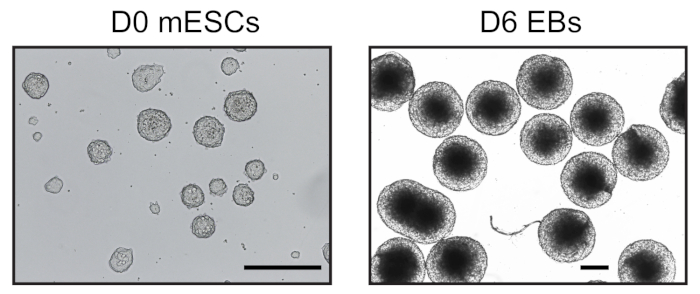
図1:mESCおよび派生胚体の代表的な画像。0日目 mESC は、無血清条件で培養した(左)と6日目の均質な6日目のEB(右)を逆顕微鏡で観察した。スケールバー= 500 μmこの図の大きなバージョンを表示するには、ここをクリックしてください。
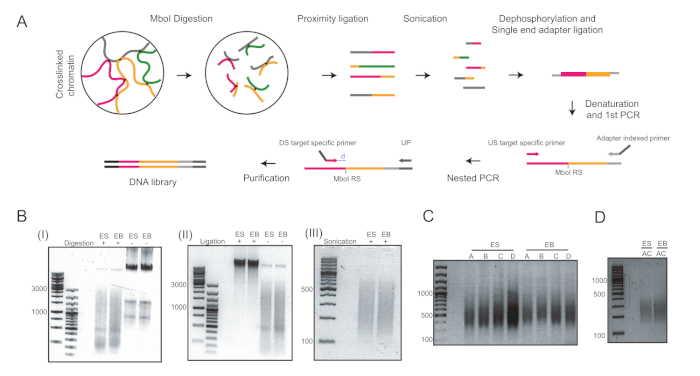
図2:プロトコルの主なステップの4Cワークフローと代表的な画像。(A) 定量4Cの概略的ワークフロー。RS = 制限サイト;US = アップストリーム;DS = ダウンストリーム;UP = ユニバーサルプライマー;D=RSとDSの間の距離は理想的には5-15 bpであるB必要があります(B)MboI消化クロマチン(I)の例、核結紮クロマチン(II)、および超音波クロマチン(III))。左側の数字は、各サンプルのDNAラダーランによって決定されるDNAサイズを示しています。(C)2つの遺伝子座におけるPCR増幅の例:Pou5f1(プライマーAおよびB)およびT(プライマーCおよびD)。(D) プライマーAおよびC.ES=胚性幹細胞を用いたPou5f1およびT遺伝子座におけるマルチプレックスPCR増幅の例;EB = 胚体。この図の大きなバージョンを表示するには、ここをクリックしてください。
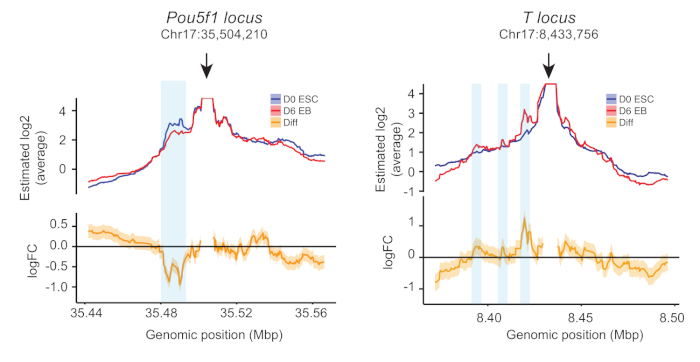
図3:4Cプロファイルの例MESCおよび6日目EBSでアッセイされたPou5f1およびT遺伝子プロモーターに位置する餌の定量4Cプロファイル。トップパネルは、2つの独立した生物学的複製から生成された平均接触のプロットを示しています。下部のパネルには、6 日目の EB と mESC の平均接触フォールド変化(2 つの反復の平均)が表示されます。水色のボックスは、分化中の動的変化を伴うエンハンサーの位置を示します。天ら24から適応図 .この図の大きなバージョンを表示するには、ここをクリックしてください。
| 5mLの場合 | |
| 1M トリス-HCl、pH8.0 | 50 μL |
| 5M ナクル | 10 μL |
| 10% イゲパル CA630 | 100 μL |
| 50xロシュ完全プロテアーゼ阻害剤 | 100 μL |
| ミリクウォーター | 4.74 mL |
表1:リシスバッファ。
| 1000μLの場合 | |
| ミリクウォーター | 869 μL |
| 10X NEB T4 DNAリガーゼバッファー | 120 μL |
| 20mg/mL 牛血清アルブミン | 6 μL |
| 2000 U/μL T4 DNAリガーゼ | 5 μL |
表2:ライゲーションマスターミックス調製。
| 15 μLの場合 | |
| 5Xクイックライゲーション反応バッファー | 10 μL |
| ネブネクストアダプター | 3 μL |
| クイックT4 DNAリガーゼ | 2 μL |
表3:アダプターライゲーション反応。
| PCR のセットアップ | |
| アダプター・リガテッド・ライブラリー・オン・デッド | 10 μL |
| PCRグレード水 | 20.25 μL |
| 10 μM ターゲット特定プライマー | 3.75 μL |
| 10 μM NEB インデックスプライマー | 3.75 μL |
| ヘラクサーゼII 5Xバッファー | 10 μL |
| 10 Mm dNTPs | 1.25 μL |
| ヘラクサーゼIIポリメラーゼ | 1 μL |
| 総量 | 50 μL |
| PCRプログラム | |
| ステップ1:98°C - 2分 | |
| ステップ2:98°C - 20年代 | |
| ステップ3:65°C - 30年代 | |
| ステップ4:72°C - 45s | |
| ステップ 5: ステップ 2 に進み、合計 15 ~ 18 サイクルを作成します。 | |
| ステップ6:72°C - 3分 | |
| ステップ7:4 °C – ホールド |
表4:4Cクロマチン相互作用ライブラリ増幅、第1PCR。
| ネストされた PCR のセットアップ | |
| 最初の PCR からの DNA 断片 | 10 μL |
| PCRグレード水 | 20.25 μL |
| 10 μM特異的プライマー+P5 イルミナプライマー | 3.75 μL |
| 10 μM P7 イルミナプライマー | 3.75 μL |
| ヘラクサーゼII 5Xバッファー | 10 μL |
| 10 Mm dNTPs | 1.25 μL |
| ヘラクサーゼIIポリメラーゼ | 1 μL |
| 総量 | 50 μL |
| ネストされた PCR プログラム | |
| ステップ1:98°C - 2分 | |
| ステップ2:98°C - 20年代 | |
| ステップ3:65°C - 30年代 | |
| ステップ4:72°C - 45s | |
| ステップ 5: ステップ 2 に進み、合計 15 ~ 18 サイクルを作成します。 | |
| ステップ6:72°C - 3分 | |
| ステップ7:4 °C – ホールド |
表5:4Cクロマチン相互作用ライブラリ増幅、ネストPCR。
| 名前 | シーケンス (5'-3') |
| DS-Oct4-A | アトガタッケグッカッカッハクトガッカクトクトクトクトトゥマクタチャッカガッCG CTCTTCCGATCTTGCAアガタアターアターガアアッカアッカカッパッカガクチャグ |
| アメリカ-10月4日-A | TCTTGCAアガガタアクタアクサアッカアッカガッコッガック |
| DS-Oct4-B | アトガタッケグッカッカッハクトガッカクトクトクトクトトゥマクタチャッカガッCG CTCTTCCGATCTGTGTGTッカガッグGCTGGAGGGGGGCT |
| アメリカ-10月4-B | アカッグッグググガッググッカGCAGGGCT |
| DS-T-C | アトガタッケグッカッカッハクトガッカクトクトクトクトトゥマクタチャッカガッCG CTCTTCCGATCTCCGTCCCTGCACATTCGCAAGGAGC |
| US-T-C | ガタケクトッヒクトクトゥクトゥクトゥクタットGCACATTCGCCA |
| DS-T-D | アトガタッケグッカッカッハクトガッカクトクトクトクトトゥマクタチャッカガッCG CTCTTCCGGCTCTTGGGGGGGTGTCAGGGGGGGGGGGAGGGGAGGGGAG |
| 米国-T-D | GCTGAGGCTTGGGGGCCAAGGAGACC |
| アップ-4C | カグカガガッガッカッカッガ |
| アダップ-i1 | カーグカガガッガッガガガガツクガクトガクトガクトガクトカガガ CGTGTGCTCTTGCATC |
| アダップ-i2 | カーグカガガアッカガッカガガガカタカトCGGTGTGTTCAGA CGTGTGCTCTTGCATC |
| アダップ-i3 | カーグカガガアッガッカガガガガガッガッガテクターガガグッカガガガガ CGTGTGCTCTTGCATC |
| アダップ-i4 | カーグカガガッガッガッカガガトッガクトクトガクトッカガッカガガ CGTGTGCTCTTGCATC |
表6:4Cライブラリの準備に使用するプライマー
ディスカッション
吊り落とし培養法は、追加の増殖因子やサイトカインを必要とせず、予め所定数のmESCs5からEBの均質集団を再現的5に生成する。ここでは、EB分化モデルにおける系統特異的転写因子のエンハンサー・プロモーター接触を定量化するUMI-4Cアプローチから適応した定量4Cのプロトコルについて説明する。EB分化中にPou5f1およびT遺伝子のプロモーターに接触するクロマチン領域を動的に同定した。POU5f1は、EB分化中にダウンレギュレートされ、Pou5f1プロモーターとその遠位エンハンサーとの接触頻度が減少した。逆に、Eb分化中にTをアップレギュレートし、プロモーターとの接触周波数が減少する3つのエンハンサーを同定した(図3)。同定を確認するために、活性ヒストンマークH3K27acのクロマチン免疫沈降(ChIP)アッセイを24を行うことができるが、このヒストンマークがインアクティベート11の間に増強活性化と関連することが示されており、エンハンサーがこのマークを失う。
標準の4C技術は、特定のゲノム部位25のクロマチン接触プロファイルを調査するために広く使用されている。しかし、PCRフラグメントサイズの不均一性によってもたらされる偏りやPCR重複を区別できないため、このアプローチは、広範な正規化26、27、2828の後でも定量的に解釈することは困難です。26,27我々の定量4C法は、従来の4Cアプローチ23の制限を回避するために超音波処理およびネストライゲーション媒介PCRステップを用いて単一分子の定量を可能にするUMI-4C技術とほぼ同一である。しかし、一意の分子識別子を使用するUMI-4Cとは異なり、当社の定量4Cプロトコルは、超音波処理ステップによって生成された特異的DNA破断に基づいて単一分子の定量を可能にします。当社のプロトコルは、市販のDNAライブラリ調製キットと互換性を持ち、固有の分子識別子を持つプライマーの必要性を排除します。
私たちのプロトコルは考慮されるべきいくつかの重要なステップを含みます。古典的な4C法28と同様に、我々のプロトコルの重要な要因は、3C分子の調製中の消化および結紮の効率である。低い消化/結紮効率は、目的の断片との相互作用の複雑さを劇的に減少させ、その結果、分解能を低下させる可能性があります。先に説明した23のように、プロトコルのもう一つの重要なステップは、ライブラリ増幅のためのプライマーの設計である。第2のPCR反応プライマーは、尋問された制限部位から5〜15ntに配置されるべきである。75 nt シーケンス読み込みでは、マッピング用のキャプチャ長の残り 40 nt 以上が可能になります。最初のPCR反応で使用されるプライマーは、重なりのない第2プライマーの上流に設計されるべきであり、両方とも効率的なDNA増幅を確実にするのに十分な特異的であるべきである。多重化の場合、プライマーは、60〜65°Cの融解温度(Tm)を目指して、独立して設計する必要があります。また、他の3C技術については、定量4C法の分解能は、プロトコル25で用いられる制限酵素によって決定される。このプロトコルは、4 bp認識部位MboIを有する制限酵素を使用する。この酵素の最大分解能は約500bpですが、これは非常に遺伝子組み込みであり、めったに達成されません。別の制限は、同じ制限フラグメント内にある要素間で発生する相互作用は検出できないことです。さらに、1つの制限部位の距離で発生する相互作用は、未消化の背景と区別することはできません。結紮前の充填工程を使用すると、これらの相互作用の検出が可能になる可能性があります。
定量4Cは、標的座のクロマチン接触を尋問するのに理想的である。ただし、特定の PCR 増幅ステップでは、同時に調査できる遺伝子数が制限されます。標的遺伝子数を増やす方法は、PCRステップを多重化して複数のターゲットを同時に増幅することですが、実装前に使用されるプライマーの互換性とテストが必要です。プロモーターにおけるクロマチンアーキテクチャのグローバルな変化が望ましい場合、Hi-C、PC Hi-C、またはHiChIPのようなゲノム全体のアプローチは、より適切な29、30、3130,31であろう。29
開示事項
著者らは開示するものは何もない。
謝辞
F・ル・ディリー、R・シュタトゥーダーズ、グラーフ研究所のメンバーの皆さんに、助言と議論に感謝します。G.S.はマリー・スクロドフスカ・キュリー・フェローシップ(H2020-MSCA-IF-2016、miRStem)、T.V.Tはフアン・デ・ラ・シエルバポスドク・フェローシップ(MINECO、FJCI-2014-22946)によって支えられました。この研究は、第7回枠組みプログラムFP7(ERCシナジーグラント4Dゲノム、T.G.への助成金契約609989)、スペイン経済産業競争力省(MEIC)、EMBLパートナーシップ、セントロ・デ・エクセレンシア・セベロ・オチョア2013-2017およびCERCAプログラムジェネラリタ・デ・カタルーニャの下で欧州研究評議会によって支持されました。
資料
| Name | Company | Catalog Number | Comments |
| 0.1% EmbryoMax gelatin | EMD Millipore | ES-006-B | Cell culture |
| 0.25% Trypsin-EDTA | 25200072 | ||
| AMPure XP | Beckman Coulter | 10136224 | 4C/DNA purification |
| B27 supplement | Gibco | 17504044 | Cell culture |
| Beta-mercaptoethanol | Gibco | 31350010 | Cell culture |
| Bioruptor Pico | Diagencode | B01060010 | 4C/sonication |
| BSA | NEB | B9000S | 4C |
| CHIR99021 | Selleck Chemicals | S1263 | Cell culture |
| CIP | NEB | M0212 | 4C |
| cOmplete Protease Inhibitor Cocktail | Roche | 4693116001 | 4C |
| DMEM/F12 medium | Gibco | 11320033 | Cell culture |
| dNTP | NEB | N0447S | 4C |
| ESGRO Leukaemia Inhibitory Factor (LIF) | EMD Millipore | ESG1107 | Cell culture |
| Formaldehyde solution (37%) | Sigma | 252549-25ML | 4C |
| Glycin | Sigma | GE17-1323-01 | 4C |
| Glycogen | ThermoFischer | R0551 | 4C |
| Herculase II Fusion DNA polymerase | Agilent | 600675 | 4C |
| IGEPAL CA-630 | Sigma | I3021-50ML | 4C |
| Knockout DMEM | 10829018 | ||
| L-glutamine | Gibco | 25030081 | Cell culture |
| MboI | NEB | R0147M | 4C |
| MEM non-essential amino acids | Gibco | 11140050 | Cell culture |
| N2 supplement | Gibco | A1370701 | Cell culture |
| NEBNext DNA Library prep | NEB | E6040 | 4C |
| NEBuffer 2.1 | NEB | B7202S | 4C/digestion |
| Neurobasal medium | Gibco | 21103049 | Cell culture |
| PD0325901 | Selleck Chemicals | S1036 | Cell culture |
| Penicillin Streptomycin | Gibco | 15140122 | Cell culture |
| Proteinase K | NEB | P8107S | 4C |
| Qubit 4 Fluorometer | ThermoFischer | Q33238 | 4C |
| Qubit dsDNA HS Assay Kit | ThermoFischer | Q32851 | 4C |
| RNase A | ThermoFischer | EN0531 | 4C |
| Sodium pyruvate solution | Gibco | 11360070 | Cell culture |
| StemPro Accutase Cell Dissociation Reagent | Gibco | A1110501 | Cell culture |
| T4 DNA Ligase Reaction Buffer | NEB | B0202S | 4C |
| T4 DNA Ligase Reaction Buffer | NEB | M0202M | 4C |
参考文献
- Evans, M. J., Kaufman, M. H. Establishment in culture of pluripotential cells from mouse embryos. Nature. 292 (5819), 154-156 (1981).
- Martello, G., Smith, A. The nature of embryonic stem cells. Annual Review Cell and Developmental Biology. 30, 647-675 (2014).
- Martin, G. R. Isolation of a pluripotent cell line from early mouse embryos cultured in medium conditioned by teratocarcinoma stem cells. Proceedings of the National Academy of Science U. S. A. 78 (12), 7634-7638 (1981).
- Loh, K. M., Lim, B., Ang, L. T. Ex uno plures: molecular designs for embryonic pluripotency. Physiological Reviews. 95 (1), 245-295 (2015).
- Sheridan, S. D., Surampudi, V., Rao, R. R. Analysis of embryoid bodies derived from human induced pluripotent stem cells as a means to assess pluripotency. Stem Cells International. 2012, 738910(2012).
- Gaszner, M., Felsenfeld, G. Insulators: exploiting transcriptional and epigenetic mechanisms. Nature Reviews in Genetics. 7 (9), 703-713 (2006).
- Lenhard, B., Sandelin, A., Carninci, P. Metazoan promoters: emerging characteristics and insights into transcriptional regulation. Nature Reviews in Genetics. 13 (4), 233-245 (2012).
- Long, H. K., Prescott, S. L., Wysocka, J. Ever-Changing Landscapes: Transcriptional Enhancers in Development and Evolution. Cell. 167 (5), 1170-1187 (2016).
- Schoenfelder, S., Fraser, P. Long-range enhancer-promoter contacts in gene expression control. Nature Reviews in Genetics. 20 (8), 437-455 (2019).
- Spitz, F., Furlong, E. E. Transcription factors: from enhancer binding to developmental control. Nature Reviews in Genetics. 13 (9), 613-626 (2012).
- Creyghton, M. P., et al. Histone H3K27ac separates active from poised enhancers and predicts developmental state. Proceedings of the National Academy of Sciences U. S. A. 107 (50), 21931-21936 (2010).
- Heintzman, N. D., et al. Histone modifications at human enhancers reflect global cell-type-specific gene expression. Nature. 459 (7243), 108-112 (2009).
- Klemm, S. L., Shipony, Z., Greenleaf, W. J. Chromatin accessibility and the regulatory epigenome. Nature Reviews in Genetics. 20 (4), 207-220 (2019).
- Rada-Iglesias, A., et al. A unique chromatin signature uncovers early developmental enhancers in humans. Nature. 470 (7333), 279-283 (2011).
- Lettice, L. A., et al. Development of five digits is controlled by a bipartite long-range cis-regulator. Development. 141 (8), 1715-1725 (2014).
- Dekker, J., Rippe, K., Dekker, M., Kleckner, N. Capturing chromosome conformation. Science. 295 (5558), 1306-1311 (2002).
- de Wit, E., de Laat, W. A decade of 3C technologies: insights into nuclear organization. Genes and Development. 26 (1), 11-24 (2012).
- Lieberman-Aiden, E., et al. Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326 (5950), 289-293 (2009).
- Simonis, M., et al. Nuclear organization of active and inactive chromatin domains uncovered by chromosome conformation capture-on-chip (4C). Nature Genetics. 38 (11), 1348-1354 (2006).
- Splinter, E., de Wit, E., van de Werken, H. J., Klous, P., de Laat, W. Determining long-range chromatin interactions for selected genomic sites using 4C-seq technology: from fixation to computation. Methods. 58 (3), 221-230 (2012).
- Stadhouders, R., et al. Multiplexed chromosome conformation capture sequencing for rapid genome-scale high-resolution detection of long-range chromatin interactions. Nature Protocols. 8 (3), 509-524 (2013).
- van de Werken, H. J., et al. Robust 4C-seq data analysis to screen for regulatory DNA interactions. Nature Methods. 9 (10), 969-972 (2012).
- Schwartzman, O., et al. UMI-4C for quantitative and targeted chromosomal contact profiling. Nature Methods. 13 (8), 685-691 (2016).
- Tian, T. V., et al. Whsc1 links pluripotency exit with mesendoderm specification. Nature Cell Biology. 21 (7), 824-834 (2019).
- Chen, H., et al. Dynamic interplay between enhancer-promoter topology and gene activity. Nature Genetics. 50 (9), 1296-1303 (2018).
- Apostolou, E., et al. Genome-wide chromatin interactions of the Nanog locus in pluripotency, differentiation, and reprogramming. Cell Stem Cell. 12 (6), 699-712 (2013).
- de Wit, E., et al. The pluripotent genome in three dimensions is shaped around pluripotency factors. Nature. 501 (7466), 227-231 (2013).
- Krijger, P. H. L., Geeven, G., Bianchi, V., Hilvering, C. R. E., de Laat, W. 4C-seq from beginning to end: A detailed protocol for sample preparation and data analysis. Methods. , (2019).
- Mumbach, M. R., et al. HiChIP: efficient and sensitive analysis of protein-directed genome architecture. Nature Methods. 13 (11), 919-922 (2016).
- Rao, S. S., et al. A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159 (7), 1665-1680 (2014).
- Schoenfelder, S., Javierre, B. M., Furlan-Magaril, M., Wingett, S. W., Fraser, P. Promoter Capture Hi-C: High-resolution, Genome-wide Profiling of Promoter Interactions. Journal of Visualized Experiments. (136), e57320(2018).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved