このコンテンツを視聴するには、JoVE 購読が必要です。 サインイン又は無料トライアルを申し込む。
Method Article
脂質膜動態と膜タンパク質相互作用に対するユニークなプローブとしての中性子スピンエコー分光法
Erratum Notice
要約
本論文では、脂質膜の中性子スピンエコー(NSE)研究におけるサンプル調製、データ削減、データ解析のプロトコルについて述べる。脂質の重水素標識は、重要な生物学的プロセスが起こるメソスコピック長および時間スケール上の異なる膜動力学へのアクセスを可能にする。
要約
脂質二重層は細胞膜の主なマトリックスを形成し、栄養素交換、タンパク質膜相互作用、およびウイルスの芽出しのための主要なプラットフォームであり、他の重要な細胞プロセスの中で。効率的な生物学的活性のために、細胞膜は細胞とそのコンパートメントの完全性を維持するのに十分な剛性でありながら、タンパク質および機能ドメインなどの膜成分が拡散し相互作用するのに十分な流体であるべきである。弾性と流体膜の特性の微妙なバランス、および生物学的機能への影響は、膜の変形やタンパク結合イベントなどの主要な生物学的プロセスのメソスコピック長さと時間スケールに対する集合的な膜ダイナミクスのより良い理解を必要とします。このダイナミックレンジを効果的に探査できる技術の中には、中性子スピンエコー(NSE)分光法があります。重水素ラベリングと組み合わせることで、NSEは、曲げや厚さの変動だけでなく、選択された膜の特徴のメソスコピックダイナミクスに直接アクセスするために使用することができます。本論文では、NSE技術の簡単な説明を提供し、サンプル調製および重水素化スキームの詳細を含むリポソーム膜に関するNSE実験を行う手順と、データ収集と削減の手順を概説する。また、曲げ剛性率、面積圧縮率、面内粘度などの主要な膜パラメータを抽出するためのデータ解析方法も紹介しています。NSE研究の生物学的重要性を説明するために、NSEによってプローブされた膜現象の選択例、すなわち、膜曲げ剛性に対する添加剤の影響、膜の変動に及ぼすドメイン形成の影響、および膜-タンパク質相互作用の動的シグネチャについて考察する。
概要
細胞膜とその機能の理解は、ここ数十年で著しく進化してきました。細胞境界とハウス膜タンパク質1を定義する受動脂質二重層としての細胞膜の以前の見解は、細胞シグナル伝達、分子交換、およびタンパク質機能を含む重要な生物学的プロセスを調節する上で脂質二重層が重要な役割を果たす動的モデルへと変貌し、2、3、4、5、6と称する。細胞膜は非常に動的であり、常に再形成と分子再分配を受けているという認識は、膜7、8、9の平衡構造を超えた科学的探求を促している。したがって、生物学的およびバイオアインスパイアされた脂質膜の様々な動的モードを研究するために、複数のアプローチが開発されてきました。現在までに、これらの研究の大半は、主に拡散性分子運動10、11、12、13および巨視的形状変動14、15、16に焦点を当て、中間膜ダイナミクス、すなわち脂質分子の数10〜100sからなる脂質集合体の集合的変動を理解する上で大きなギャップを残している。これらのダイナミクスは、数十から数100Åの長さのスケールと数百nsに対する時間スケール(図1を参照)で発生します。これらのスケールでは、主要な生物学的活性が膜レベル17で行われます。これには、ウイルスの出芽18、チャネル・ギャティング19、および膜タンパク質相互作用20が含まれる。また、膜タンパク質21,22のエネルギーランドスケープは、タンパク質の立体構造変化(その調節的役割に必要)が集団膜変動のns時間スケール23で起こることを示し、細胞膜の生物学的機能におけるメソスコピックダイナミクスの重要性をさらに強調することを指摘する。本論文では、脂質膜における2つの主要なメソスコピック動的モード、すなわち曲げ変動と厚さ変動に焦点を当てています。
これらの変動モードを直接探査する際の主な課題は、標準分光法を用いて空間的スケールと時間スケールに同時にアクセスすることの難しさです。もう一つの課題は、直接接触技術が16を測定するために意図されているのと同じ変動に影響を与える可能性があるということです。これは、生物学的膜24,25の組成および構造の複雑さによってさらに悪化し、脂質ドメイン形成26、27、28、29、30および膜非対称性31、32、33を含む非均質な膜特徴を生じ、異なる膜特徴のダイナミクスを理解するための選択的プローブを要求する。幸いなことに、これらの課題は、本質的に必要な長さと時間スケールにアクセスする中性子スピンエコー(NSE)のような非侵襲的な中性子分光法法で克服することができ、さらにそれらの物理化学的環境34を変更することなく、選択的膜特徴の研究を可能にする。実際、ここ数年NSE分光法は、集団膜動態35のユニークで強力なプローブに進化しました。脂質膜に関するNSE研究の結果は、機械的36、37、粘弾性38、脂質膜の39の特性に新たな洞察を生み出し、生物学的機能40,41におけるその潜在的役割に新たな光を当てた。
NSE分光法は、メゼイ42が最初に提案した干渉計器の設計に基づいており、一連のスピンフリッパーと磁気コイルを使用して中性子が機器を横断する中性子スピンの歳差を制御します。この設計は、サンプル位置に対する磁界要素の磁気ミラーリングにかかっています(図1A)。これは、中性子と試料の間のエネルギー交換がない場合、中性子は、装置の最初と後半に反対方向に同じ数のスピン歳差を実行することを意味します(2つの歳差コイル間のπフリッパーに注意してください)。その結果、中性子の最終的なスピン状態は、初期状態に対して変化しないままである-スピンエコーと呼ばれる現象(図1Aの透明中性子を参照)。しかし、中性子がサンプルとエネルギー相互作用すると、エネルギー交換によって装置の後半のスピンの間圧数が変わり、最終的なスピン状態が異なる(図1Aを参照)。これは、この論文の後半で示されるように、偏光の損失として実験的に検出される。NSEテクニックの詳細については、専用の技術論文42、43、44、45を参照してください。
ここでは、NSEでアクセス可能な長さと時間スケールの概算を提供する簡単な説明を提示します。長さスケールは、達成可能な波ベクトル伝達の範囲によって決定され、Q = 4π sinθ/λ、2θは散乱角、λは中性子波長である。Qが分光計の第2アームの波長範囲と回転の程度によって設定されていることがわかります(図1Aを参照)。NSE分光計の典型的なQ-範囲は、〜0.02-2 Å-146、47、および最近のアップグレード48、49で0.01-4 Å-1まで、〜1-600 Åの空間スケールに対応しています。一方、アクセス可能な時間スケールは、磁気陽イオンコイル内で中性子が獲得した総歳差角(または位相)から計算され、50: と見なされます 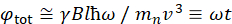 。この式では、tは、
。この式では、tは、 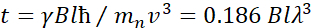
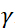 中性子ジャイロ磁性比、
中性子ジャイロ磁性比、  コイル長、および
コイル長、および 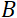 コイルの磁場の強さとして定義されるフーリエ時間です。フーリエ時間は、計測器の形状、磁場強度、中性子波長に厳密に依存する量であることを指摘する価値があります。例えば、波長=8Åの中性子
コイルの磁場の強さとして定義されるフーリエ時間です。フーリエ時間は、計測器の形状、磁場強度、中性子波長に厳密に依存する量であることを指摘する価値があります。例えば、波長=8Åの中性子 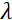 と
と  =1.2mと
=1.2mと 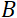 =0.4Tの計器設定を使用して、フーリエ時間はt~50nsと計算されます。 実験的には、フーリエ時間は、p景気後退コイルの電流(すなわち、磁場強度)を変化させることによって調整されるか、異なる中性子波長を使用して、〜1 ps〜100nsの典型的なNSE時間スケールをもたらす。しかし、NSE分光計の最近のアップグレードにより、より長いフーリエ時間へのアクセスが可能になりました。 オークリッジ国立研究所48のハインツ・マイヤー・ライプニッツ・ツェントルム51とSNS-NSE分光計のJ-NSE-フェニックス分光計では約~400ns、インスティトゥート・ラウ・ランゲビン(ILL)のIN15 NSE分光計では最大1,000nsまで。
=0.4Tの計器設定を使用して、フーリエ時間はt~50nsと計算されます。 実験的には、フーリエ時間は、p景気後退コイルの電流(すなわち、磁場強度)を変化させることによって調整されるか、異なる中性子波長を使用して、〜1 ps〜100nsの典型的なNSE時間スケールをもたらす。しかし、NSE分光計の最近のアップグレードにより、より長いフーリエ時間へのアクセスが可能になりました。 オークリッジ国立研究所48のハインツ・マイヤー・ライプニッツ・ツェントルム51とSNS-NSE分光計のJ-NSE-フェニックス分光計では約~400ns、インスティトゥート・ラウ・ランゲビン(ILL)のIN15 NSE分光計では最大1,000nsまで。
膜動態の長さと時間スケールへの直接アクセスに加えて、NSEは中性子体同位体感度52の固有の能力を有する。具体的には、生物系において最も豊富な元素である水素同位体と異なった相互作用をする中性子の能力は、プロトニウムが重水素によって置換される場合に、異なる中性子散乱長さ密度、34またはNSLD(屈折の光学インデックスに相当する)をもたらす。これにより、特定の膜の特徴を強調したり、他の特徴を隠すために一般的に使用されるコントラストバリエーションと呼ばれるアプローチが可能になります。コントラスト変動/一致の頻繁な適用は、水の置換です (NSLD = -0.56 × -0.56 × 10-6 Å-2)重水またはD2O (NSLD = 6.4 × 10-6 Å-2)を使用して、プロチアード脂質膜からの中性子信号を増幅します (NSLD ~ 0 × 10-6 Å-2)。このアプローチは、膜のヘッドグループ領域へのD2Oの浸透が膜の厚さ(図2A、左パネルを参照)および異なる脂質サブグループの位置をより高度なモデルが適用される53,54の正確な決定を可能にするので、膜構造の研究において非常に効果的である。本論文では、バイオミメティック膜の集合的ダイナミクスの研究におけるコントラスト変動の使用と、膜の特徴の選択に関する例をいくつか強調する。
ここでは、動的および機能的膜特性に関する独自の洞察を提供するNSEの有効性を、リポソーム懸濁液の形態で自立膜中質動態に重点を置いたモデルおよび生物学的に関連する脂質膜系に関するNSE研究の具体的な例を通して示されている。面内膜ダイナミクスのNSE測定では、読者は放牧発生率中性子スピンエコー分光法(GINSES)55、56および整列した多層膜スタック57、58、59、60の他の研究に関する専用の出版物と呼ばれる。
簡潔にするために、この論文は、1,2-ジミリストイル-sn-グリセロ-3-ホスホコリン(DMPC)および1,2-distearoyl-sn-グリセロ-3-ホスホコリン(DSPC)混合物の十分に研究されたドメイン形成、または相分離の脂質二重層系に示された3つの異なる膜重膜スキームを強調する。2つの脂質は、炭化水素鎖長(DSPCの18炭素/テールの14炭素/テール)とゲル流体転移温度(Tm、DMPC = 23°C対Tm、DSPC=55°C)の不一致が特徴です。この結果、DMPC:DSPC膜における、混合物63の上下遷移温度間の温度における横相分離が生じる。ここで考慮される重曲スキームは、リポソーム膜のNSE測定でアクセス可能なさまざまな動的モード、すなわち、曲げ変動、厚さ変動、および側面ドメインの選択的曲げ/厚さ変動を実証するために選択されます。DMPC:DSPCバイレイヤーは、DMPCとDSPCの商業的に入手可能なプロティエートおよび透過型の変異体を使用して、70:30のモル画分で調製されたDMPC:DSPC二重層について報告されます。すべてのサンプル調製ステップは、4 mLのリポソーム懸濁液に基づいており、D2Oでは、50mg/mLの脂質濃度を有し、全脂質質量のMtot= 200mgの試料に基づいている。
プロトコル
1. 実験に必要な重極化スキーム
- 曲げゆらゆら測定のために、D2 O(D 99.9%)またはD2O−バッファ(例えば、H2Oの代わりにD2Oで調製されたリン酸塩緩衝液)で完全にプロチオートリポソームを作る。完全にプロピテられたDMPC(C36H72NO8P)およびDSPC(C44H88NO8P)を
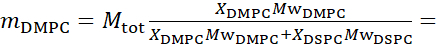 133.4 mgで使用し、 ここで、XDMPCとXDSPCは DMPC と DSPC のモル分率であり、ここではそれぞれ 0.7 と 0.3 に設定され、MwDMPCと MwDSPCはそれぞれ 677.9 g/mol と 790.1 g/mol で与えられるモル重量です。同様に、mDSPC = 66.6 mg.この重水素化スキームは、膜(NSLD~0×10-6 Å-2)と重水素化緩衝液(NSLD~6.4×10-6 Å-2)との散乱コントラストを高め、膜起振から信号を増幅する(図2A左パネル参照)。
133.4 mgで使用し、 ここで、XDMPCとXDSPCは DMPC と DSPC のモル分率であり、ここではそれぞれ 0.7 と 0.3 に設定され、MwDMPCと MwDSPCはそれぞれ 677.9 g/mol と 790.1 g/mol で与えられるモル重量です。同様に、mDSPC = 66.6 mg.この重水素化スキームは、膜(NSLD~0×10-6 Å-2)と重水素化緩衝液(NSLD~6.4×10-6 Å-2)との散乱コントラストを高め、膜起振から信号を増幅する(図2A左パネル参照)。 - 選択された横膜の特徴の曲げダイナミクスを測定するために、 例えば、位相分離DMPC:DSPC膜におけるマトリックスダイナミクスは、プロチッドDMPC(C36H72NO8P)および重水素化、DSPC-d83(C44H5NO 8 PD83、Mw873.7 g/mol)を使用し、m DMPC= 128.8 mgおよびmDSPC-d83mg=71mg= この重み付け方式は望ましくないDSPCリッチドメインからの散乱を最小にし、DMPCリッチマトリックスからの曲げ変動の選択的測定を可能にする(図2B中間を参照)。
注:特定のコントラストマッチングスキームに必要な最適な脂質重定を見つけるために、NIST中性子研究センター64が開発したものなど、利用可能なウェブベースの散乱長密度(SLD)計算機を利用します。これらのウェブベースのインターフェースは、脂質混合物の様々な程度と脂質のSLDの簡単な計算のためのユーザーフレンドリーなツールが装備されています。 - NSEの平均膜厚変動(横コントラストなし)の測定では、構成脂質の尾重化変異体を使用し、 すなわち、DMPC-d54(C36H18NO8PD 54、732.3 g/mol)およびDSPC-d70(C44H18NO8PD70、860.1g/mol)35、38、mDM-d54 = 133.0mgおよびm DSPC-d70mg=この造影スキーム(図2A、右パネル)は、脂質ヘッドグループ(NSLD~4.5×10-6Å-2)から、膜厚の変動の検出を可能にする重水素緩衝液(NSLD~6.6×10-6Å-2)とのコントラストマッチングにより、脂質ヘッドグループからの散乱信号を増幅する。
- 選択膜コンパートメントの厚さ変動研究の場合、 例えば、DMPCリッチマトリックスは、突起DMPC脂質を尾重数の類似体、すなわちDMPC-d54と置き換えることによってステップ1.2で説明した同じ戦略を使用し、DSPCリッチドメインが重水素化された緩衝液と対比され、一次散乱信号がテール・デレートされたマトリックスのヘッドグループ領域からである。
2. 押出用脂質懸濁液の調製
- サンプルの構成に応じて、サンプル内の各構成の質量を計算します。経験則として、複数の分子成分を含むサンプルの場合、成分の質量はモル分率、Xiによって重み付けされ
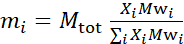 、Mtotが総質量である場合、ここで200mgに設定されるようなすべての成分にわたって正規化されます。異なる重水素化スキームを持つ DMPC-DSPC 脂質二重層については、上記の例を参照してください。
、Mtotが総質量である場合、ここで200mgに設定されるようなすべての成分にわたって正規化されます。異なる重水素化スキームを持つ DMPC-DSPC 脂質二重層については、上記の例を参照してください。 - デジタルセミマイクロバランスを使用して、計算された脂質の質量(タンパク質、ナノ粒子など)を計量し、バイアルまたは丸底フラスコに追加します - バイアルまたはフラスコを事前に計量することを忘れないでください。フード内部で手動で混合して重量を量った部品を溶解するために溶媒の1 mLを加えます。純粋な脂質サンプルの場合は、クロロホルムまたはエタノールを使用してください。追加の非脂質成分(例えば、ナノ粒子)を含むサンプルについては、すべての成分を分散させる共通の溶媒を選択してください。
- 少量の脂質量(<10mg)の場合は、ストック溶液を調製し、必要な量を混合物にピペットします。
注:以下に説明する溶剤乾燥工程を著しく遅くするので、過剰な量の溶剤を追加しないでください。
- 少量の脂質量(<10mg)の場合は、ストック溶液を調製し、必要な量を混合物にピペットします。
- 脂質溶液を、フードの内側に乾燥させ、不活性ガス(例えば、窒素、アルゴン)をバイアル中で緩やかに流し、バイアルをゆっくりと斜め回転させます。バイアルを傾けた位置に保ち、バイアル壁に乾燥した脂質の薄膜を作り、乾燥さえ可能にします。35°Cの水浴にバイアルを断続的に入れ、蒸発媒介冷却を回避し、溶媒の蒸発を遅くします。
- バイアルを約35°Cの真空オーブンに一晩入れ、残留溶媒を完全に除去します。不飽和脂質の場合は、不活性ガスで真空をパージして酸化を最小限に抑えます。
- 完全な溶媒除去を確実にするために、脂質乾燥後にバイアルを秤量し、測定量を超える材料量を超える余分な質量がないことを確認します。乾燥後に測定された質量からバイアルの質量を差し引いてこれを行います。余分な質量がある場合は、別の6時間真空下でサンプルを乾燥させます。必要に応じてこのプロセスを繰り返します。
- 脂質膜を4 mLのD2OまたはD2O-バッファで水和し、50mg/mLの脂質濃度を得た。DMPC-DSPC混合物のような高い転移温度を有する脂質の場合は、バッファーを転移温度(60°C)以上に加熱し、均一に混合を確保します。
注: NSE 実験では比較的大きなサンプル量(〜4 mL)が必要なため、必要なバッファーの半分、すなわち 2 mL を使用してサンプルの水分補給を行い、サンプルあたりの押出量を最小限に抑えることを検討してください(セクション3参照)。この場合は、バッファーポスト押出の残りの半分を追加します。押し出しに使用されるシリンジの容量は1 mLに制限されています。したがって、4 mLのバッファで水分補給を行うには、4セットの押し出しが必要になります。 - 水和した脂質溶液を、脂質膜が完全に溶解し、バイアルの壁に見えなくなるまで、渦液を混合する。この段階では、水和脂質はマルチラメラ小胞を形成し、ミクロンサイズのマルチラメラスタックを形成し、懸濁液は乳白色に見える。
- 脂質スタックの破壊を容易にし、多層減少を低減するために、完全に凍結するまで、水和脂質溶液のバイアルをラボグレードの冷凍庫(好ましくは-80°Cフリーザー)に入れ、その後、バイアルを35°Cの水浴に移して、脂質溶液が完全に解凍されるまで、5回の凍結/解凍サイクルを行います。解凍溶液を均質になるまで渦液。さらに 4 回繰り返します。
注:あるいは、ドライアイス浴はアセトンとドライアイスを組み合わせることで、急速な凍結に備えることができます。
3. 水和脂質溶液の押出
- 2つの膜支持体の間にポリカーボネート膜を使用して押出機のセットアップを組み立て、各側に2つの紙フィルターを追加して、サポートを追加します。ターゲットリポソームサイズに一致する孔径のポリカーボネート膜を使用します(NSE実験では一般的な孔径は50nmと100nmで、通常、直径100nmのリポソームは膜の変動を少なくすることができますが、曲率研究には50nmのリポソームが小さい。アセンブリを完成させる前にポリカーボネート膜が十分に伸張していることを確認し、外部押出機ケーシングを締めます。
- 気密性ガラス注射器を用いて、D2OまたはD2O-バッファの約0.3 mLを膜集合体を通して、ポリカーボネート膜を水和する。サンプル調製で使用されるのと同じバッファを利用します。少なくとも10分間放置し、サンプルを導入する前にバッファーを完全に吸い出します。
- 1 mL 気密シリンジに調製した脂質溶液を充填し、押出機装置の一端に挿入します。次に、反対の端に空のシリンジを挿入します。シリンジが押出機アセンブリに接続されたら、押出機ブロックに配置します。
- 押出に対して高温が必要な場合、高転移温度を有する飽和脂質の場合(例えば、DSPC、Tm=55°C)のように、押出機加熱ブロックを脂質転移温度より上(例えば、60°C)上に予熱し、熱いプレートに加熱ブロックを配置するか、または図3Aに示すように循環浴を使用して下さい。
注:このステップは、脂質の均一な混合を確実にし、押出中に極端な圧力を加えることを避けるために重要であり、ポリカーボネート膜を破裂させる可能性があります。低転移温度(<25°C)の脂質サンプルについては、室温で押出を行います。 - 脂質溶液を押し出すために、 図3Aに示すように、プログラム可能なシリンジポンプに設定された押出機をアルミニウム/スチールフレームに取り付けます。温度制御の押出の場合は、流体チャネルを備えたカスタムメイドの押出機ベースを追加し、循環水浴に取り付けます。
- メーカーのマニュアルに従って15-20押出サイクルを実行するようにシリンジポンプをプログラムします。押し出すと、脂質溶液の色が乳白色から透明オパールブルー(図3B、C)に変化し、期待通り可視光の波長よりも小さい最終的なリポソームサイズを示す。図3Aに示すシリンジポンプの種類については、以下の手順に従います。
- ポンプの設定を調整して開始します。 [Rate] ボタンを押したまま押し出し速度(50.99 mL/h)を入力し、[ 直径] ボタンを押して、シリンジ径(4.606mm)を入力します。画面上の各桁の下にある上向き矢印を使用して、その数字の値を変更します。
- サンプルシリンジを持つ押出機セットを右に置きます( 図3Aを参照)。 取り消し 灯が点灯するまで[取り消し]ボタンを押します。 [Start] を押して、サンプルが左(空)のシリンジに分配されるのを待ちます。
- サンプル(右)のシリンジが完全に空になる直前に [停止 ]ボタンを押します。分配されたボリュームを記録し、押し出しサイクルをプログラムするためにそれを使用します。フェーズ 1 (PH:01) が画面に表示されるまで 、レート ボタンを押したままにします。 ボリューム ボタンを押して、先ほど録音した分配ボリュームを入力します。このフェーズでは、取り消しライトがオフになっていることを確認します。
- もう一度 Rate ボタンを押し、右端の上矢印を使用してフェーズ 2 (PH:02) にアクセスします。 ボリューム を押して、先ほど記録した分配ボリュームと同じ値を入力します。このフェーズでは、取り消しライトが点灯するまで [取り消し] ボタンを押します。
- このサイクルを繰り返す場合は 、Rate ボタンをもう一度押し、右端の上矢印を使用してフェーズ 3 (PH:03) にアクセスします。LP:SE が画面に表示されるまで 音量 ボタンを押し、20に設定します。これは、ポンプが実行するループまたは繰り返しの数です。最後に 、Rate ボタンを押し、フェーズ4(PH:04)にアクセスし、 音量 ボタンを押して 停止 機能に到達します。ポンプは自動押し出し用に設定されました。
- [開始]を押して押し出しサイクルを開始します。
- 押し出された脂質懸濁液を含むシリンジをクリーンバイアルで空にし、保管または測定の準備を行います。高融解温度の脂質サンプルの場合、測定されるまで流体相転移の上にサンプルを保存します。それ以外の場合は、室温でサンプルを保持します。
- 凍結すると小胞が破裂するので、押し出されたサンプルを凍結しないでください(サスペンションは再び乳白色になります)。
4. サンプルのNSE測定と収集したデータの削減
- NSE実験の前に、適切なサンプル品質を確保するために利用可能な方法を使用してステップ3.7からの押し出しリポソームサンプルを特徴付ける。NSE実験のためのリポソーム懸濁液の品質を評価するために使用できる潜在的な炭化法のリストは、例えば、サイズ分布、多層性、横膜構造、議論のセクションに含まれています。
- 実験に必要なQ範囲と対応する計測器の設定を決定します。脂質二重層の曲げ剛性測定には、Q範囲~(0.04 - 0.2) Å-1を使用します。膜の厚さ変動の研究では、膜の厚さ35、66、67に対応する~(0.04 -0.2) Å-1のQ範囲を使用します。
注: 実験開始前に、実験の設定について計測器の科学者と話し合ってください。前述のように、選択的に重膜のように散乱信号の事前情報が利用できない場合は特に、試料のSANS特性評価が必要である。あるいは、NSE計測器上の限られたQ範囲で静的(回折とも呼ばれる)測定を実行し、SANSと比較してそのような測定にかかる時間が大幅に長くなるという点に注意してください。 - 注射器または転写ピペットを使用して、NSEビームラインで入手可能な指定サンプルセルに押し出されたリポソーム懸濁液をロードします。標準的なNSEサンプルセルの厚さは1、2、3、4mmです。不コヒーレントなバックグラウンド信号を妥当な強度に保ちながら散乱信号を最適化する方法で、細胞の厚さを選択します。
注: 経験則として、重水素化バッファー内のプロトリュート リポソームに 1 mm または 2 mm のパス長を持つサンプル セルを使用してください。重体のレベルが高いリポソーム(例えば、尾コントラストマッチリポソームまたは単一のプロティアードリーフレットを持つアサイムメトリックリポソーム)の場合は、より厚いサンプルセル(例えば、3または4mmのパス長)を使用して、サンプルがより多く利用可能な場合は、コストがかかる場合があります。 - バッファーに対して同じサンプル セルを準備します。リポソーム懸濁液と同様の緩衝液を使用する。バッファーの測定は、強度の正規化とバックグラウンド(BKG)補正に必要です。
- サンプルセルをNSE分光計のサンプルホルダーに入れ、測定実行をプログラムし、エコーデータを収集します。初めてNSEユーザーが使用する場合は、測定のプログラミングについて計測器の科学者に相談してください。
- データ削減に必要な 2 つの追加の測定セットを実行します: 解像度 (R) と伝送 (T) 測定。
- 同じ設定で実行する弾性散乱基準(例えば、炭素)で解像度(R)測定を行います。すなわち、サンプルおよびバッファーの測定値と同じ波形とフーリエ倍。
- サンプルとバッファで伝送(T)測定を行い、送信された中性子ビームの強度を計算します(下記のステップ4.9を参照)。透過は、開いたビームの中性子数で割ったサンプルまたはバッファーからの中性子数の比率(つまり、空のサンプル位置)として計算されます。
- 測定を行うNSE分光計専用のデータ削減ソフトウェアを使用して、収集したデータを削減します。
注: 分光計が異なると、ソフトウェアやユーザーインターフェイスが異なる場合があります。以下は、データ分析と可視化環境(DAVE)を用いたNSEデータ削減の例です。68 NIST中性子研究センターでNSE分光計用に特別に書かれたソフトウェア。- DAVEソフトウェアを開き、データ削減メニューから [NSEデータ を削減]を選択します。いくつかのポップアップ ウィンドウが表示されます。
- ファイルメニューから.echoファイルを開いて、異なるQ値でデータファイルをアップロードします。これらのファイルは、スピンエコー信号を持つ生データファイルに対応し、ファイル名に拡張子.echoを持っています。ファイルのアップロードが完了すると、使用可能なデータセットの下にファイルが表示されます。
- 選択したファイルを右クリックし、対応する測定に従ってラベルを付けます。例えば、サンプル、セル(空のセルまたはバッファの場合)、または解像度。
- 検出器のピクフルを2 x 2にグループ化し、[ データセット ]タブを使用して信号対雑音比を改善します。すべてのファイルに同じビン分割を適用します。つまり、解像度、セル、およびサンプル。
- すべてのピクセルグループのデータを検査し、キーボードのmキーを押して、信号の悪い方(図4Bを参照)をマスクします。ポップアップ ウィンドウにアクセスするには、Enterキーを押して、Fourier 回またはその後のすべてのフーリエ時間に同じマスクを適用します。また、データの削減中の任意の時点で、個々のピクセルに適用することもできます。マスクされたピクセルは緑色に変わります。
- 収集されたデータがエコー信号、すなわち位相電流の点でコサイン関数の形を、各検出器ピクセルにわたって確認します( 図4Aを参照)。
注: 位相電流は中性子スピンの後乗角に比例します。したがって、 図 4Aに示すように、位相電流を位相角度として表すのが一般的です。パルス発生源の測定では、データに追加の飛行時間が適用され、中性子パルス内の入射中性子波長の関数としてエコー信号が得られます。 - まず、解像度ファイルを取り付けます。アップロードしたファイルの一覧から解像度のファイルを選択し、ファイルを右クリックします。ポップアップメニューから「 フィット操作:エコー(解像度)を調整」を 選択します。
- エコー信号の適合性が、ステップ 4.8 で必要なパラメータ Aを含む多くのフィッティングパラメータを生成することを確認します。適合値は、次の式を使用して自動的に実行されます。
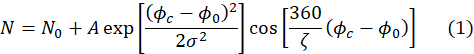
ここで、ζエコー信号の周期(すなわち、図4Aのコサイン関数)は、入射中性子ビームの平均波長と波長広がりによって決定されるガウスエンベロープの幅σ、Φcは位相電流、及びΦ0は中性子50が経験するフィールドパスに依存するエコーポイントである。サンプルに関する物理情報は、式(1)のコサイン関数の振幅Aに符号化される。
注: ガウスエンベロープの幅は、計測器の科学者があらかじめ定義した値に基づいて決まるので、変更しないでください。他のパラメータは、各ピクセルに対する特定のエコー信号に適合する変数です。 - 各ピクセルをクリックして適合結果を検査し、結果として得られる適合パラメータ、適合の品質、適合度の平均平方偏差を表示します。検出器全体で各継ぎ手パラメータに関連付けられたエラーを検査するには、[ イメージ オプション] を選択し、目的の継ぎ手パラメータを選択します。これにより、各ピクセルに対するフィッティング パラメータの値を持つマップが生成されます。検出器の画像を右クリックします。選択したフィッティングパラメータのエラーバーマップを示すポップアップウィンドウが表示されます。
- 特定のピクセルに対する適合が不十分な場合(例えば、大きな誤差範囲を持つパラミーターを適合させる)、その特定のピクセルの上に信号を再フィットします。そのピクセルを選択し、[フィット]タブを押して、[ピクセルの調整]を押します。「フィッティング」タブでフェーズ(Φ0)と期間(ζ)の新しい開始パラメータを入力して、より満足のいくフィット感を得ます。
注: 適合相をフーリエ時間の関数としてプロットすると便利です。これを行うには、メインプロットウィンドウに移動し、 フィット相対フーリエ時間を選択します。このプロットは、滑らかで連続的である必要があります。このプロットの不連続性を検査し、対応するピクセルを再調整します。
- アップロードされたファイルリストとラベル付きファイルリストから対応するファイルを選択して、サンプルファイルまたはセルファイルを削減します。
- 手順 4.7.5 で説明したように、すべてのピクセルを検査し、統計情報が正しくないものをマスクします。
- ファイルを右クリックし、[ フィット操作: フェーズのインポート (サンプル、セル)]を選択します。これにより、フェーズと適用されたマスクが解像度ファイルからインポートされます。
- 解像度ファイル(手順 4.7.8-4.7.10)で前述したのと同じ手順を使用してエコー信号を適合します。サンプルファイルとセルファイルのフィッティングでは、解像度フィットから読み込んだ期間とエコーフェーズポイントの値を変更しないでください。これらのパラメータはインストゥルメンタルの設定に依存し、サンプルによって異なるべきではありません。
- データ削減に進む前に、すべてのデータ ファイルに対してビーム中心を入力します。データ ファイルを選択し、[ 一般 ]タブに移動し、X と Y の梁中心の値を入力します。これらの値は、実験中に記録されます。
- サンプル、セル、解像度の各ファイルに適合したら、後でデータ分析と解釈に使用する正規化された中間スキャッタリング関数を計算します。これを行うには、適合ファイルのリストから削減するサンプルファイルを右クリックし、ポップアップメニューから 「I(Q)を計算 」を選択します。ウィンドウが表示され、解像度とセル (バッファー) ファイルのエントリオプションと Q アークの数 (ステップ 4.9 を参照) が表示されます。必要な情報をすべて入力したら 、[OK]ボタン を押します。結果は新しいウィンドウに表示されます。
注:データ削減は、正規化された中間散乱関数69を得るために、次の式に従って行われます。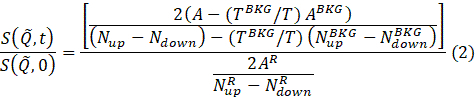
ここで、Tはフーリエ時間、NアップとNダウン はノンスピンフリップおよびスピンフリップ構成(それぞれπ/2フリッパーオフとπフリッパーオフとオンで測定)の中性子数であり、上付き文字BKGとRは、それぞれステップ4.4と4.6で定義されているように、背景と解像度の測定値に対応します。なお、ビーム偏光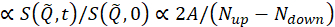 は、このように中性子と試料との間のエネルギー交換によるスピン状態の変化が、偏光(一体から)の低下として検出される。
は、このように中性子と試料との間のエネルギー交換によるスピン状態の変化が、偏光(一体から)の低下として検出される。
- 最後に、図4Bに示すように検出器のピクセルをQ-アークにグループ化し、正規化された中間散乱関数のQ-依存性を求める、S(Q、t)/S(Q,0)。 これは、技術的にはデータビニングと呼ばれ、サンプルからのカウント統計とグループ化されたピクセル上のデータの予想標準偏差を考慮して、慎重に行う必要があります。
- サンプルを強く散乱する場合は、結果として得られた中間散乱関数S(Q、t)/S(Q,0)上で合理的な誤差バーを維持しながら、検出器をより多くのQアークに分割します。 これにより、Q データポイントが多くなり、以下で説明するデータ分析手順に重要です。サンプルを弱く散乱させる場合、過度のビン分割は減衰信号の不良、すなわちS(Q、t)/S(Q,0)の大きな誤差範囲で発生し、大きな不確実性を生じる可能性があることに注意してください。
5. データ分析と解釈
- 正規化された中間散乱関数に適合する、S(Q、t)/S(Q,0)は、上記のデータ減少から得られた伸縮指数関数に2/3 70の伸縮指数関数を行う。
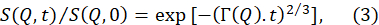
注: これらの適合の例は 、図 5B に示されています。S(Q,t) / S(Q,0) を式 (3) に適合すると、Q依存緩和率Γ(Q) が得られます。 - Qの関数としてΓ(Q)をプロットし、適切なモデルに適合して、関連する膜パラメータを抽出します。
結果
ベンド変動にアクセスするNSE研究は、通常、~(0.04 - 0.2)Å-1のQ範囲で行われます。このQ範囲は、膜の厚さとリポソーム半径の中間長さスケールに対応し、曲げダイナミクスが支配的です。拡張されたQ-範囲を超える測定は、リポソーム拡散および膜内動態を含む追加の動的モードへのアクセスを提供することができる。NSEがアクセスする膜動態のクロスオーバーの詳細については、こ?...
ディスカッション
NSEは、様々な条件下での脂質膜のメソスコピックダイナミクスを測定する上で強力かつユニークな技術です。NSEの有効利用は、サンプルの品質、中性子のコントラスト、および特定のサンプルに対してプローブ可能なアクセス可能なダイナミクスの範囲に依存します。したがって、NSE実験を成功させ、高品質なデータを収集するには、いくつかの重要なステップが必要です。NSE実験中に中性?...
開示事項
著者らは利益相反を宣言せず、開示するものは何もない。
謝辞
R.アシュカーはM.ナガオ、L.-Rに感謝します。スティンタシウ、およびP.ゾルニエツクは、多くの有用な議論とそれぞれのビームライン上のNSE実験で頻繁に支援を行う。著者らは、NISTおよびORNLにおける中性子スピンエコー分光計の使用を認めている。NISTのNSE分光計は、国立標準技術研究所と国立科学財団とのパートナーシップである高解像度中性子散乱センターによって支えられている。DMR-1508249.ORNLのスマレーション中性子源のNSE分光計は、米国エネルギー省基礎エネルギー科学局の科学ユーザー施設部門によって支えられている。オークリッジ国立研究所は、米国DOE契約No.の下でUT-Battelle,LLCによって管理されています。DE-AC05-00OR22725.
資料
| Name | Company | Catalog Number | Comments |
| Chloroform (biotech grade) | Sigma Aldrich | 496189 | Biotech. grade, ≥99.8%, contains 0.5-1.0% ethanol as stabilizer |
| Circulating water bath | Julabo | SE-12 | Heating Circulator with smart pump, programmable temperature settings, and external sensor connection for measurement and control |
| Deuterium Oxide | Cambridge Isotopes Laboratories | DLM-4 | Deuterated water; Heavy water (D2O) (D, 99.9%) |
| Digital Semi-Microbalance | Mettler Toledo | MS105 | Semi-micro balance with 120 g capacity, 0.01 mg readability, high resolution weighing cell, ergonomic doors, and pipette-check application |
| Ethanol (molecular biology grade) | Sigma Aldrich | E7023 | 200 proof ethanol for molecular biology applications |
| Glass Pipets | VWR | 36360-536 | Disposable Soda Lime glass Pasteur pipets |
| Glass Vials | Thermo Scientific | B7990-1 | Borosilicate glass vials with PTFE/Silione septum caps |
| Lab grade freezer | Fisher Scientific | IU2886D | Ultra-low temprature freezer (-86 to -50 C) for long-term storage of lipids and proteins |
| Lipids (protaited or perdeuterated) | Avanti Polar Lipids | varies by lipid | Lipids can be purchased from Avanti in powder form or in a chloroform solution with the required amounts and deuteration schemes. |
| Millipore water purifier | Millipore Sigma | ZRQSVP3US | Direct-Q® 3 UV Water Purification System which deliver both pure and ultrapure water with a built-in UV lamp to reduce the levels of organics for biological applications |
| Mini Extruder Set | Avanti Polar Lipids | 610020 | Mini-extruder set includes mini-extruder, heating block, 2 GasTight Syringes, and 2 O-rings, Polycarbonate Membranes, and Filter Supports |
| Quick Connect Fittings | Grainger | 2YDA1 and 2YDA7 | Push-button tube fittings for QuickConnect water circulation applications, e.g. high temperature vesicle extrusion |
| Syringe Pump | SyringePump.com | New Era-1000 | Fully programmable syringe pump for infusion and withdrawal; programs up to 41 pumping phases with adjustable pumping rates, dispensed volumes, and extrusion cycles |
| Ultrasonic bath | Fisher Scientific | CPX2800 | Temperature controlled ultra sonic bath with programmable functionality for degassing and ultrasonic applications |
| Vacuum Oven | Thermo Scientific | 3608 | 0.7 cu ft vaccum oven with built-in-high-limit thermostat guards against overheating |
| Vortex Mixer | Fisher Scientific | 02-215-414 | Variable speed, analog control that allows low rpm start-up for gentle shaking or high-speed mixing for vigorous vortexing of samples |
参考文献
- Singer, S. J., Nicolson, G. L. The fluid mosaic model of the structure of cell membranes. Science. 175 (4023), 720-731 (1972).
- Andersen, O. S., Koeppe, R. E. Bilayer thickness and membrane protein function: an energetic perspective. Annual Review of Biophysics and Biomolecular Structure. 36, 107-130 (2007).
- Lundbæk, J. A., Collingwood, S. A., Ingólfsson, H. I., Kapoor, R., Andersen, O. S. Lipid bilayer regulation of membrane protein function: gramicidin channels as molecular force probes. Journal of The Royal Society Interface. 7 (44), 373-395 (2010).
- Bradley, R. P., Radhakrishnan, R. Curvature-undulation coupling as a basis for curvature sensing and generation in bilayer membranes. Proceedings of the National Academy of Sciences of the United States of America. 113 (35), 117-124 (2016).
- Perozo, E., Cortes, D. M., Sompornpisut, P., Kloda, A., Martinac, B. Open channel structure of MscL and the gating mechanism of mechanosensitive channels. Nature. 418 (6901), 942-948 (2002).
- Jensen, M. &. #. 2. 1. 6. ;., Mouritsen, O. G. Lipids do influence protein function-the hydrophobic matching hypothesis revisited. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1666 (1-2), 205-226 (2004).
- Rajendran, L., Simons, K. Lipid rafts and membrane dynamics. Journal of Cell Science. 118 (6), 1099-1102 (2005).
- Katchalsky, A., Spangler, R. Dynamics of membrane processes. Quarterly Reviews of Biophysics. 1 (2), 127-175 (1968).
- Rheinstädter, M. C. Collective molecular dynamics in proteins and membranes (Review). Biointerphases. 3 (2), 83-90 (2008).
- Fujiwara, T., Ritchie, K., Murakoshi, H., Jacobson, K., Kusumi, A. Phospholipids undergo hop diffusion in compartmentalized cell membrane. The Journal of Cell Biology. 157 (6), 1071-1082 (2002).
- Hac, A. E., Seeger, H. M., Fidorra, M., Heimburg, T. Diffusion in two-component lipid membranes--a fluorescence correlation spectroscopy and monte carlo simulation study. Biophysical Journal. 88 (1), 317-333 (2005).
- Heinrich, M., Tian, A., Esposito, C., Baumgart, T. Dynamic sorting of lipids and proteins in membrane tubes with a moving phase boundary. Proceedings of the National Academy of Sciences of the United States of America. 107 (16), 7208-7213 (2010).
- Hormel, T. T., Kurihara, S. Q., Brennan, M. K., Wozniak, M. C., Parthasarathy, R. Measuring lipid membrane viscosity using rotational and translational probe diffusion. Physical Review Letters. 112 (18), 188101 (2014).
- Dimova, R. Recent developments in the field of bending rigidity measurements on membranes. Advances in Colloid and Interface Science. 208, 225-234 (2014).
- Bassereau, P., Sorre, B., Lévy, A. Bending lipid membranes: Experiments after W. Helfrich's model. Advances in Colloid and Interface Science. 208, 47-57 (2014).
- Monzel, C., Sengupta, K. Measuring shape fluctuations in biological membranes. Journal of Physics D: Applied Physics. 49 (24), 243002 (2016).
- Deserno, M. Mesoscopic membrane physics: concepts, simulations, and selected applications. Macromolecular Rapid Communications. 30 (9-10), 752-771 (2009).
- Reynwar, B. J., et al. Aggregation and vesiculation of membrane proteins by curvature-mediated interactions. Nature. 447 (7143), 461-464 (2007).
- Haswell, E. S., Phillips, R., Rees, D. C. Mechanosensitive channels: what can they do and how do they do it. Structure. 19 (10), 1356-1369 (2011).
- Phillips, R., Ursell, T., Wiggins, P., Sens, P. Emerging roles for lipids in shaping membrane-protein function. Nature. 459 (7245), 379-385 (2009).
- Dill, K. A., Chan, H. S. From Levinthal to pathways to funnels. Nature Structural Biology. 4 (1), 10-19 (1997).
- Henzler-Wildman, K., Kern, D. Dynamic personalities of proteins. Nature. 450 (7172), 964-972 (2007).
- Grimaldo, M., Roosen-Runge, F., Zhang, F., Schreiber, F., Seydel, T. Dynamics of proteins in solution. Quarterly Reviews of Biophysics. 52, 7 (2019).
- Lyman, E., Hsieh, C. -. L., Eggeling, C. From dynamics to membrane organization: experimental breakthroughs occasion a "modeling manifesto". Biophysical Journal. 115 (4), 595-604 (2018).
- Arriaga, L. R., et al. Dissipative curvature fluctuations in bilayer vesicles: Coexistence of pure-bending and hybrid curvature-compression modes. The European Physical Journal. E, Soft Matter. 31 (1), 105-113 (2010).
- Honerkamp-Smith, A. R., Veatch, S. L., Keller, S. L. An introduction to critical points for biophysicists; observations of compositional heterogeneity in lipid membranes. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1788 (1), 53-63 (2009).
- Veatch, S. L., Keller, S. L. Organization in lipid membranes containing cholesterol. Physical Review Letters. 89 (26), 268101 (2002).
- Heberle, F. A., et al. Bilayer thickness mismatch controls domain size in model membranes. Journal of the American Chemical Society. 135 (18), 6853-6859 (2013).
- Nickels, J. D., et al. The in vivo structure of biological membranes and evidence for lipid domains. PLOS Biology. 15 (5), 2002214 (2017).
- Simons, K., Ikonen, E. Functional rafts in cell membranes. Nature. 387 (6633), 569-572 (1997).
- van Meer, G., Voelker, D. R., Feigenson, G. W. Membrane lipids: where they are and how they behave. Nature Reviews. Molecular Cell Biology. 9 (2), 112-124 (2008).
- Liu, S. -. L., et al. Orthogonal lipid sensors identify transbilayer asymmetry of plasma membrane cholesterol. Nature Chemical Biology. 13, 268 (2016).
- Rothman, J., Lenard, J. Membrane asymmetry. Science. 195 (4280), 743-753 (1977).
- Ashkar, R., et al. Neutron scattering in the biological sciences: progress and prospects. Acta Crystallographica Section D. 74 (12), 1129-1168 (2018).
- Woodka, A. C., Butler, P. D., Porcar, L., Farago, B., Nagao, M. Lipid bilayers and membrane dynamics: insight into thickness fluctuations. Physical Review Letters. 109 (5), 058102 (2012).
- Chakraborty, S., et al. How cholesterol stiffens unsaturated lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (36), 21896-21905 (2020).
- Arriaga, L. R., et al. Stiffening effect of cholesterol on disordered lipid phases: a combined neutron spin echo + dynamic light scattering analysis of the bending elasticity of large unilamellar vesicles. Biophysical Journal. 96 (9), 3629-3637 (2009).
- Nagao, M., Kelley, E. G., Ashkar, R., Bradbury, R., Butler, P. D. Probing elastic and viscous properties of phospholipid bilayers using neutron spin echo spectroscopy. The Journal of Physical Chemistry Letters. 8 (19), 4679-4684 (2017).
- Kelley, E. G., Butler, P. D., Ashkar, R., Bradbury, R., Nagao, M. Scaling relationships for the elastic moduli and viscosity of mixed lipid membranes. Proceedings of the National Academy of Sciences of the United States of America. 117 (38), 23365-23373 (2020).
- Rickeard, B. W., et al. Transverse lipid organization dictates bending fluctuations in model plasma membranes. Nanoscale. 12 (3), 1438-1447 (2020).
- Nickels, J. D., et al. Mechanical properties of nanoscopic lipid domains. Journal of the American Chemical Society. 137 (50), 15772-15780 (2015).
- Mezei, F. Neutron spin echo: A new concept in polarized thermal neutron techniques. Zeitschrift für Physik A Hadrons and Nuclei. 255 (2), 146-160 (1972).
- Hayter, J. B., Penfold, J. Neutron spin-echo integral transform spectroscopy. Zeitschrift für Physik B Condensed Matter. 35 (2), 199-205 (1979).
- Monkenbusch, M., Richter, D., Imae, T., Kanaya, T., Furusaka, M., Torikai, N. . Neutrons in Soft Matter. , 147-182 (2011).
- Pynn, R., Mezei, F., Pappas, C., Gutberlet, T. . Neutron Spin Echo. , 159-177 (2003).
- Holderer, O., et al. The JCNS neutron spin-echo spectrometer J-NSE at the FRM II. Measurement Science and Technology. 19 (3), 034022 (2008).
- Schleger, P., et al. The long-wavelength neutron spin-echo spectrometer IN15 at the Institut Laue-Langevin. Physica B: Condensed Matter. 241-243, 164-165 (1997).
- Holderer, O., Zolnierczuk, P., Pasini, S., Stingaciu, L., Monkenbusch, M. A better view through new glasses: Developments at the Jülich neutron spin echo spectrometers. Physica B: Condensed Matter. 562, 9-12 (2019).
- Farago, B., et al. The IN15 upgrade. Neutron News. 26 (3), 15-17 (2015).
- Ashkar, R. Selective dynamics in polymeric materials: Insights from quasi-elastic neutron scattering spectroscopy. Journal of Applied Physics. 127 (15), 151101 (2020).
- Pasini, S., Holderer, O., Kozielewski, T., Richter, D., Phoenix Monkenbusch, M. J-NSE- Phoenix, a neutron spin-echo spectrometer with optimized superconducting precession coils at the MLZ in Garching. Review of Scientific Instruments. 90 (4), 043107 (2019).
- Svergun, D. I., Koch, M. H. J., Timmins, P. A., May, R. P. . Small Angle X-Ray and Neutron Scattering from Solutions of Biological Macromolecules. , (2013).
- Eicher, B., et al. Joint small-angle X-ray and neutron scattering data analysis of asymmetric lipid vesicles. Journal of Applied Crystallography. 50 (2), 419-429 (2017).
- Heberle, F. A., et al. Model-based approaches for the determination of lipid bilayer structure from small-angle neutron and X-ray scattering data. European Biophysics Journal. 41 (10), 875-890 (2012).
- Jaksch, S., Koutsioubas, A., Mattauch, S., Holderer, O., Frielinghaus, H. Long-range excitations in phospholipid membranes. Chemistry and Physics of Lipids. 225, 104788 (2019).
- Jaksch, S., et al. Influence of ibuprofen on phospholipid membranes. Physical Review E. 91 (2), 022716 (2015).
- Armstrong, C. L., et al. Effect of cholesterol on the lateral nanoscale dynamics of fluid membranes. European Biophysics Journal. 41 (10), 901-913 (2012).
- Rheinstädter, M. C., Häußler, W., Salditt, T. Dispersion relation of lipid membrane shape fluctuations by neutron spin-echo spectrometry. Physical Review Letters. 97 (4), 048103 (2006).
- Armstrong, C. L., Häußler, W., Seydel, T., Katsaras, J., Rheinstädter, M. C. Nanosecond lipid dynamics in membranes containing cholesterol. Soft Matter. 10 (15), 2600-2611 (2014).
- Nickels, J. D., et al. Lipid rafts: buffers of cell membrane physical properties. The Journal of Physical Chemistry B. 123 (9), 2050-2056 (2019).
- Michonova-Alexova, E. I., Sugár, I. P. Component and state separation in DMPC/DSPC lipid bilayers: a Monte Carlo simulation study. Biophysical Journal. 83 (4), 1820-1833 (2002).
- Sugár, I. P., Thompson, T. E., Biltonen, R. L. Monte Carlo simulation of two-component bilayers: DMPC/DSPC mixtures. Biophysical Journal. 76 (4), 2099-2110 (1999).
- Mabrey, S., Sturtevant, J. M. Investigation of phase transitions of lipids and lipid mixtures by sensitivity differential scanning calorimetry. Proceedings of the National Academy of Sciences. 73 (11), 3862-3866 (1976).
- . Neutron activation and scattering calculator Available from: https://www.ncnr.nist.gov/resources/activation/ (2021)
- Scott, H. L., et al. On the mechanism of bilayer separation by extrusion, or why your LUVs are not really unilamellar. Biophysical Journal. 117 (8), 1381-1386 (2019).
- Ashkar, R., et al. Tuning membrane thickness fluctuations in model lipid bilayers. Biophysical Journal. 109 (1), 106-112 (2015).
- Carrillo, J. -. M. Y., Katsaras, J., Sumpter, B. G., Ashkar, R. A computational approach for modeling neutron scattering data from lipid bilayers. Journal of Chemical Theory and Computation. 13 (2), 916-925 (2017).
- Azuah, R. T. DAVE: a comprehensive software suite for the reduction, visualization, and analysis of low energy neutron spectroscopic data. Journal of Research of the National Institute of Standards and Technology. 114 (6), 341-358 (2009).
- Van Hove, L. Correlations in space and time and born approximation scattering in systems of interacting particles. Physical Review. 95 (1), 249-262 (1954).
- Zilman, A. G., Granek, R. Undulations and dynamic structure factor of membranes. Physical Review Letters. 77 (23), 4788-4791 (1996).
- Kelley, E. G., Butler, P. D., Nagao, M. . Collective dynamics in model biological membranes measured by neutron spin echo spectroscopy. , 131-176 (2019).
- Zheng, Y., Michihiro, N., Dobrin, P. B. Bending elasticity of saturated and monounsaturated phospholipid membranes studied by the neutron spin echo technique. Journal of Physics: Condensed Matter. 21 (15), 155104 (2009).
- Sharma, V. K., Qian, S. Effect of an antimicrobial peptide on lateral segregation of lipids: a structure and dynamics study by neutron scattering. Langmuir. 35 (11), 4152-4160 (2019).
- Boggara, M. B., Faraone, A., Krishnamoorti, R. Effect of pH and Ibuprofen on the Phospholipid Bilayer Bending Modulus. The Journal of Physical Chemistry B. 114 (24), 8061-8066 (2010).
- Lee, J. -. H., et al. Thermal fluctuation and elasticity of lipid vesicles interacting with pore-forming peptides. Physical Review Letters. 105 (3), 038101 (2010).
- Chakraborty, S., Abbasi, A., Bothun, G. D., Nagao, M., Kitchens, C. L. Phospholipid bilayer softening due to hydrophobic gold nanoparticle inclusions. Langmuir. 34 (44), 13416-13425 (2018).
- Hoffmann, I., et al. Softening of phospholipid membranes by the adhesion of silica nanoparticles - as seen by neutron spin-echo (NSE). Nanoscale. 6 (12), 6945-6952 (2014).
- Watson, M. C., Brown, F. L. H. Interpreting membrane scattering experiments at the mesoscale: the contribution of dissipation within the bilayer. Biophysical Journal. 98 (6), 9-11 (2010).
- Seifert, U., Langer, S. A. Viscous modes of fluid bilayer membranes. Europhysics Letters (EPL). 23 (1), 71-76 (1993).
- Bingham, R. J., Smye, S. W., Olmsted, P. D. Dynamics of an asymmetric bilayer lipid membrane in a viscous solvent. EPL (Europhysics Letters). 111 (1), 18004 (2015).
- Rawicz, W., Olbrich, K. C., McIntosh, T., Needham, D., Evans, E. Effect of chain length and unsaturation on elasticity of lipid bilayers. Biophysical Journal. 79 (1), 328-339 (2000).
- Doktorova, M., LeVine, M. V., Khelashvili, G., Weinstein, H. A new computational method for membrane compressibility: bilayer mechanical thickness revisited. Biophysical Journal. 116 (3), 487-502 (2019).
- Evans, E., Needham, D. Physical properties of surfactant bilayer membranes: thermal transitions, elasticity, rigidity, cohesion and colloidal interactions. The Journal of Physical Chemistry. 91 (16), 4219-4228 (1987).
- Lesieur, S., Grabielle-Madelmont, C., Paternostre, M. T., Ollivon, M. Size analysis and stability study of lipid vesicles by high-performance gel exclusion chromatography, turbidity, and dynamic light scattering. Analytical Biochemistry. 192 (2), 334-343 (1991).
- Heberle, F. A., et al. Direct label-free imaging of nanodomains in biomimetic and biological membranes by cryogenic electron microscopy. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19943-19952 (2020).
- Cornell, C. E., Mileant, A., Thakkar, N., Lee, K. K., Keller, S. L. Direct imaging of liquid domains in membranes by cryo-electron tomography. Proceedings of the National Academy of Sciences of the United States of America. 117 (33), 19713-19719 (2020).
- Yao, X., Fan, X., Yan, N. Cryo-EM analysis of a membrane protein embedded in the liposome. Proceedings of the National Academy of Sciences of the United States of America. 117 (31), 18497-18503 (2020).
- Kučerka, N., Nieh, M. -. P., Katsaras, J. Fluid phase lipid areas and bilayer thicknesses of commonly used phosphatidylcholines as a function of temperature. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1808 (11), 2761-2771 (2011).
- Nielsen, J. E., Bjørnestad, V. A., Lund, R. Resolving the structural interactions between antimicrobial peptides and lipid membranes using small-angle scattering methods: the case of indolicidin. Soft Matter. 14 (43), 8750-8763 (2018).
- Kučerka, N., et al. Lipid bilayer structure determined by the simultaneous analysis of neutron and X-ray scattering data. Biophysical Journal. 95 (5), 2356-2367 (2008).
- Kelley, E. G., Butler, P. D., Nagao, M. Scaling of lipid membrane rigidity with domain area fraction. Soft Matter. 15 (13), 2762-2767 (2019).
- Brüning, B. -. A., et al. Bilayer undulation dynamics in unilamellar phospholipid vesicles: Effect of temperature, cholesterol and trehalose. Biochimica et Biophysica Acta (BBA) - Biomembranes. 1838 (10), 2412-2419 (2014).
- Kučerka, N., et al. Areas of monounsaturated diacylphosphatidylcholines. Biophysical Journal. 97 (7), 1926-1932 (2009).
- Sharma, V. K., Mamontov, E., Anunciado, D. B., O'Neill, H., Urban, V. S. Effect of antimicrobial peptide on the dynamics of phosphocholine membrane: role of cholesterol and physical state of bilayer. Soft Matter. 11 (34), 6755-6767 (2015).
- Kelley, E. G., Butler, P. D., Nagao, M. Collective dynamics in lipid membranes containing transmembrane peptides. Soft Matter. , (2021).
- Yu, J., et al. Structure and dynamics of lipid membranes interacting with antivirulence end-phosphorylated polyethylene glycol block copolymers. Soft Matter. 16 (4), 983-989 (2020).
- Stingaciu, L. -. R., et al. Revealing the dynamics of thylakoid membranes in living cyanobacterial cells. Scientific Reports. 6 (1), 19627 (2016).
- Stingaciu, L. -. R., O'Neill, H. M., Liberton, M., Pakrasi, H. B., Urban, V. S. Influence of chemically disrupted photosynthesis on cyanobacterial thylakoid dynamics in synechocystis sp. PCC 6803. Scientific Reports. 9 (1), 5711 (2019).
- Miller, I. R. Energetics of fluctuation in lipid bilayer thickness. Biophysical Journal. 45 (3), 643-644 (1984).
- Nagao, M. Observation of local thickness fluctuations in surfactant membranes using neutron spin echo. Physical Review E. 80 (3), 031606 (2009).
Erratum
Formal Correction: Erratum: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions
Posted by JoVE Editors on 8/06/2021. Citeable Link.
An erratum was issued for: Neutron Spin Echo Spectroscopy as a Unique Probe for Lipid Membrane Dynamics and Membrane-Protein Interactions. The Introduction, Protocol, and Representative Results sections have been updated.
In the Introduction, the fith pargraph was updated from:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 2 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
to:
Besides direct access to the length and time scale of membrane dynamics, NSE has the inherent capabilities of neutron isotope sensitivity52. Specifically, the ability of neutrons to interact differently with the isotopes of hydrogen, the most abundant element in biological systems, results in a different neutron scattering length density,34 or NSLD (the equivalent of the optical index of refraction50), when protium is substituted by deuterium. This enables an approach known as contrast variation, which is commonly used to highlight specific membrane features or conceal others — the latter scenario is referred to as contrast matching. A frequent application of contrast variation/matching is the substitution of water (NSLD = -0.56 × 10-6 Å-2) by heavy water or D2O (NSLD = 6.4 × 10-6 Å-2) to amplify the neutron signal from protiated lipid membranes (NSLD ~ 0 × 10-6 Å-2). This approach is highly effective in studies of membrane structure because the penetration of D2O into the headgroup region of the membrane allows accurate determination of the membrane thicknesses (see Figure 2A, left panel) and of the location of different lipid subgroups when more sophisticated models are applied53,54. This paper highlights some examples on the use of contrast variation for studies of collective dynamics in biomimetic membranes and select membrane features.
In the Protocol, step 1.1 was updated from:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 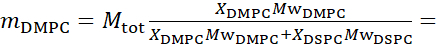 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 2 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
to:
For bending fluctuation measurements, make fully protiated liposomes in D2O (D 99.9%) or D2O-buffer (e.g., phosphate buffer prepared with D2O instead of H2O). Use fully protiated DMPC (C36H72NO8P) and DSPC (C44H88NO8P) with 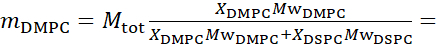 133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
133.4 mg, where XDMPC and XDSPC are the mole fractions of DMPC and DSPC, here set to 0.7 and 0.3, respectively, and MwDMPC and MwDSPC are the molar weights given by 677.9 g/mol and 790.1 g/mol, respectively. Similarly, mDSPC = 66.6 mg. This deuteration scheme increases the scattering contrast between the membrane (NSLD ~ 0 × 10-6 Å-2) and the deuterated buffer (NSLD ~ 6.4 × 10-6 Å-2) and amplifies the signal from membrane undulations (see Figure 2A left panel).
In the Representative Results, the fist pagargaph was updted from:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 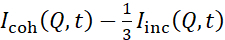 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~2 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
to:
NSE studies accessing bending fluctuations are typically performed over a Q-range of ~ (0.04 - 0.2) Å-1. This Q-range corresponds to intermediate length scales between the membrane thickness and the liposomal radius, where bending dynamics dominate. Measurement over an extended Q-range can give access to additional dynamic modes, including liposomal diffusion and intramembrane dynamics. For more details on the cross-over in membrane dynamics accessed by NSE, check these relevant publications25,71. It is important to emphasize that NSE signals are proportional to: 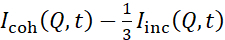 , where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
, where Icoh and Iinc are, respectively, the coherent and incoherent scattering intensity from the sample. Therefore, it is advisable to prepare NSE liposomal samples in deuterated buffers (i.e., buffers prepared with D2O instead of H2O) to minimize the incoherent scattering signal, mainly contributed by the hydrogen content of the sample. However, in some cases intermediate deuteration schemes (i.e., using mixtures of D2O and H2O) might be necessary to obtain optimal contrast conditions. Typically, NSE measurements of membrane bending fluctuations are performed on fully protiated liposomes in deuterated buffer, referred to as fully contrasted liposomes in Figure 5. This deuteration scheme results in a large NSLD difference between the membrane core (~0 × 10-6 Å-2) and its deuterated fluid environment (~6.4 × 10-6 Å-2), which significantly enhances the scattering signal from the liposomal membranes and improves the measurement statistics of bending dynamics. This contrast scheme (Figure 2A left panel) is frequently utilized in studies of bending rigidity of lipid membranes with single38,72 and multiple39,66 lipid components and in studies of membrane softening/stiffening by biological inclusions (e.g., cholesterol, drug molecules, peptides/proteins)36,37,73,74,75, and synthetic additives (e.g., nanoparticles)76,77.
In the Representative Reults, Figure 2 was updated from:
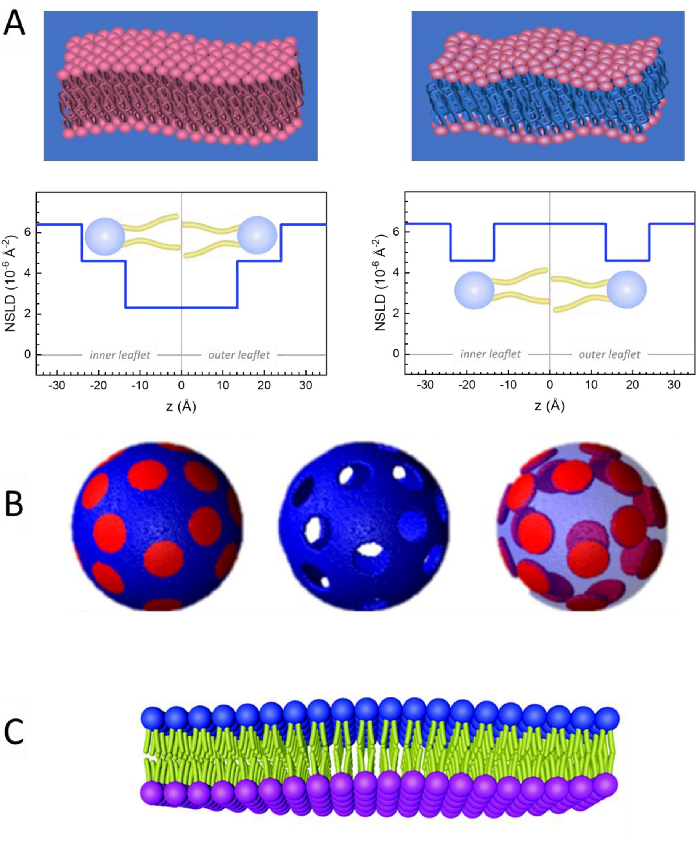
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the headgroup (~2 × 10-2 Å-2) and tail region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
to:
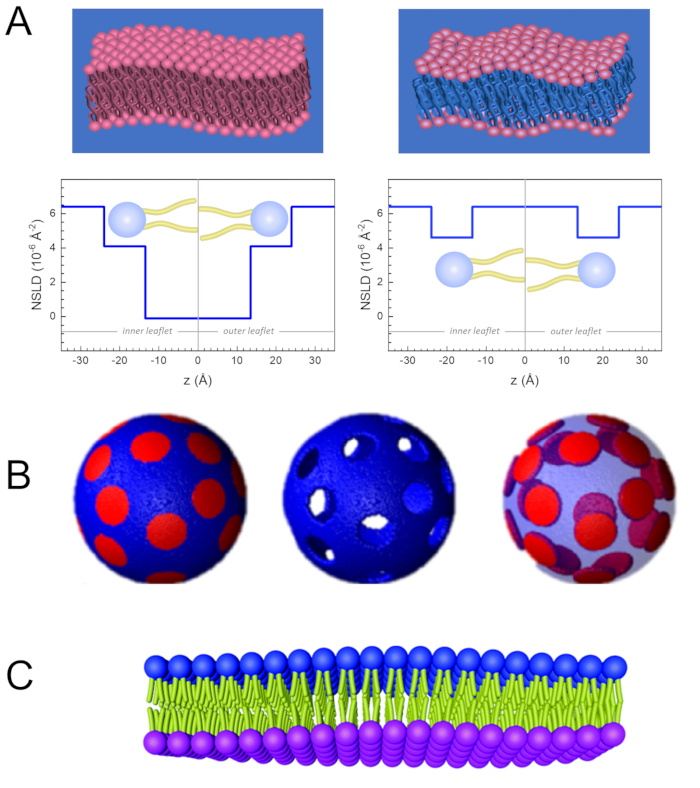
Figure 2: Examples of possible deuteration schemes in NSE experiments on lipid membranes. (A) Left: Fully contrasted membranes, e.g., protiated membranes in deuterated buffer, showing the NSLD profile along the normal to the membrane surface. The difference in the NSLD between the tail region (~0 × 10-2 Å-2) and headgroup region (~4.5 × 10-6 Å-2) of the membrane is due to the headgroup hydration with deuterated buffer. Right: Tail-contrast matched membranes such that the hydrocarbon tail region of the membrane has the same NSLD as the buffer, as shown in the corresponding NSLD profile along the membrane normal. (B) Domain-forming membranes with two neutron contrast schemes where the domains (center) or the matrix (left) are contrast-matched to the buffer, enabling selective studies of matrix or domain dynamics, respectively. This figure has been modified from Nickels et al., JACS 201541. (C) Asymmetric membranes prepared by cyclodextrin exchange between protiated and deuterated lipid vesicles, resulting in the deuteration of one membrane leaflet while keeping the other leaflet protiated. This allows studies of the bending dynamics of the protiated leaflet and provides insights into the mechanical coupling between opposing leaflets in asymmetric membranes. This figure has been modified from Rickeard et al., Nanoscale 202040. Please click here to view a larger version of this figure.
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved