Method Article
ウイルス感染時のm6Aおよびm5Cエピトランスクリプトームの探索:HIVの例
要約
ウイルス感染におけるRNA修飾の役割は、検討され始めたばかりで、新しいウイルス宿主相互作用メカニズムを強調する可能性がある。本研究では、ウイルス感染の文脈におけるm6Aおよびm5C RNA修飾を調査するパイプラインを提供する。
要約
生物学的プロセスにおけるRNA修飾の役割は、ここ数年で研究の増加の焦点であり、今日ではエピトランスクリプトロミクスとして知られています。とりわけ、N6-メチルデノシン(m6A)および5-メチルシトシン(m5C)RNA修飾はmRNA分子に記載されており、細胞プロセスを調節する役割を有し得る。エピトランスクリプトミクスは、ウイルス感染を含む任意の化学物質または生物学的薬剤への暴露によって改変または変調することもできるので、トランスクリプト分析に加えて考慮されなければならない新しい調節層である。
ここでは、ヒト免疫不全ウイルス(HIV)に感染している細胞のm6Aとm5Cマークの関節細胞およびウイルスエピトランストランスクリプトの風景を同時に分析できるワークフローを提示する。HIV感染細胞と非感染細胞からのmRNA単離および断片化に際して、RNA免疫沈降法であるMeRIP-Seqを用いて、m6Aマークを含むRNA断片とバイサルファイト変換ベースの技術であるBS-Seqを濃縮し、一塩基分解能でm5Cマークを同定しました。メチル化特異的なキャプチャの際、RNAライブラリはハイスループットシーケンシング用に用意されています。また、基礎的発現プロファイルとは無関係に微分メチル化(DM)転写物を同定する専用のバイオインフォマティクスパイプラインも開発しました。
全体として、この方法論は複数のエピトランストランスクリプトマークを同時に探索することを可能にし、ウイルス感染または他の細胞摂動時にDM転写物のアトラスを提供する。このアプローチは、ウイルス複製を促進または制限する細胞因子などの新しいプレーヤーと細胞応答の新しいメカニズムを識別する新しい機会を提供します。
概要
RNA分子が改変できることは長い間知られており、150以上の転写後修飾がこれまでに1に記載されている。これらは、主にメチル基である化学基を、RNA分子のピリミジンおよびプリン環の実質的に任意の位置に添加する2を含む。このような転写後修飾は、伝達RNA(tRNA)およびリボソームRNA(rRNA)において高度に富化することが既に示されており、最近ではmRNA分子にも説明されている。
次世代シーケンシング(NGS)などの新技術の台頭と、明確な化学修飾を認識する特定の抗体の産生は、トランスクリプトーム全体のレベルでの特定の化学修飾の場所と頻度の調査を初めて可能にしました。これらの進歩はRNA修飾のよりよい理解とmRNA分子3,4上のいくつかの修飾のマッピングにつながっている。
エピジェネティクスは、トランスクリプトーム調節におけるDNAの役割とヒストン修飾を調査する一方で、同様の方法でエピトランストランスクリプト法はRNA修飾とその役割に焦点を当てています。エピトランスクリプト法修飾の調査は、様々な細胞プロセス(RNAスプライシング、エクスポート、安定性および翻訳)を調整する新しい調節機構を強調する新しい機会を提供する。したがって、最近の研究では、細胞およびウイルスの両方のRNA6におけるウイルス感染時に多くのエピトランストランスプトミック修飾が明らかになったことは大きな驚きではなかった。これまでに調査されたウイルスには、DNA ウイルスと RNA ウイルスの両方が含まれます。その中でも、HIVは先駆的な例と考えることができます。全体として、ウイルス感染の文脈におけるRNAメチル化の発見は、ウイルス発現または複製の未記述的なメカニズムの調査を可能にし、したがってそれらを制御するための新しいツールおよび標的を提供するかもしれない7。
HIVエピトランスクリプノミクスの分野では、ウイルス転写物の改変が広く検討されており、この改変の存在がウイルス複製に有益であった8,9,10,11,12,13であることが示されている。現在までに様々な技術を用いて、トランスクリプトームワイドレベルでエピトランスクリプソメマークを検出することができる。m6A同定に最も使用される技術は、MeRIP-SeqやmiCLIPなどの免疫沈殿技術に依存しています。MeRIP-Seqは、メチル化残基を含む断片を捕捉するためにRNA断片化に依存していますが、miCLIPはRNA抗体UV架橋時のα-m6A抗体特異的シグネチャ突然変異の生成に基づいており、より正確なマッピングを可能にします。
m5C修飾の検出は、m6A検出(m5C RIP)に類似した抗体ベースの技術、バイサルファイト変換、AZA-IPまたはmiCLIPによって達成できます。アザIPとm5C miCLIPの両方がRNAメチル化を経ながらRNAを標的とする餌として特異的なメチルトランスファー酵素を使用します。Aza-IPでは、標的細胞が5-アザシチジンにさらされ、その結果、シチジンアナログ5-アザシチジン部位が新生RNAにランダムに導入される。miCLIPでは、NSun2メチルトランスファー酵素が遺伝子組み換えされ、C271A突然変異14,15を収容する。
本研究では、HIVをモデルとして、感染細胞におけるm6Aとm5C修飾の二重特性化に焦点を当てます。方法論的最適化では、メチル化RNA免疫沈降(MeRIP)とRNAバイサルファイト変換(BS)を組み合わせたワークフローを開発し、細胞およびウイルスの両方のコンテキストで、トランストランスクリプトーム全体のレベルでm6Aとm5Cエピトランスクリプトマークを同時に探査することを可能にしました。このワークフローは、細胞RNA抽出物と、ウイルス粒子から単離されたRNA上で実装することができます。
メチル化RNA免疫沈降(MeRIP)16アプローチにより、転写体全体のレベルでm6Aの調査が十分に確立されており、m6A特異的抗体の配列が、現在までに市販されています。この方法は、m6A特異的抗体を用いたm6A含有RNA片の選択的捕捉に含まれる。この技術の2つの大きな欠点は、(i)RNA断片のサイズに大きく依存し、メチル化残基を含む近似位置および領域を提供する限られた分解能であり、(ii)分析を行うために必要な大量の材料を提供する。以下の最適化されたプロトコルでは、フラグメントサイズを約150ntに標準化し、出発物質の量を、現在推奨される出発物質量であるポリ-A選択RNAの10μgから、ポリA選択RNAのわずか1μgに減らしました。また、フェノールベースの技術やプロテナーゼKを用いた従来の非特異的溶出法の代わりにm6Aペプチドを用いた溶出法を用いて、特定の抗体に結合したm6A RNA断片の回収効率を最大化しました。しかしながら、このRIPベースアッセイの主な制限は、正確な修飾されたAヌクレオチドの同定を可能にしない最適ではない分解能のままである。
m5Cマークの解析は、m5C特異的抗体を用いたRIPベースの方法とRNAバイサルファイト変換の2つの異なるアプローチを使用して現在行うことができます。RIPはメチル化残基の同定に関する解像度が限られているため、一塩基分解能を提供できる亜硫酸水素変換を用いた。バイサルファイト(BS)へのRNA曝露は、シトシン脱アミノ化をもたらし、それによってシトシン残基をウラシルに変換する。したがって、RNAバイサルファイト変換反応の間、すべての非メチル化シトシンは脱アミン化され、ウラシルに変換され、シトシンの位置5にメチル基が存在すると保護効果を有し、BS誘導脱アミノ化を防止し、シトシン残基を保存する。BSベースのアプローチは、単一塩基分解能でm5C修飾ヌクレオチドの検出と、各転写物のメチル化頻度の評価を可能にし、m5C修飾ダイナミクス18に関する洞察を提供します。しかし、この技術の主な制限は、メチル化残基の偽陽性率に依存する。確かに、BS変換は、アクセス可能なC残基を有する一本鎖RNAに有効である。しかし、緊密なRNA二次構造の存在は、N5C位置をマスクし、BS変換を妨げ、U残基に変換されない非メチル化C残基、したがって偽陽性をもたらす可能性があります。この問題を回避し、偽陽性率を最小限に抑えるために、3ラウンドの変性と亜硫酸水素変換サイクル19を適用しました。また、サンプルに2つのコントロールを導入して、バイサルファイト変換効率の推定を可能にしました:ERCCシーケンシングコントロール(非メチル化標準化および市販シーケンス)20とポリA枯渇RNAをスパイクインして、一方でバイサルファイト変換率を評価し、RT-PCRによって既知でよく保存されたメチル化部位の存在を確認します。 28SリボソームRNAに対して他方の21.
ウイルス学の分野では、これら2つのエピトランスクリプト法と次世代シーケンシングと正確なバイオインフォマティクス分析を組み合させることで、m6Aおよびm5Cダイナミクスの詳細な研究が可能になります(すなわち、ウイルス感染時に起こり得るRNA改変一時的な変化は、臨床使用に対する新しい治療関連の標的の配列を明らかにすることができる)。
プロトコル
1. 細胞の調製
注:細胞の種類とRNA含有量によって、細胞の開始数は異なる場合があります。
- 全RNAの200〜500 μgまたはポリA選択RNAの5〜7 μgの間で得られる十分な細胞を持っている。例えば、50 x 106 SupT1細胞はフェノール系試薬を用いて抽出すると約500μgの総RNAを得る必要があり、テストされた個々の条件に必要です。
- 実験計画に従って必要な数の細胞を準備し、テストされた条件の数(感染、タイムポイント、治療)に従って。実験が感染後24時間で非感染細胞とHIV感染細胞を得ることを目的としている場合、合計100 x 106 細胞が必要であり、半分は非感染状態、半分は感染状態である。
2. RNA抽出
- 細胞から: フェノールクロロホルムを用いて RNA を抽出
- 各条件について、遠心分離により細胞(例えば50 x 106)を収集し、上清を捨てる。
- 50 x 106 細胞ペレットに5mLのフェノール系試薬を加え、数回上下にピペットで混合します。
- 完全なリシスを可能にするために室温で5分間インキュベートする。Lysed細胞は、-80°Cで保存することも、直接処理することもできます。
注:必要に応じて、細胞はまた、1.5 mLチューブ内のチューブあたり10 x 106 細胞のアリコートに分割することができ、より便利な貯蔵のためのフェノールベースの試薬の1 mLでlysed。 - クロロホルムを1mL加え、反転して混ぜます。
- 室温で3分間インキュベートします。
- 2,000 x g および 4 °C で 15 分間の遠心分離機。
- 水相(上相)をピペットアウトし、新しいチューブに移します。45°でチューブを釣り、慎重に溶液をピペットアウトすることによって水相を移し終えます。
注:水相の量はサンプルによって異なる場合がありますが、サンプルに加えられるクロロホルムの量 (すなわち、 1mL)に近いはずです。 相間または有機層を移さない! 位相ロックチューブまたはフェーズメーカーチューブを使用すると、このプロセスを容易にすることができます。 - 水相に100%分子グレードのイソプロパノールの0.5 mLを加えます。
- -80°Cで1時間インキュベートし、RNA沈殿を可能にします。
- 遠心分離機は12,000xgで10分間、4°Cで沈殿したRNAをペレット化した。
- 上清を捨て、75%分子生物学グレードエタノールの1mLでRNAペレットを再懸濁します。渦は簡単に。
- 7,500xgと4°Cで5分間遠心分離機を使用し、上清を捨てます。
- ペレットを15分間エアドライします。
- ペレットを20μLのRNaseフリー水で再懸濁し、新しいチューブに移します。
- 20 μLの水で空のチューブを洗浄してRNA回収を最大化し、最初の20 μLボリュームでプールします。
- 分光光度計でRNA全体を定量化し、フラグメントアナライザでRNAの品質を評価します。
- ウイルス粒子から: カラムベースのウイルスRNA抽出キットによるRNA抽出
注:フェノールベースの試薬を用いたウイルス粒子からのRNA抽出は、低品質のウイルスRNAおよび低品質のライブラリーで生じます。カラムベースのRNA抽出が好まれるべきである。RNAの溶出および回収にキャリアRNAを用いたRNA抽出キットは、この手順には適しておらず、避けるべきです。HIV RNAはポリ-アデニル化されているため、さらなるmRNA分離を伴う直接RNA抽出は、MeRIP-SeqおよびBS-Seqパイプラインに入るのに十分です。通常、普遍的に感染した細胞からのウイルス上清の1〜2 mLは、ワークフロー全体を実行するのに十分なRNAを提供する必要があります。- 30 mLのリシスバッファーにβ-メルカプトエタノールを150 μL添加してバッファーを調製します。100%エタノールの96 mLを加えることによってウイルス洗浄緩衝液を再構成する。
- 細胞RNA汚染を最小限に抑えるために、ウイルス含有上清および遠心分離機をペレット細胞の破片に収集する。
- 1mLのウイルス上清を15mLチューブに移す。
- ウイルスサンプルの1 mLにウイルスRNAバッファーの3 mLを加え、渦によって混合する。
- 700 μLのサンプルをカラムに移し、コレクションチューブに挿入します。
- 室温で13,000 x gで2分間遠心分離機。
- フロースルーを破棄します。
- サンプル全体が処理されるまで上記の3つの手順を繰り返し、すべてのRNAをシリカベースマトリックスカラムに取り込みます。
- 500 μL のウイルス洗浄バッファーをカラムに追加します。
- 室温で10,000 x gで1分間の遠心分離機。フロースルーを破棄します。
- 200 μL のウイルス洗浄バッファーをカラムに追加します。
- 室温で10,000 x gで1分間の遠心分離機。フロースルーを破棄します。
- 空のコレクション チューブに列を配置します。
- 遠心分離機は室温で10,000 x gで1分間、残りの洗浄バッファー汚染物質をさらに廃棄する。
- 慎重に1.5 mLチューブにカラムを移します。
- DNase/RNaseフリー水20μLをカラムマトリックスの中心に直接加え、遠心分離機を室温で30秒の場合は10,000 x gで追加します。
- さらに10 μLのDNase/RNaseフリー水をカラムマトリックスの中心に直接加え、遠心分離機を30秒で再度追加します。
- 分光光度計でRNA全体を定量化し、フラグメントアナライザでRNAの品質を評価します。
注:RNA抽出は、取得したRNAの品質が高い場合、RNAの完全性/品質番号が9>であれば、任意の方法で行うことができます。全RNAは、さらに処理されるまで-80°Cで保存することができます。
3. オリゴ(dT)25によるポリA選択によるmRNA分離
注:細胞抽出物にメチル化リボソームRNAが存在するため、rRNAの枯渇によってポリARNAを分離するか、ポリA陽性選択によって優先的にポリARNAを分離することが強く推奨されます。このステップは任意であり、より高い解像度でシーケンシング結果を得るために、細胞RNAサンプルに対してのみ実行されるべきである。非ポリアデニル化ウイルスRNAのメチル化を分析する場合、ポリA選択ではなくrRNA枯渇を好むか、最終的には全RNAに対する分析を行います。
-
ポリAキャプチャのためのビーズの準備
- オリゴ(dT)25 磁性ビーズストックバイアルを>30sのボルテックスで再中断します。
- 200 μL の磁気ビーズを 1.5 mL チューブに移します。処理するRNAサンプルの合計量に応じて、マグネットビーズを使用してチューブの数を準備します。
注:ダイナビーズストックソリューション200 μLのチューブ1本は1mgのビーズに対応し、総RNAの75 μgのサンプルを収容できます。 - チューブを磁石の上に1分間置き、上清を捨てます。マグネットからチューブを取り外します。
- 結合バッファー (20 mM トリス-HCl、pH 7.5、1.0 M LiCl、2 mM EDTA) を 1 mL 追加し、ボルテックスで再中断します。マグネットにチューブを1分間置き、上清を捨てます。マグネットからチューブを取り外します。繰り返す。
- 洗浄した磁気ビーズを、100 μLの結合バッファーに再懸濁します。
-
トータルRNA製剤
- RNaseフリー水で、最終的な濃度0.75μg/μLでRNAの総量を希釈します(75 μg/100 μL)。
注:RNAの濃度が低い場合は、ボリュームを変更せずに以下の手順に従ってください。 - アリコートは、チューブあたり100 μLのRNAサンプルを分配することにより、複数のチューブ内のRNA全体を分配します。
- 各RNAサンプルに100μLの結合バッファーを加えます。
- RNA全体を65°Cに加熱し、2分間加熱して二次構造を破壊します。
- 次のステップに進む準備ができるまで、すぐに氷の上に置きます。
注:インキュベーション時間は、処理するサンプルの数によって異なる場合がありますが、RNAの分解を避けるために1時間を超えないようにしてください。
- RNaseフリー水で、最終的な濃度0.75μg/μLでRNAの総量を希釈します(75 μg/100 μL)。
-
ポリA選択
- 各RNAチューブ(ステップ3.2から)に、洗浄された磁気ビーズを100μL加えます(ステップ3.1から)。
- 上下にピペットを入れ、室温で15分間回転するホイールにバインドできるように徹底的に混ぜます。
- すべてのチューブを開き、磁石の上に1分間置き、上清をすべて慎重に取り除きます。
- ポリA最終回収を改善するために、新しいチューブ内の上澄み物を回収し、RNAキャプチャの第2ラウンド(ステップ3.3.14)のために脇に置きます。
- マグネットからチューブを取り出し、200 μLの洗浄バッファー(10 mM Tris-HCl、pH 7.5、0.15 M LiCl、1 mM EDTA)を加えます。慎重に4〜5回ピペットで混ぜます。
- マグネットにチューブを1分間置き、上清を捨てます。
- 洗浄手順を 1 回繰り返します(ステップ 3.3.5 および 3.3.6 を繰り返します)。
- 20 μLの氷冷 10 mM Tris-HCl を加え、ビーズからポリ A RNA を溶出します。
- 80°Cで2分間インキュベートします。
- チューブをマグネットに置き、ポリARNAを含む上清を新しいRNaseフリーチューブに素早く移します。チューブを氷の上に置きます。
- 溶出ステップ(ステップ3.3.8~3.3.10)を繰り返して歩留まりを増やします。
- 200 μL の洗浄バッファーで同じビーズを 1 回洗います。慎重に4〜5回ピペットで混ぜます。
- 磁石の上に1分間置き、洗浄バッファーを捨てます。
- ステップ 3.3.4 からのフロースルーをビーズに追加し、バインドから溶出までの手順を繰り返します (ステップ 3.3.2 ~ 3.3.10)。今のところ、RNA溶出物を別々のチューブに入れておいてください。
注: 必要に応じて、コントロールとして使用できるように、手順 3.3.4 に相当する上清を新しいチューブに残します。手順の最後に、エタノール沈殿またはカラムベースの選択方法(すなわち、RNAクリーンおよび濃縮器)によってRNAを精製し、濃縮する。このサンプルは、ポリAが枯渇したRNAサンプルに対応し、バイサルファイト変換の制御として使用できます(ステップ8.2.2)。 - 分光光度計で溶出したRNAを定量化し、2 μLアリコートを保持して、フラグメントアナライザでRNAの品質をさらに評価します。
注:ポリA RNAは必要になるまで-80°Cで保存することができます。
4. RNA ワークフロー
- 細胞ポリARNA(mRNA)とウイルスRNAサンプルを2つのアリコートに分割し、それぞれのエピトランストランスクリプト分析パイプライン専用にします。
(i) MeRIP-Seqおよび入力制御用の細胞mRNAまたは1μgのウイルスRNAの5 μg(ステップ5~7、およびステップ9に進む)
(ii) BS-Seq用の細胞mRNAまたは500ngのウイルスRNAの1μg(ステップ8および9に進む)。
5. RNA フラグメンテーション
注: RNA の断片化は RNA 断片化試薬で行われ、MeRIP-Seq および制御 RNA サンプル用に使用されます。これは、100から200 ntの範囲のフラグメントを取得するために慎重に最適化する必要がある非常に重要なステップです。
- mRNA/チューブの18 μLでmRNAの総容量を0.2 mL PCRチューブに分割します。
注:迅速に作業してください。再現性のある結果を得るために、一度に8つ以上のサンプルで作業しないでください。ボリュームをスケールアップしても、再現性と均一な断片化は保証されません。 - サーモサイクラーを70°Cで温めます。
- 各PCRチューブの縁に2μLの断片化試薬を加えます。
- チューブを閉じてスピンダウンします(試薬が8本のチューブに対して同時にRNAと接触するようにします)。
- 予熱サーモサイクラーで70°Cでサンプルを15分間インキュベートします。
- インキュベーションが終わるとすぐに、各チューブに2μLのStop溶液を素早く加えます。
- スピンダウンし、次のステップに進む準備ができるまで氷の上に座らせてください。
注:インキュベーション時間は、処理するサンプルの数によって異なる場合がありますが、RNAの分解を避けるために1時間を超えないようにしてください。 - すべてのサンプルに対して手順を繰り返します(アリコートが 8 個を超える場合)。
- チューブを一緒にプールし、RNAクリーンおよびコンセントレータキット(ステップ6)またはカスタマイズされたカラムベースキットでRNA精製に進み、バッファーを取り除き、水中のきれいな断片化したRNAを回収します。
6. RNA精製
注:このステップは、エタノール沈殿またはカラムベースのRNA精製および濃縮方法(すなわち、RNAクリーンおよび濃縮器)の任意の種類で行うことができます。
- 精製したRNAをDNase/RNaseフリー水の総体積50~75μLで溶出または再中断します。
注: カラムベースの方法を使用する場合、最大の回復を確実にするために 2 ラウンドの溶出を強くお勧めします。 - 分光光度計で精製した断片化したmRNAを定量化し、フラグメント分析装置でRNAの品質を評価します。
- 断片化した mRNA の 100 ng をライブラリの準備とシーケンシングの入力制御として保持します (ステップ 9 に進みます)。残りの断片化されたmRNA(最小2.5 μg)はMeRIPに使用できます(ステップ7.2に進みます)。
7. メリップ
注:各免疫沈降(IP)に、特定の抗m6A抗体(試験条件)を使用するか、または抗IgG抗体(陰性対照)を使用して、フラグメント化mRNAの最小値が必要です。
- 免疫沈降のための磁気ビーズ調製
- 各サンプルに対して、800 μLのmRNA IPバッファー5x(50 mM Tris-HCl pH 7.4,750 mM NaCl,0.5%イゲパルCA-630、ヌクレアーゼフリー水)を3.2 mLの核無水で希釈して、新しい円錐形チューブに4mLの1x IPバッファーを調製します。
注:少なくとも2つの反応が必要です(1つのテストと1つのIgGコントロール)。 - チューブを氷の上に置きます。
- 所望のIP反応の数に応じて、適切な数の1.5 mLマイクロ遠心分離管にラベルを付けます。
抗m6A抗体のnチューブ(試験)。
通常マウスIgGのためのnチューブ(陰性制御)。 - 反転とボルテックスにより磁気ビーズ(マグナChIPプロテインA/Gなど)を再懸濁します。ビーズの塊は表示されません。
- 計画された各反応に対して、 25 μL の磁気ビーズをマイクロ遠心チューブに移します。
- 使用されるビーズの元のボリューム(すなわち、 25 μLの磁気ビーズあたり1x IPバッファの250 μL)に関して、1x IPバッファ(ステップ7.1.1から)を10倍追加します。
- ビーズを数回上下に軽くピペットして混ぜ、完全な再懸濁液を取り付けます。
- チューブを磁気分離器に1分間置きます。
- 上清を取り出して捨て、磁気ビーズを吸引しないようにします。マグネットからチューブを取り外します。
- 洗浄手順を繰り返します(ステップ7.1.6~7.1.9)。
- 元の磁気ビーズの体積25μLあたり1x IPバッファの100 μLでビーズを再中断します。
- 磁気ビーズの元の容積の25 μLあたり5 μLの抗体(1 μg/μL)を加える。
nチューブ(試験)と抗m6A抗体(クローン17-3-4-1)[1 μg/μL]。
nチューブ(陰性制御)とノーマルマウスIgG(1 μg/μL) - 回転ホイールに室温で30分間インキュベートし、抗体を磁気ビーズと共役にします。
- チューブを磁気分離器に1分間置きます。上清を捨てます。マグネットからチューブを取り外し、抗体ビーズ混合物を1x IPバッファの100 μLで再懸濁します。
- 各サンプルに対して、800 μLのmRNA IPバッファー5x(50 mM Tris-HCl pH 7.4,750 mM NaCl,0.5%イゲパルCA-630、ヌクレアーゼフリー水)を3.2 mLの核無水で希釈して、新しい円錐形チューブに4mLの1x IPバッファーを調製します。
- RNA免疫沈降 (RIP)
- 各2.5 μg mRNAサンプルに対して500 μLのRIP反応混合物を次のように調製します:断片化されたRNAの100 μLで2.5 μg(ステップ6.12から)。ヌクレアーゼフリー水295μL;5 μL の 40 U/μL RNase 阻害剤;5x IPバッファの100 μL。
- 各抗体ビーズ混合物に500 μLのRIP反応混合物を加えます(ステップ7.1.14から約100 μL)。ビーズを完全に再中断するために数回穏やかにピペットで混ぜます。氷の上に置きます。
- 4 °Cで2時間回転ホイール上のすべてのRIPチューブをインキュベートします。
- MeRIP反応を遠心分離して、キャップとチューブの側面から液滴をスピンダウンします。チューブを磁気セパレータに1分間置きます。
- 新しい遠心管に上清を移し、磁気ビーズを邪魔しないように注意してください。
注: RIP の効率を確認するために制御としてフロースルーを保持できます(手順 7.3.9 に進みます)。 - マグネットからチューブを取り外します。500 μLのコールド 1x IP バッファを加えてビーズを洗浄します。ビーズを数回軽くピペットしてビーズを混ぜて完全に再中断します。
- チューブを磁性分離器に1分間置き、上清を捨てます。
- 洗浄手順(ステップ7.2.6~7.2.7)を2回繰り返し、合計3回の洗浄を行います。
- チューブを氷の上に置き、すぐに溶出に進みます。
- 溶出
- 10 mgのN6-メチルアデノシン、5'一リン酸ナトリウム塩(m6A)を1.3 mLのヌクレアーゼフリー水に溶解して20 mM m6A溶液を調製します。150 μL のアリコートを準備し、-20 °Cで保管します。
- 各サンプル(試験および制御):5x IPバッファの45 μL、20 mM m6Aの75 μL、40U/μL RNase阻害剤の3.5 μL、ヌクレアーゼフリー水101.5 μLの各成分を混合して溶出バッファー225 μLを調製します。
- 溶出バッファーの 100 μL (ステップ 7.3.2 から) をビーズに追加します(ステップ 7.2.9 から)。ビーズを完全に再中断するために数回穏やかにピペットで混ぜます。
- 4 °Cでロッカーに連続的な揺れで1時間すべてのチューブをインキュベートします。
- RIP反応を遠心分離して、キャップとチューブの側面から液滴をスピンダウンします。チューブを磁気セパレータに1分間置きます。
- 溶出したRNA断片を含む上清を新しい1.5 mLマイクロ遠心分離チューブに移します。それは、バックグラウンドノイズを増加させるので、ビーズを吸引しないように注意してください。
- 溶出バッファーを再び100 μL添加し、4°Cで1hインキュベートし、磁気分離後に溶出液を回収することにより、溶出工程(7.3.3~7.3.6)を繰り返します。
- 同じサンプルからすべての溶出液を組み合わせます(溶出量は200μLでなければなりません)。
- 溶出したRNAとフロースルー(オプション、ステップ7.2.5から)をエタノール沈殿またはカラムベースの選択方法(すなわち、RNAクリーンおよびコンセントレータ)によって精製する。
- 高感度検出キットを使用して、フラグメントアナライザを使用して、RNA量とフロースルーおよび溶出したサンプルの品質を評価します。RNAの品質が満足のいく場合は、ライブラリ調製およびハイスループットシーケンシング(ステップ9)に進みます。
注: MeRIP で回収される RNA の量は非常に少なく、定量化を確実にするためには高感度検出キットが必要です。バイオアナライザーが利用できない場合は、ライブラリの準備に盲目的に進むことができます。
8. RNAバイサルファイト変換
- 制御および試薬の準備
- ERCCミックススパイクインコントロール:メーカーの指示に従ってERCCミックスを追加し、0.5 μLの希釈されていないERCCミックスをmRNAの500 ngに添加することを推奨します。この制御は、バイサルファイト変換の効率を評価するのに役立ちます。
- スパイクポリA枯渇RNA(ステップ3.3.14から)比1/1000(すなわち、 500 ngのmRNAに対するポリA枯渇RNAの500pg)。このサンプルはリボソームRNAに富んでおり、バイサルファイト変換のための陽性対照である28S rRNAを含む必要があります。
注:総RNAは、ポリA枯渇RNAの代わりに正のコントロールとして使用することもできます。 - RNAメチル化キット(例えば、Zymo EZ)でバイサルファイト変換を行います。
- RNAウォッシュバッファー:使用前に100%エタノール(または95%エタノールの52 mL)を12mLのRNA洗浄バッファー濃縮物に加えます。
- バイサルフィット変換
注:バイサルファイト変換は、下記の製造者の手順に従って市販のRNAバイサルファイト変換キットで行った。- 0.2 mL PCRチューブに、1000 ngのmRNA(または300〜1000 ngの間)を加えます。スパイクインコントロールを追加:ERCCミックスの1 μL(ステップ8.1.1)とポリA枯渇RNAの1000 pg(ステップ8.1.2)。DNase/RNaseフリーの水で最大20 μLのボリュームを完成させます。
- 各20 μL RNA サンプルに 130 μL の RNA 変換試薬を加えます。
- サンプルを上下にピペットで混ぜます。
- チューブのキャップまたは側面に液滴がないことを確認するために、短時間回転します。
- PCRチューブをサーマルサイクラーに入れ、70°Cで5分間変性する:54 °Cで45分間の変換。合計3サイクルの変性と変換ステップを繰り返します。4°Cで無期限に保持します。
注:変性と亜硫酸水素変換の3サイクルは、サンプルの完全な亜硫酸水素変換を保証します。サンプルは-80 °Cで保存することも、直接処理することもできます。 - 列内の逆行を続行します。カラムを空のコレクションチューブに入れ、250 μL の RNA 結合バッファーをカラムに追加します。
- サンプル(ステップ8.2.5から約150 μL)をRNA結合バッファーを含むカラムにロードし、上下にピペットを入れて混ぜます。
- カラム内のサンプルRNA結合バッファー混合物に、95~100%エタノールの400 μLを加えます。キャップを閉じ、列を数回反転してすぐに混ぜます。
- 30 s のフルスピード(≥ 10,000 x g) での遠心分離機。フロースルーを破棄します。
- カラムに200μLのRNAウォッシュバッファーを加え、遠心分離機を30秒のフルスピードで追加します。
- カラムに200μLのRNAデスルフォネーションバッファーを加え、室温で30分間インキュベートします。インキュベーション後、遠心分離機は30sの全速速度で。フロースルーを破棄します。
- 400 μL の RNA ウォッシュ バッファーをカラムに加え、遠心分離機を 30 s のフルスピードで追加します。さらに400 μLのRNA洗浄バッファーで洗浄ステップを繰り返します。フロースルーを破棄します。
- 空にしたコレクションチューブの柱を全速速度で2分間遠心分離します。カラムをRNaseフリーチューブに移します。
- DNase/RNaseフリー水≥10μLをカラムマトリックスに直接加え、室温で1分間インキュベートします。30 sのための全速力で遠心分離機。
注:通常、20 μLのボリュームで溶出します。溶出したRNAは、すぐに使用するか、または-20°Cで最大3ヶ月間保存することができます。長期保存の場合は、-80°Cに保ちます。 - RNAの品質と量のフラグメント分析装置評価のために2.5 μLを取り出し、ライブラリの調製およびハイスループットシーケンシングに進みます(ステップ9)。
- バイサルファイト変換制御の効率を制御するために、変換されたRNAの4 μLを取ります(ステップ8.3)。
- RT-PCRによるバイサルファイト変換制御
注: この手順により、シーケンス処理に進む前にバイサルフィット変換が成功したことを確認します。 ホモサピエンス からの28SリボソームRNAは、RNAメチル化分析のための陽性対照として使用され、位置4447におけるC残基(GenBank加盟#NR_003287)が100%メチル化されていると説明されている。
プライマーシーケンス:
H 28SF プライマー: 5'-GGGGTTTTAYGATTTTTTTTTGGG-3'
H 28SR プライマー: 5'-CCAACTCACRTCCCタッタアタアターアアアク-3'- 大容量cDNA逆転写キットを使用して、逆転写(RT)反応ミックスを準備します。氷の上にキットコンポーネントを解凍し、次のように氷の上にRTマスターミックスを準備します。
バイサルファイト変換RNAの4 μL(ステップ8.2.14から):
10xRTバッファの2 μL
0.8 μL の 25x dNTP ミックス [100 mM]
10x RTランダムプライマーの2 μL
マルチスクライブ逆転写酵素 1 μL
1 μL の RNase 阻害剤
ヌクレアーゼフリーH2O 9.2 μL
注:各RT反応には、0.2 mL PCRチューブに20 μLの最終体積が含まれている必要があります。 - 次のRTプログラムとサーマルサイクラーにチューブを入れて:10分間25°C。37 °C 120分の間;85°C 5分間。その後、4 °Cで無期限に。
- PCR のリプリーディング酵素を使用して、28S rRNA を特異的に増幅する PCR 反応を準備します。氷の上にキットコンポーネントを解凍し、穏やかに渦巻きと短い遠心分離機。次のように、氷の上または氷冷金属プレートホルダーにPCRマスターミックスを準備します。
0.6 μL 10 μM H 28SF プライマー
0.6 μL 10 μM H 28SF プライマー
テンプレート cDNA の 6.5 μL
22.5 μLのDNAポリメラーゼマスターミックス
注:各PCR反応には、0.2 mL PCRチューブに20 μLの最終体積が含まれている必要があります。 - 次のPCRプログラムでサーマルサイクラーにチューブを入れます:5分間95°Cで初期変性。変性の45サイクル(15sのための95°C)、アニーリング(30sのための57°C)、および伸び(15sのための72°C)、72°Cで10分間の最終伸び、そして4°Cで無期限に保持する。
- 2%アガロースゲルで10μLの反応を実行します。予想されるバンドサイズは130-200 bpです。
- 大容量cDNA逆転写キットを使用して、逆転写(RT)反応ミックスを準備します。氷の上にキットコンポーネントを解凍し、次のように氷の上にRTマスターミックスを準備します。
- PCR産物のシーケンシング
- 酵素およびdNTP残基を除去し、少なくとも20μLのDNase/RNaseフリー水で増幅されたDNAを溶出させるカラムベースの方法でPCR製品を精製します。
- 分光光度計で精製したDNAを定量化します。
- シーケンシング反応
- PCR産物/シーケンシング反応の40 ngを使用してください。
- H28SFおよびH 28SRプライマーを使用して両方向に配列する。
- 配列を公知の非変換配列(28SリボソームN5(RNA28SN5)と一致させる。位置C4447にC残基の存在を確認し、他の場所でCの代わりにT残基を確認してください。
9. ライブラリの準備とハイスループットシーケンス
- mRNAキット(例えば、イルミナTruSeq Stranded)を使用してシーケンシング用のライブラリを準備し、Elute-Prime-Fragmentステップでプロトコルを開始し、メーカーの指示に従います。
- しかし、入力RNA-SeqおよびMeRIP-Seqサンプルについては、サンプルを80°Cで2分間インキュベートし、素数のみで、さらに断片しません。
- イルミナプラットフォームを使用してシーケンシングを実行します。シーケンス化反応は、100 nt の長さの最小値を持つ、単一または対端の好みおよび実験計画に従って行うことができます。
10. バイオインフォマティクス分析
- m6A データ処理
- FASTQC24 を実行して、m6A の読み取り品質を評価し、シーケンスから FASTQ ファイルを入力します。
- Atropos25 を実行して、読み取りから低品質のエンド シーケンスとアダプター シーケンスをトリミングします。Atropos の実行中に次のパラメーターを設定します。
- 次のアダプターシーケンスを削除します: アガトCGGAAGAGGAG, CTCTTCCATCT, AACCTTTCCCT, アガッガグCG, アグガアガグットgtt, CGCTTCCGATCT.
- メーカー(https://support.illumina.com/downloads/illumina-adapter-sequences-document-1000000002694.html)で指定された低品質の端をトリミングするには、次の「Phred 品質カットオフ」5 を使用します。
- トリミング後に読み取りの最小長を使用します: 25 基本ペア。
- GRh38ヒトゲノムとHIV[統合線形pNL4-3Env-GFP]参照をFASTA形式でマージします。
- マージされた参照に HISAT226 を使用してインデックスを付けます。
- 索引付き参照に合わせて、トリミングされた読み取りで HISAT2 を実行します。デフォルトの HISAT パラメータを使用します。
- 整列された読み取り値を SAMtools27 でソートし、インデックスを付けます。
- SAMtools stat および Qualimap 228 を実行して、順序付きライブラリーの後アライメント品質チェックを行います。
- 必要に応じて、multiQC29 を使用して、前のステップから品質測定を収集および要約します。
- HIVゲノムは5'LTRおよび3'LTRに相同634bp配列を有する:SAMtoolsを用いて5'LTRから対応する3'LTR領域への再整列マルチマッピング読み取り。
- m6A ピークを識別するには、ピークコールソフトウェア MACS230(v 2.1.2)を実行します。ピークコールが遺伝子発現レベルの影響を受ける可能性があり、短いエキソンがピークと誤って呼ばれる可能性があるため、RNA-Seqデータ上で正しく機能するために、MACS2実行パラメータを慎重に選択します。したがって、入力信号は、MACS2によって日常的にDNAベースのデータに適用される平滑化を行わずに、m6A信号から差し引かれなければならない。MACS2 から 'callpeak' サブコマンドに次のパラメータを適用します。
-keep-dup auto (重複読み取りに向けて MACS2 の動作を制御し、'auto' を使用すると、MACS は p 値カットオフとして 1e-5 を使用した二項分布に基づいて、まったく同じ場所での最大読み取り数を計算できます)
-g 2.7e9 (ヒトゲノムのサイズ(bp)
-q 0.01 (有意なピークを呼び出す最小 FDR カットオフ)
-nomodel(ChIP-Seq実験に合わせたシフトモデルの構築をバイパスする)
-スローカル 0
-llocal 0 (このパラメータと前のパラメータを 0 に設定すると、MACS2 は、平滑化せずに m6A からの入力読み取りを直接減算できます)
-extsize 100 (bp の平均フラグメント長)
-B - 感染サンプルと非感染サンプルを比較するには、MACS2 のサブコマンドである 'bdgdiff' を呼び出す差分ピークを実行します。'bdgdiff' は、前のステップで 'callpeak' によって生成された bedGraph ファイルを入力として取ります。各時点で、感染サンプルと非感染サンプルの比較を'bdgdiff'で実行し、m6A信号からそれぞれの入力信号を減算し、追加のパラメータを提供します: -g 60 -l 120。
- m5C データ処理
- 次のパラメーターを使用して、Raw 読み取りからアダプター シーケンスをトリムするには、Cutadapt31 を実行します。
アダプター 「アガトグガガカカッククトクガック」
-最小長 =25。 - シーケンスプロトコルが逆鎖からの読み取りを生成するので、seqkit32 を使用してトリムされた読み取りを逆補完します。
- FastQC を実行して読み取り品質を確認します。
- GRh38ヒトゲノムとHIV[統合線形pNL4-3Env-GFP]参照をFASTA形式でマージします。
- マージされた参照に、meRanTK パッケージ33 からアプリケーション meRanGh を使用してインデックスを作成します。
- 次のパラメータを使用して、meRanGh と位置合わせします。
-UN マップされていない読み取りを出力ファイルに書き込む
-MM を使用すると、出力ファイルに書き込まれるマルチマップ読み取りを有効にします。
ベッドグラフで出力用-bg
-mbgc 10 フィルターはカバレッジによって報告された領域 (カバレッジの少なくとも 10 読み取り) - HIVゲノムは5'LTRおよび3'LTRに相同634bp配列を有する:SAMtoolsを用いて5'LTRから対応する3'LTR領域へのマルチマッピング読み取り結果を再整列させる。
- 次のパラメータを指定して、meRanTK が提供する meRanCall ツールを使用して、メチル化呼び出しを実行します。
-rl = 126,読み取り長
-ei = 0.1、メチル化率p値計算の誤差間隔
-cr = 0.99、変換が期待される - 各サンプルのサイズ係数を見積もるためのMeRanTKのユーティリティ estimateSizeFactors.pl を実行します。サイズ係数は、次のステップでパラメータとして使用されます。
- MeRanCompare を実行して、非感染と感染した時点 12、24、および 36 時間の微分メチル化分析を行います。次のパラメータが適用されます: 前のステップからのレポートおよびサイズ係数の最小しきい値として、有意値 .01 を指定します。
- 次のパラメーターを使用して、Raw 読み取りからアダプター シーケンスをトリムするには、Cutadapt31 を実行します。
結果
このワークフローは、HIV感染のコンテキストでm6Aおよびm5Cメチル化の役割を調査するのに有用であることが証明されています。このために、HIVに感染するか、未治療のまま残したCD4+ T細胞線モデル(SupT1)を使用しました。1条件あたり5,000万個の細胞でワークフローを開始し、RNA品質番号10の平均500μgのRNAを得た(図1A-B)。ポリA選択時に、1つの条件(全RNAの約2%を表す)のmRNAの10〜12μgの間で取り出しました(図1B)。この時点で、MeRIP-Seqパイプラインには5μgのポリA選択RNA、BS-Seqパイプラインには1μgを使用しました。HIV RNAはポリアデニル化されているため、それ以上のアクションは必要なく、MeRIP-SeqおよびBS-Seq手順を直接適用することができます。
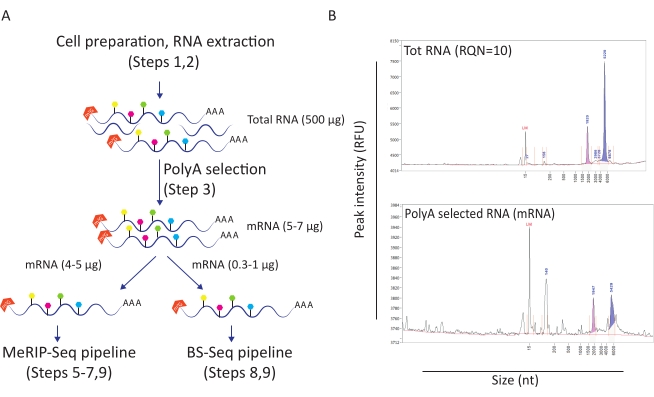
図1:下流用途のRNA調製物。A) 同時MeRIP-SeqおよびBS-SeqパイプラインのためのRNAの準備および分布を描写するワークフロー。塗りつぶされた六角形の形状は、m6A(緑色)やm5C(ピンク)などのRNA修飾タイプを表します。実験を行うために必要なRNA材料の量が示されている。 B) RNAの総抽出(上部パネル)およびポリA選択(下部パネル)で期待されるRNA分布プロファイル(サイズと量)を示す代表的な結果。サンプルは、特定のMeRIP-SeqおよびBS-Seq手順に入る前にRNAの品質を評価するために、標準感度キットを用いてフラグメント分析装置にロードしました。RQN: RNA品質番号;nt: ヌクレオチド。この図の大きなバージョンを表示するには、ここをクリックしてください。
MeRIP-Seqパイプラインは、RNA分子に沿ったm6A修飾の調査を可能にするRNA免疫沈降ベースの技術です。このために、RNAはまず断片化され、m6A特異的抗体と結合して免疫沈降および捕獲のために磁気ビーズと結合される。MeRIP濃縮RNA断片と手つかずの(入力)分数を配列決定し、m6A修飾RNA領域を同定し、m6A-メチル化された転写物を同定する(図2A)。この技術の分解能は、RNA断片化の効率に依存します。実際、短い断片はm6A残基のより精密な局在化を可能にする。ここで、細胞ポリA選択RNAおよびウイルスRNAは、20μL最終容積で15分の間にRNA断片化バッファーを有するイオンベースの断片化を行い、100〜150ntのRNA断片を得た。5 μgのmRNAから、90%の回収率に対応した4.5μgの断片化したRNAを回収しました(図2B)。100 ngの断片化した精製RNAを入力制御として使用し、直接ライブラリ調製およびシーケンシングを行った。残りのRNA(約4.4μg)は、抗体特異的抗体または抗IgG抗体に抗IgG抗体を制御するように結合したビーズを有する断片化されたRNAのインキュベーションから始まるMeRIP-Seqパイプラインに従って処理された。2.5 μgの断片化されたRNAのm6A特異的RIP(MeRIP)は、ライブラリー調製およびシーケンシングを行ったm6A濃縮材料の約15ngを取り出すことができます(図2B)。反IgG制御を用いたRIPは、予想通り、さらなる分析を可能にするのに十分なRNAを得られなかった(図2B)。
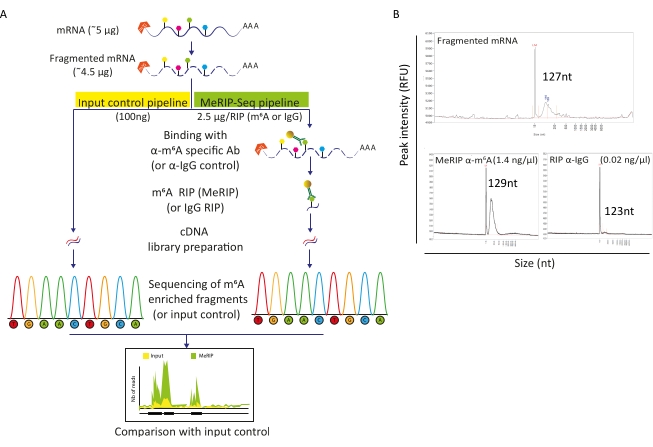
図 2: MeRIP-Seq パイプライン A) MeRIP-Seq ワークフローと入力コントロールの概略図ポリA選択の際、サンプルを120-150 nt部分に断片化し、シーケンシング(100ng、入力制御)を直接行うか、またはシーケンシング前に陰性対照として抗m6A特異的抗体または抗IgG抗体を有するRNA免疫沈降(2.5μg、RIP)に使用した。B)断片化時の期待されるRNA分布プロファイル(サイズおよび量)およびRIP(下部パネル、MeRIP:左、IgG制御:右)を示す代表的な結果。サンプルは、ライブラリー調製およびシーケンシングにさらなる処理を行う前に、RNAの品質と濃度を評価するためにフラグメント分析装置にロードした。断片化したRNA分析は、免疫沈降RNAが高感度キットを使用しながら、RNA標準感度キットを用いて行った。この図の大きなバージョンを表示するには、ここをクリックしてください。
BS-Seqパイプラインは、ヌクレオチド分解能でm5C RNA修飾を探索し、m5C-メチル化転写物の同定につながります。バイサルファイト変換時には、非メチル化シトシンはウラシルに変換され、メチル化されたシトシンは変わりません(図3A)。バイサルファイト変換手順 (すなわち、 高温および低pH)の過酷な条件により、変換されたmRNAは高度に分解される(図3B)が、これはライブラリの調製およびシーケンシングに干渉しない。バイサルファイト変換は、一本鎖RNAでのみ効率的であり、二次的な二本鎖RNA構造によって妨げられる可能性があります。C-U変換の効率を評価するために、2つのコントロールを導入しました。陽性対照として、我々は、28S rRNA23の位置C4447における高度にメチル化されたシトシンの前述の存在を利用した。メチル化部位を取り囲む200bp断片のRT-PCR増幅およびシーケンシングの際に、すべてのシトシンがウラシルに正常に変換され、それによって、変化しなかった4447位のシトシンを除くDNA配列中にチミジンとして現れるのを観察することができた。バイサルファイト変換速度の制御として、市販の合成ERCC RNA配列を用いた。この混合物は、既知の非メチル化およびポリアデニル化RNA配列のプールで構成され、様々な二次構造および長さを有する。ライブラリの調製とシーケンスの際に、変換速度を計算するためにこれらのERCC配列に焦点を当て、これはすべてのERCC配列および各サンプルの合計C残基の中で変換されたCの数を数えることによって実行することができる。我々は、99.5%の変換率を得て、バイサルファイト変換反応の効率と成功を確認した(図3D)。
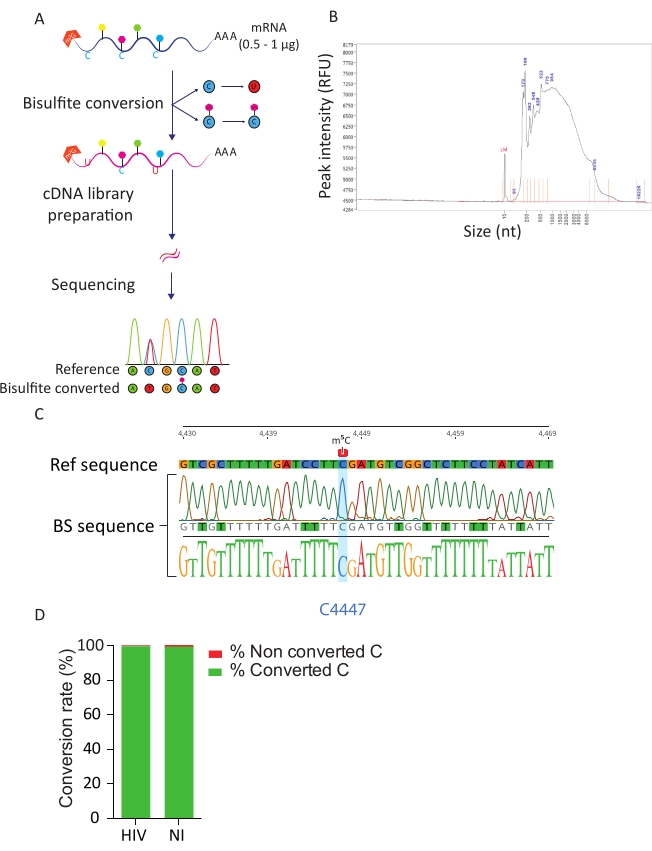
図 3: BS-Seq パイプラインA) BS-Seq ワークフローの概略表現。ポリA選択の際、サンプルはバイサルファイトにさらされ、非メチル化C残基に対してCからUへの変換(脱アミノ化による)が生じます。対照的に、メチル化C残基(m5C)は、亜硫酸水素化処理の影響を受けず、変化しないままである。 B) 標準感度キットを用いてフラグメントアナライザで解析した際のバイサルファイト変換RNA分布プロファイル(サイズと量)の代表的な結果。 C)28S rRNAの位置4447で100%メチル化Cを取り囲む領域のRT-PCRアンプリコンの代表的なシーケンシング結果を示す電解液球図(青色で強調表示)。これに対し、参照配列のC残基は、亜硫酸水素塩変換成功によるアンプリコン配列のT残基として同定された。 D) HIV感染細胞および非感染細胞におけるERCCスパイクイン配列の分析によるC-U変換率の評価平均換算率は99.5%です。 この図の大きなバージョンを表示するには、ここをクリックしてください。
M6A濃縮サンプル、バイサルファイト変換サンプルおよび入力制御は、ライブラリ調製、シーケンシングおよびバイオインフォマティクス分析のためにさらに処理される(図4)。実験計画と生物学的問題に従って、複数のバイオインフォマティクス解析を適用することができる。ここでの原理証明として、HIV感染時に誘導される微分メチル化転写物の同定に焦点を当てた1つの潜在的な応用(すなわち、微分メチル化分析)の代表的な結果を示す。簡単に言えば、ウイルスのライフサイクル中のRNAメチル化の役割をさらに理解するために、非感染細胞とHIV感染細胞の両方で、遺伝子発現レベルとは無関係に、転写産物のm6Aまたはm5Cメチル化レベルを調べた。遺伝子発現正常化の結果、感染状態に応じてZNF469転写物がm6A-メチル化されたのが分化され、実際にこの転写産物は非感染細胞ではメチル化されなかったが、HIV感染時にメチル化されたピークがいくつか示された(図5A)。m5Cに対する同様の微分メチル化分析は、PHLPP1転写物に複数のメチル化残基が含まれていることを明らかにした。この文脈では、両方の分析は、HIV感染が細胞エピトランストランスクリプトームに影響を与ことを示唆している。
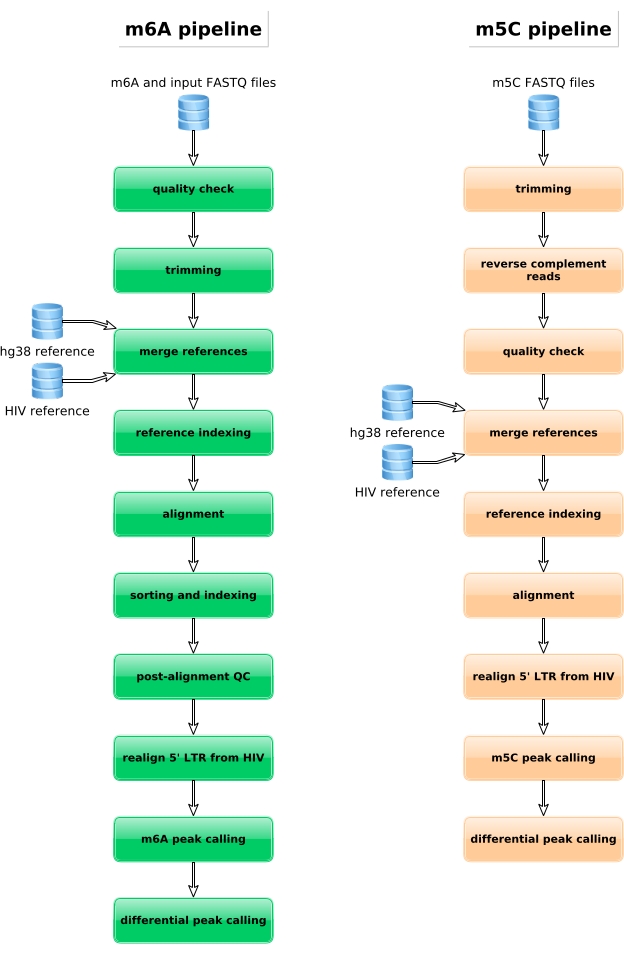
図4:m6Aおよびm5Cデータの解析のためのバイオインフォマティクスワークフローの概略表現。
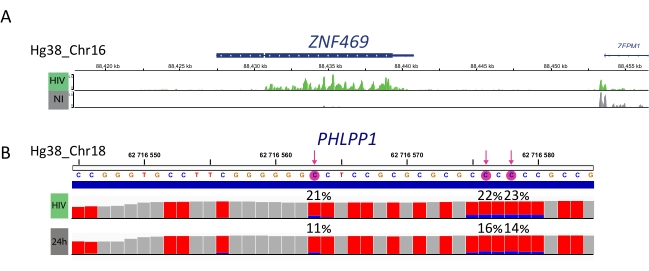
図5:感染時の微分メチル化転写物の例。A) HIV感染(緑色)および非感染(灰色)細胞におけるZNF459転写産物のm6Aメチル化を示す代表的な結果。ピーク強度(入力式減算時)は、y軸上およびX軸に沿った染色体内の位置に表示されます。微分メチル化分析は、ZFN469転写産物がHIV感染時に過メチル化されることを明らかにする。 B)HIV感染(上レーン)および非感染(下車線)細胞におけるm5Cメチル化遺伝子の代表的な結果。各バーの高さは、ヌクレオチドあたりの読み取りの数を表し、カバレッジ評価を可能にします。各C残基は赤色で表され、メチル化Cの割合は青色で表される。正確なメチル化率(%)は、各C残基より上に報告される。矢印は、IGVビューアを用いて可視化された統計的に有意な微分メチル化C.サンプルを強調した。 この図の大きなバージョンを表示するには、ここをクリックしてください。
ディスカッション
ウイルス感染におけるRNA修飾の役割は、まだほとんど知られていない。ウイルス感染の文脈におけるエピトランストランスクリプト修飾の役割をよりよく理解することは、新しい抗ウイルス治療標的の探求に寄与する可能性がある。
この研究では、感染細胞のm6Aおよびm5Cエピトランスクリプションの調査を可能にする完全なワークフローを提供する。生物学的な問題に応じて、ポリA選択RNAを出発物質として使用することをお勧めします。パイプラインは全RNAと併用できますが、rRNAと小さなRNAは高度に修飾されており、重要な数のメチル化残基が含まれていることを覚えておいてください。これにより、意味のあるシーケンス データの質と量が減少する可能性があります。
しかし、研究の焦点が非ポリアデニル化RNAである場合、RNA抽出ステップは、小さなRNA(カラムベースのRNA抽出の場合)を廃棄することを避け、パイプラインに入るためのポリボソーム枯渇技術ではなく特権リボソーム枯渇技術に適応する必要があります。
高品質のRNA、正しい断片化、適切なm6A濃縮およびBS変換されたRNA品質をライブラリ調製に確実に行うために、フラグメントアナライザまたはバイオアナライザを使用することを強くお勧めします。ただし、この装置は常に使用できるとは限りません。別の方法として、RNAの品質、mRNAおよび断片化されたRNAのサイズはまたアガロースゲルの視覚化によって評価することができる。あるいは、ライブラリーの調製は、RNA量の以前の評価なしで行うことができる。
抗体ベースのMeRIP-Seq16技術を用いて、m6Aエピトランスクリプトの風景を探索しました。この技術はRNA免疫沈降に基づいており、成功しています。ただし、一部の手順では、慎重な最適化が必要であり、重要な場合があります。m6Aメチル化は、主にコンセンサス配列RRA*CH内で起こることが説明されているが、このモチーフはmRNA分子に沿って非常に頻繁に起こるものであり、メチル化部位の正確な同定を可能にしない。したがって、RIPベースの分解能を向上させるためには、再現性と一貫性のあるRNA断片化を達成し、小さなRNA断片を生成することが重要です。このプロトコルでは、実験環境で再現可能で一貫性のある結果を提供する最適化された手順をお勧めします。ただし、この断片化の手順では、特定のサンプル機能に応じて、さらに最適化が必要になる場合があります。
近年、m6A直接シーケンシングを可能にする新しい技術が説明された。m6A RNA修飾24に遭遇した応答としてユニークなRTシグネチャを示す特異的な逆転写酵素変異体の使用に基づいています。この技術は、慎重な最適化の際に、MeRIP-Seqに直面する大きな制限を回避する可能性があります(初期材料の量を減らすと、より高い解像度を可能にします)。m5C修飾を探索するために、修飾されたC残基をヌクレオチド分解能で検出するためにバイサルファイト変換技術を使用することにしました。RNA二次構造の存在による偽陽性率を低減するために、ERCCスパイクインコントロールの使用により、変性/亜硫酸水素変換の3サイクルを行い、バイサルファイト変換速度のパフォーマンスをさらに制御しました。この技術に関連する制限の1つは、亜硫酸水素変換が非常に過酷であり、変性/亜硫酸水素塩変換の3サイクルがRNAを分解し、したがって分解能を低下させる可能性があることです。しかし、この設定では、データセットの品質を向上させるために、わずかに低い解像度に落ち着くことを選択しました。
これらの最適化と制御のおかげで、ウイルス感染、宿主病原体相互作用、または特定の治療への暴露の文脈におけるエピトランストランスクリプトの風景とその変化を調査するために利用できる信頼性の高い健全なワークフローを提供することができました。
開示事項
著者らは開示するものは何もない。
謝辞
この研究は、スイス国立科学財団(助成金31003A_166412と314730_188877)によって支援されました。
資料
| Name | Company | Catalog Number | Comments |
| AccuPrime Pfx SuperMix | Invitrogen | 12344-040 | |
| anti-m6A antibody _Clone 17-3-4-1 | Millipore | MABE1006 | |
| Chloroform | Merck | 67-66-3 | |
| ERCC | Invitrogen | 4456740 | |
| EZ RNA Methylation Kit | Zymo Research | EZR5001 | |
| Fragment analyzer RNA Kit - HS RNA Kit | Agilent | DNF-472-0500 | |
| Fragment analyzer RNA Kit - RNA Kit | Agilent | DNF-471-0500 | |
| High-Capacity cDNA Reverse Transcription Kit | Applied Biosystem | 4368814 | |
| Illumina TruSeq Stranded mRNA | Illumina | 20020594 | |
| Magnetic Beads A/G Blend | Merck | 16-663 | |
| N6-Methyladenosine, 5′-monophosphate sodium salt (m6A) | Sigma Aldrich | M2780-10MG | |
| Normal Mouse IgG | Merk | 12371 | |
| Oligo(dT)25 | Life Technologies | 61005, | |
| PCRapace | Stratec | 1020220300 | |
| Quick RNA Viral Kit | Zymo Research | 1034 | |
| RNA Clean & Concentrator | Zymo Research | R1015 | |
| RNA Fragmentation Reagent | Ambion | AM8740 | |
| RNase Inhibitor | Ambion | AM2684 | |
| Trizol | TRIzol Reagent | 15596026 |
参考文献
- Machnicka, M. A., et al. MODOMICS: a database of RNA modification pathways--2013 update. Nucleic Acids Research. 41, 262-267 (2013).
- Zaccara, S., Ries, R. J., Jaffrey, S. R. Reading, writing and erasing mRNA methylation. Nature Reviews Molecular Cell Biology. 20 (10), 608-624 (2019).
- Davalos, V., Blanco, S., Esteller, M. SnapShot: Messenger RNA Modifications. Cell. 174 (2), 498 (2018).
- Saletore, Y., et al. The birth of the Epitranscriptome: deciphering the function of RNA modifications. Genome Biology. 13 (10), 175 (2012).
- Zhao, B. S., Roundtree, I. A., He, C. Post-transcriptional gene regulation by mRNA modifications. Nature Reviews Molecular Cell Biology. 18 (1), 31-42 (2017).
- Netzband, R., Pager, C. T. Epitranscriptomic marks: Emerging modulators of RNA virus gene expression. Wiley Interdisciplinary Reviews: RNA. 11 (3), 1576 (2020).
- Pereira-Montecinos, C., Valiente-Echeverria, F., Soto-Rifo, R. Epitranscriptomic regulation of viral replication. Biochimica et Biophysica Acta. 1860 (4), 460-471 (2017).
- Lichinchi, G., et al. Dynamics of the human and viral m(6)A RNA methylomes during HIV-1 infection of T cells. Nature Microbiology. 1, 16011 (2016).
- Courtney, D. G., et al. Epitranscriptomic Addition of m(5)C to HIV-1 Transcripts Regulates Viral Gene Expression. Cell Host & Microbe. 26 (2), 217-227 (2019).
- Kennedy, E. M., et al. Posttranscriptional m(6)A Editing of HIV-1 mRNAs Enhances Viral Gene Expression. Cell Host & Microbe. 19 (5), 675-685 (2016).
- Tirumuru, N., Wu, L. HIV-1 envelope proteins up-regulate N (6)-methyladenosine levels of cellular RNA independently of viral replication. Journal of Biological Chemistry. 294 (9), 3249-3260 (2019).
- Tirumuru, N., et al. N(6)-methyladenosine of HIV-1 RNA regulates viral infection and HIV-1 Gag protein expression. Elife. 5, (2016).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. Frontiers in Virology. 1 (11), (2021).
- Khoddami, V., Cairns, B. R. Transcriptome-wide target profiling of RNA cytosine methyltransferases using the mechanism-based enrichment procedure Aza-IP. Nature Protocols. 9 (2), 337-361 (2014).
- Hussain, S., Aleksic, J., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (11), 215 (2013).
- Dominissini, D., Moshitch-Moshkovitz, S., Salmon-Divon, M., Amariglio, N., Rechavi, G. Transcriptome-wide mapping of N6-methyladenosine by m6A-seq based on immunocapturing and massively parallel sequencing. Nature Protocols. 8 (1), 176-189 (2013).
- Dominissini, D., et al. Topology of the human and mouse m6A RNA methylomes revealed by m6A-seq. Nature. 485 (7397), 201-206 (2012).
- Shobbir Hussain, J. A., Blanco, S., Dietmann, S., Frye, M. Characterizing 5-methylcytosine in the mammalian epitranscriptome. Genome Biology. 14 (215), (2013).
- Amort, T., et al. Distinct 5-methylcytosine profiles in poly(A) RNA from mouse embryonic stem cells and brain. Genome Biology. 18 (1), 1 (2017).
- Endrullat, C., Glökler, J., Franke, P., Frohme, M. Standardization and quality management in next-generation sequencing. Applied & Translational Genomics. 10, 2-9 (2016).
- Schaefer, M., Pollex, T., Hanna, K., Lyko, F. RNA cytosine methylation analysis by bisulfite sequencing. Nucleic Acids Research. 37 (2), 12 (2009).
- Cristinelli, S., Angelino, P., Janowczyk, A., Delorenzi, M., Ciuffi, A. HIV Modifies the m6A and m5C Epitranscriptomic Landscape of the Host Cell. biorxiv. 1 (11), (2021).
- Squires, J. E., et al. Widespread occurrence of 5-methylcytosine in human coding and noncoding RNA. Nucleic Acids Research. 40 (11), 5023-5033 (2012).
- Aschenbrenner, J., et al. Engineering of a DNA Polymerase for Direct m(6) A Sequencing. Angewandte Chemie (International ed. in English). 57 (2), 417-421 (2018).
- Didion, J. P., Martin, M., Collins, F. S. Atropos: specific, sensitive, and speedy trimming of sequencing reads. PeerJ. 5, 3720 (2017).
- Kim, D., Langmead, B., Salzberg, S. L. HISAT: a fast spliced aligner with low memory requirements. Nature Methods. 12 (4), 357-360 (2015).
- Li, H., et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 25 (16), 2078-2079 (2009).
- Okonechnikov, K., Conesa, A., García-Alcalde, F. Qualimap 2: advanced multi-sample quality control for high-throughput sequencing data. Bioinformatics. 32 (2), 292-294 (2016).
- Ewels, P., Magnusson, M., Lundin, S., Käller, M. MultiQC: summarize analysis results for multiple tools and samples in a single report. Bioinformatics. 32 (19), 3047-3048 (2016).
- Zhang, Y., et al. Model-based Analysis of ChIP-Seq (MACS). Genome Biology. 9 (9), 137 (2008).
- Martin, M. Cutadapt removes adapter sequences from high-throughput sequencing reads. EMBnet Journal. 17 (1), (2011).
- Shen, W., Le, S., Li, Y., Hu, F. SeqKit: A Cross-Platform and Ultrafast Toolkit for FASTA/Q File Manipulation. PLOS ONE. 11 (10), 0163962 (2016).
- Rieder, D., Amort, T., Kugler, E., Lusser, A., Trajanoski, Z. meRanTK: methylated RNA analysis ToolKit. Bioinformatics. 32 (5), 782-785 (2015).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved