Method Article
遺伝子組み換えアノフェリン蚊集団におけるインデル変異を検出するデジタル液滴PCR
要約
このプロトコルは、gRNA誘導Cas9切断およびDNA修復後の標的部位における非相同性端結合(NHEJ)事象の同定および定量化に関する分析を含む、DNA抽出からデジタル液滴PCR(ddPCR)の実験的セットアップまでのステップを提供する。この方法の他の用途としては、多型検出や遺伝子編集バリアント検証などの用途が挙げられる。
要約
最近の蚊ゲノミクスや遺伝子工学技術の進歩により、標的DNA配列の変異を大規模に検出するための迅速かつ効率的な方法が求められております。具体的には、CRISPRガイドRNA(gRNA)/Cas9媒介非相同末端結合(NHEJ)によって生成された遺伝子編集部位における挿入および欠損(インデル)の検出は、変異生成の忠実度および意図しない変化の頻度を評価するために重要である。ここでは、高スループットのNHEJ分析に適したデジタル液滴PCR(ddPCR)のプロトコルについて説明します。この方法では、個々のシーケンスの変動を識別するデータは生成されませんが、母集団内のシーケンス変動の定量的な推定値が提供されます。さらに、適切なリソースを使用すると、このプロトコルは、次世代またはサンガーシーケンシングよりも簡単に現場のラボ設定で実装することができます。ddPCRはまた、これらの方法のどちらかよりも結果のためのより速い回り時間を有し、遺伝子組み換え生物のフィールド試験中に野生集団の遺伝的変異をより迅速かつ完全に分析することを可能にする。
概要
遺伝子ドライブは、医療と農業関連の昆虫集団を制御する巨大な可能性を有する1,2,3,4,5.例えば、CRISPR CasヌクレアーゼおよびガイドRNA(gRNA)に基づく遺伝子駆動システムは、感染の減少と減らされた疾患1、4、5につながるマラリア原虫に屈折を与える形質を導入することによって、ベクター蚊集団を改変するために使用することができる。遺伝子駆動システムは、それ自体と関連する形質を、1つの相同染色体から、大きな異形性胚芽細胞の別の染色体にコピーし、これは、子孫の大半がドライブを継承し、フィールドで長期的かつ持続可能な集団改変の可能性を作成することを保証します。しかし、Cas/gRNAベースの方法の欠点の1つは、非相同的なエンド結合(NHEJ)DNA修復を通じて挿入および欠失(indel)突然変異を発生させる可能性であり、その結果、駆動抵抗性対立遺伝子が生成され、集団中の十分な頻度に蓄積されると、駆動系が広がるのを止めることができる。.このプロトコルはCas/gRNAベースの遺伝子駆動中に、集団レベルと個人レベルの両方でインデル突然変異の有病率と相対的量を決定できる、ハイスループットと信頼性の高い方法を詳述しています。
次世代シーケンシング(NGS)法は、比類のないシーケンシングの解決を提供します。しかし、NGS に関連するコストと技術要件は、日常的なテストを禁止し、インデル6、7、8を評価するためのハイスループット方式としての使用を制限します。従来のPCR定量法は、ゲノムインデルの標準的な評価手順として長い間使用されてきました。しかし、これらの方法は、労力を要し、データの調達に時間がかかり、変動性が高い。デジタル・ドロップレットPCR(ddPCR)は、一部のアプリケーションではサンガーシーケンシングよりも変異の検出に敏感であることが証明されており、他の6、7、8、9ではNGSよりも低い検出限界を有する。さらに、結果を得るためのサンプルセットとターンアラウンド時間を評価するためのコストは、それぞれ、サンガーシーケンシングまたはNGS9のいずれかよりもddPCRのために、より安価かつ迅速である。デュアルプローブシステムを使用して、ドロップオフアッセイは、gRNA指向ターゲットCas9カット部位での野生型(WT)配列の欠如に基づいてNHEJ対立遺伝子を識別します。このアッセイでは、Cas/gRNAベース系系の予測切断部位を含む短いアンプリコンが、特異的プライマー対で増幅される。1つの蛍光プローブはアンプリコンの保存領域に結合するように設計されており、別の蛍光プローブは切断部位のWT配列を認識する。NHEJアレーレの存在下では、後者はアンプリコンに結合しない。
ddPCR の使用は、削除、単一ベースペアの違いおよび挿入をターゲットにプライマーを設計する機能を提供し、蚊の集団分析における NHEJ プロファイリングを可能にします9.これらの魅力的な特徴を踏まえて、蚊のCas/gRNAベースの遺伝子駆動システムから生成されたインデルのハイスループット検出のためのddPCRプロトコルを作成しました。
プロトコル
1. DNA抽出
- 500 μL の NLS と 1 サンプル当たり 120 μL の比率で、EDTA/核ライシスバッファー (EDTA/NLS) を準備します。複数のサンプルのスケールアップ。氷の上で混合物を冷やします。
注:溶液は、ボリュームに応じて冷やすと2〜5分で曇りになります。 - 冷却されたEDTA / NLSの600 μLで満たされた1.5 mLマイクロ遠心分離管で10-15 sのための機械ホモジナイザーを使用して蚊のサンプルを均質化;よく混ぜます。
- 20 mg/mL の 20 mg/mL の 17.5 μL をチューブに加え、十分に混ぜます。
- 55°Cで一晩インキュベートする。 あるいは、55°Cで3時間、サンプルを1時間ごとに振り、渦を入れインキュベートする。
- 200 μLのタンパク質沈殿液を室温サンプルに加え、渦を20秒に激しく加えます。
- サンプルを氷の上で5分間冷やします。
- サンプルをペレットタンパク質に遠心分離して、15,890 RCFで4分間用いた。
- DNAを含む上澄み物を慎重に吸引し、600 μLのイソプロパノールを含むクリーンな1.5 mLマイクロ遠心チューブに移します。
- チューブを5~10回反転して溶液を軽く混ぜます。15,890 RCFで1分間の遠心分離機。DNAペレットを保存しながら上清を慎重にデカントします。
- 600 μLの室温 70% エタノールを加えます。ペレット化したDNAを、チューブを軽く反転させて洗浄します。
- 15,890 RCFで1分間の遠心分離機。ガラスピペットチップを使用して吸引して上清を慎重に除去します。
- チューブをきれいな吸収性の紙に反転させ、ペレットを10〜15分間空気乾燥させます。
- DNAをPCRグレードの水で再懸濁します。1個の蚊のサンプルにつき20μL、10個のプールされた蚊に100 μLを使用してください。
注:市販のキットを使用した蚊のサンプルのDNA抽出方法( 材料表を参照)は、組織培養細胞および動物組織プロトコルからゲノムDNAを分離するメーカーから適応されています。
2. ddPCR反応と液滴生成製剤
- フルオメーターを使用してDNAを定量化します。
注:ドロップオフアッセイでは、ターゲットカット部位のWT配列に結合し、ターゲットサイトで欠失または挿入がある場合はアニール(ドロップオフ)しないHEXラベリングプローブでNHEJイベントを検出するように設計された、反応ごとに3,000〜30,000のハプロイドゲノムコピーの範囲を使用することをお勧めします。 NHEJ バリアントの存在を示します。 - 抽出物中のdnaのハプロイドゲノム重量と濃度を用いてコピー数を計算する。これは、抽出されたDNAの濃度と全DNA質量を得るために使用される体積を掛け、それをハプロイドゲノム重量で割ることによって行われる。推奨されるハプロイドゲノムコピー範囲内に入るために必要に応じて、追加されたボリュームが1〜10 μLの間にあることを確認します。
注:1つの アノフェレスガンビアハ プロイドゲノムは、成虫蚊10あたり0.27 pgであると推定される。 - プライマーとプローブを設計します。150-400 bpのアンプリコンを生成するgRNA標的部位の5'-および3'末端に隣接する55〜60°Cの範囲のプライマー融解温度(Tm)を有する前方および逆オリゴヌクレオチドプライマーを設計する。
- 16進数(ヘキサクロロ-フルオレセイン)-NHEJ検出用プローブ:標的部位に相補的な長さが15~20bpのオリゴヌクレオチドを設計し、HEXプローブを5'エンドに、BHQ1(BLackホールクエンチャー1)を3'末端に加えます。加水分解プローブのTmは、プライマーのTmよりも3〜10°C高くする必要があります。
- FAM(6-カルボキシフルオレセイン)標識プローブ(参照WT用):オリゴヌクレオチドを、標的部位から遠く離れた保存ゲノム部位(約25bp)に補長のオリゴヌクレオチドを設計し、FAMプローブを5'末端に、BHQ1を3'末端に追加します。加水分解プローブのTmは、プライマーのTmよりも3〜10°C高くする必要があります。
3. PCR反応の準備
- ddPCR サンプルミックスの 25 μL を次のコンポーネントで準備します: プローブ用 ddPCR スーパーミックス (UTP なし): 12.5 μL、 フォワードおよびリバースプライマー(10 μM):1 μL、HEX/FAMプローブ(10 μM):それぞれ0.625 μL、DNA:1-5 μL(3,750-37,500 haploidゲノムコピー)および水:最大25μL。
- 反応を、渦巻きまたは還流ピペット(上下)(20倍)で十分に混合します。
注:反応が96ウェルプレートにある場合は、気泡の形成を避けるために渦を巻くのではなく、体積全体を上下に20回ピペットします。 - サンプルを簡単に遠心分離して、混合物をチューブの底部またはウェルで沈着させる。
注:液滴生成のための反応が室温であることを確認してください。各カートリッジに余分な/未使用のウェルを用意するために、1xのddPCRミックスを準備します(各カートリッジには8つのウェルがあります)。
4. 液滴発生
- 50 μL のマルチチャンネルピペットを使用して、ddPCR サンプルの 20 μL をカートリッジの中央の行に混合します(図 1A、上)。
- オイルの70 μLを下の列に積み込みます。未使用のウェルに1x ddPCRスーパーミックスの20 μLをロードします。
注意:気泡を導入しないでください。 - ガスケットをエッジのみに触れ、中央の凹面領域を避けます(図1B)。
- プレートを液滴発生器にしっかりと置き、カバーを閉じて実行を開始します。
- マルチチャンネルピペットを使用して、カートリッジの上段(図1A、下)から96ウェルプレートにエマーシオンミックスの40 μLを移します。
- 45~30°の角度で3~5sの液体サンプルを描きます。混合物を45°の角度で3s以上ゆっくりとウェルの側面に出して、側面に滴り落ちるようにします。すべての液体を排出するためにピペットの第二の停止(完全な追放)に行くのは大丈夫です。
- ホイルヒートシールを使用し、180°Cで5sのプレートを密封します。
5. PCR
- 密封プレートをサーモサイクラー(図1C)に入れ、NHEJドロップオフガイドラインに従う場合は、推奨PCR条件を次のように設定します。
- 95°Cで10分間の初期変性。
- 変性する場合は94°Cの40サイクル、アニールに1分間55°C、延長する場合は60°Cを2分間設定します。
- 98°Cで10分間保持します。
- 4 °Cで保持します。
注: 特定のプライマーおよびプローブセットのアニーリング温度は、熱勾配を使用して最適化できます。すべてのステップに2°C/sのランプレートを使用してください。PCR条件は、各実験計画とセットアップに応じて調整する必要があります。
6. 液滴の読み取り
- プレートを左上にA-1(平滑化されたコーナー、他の3つは縁取り)で液滴リーダーにしっかりと置きます(図1D)。
- プログラムでプレートを設定する: 既知の参照チャネルとして FAM を指定し、HEX を未知のチャネルとして指定する (図 2A)。
- 直接定量として液滴読み取り実験を実行します。実行が完了したら、分析の [実験の種類] を[ドロップオフ(DOF)] に変更します (図 2A)。
7. 分析
- 正しい実験パラメータを指定します (図 2A):サンプル情報,SuperMix,ターゲット名(WTまたはNHEJ),ターゲットタイプ(参照または不明)),信号Ch1(HEXまたはFAM)、シグナルCh2(HEXまたはFAM)、および手動で液滴数のしきい値を設定します(信頼できる結果を得るには10,000以上を推奨)。ソフトウェアは、指定されたパラメータを使用してほとんどの解析を実行します。
- ドロップレットタブで ドロップレット 数を確認します。すべてが 10,000 を超えているようにしてください (図 2B)。
- 陰性からの効率的な信号分離のために1 D振幅を点検して下さい。
- プレート エディタでプレート全体をハイライト表示し、[ 実験タイプ ] を [ドロップ オフ]に設定します。
- WT ターゲットを 参照として設定し、FAM 用のチャネル 1 を指定し、HEX 用にチャネル 2 を指定します。
- NHEJ ターゲットを不明として設定し、FAM 用のチャネル 1 とチャネル 2 を none に指定します。
- [2D 振幅] タブで、各サンプルのグラフ ツールを使用して クラスターのしきい値 を設定します。WT クラスターに関連付けられているテールを検討してください。これは、NHEJアッセイ(図3A)では正常です。
注: [比率]タブで、グラフの右上にある歯車アイコンをクリックします。[分数の豊かさ]を選択します。グラフは、NHEJ イベントの割合に対応するポイントをプロットします (図 3B)。
結果
この手順の適用は、カルバラル・レハラスーら9に表示されます。ddPCRドロップオフアッセイは、WTとインデル配列を識別するために2つの蛍光プローブを利用します:FAMプローブはアンプリコン内の保存された配列に結合し、HEXプローブは標的部位のWT配列を標的とします(図4A)。インデルの存在下では、HEXプローブは結合しません。代表的な結果は、カルバラ・レハラスースらの図2、表1、表2等に記載されている。このプロトコルを使用して、ddPCRは、CRISPR-Cas9誘導NHEJ事象の多種多様を検出し、個々のまたはプールされたサンプルでNHEJ周波数を定量化することが実証されています。10個の蚊の15種類のプールサンプルにはそれぞれ様々なNHEJ対立体が含まれていました(カルバラ・レハラズスの表29)。これらは、ここで示したプロトコルとパラメータを使用してddPCRで分析した。表19の結果は、15個のサンプルすべてが、ドロップオフアッセイによって識別された100%インデル対立差を運んだことを示している(図4B)。別の実験では、異なるNHEJパーセンテージを有するWT蚊およびNHEJ蚊の11個のプールサンプル(0%、10%、20%、30%、40%、50%、60%、70%、80%、90%、および100%)をこのddPCRプロトコルで調べた(Carballar- Lejarazúら9)は、同定された割合がアンプリコン分析によるインデル検出の比較技術に近いことを示した(図4C)。

図1: 実験的なセットアップと手順 (A)液滴生成用カートリッジの準備。(トップ)サンプルはカートリッジの中央の列に充填され、オイルは一番下の列に充填されます。(下)液滴発生後の乳化液滴で満たされた一番上の行。(B)カートリッジをサンプルで満たし、ガスケットで覆われたドロップレット発生器。(C)96ウェルプレートは、サーモサイクラーのホイルシールで覆われています。(D) 96ウェルプレートを固定するために、金属カバーをプレートの上に掛けた状態で、ドロップレットリーダー。 この図の大きなバージョンを表示するには、ここをクリックしてください。
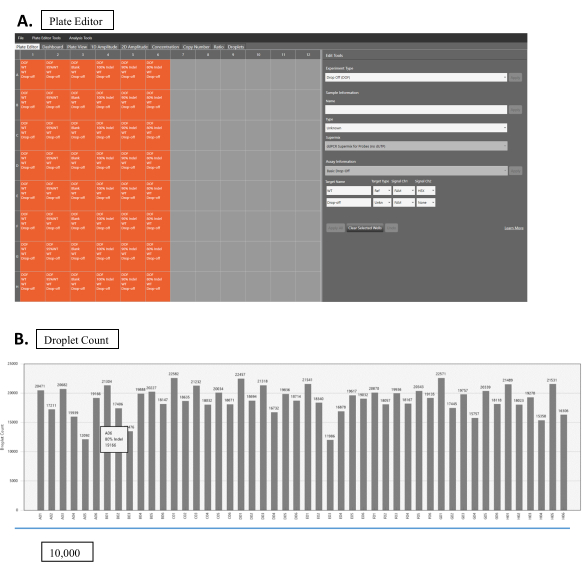
図2:液滴読み取り(A) 液滴読み取り用のソフトウェアインターフェイス。オレンジ色のボックスは、サンプルとウェルを示しています。灰色の箱は空の井戸です。実験用パラメータは、[編集ツール]パネル(右側)で設定します。各サンプルは、それぞれのサンプルボックスをクリックして編集できます。実験タイプで[ドロップオフ(DOF)] を選択します。サンプル情報に、サンプルの名前と種類、およびSuperMixの適切な情報を入力します。アッセイ情報の基本ドロップオフを選択します。WT サンプルでは、[ターゲット名]、ターゲットタイプの参照、[シグナル Ch1]および[Ch2]の [FAM] および [HEX] の両方をそれぞれ選択します。NHEJ サンプルの場合は、ターゲット名に適切な名前を入力し、ターゲットの種類に対して [不明] を選択し、[シグナル Ch1]の [FAM] を選択します。シグナル Ch2を [なし] のままにします。(B)複数サンプルのドロップレット数の結果。この図の大きなバージョンを表示するには、ここをクリックしてください。
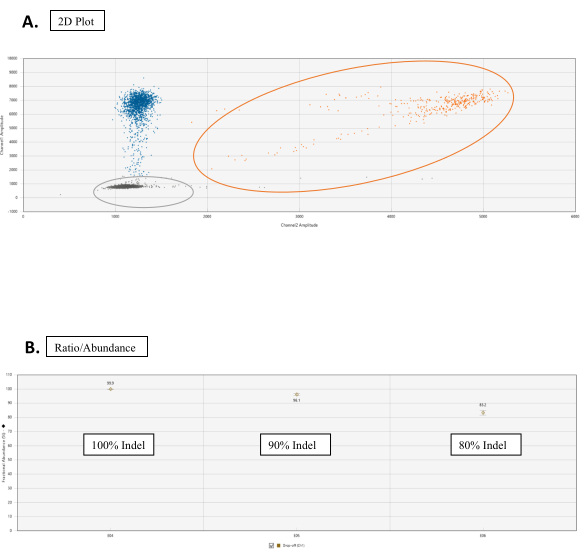
図3:ドロップオフアッセイ分析(A)WTおよびNHEJ対立の液滴数のクラスター2Dプロット。[2D 振幅]タブでは、デフォルトですべての液滴が分類解除されます。この図では、色を手動で割り当て、区別します。オレンジ色のドットクラスターは、それぞれ参照配列およびターゲット部位配列におけるFAMおよびHEXプローブの両方の結合によって得られるWTアリール数である。青い点は、参照シーケンスに FAM 結合を持つが、ターゲット部位シーケンスで HEX 結合を持たない(したがって、HEX のドロップオフ)、液滴のスコアを表します。灰色のドットは、FAM または HEX バインディングを持たない空の液滴です。(B) NHEJイベントの比率/存在量グラフ。[比率] タブで、NHEJ イベントの対応する割合を持つグラフの[分数の豊富さ] を選択します。この図の大きなバージョンを表示するには、ここをクリックしてください。
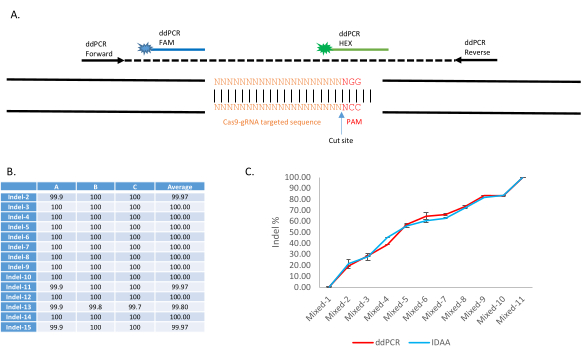
図4:トランスジェニック・アノフェレス・ステファニシ線AsMCRkh1における非相同のエンド結合同定および定量化に対するddPCRによるドロップオフアッセイの適用(A) デュアルプローブシステムを用いた標的DNA部位の変異を検出するためのddPCRドロップオフアッセイの概略的な提示。150-400 bpのアンプリコンは、フォワードおよびリバースプライマーで増幅される。FAM標識プローブは、アンプリコンの保存された配列に結合するように設計されていますが、HEX標識プローブはWT gRNA標的部位に結合するように設計されています。(B) ddPCR を用いたさまざまなタイプのインデルの検出。挿入、欠失、置換を含む様々なタイプのインデルを含む10個のAsMCRkh1蚊の15個のプールを、ddPCRドロップオフアッセイで分析した。突然変異および配列の詳細は、カルバラールらの表2および表S3に記載されている。9.(C) 様々な比率の AsMCRkh1 と WT 蚊の混合サンプルにおける NHEJ の定量化 (10:0, 9:1, 8:2, 7:3, 6:4, 5:5, 4:6, 3:7, 2:8, 1:9, 0:10) ddPCR を用いたインデル検出法と比較した方法カルバラール・レハラスーらバイオテクニックから適応した画像.68(4):172-179 (2020)9.この図の大きなバージョンを表示するには、ここをクリックしてください。
| プライマー/プローブ | シーケンス (5' » 3') |
| ddPCR フォワードプライマー | アガットカートグCGACCG |
| ddPCR リバースプライマー | アクトクトットゥグトガアカ |
| ddPCR HEX プローブ (BHQ1) | [ヘックス]-TTCTACGGGCGC-[BHQ1] |
| DDPCR FAM プローブ (BHQ1) | [6FAM]-CCACGTGGGATCGAAGG-[BHQ1] |
| HEX:ヘキサクロロフルオレセイン、FAM:6カルボキシフルオレセイン、BHQ:ブラックホールクエンチャー | |
表1:プライマーとプローブの配列。
ディスカッション
デジタル液滴PCRは、Cas/gRNAベースの遺伝子駆動システムにおけるNHEJ事象に起因するインデル対立遺伝子の存在を決定するための効率的な方法であり、個体または集団におけるこれらの対立遺伝子の頻度を定量化することを可能にする。プロトコルのいくつかのステップは、信頼性の高い結果を達成するために特別な注意を払って従う必要があります。まず、ゲノムDNA抽出は、高品質で十分な量を確保するために慎重に行う必要があります。良好な抽出により、反応ごとにハプロイドゲノムコピーを正確に決定することができます。我々の経験では、市販のキット( 材料表を参照)は一貫して高品質のDNA抽出を提供しました。しかし、個々の蚊の抽出は、DNAペレットが視覚化しにくくなり、注意しないと上澄みで簡単に吸い上げられるため、特に困難であることが証明されます。第二に、プライマーとプローブは慎重に設計する必要があります。ddPCR 実験を完了する前に、設計されたプライマーが、最初に従来の PCR を実行し、ゲル電気泳動を介して単一の製品を視覚化することによって、単一の PCR 製品が得られるようにしてください。また、FAMプローブのリファレンスは、高度に保存されたシーケンスを補完するように設計する必要があります。これにより、多様な集団全体でWT対立体を正確に検出することができます。各ユニークな実験のプライマー/プローブの組み合わせは、異なるサーモサイクラー条件を持つため、熱勾配を使用してそれらの条件を最適化することをお勧めします。
サンガーシーケンシングやNGSなど、インデルを識別するための他の方法が存在します。サンガーシーケンシングは、検出の下限と新しい変種を識別するための低い発見力を有するため制限されています。サンガーシーケンシングも労働集約的で、ハイスループットではありません。サンガーシーケンシングと比較すると、NGS には低感度、検出パワー、スループットの制限が同じではありません。NGSのもう一つの利点は、一塩基多型(SNPs)から再配列に様々な変異を検出する能力です。しかし、NGSは、目的のターゲット領域が1つしかなく、より大きなゲノム全体の分析に最も適しているため、Cas9/gRNA関連インデルを決定するアプリケーションにおいて、よりコストと時間のかかる方法です。前述の方法と比較して、ddPCRは高スループットであり、迅速な回転時間を有する。ddPCRの材料および器具が社内で入手可能な場合、96サンプルは1〜2日以内に処理することができ、Cas9/gRNA改変生物の大規模な試験の迅速な分析に適しています。
ddPCR には多くの利点がありますが、制限もあります。第1に、ddPCR装置は独立した実験室環境で頻繁に利用できるわけではない。ddPCR装置は、大規模な研究機関で共同で利用できるかもしれませんが、これは機関外でのデータ生成と分析の容易さを可能にしません。第二に、代替とは異なり、ddPCRは、同定されたインデル突然変異の個々の固有配列を提供しない。デジタル液滴PCRは、集団内のインデル突然変異の頻度を提供するが、配列がなければ、存在するインデルが対象遺伝子の機能を保存または阻害する可能性が高いかを判断できない。ddPCR法は、ネイティブ集団へのトランスジーン導入の頻度と集団内のインデルの生成をリアルタイムに近い状態で効率的に決定できるため、Cas9/gRNAベースのドライブ生物のフィールドリリース試験後に野生集団を分析するのに最も適している可能性があります。ddPCR の迅速な回転時間のため、材料がローカルで入手可能な場合、毎週フィールドトライアル領域でサンプリングを実行し、母集団を分析することが可能です。ddPCR機器の購入、インポート、セットアップのスタートアップコストは、リモートラボでは高くなりますが、ドライブシステムから変更を受けているため、野生の集団を厳密に評価できる利点はコストを正当化します。
開示事項
著者は開示を持っていません。
謝辞
資金は、カリフォルニア大学アーバインマラリアイニシアチブによって提供されました.AAJはカリフォルニア大学アーバイン校のドナルド・ブレン教授です。
資料
| Name | Company | Catalog Number | Comments |
| Reagents | |||
| ddPCR Super Mix for Probes (no dUTP) | Bio-Rad | 1863024 | |
| DNA extraction reagent (e.g. Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| EDTA (pH 8.0) | Invitrogen | AM9260G | |
| Ethanol, 200 Proof | Thermo Fisher Scientific | A4094 | |
| Isopropanol (Certified ACS) | Thermo Fisher Scientific | A416-500 | |
| Nuclei Lysis Solution (NLS) (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| PCR-grade Water | Any certified PCR-grade water can be used | ||
| Protein Precipitation Solution (Wizard Genomic DNA Purification kit) | Promega | A1120 | |
| Proteinase K 20 mg/mL | Thermo Fisher Scientific | AM2546 | |
| Materials | |||
| ddPCR 96-Well Plate | Bio-Rad | 12001925 | |
| Droplet Generator DG8 Cartridge and Gaskets | Bio-Rad | 1864007 | |
| Droplet Generation Oil for probes | Bio-Rad | 1863005 | |
| Fluorescent probes (e.g. FAM/HEX probes) | Sigma-Aldrich | N/A | Probes are experiment specific and can be purchased from any certified seller available. |
| Forward and Reverse oligonucleotide primers | Sigma-Aldrich | N/A | Primers are experiment specific and can be purchased from any certified seller available. |
| Equipment | |||
| C1000 Touch Thermal Cycler | Bio-Rad | 1851148 | Can use other Thermo cycler with gradient function and deep well |
参考文献
- Gantz, V. M., et al. Highly efficient Cas9-mediated gene drive for population modification of the malaria vector mosquito Anopheles stephensi. Proceedings of the National Academy of Sciences of the United States of America. 112 (49), 6736-6743 (2015).
- Hammond, A., et al. A CRISPR-Cas9 gene drive system targeting female reproduction in the malaria mosquito vector Anopheles gambiae. Nature Biotechnology. 34, 78-83 (2016).
- Courtier-Orgogozo, V., Morizot, B., Boëte, C. Agricultural pest control with CRISPR-based gene drive: time for public debate: Should we use gene drive for pest control. EMBO Reports. 18 (6), 878-880 (2017).
- Carballar-Lejarazú, R., et al. Next-generation gene drive for population modification of the malaria vector mosquito, Anopheles gambiae. Proceedings of the National Academy of Sciences of the United States of America. 117 (37), 22805-22814 (2020).
- Adolfi, A., et al. Efficient population modification gene-drive rescue system in the malaria mosquito Anopheles stephensi. Nature Communications. 11, 5553 (2020).
- Wang, Z., et al. Comparison of droplet digital PCR and direct Sanger sequencing for the detection of the BRAFV600E mutation in papillary thyroid carcinoma. Journal of Clinical Laboratory Analysis. 33 (6), 22902 (2019).
- Dong, L., Wang, S., Fu, B., Wang, J. Evaluation of droplet digital PCR and next generation sequencing for characterizing DNA reference material for KRAS mutation detection. Scientific Reports. 8 (1), 9650 (2018).
- Bell, C. C., Magor, G. W., Gillinder, K. R., Perkins, A. C. A high-throughput screening strategy for detecting CRISPR_Cas9 induced mutations using next-generation sequencing. BMC Genomics. 15 (1), 1002 (2014).
- Carballar-Lejarazú, R., Kelsey, A., Pham, T. B., Bennett, E. P., James, A. A. Digital droplet PCR and IDAA for the detection of CRISPR indel edits in the malaria species Anopheles stephensi. Biotechniques. 68 (4), 172-179 (2020).
- Holt, R. A., et al. The genome sequence of the malaria mosquito Anopheles gambiae. Science. 298 (5591), 129-149 (2002).
転載および許可
このJoVE論文のテキスト又は図を再利用するための許可を申請します
許可を申請さらに記事を探す
This article has been published
Video Coming Soon
Copyright © 2023 MyJoVE Corporation. All rights reserved